Method Article
Einzigen Molekül Fluoreszenz In Situ Hybridisierung (SmFISH) Analyse in Knospen Hefe vegetativen Wachstums und Meiose
In diesem Artikel
Zusammenfassung
Dieses Molekül Fluoreszenz in Situ Hybridisierung Protokoll ist optimiert, um die Anzahl der RNA-Moleküle in der angehenden Hefe während des vegetativen Wachstums und Meiose zu quantifizieren.
Zusammenfassung
Einzelnes Molekül Fluoreszenz in Situ Hybridisierung (SmFISH) ist eine leistungsstarke Technik, Genexpression in Einzelzellen aufgrund seiner Fähigkeit, zu erkennen und zählen die einzelnen RNA-Moleküle zu studieren. Ergänzend zu deep Sequencing-basierte Methoden, SmFISH liefert Informationen über die Zell-Zell-Variation im Transkript Überfluss und die subzelluläre Lokalisation einer bestimmten RNA. Vor kurzem haben wir SmFISH verwendet, um die Expression des Gens NDC80 während der Meiose in der angehenden Hefe zu studieren, in welche zwei Niederschrift Isoformen vorhanden und kurze Niederschrift Isoform hat seine gesamte Sequenz mit der langen Isoform geteilt. Um jede Abschrift Isoform selbstbewusst zu identifizieren, wir optimierte bekannt SmFISH Protokolle und erzielt hohe Konsistenz und Qualität der SmFISH Daten für die Proben während angehende erworben Hefe Meiose. Hier beschreiben wir diese optimierte Protokoll, die Kriterien, die wir verwenden, um festzustellen, ob hoher Datenqualität SmFISH gewonnen wird, und ein paar Tipps für die Durchführung dieses Protokolls in anderen Hefestämme und Wachstumsbedingungen.
Einleitung
Dynamische Regulation der Genexpression treibt die Entwicklung eines Organismus, sowie seine Reaktion auf Umwelteinflüsse, Infektionen und Veränderungen im Stoffwechsel. Studien, die sich auf transcriptional Regelung setzen auf Technologien, die RNA Fülle zu messen. Eine solche Methode, als einzelnes Molekül Fluoreszenz in Situ Hybridisierung (SmFISH) dient zur Erkennung von einzelnen RNA-Moleküle in einzelne Zellen1,2. Diese Methode ermöglicht die Messung der Zell-Zell-Variabilität in der Genexpression und Bestimmung der intrazellulären RNA-Lokalisierung.
In der am häufigsten verwendeten SmFISH-Technik erfordert Erkennung von einem einzelnen RNA-Molekül mehrere kurze DNA-Sonden (oft ~ 48 20-Mer Sonden), die komplementär zu der Ziel-RNA und sind auf der gleichen Fluoreszenzfarbstoff konjugiert. Bindung von einzelnen fluoreszierende Sonden führt zu schwaches Signal, aber das Signal aus dem Ensemble der Sonden ist robust. Diese Funktion verbessert das Signal-Rausch-Verhältnis, denn obwohl eine einzelne Sonde Ziel Bindung aufweisen kann, solches Signal erwartet wird sehr schwach im Vergleich zu der der Ziel-RNA-Molekül3. Innerhalb Fehler erkennen kann die Anzahl der RNA-Moleküle gezählt und verglichen, in ganz verschiedenen Wachstumsbedingungen und unter verschiedenen Mutanten.
Seit seiner ersten Entwicklung ist SmFISH angepasst worden, um verschiedene Aspekte der Gene Expression4, z. B. Transkription Dehnung1,2,5,6, transkriptionelle platzen7 Spleißen studieren , 8 , 9, intrazelluläre allelic Ausdruck10,11,12und RNA Lokalisierung13,14,15. Vor kurzem haben wir diese Methode verwendet, um die Expression von zwei Transkript Isoformen des gleichen Gens, in dem studieren der kurzen Transkript Isoform (NDC80ORF) hat seine gesamte Sequenz gemeinsam mit der langen Isoform (NDC80Luti )16. Zur eindeutigen Identifizierung der beiden Isoformen von mRNA, wir verwendeten Sonde zweierlei: ein Satz bezieht sich auf die einzigartige Abfolge der NDC80Lutiund die andere Gruppe, zu einem anderen Fluoreszenzfarbstoff konjugiert bindet an die gemeinsame Region der beiden Isoformen. NDC80Luti RNA wird als ein colocalized Ort mit beide fluoreszierende Signale identifiziert, während die NDC80ORF RNA ist derjenige, der nur das Signal aus dem gemeinsamen Sonde Set enthält. Da die Anzahl der der NDC80ORF Abschriften wird berechnet, indem eine "Subtraktion" Methode, hohe Effizienz der Sonde Hybridisierung und ein hohes Signal-Rausch-Verhältnis sind notwendig, um selbstbewusst SmFISH Flecken erkennen und reduzieren Fehler Ausbreitung.
Dieses Whitepaper beschreibt ein optimiertes Protokoll für die Durchführung von SmFISH in der angehenden Hefe Saccharomyces Cerevisiae. In dieses Protokoll, die Zellenzahl, Fixierzeit, Dauer der Verdauung, Verdauung Puffer, Sonde Konzentration und Hybridisierung Puffer für die SmFISH Experimente verwendet wurden optimiert für den SK1 Stamm Hintergrund S. Cerevisiae unterziehen vegetative Wachstum oder Meiose. Allerdings haben wir auch in diesem Manuskript beachten: (1) die Methode zum Überprüfen der Qualität der SmFISH Daten nach der Bildaufnahme und (2) die Schritte in das Protokoll, die zusätzliche Optimierung für verschiedene Belastungen Hintergründe und Wachstumsbedingungen erfordern.
Protokoll
Hinweis: Alle Puffer und in diesem Protokoll verwendeten Medien sind in Tabelle 1aufgeführt. Die Herstellerinformationen für Reagenzien wird in der Tabelle der Materialienaufgeführt.
Tag 1/Tag 1-2:
Hinweis: Für die vegetative Kultur wachsen Sie Zellen zu einem OD-600 von 0,4 bis 0,6 in ein bevorzugtes Medium. Induzieren Sie für die meiotische Kultur Zellen Meiose eine bevorzugte Methode (in der Regel in einem OD-600 von 1,85 Kultivierung) zu unterziehen.
1. Probe Fixierung und Verdauung
- Insgesamt ~3.5 OD600 Zellen in 3 % Formaldehyd zu beheben.
- Fügen Sie für vegetative Kultur 5,52 mL Kultur zu 480 µL 37 % Formaldehyd in 15 mL konische Röhrchen hinzu.
- Meiotische Kultur fügen Sie 1840 µL der Kultur zu 160 µL 37 % Formaldehyd in 2-mL Mikrozentrifugenröhrchen hinzu. Invertieren ~ 5 Mal zu mischen.
Achtung: Formaldehyd ist giftig. Handhabung und Entsorgung nach institutionellen Regelungen.
- Eine Bandage bei Raumtemperatur für 20 min Rohre aufsetzen. Für meiotische Proben nach der Fixierung bei Raumtemperatur für 20 min Befestigung über Nacht bei 4 ° c weiter, drehen.
Hinweis: Die Übernachtung Fixierung erhöht die Reproduzierbarkeit und die Qualität der SmFISH Daten für meiotische Proben. Fixierzeit sollte optimiert werden. - Während der Proben beheben, tauen Sie 200 mM Vanadyl Ribonucleoside komplexe (VRC) bei 65 ° c für mindestens 10 min auf.
- Verdauung-master-Mix in einem 15-mL-Tube vorzubereiten: 425 µL Puffer B mix für 1 Probe mit 40 µL 200 mM VRC (erwärmt auf 65 ° c); 2125 µL Puffer B mix für 5 Proben mit 200 µL 200 mM VRC (erwärmt auf 65 ° c). Vortex ~ 5 s voll Aufschwemmen der VRC vor dem Hinzufügen zu den master-Mix, der leicht bräunlich grün nach VRC Zugabe erscheint.
Hinweis: Zugabe von VRC während der Verdauung verbessert die Konsistenz der SmFISH Ergebnisse, möglicherweise durch Hemmung der Nuklease Verunreinigung durch die Zymolyase-Mischung, die aus Rohöl Extrakt gereinigt wird eingeführt. Die Höhe der VRC erforderlich bei diesem Schritt sollte optimiert werden. - Zentrifugieren Sie für vegetative Proben Schläuche am ~ 1057 X g 3 Minuten lang. Für meiotische Proben (nach der Übernachtung Fixierung) Zentrifugieren bei 21.000 x g für 1,5 min. Dekantieren oder Aspirieren des Überstands zu Formaldehyd Verschwendung.
Hinweis: Die Zentrifugation Schritte sind bei Raumtemperatur durchgeführt. - Aufschwemmen Sie Zellen in 1,5 mL kaltem Puffer B durch pipettieren rauf und runter oder durch Umkehrung der Rohre und streichen um zu mischen. Übertragen Sie vegetative Proben auf 2-mL-Tuben nach Wiederfreisetzung.
- Zentrifuge bei 21.000 x g für 1,5 min. entfernen Sie den Großteil der Flüssigkeit durch Vakuum Aspiration oder pipettieren, ~ 100 µL hinterlässt.
- Aufschwemmen der Zellen in 1,5 mL kaltem Puffer B.
- Zentrifuge bei 21.000 x g für 1,5 min. entfernen Sie den Großteil der Flüssigkeit durch Vakuum Aspiration oder pipettieren, ~ 100 µL hinterlässt.
- Aufschwemmen Sie Zellen in 1,5 mL kaltem Puffer B. Zentrifuge bei 21.000 x g für 1,5 min. Absaugen der Flüssigkeit vollständig durch Vakuum oder pipettieren.
- Aufzuwirbeln Sie Zellen in 425 µL Verdauung master-Mix und kurz Vortex zu Aufschwemmen. Jedes Rohr 5 µL 100 t 10 mg/mL Zymolyase hinzufügen.
Hinweis: Vortex Zymolyase jedes Mal, wenn die Rohre vor hinzufügen. Fügen Sie Zymolyase für jedes Rohr einzeln hinzu, anstatt zu den master-Mix. Beide Schritte helfen Verdauung Konsistenz unter den Röhren zu gewährleisten, weil Zymolyase schnell ausfällt. - Vortex 2-3 s zu mischen. Legen Sie die Röhren auf eine Bandage, und bei 30 ° c für 15-30 min zu verdauen. Für vegetativen Zellen und meiotischen Anfangsphase in der Regel dauert es ca. 15 min und meiotischen Divisionen und meiotischen Prophase dauert in der Regel ~ 30 min.
Hinweis: Überprüfen Sie auf dem Mikroskop alle 5 min nach 15 min und stoppen Sie die Verdauung zu, wenn ~ 80 % der Zellen nicht transparent und nicht-Refraktive erscheinen. Von diesem Zeitpunkt an Zellen sind sehr zerbrechlich. Handhaben Sie Zellen vorsichtig und vermeiden Sie die Verwendung von Vakuum Aspiration oder Vortexen. - Zentrifugieren Sie die Rohre bei ~ 376 X g für 3 min. entfernen die Flüssigkeit vollständig durch pipettieren.
- Sanft Aufschwemmen Zellen mit 1 mL Puffer B durch pipettieren oben und unten 1 - 2 Mal zu mischen.
- Zentrifugieren Sie die Rohre bei ~ 376 X g für 3 min. entfernen Puffer B Flüssigkeit durch pipettieren. Aufschwemmen Sie sanft Zellen in 1 mL 70 % igem Ethanol (mit RNase-freies Wasser verdünnt).
- 3,5-4 Stunden bei Raumtemperatur inkubieren.
2. Hybridisierung
- Formamid auf Raumtemperatur bringen (für 50-mL-aliquoten, dauert es ca. 30 min im Wasserbad).
Hinweis: Öffnen Sie die Formamid-Flasche nicht, bis die Flasche Raumtemperatur zur Vermeidung von Oxidation von Formamid erreicht.
Achtung: Formamid ist giftig. Handhabung und Entsorgung nach institutionellen Regelungen. - 10 % Formamid Waschpuffer in einem 15 mL konische Röhrchen vorzubereiten.
- Zentrifugieren Sie die Rohre bei ~ 376 X g für 3 min. entfernen 500 µL 70 % Ethanol durch pipettieren. Sanft nach oben und unten zu Aufschwemmen der verbleibenden Zellen, und übertragen Sie dann die Zellen auf niedrigem Kraftschlußbeiwert Röhrchen pipettieren.
Hinweis: Mit niedrigem Kraftschlußbeiwert Rohre stark Zellverlust reduziert, bei nachfolgenden Wäschen. - Zentrifugieren Sie die Rohre wieder bei ~ 376 X g für 3 min. entfernen das Ethanol durch pipettieren.
- Hinzufügen 1 mL 10 % Formamid Waschpuffer und sanft pipettieren rauf und runter 2-3mal Aufschwemmen der Zellen.
- Lassen Sie die Zellen, die bei Raumtemperatur für ~ 20 min sitzen, während der Vorbereitung der Hybridisierung-Lösung. Vermischen Sie für 1 Probe 50 µL der Hybridisierung Puffer (Raum Temp), 5 µL 200 mM VRC (erwärmt auf 65 ° c) und 1 µL jede Sonde (200 nM Final). Vermischen Sie für 5 Proben 250 µL Hybridisierung Puffer (Raum Temp), 25 µL 200 mM VRC (erwärmt auf 65 ° c) und 5 µL jede Sonde (200 nM Final).
- Tauen Sie Hybridisierung Puffer (bei-20 ° c eingefroren) auf Raumtemperatur vor dem Öffnen der Tube um Formamid Oxidation zu verhindern.
- Nach Zugabe von 200 mM VRC, Wirbel für 5-10 s vor dem Hinzufügen der Sonden sind die entwickelt und kommerziell erworben und gemäß den Anweisungen des Herstellers rekonstituiert.
- Wenn zwei Sonden Co inkubierten sind, fügen Sie 1 µL der 01:10 Verdünnung der Sonde #1 bestand, sowie 1 µL der 01:10 Verdünnung der Sonde #2 Lager, zu einer Endverdünnung von ~ 1: 500 für jede Sonde. Die Konzentration für die beste Signal-Rausch-Verhältnis muss optimiert (siehe Diskussion für Details).
- Zentrifugieren Sie Proben bei ~ 376 X g für 3 min. Entfernen des Überstands in Sondermüll durch pipettieren.
- Jedes Rohr mindestens 50 µL der Hybridisierung Lösung hinzufügen. Streichen Sie die Rohre zu mischen.
- Mindestens 16 Stunden im Dunkeln bei 30 ° c auf eine Bandage inkubieren.
2/3 Tag
3. Waschen und imaging
- Bringen Sie Formamid auf Raumtemperatur.
- 10 % Formamid Waschpuffer (FWB) in einem 15 mL konische Röhrchen vorzubereiten.
- Entfernen Sie die Röhren aus der Bandage und legen Sie sie in ein Feld Folie abgedeckt, um vor Licht zu schützen.
- Zentrifugieren Sie Proben bei ~ 376 X g für 3 min. Entfernen des Überstands in Sondermüll durch pipettieren.
- In 1 mL 10 % FWB durch sanft auf und ab pipettieren Aufschwemmen 2-3 Mal.
- Inkubation bei 30 ° c für 30 min (nicht rotierend) im Feld Folie abgedeckt.
- Zentrifugieren Sie die Proben bei ~ 376 X g für 3 min. Überstands um gefährliche Abfälle durch pipettieren entfernen und verlassen Sie ~ 50 µL zu.
- Unterdessen bereiten DAPI/FWB in einem 15 mL konische Röhrchen: 1000 µL 10 % Formamid Waschpuffer mit 1 µL von 5 mg/mL DAPI für 1 Probe vermischen. Mischen Sie für 10 Proben 10 mL 10 % Formamid Waschpuffer mit 10 µL von 5 mg/mL DAPI. In 1 mL DAPI/FWB durch sanft auf und ab pipettieren Aufschwemmen 2-3 Mal.
- Inkubation bei 30 ° c für 30 min (nicht rotierend) im Feld Folie abgedeckt.
- Anti-Bleichmittel Reagenzien auf Eis Auftauen.
- Zentrifugieren Sie Proben bei ~ 376 X g für 3 min. Entfernen des Überstands vollständig durch pipettieren. Bei Bedarf wieder entfernen Sie alle des Überstands Zentrifugieren.
- Für Proben, die nicht sofort abgebildet sind, erneut das Pellet in 50 µL GLOX-Puffer ohne Enzyme. Pipettieren rauf und runter 3-bis 4-Mal zu mischen.
- Halten Sie alle unimaged Proben im Feld Folie bedeckt bei 4 ° c bis bereit zu Bild. Wenn Sie bereit zum Bild, die Proben bei ~ 376 X g für 2 min zentrifugieren und überstand vollständig durch pipettieren entfernen. Aufschwemmen Sie in GLOX mit Enzymen wie unten.
- Fügen Sie für die Proben sofort abgebildet wird 15-20 µL GLOX-Puffer mit Enzymen. Pipettieren Sie sanft auf und ab zu mischen.
Hinweis: Das Volume hinzugefügt kann variieren abhängig von der Größe des Pellet-Zelle. Wir empfehlen resuspending das Pellet in 15 µL GLOX-Puffer mit Enzymen und prüfen die Zelldichte am Mikroskop. Wenn Zellen sind zu dicht (Zellen verklumpen übereinander), fügen Sie zusätzliche GLOX-Puffer mit Enzymen. Wenn Zellen zu spärlich sind, Zentrifugieren der Probenmaterials und ~ 5 µL Puffer zu entfernen. - Pipettieren 5 µL auf ein Deckglas (18 mm x 18 mm, Nr. 1), und setzen Sie das Deckglas auf einer Folie.
- Setzen Sie eine Labor wischen auf der Folie platziert das Deckglas. Drücken Sie vorsichtig auf das Labor-Tuch, Folie festlegen (siehe flüssige Abspringen von allen vier Rändern der das Deckglas sollte).
- Übertragen der Folie auf das Mikroskop-Zimmer in einer Box von Alu-Folie abgedeckt.
- Bild mit einem Weitfeld-Fluoreszenzmikroskop mit starker Vergrößerung (60-100 X) und hoher numerischer Apertur.
Hinweis: Um die maximale Anzahl der Photonen, die durch die SmFISH Sonden zu sammeln, sind konfokale Mikroskope nicht empfohlen, da sie die Menge des Lichtes zu erhebenden erheblich einschränken. Hier die Daten auf einem Mikroskop, ausgestattet mit einem 100 X 1,4 NA Ziel, Verwendung von Filtern CY5 (EX632/22, EM679/34) abgebildet bei 1,3 s, 100 % T; TRITC (EX542/27, EM597/45), 1.3 s, 100 % T; und DAPI (EX390/18, EM435/48), 0,05-0,1 s, 32-50 % T. Hellfeld Referenzbild sollte auch erworben werden. Erwerben von 15-25 Scheiben mit einer Schrittweite von 0,1-0,2 µm, von ganz unten im Mittelpunkt das Sichtfeld zu ganz oben, um sicherzustellen, dass alle die RNA-Spots entfallen.
(4) Bildanalyse
- Analysieren Sie die SmFISH-Daten mit veröffentlichten SmFISH Analyse-Tools wie Fisch-Quant17 und StarSearch2oder kundenspezifischen Programmen, je nach der genauen Anforderungen des Experiments.
Hinweis: Eine gute Analyse-Pipeline soll erlauben den Benutzern, eine Schwelle, die wahre RNA-Spots vom Hintergrund zu trennen, und Ausgang Statistiken wie die X, Y und Z Koordinaten (optionalen) und die Intensität von jedem Fleck, die Anzahl der Plätze in jeder Zelle zu bestimmen , und eventuell Anpassung als ein Weg, um Fehlalarme herausfiltern.
Ergebnisse
Zu bewerten, wie gut wir das SmFISH-Protokoll gearbeitet (skizziert in Abbildung 1), eine Reihe von 54 Sonden entwickelt, die den offenen Leseraster des Gens NDC80 Fliesen (Abb. 2A, oben). Die Sonde befindet sich an der 5'-Ende bezeichnet man als Sonde 1; dem andern, Sonde 2; und der dritte, Probe 3, etc.. Die 27 Sonden zugewiesen eine ungerade Anzahl (Probe 1, 3, 5,...) sind alle konjugiert, der Fluoreszenzfarbstoff CAL Fluor 590 (CF590); und die 27 Sonden zugewiesen eine gerade Zahl (Sonde 2, 4, 6...), um den Quasar 670 (Q670) Fluoreszenzfarbstoff konjugiert. Daher sind diese wechselnden Sonde Sätze oft als "Odd/even" Sonden bezeichnet. Bei der Hybridisierung sollten beide Sonde das gleiche Protokoll kennzeichnen.
Nach Spoterkennung haben wir ein paar Messungen zur Beurteilung der Qualität des SmFISH-Datensatz verwendet. Der erste war der Grad und die Qualität der ns1 für die ungerade/gerade Sonden. In unserem Fall colocalized 88 % aller SmFISH-Spots (Abbildung 2A und 2 b), mit mehr als 95 % der Flecken gepaart innerhalb von 2 Pixeln voneinander entfernt (Abbildung 2, gepaart), die innerhalb der Erwartungswert gegeben jeden chromatischen und erkennen Abweichung zwischen den beiden Leuchtstofflampen Kanälen. Im Vergleich dazu weniger als 10 % der unpaaren Flecken hatte einen nächster Nachbar Abstand von weniger als 2 Pixel, zeigen, dass die Wahrscheinlichkeit von falsch vor Ort zwei gering ist (Abbildung 2). 12 % der unpaaren Spots wurden gleichmäßig zwischen den beiden Kanälen unterteilt (Abb. 2 b, vgl. CF590-nur mit Q670 nur); und so schlossen wir, dass für dieses 2,4 kb-gen mit einer Reihe von Ausdruck zwischen 0 und 45 Transkripte pro Zelle ~ 94 % der RNA-Moleküle in jedem fluoreszierende Kanal genau erfasst wurden. Wenn das SmFISH-Protokoll suboptimal wäre, würde man (1) eine größere Bruchteil der SmFISH Spots mit nur einem der beiden fluoreszierende Signale (colocalized) und/oder (2) Zellen mit einem sehr niedrigen Signal-Rausch-Verhältnis oder kein Signal überhaupt beobachten.
Wir als nächstes gefragt, ob die Erkennung des SmFISH-Signals in Bezug auf die Gesamtzahl der RNA-Moleküle pro Zelle voreingenommen war. In einer Bevölkerung liegt die Gesamtzahl der einer bestimmten RNA in jeder Zelle in einer Verteilung mit einigen Zellen beherbergen weitere RNA-Moleküle als andere. Eine gute SmFISH-Protokoll sollte die RNA robust unabhängig davon erkennen, ob eine hohe Zahl oder geringe Anzahl von RNA-Moleküle in jeder Zelle vorhanden ist. Um dies, für jede Zelle zu testen, haben wir den Bruch der SmFISH Flecken mit colocalized Signalen und der Bruch mit nur einem der beiden fluoreszierende Signale berechnet. Nach Gruppierung der Zellen, die die gleiche Anzahl von insgesamt Flecken pro Zelle hatte, wir berechnen den durchschnittlichen Anteil der colocalized (gepaart) oder nicht colocalized (nur für CF590 oder Q670-only) Flecken in einer bestimmten Gruppe, und dieser Durchschnitt in Abhängigkeit von der Gesamtzahl der im Diagramm Flecken pro Zelle (Abbildung 3). Für jede Kategorie von Spots ähnelten sich die Fraktionen über den gesamten Bereich der Gesamtzahl der Flecken pro Zelle. Vergleicht man die Zellen mit insgesamt 20 fluoreszierenden Flecken pro Zelle im Vergleich zu denen mit insgesamt 30 Punkte, waren durchschnittlich Bruchteile der colocalized Spots zum Beispiel ähnlich. Dieses Ergebnis vorgeschlagen, dass unser Protokoll eine Reihe von RNA-Moleküle in einer Zelle (bis mindestens 40 ~ Moleküle pro Zelle) erkennen könnte. Wenn das Protokoll suboptimal gearbeitet, könnte man eine Tendenz beobachten. Zum Beispiel könnte der Anteil der Spots mit nur einem der beiden fluoreszierende Signale als die Gesamtzahl der Flecken pro Zelle erhöht erhöhen.
Mit dieser optimierten Protokoll, untersuchten wir den Ausdruck des NDC80 -Gens, die in verschiedenen Wachstumsbedingungen ein Kinetochor-Protein kodiert. Das NDC80 -gen drückt zwei mRNA-Isoformen: lange undecodierten Abschrift Isoform, NDC80Luti, hat ~ 400 Basenpaaren die Erweiterung am 5'-Ende im Vergleich zu den kurzen NDC80ORF Isoform, aber beide Protokolle teilen der kodierenden Region des NDC80 -Gens (siehe schematisch in Abbildung 4A). Wir haben zwei Sätze von Sonden entwickelt: die CF 590 Satz bindet in der Region einzigartige 5' NDC80Luti; und das Q-670-Set bindet an die gemeinsame Region NDC80Luti und NDC80ORF. Die NDC80Luti Transkripte wurden erkannt, als die SmFISH Flecken, wo die Signale beider Sätze colocalized, Sonde, während, die NDC80ORF Abschriften wurden diejenigen mit dem Signal nur aus Q-670. Aufgrund der Größe des einzigartigen Segments NDC80Lutikönnten wir nur 20 Oligonukleotid Sonden entlang dieser Region Fliese ist weniger als die empfohlenen 30 bis 48-Sonden. Wir ergibt sich also erst wenn diese Sonde Serie robust RNA in unser optimiertes Protokoll erkennen könnte. Für entweder fluoreszierende Kanal wir grafisch das Signal-Rausch-Verhältnis (SNR, definiert als die Varianz der Pixel rund um einen Platz im Vergleich zu den vor Ort Intensität) gegen das Signal für jeden SmFISH Fleck erkannt, und erzeugt ein Streudiagramm für alle spots in das gesamte Sichtfeld (Beispielbilder Abbildung 4a) und Grundstücke in Abbildung 4 bidentifiziert. Zwei verschiedene Populationen wurden für beide der Q-670 eindeutig identifiziert und die CF-590-Sonde setzt (optimiert, Abbildung 4 b), was darauf hindeutet, dass die wahre SmFISH Spots (innerhalb des grauen Bereichs) aus dem Hintergrundsignal getrennt werden könnte. Beachten Sie, dass eine solche Trennung in suboptimalen Zustand, weniger offensichtliche (Suboptimal, Abbildung 4 b) war.
Wir diese zwei Sonde Sets verwendet, um die Expression von NDC80Luti studieren und NDC80ORFwährend des vegetativen Wachstums und Meiose. In den vegetativen Zellen robustes Signal aus dem Q-670-Sonde-Set wurde erkannt, aber nicht von der CF-590 Sonde gesetzt (Abbildung 5A, vegetative), Vereinbarung mit der Beobachtung von Nordbeflecken, die nur die kurze Isoform während des vegetativen Wachstums16 zum Ausdruck kam . Dieses Ergebnis auch vorgeschlagen, dass die CF 590 Sonde spezifisch, nachgiebig niedrigen Hintergrund in unserem optimierten SmFISH-Protokoll ist. Im Gegensatz dazu robustes Signal aus beide Sonde in meiotischen Prophase erkannt wurde, und hatte die Mehrheit der Spots Signal (Abb. 5A, meiotischen Prophase) colocalized. Nordfleck und analysiert (Abb. 5 b) bestätigt diese Beobachtung, dass die lange Isoform NDC80Luti speziell in Meiose zum Ausdruck kam. Diese beiden Arten von mRNAs erkannt wurden, in das Zytoplasma (außerhalb der DAPI-Region), was darauf hindeutet, dass beide aus dem Zellkern, Einklang mit dem Ribosom Profilerstellung Daten18exportiert wurden.
Um festzustellen, ob ausreichende Daten genau entfallen die biologischen Variation untrennbar mit unserem Datensatz erhoben wurden, führten wir bootstrap Analyse anhand von Statistiken der einzelnen Zellen, die enthalten (1) den Anteil der colocalized Flecken pro Zelle und (2) der Anteil der Flecken mit einem der beiden fluoreszierende Signale (CF 590 nur und nur Q-670). In dieser Analyse eine Programm nach dem Zufallsprinzip ausgewählt eine Zelle aus den 437 quantifizierten Zellen für 500 Iterationen. Als nächstes wurden der Mittelwert und die Varianz der jeweiligen Statistik verknüpft mit diesen 500 ausgewählten Zellen berechnet. Dieser Prozess wiederholte sich dann für die Auswahl nach dem Zufallsprinzip zwei Zellen aus allen Zellen, ohne Ersatz; und dann für die Auswahl nach dem Zufallsprinzip drei Zellen, etc.. bis zu einer Hälfte der gesamten Datensatz Größe erreicht wurde. Die Varianz in den Daten nach ~ 40 Zellen, darauf hindeutet, dass nach diesem Punkt die meisten die Variation in der Zwischenzeit untrennbar mit der Daten anstatt ein Artefakt der Undersampling ist Plateau (Abbildung 6, inset). Dieser Effekt wurde noch deutlicher, wenn wir die Stichprobenvarianz dividiert durch die Stichprobe-Mittelwert, in Abhängigkeit von der Anzahl der Zellen in der Stichprobe jedes Iterationszyklus (Abbildung 6) grafisch dargestellt. Mit den Daten angezeigt wurde die Änderung der Varianz i.e. klein nach ~ 60 Zellen. Die Anzahl der Zellen in einem SmFISH Experiment quantifiziert übersteigt die minimale Anzahl von Zellen erforderlich, um einen stabilen Mittelwert und einem Plateau der Varianz zu erreichen. In unserem Fall quantifizieren wir über 95 Zellen pro Probe pro replizieren. Die Gesamtzahl der Zellen (> 400) übertraf auch die minimale Anzahl von Zellen (~ 60) erforderlich, um den Mittelwert der Grundgesamtheit widerspiegeln.
Mit einem maßgeschneiderten Matlab-Programm16, vegetative Zellen wurden gefunden, um einen Median von 5 haben NDC80ORF Abschriften pro Zelle, während in meiotischen Prophase Zellen, der Median, 4 Transkripte pro Zelle (zweiseitige Wilcoxon deutlich gesunken Summe der Rank-Test, p = 0.026) (Abbildung 7). Die mittlere Anzahl der NDC80Luti Transkripte pro Zelle war 21 Transkripte und 100 % der Zellen ausgedrückt NDC80Luti Transkripte. Wir grafisch der Anteil der Zellen mit einer bestimmten Anzahl von Abschriften als eine schrittweise, relative Häufigkeit Histogramm, weil die Anzahl der mRNA-Moleküle eine diskrete Menge ist. Das größte Fach jeder Histogramm wurde auf die gleiche Höhe normalisiert.

Abbildung 1: Flussdiagramm des Protokolls SmFISH. Zellen werden mit Formaldehyd bei Raumtemperatur für 20 min oder bei 4 ° c über Nacht für vegetatives Wachstum und meiotischen Proben bzw. fixiert. Nach dem Waschen in Puffer B dreimal, werden Zellen von Zymolyase verdaut, bis ~ 70-90 % der Zellen verdaut werden. Die verdauten Proben sind dann gewaschen, einmal mit Puffer B, die Zymolyase zu entfernen, und anschließend permeabilized in 70 % igem Ethanol (EtOH) stundenlang ~3.5. Zur Vorbereitung der Hybridisierung sind Proben zunächst in 10 % Formamid Waschpuffer (FWB) für ~ 20 min inkubiert. Als nächstes sind die Proben in ~ 50 µL der Hybridisierung-Lösung, Nukleinsäuretablette, enthält die fluoreszierenden Sonden für Übernachtung Inkubation im Dunkeln bei 30 ° c. Die Proben sind nach Hybridisierung in 10 % FWB für 30 min abzuwaschen die überschüssige Sonden inkubiert und dann in 10 % FWB mit 4', 6-Diamidino-2-Phenylindole (DAPI), die DNA zu beflecken inkubiert. Für die Probe unmittelbar nach der Inkubation FWB/DAPI abgebildet ist die Probe im GLOX Puffer ergänzt mit Katalase, Trolox und Glukose-Oxidase (GLOX + Enz); Nukleinsäuretablette in der Erwägung, dass die anderen Proben sind Nukleinsäuretablette im GLOX Puffer ohne Enzyme, und bei 4 ° c gelagert bis zu ~ 3 Stunden. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2: Beurteilung der SmFISH Qualität mit ungerade/gerade Sonden. (A) oben: Schaltplan für die ungerade/gerade Sonde-Sets. Vierundfünfzig Oligonukleotid Sonden des NDC80 -Gens mit Ziegeln zu decken wurden entwickelt. Die ungeraden Sonden wurden mit einem Fluorophore (CF590, in Magenta dargestellt) und die geraden Sonden mit einem anderen Fluorophor (Q670, grün dargestellt) beschriftet. Unten: Vertreter SmFISH Bilder von meiotischen Prophase Zellen mithilfe der ungerade/gerade Sonde erworben. 6 Stunden nachdem Zellen (UB8144) eine Zeit auf Sporenbildung Medium übertragen wurden als diese Zellen in meiotischen Prophase verhaftet wurden, wurden Proben genommen. DNA wurde mit DAPI (blau dargestellt) gebeizt. Bilder werden als die maximale Intensität Projektionen der Z-Stapel angezeigt. Maßstabsleiste: erhalten die ungerade/gerade Sonde-Sets mit 5 µm. (B) Prozentsatz der gepaart oder ungepaart SmFISH Flecken. Insgesamt 428 meiotischen Prophase Zellen wurden analysiert, pooling von zwei unabhängigen Experimenten. Diese Zahl ist aus Abbildung 2geändert-Figur Zuschlag 4 von Chen Et Al. 16 (C) A kumulative Dichte Funktion (CDF) des Abstandes zwischen je zwei gekoppelten Flecken und der Abstand zwischen der nächste Nachbar ein Ungepaartes Spot. Für jeden gefundenen Fleck in einem fluoreszierenden Kanal, der "k nächsten Nachbarn" Algorithmus wurde angewandt, um am nächsten gefundenen Ort zu identifizieren – und die Entfernung zu diesem Punkt — in den ergänzenden Kanal. Lokalisierungen, die gegenseitige nächste Nachbarn waren galten gekoppelt werden, und eine neue Liste generiert wurde, Aufnahme der gekoppelten, nur CF590 und Q670 nur-Erkennungen. Für jede Kategorie von Erkennungen war ein CDF-Histogramm der Abstände in Matlab, bestätigt, dass richtig gekoppelte Flecken in der Tat viel näher in ferne als solche ohne eine entsprechende Stelle in den anderen Kanal wurden aufgetragen. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: Bruchteile von gepaarten und ungepaarten Flecken, in Abhängigkeit von der Gesamt-RNS pro Zelle Nummer. Um zu testen ob die Erkennung und Paarung der SmFISH Flecken auf verschiedenen Ausdruck Ebenen befangen, wurden einzelne Zellen durch total RNA Ausdruck gruppiert. Die mittlere Bruchteile von gekoppelten, nur CF590 und Q670 nur-Erkennungen wurden für jede Gruppe von Zellen berechnet. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4: Qualitätsbeurteilung von SmFISH Daten mit den Sonden für NDC80Luti entworfen und NDC80ORF mRNAs. Die Q-670-Sonden (grün dargestellt) in der gemeinsamen Region geteilt zwischen NDC80Luti hybridisieren und NDC80ORF mRNAs, während die CF-590-Sonden (in Magenta dargestellt) in der Region einzigartige 5' NDC80Luti hybridisieren . (A) Vertreter SmFISH Bilder von NDC80Luti und NDC80ORF meiotischen Prophase erwarb unter im Vergleich zu suboptimalen Bedingungen optimiert. UB8144-Stämme (optimiert Zustand) und UB1337 (suboptimalen Zustand) induzierte Meiose unterziehen und während meiotischen Prophase behoben wurden. Diese beiden Stämme beherbergen verschiedene Synchronisation Systeme zur Meiose zu induzieren, aber die Verwendung von entweder System hat keinen Einfluss auf SmFISH Qualität (Daten nicht gezeigt). In den suboptimalen Zustand wurden Zellen bei Raumtemperatur für 20 min (keine Übernachtung Fixierung), verdaut mit Zymolyase ohne VRC ergänzt und in einer niedrigeren Konzentration des VRC hybridisiert fixiert. Bilder werden als die maximale Intensität Projektionen der Z-Stapel angezeigt. DNA wurde mit DAPI (blau dargestellt) gebeizt. Maßstab: 5 µm. (B) Scatterplots Anzeige das Signal-Rausch-Verhältnis (SNR) und das Signal von jedem SmFISH vor Ort in das Sichtfeld erkannt dargestellt in Abbildung 4A, für entweder fluoreszierende Kanal sowie die optimierte oder suboptimalen Bedingungen. Im optimierten Zustand lebten zwei SmFISH Flecken vorhanden. Die wahre SmFISH Spots befanden sich in der Grauzone, trennt das Hintergrundsignal. In suboptimalen Zustand einzelnen mRNAs waren hart, von Auge zu unterscheiden, und die Trennung zwischen den wahren Spots und Hintergrund nach dem Ausführen der Spoterkennung Software war weniger offensichtlich. Bitte klicken Sie hier für eine größere Version dieser Figur.
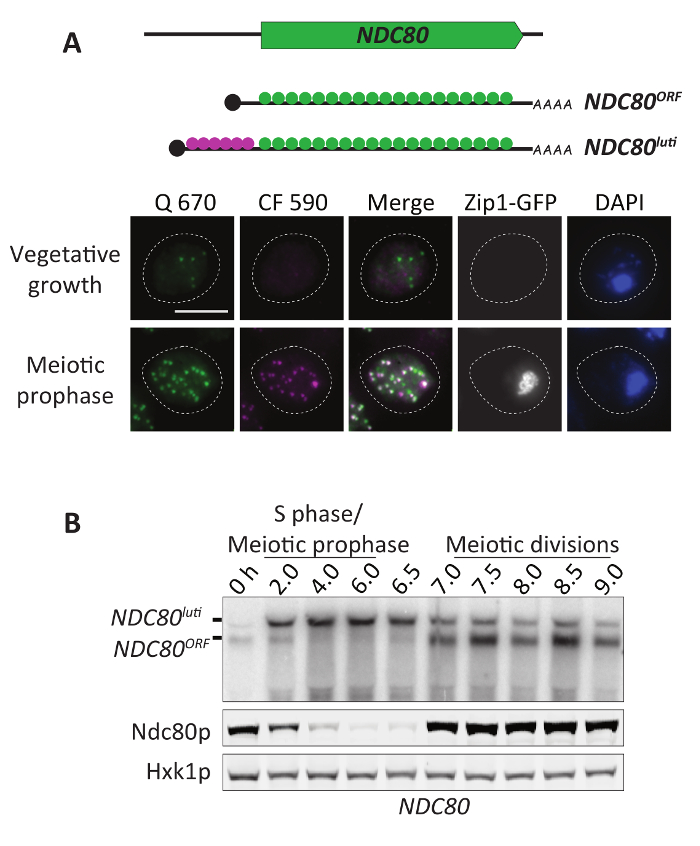
Abbildung 5: Ausdruck der NDC80Luti und NDC80ORF Abschriften sind zeitlich gesteuert. (A) Vertreter SmFISH Bilder von NDC80Luti und NDC80ORF während des vegetativen Wachstums und Meiose. Wenn Zellen (UB8144) exponentiell wachsende im Reich Nährmedium wurden, wurden die vegetative Proben genommen. 6 Stunden nachdem Zellen (UB8144) eine Zeit auf Sporenbildung Medium übertragen wurden als diese Zellen in meiotischen Prophase verhaftet wurden, wurden meiotischen Prophase Proben genommen. Die Q-670-Sonden (grün dargestellt) in der gemeinsamen Region geteilt zwischen NDC80Luti hybridisieren und NDC80ORF mRNAs, während die CF-590-Sonden (in Magenta dargestellt), der einzigartige 5' Region NDC80hybridisieren Luti . DNA wurde mit DAPI (blau dargestellt) gebeizt. Jede Zelle wurde durch seine Zip1-GFP-Signal, ein Marker für meiotischen Prophase inszeniert. Unser SmFISH-Protokoll bewahrt starkes GFP Signal ohne weitere Modifikation. Vegetatives Wachstum: Zip1-GFP negativ. Meiotischen Prophase: Zip1-GFP positiv. Bilder werden als die maximale Intensität Projektionen der Z-Stapel angezeigt. Maßstab: 5 µm. Diese Zahl ist aus Abbildung 2 von Chen Et Al. geändert. 16. (B) NDC80ORF, NDC80Lutiund Ndc80 Protein (Ndc80p) Ebene während der Meiose (UB4074). NDC80Luti und NDC80ORF Ebenen wurden durch Nordfleck, und Ndc80p Ebene wurde durch Anti-V5 Immunoblot zu den angegebenen Zeitpunkten bestimmt. Hxk1p, Ladekontrolle für Immunoblot. Als UB8144 Stamm, birgt auch diese Sorte der pGAL-NDT80 GAL4-ER Synchronisation System19,20. Zellen wurden auf 0 Stunden auf Sporenbildung Medium übertragen und durch β-Östradiol zusätzlich 6 Stunden später aus der Pachytene Haft entlassen. Die NDC80Luti Abschrift in S/meiotischen Prophase robust erkannt wurde, während die NDC80ORF Transkripte war überwiegend vor der meiotischen Eintrag (0 Stunden) und während die meiotischen Divisionen (7-9 Stunden). Diese Zahl ist aus Abbildung 6J von Chen Et Al. geändert. 16 Bitte klicken Sie hier für eine größere Version dieser Figur.
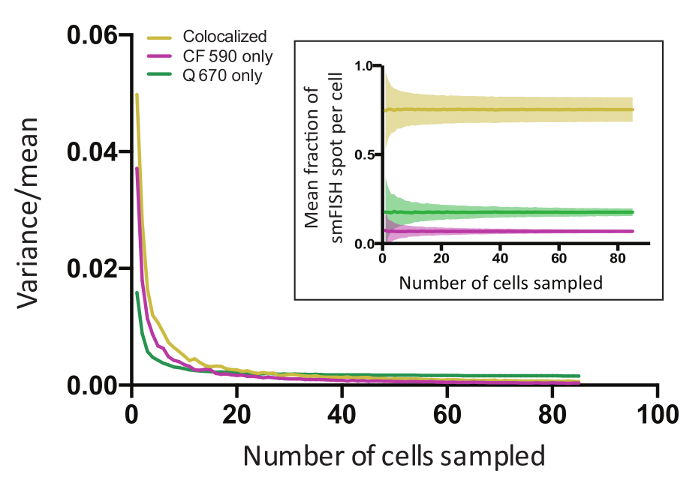
Abbildung 6: Bootstrap-Analyse für die meiotischen Prophase Proben gezeigt Abbildung 5 . Aller quantifizierten Zellen wurden zusammengefasst und eine bestimmte Anzahl (n) der Zellen wurden nach dem Zufallsprinzip 500 mal probieren. Der Mittelwert und 95 % Konfidenzintervall wurden für den Bruchteil der gepaarten und ungepaarten mRNA pro Zelle berechnet. Diese Daten wurden für jede Wahl die Zahl n (kleines Foto) aufgetragen. Das Plateau in Varianz wurde visualisiert, indem Plotten der Stichprobenvarianz dividiert durch den Mittelwert in Abhängigkeit von der Anzahl der Zellen (n) abgetastet. Die Gesamtzahl der Zellen gemessen (437) überstiegen die Nummer auf dem Plateau des Fehlers (~ 60 Zellen), darauf hinweist, dass zusätzliche Daten nicht das Vertrauen in die Messungen verbessern würde. Bitte klicken Sie hier für eine größere Version dieser Figur.
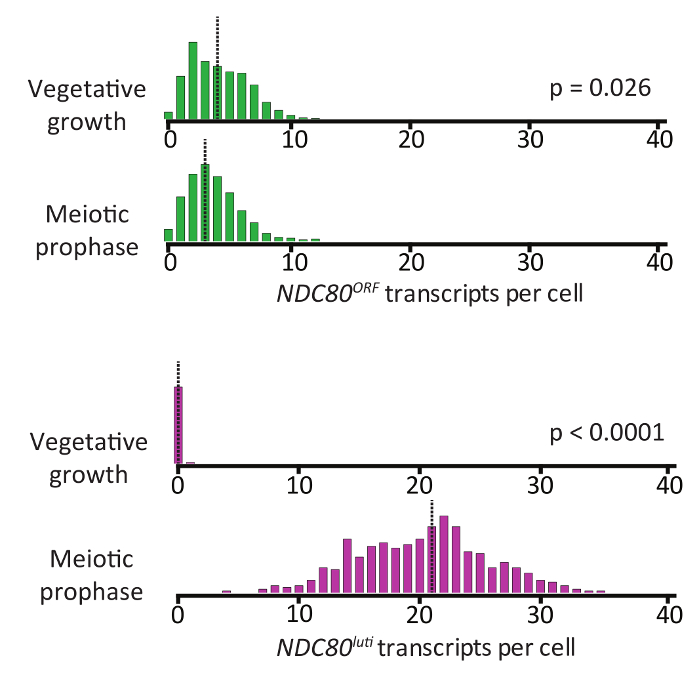
Abbildung 7: Quantifizierung der SmFISH Daten in Abbildung 5 , als die relative Häufigkeit Histogramme der Zellen mit einer bestimmten Anzahl von NDC80Luti gezeichnete und NDC80ORF Abschriften pro Zelle, mit den Daten aus drei unabhängigen Experimenten gebündelt. Die gestrichelte Linie gibt die mittlere Anzahl der NDC80Luti und NDC80ORF Abschriften pro Zelle. Jede Histogramm war normalisiert, so dass die maximale bin Höhe der gleichen quer durch die Histogramme. Eine Gesamtanzahl von 637 Zellen wurden für vegetatives Wachstum und 437 für meiotischen Prophase analysiert. Zweiseitige Wilcoxon Rang Summe durchgeführt wurde für NDC80Luti und NDC80ORF, vegetative und meiotischen Prophase Proben zu vergleichen. Diese Zahl wird von Figur 2D von Chen Et Al. geändert. 16 Bitte klicken Sie hier für eine größere Version dieser Figur.
| Puffer B (1 L Brühe: 1,2 M Sorbit; 0,1 M Kalium-Phosphat-Puffer pH 7,5) | 1 x |
| Nuklease-freies Wasser | 500 mL |
| Sorbit | 218,6 g |
| KH2PO4 | 2,18 g |
| K2HPO4 | 14,62 g |
| Nuklease-freies Wasser | 1 L Endvolumen bringen |
| * Lagerung bei 4 ° c in 50-mL-aliquoten nach Filter sterilisieren | |
| Zymolyase 100 t (10 mg/mL) | |
| * Lagerung bei-20 ° c in Aliquote | |
| 10 mg Zymolyase Pulver in 1 mL MilliQ Wasser auflösen | |
| E. coli tRNA (10 mg/mL) | |
| * Lagerung bei-20 ° c in Aliquote | |
| 10 mg tRNA Pulver in 1 mL MilliQ Wasser auflösen | |
| 70 % igem Ethanol (50 mL) | 1 x (mL) |
| Reinem ethanol | 35 |
| Nuklease-freies Wasser | 15 |
| * Bei Raumtemperatur lagern | |
| Hybridisierung-Puffer (10 mL) | 1 x (mL) |
| 50 % Dextran Sulfat | 2 |
| E. Coli tRNA (10 mg/mL) | 1 |
| 200 mM Vanadyl Ribonucleoside Komplex (VRC) | 0.1 |
| BSA, 50 mg/mL | 0,04 |
| 20 X SSC | 1 |
| Formamid | 1 |
| Nuklease-freies Wasser | 4,86 |
| * Bei-20 ° c in 250 µL oder 500 µL Aliquots Lagern | |
| 10 % Formamid Waschpuffer (10 mL) | 1 x (mL) |
| Formamid bei Raumtemperatur | 1 |
| 20 X SSC | 1 |
| Nuklease-freies Wasser | 8 |
| * machen Sie frisch, Wirbel für 20-30 Sekunden mischen | |
| 10 % Glukoselösung | |
| * Lagerung bei 4 ° c in Aliquote nach Filter sterilisieren | |
| 1 g Glukose in 10 mL Nuklease-freies Wasser auflösen | |
| Glukose-oxidase | |
| * Lagerung bei-20 ° c in Aliquote | |
| Auflösen von 3,7 mg Glukose-Oxidase in 1 mL 50 mM NaOAc, pH 5 | |
| Anti-Bleichmittel (GLOX) Puffer ohne Enzyme (1 mL) | 1 x (µL) |
| 10 % Glukose im Nuklease-freies Wasser | 40 |
| 1 M Tris, pH 8,0 | 10 |
| 20 X SSC | 100 |
| Nuklease-freies Wasser | 850 |
| * frisch machen, können auch Aliquote bei 4 ° c Lagern | |
| Anti-Bleichmittel (GLOX) Puffer mit Enzymen (50 µL) | 1 x (µL) |
| Katalase (Vortex gelinde gesagt, es setzt sich leicht) | 0,5 |
| Glukose-oxidase | 0,5 |
| 100 mM Trolox (gelöst in Ethanol) | 1 |
| GLOX Puffer | 50 |
| * frisch machen jedes Mal, auf dem Eis vorbereiten, können für ca. 2-3 Stunden bei 4 ° c gelagert werden |
Tabelle 1. Puffer und Medien
Diskussion
Das Protokoll hier vorgestellten stammt von anderen veröffentlichten SmFISH Protokolle2,3,21,22,23, und ist speziell für den Hefe-Stamm Hintergrund SK1 optimiert. Die Parameter optimiert enthalten Zellzahl, Fixierung Dauer, Zentrifugation Geschwindigkeit und Dauer, Dauer der Verdauung, Verdauung Puffer, Sonde Konzentration und Hybridisierung Puffer. Andere Parameter wie Hybridisierung Temperatur und Dauer wurden nicht optimiert. In diesem Abschnitt haben wir ein paar Hinweise, die helfen würden, dieses Protokoll Hefe-Stamm-Hintergrund und die Wachstumsbedingungen von Interesse anzupassen.
Die Zellwand der angehende Hefe ist eine große Herausforderung gegen hochwertige SmFISH Bilder in der angehenden Hefe zu erhalten, weil die Zellwand Sonde Eindringen verhindert. Unvollständige Verdauung der Zellwand von Zymolyase führt zu ineffizienter Hybridisierung von Sonden und hohe Variabilität von Zelle zu Zelle im Signal. Aber können über Verdauung Zellen zu schwach zu machen, führt zu erheblichen Zell-Verlust während der Waschschritte und Zelle platzt während der Vorbereitung der Folie für die Bildgebung. Daher ist es entscheidend, die Dauer der Verdauung zu optimieren. Wir empfehlen die Durchführung eines pilot SmFISH Experiments durch Verdauung derselben Probe für unterschiedlich lange. In unserem Fall haben wir die besten SmFISH Daten Wenn wir die Verdauung hielten bei ~ 80 % der Zellen nicht Refraktive wurde. In der Regel können für dasselbe Wachstum, verschiedene genetische mutierte Stämme mit ähnlicher Timing verdaut werden. Allerdings unterscheidet sich die Dauer der Verdauung Vergleich unter verschiedenen Wachstumsbedingungen und verschiedenen Phasen der Meiose in der angehenden Hefe. Sobald der Zeitpunkt bestimmt ist, ist die Qualität der SmFISH reproduzierbar. Hinweis kann für mutierte Stämme mit defekten Zellwand-Synthese und/oder Zusammensetzung, Verdauung in weniger als 15 min. Einsatz verschiedener Chargen Zymolyase durchgeführt werden kann auch leicht die Verdauung Timing ändern.
Optimale Sonde Konzentration ist erforderlich, um ein hohes Signal-Rausch-Verhältnis zu erreichen. Wir entwarfen und unsere SmFISH Sonden im Handel gekauft. Gute Sonden (oft 20-Mers) sollte einen Prozentsatz von GC von 35 % bis 45 %, ein Mindestabstand von 2 Basenpaare zwischen Fühler und geringe Kreuzreaktivität haben. Wir einen Web-basiertes Sonde Designer vom Hersteller verwendet, um eine Liste der Sonden generieren, und gäbe es genügend Sonden zur Auswahl, wir würde Nutzen des BLAST-Algorithmus in der Saccharomyces-Genom-Datenbank die Sonden mit mehr als 17 Basenpaaren überlappt zu beseitigen mit anderen genomischen Regionen. Wir entschieden uns für mehr photostabilen Fluoreszenzfarbstoff (CAL-Fluor-590) für die Sonde gesetzt, die die einzigartige Region NDC80Luti (CF 590) glüht, weil in dieser Reihe, die Anzahl der Sonden, die alle der oben genannten Kriterien (20 Proben) zufrieden niedriger als war die minimale Anzahl der vom Hersteller empfohlenen (~ 25-Sonden). Nach dem Neuaufbau der Sonde Lösung nach den Anweisungen des Herstellers, wir machten eine 01:10 Verdünnung der Stammlösung und die verdünnte Lösung in 5 µL Aliquots bei-20 ° c gelagert. Jedes aliquoten wurde nur ein einziges Mal verwendet. Für die Optimierung sollte man serielle Verdünnungen ab dem 01:10 verdünnt, Lösung und testen, welche Konzentration das beste Signal-Rausch-Verhältnis ergibt. Wir empfehlen, dass eine Verdünnungsreihe 1: 250, 1: 500, 1: 1000 und 1: 2000 aus dem ursprünglichen bestand. In unserem Fall führte ein Verdünnungsfaktor von 1: 500 das beste Signal-Rausch-Verhältnis für die CF-590 und Q-670-Sonde-Sets.
Optimale Fixierung ist auch entscheidend für erfolgreiche SmFISH in der angehenden Hefe. Wir fanden, dass über Nacht Fixierung bei 4 ° c, anstatt Befestigung bei Raumtemperatur für 20 min, verbessert die Konsistenz und Qualität der SmFISH Ergebnisse für meiotische Proben. Obwohl wir nicht getestet haben, wie über Nacht Fixierung vegetative Proben auswirken könnten, Fixierung bei Raumtemperatur gut für dieses Wachstum Zustand gearbeitet. Also, um das SmFISH-Protokoll für neue Stämme oder Wachstumsbedingungen zu optimieren, empfehlen wir beginnend mit kurze Fixierzeit bei Raumtemperatur und Erhöhung der Fixierzeit, wenn hohe Variabilität des Signals aufgetreten ist.
Im Vergleich zu anderen veröffentlichten Protokollen3,22,23, nutzt unser Protokoll die RNase-Inhibitor VRC während der Verdauung und eine höhere Konzentration von VRC während Hybridisierung. Ergänzung der VRC in diesen zwei Schritten verbessert die Konsistenz der SmFISH Ergebnisse, möglicherweise durch eine bessere Erhaltung der RNA-Moleküle gegen Nuklease Aktivität, die durch die Zymolyase-Mischung eingeführt werden können (das Enzym wird aus groben Extrakten gereinigt und RNase enthalten kann (Verunreinigungen). Daher empfehlen wir die Menge der VRC wie entnehmen Sie bitte unserem Protokoll oder sogar höhere Konzentrationen von VRC für Optimierung.
Ein erheblicher Teil der Zellen kann während Waschschritten in Puffer B und die Formamid Waschpuffer verloren. Um Zellverlust zu reduzieren, könnte man die Zentrifugation Geschwindigkeit erhöhen. In unserem Fall wäscht mit einer hohen Geschwindigkeit (21.000 x g) zu unseren Proben innerhalb der Puffer B Pellets deutlich reduzierten Zellverlust. Jedoch werden die Zellen sehr zerbrechlich nach Zymolyase Verdauung, daher Zentrifugation Geschwindigkeit ändern nicht empfohlen. Stattdessen empfehlen wir die niedrigem Kraftschlußbeiwert Rohre aus USA, die wesentlich dazu beitragen, die Zellen während der Wäschen in der Formamid Waschpuffer pellet. Insgesamt kann unser Protokoll konsequent eine Monolage Zellen dicht genug für die effiziente Bildgebung generieren. In der Regel 7 Gesichtsfelder nachgeben sollte > 130 Zellen geeignet für Quantifizierung.
Zu guter Letzt ist es wichtig zu bestimmen, die optimale Parameter für und die Ausgänge von Bildanalyse erforderlich. Um SmFISH Flecken zu erkennen, veröffentlichten Analyse-Programme häufig filtern die raw-Bilder mit "glockenförmig" Kernel um Hintergrundsignal zu entfernen, und fordern Sie die Benutzer zu bestimmen, das Signal-Rausch-Verhältnis, für jeden Satz von Bildern2,17zu verwenden. Leider gibt es derzeit kein einheitlichen Standard die richtigen Parametern bestimmt, und so einige empirische Tests diese unterschiedlichen Einstellungen ist notwendig. Zur Einstellung der Parameter für jeden dieser Schritte benötigt, braucht man iterativ unterschiedliche Parameter eingeben und überprüfen, wie gut die Ergebnisse aus jedem Satz entspricht, die von der manuellen Zählung in ein paar repräsentative Zellen. Sobald eine Reihe von Parametern gefunden wird, kann es für den Großteil der Bilder, die trotz unterschiedlichen Wachstumsbedingungen und genetische Hintergründe verwendet werden.
Darüber hinaus kann man testen, ob maximale Intensität Projektion der SmFISH Bilder vor der Quantifizierung22durchgeführt werden kann. Dieser Schritt den spot Erkennungsalgorithmus vereinfacht und reduziert bildgebenden Zeit, allerdings auf Kosten der potenziell nützliche Informationen zu einzelnen Flecken, wie ihre subzelluläre Lokalisation. In unserem Fall war die Anzahl der mRNA-Moleküle, die durch das NDC80 -gen produziert niedrig genug, dass die Flecken gut, nach dieser Bearbeitung (nicht selten getrennt wurden für Transkripte in angehende-3,7 Hefe). In Fällen, in denen die ns1 Analyse entscheidend ist, muss die Analyse Pipeline zur Bestimmung der Position jeder SmFISH Stelle in jedem Kanal ns1 beurteilen. Je nach den konkreten Fragen gestellt, weitere Informationen wie die Intensität von jedem Fleck möglicherweise auch die Pipeline zur weiteren Analyse entnommen werden. Optimierung des Schlüssels Schritte in das Protokoll und die Bild-Analyse-Pipeline ist entscheidend bei der Beschaffung von hohen Qualität der SmFISH Daten, die Frage von Interesse zu studieren.
Offenlegungen
Die Autoren haben keinen Interessenkonflikt, offen zu legen.
Danksagungen
Wir bedanken uns bei Anne Dodson und Stephanie Heinrich für Ratschläge, wie Sie das SmFISH-Protokoll zu optimieren, Xavier Darzacq für helfen bei der Analyseplattform, Haiyan Huang für Anregungen über die statistische Auswertung. Diese Arbeit wurde mit Mitteln der March of Dimes (5-FY15-99), Pew Charitable Trusts (00027344), Damon Runyon Cancer Research Foundation (35-15) und Glenn Foundation zu EÜ und NSF Graduate Research Fellowship Grant No unterstützt. DGE-1106400, JC.
Materialien
| Name | Company | Catalog Number | Comments |
| BSA, RNase-free (50 mg/mL) | Ambion | AM2616 | Store at -20 °C. |
| Catalase | Sigma | C3515 | Store at 4 °C for short-term. Vortex before use. |
| DAPI | Sigma | D9564 | Store at -20 °C after reconstitution in water. Protect from light. |
| 50% Dextran Sulfate | Milli Pore | S4030 | Store at room temperature. Very viscous liquid. Handle with patience. |
| E. coli tRNA | Sigma | R4251 | Store at -20 °C in aliquots after reconstitution in water. |
| Ethanol (100%, 200 proof) | various | Flammable. | |
| 37% Formaldehyde | Fisher | F79-500 | Store at room temperature. Toxic. Use and dispose with caution. |
| Formamide | Ambion | AM9342 | Store at 4 °C. Open after the temperature equilibrates to room temperature in order to prevent oxidation. Toxic. Use and dispose with caution. |
| Glucose | various | Store at 4 °C after dissolving in nuclease-free water. | |
| Glucose oxidase | Sigma | G2133 | Store at -20 °C in aliquots after reconstitution in 50 mM NaOAc, pH 5. |
| Nuclease-free water | Ambion | AM9932 | Store at room temperature. |
| Potassium phosphate (monobasic and dibasic) | various | Store at room temperature. | |
| Probe library, diluted in TE, pH 8.0 | Biosearch Technologies | Store at -20 °C in aliquots (5 µL) after reconstitution in TE pH 8.0, following the instructions from the manufacturer. Protect from light. | |
| Sorbitol | various | Store at room temperature. | |
| 20X SSC | Ambion | AM9763 | Store at room temperature. |
| TE, pH 8.0 | Ambion | AM9849 | Store at room temperature. |
| 1 M Tris, pH 8.0 | Ambion | AM9856 | Store at room temperature. |
| Trolox | Sigma | 238813 | Store at -20 °C in aliquots. |
| 200 mM Vanadyl ribonucleoside complex (VRC) | NEB | S1402S | Store at -20 °C in aliquots after reconstitution, following the instructions from the manufacturer. |
| Zymolyase 100T | MP Biomedicals | 08320932 | Store at -20 °C in aliquots after reconstitution in water. |
| Low-adhesion tubes | USA Scientific | 1415-2600 | Store at room temperature. |
Referenzen
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of single RNA transcripts in situ. Science. 280 (5363), 585-590 (1998).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat Methods. 5 (10), 877-879 (2008).
- Rahman, S., Zenklusen, D. Single-molecule resolution fluorescent in situ hybridization (smFISH) in the yeast S. cerevisiae. Methods Mol Biol. 1042, 33-46 (2013).
- Gaspar, I., Ephrussi, A. Strength in numbers: quantitative single-molecule RNA detection assays. Wiley Interdiscip Rev Dev Biol. 4 (2), 135-150 (2015).
- Vargas, D. Y., et al. Single-molecule imaging of transcriptionally coupled and uncoupled splicing. Cell. 147 (5), 1054-1065 (2011).
- Waks, Z., Klein, A. M., Silver, P. A. Cell-to-cell variability of alternative RNA splicing. Mol Syst Biol. 7, 506(2011).
- Zenklusen, D., Larson, D. R., Singer, R. H. Single-RNA counting reveals alternative modes of gene expression in yeast. Nat Struct Mol Biol. 15 (12), 1263-1271 (2008).
- Raj, A., Peskin, C. S., Tranchina, D., Vargas, D. Y., Tyagi, S. Stochastic mRNA synthesis in mammalian cells. PLoS Biol. 4 (10), e309(2006).
- Senecal, A., et al. Transcription factors modulate c-Fos transcriptional bursts. Cell Rep. 8 (1), 75-83 (2014).
- Levesque, M. J., Ginart, P., Wei, Y., Raj, A. Visualizing SNVs to quantify allele-specific expression in single cells. Nat Methods. 10 (9), 865-867 (2013).
- Hansen, C. H., van Oudenaarden, A. Allele-specific detection of single mRNA molecules in situ. Nat Methods. 10 (9), 869-871 (2013).
- Ginart, P., et al. Visualizing allele-specific expression in single cells reveals epigenetic mosaicism in an H19 loss-of-imprinting mutant. Genes Dev. 30 (5), 567-578 (2016).
- Long, R. M., et al. Mating type switching in yeast controlled by asymmetric localization of ASH1 mRNA. Science. 277 (5324), 383-387 (1997).
- Park, H. Y., Trcek, T., Wells, A. L., Chao, J. A., Singer, R. H. An unbiased analysis method to quantify mRNA localization reveals its correlation with cell motility. Cell Rep. 1 (2), 179-184 (2012).
- Jourdren, L., Delaveau, T., Marquenet, E., Jacq, C., Garcia, M. CORSEN, a new software dedicated to microscope-based 3D distance measurements: mRNA-mitochondria distance, from single-cell to population analyses. RNA. 16 (7), 1301-1307 (2010).
- Chen, J., et al. Kinetochore inactivation by expression of a repressive mRNA. eLife. 6, e27417(2017).
- Mueller, F., et al. FISH-quant: automatic counting of transcripts in 3D FISH images. Nat Methods. 10 (4), 277-278 (2013).
- Brar, G. A., et al. High-resolution view of the yeast meiotic program revealed by ribosome profiling. Science. 335 (6068), 552-557 (2012).
- Carlile, T. M., Amon, A. Meiosis I is established through division-specific translational control of a cyclin. Cell. 133 (2), 280-291 (2008).
- Benjamin, K. R., Zhang, C., Shokat, K. M., Herskowitz, I. Control of landmark events in meiosis by the CDK Cdc28 and the meiosis-specific kinase Ime2. Genes Dev. 17 (12), 1524-1539 (2003).
- Dodson, A. E., Rine, J. Heritable capture of heterochromatin dynamics in Saccharomyces cerevisiae. eLife. 4, e05007(2015).
- Trcek, T., et al. Single-mRNA counting using fluorescent in situ hybridization in budding yeast. Nat Protoc. 7 (2), 408-419 (2012).
- Youk, H., Raj, A., van Oudenaarden, A. Imaging single mRNA molecules in yeast. Methods Enzymol. 470, 429-446 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten