Method Article
Optical Microscopy Scatter auf zweidimensionalen Gabor-Filtern
In diesem Artikel
Zusammenfassung
Wir zeigen eine Dunkelfeld-Mikroskopie-Methode auf Gabor-ähnliche Filterung subzellulären Dynamik in einzelnen lebenden Zellen zu messen basiert. Die Technik ist empfindlich auf Veränderungen in der Struktur der Organellen, wie Mitochondrien Fragmentierung.
Zusammenfassung
Wir zeigen eine mikroskopische Instrument, das subzelluläre Textur aus Organellen Morphologie und Organisation innerhalb ungefärbten lebenden Zellen messen können. Das vorgeschlagene Instrument erweitert die Empfindlichkeit des label-free optische Mikroskopie an nanoskaligen Veränderungen in Organellen Größe und Form und kann verwendet werden, um die Untersuchung der Struktur-Funktions-Beziehung in Bezug auf Organelle Dynamik zugrunde liegenden fundamentalen biologischen Prozessen wie der programmierte Zelltod oder zellulären beschleunigen Differenzierung. Das Mikroskop kann leicht auf die bestehenden Mikroskopie-Plattformen implementiert werden und können daher für einzelne Laboratorien, in denen Wissenschaftler implementieren und können mit dem vorgeschlagenen Verfahren mit unbeschränktem Zugriff verbreitet werden.
Die vorgeschlagene Technik ist in der Lage, subzelluläre Struktur durch die Beobachtung der Zelle durch zweidimensionale optische Gabor-Filter zu charakterisieren. Diese Filter können zu spüren, mit nanoskaligen (10 ist der nm) Empfindlichkeit, spezifischen morphologischen Eigenschaften in Bezug auf die Größe und Orientierung des nicht-sphärischen subzellulären Organellen abgestimmt werden. Während auf dem Kontrast von elastischen Streuung verursacht haben, ist die Technik nicht auf einer detaillierten inverse Streu-Modell oder Mie-Theorie stützen, um morphometrische Messungen zu extrahieren. Diese Technik ist daher für nicht-sphärische Organellen in denen eine genaue theoretische Beschreibung Streuung nicht einfach gegeben ist, und bietet unverwechselbare morphometrischen Parameter, die innerhalb ungefärbten lebenden Zellen gewonnen werden, um ihre Funktion zu beurteilen können. Die Technik ist vorteilhaft im Vergleich mit der digitalen Bildverarbeitung, dass es wirkt sich direkt auf das Objekt-Feld zu verwandeln, anstatt die diskretisierten Objekts Intensität. Es kommt nicht auf hohe Bildqualität Abtastraten verlassen und kann daher verwendet werden, um schnell Bildschirm morphologische Aktivität in Hunderten von Zellen zu einem Zeitpunkt, somit erheblich erleichtert das Studium der Organellen-Struktur über einzelne Organellen Segmentierung und Rekonstruktion durch Fluoreszenz konfokale Mikroskopie von stark vergrößerten digitalen Bildern der begrenzten Bereichen zu sehen.
In dieser Demonstration zeigen wir Daten aus einem marinen Kieselalgen, die Methodik zu veranschaulichen. Wir zeigen auch vorläufige Daten aus lebenden Zellen gesammelt, um eine Idee, wie das Verfahren kann in einem relevanten biologischen Kontext angewendet werden geben.
Protokoll
1. Getting die Zellen bereit
- Die Zellen, die den Tag vergoldet waren, bevor mit Mitotracker grün für Fluoreszenz-Bildgebung der Mitochondrien gekennzeichnet werden müssen.
- Entfernen 100 uM Stammlösung Mitotracker grün in DMSO hergestellt zuvor aus dem 4 ° C Gefrierschrank, und auf Raumtemperatur erwärmt mit der Hand. Auch nehmen bovine Endothelzellen (BAEC) Zellkulturmedium auch vorher vorbereitet und warm bis 37 ° C im Wasserbad Tischgerät.
- Sobald die Mitotracker und Kulturmedium erwärmt werden, statt diese in die Haube achten Sie darauf, Ihre behandschuhten Hände und alle äußeren Oberflächen der Behälter mit 70% Ethanol-Lösung zu sterilisieren. Nicht auf der Motorhaube Ampel, als Fluoreszenzmarker ist lichtempfindlich und wird schnell in Raumbeleuchtung photobleach.
- Die richtige Konzentration für die mitochondriale Kennzeichnung ist sehr wichtig. Zu wenig wird nicht label den Mitochondrien wirksam, während zu viel Mitotracker toxische Wirkungen haben kann. Eine Konzentration von 100 nM von Mitotracker für 45 Minuten mit den Zellen inkubiert gut funktioniert. Bereiten Sie diese Konzentration durch Zugabe von 100 ul der Mitotracker Lager zu 10 ml Kulturmedium in einer 15 ml Tube. Dadurch wird viel für mindestens ein Experiment.
- Ersetzen Sie das vorhandene Medium mit der Bezeichnung Medium durch Absaugen des alten Mediums mit einer Pasteurpipette mit dem Vakuum-Leitung. Dann sofort 2 ml des markierten Mediums zu jedem besetzten Kultur auch in der 6-Well-Platte.
- Da die Fluoreszenz-Markierung ist lichtempfindlich, ersetzen Sie die Zellen in den Inkubator schnell ohne dass eine direkte Lichteinstrahlung. Abdecken der 6-Well-Platte mit den Händen arbeitet gut für diese. Die Zellen werden im Brutschrank für 45 Minuten zu bleiben.
2. Getting den optischen Aufbau bereit
- Während die Zellen in den Inkubator zu warten, müssen wir uns auf den optischen Aufbau einzuschalten. In der Optik Raum, zuerst auf die Quecksilber-Lampe, gefolgt von dem Computer, das Mikroskop, die Kameras und des Lasers. Dann stecken Sie in der digitalen Mikrospiegel (DMD) und die Spinnen-Diffusor.
- Vergewissern Sie sich, dass die optische starten, indem Sie durch das Mikroskop-Okular, um sicherzustellen, dass das Sichtfeld hell wird durch das Laserlicht beleuchtet ausgerichtet ist.
- Reinigen Sie das Objektiv durch Falten ein Stück Linse Papier in einem engen Platz und greifen eng mit hemostat. Tauchen Sie die Linse Papier in Ammoniak-freien Glas Reinigungslösung auf eine winzige Menge in das Papier aufzunehmen. Rap der hemostat auf Ihrer freien Hand mehrmals zu entfernen überschüssige. Wischen Sie das Ziel durch fest Anwendung einer sauberen, kontinuierlichen Streichen über das Ziel von einem Ende zum anderen, geht über die Linse in der Mitte. Nicht wieder streichen oder zu scheuern. Entsorgen Sie verbrauchte Papier.
- So laden Sie die Probe, legen Sie das Fadenkreuz über dem 63x Ölimmersionsobjektiv durch Fallenlassen 1-2 kleine Tropfen Immersionsöl vor dem Ziel, während das Ziel ist den ganzen Weg hinunter. Dann legen Sie das Fadenkreuz in die Bühne. Dann heben das Ziel, so dass das Öl "greift" der Probe. Schwerpunkt der Probe in das Okular.
- So richten Sie den Kondensator, stellen Sie den Kondensator Höhe, so dass es im Zentrum von Kohler Beleuchtung wird durch die Fokussierung des sechseckigen Rand des Kondensators Feldblende ausgerichtet. Zentrum der Kondensator Feld über das Sichtfeld gegebenenfalls zu stoppen, indem Sie den Kondensator Zentriernoppen. Der Kondensator Blende geschlossen werden.
- Starten Sie das Programm IPLab und geben Sie die Einstellungen auf die RoperScientific Cascade 512 Kamera zu bedienen. Bestätigen Sie, dass die Kamera so eingestellt ist, Transfer-Modus Rahmen. Starten Sie die Live-Vorschau, indem Sie den "Acquire Fokus"-Befehl. Setzen Sie den Index Präfix und Speicherort für die Bilder gespeichert werden.
- Starten Sie das Programm RSImage und geben Sie die Einstellungen auf die CoolSnap Programm zu betreiben. Clocking-Modus sollte auf normal eingestellt werden.
- Starten Sie den DMD-Software und-ort der Dunkelfeld-Iris auf der Skript-Menü, gefolgt von den "Laden und Reset"-Befehl und führen Sie das Skript.
- Senden Sie das Licht auf den DMD-und Cascade 512 Kamera, indem Sie das Mikroskop Optovar und Ansichtsfenster, um LSM. Dadurch wird das Bild durch die DMD und die ausgerichteten Optik zu senden, die Projektion der DF Bild auf den CCD. Das Dunkelfeld (DF) sollte auf dem Live-Vorschau bereits im Gange erscheinen in IPLab. Stellen Sie die Fokussierung des Mikroskops, wenn notwendig, um das Bild auf dem Live-Vorschau zu konzentrieren.
- Machen Sie einen Schnappschuss von dem Gebiet der Ansicht mit dem "Erwerb von einzelnen"-Befehl. Stellen Sie die Belichtungszeit hoch genug, um mindestens 10000 Grafen von Signal im Bild zu gewährleisten. Nach dem Erwerb, verwenden Sie die "Speichern als indizierte"-Befehl, um das Bild auf der Festplatte zu speichern. Dieses Bild des Rasters misst die Größe des Field of View (FOV).
- Bewegen Sie nun das Fadenkreuz Probe, so dass das Fadenkreuz über den FOV ist, so dass nur Hintergrund sichtbar ist. Erwerben Sie ein Hintergrundbild des Feldes zu einemlange genug, Belichtungszeit, um sicherzustellen, dass mindestens 5000 Digits des Signals erfasst wird. Dieses Bild wird im Hintergrund Subtraktion des ungefilterten Bilder zu unterstützen.
3. Lädt die Filterbank und mit dem Setup zu filternden-Hintergrund-Bilder erwerben
- Jetzt müssen wir Gabor-gefilterte Bilder der Hintergrund zu erwerben. Laden Sie die Gabor-Filterbank Skript, um die DMD Control Software. Führen Sie das gesamte Skript, um die Filter, um die Onboard-Speicher des DMD-Puffer; könnte dies einige Minuten dauern.
- Sobald das gesamte Skript wird gepuffert, können wir jetzt zu erwerben gefilterte Bilder der Hintergrund. Verwenden Sie die Start-und Stop Marker innerhalb des DMD-Software, die DMD anweisen, nur eine Reihe von Filtern entsprechend einer Gabor-like-Filter in einer Zeit, zu laden, und führen Sie das Skript. Die Live-Vorschau sollten aus dunklen Feld, um das gefilterte Bild für diesen Filter zu ändern.
- Öffnen Sie den Erwerb script in IPLab von der Festplatte. Stellen Sie die Belichtungszeit zu gewährleisten, dass mindestens 2000 zählt der Signal erworben werden. Da die DMD-Skript ausgeführt wird, brechen Sie die Live-Vorschau in IPLab und führen den Erwerb Skript. Dadurch wird automatisch erwerben, zu indizieren und speichern Sie die gefilterten Bild auf der Festplatte.
- Sobald das erste Bild aufgenommen wird, stoppen Sie das Skript läuft in der DMD-Software und löschen Sie die verwendete Befehle aus dem Skript. Ersetzen Sie die Start-und Stop Marker am Anfang und am Ende des nächsten Filters eingestellt. Wiederholen Sie die Übernahme in IPLab.
- Wiederholen Sie Schritt 3.4, bis die gesamte Filterbank verwendet worden ist und alle gefilterten Bilder wurden erfasst und gespeichert.
4. Ausplattieren der Zellen
- Inzwischen werden die Zellen bald bereit sein, Platte für das Experiment. Stecken Sie den Lötkolben an das Labor Labortisch. Entfernen Sie die L15 Betrachtungsmedium und Wärme bis 37 ° C. Machen Sie einen Arbeitsplatz mit einem Papiertuch und einem Kimwipe. Machen Sie mehrere Dochte durch Reißen und zwirbelte Kimwipes. Die Dochte werden bei der Übertragung von Flüssigkeit und von der Zelle Platte zu helfen.
- Danach müssen wir auf den Teller der Probe. Wir verwenden das bearbeitete Metall Probenhalter auf den Teller unserer Zellen, was eine "Deckglas Sandwich" mit der Metallplatte dazwischen. Tragen Sie eine dünne Wulst Vakuumfett aus einer Spritze um den oberen Rand der Metallplatte Loch Ausweitung etwa auf halbem Weg zu den Enden der Rillen auf jeder Seite. Drücken Sie vorsichtig einen sauberen nicht. 1 Deckglas auf das Fett. Drehen Sie die Platte über und Fett um das Loch. Schalten Sie die Raumbeleuchtung.
- Jetzt bekommen wir die Zellen aus dem Inkubator, die Handhabung des Inkubators Inhalte mit Nitril-Untersuchungshandschuhe mit 70% Ethanol sterilisiert. Entfernen Sie die Zelle Platte aus dem Inkubator, den Atem anzuhalten, während der Inkubator Tür ist offen. Seien Sie vorsichtig, um die Exposition gegenüber Raumlicht zu minimieren.
- Entfernen Sie das Deckglas, das für das Experiment aus den Sechs-Well-Platte verwendet werden soll, stellt fest, dass die Seite, die offen in der Brunnen war die Seite mit den Zellen verbunden ist. Vorsichtig trocken das Deckglas auf beiden Seiten, bis es fast vollständig trocken ist, während die Verfolgung von welcher Seite der Deckglas hat der Zellen. Dann drücken Sie die Deckglas-, Zell-Seite nach unten in die gefettete Blech über dem Guckloch, um sicherzustellen, dass keine Luft Lücken innerhalb der Fettschicht bleiben. Das Fett muss bilden eine wasserdichte Abdichtung, damit für uns die Zellen mit dem L15-Medium zu laden. Sobald Sie sicher sind, dafür sind, drehen Sie die Platte wieder um.
- Pipettieren Sie die L15 Medium in der plattierten Zellen, indem sie den flüssigen durch die Nut zwischen dem oberen Deckglas und der Metallplatte. Pipettieren 200 ul in einer Zeit gut funktioniert. Der erste Band Pipette füllen den Raum zwischen den Deckgläschen mit der Flüssigkeit sich fast in die Nut auf der anderen Seite angeordnet.
- Pipette weitere 200 ul Medium in der plattierten Zellen, aber dieses Mal, halten einen Docht an der gegen Hain, so dass das Medium von einer Seite zur anderen fließt. Dies spült die Zellen und entfernt alle Spuren des alten Mediums. Achten Sie darauf, keine Blasenbildung in der Flüssigkeit bei diesem Schritt zu verhindern. Wiederholen Sie diesen Vorgang 2-3 mal mit einem neuen Docht für jeden spülen.
- Denken Sie daran, den Lötkolben wir eingesteckt? Jetzt ist die Zeit daran gewöhnt. Drehen Sie die Platte auf den Kopf noch einmal die Unterstützung der Platte von den Rändern, so dass Flüssigkeit in die Zelle Reservoir gefangen und kann nicht abtropfen. Tauchen Sie den Lötkolben in die valap Becher. Dies wird schnell schmelzen einige der valap die dann an der Lötspitze wird klammern. Tragen Sie die geschmolzene valap um die Ränder des unteren Deckglas (die jetzt nach oben) mit der Lötspitze als Applikator. Setzen Sie das Eintauchen und die Anwendung bis ein Sie fahren den ganzen Weg um das Deckglas Umfang, Abdichtung des Deckglases auf die Metallplatte.
- Die untere Deckglas hat wachsenden Zellen auf, und können Rückstände aus den getrockneten bis mittelfristig auf die offene Seite zu haben. Reinigen Sie alle Rückstände aus dem Deckglas Oberfläche durch balling eine Kimwipe und cleaning das Deckglas in einem einzigen gleitenden Bewegung ähnlich wie die Reinigung der Zielsetzung. Dies gewährleistet, dass das Deckglas saubersten in der Mitte, wo es angezeigt werden wird.
- Ziehen Sie den Lötkolben und geben die 6-Well-Platte in den Inkubator unter Beachtung der gleichen Containment und Sterilität Verfahren. Nehmen Sie die plattierten Zellen der Optik-Labor und montieren auf das Ziel wie in den Schritten 2.4 und 2.5 beschrieben.
5. Die Durchführung des Experiments
- Suchen Sie ein schönes Feld der gesund aussehende Zellen.
- Erwerben Sie ein dunkles Feld Bild des Sichtfeldes. Richten Sie das Mikroskop in Differential-Interferenz-Kontrast (DIC) und erwerben ein DIC Bild. Stellen Sie sicher, dass die Belichtungszeiten lang genug, um sicherzustellen, dass das Signal ist ausreichend sind.
- Nun müssen wir das fluoreszierende Bilder auf der anderen Kamera zu erwerben. Um DIC Bilder auf der CoolSnap, verwenden wir eine blaue LED an den Kondensator angeschlossen, ersetzt sie und entfernen sie als notwendig. Während das Mikroskop ist immer noch in DIC ausgerichtet, senden das Licht auf die CoolSnap, indem Sie das Mikroskop Optovar zu 1.0x und Ansichtsfenster zu 100% Fernglas. Umleiten das Bild vom Okular zur Kamera. Setzen Sie die LED über den Kondensator auf das Feld zu beleuchten und eine Vorschau des FOV in RSimage und passen Feinfokussierung, wenn nötig. Erwerben Sie eine DIC Bild und auf der Festplatte speichern. Beachten Sie, wie die FOV unterscheidet sich von dem von der Cascade-Kamera gewonnen wird. Diese Bilder werden während der Analyse-Phase nach dem Experiment registriert werden.
- Besorgen Sie sich ein Fluoreszenz-Bild durch die Anpassung der Filter-Würfel, die Fluorescein FILTERCUBE. Erwerben Sie ein Bild durch kurzes Einschalten der Fluoreszenzanregung mit dem Mikroskop und dann schalten Sie ihn aus, sobald die Übernahme abgeschlossen ist. Da wir die Probe fokussiert in DIC wird die Fluoreszenz Bild fokussiert sowie. Das spart Belichtungszeit in der Fluoreszenz und damit Verlangsamung Ausbleichen. Speichern Sie die Fluoreszenz image to disk.
- Nun müssen wir die gefilterten Bilder zu erwerben. Setzen Sie das Mikroskop Dunkelfeld und senden das Licht durch die LSM-Port wie in 2.9.
- Führen Sie das gesamte Gabor Filterbank Skript als in 3,3-3,5. Wir haben nun die Datenerfassung für einen Zeitpunkt abgeschlossen.
6. Switching das Medium, um die Zellen zu Staurosporin (STS) setzen, und die Aufrechterhaltung des Mediums während des gesamten Experiments
- Während die Zellen noch auf der Bühne und ohne Störung des Sichtfeldes, schalten Sie die regelmäßigen L-15-Medium für das gleiche mit einer 1 nM Lösung von STS aus einer 4 mM Stammlösung von STS in DMSO hergestellt. Verwenden Sie den Docht in den Schritten 4.6 beschrieben, die Medien wechseln.
- Jetzt wiederholen wir die Schritte 5,2-5,6 für spätere Zeitpunkte. Wir wiederholen diesen Vorgang, bis das Experiment abgeschlossen ist.
- Im Laufe des Experiments werden mehr Medium hinzugefügt, so dass die Probe nicht austrocknen tut. Dies ist durch Pipettieren Medium in den Hain der Zellplatte ohne Entfernen von der Bühne und ohne Störung des FOV durchgeführt.
7. Repräsentative Ergebnisse
Am Ende des Experiments werden die erhobenen Daten umfassen eine große Anzahl von gefilterten Bilder, die verarbeitet werden, um die subzelluläre strukturelle Daten zu extrahieren werden müssen. Zwei Beispiele sind für einen optischen Filter Bank, bestehend aus 9 Gabor-wie Filter mit Filter Zeitraum S = 0.95μm, Gauß-Umschlag Standardabweichung s = S / 2 = 0.45, und Orientierungen Φ = 0 ° bis Φ = 160 ° in 20 gezeigt, °-Schritten. (Siehe auch [1] für weitere Details).
Beispiel 1: Marine Diatomee
Wir erstmaligen Anwendung unserer Orientierung empfindlichen Filter Bank zu einer marinen Kieselalgen Probe (Carolina Biological Supply Company) mit orientierten Features, die deutlich sichtbar in Dunkelfeld-(DF)-Bildgebung (Abb. 1). Die optisch gefiltert Bilder sind neben dem ungefilterten Bild der Probe zum Vergleich gezeigt.

Abbildung 1: Dunkelfeld (DF) und optisch gefilterte Bild der marinen Kieselalgen. Wir werden die Kieselalgen in der unteren rechten Ecke des Bilds (weißer Pfeil in der linken die meisten Panel) zu analysieren.
Der Satz von neun Gabor-gefilterte Bilder der Kieselalge verarbeitet wurden Pixel-by-Pixel für Objektorientierung und Rundheit. Verarbeitung bestand aus (1) Zusammenfassung gemessenen Reaktionen aller neun Gabor-gefilterte Bilder bei jedem Pixel die gesamte Größe des Signals Reaktion dadurch Kodierung Reaktion Bedeutung zu bestimmen, und (2) Suche nach der Gabor-Filter Orientierung, Φ, bei denen die Antwort maximiert und unter das Verhältnis dieser maximale Reaktion auf die durchschnittliche Antwortzeit für alle Winkel so kodiert, inwieweit die Objekte an jedem Pixel eine bevorzugte Richtung. Der Grad der Orientierung ist eng mit der geometrischen Seitenverhältnis des Partikels. In Abb.. 2B, the Gesamtansprechrate der Pixel, um den Filter Bank (Parameter 1) und der Grad der Orientierung oder Seitenverhältnis (Parameter 2) sind in der Farbsättigung und Farbton kodiert bzw.. Ein Seitenverhältnis nahe 1 (blau) ist in Bereichen, in denen es keine bevorzugte Reaktion Winkel, während größere Werte (rot), in welchen Bereichen eine höhere bevorzugten Winkel Reaktion vorhanden ist anzugeben. Unterbau Partikel Orientierung ist in einem Köcher Plot (Abb. 2C), wobei jede Linie eng mit den zugrunde liegenden lokalen Objektorientierung sichtbar in ungefilterten Dunkelfeld-(Abb. 2A) vereinbart kodiert.

Abbildung 2: A: Dunkelfeld Bild von Kieselalgen. B: Objektorientierung Bild. Farbskala zeigt Grad der Orientierung (aspect ratio), während Helligkeit kodiert Bedeutung der insgesamt Gabor-Filters. C: Orientierung von Objekten mit Antwort Intensität ≥ 10% der maximalen. Line-Segment zeigt die entsprechende Struktur der langen Achse.
Beispiel 2: apoptotischen Zellen
Hier zeigen wir gefilterte Bilder von Rinder-Endothelzellen mit Staurosporin (STS), die in der gleichen Weise wie die Kieselalge verarbeitet wurden behandelt. Abb.. 3 zeigt eine ungefilterte Dunkelfeld-(DF) Bild der Zellen zusammen mit den neun gefilterte Bilder zum Zeitpunkt T =- 180 min. vor STS Behandlung.
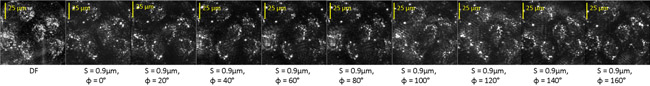
Abbildung 3: Dunkelfeld (DF) und optisch gefiltert Bilder von einem Feld mit mehreren lebenden Endothelzellen.
Die gefilterte Bilder wurden anschließend alle 20 Minuten für einen Zeitraum von drei Stunden nach STS Behandlung erworben. Abb.. 4a zeigt ein Seitenverhältnis Karte der Zellen als eine Funktion der Zeit. In diesem Fall wird der Farbton entspricht dem Grad der Orientierung (beschriftet Orientiertheit) wie für den Farbton in Abb.. 2b oben. Allerdings war das Seitenverhältnis Helligkeit nicht von der durchschnittlichen, gewichteten Filter. Durch die Registrierung unserer Seitenverhältnis Karten mit Fluoreszenz-Aufnahmen der markierten Mitochondrien in diesen Zellen (Abb. 4b) haben wir festgestellt, dass die gemessene Seitenverhältnis Drop auf die zellulären Regionen mit Mitochondrien beschränkt war und war gleichzeitig mit der mitochondrialen Fragmentierung, die direkt konnte beobachtet werden, die Fluoreszenz-Aufnahmen von den gleichen Zellen. Abb.. 5 zeigt Zeitdiagramme der Darstellung der Veränderung des Seitenverhältnisses als Funktion der Zeit in den Zellen eine Apoptose. Innerhalb jeder Zelle gibt es einen Rückgang in Seitenverhältnis bei T = 60-100 min in den Regionen, die mit fluoreszierenden Mitochondrien registrieren, aber nicht in Regionen, die mit dem dim Hintergrundfluoreszenz Bereichen zu registrieren.
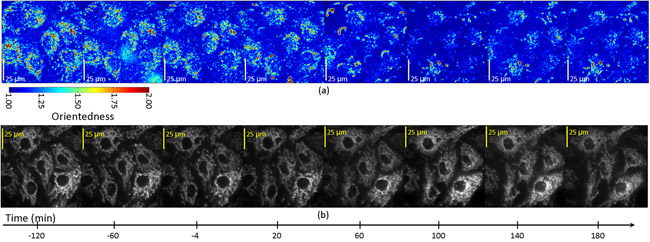
Abbildung 4: Das Seitenverhältnis (a) und Fluoreszenz (b) Bilder von Endothelzellen mit dem Apoptose-Induktor, Staurosporin behandelt.
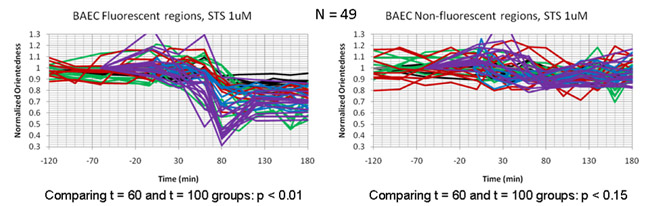
Abbildung 5: Time Grundstücke Vergleich der Abnahme der Teilchen-Seitenverhältnis (Orientiertheit) in Endothelzellen mit Staurosporin behandelt. Die einzelnen Spuren zu repräsentieren Zeit Parzellen innerhalb einzelner Zellen. Der Rückgang der Orientiertheit ist es, die Regionen der Zellen, die mit fluoreszierenden Mitochondrien (links) registrieren beschränkt und fehlt im übrigen Hintergrundfluoreszenz Regionen (rechts).
Jetzt, da wir festgestellt haben, dass das Seitenverhältnis Drop mitochondriale Fragmentierung entspricht, können wir die Apoptose in diesen Zellen, messen Sie die Fragmentierung der Nutzung unserer optischer Streuung Methode, ohne die Zellen zu markieren, und studieren die Wirkung von verschiedenen genetischen und experimentellen Bedingungen auf dieser Dynamik.
Diskussion
Das oben beschriebene Verfahren liefert morphometrische Karten des Objekts, das Partikel-Größe oder Ausrichtung zum Beispiel codieren kann. Diese strukturelle Information kann auf verschiedene Arten genutzt werden:
- Es kann als Einstieg, um Gewebe oder Zellen Regionen, die während einer bestimmten Behandlung verändert werden und dann weiter zu analysieren diesen Regionen mit spezifischen molekularen und biochemischen Assays zu identifizieren verwendet werden.
- Es kann in Verbindung mit Fluoreszenz verwendet werden, um eine multimodale Analyse der Zellen, die eine gleichzeitige Verständnis der molekularen Aktivität (durch Fluoreszenz) kombiniert mit quantitativen Charakterisierung der subzellulären Struktur (mit dieser Methode) ergeben.
- Sobald eine bestimmte strukturelle Antwort mit einem bestimmten Organellen Aktivität in einem bestimmten biologischen Prozess (z. B. Apoptose) korreliert ist, kann die Methode ohne Fluoreszenz-Labels zum Screening auf verschiedene genetische und experimentelle Bedingungen, die diese strukturelle Antwort verwendet werden.
Bis heute haben wir gezeigt, dass die Methode empfindlich auf Unterschiede in der Partikelgröße in der Größenordnung von 30-50nm [2] ist. Wir haben gezeigt, dass die Methode empfindlich auf Veränderungen in der Teilchenphysik Orientierung und Seitenverhältnis ist [3] und zu einer Abnahme der Partikelgröße Seitenverhältnis im Einklang mit der Apoptose-induzierten mitochondrialen Fragmentierung (Abb. 4-5 oben).
Die Ergebnisse von Beispiel 2 lassen vermuten, dass unsere Methode dynamische Messungen der zellulären Funktion, die in Bezug auf spezifische Organellen Funktion interpretiert werden können und die auch ohne Fluoreszenz-Labels oder exogene Farbstoffe gesammelt werden lässt. Allerdings war zunächst die Validierung der gemessenen Reaktionen gegen spezifisch markierten Organellen notwendig. Nach dieser ersten Prüfung abgeschlossen ist, kann Organellen Dynamik direkt sondiert mit unserem Label-freie Methode.
Die Anwendung der Methode auf mehrere zelluläre Bedingungen und Korrelation unserer dynamischen Strukturwandel Messung mit der Lage der verschiedenen Organellen innerhalb der Zelle, wie wir er mit Mitochondrien, kann letztlich zu einer Bibliothek von dynamischen Strukturwandel Verhaltensweisen, die eindeutig charakterisieren können spezifische zelluläre Zustände führen ( z. B. Apoptose, oxidative und metabolischen Stress, Entzündungsreaktionen etc.) .. Diese Informationen könnten weiter in eine "Zelle Zustand Analyzer", die in einer Vielzahl von Anwendungen, einschließlich der Wirkstoffforschung in klinischen Zellanalyse verwendet werden können eingebunden werden.
Es ist wichtig zu beachten, dass die Methode hier skizzierte einen allgemeinen Ansatz, bei denen optische Streuung von Daten in eine mikroskopische Imaging-System erworben benutzt, um bestimmte subzelluläre Dynamik Extrakt kann darstellt. Allerdings kann die spezielle Instrumentierung signifikant verbessert werden. Insbesondere kann die aktuelle Auswahl von Spatial Light Modulator für räumliche Filterung nicht optimal sein, um die Effizienz der Datenerfassung, Ortsfrequenz Auflösung und optisches Signal Durchsatz zu maximieren. Chromatische und geometrischen Aberrationen mit der digitalen Mikrospiegel-Gerät verwendet hier verknüpft sind in [3] diskutiert. Wir untersuchen derzeit die potenziellen Vorteile und Nachteile einer state-of-the-art Flüssigkristallanzeige statt der DMD, um diese Probleme zu mildern. Darüber hinaus wird das Gerät für die Datenerfassung, sind räumliche Filterung und Mikroskop-Steuerung getrennt durch den menschlichen Benutzer betätigt. Dies schränkt Akquisition Zeit, wo eine große Anzahl von gefilterten Bilder müssen vor der Verarbeitung gesammelt werden. So ist die Automatisierung des Setup-Gebot, um die Messzeit zu der Dauer der CCD Exposition, die voraussichtlich bis 10 ist von Millisekunden pro gefilterte Bild für eine ausreichende Signal-Rausch zu erreichen ist. Dieser Anstieg der zeitlichen Auflösung wird uns auch ermöglichen, genauer Charakterisierung der strukturellen Dynamik von Organellen in lebenden Zellen. Wir sind daher aktiv an der Entwicklung eines maßgeschneiderten grafischen Benutzeroberfläche, die die Kontrolle über die Hardware zu vereinheitlichen und zu rationalisieren kann die Betätigung seiner Komponenten, einschließlich Mikroskop kontrollieren, CCD Bildaufnahme und optischen Filtern an der Spatial Light Modulator.
Offenlegungen
Danksagungen
Die Mikrospiegel-Gerät in dieser Untersuchung wurde von Whitaker Foundation Grant RG-02 bis 0682 zu N. Boustany finanziert. Laufende Arbeiten von zu gewähren NSF-DBI-0852857 bis N. Boustany finanziert. RM Pasternack wurde teilweise durch eine Rutgers Presidential Graduate Fellowship unterstützt. Wir möchten auch Dr. E. Weiß für die iBMK Zellen in unseren Studien und Dr. DN Metaxas für nützliche Diskussion über optische Filterung Strategien danken.
Materialien
| Name | Company | Catalog Number | Comments |
| DMEM | Invitrogen | Low glucose DMEM | |
| Liebowitz L15 medium | Invitrogen | Without phenol red | |
| L-glutamine | Invitrogen | ||
| Mitotracker Green | Invitrogen | ||
| Bovine Brain Extract | Clonetics | ||
| Fetal Bovine Serum | Gemini Bio Products | ||
| Heparin | Sigma-Aldrich | ||
| Staurosporine | Sigma-Aldrich | ||
| Dymethylsulfoxide | Sigma-Aldrich | ||
| Inverted microscope | Carl Zeiss, Inc. | Axiovert 200M | |
| DMD | Texas Instruments | TI 0.7 XGA DMD 1100 | |
| CCD | Roper Scientific | Cascase 512B | High (16 bit) dynamic range CCD |
| CCD | Roper Scientific | Coolsnap cf |
Referenzen
- Pasternack, R. M., Qian, Z., Zheng, J. -Y., Metaxas, D. N., White, E., Boustany, N. N. Measurement of Subcellular Texture by Optical Gabor-Like Filtering with a Digitial Micromirror Device. Optics Letters. 33 (19), 2209-2211 (2008).
- Pasternack, R. M., Qian, Z., Zheng, J. -Y., Metaxas, D. N., Boustany, N. N. Highly sensitive size discrimination of submicron objects using optical Fourier filtering based on two-dimensional Gabor filters. Optics Express. 17 (14), 12001-12012 (2009).
- Zheng, J. -Y., Pasternack, R. M., Boustany, N. N. Optical scatter imaging with a digital micromirror device. Optics Express. 17 (22), 20401-20414 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten