Method Article
演示序列比对以预测跨物种敏感性的工具,用于快速评估蛋白质保存
摘要
在这里,我们提出了一个协议,以利用最新版本的美国环境保护署序列比对来预测跨物种敏感性(SeqAPASS)工具。该协议演示了在线工具的应用,以快速分析蛋白质保存,并提供可定制且易于解释的跨物种化学敏感性预测。
摘要
美国环境保护署跨物种敏感性预测序列比对(SeqAPASS)工具是一种快速,免费提供的在线筛选应用程序,允许研究人员和监管机构推断跨物种的毒性信息。对于模型系统中的生物靶标,如人类细胞、小鼠、大鼠和斑马鱼,可以使用各种化学物质的毒性数据。通过评估蛋白质靶标保守性,该工具可用于将从此类模型系统生成的数据外推到数千种缺乏毒性数据的其他物种,从而预测相对内在化学敏感性。该工具的最新版本(版本 2.0-6.1)包含新功能,允许快速合成、解释和使用数据以进行发布以及演示质量的图形。
这些功能包括可定制的数据可视化和全面的摘要报告,旨在汇总SeqAPASS数据,以便于解释。本文描述了指导用户提交作业、浏览各种级别的蛋白质序列比较以及解释和显示结果数据的协议。重点介绍了 SeqAPASS v2.0-6.0 的新功能。此外,还描述了使用此工具专注于转甲状腺素蛋白和阿片受体蛋白质保存的两个用例。最后,讨论了SeqAPASS的优势和局限性,以定义该工具的适用范围,并强调跨物种外推的不同应用。
引言
传统上,毒理学领域严重依赖使用全动物测试来提供化学安全评估所需的数据。此类方法通常成本高昂且需要大量资源。然而,由于目前使用的化学品数量众多,新化学品的开发速度很快,全球范围内都认识到需要更有效的化学品筛选方法1,2。这种需求以及由此产生的从动物试验的范式转变导致了许多新方法的开发,包括高通量筛选测定、高通量转录组学、二代测序和计算建模,这些都是有前途的替代测试策略3,4。
评估可能受化学品暴露影响的物种多样性的化学安全性一直是一个持久的挑战,不仅要进行传统的毒性测试,还要使用新的方法。比较和预测毒理学的进步为理解不同物种的相对敏感性提供了框架,计算方法的技术进步继续提高这些方法的适用性。在过去的十年中,已经讨论了几种策略,这些策略利用现有的基因和蛋白质序列数据库以及特定化学分子靶点的知识,以支持跨物种外推的预测方法,并增强典型模式生物以外的化学安全性评估5,6,7,8。
为了将科学转化为行动,在这些预测毒理学基础研究的基础上,优先考虑化学测试工作并支持决策,创建了美国环境保护署序列比对以预测跨物种敏感性(SeqAPASS)工具。该工具是一个公共且免费提供的基于Web的应用程序,它使用不断扩展的蛋白质序列信息的公共存储库来预测物种多样性的化学敏感性9。基于可以通过评估该化学品的已知蛋白质靶标的保守性来确定物种对特定化学物质的相对内在敏感性的原理,该工具可快速比较具有已知敏感性的物种的蛋白质氨基酸序列与具有现有蛋白质序列数据的所有物种。该评估通过三个级别的分析完成,包括(1)伯爵氨基酸序列,(2)功能结构域和(3)关键氨基酸残基比较,每个级别都需要更深入地了解化学 - 蛋白质相互作用,并在敏感性预测中提供更高的分类分辨率。SeqAPASS的一个主要优势是,用户可以根据感兴趣的化学-蛋白质或蛋白质-蛋白质相互作用的可用信息量,通过添加额外的证据线来定制和完善他们的评估。
第一个版本于2016年发布,允许用户以简化的方式评估伯代氨基酸序列和功能结构域,以预测化学敏感性,并且包含最少的数据可视化功能(表1)。个体氨基酸差异已被证明是化学 - 蛋白质相互作用跨物种差异的重要决定因素,这会影响物种的化学敏感性10,11,12。因此,开发了后续版本以考虑对直接化学相互作用很重要的关键氨基酸13。为了响应利益相关者和用户的反馈,该工具每年发布一次版本,增加了新功能,旨在满足研究人员和监管界应对跨物种外推挑战的需求(表1)。2020 年推出的 SeqAPASS 5.0 版本带来了以用户为中心的功能,这些功能包括数据可视化和数据合成选项、外部链接、汇总表和报告选项以及图形功能。总体而言,该版本的新属性和功能改进了数据合成、外部数据库之间的互操作性以及预测跨物种敏感性的数据解释的便利性。
研究方案
1. 入门
注意:此处介绍的协议侧重于工具实用程序和关键功能。有关方法、特性和组件的详细说明,请参见网站上的综合用户指南(表1)。
表 1:SeqAPASS 工具的演变。 从初始部署添加到 SeqAPASS 工具的功能和更新列表。缩写:seqAPASS = 序列比对以预测跨物种敏感性;ECOTOX = 生态毒素学知识库。 请按此下载此表格。
- 转到使用 https://seqapass.epa.gov/seqapass 浏览器。选择"登录"以使用现有帐户,或按照说明创建 SeqAPASS 帐户,这将允许用户运行、存储、访问和自定义其已完成的作业。
- 在进行分析之前,首先通过查看现有文献或现有数据来确定感兴趣的蛋白质以及目标或敏感物种(图1)。由于 SeqAPASS 包含指向外部资源的链接以帮助识别查询蛋白质,请单击"识别蛋白质靶标"下的下拉按钮以访问相关资源。

图 1:SeqAPASS 问题表述:成功分析所需的初步信息的示意图。缩写:seqAPASS = 序列比对以预测跨物种敏感性;LBD = 配体结合结构域。请点击此处查看此图的大图。
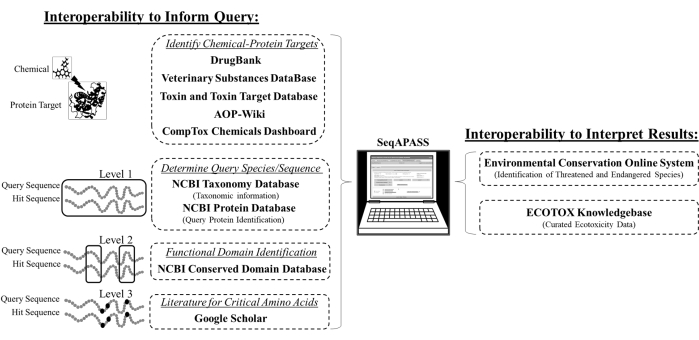
图 2:跨数据库的 SeqAPASS 互操作性。 集成到SeqAPASS中的外部工具,数据库和资源的示意图。缩写:seqAPASS = 序列比对以预测跨物种敏感性;AOP = 不良结局途径;NCBI = 国家生物技术信息中心;ECOTOX = 生态毒素学知识库。 请点击此处查看此图的大图。
表 2:集成到 SeqAPASS 工具中的链接、资源和工具。 SeqAPASS 工具中利用的各种数据源、链接和资源的列表。缩写:seqAPASS = 序列比对,用于预测跨物种的敏感性。 请按此下载此表格。
2. 开发和运行 SeqAPASS 查询:级别 1
注意:在 1 级分析中,将查询蛋白的整个主要氨基酸序列与具有可用序列信息的所有物种的主要氨基酸序列进行比较。该工具使用算法来挖掘、收集和编译公开可用的数据,以快速对齐和比较跨物种的氨基酸序列。后端存储来自国家生物技术信息中心(NCBI)数据库的信息,并战略性地利用蛋白质基本局部比对搜索工具(BLASTp)54 和基于约束的多重比对工具(COBALT)55的独立版本。
- 在" 比较初级氨基酸序列"下,单击 "按物种" 或 "按种质"。使用" 按物种" 选项键入或从物种列表中选择感兴趣的蛋白质靶标。
- 通过在"按加入"文本框中输入种质,直接提交蛋白质种质(即NCBI蛋白质ID)。
- 选择" 请求运行 "以提交查询。提交后,等待浏览器窗口右上角出现指示提交成功的通知。
- 选择页面顶部的"SeqAPASS 运行状态"选项卡,以显示在该用户帐户下执行的所有 SeqAPASS 运行 的列表,并检查完成百分比。
- 单击 刷新数据 ,同时选择相应的单选按钮以检查级别 2 和级别 3 运行的状态。
- 选择页面顶部的" 查看 SeqAPASS 报告"选项卡以访问在该帐户下完成的所有报告的列表。
- 在 "查看 SeqAPASS 报告 "选项卡中,选择感兴趣的查询蛋白。单击 "请求所选 报告"以打开"1 级查询蛋白质信息"页面并查看结果、数据自定义选项、可视化和摘要报告。
- 默认情况下,选择" 查看报表 "以在 Web 浏览器中查看数据。或者,选择 "保存报表 "以将原始数据下载为.zip文件。
注意:1 级分析所需的时间会有所不同(版本 5.1 的平均时间为 23 分钟),具体取决于该时间点的全球用户需求、提交到队列的作业数以及提交的作业存在的蛋白质信息量。如果之前已经完成了蛋白质靶标,则数据将在提交后的几秒钟内可用。
3. 开发和运行 SeqAPASS 查询:级别 2
注意:由于整个蛋白质序列不直接参与化学相互作用,因此2级分析仅比较功能域的氨基酸序列,以在较低的分类学等级(例如,类,目,科)下进行敏感性预测。
- 在" 1 级查询蛋白质信息" 页中,单击 2 级标题旁边的加号 + 以填充" 2 级 查询"菜单。
- 确定目标蛋白质(查询蛋白质)中的适当结构域。
- 如果尚未识别域,请单击指向NCBI保守域数据库(CDD)(表1)的集成链接,该链接有助于识别适当的域选择。
注意:通常,在级别 2 中仅选择特定的命中域作为查询。
- 如果尚未识别域,请单击指向NCBI保守域数据库(CDD)(表1)的集成链接,该链接有助于识别适当的域选择。
- 单击" 选择结构域" 框以自动填充查询蛋白的功能域列表。
- 从下拉列表中选择域 加入 ,然后单击" 请求域运行 "按钮启动级别 2 查询。提交后,等待出现指示提交成功的通知。
- 单击"刷新级别 2"和"级别 3"运行以填充 级别 2 数据,这些数据将在提交后的几秒钟内可用。
- 在"查看二级数据"下,从下拉列表中选择已完成的域加入,然后单击"查看二级数据"按钮以在新页面中打开结果。
4. 访问和理解数据:SeqAPASS 级别 1 和级别 2
- 滚动到 "查询蛋白质信息 "页面的底部以查看结果报告 - 默认情况下, 主要报告 包含 1 级和 2 级分析。选择完整报告单选按钮以查看提供所有序列命中和对齐指标的更详细 报告 。单击两份报告中相应的 种质/ID/名称 ,以访问NCBI数据库中的透明蛋白质比对和分类信息。
- 滚动到结果表的右侧以查看 ECOTOX 列。单击生态毒素学知识库 (ECOTOX) 的链接 ,快速收集具有易感性预测的物种的相应毒性数据。
注意:ECOTOX是一个全面的,公开可用的知识库,为水生和陆地植物和野生动物提供单一的化学毒性数据。SeqAPASS v6.0 包含一个 ECOTOX 小部件,可按感兴趣的化学物质和物种更快地连接相关的 ECOTOX 数据。 - 单击 下载 表格将表格另存为电子表格文件。单击"查看摘要报告"按钮可查看和下载按分类组排序的数据的 摘要报告 表。
注意:数据摘要表可用于主要报告和完整报告,并提供给定目标的预测概述。
5. 操作数据设置:SeqAPASS 级别 1 和级别 2
注意:在1级和2级分析中,假设蛋白质相似性越大,化学物质以与查询物种/蛋白质类似的方式与蛋白质相互作用的可能性就越大,使它们容易受到具有该分子靶标的化学物质的潜在影响。由于这些数据的相似性,理解1级和2级数据的步骤一起概述在一个协议中。
- 请参阅 查询蛋白质信息 顶部的子菜单,以访问和操作报告设置,并对大多数分析的所有报告选项使用默认设置。如果有科学理由更改默认设置,请按照以下可选步骤操作:
- (可选)单击"敏感性截止值"旁边的加号 +,在新选项卡中查看和调整敏感性截止设置。从下拉列表中选择新的截止值,或输入用户定义的截止值。
- (可选)如果需要默认值以外的其他内容,请更改 E 值 (预计会偶然发生的不同对齐方式的数量)字段中的数字。
注意:任何E值大于框中数字的蛋白质都将从主要报告中消除。 - (可选)使用" 按 分类组排序"选项选择要在结果表的 "筛选的分类组" 列中显示的分类层次结构级别。
注意:更改分类层次结构还将根据在截止值上方找到的每个过滤组中的物种来改变易感性的预测。 - (可选)如果需要默认以外的其他内容,请更改"公共结构域"字段(蛋白质必须与查询蛋白质共享多少个 公共结构 域才能包含在结果中)。
注意:由于默认设置为 1,因此将排除与查询蛋白不共享至少一个公共结构域的任何序列。 - (可选)仅当相似百分比大于或等于临界值或命中被标识为正交同源物候选项时,才在物种读取下选择否以返回 Y 的敏感性预测。
注意:此设置默认为"是",这意味着将报告所有直系同源候选物种、药敏率临界值上方列出的所有物种以及同一分类组低于临界值的所有物种的敏感性预测 Y,其中一个或多个物种高于临界值。
- 单击"下载 当前报告设置 "按钮以下载捕获当前应用设置的文件。
注意:所选的特定评估级别(1、2 或 3)将决定报告中显示的设置。
6. 可视化数据:序列APASS 级别 1 和级别 2
- 单击可视化旁边的加号 +,然后单击可视化数据按钮以打开一个单独的选项卡,其中显示用户定义的信息以及用于选择结果交互式图形的选项。
- 单击箱线图以打开交互式箱线图和绘图控件,并允许 箱线图 可视化效果主动更新以反映对数据表的更改并提供具有出版和演示质量的图形。
注意: 默认箱线图在 x 轴上显示物种组,在 y 轴上显示相似百分比。箱线图显示易感性临界值(虚线)、与查询物种相比,物种间相似度百分比、每个分类组的平均值和中位数以及第 25 和第 75 个百分位数和四分位距。根据分析目标和用户需求,可以通过以下可选步骤修改许多箱线图特征。- (可选)若要自定义显示的分类组,请参阅"控件"部分下的"分类组"框。通过滚动名称并选择 x 或使用分类组下拉菜单来删除组。
- (可选)要添加图例以精确定位感兴趣的物种或特定的预定义组(例如,濒危或受威胁的物种),请将鼠标悬停在 x 轴上的 分类组名称 上以激活一个弹出框,其中列出了按最高相似百分比排序的前三个物种。将鼠标悬停在图例中的物种上可生成包含相应 物种 信息的弹出框。单击特定分类组的 框 以生成可下载的汇总表,列出物种和预测。
- 单击 下载箱线图 以选择文件类型、自定义宽度/高度分辨率并保存可视化效果。
7. 开发和运行 SeqAPASS 分析:级别 3
注意:3级分析评估查询蛋白质中用户鉴定的氨基酸残基,并快速比较不同物种这些残基的保存情况。假设这些残基保守的物种更有可能以与模板物种/蛋白质类似的方式与化学品相互作用。由于 Level 3 侧重于单个氨基酸,因此只有在对化学-蛋白质或蛋白质-蛋白质相互作用至关重要的氨基酸残基有详细的了解时,才能进行分析。
- 单击"1 级查询蛋白质信息"页面上 3 级标题旁边的加号 +,以填充"3 级查询"菜单。
- 单击参考文献浏览器旁边的加号 + 以打开参考文献浏览器工具,该工具会生成预定义的布尔字符串来查询可用的文献,并帮助用户识别适当的文献,以支持鉴定用于 3 级评估的关键氨基酸(表 2 和图 2)。
- (可选)查询蛋白质自动填充后,使用 "添加蛋白质名称 "函数添加其他蛋白质。
- 点击 生成 Google 学术 搜索链接以打开一个弹出窗口,其中包含自动生成的搜索字符串,其中包含相关搜索字词。
- 点击 搜索 Google 学术 搜索,使用搜索字符串查询文献数据库。
- 或者,单击复制到 剪贴板 ,然后通过使用 引用资源管理器中的函数添加或删除术语来自定义搜索字符串。
8. 使用鉴定的文献鉴定关键氨基酸残基
- 在"3 级查询"菜单中选择用户选择的物种将与其对齐的 模板序列 。
注意:此模板序列通常根据鉴定关键氨基酸的文献进行选择,并且可以与1级和2级中查询的物种相同或不同。- (可选)使用 "其他 比较"框可以比较" 主要/完整报告 "表中未显示的任何种质/序列。
- 在输入 3 级运行名称文本框中输入级别 3 运行的用户定义名称,以标识已完成的级别 3 运行 。为每个评估选择一个唯一的名称。
- 在"选择分类组"字段中选择感兴趣的 分类组 。选择一个分类组以按该分类组自动筛选表。
- 在结果表中,手动单击要与模板序列对齐 的任何物种旁边的复选框 。
注意:为确保适当对齐,应一次将一个分类组与模板进行比较。仅为感兴趣的物种选择类似注释的蛋白质。在选择序列进行比较时,重要的是要注意某些序列(例如, 假设的、低质量的或部分的)。除非有透明的理由,否则最好排除这些序列,因为它们可能会由于不完整或不适当的序列信息而扭曲预测。 - 重复步骤以对齐所有感兴趣的分类组。
- 在所有物种对齐后,单击刷新 级别 2 和 3 运行 ,以使用已完成的级别 3 作业填充 "选择 3 级运行名称 "菜单,并立即从级别 3 对齐获取数据。
- 单击 合并级别 3 数据 以合并来自多个分类组的对齐方式。
- 或者,要查看单个报表,请在" 选择要查看的查询" 下选择用户定义的名称,然后单击" 查看级别 3 数据"。
- 在合并 3 级报告菜单中选择要用作氨基酸残基比较基础的 3 级模板,然后单击下一步。
- 在"级别 3 作业"中,选择要比较的 已完成作业 ,然后单击 "下一步"。如果需要,使用 "顺序级别 3 作业 "功能对分类组重新排序。单击" 查看级别 3 数据 "以生成级别 3 报告页面,其中组合的分类组对齐。
- 通过在"输入氨基酸残基位置"框中键入以逗号分隔的氨基酸位置,然后选择"复制到残基列表",为模板物种选择先前标识的氨基酸位置。直接从穿梭盒中选择模板序列中的残基。
- 单击 更新报告 以刷新页面并显示 3 级敏感性预测。
注意:Level 3使用一组简单的规则,这些规则源自侧链功能特性(例如,脂肪族,芳香族)和分子尺寸(分子量差异>30 g / mol)的基本描述符,以确定关键位置的差异是否可能影响蛋白质相互作用13。
9. 可视化 3 级 SeqAPASS 数据
注意:与以前的级别一样,提供主要和完整报告。除了与 1 级和 2 级数据相同的数据外,主要报告还显示氨基酸位置、缩写以及与模板预测相似的是/否 (Y/N) 敏感性。同样,完整报告包含有关氨基酸侧链分类和分子量的信息。
- 在" 3 级报告"页上,滚动到底部以查看结果报告。单击报告底部的下载表以保存 表 。
- 单击"查看 3 级摘要报告"以查看和下载按分类组排序的数据的摘要报告表。单击"级别 3 报告"页面上"可视化"旁边的加号 +,打开一个单独的浏览器选项卡,其中显示用户定义的信息和以交互式热图形式查看结果的选项。
- 单击可视化信息页面上的热图以打开交互式图形和控件,并允许热图可视化效果主动更新以反映对数据表的更改。执行以下可选步骤以自定义热图。
- (可选)选择"报告选项"可在"简单报告"(显示氨基酸位置、一个字母缩写和氨基酸相似性)或"完整报告"(显示所选每个氨基酸的详细信息)之间切换。
- (可选)选择 "报告选项 "以更改物种的显示方式,按 通用名称 或 学名显示。
注意:在 简单报告中,氨基酸被分类为模板氨基酸的完全匹配(深蓝色)、部分匹配(浅蓝色,仅满足一个标准的替换)或不匹配(黄色,替代不符合任何标准)。 完整报告 将比较显示为"总匹配"(深蓝色)或"非匹配"(黄色)。 - (可选)选择 可选选择 以突出显示有用信息,例如直系同源候选物种、受威胁物种、濒危物种或常见模式生物。
- (可选)选择 "热图设置 "以选择其他自定义选项,包括添加或删除列、图例和文本。
- 单击 下载箱线图 以选择文件类型并保存可视化效果。
10. SeqAPASS结果的解释:蛋白质保存的证据线
注意:为了便于解释,此工具包括一个 决策摘要 报告(DS报告),旨在整合跨级别的数据。DS报告包含用户选择的结果(即数据表和/或可视化),并允许同时快速评估多个物种的多个级别的敏感性预测。
- 从结果或数据可视化页面单击将 级别 # 推送到 DS 报告,然后等待数据"推送"并且 DS 报告 选项卡变为活动状态。
注意:如果结果或任何更改尚未推送到 DS 报告,则 推送级别 # 到 DS 报告 将保持活动状态,直到选中为止。如果设置已更改,则在将更改推送到报表之前,将显示文本" 单击以推送新更改 "。在评估期间,可以随时将可视化效果推送到 DS 报告。 - 随时选择"DS 报告"选项卡以访问 DS 页面。
注意:对于在1级对齐的所有物种, 最终决策摘要报告 表包含每项分析的重要数据和敏感性预测。如果DS表中的物种未包含在3级报告中,但在1级和/或2级作业中发现,则表中的单元格将获得3级敏感性预测的不适用(NA)指定。
结果
为了演示SeqAPASS工具的应用并突出新功能,描述了两个案例研究,代表了蛋白质保守预测不同物种(人甲状腺素转运蛋白)的化学敏感性存在差异并且没有差异(μ阿片受体[MOR])的情况。这些例子中的第一个涉及蛋白质序列/结构比较,以预测不良结果途径的适用性领域(AOP,定义见 表2 ),而第二个则侧重于开发与废水中存在的阿片类药物的跨物种敏感性相关的研究假设。这些案例研究中描述的基本方法可以应用于任何化学品,并证明该工具在决策和研究方面的广泛用途。
甲状腺激素对正常的生长和发育至关重要。它们在甲状腺中合成并分泌到血液中,在那里它们与分布蛋白结合并在整个身体中循环14,15,16,17,18。最近的研究表明,环境污染物,如多氯联苯(PCB)、多溴联苯醚(PBDEs)以及全氟和多氟烷基物质(PFAS),可以竞争性地与分布蛋白转甲状腺素蛋白(TTR)结合并破坏正常的甲状腺过程19,20,21,22,23,24,25.已经开发出一种AOP,描述了与TTR的竞争性结合,导致人类神经发育毒性(https://aopwiki.org/aops/152)。虽然有证据表明这种AOP也适用于啮齿动物,但对其他分类群的适用性尚待确定。由于TTR结合化学物质存在于环境中,因此了解该AOP的分类相关性非常重要,这一挑战可以通过SeqAPASS分析部分解决。使用该工具的问题制定策略,分析的目标可以陈述如下:在知道TTR结合化合物导致人类不良后果的情况下,预计哪些分类群具有相似的易感性?
人转甲状腺素蛋白具有很好的表征,并且已知有几种经过充分研究的配体在人TTR(hTTR)结合位点结合,使其成为SeqAPASS分析的最佳靶标8,9,13。使用人甲状腺素转运蛋白的NCBI加入物P02766.1,使用默认设置进行1级分析。1 级分析的结果将相似百分比临界值设定为 49%,哺乳动物(哺乳动物)、鸟类(Aves)、爬行动物(睾丸、鳞龙、鳄鱼)、两栖动物(两栖动物)和大多数鱼类(放线鸟、腔肠动物、克拉迪斯塔、软骨鱼)都高于这一临界值(图 3)。因此,来自这些分类组的所有物种都导致了"Y"的敏感性预测(即,是),并且可能对已知与hTTR相互作用的化学物质敏感(图3和补充文件1)。
对于功能结构域的2级评估,使用NCBI保守结构域数据库将TR_THY(加入smart00095)识别为包含从残基27到147的TTR亚基蛋白成熟链的保守结构域。由于NCBI中报道的TTR蛋白质序列包括与当前分析无关的20个氨基酸前片段,因此将比较重点放在成熟链上为跨物种保存该蛋白质提供了额外的,更具体的证据线。从 2 级评估中,报告的相似性临界值百分比为 58%,哺乳动物、鸟类、爬行动物、两栖动物和大多数鱼类再次低于此临界值(图 4)。因此,SeqAPASS得出了来自这些分类群的物种的敏感性预测为"Y"(即是),表明它们可能对与hTTR蛋白相互作用的化学物质敏感(图4 和 补充文件1)。总体而言,1级和2级分析的结果表明,大多数脊椎动物物种共享hTTR的保护,并且可能对已知与这种蛋白质相互作用的化学物质敏感。
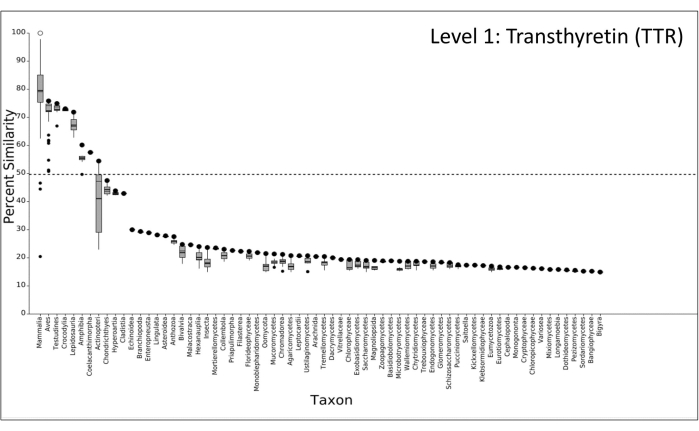
图 3:跨分类组的甲状腺素转运蛋白保护的 SeqAPASS 1 级分析,以及与人类蛋白质相关的可用序列信息。 蛋白质氨基酸序列的相似度百分比显示在Y轴上;分类组显示在 X 轴上。开放圆圈 (○) 表示查询序列,闭圈 (●) 表示分类组中相似度最高的物种。在图中,每个框的顶部和底部表示第 75个 和第 25个 百分位数,晶须延伸到四分位数范围的 1.5 倍,平均值和中值由框上的水平黑线表示。虚线表示敏感性预测的截止值。缩写:TTR = 甲状腺素转运蛋白;SeqAPASS = 序列比对以预测跨物种敏感性。 请点击此处查看此图的大图。

图 4:跨分类组的甲状腺素转运蛋白受体配体结合结构域保守的 SeqAPASS 2 级分析,具有与人类蛋白质 LBD 相关的可用序列信息。 配体结合域氨基酸序列的相似度百分比显示在Y轴上;分类组显示在 X 轴上。开放圆圈 (○) 表示查询序列,闭圈 (●) 表示分类组中相似度最高的物种。在图中,每个框的顶部和底部表示第 75个 和第 25个 百分位数,晶须延伸到四分位数范围的 1.5 倍,平均值和中值由框上的水平黑线表示。虚线表示敏感性预测的截止值。缩写:TTR = 甲状腺素转运蛋白;SeqAPASS = 序列比对以预测跨物种敏感性。 请点击此处查看此图的大图。
通过分子建模分析和蛋白质晶体学研究,在TTR的结合区鉴定出氨基酸,这些氨基酸预计与内源性配体3,3′,5,5′-四碘-L-甲状腺原氨酸(T4,PDB 2ROX)以及三种环境化学物质相互作用:全氟辛烷磺酸(PFOS,PDB 5JIM),四溴双酚A(TBBPA,PDB 5HJG)和己烯雌酚(DES,PDB 1TZ8)19, 21,22,26.氨基酸残基Lys35,Ser137,Leu130,Ala128,Ala129和Thr139都被鉴定为通过直接氢键相互作用或范德华相互作用在蛋白质 - 配体相互作用中起关键作用。使用hTTR作为模板序列,并排除非同源,假设,部分和低质量序列,在跨物种的3级分析中评估了这六个氨基酸残基(补充文件1)。由于先前确定TTR仅在脊椎动物物种中保存,因此无脊椎动物物种被排除在分析之外(图3和图4)。此外,重要的是要注意,文献中报道的氨基酸位置排除了成熟hTTR蛋白中不存在的20个氨基酸前段,因此,在Level 3中提交的位置与文献中报告的位置进行了调整,以确保与所选模板蛋白15(补充文件1)准确对齐。
在TTR的3级分析中,选择了294种脊椎动物物种(哺乳动物,鸟类,两栖动物,爬行动物和鱼类)进行对齐。在评估的物种中,18种显示出关键氨基酸的差异,导致敏感性预测为"N"(即,否)。有趣的是,五种海洋哺乳动物在位置2(128A)出现氨基酸取代,而四种鱼类在位置2(128A)或位置6(139T)表现出取代(图5)。由于这些氨基酸在TTR结合通道中的蛋白质-配体相互作用中起重要作用,这些数据表明TTR配体在这些物种中的相互作用可能不同,并且会导致相对于人类不同的化学敏感性。

图 5:对 TTR 化学结合很重要的氨基酸残基守恒的 SeqAPASS 3 级分析。 (A) 3级汇总表,显示所有分类群中具有可用序列数据的物种数量,预测为类似易感物种的数量(Y),以及预测不具有类似易感的物种数量(N)。(B) 3 级热图显示预测相对于人转甲状腺素蛋白 TTR 蛋白不具有类似敏感性的选定物种,显示完整、部分和不匹配的氨基酸。缩写:TTR = 甲状腺素转运蛋白;SeqAPASS = 序列比对以预测跨物种敏感性。 请点击此处查看此图的大图。
为了响应利益相关者和用户的反馈,SeqAPASS工具设计了新功能并将其纳入其中,包括连接到不同应用程序的经验数据的能力。该工具与 ECOTOX 知识库(表 1)的互操作性是通过在 ECOTOX 中存在的种质的 1 级和 2 级数据表中嵌入外部链接并在工具中创建 ECOTOX 小部件以直接过滤到 ECOTOX 中最相关的毒性数据来实现的。通过链接和小部件,用户可以快速查询ECOTOX并收集具有SeqAPASS敏感性预测的物种的相应毒性数据。目前,SeqAPASS预测通过化学压力源和物种与ECOTOX相关联;然而,毒性数据尚未与特定的基因/蛋白质相关联,这将允许与SeqAPASS中感兴趣的特定终点/分子靶标直接连接。虽然将预测与基于化学应激源的毒性数据联系起来并不理想,因为数据可能不特定于给定的途径,但建立连接以将结果汇集在一起是第一步。作为SeqAPASS-ECOTOX集成的第一次迭代,当前的方法为用户提供了广泛的化学应激源和物种的所有可用毒性数据。这些数据与SeqAPASS预测相结合,可以提供广泛的背景(脊椎动物与无脊椎动物),并且可以在AOP框架的背景下考虑。
TTR为检查这种联系提供了一个很好的案例,因为现有的AOP(AOP 152)为解释潜在相关的ECOTOX毒性数据提供了背景。从SeqAPASS Level 3中检查的配体开始,收集了已知与TTR配体结合域相互作用的四种化学物质(己烯雌酚[DES],全氟己烷酸[PFHxA],全氟辛烷磺酸[PFOS]和四溴双酚A [TBBPA])的环境毒性数据19,21,23,24.对于每种化学品,使用自定义搜索参数(补充文件1)通过化学文摘社(CAS)编号查询ECOTOX的水生和陆地数据。数据被过滤到感兴趣的物种组(两栖动物,鸟类,鱼类,无脊椎动物,哺乳动物,爬行动物)。在过滤的查询结果中,计算并实施研究的最小和最大效应浓度的平均值,作为未报告平均效应浓度值的任何命中的平均值的近似值(图6A和补充文件1)。在单一化学品的背景下,进行了Kruskal-Wallis检验,以比较不同分类组的平均效应浓度,因为数据不符合方差分析检验假设。然后使用邓恩检验对所有化学物质进行事后成对比较测试,因为分类组由不相等的样本量组成。水生和陆地结果分别分析,因为两种类型的暴露之间的数据没有直接可比性。在ECOTOX中,可以选择两栖动物,鸟类,无脊椎动物和鱼类的水生毒性数据(图6A)。选定化学品的陆地毒性数据仅可用于哺乳动物和DES(补充文件1)。

图 6:将 SeqAPASS 结果与经验数据联系起来 。 (A)跨分类组的平均效应浓度,ECOTOXicicic知识库中提供了已知与人类TTR蛋白结合的特定化学物质的数据。(B)每个SeqAPASS分析中包含的物种数量与可获得ECOTOX数据的物种重叠。在面板 A 中,x 轴上的括号表示聚合数据的查询命中数。星号表示在单一化学品的背景下物种组之间具有显著差异的效应浓度对(邓恩检验,p < 0.05),其中星号数越高表示显著性水平越强(*: p < 0.05; **: p < 0.01; ***: p < 0.001; ****: p < 0.0001)。每个框内的中心线表示中位数,框边缘表示四分位数间距。晶须延伸至四分位距的 1.5 倍。超出该范围的异常值显示为单个点。缩写:TTR = 甲状腺素转运蛋白;SeqAPASS = 序列比对以预测跨物种易感性;ECOTOX = 生态毒素学知识库。 请点击此处查看此图的大图。
总体而言,这些数据表明,所评估的化学物质的生物活性发生在脊椎动物物种中,但不发生在无脊椎动物中。虽然ECOTOX中缺乏生物靶标和途径信息使得无法将这些经验数据直接与TTR联系起来,但这些结果确实支持了SeqAPASS的预测,即无脊椎动物物种不具有易感性。所有有数据可查的脊椎动物物种都表明对全氟辛烷磺酸和TBBPA都易感,尽管鱼类和鸟类的平均效应浓度明显高于两栖动物。这些数据表明,分类群之间的敏感性可能存在差异,这可能归因于生物途径差异(包括TTR)。值得注意的是,代谢和排泄等其他变量也可能在敏感性差异中发挥作用。就全氟辛烷磺酸和全氟辛烷磺酸而言,鱼类明显比无脊椎动物和鸟类更敏感,就DES而言,两栖动物的平均效应浓度明显高于无脊椎动物。同样,这些数据支持我们的SeqAPASS预测,即无脊椎动物与脊椎动物物种没有共同的易感性。在使用该工具评估的所有物种中,只有少数物种具有四种感兴趣的化学品的相应ECOTOX数据(图6B,补充表S1和补充表S2)。对于那些缺乏顶端数据的物种,SeqAPASS对易感性的预测增加了额外的证据线,表明相关物种的行为可能与具有顶端数据的物种相似。所有SeqAPASS和ECOTOX分析的数据可在补充文件1中找到。
根据疾病控制和预防中心(CDC)的数据,2017年,阿片类药物导致美国约47,600人因过量服用而死亡,这一数字继续上升27人。美国的废水厂在全国范围内受到美国环境保护署的国家污染物排放消除系统的监管,该系统不需要在其排放中测试阿片类药物或其他药物28。近年来,已经努力使用基于废水的流行病学作为绘制社区阿片类药物使用情况的工具。阿片类药物监测工作在废水中检测到浓度高达1.27微克/升,在地表水中检测到浓度高达0.7微克/升29,30。最近评估阿片类药物暴露对鱼类影响的毒性研究报告了成瘾行为的发展和不利的免疫影响(例如,更高的感染率,免疫基因的下调)31,32,33。总体而言,这些研究表明存在不良环境阿片类药物暴露的可能性,并强调了了解这些化学物质对水生物种构成的风险的重要性。鉴于可能在环境中遇到这些化合物的物种范围,使用SeqAPASS识别潜在的易感物种对于确定测试或监测工作的优先级可能很重要。
MOR是治疗疼痛的主要阿片类药物靶标,并负责人类阿片类生物碱的强大镇痛和成瘾特性34,35。由于该受体对人类健康的重要性,MOR配体是众所周知的,并且可以使用高质量的X射线晶体学研究,使该靶标成为SeqAPASS分析的理想选择8,9,13。使用人μ阿片受体的NCBI加入物ACM90349.1,使用默认设置进行1级分析。1级的易感性临界值确定为55%,哺乳动物(哺乳动物)、鸟类(Aves)、爬行动物(睾丸、鳞龙、鳄鱼)、两栖动物(两栖动物)和大多数鱼类(放线鸟、腔肠龙、克拉迪斯塔、软骨鱼)的相似性百分比高于此临界值;因此,来自这些分类群的物种导致易感性预测为"Y"(即是),表明它们可能对已知与人类MOR相互作用的化学物质敏感(图7和补充文件1)。使用NCBI保守结构域数据库,7tmA_Mu_opioid_R被鉴定为功能结构域(加入cd15090),包括MOR蛋白从133到411的所有七个螺旋,包括一个假定的配体结合位点。与 1 级相比,2 级结果确定了更高的易感性临界值,相似度为 88%,哺乳动物、鸟类、爬行动物、两栖动物和大多数鱼类都高于此临界值,导致易感性预测为"Y"表示是(图 8)。总体而言,1级和2级分析的结果表明,大多数脊椎动物物种共享MOR的保护,并且可能容易受到已知与人类MOR相互作用的化学物质的影响。
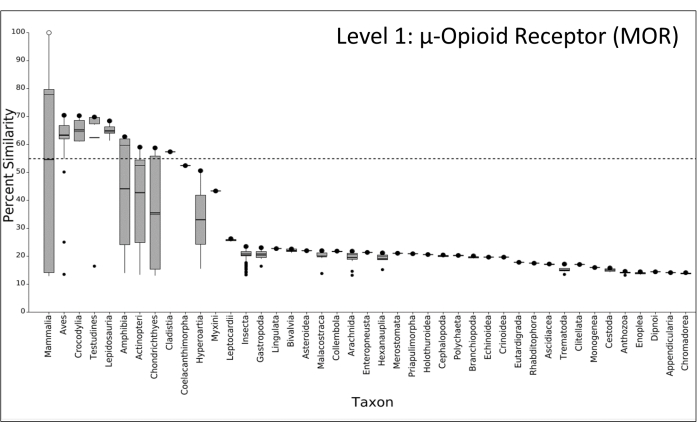
图 7:跨分类组μ阿片受体保护的 SeqAPASS 1 级分析,以及与人类蛋白质相关的可用序列信息。 蛋白质氨基酸序列的相似度百分比显示在Y轴上;分类组显示在 X 轴上。开放圆圈 (○) 表示查询序列,闭圈 (●) 表示分类组中相似度最高的物种。在图中,每个框的顶部和底部表示第 75 个和第 25 个百分位数,晶须延伸到四分位数间距的 1.5 倍,平均值和中值由框上的水平黑线表示。虚线表示敏感性预测的截止值。缩写:MOR = μ-阿片受体;SeqAPASS = 序列比对以预测跨物种敏感性。 请点击此处查看此图的大图。

图 8:相对于人类蛋白质结构域的阿片受体配体结合结构域保守μ的 SeqAPASS 2 级分析。 配体结合域氨基酸序列的相似度百分比显示在Y轴上;分类组显示在 X 轴上。开放圆圈 (○) 表示查询序列,闭圈 (●) 表示分类组中相似度最高的物种。在图中,每个框的顶部和底部表示第 75 个和第 25 个百分位数,晶须延伸到四分位数间距的 1.5 倍,平均值和中值由框上的水平黑线表示。虚线表示敏感性预测的截止值。缩写:MOR = μ-阿片受体;SeqAPASS = 序列比对以预测跨物种敏感性。 请点击此处查看此图的大图。
通过分子建模和蛋白质晶体学研究的分析,在MOR的结合区域中鉴定出氨基酸,这些氨基酸预计与已知配体相互作用。尽管与阿片受体结合良好的各种配体导致复杂的药理学,但观察到一些一致的配体 - 蛋白质相互作用36,37。基于分子对接到各种MOR晶体结构,包括吗啡和芬太尼,高亲和力MOR激动剂,与D147,Y148,M151,W293,I296,H297,V300,I322和Y326相互作用36,38。残基D147、Y148、M151和H297也与吗啡激动剂BU72结合的MOR晶体结构有关,而D147、M151、H297和Y326在与不可逆吗啡拮抗剂β-funaltrexam37结合中也至关重要。考虑到这些证据线,选择九个残基(D147、Y148、M151、W293、I296、H297、V300、I322、Y326)进行3级评估,使用人类MOR作为模板序列,排除部分、预测、假设和低质量序列。重要的是,文献中报道的氨基酸位置排除了相对于NCBI蛋白加入的64个氨基酸片段,因此,选择3级的位置来代表那些与模板序列对齐以获得正确比对序列的位置。
在对人类MOR的3级分析中,评估了脊椎动物物种(哺乳动物,鸟类,两栖动物,爬行动物和鱼类)的284个物种。在评估的所有物种中,基于侧链分类和分子量,九种氨基酸要么完全匹配,要么部分匹配;因此,所有评估的物种的敏感性预测为"Y"为"是"(表3和补充文件1)。由于这些氨基酸在强MOR激动剂和强拮抗剂的结合中都很重要,这些数据表明,靶向人类μ阿片受体的阿片类化合物可能与脊椎动物物种的受体发生类似的相互作用。尽管迄今为止,ECOTOX知识库中关于阿片类化合物的经验数据很少,但几项研究表明,鱼类可能易感31,32,33。总体而言,SeqAPASS的结果表明,MOR调节化学品对跨物种的环境影响更大,这表明更多的研究和监测可能是有价值的。所有分析的数据可在补充文件1中找到。
表3:SeqAPASS 3级分析,用于对与μ阿片受体的化学结合很重要的氨基酸残基的保存。 汇总表显示了所有分类群中具有可用序列数据的物种数量、预测为类似易感物种的数量 (Y)、预测不具有类似易感性的物种数量,以及完整、部分和不匹配的氨基酸。缩写:seqAPASS = 序列比对,用于预测跨物种的敏感性。 请按此下载此表格。
补充表S1:已知与人甲状腺素蛋白结合的四种感兴趣的化学物质的可用ECOTOX数据的物种。每种化学品的可用数据与SeqAPASS对1级,2级和3级相似敏感性的预测一致。与人转甲状腺素蛋白序列相关的所有SeqAPASS预测。缩写:seqAPASS = 序列比对以预测跨物种敏感性;DES = 己烯雌酚;PFHxA = 全氟己酸;全氟辛烷磺酸=全氟辛烷磺酸;TBBPA = 四溴双酚 A. 请点击此处下载此表。
补充表S2:在SeqAPASS和ECOTOX评估中具有可用数据的物种总数,用于已知与人转甲状腺素蛋白结合的选定化学物质。SeqAPASS提供了预测许多物种的物种易感性的方法,这些物种没有经验毒性数据。 请按此下载此表格。
补充文件1:所有代表性结果的SeqAPASS和ECOTOX数据。文件包含ToC,后跟以下数据表:选项卡1-hTTR序列APASS结果级别1,选项卡2-hTTR序列APASS结果级别2,选项卡3-hTTR序列APASS结果级别3,选项卡4-DES的EcoTox数据,选项卡5-PFHxA的EcoTox数据,选项卡7-TBBPA的EcoTox数据,选项卡8-EcoTox组平均值计算, 选项卡 9-序列APASS EcoTox 数据比较、选项卡 10-hMOR 序列APASS 结果级别 1、选项卡 11-hMOR 序列APASS 结果级别 2 和选项卡 12-μ-hMOR 序列APASS 结果级别 3。缩写:seqAPASS = 序列比对以预测跨物种敏感性;目录 = 目录;hTTR = 人甲状腺素转运蛋白;ECOTOX = 生态毒理学知识库;DES = 己烯雌酚;全氟辛烷磺酸=全氟辛烷磺酸;PFHxA = 全氟己酸;TBBPA=四溴双酚A;hMOR = 人 μ-阿片受体。 请点击此处下载此文件。
讨论
人们普遍认识到,对足够的物种进行经验测试以捕获可能暴露于毒理学相关化学物质的生物体的基因组、表型、生理和行为多样性是不可行的。SeqAPASS的目标是最大限度地利用现有和不断扩展的蛋白质序列和结构数据,通过分子水平的比较,帮助和告知从测试生物体到数百或数千种其他物种的化学毒性数据/知识的外推。SeqAPASS工具旨在通过简化和快速的分析来降低科学家,风险评估人员和监管机构蛋白质序列比较的复杂性,其中包括透明生成和可下载的汇总表,交互式数据可视化以及轻松识别受威胁和濒危物种以及常见模式生物。此处描述了运行 SeqAPASS 级别 1、2 和 3 的协议,以评估初级氨基酸序列相似性、功能域保守以及参与化学-蛋白质和蛋白质-蛋白质相互作用的关键氨基酸。从每个级别的SeqAPASS分析中收集的证据线预测了跨物种的化学敏感性,提供了一致且易于解释的数据。迄今为止,该工具已被广泛用于广泛的应用,包括鉴定与某些受体结合的化学物质以及评估具有哺乳动物系统的脊椎动物生态受体的读取潜力。此外,这里还介绍了两个专注于甲状腺激素分布蛋白、TTR 和 MOR 的案例研究,以演示 SeqAPASS v2 至 v6 的新特性和功能。
与任何计算方法一样,在SeqAPASS工具中生成物种敏感性预测的能力高度依赖于适当参数8,9,13的输入。因此,至关重要的是,在进行分析之前,要进行问题制定步骤,以调查现有数据和文献,以寻找预定目标。在了解蛋白质靶标的情况下开始分析,用户可以识别适当的蛋白质加入数和高质量序列。同样,对敏感或目标物种或用于测定或AOP开发的模式生物的了解可确保选择与所有其他物种进行比较的适当查询物种。为2级选择功能域,为3级选择关键氨基酸残基也是关键步骤,需要用户确定适当的输入参数以生成预测。由于需要预先存在的化学-蛋白质相互作用知识,最新版本的SeqAPASS工具集成了用户友好的资源,旨在帮助指导用户获取启动查询的相关信息(例如,指向其他工具的链接)(表2和图2)。此外,弹出信息消息和警报已集成到工具中,以指导用户完成分析并帮助通知用户需要解决的任何错误。
化学-生物相互作用的复杂性是SeqAPASS工具的局限性。在跨物种推断毒性数据时,分子靶标的守恒是需要考虑的众多因素之一。在考虑化学毒性时,化学物质的吸附,分布,代谢和排泄(ADME)至关重要,因为化学物质可以通过这些过程被激活或解毒39,40。其他因素,如化学暴露途径,生物体生命阶段和生活史以及饮食,也可以在确定跨物种的化学敏感性方面发挥重要作用41,42。为了解决这一限制,重要的是要了解SeqAPASS在预测化学敏感性时提出的主要问题:化学物质的蛋白质靶标是否可能存在于另一个物种中,以便化学物质起作用?这个问题可以通过确定直系同源物候选物并考虑相对于已知敏感或目标物种跨物种保护该目标来解决。该信息可用作跨物种外推的证据线,并整合到其他证据流中(例如,暴露的可能性),以更好地了解物种对化学应激源的易感性。SeqAPASS的更新包含了与外部工具的集成链接,包括美国EPA ECOTOX知识库43 和美国鱼类和野生动物管理局环境保护在线系统(ECOS)44。通过连接到这些数据库,SeqAPASS用户可以轻松访问经验化学毒性数据,以便与基于序列的预测进行比较,并具有识别可能具有保护状态的物种的方法。
SeqAPASS工具为内在敏感性的计算预测提供了一个基于科学的平台,这些预测得到了进化生物学概念和将预测与可用经验结果进行比较的案例的支持。此外,SeqAPASS是免费的,并且可以在支持良好的基于Web的平台上公开获得,该平台可广泛访问(https://seqapass.epa.gov/seqapass/)。由于该工具利用了来自现有数据库的序列数据和蛋白质信息,因此随着测序技术的进步以及新物种的基因组被测序和注释,它预测更广泛物种多样性的化学敏感性的能力正在不断提高。尽管这在数据可用性方面提供了明显的优势,但它也存在一个局限性,即公开可用的序列信息可能会受到某些物种的蛋白质序列质量不一致、注释差和不完整的影响。然而,生物信息学中的组学技术和方法正在迅速发展,因此,随着时间的推移,序列管理和质量可能会继续提高。
SeqAPASS工具的一个主要目标是透明度,以链接的形式提供对后端集成的所有数据源和工具的访问。这种透明度使用户能够快速访问NCBI的序列或分类信息的原始来源。此工具的适用范围由进行有意义的分析所需的信息定义。由于了解已知敏感或目标物种中的化学-蛋白质或蛋白质-蛋白质相互作用是开始查询的关键要素,因此必须承认,在没有这些信息的情况下进行的查询是没有意义的。此外,具有多个未定义生物靶标或与不同靶标相互作用且效力不同的化学品也对当前形式的工具提出了挑战和限制。预计随着生物信息学,计算建模以及基于细胞的高通量筛选和转录组学的改进,将继续阐明化学空间多样性中有关与特定蛋白质相互作用的更多知识。预计将SeqAPASS应用于物种外推的更广泛挑战的能力,相对于了解物种多样性中不利化学效应的可能性,将继续提高。
总之,SeqAPASS工具是一个可访问的平台,可以轻松应用分子信息来解决化学品安全评估中跨物种外推的巨大挑战。虽然这里强调的例子侧重于生成化学敏感性的预测,但结果也有助于理解生物途径的整体保护。该工具汇集了不同的证据线,便于访问多个平台和数据库,有助于为化学测试的优先级和资源分配建立透明的案例。随着科学和生物信息能力的不断发展,该工具的功能和实用性将继续增长和改进,以满足研究和监管界的需求,同时减少跨物种评估所需的资源。
披露声明
作者没有利益冲突需要披露。
致谢
作者感谢Daniel L. Villeneuve博士(美国环保署计算毒理学和暴露中心)和Jon A. Doering博士(路易斯安那州立大学环境科学系)对手稿的早期草稿提供评论。这项工作得到了美国环境保护署的支持。本文中表达的观点是作者的观点,不一定反映美国环境保护署的观点或政策,提及的商品名称或商业产品也不表示联邦政府的认可。
材料
| Name | Company | Catalog Number | Comments |
| Spreadsheet program | N/A | N/A | Any program that can be used to view and work with csv files (e.g. Microsoft Excel, OpenOffice Calc, Google Docs) can be used to access data export files. |
| Basic computing setup and internet access | N/A | N/A | SeqAPASS is a free, online tool that can be easily used via an internet connection. No software downloads are required. |
参考文献
- Krewski, D., et al. Toxicity testing in the 21st century: a vision and a strategy. Journal of Toxicology and Environmental Health, Part B. 13 (2-4), 51-138 (2010).
- Wang, Z., Walker, G. W., Muir, D. C. G., Nagatani-Yoshida, K. Toward a global understanding of chemical pollution: A first comprehensive analysis of national and regional chemical inventories. Environmental Science & Technology. 54 (5), 2575-2584 (2020).
- Brooks, B. W., et al. Toxicology advances for 21st century chemical pollution. One Earth. 2 (4), 312-316 (2020).
- Kostal, J., Voutchkova-Kostal, A. Going all in: A strategic investment in in silico toxicology. Chemical Research in Toxicology. 33 (4), 880-888 (2020).
- Cheng, W., Doering, J. A., LaLone, C., Ng, C. Integrative computational approaches to inform relative bioaccumulation potential of per- and polyfluoroalkyl substances (PFAS) across species. Toxicology Sciences. 180 (2), 212-223 (2021).
- Kostich, M. S., Lazorchak, J. M. Risks to aquatic organisms posed by human pharmaceutical use. Science of the Total Environment. 389 (2-3), 329-339 (2008).
- Gunnarsson, L., Jauhiainen, A., Kristiansson, E., Nerman, O., Larsson, D. G. Evolutionary conservation of human drug targets in organisms used for environmental risk assessments. Environmental Science & Technology. 42 (15), 5807-5813 (2008).
- LaLone, C. A., et al. Evidence for cross species extrapolation of mammalian-based high-throughput screening assay results. Environmental Science & Technology. 52 (23), 13960-13971 (2018).
- LaLone, C. A., et al. Editor's highlight: Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS): A web-based tool for addressing the challenges of cross-species extrapolation of chemical toxicity. Toxicology Sciences. 153 (2), 228-245 (2016).
- Head, J. A., Hahn, M. E., Kennedy, S. W. Key amino acids in the aryl hydrocarbon receptor predict dioxin sensitivity in avian species. Environmental Science & Technology. 42 (19), 7535-7541 (2008).
- Bass, C., et al. Mutation of a nicotinic acetylcholine receptor β subunit is associated with resistance to neonicotinoid insecticides in the aphid Myzus persicae. BMC Neuroscience. 12, 51-51 (2011).
- Erdmanis, L., et al. Association of neonicotinoid insensitivity with a conserved residue in the loop d binding region of the tick nicotinic acetylcholine receptor. Biochemistry. 51 (23), 4627-4629 (2012).
- Doering, J. A., et al. et al. In silico site-directed mutagenesis informs species-specific predictions of chemical susceptibility derived from the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) tool. Toxicology Sciences. 166 (1), 131-145 (2018).
- Noyes, P. D., et al. Evaluating chemicals for thyroid disruption: Opportunities and challenges with in vitro testing and adverse outcome pathway approaches. Environmental Health Perspectives. 127 (9), 95001 (2019).
- Park, G. Y., Jamerlan, A., Shim, K. H., An, S. S. A. Diagnostic and treatment approaches involving transthyretin in amyloidogenic diseases. Int J Mol Sci. 20 (12), 2982 (2019).
- Rabah, S. A., Gowan, I. L., Pagnin, M., Osman, N., Richardson, S. J. Thyroid hormone distributor proteins during development in vertebrates. Front Endocrinol (Lausane). 10, 506 (2019).
- Richardson, S. J. Cell and molecular biology of transthyretin and thyroid hormones. International Review of Cytology. 258, 137-193 (2007).
- Yamauchi, K., Ishihara, A., Richardson, S. J., Cody, V. Transthyretin and Endocrine Disruptors. Recent Advances in Transthyretin Evolution, Structure and Biological Functions. , 159-171 (2009).
- Iakovleva, I., et al. Tetrabromobisphenol A is an efficient stabilizer of the transthyretin tetramer. PLoS One. 11 (4), 0153529 (2016).
- Ishihara, A., Sawatsubashi, S., Yamauchi, K. Endocrine disrupting chemicals: Interference of thyroid hormone binding to transthyretins and to thyroid hormone receptors. Molecular and Cellular Endocrinology. 199 (1), 105-117 (2003).
- Kar, S., Sepúlveda, M. S., Roy, K., Leszczynski, J. Endocrine-disrupting activity of per- and polyfluoroalkyl substances: Exploring combined approaches of ligand and structure based modeling. Chemosphere. 184, 514-523 (2017).
- Morais-de-Sa, E., Pereira, P. J., Saraiva, M. J., Damas, A. M. The crystal structure of transthyretin in complex with diethylstilbestrol: A promising template for the design of amyloid inhibitors. Journal of Biological Chemistry. 279 (51), 53483-53490 (2004).
- Morgado, I., Campinho, M. A., Costa, R., Jacinto, R., Power, D. M. Disruption of the thyroid system by diethylstilbestrol and ioxynil in the sea bream (Sparus aurata). Aquatic Toxicology. 92 (4), 271-280 (2009).
- Yamauchi, K., Prapunpoj, P., Richardson, S. J. Effect of diethylstilbestrol on thyroid hormone binding to amphibian transthyretins. General and Comparative Endocrinology. 119 (3), 329-339 (2000).
- Zhang, J., et al. Structure-based virtual screening protocol for in silico identification of potential thyroid disrupting chemicals targeting transthyretin. Environmental Science & Technology. 50 (21), 11984-11993 (2016).
- Ren, X. M., et al. Binding interactions of perfluoroalkyl substances with thyroid hormone transport proteins and potential toxicological implications. Toxicology. 366-367, 32-42 (2016).
- Wilson, N., Mbabazi, K., Seth, P., Smith, H., Davis, N. L. Drug and opioid-involved overdose deaths - United States, 2017-2018. Morbidity and Mortality Weekly Report. 69 (11), 290-297 (2020).
- National Pollutant Discharge Elimination System (NPDES). United States Environmental Protection Agency Available from: https://www.epa.gov/npdes/npdes-resources (2018)
- Duvallet, C., Hayes, B. D., Erickson, T. B., Chai, P. R., Matus, M. Mapping community opioid exposure through wastewater-based epidemiology as a means to engage pharmacies in harm reduction efforts. Preventing Chronic Disease. 17, 200053 (2020).
- Gushgari, A. J., Venkatesan, A. K., Chen, J., Steele, J. C., Halden, R. U. Long-term tracking of opioid consumption in two United States cities using wastewater-based epidemiology approach. Water Research. 161, 171-180 (2019).
- Lau, B., Bretaud, S., Huang, Y., Lin, E., Guo, S. Dissociation of food and opiate preference by a genetic mutation in zebrafish. Genes Brain Behave. 5 (7), 497-505 (2006).
- Bossé, G. D., Peterson, R. T. Development of an opioid self-administration assay to study drug seeking in zebrafish. Behavioural Brain Research. 335, 158-166 (2017).
- Mottaz, H., et al. Dose-dependent effects of morphine on lipopolysaccharide (LPS)-induced inflammation, and involvement of multixenobiotic resistance (MXR) transporters in LPS efflux in teleost fish. Environmental Pollution. 221, 105-115 (2017).
- Manglik, A., et al. Crystal structure of the µ-opioid receptor bound to a morphinan antagonist. Nature. 485 (7398), 321-326 (2012).
- Comer, S. D., Cahill, C. M. Fentanyl: Receptor pharmacology, abuse potential, and implications for treatment. Neuroscience & Biobehavioral Reviews. 106, 49-57 (2019).
- Podlewska, S., Bugno, R., Kudla, L., Bojarski, A. J., Przewlocki, R. Molecular modeling of µ opioid receptor ligands with various functional properties: PZM21, SR-17018, morphine, and fentanyl-simulated interaction patterns confronted with experimental data. Molecules. 25 (20), 4636 (2020).
- Huang, W., et al. Structural insights into µ-opioid receptor activation. Nature. 524 (7565), 315-321 (2015).
- Lipiński, P. F. J., et al. Fentanyl family at the mu-opioid receptor: Uniform assessment of binding and computational analysis. Molecules. 24 (4), 740 (2019).
- Boland, L. A., Angles, J. M. Feline permethrin toxicity: Retrospective study of 42 cases. Journal of Feline Medicine and Surgery. 12 (2), 61-71 (2010).
- Stevenson, B. J., Pignatelli, P., Nikou, D., Paine, M. J. Pinpointing P450s associated with pyrethroid metabolism in the dengue vector, Aedes aegypti: developing new tools to combat insecticide resistance. PLoS Neglected Tropical Diseases. 6 (3), 1595 (2012).
- Ankley, G. T., Gray, L. E. Cross-species conservation of endocrine pathways: A critical analysis of tier 1 fish and rat screening assays with 12 model chemicals. Environmental Toxicology and Chemistry. 32 (5), 1084-1087 (2013).
- Meteyer, C. U., Rideout, B. A., Gilbert, M., Shivaprasad, H. L., Oaks, J. L. Pathology and proposed pathophysiology of diclofenac poisoning in free-living and experimentally exposed oriental white-backed vultures (Gyps bengalensis). Journal of Wildlife Diseases. 41 (4), 707-716 (2005).
- ECOTOX User Guide: ECOTOXicology Knowledgebase System. EPA, United States Environmental Protection Agency Available from: https://cfpub.epa.gov/ecotox/index.cfm (2021)
- ECOS Environmental Conservation Online System. U.S. Fish & Wildlife Service Available from: https://ecos.fws.gov/ecp/ (2021)
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。