Method Article
Ensaio de número mais provável modificado para quantificar Salmonella em produtos de frango crus e prontos para cozinhar
Neste Artigo
Resumo
Quantificar com precisão a Salmonella em aves em níveis baixos é um desafio industrial e regulatório atual. Este protocolo descreve um ensaio MPN que permite a quantificação de Salmonella em produtos avícolas crus e prontos para cozinhar. Este método é rápido, sensível e se alinha com as diretrizes do FSIS, aumentando a segurança alimentar e apoiando os esforços de saúde pública.
Resumo
A salmonela é uma das principais causas de doenças transmitidas por alimentos nos Estados Unidos, principalmente em produtos avícolas. Os métodos tradicionais de detecção de Salmonella se concentram na prevalência e não na quantificação, o que limita sua utilidade na avaliação dos níveis e riscos de contaminação. Este estudo apresenta um novo ensaio de número mais provável (MPN) projetado para quantificar Salmonella em produtos avícolas prontos para cozinhar, como frango cordon bleu. O método envolve lavar a amostra de aves, concentrar o enxágue por centrifugação e diluí-lo em série em um bloco de 48 poços. O ensaio MPN é integrado ao método de amplificação isotérmica mediada por loop (LAMP) para fornecer uma quantificação sensível, precisa e rápida da contaminação por Salmonella dentro do mesmo período de tempo que os protocolos existentes do Serviço de Inspeção e Segurança Alimentar (FSIS). Os resultados mostram uma forte correlação linear entre as medidas de MPN-LAMP e os níveis teóricos de inoculação (R² = 0,933). No entanto, a variabilidade em concentrações mais baixas destaca os desafios na detecção precisa de Salmonella nesses níveis, com o limite inferior prático de detecção estimado em aproximadamente 300 UFC / g. Refinamentos potenciais para melhorar a aplicabilidade do protocolo incluem aumentar a quantidade amostrada para melhorar ainda mais o limite de detecção, otimizar formulações de meios de enriquecimento e expandir a detecção molecular para atingir vários sorovares de Salmonella . No geral, este estudo apresenta uma ferramenta prática para a indústria alimentícia, permitindo a quantificação confiável da contaminação por Salmonella em produtos avícolas, contribuindo para melhorar a segurança alimentar e a saúde pública.
Introdução
Como uma das principais causas de doenças transmitidas por alimentos, hospitalização e morte nos EUA, a Salmonella tem um impacto significativo na saúde pública e na economia. A carga econômica estimada do patógeno somente em 2013 foi de US $ 3,67 bilhões1. Embora as recentes iniciativas regulatórias visem reduzir a salmonelose em 25% até 20302, as lacunas nas estratégias atuais de detecção e mitigação permanecem evidentes, particularmente no alinhamento da vigilância da planta de processamento com os resultados de saúde pública3 .
Produtos avícolas congelados prontos para cozinhar, que foram implicados em vários surtos de Salmonella , são uma preocupação significativa para a saúde pública. Em resposta, o Serviço de Inspeção e Segurança Alimentar (FSIS) classificou a Salmonella como adulterante nesses produtos. Atualmente, o Guia de Laboratório de Microbiologia do FSIS (MLG) 4.15 concentra-se exclusivamente na determinação da prevalência de Salmonella em produtos avícolas4. De acordo com essa diretriz, as amostras coletadas são enriquecidas por 18 a 24 horas e, em seguida, rastreadas usando o Sistema de Detecção Molecular (MDS), que identifica a presença ou ausência de Salmonella , mas não oferece informações sobre o nível de contaminação. Embora essa abordagem seja valiosa para detectar a presença de patógenos, ela não fornece informações quantitativas que poderiam ajudar os processadores de alimentos a avaliar os riscos de contaminação com mais precisão e tomar ações corretivas direcionadas.
Neste estudo, desenvolvemos um método para aumentar a detecção da prevalência à quantificação de patógenos microbianos. Ele foi projetado para integração perfeita em processos existentes para detectar Salmonella em produtos avícolas com o mínimo de interrupção nos protocolos FSIS atuais. Em vez de simplesmente enriquecer a amostra a granel, o método começa lavando os produtos avícolas usando meios consistentes com os métodos FSIS atuais. O enxágue é então distribuído na primeira coluna de um bloco de poço de 48 de profundidade. Diluições seriais são realizadas nas cinco colunas restantes e o bloco é incubado por 18-24 h, alinhando-se com o protocolo MLG 4.15. Após a incubação, os poços são testados para Salmonella, e os resultados são usados para calcular o número mais provável (MPN)5,6. Essa abordagem permite a quantificação da contaminação dentro do mesmo período de tempo que o processo FSIS atual, tornando-o uma opção prática para uso industrial e regulatório. A Figura 1 mostra um diagrama de blocos resumindo o ensaio MPN modificado. A figura inclui fotografias tiradas em etapas específicas, o bloco de 48 poços utilizado para diluição e crescimento de réplicas e as três técnicas usadas como referência para avaliar o número mais provável de Salmonella presente em frango moído. Na primeira fase deste estudo, utilizamos frango moído irradiado para minimizar o impacto da microflora de fundo e a incerteza das medições em relação ao inóculo verificado antes de aplicar o protocolo a amostras de frango não irradiadas.
Protocolo
NOTA: Todo o trabalho associado a este protocolo deve ser conduzido em um laboratório de Nível de Biossegurança 2 (BSL-2). Quando apropriado, este protocolo deve ser conduzido dentro de um gabinete de segurança biológica (BSC) para manter as condições assépticas e minimizar o risco de contaminação da amostra ou exposição do operador a patógenos microbianos. Ao transferir amostras para fora do BSC, use recipientes lacrados para manter a integridade da amostra e evitar derramamento em caso de quedas acidentais. De preferência, componentes descartáveis devem ser usados durante todo o procedimento para mitigar a possibilidade de contaminação cruzada. Nos casos em que os descartáveis não são viáveis, certifique-se de que todos os equipamentos e materiais sejam estéreis antes do uso. A gestão adequada dos resíduos é crucial; Todos os componentes descartáveis usados devem ser descartados como resíduos de risco biológico. Autoclave materiais reutilizáveis antes da reutilização para garantir a esterilização e contenção adequadas de materiais potencialmente perigosos. Aderir a essas precauções não apenas protege a integridade da amostra, mas também minimiza o risco de exposição do operador a patógenos microbianos.
1. Preparação de amostras de carne
- Aquisição e processamento de amostras de carne
- Carne fresca
- Adquira frango moído no departamento de carnes frescas dos varejistas locais. Transfira todas as amostras para armazenamento a 4 °C e processe dentro de 24 horas após o recebimento. Divida assepticamente a carne em amostras de 25 g.
- Selar a vácuo e irradiar a amostra. Aqui, o Texas A&M AgriLife National Center for Electron Beam Research irradiou carne submetida a uma dose de ~ 25 kGy.
NOTA: Embora a irradiação tenha sido usada como medida de controle neste estudo para garantir a eliminação da microflora de fundo, ela não é um pré-requisito para o protocolo em aplicações práticas, conforme mostrado pelos produtos prontos para cozinhar não irradiados usados na seção subsequente. Em ambientes de campo, métodos alternativos, como meios seletivos ou especificidade de diagnósticos moleculares, podem abordar a interferência potencial de microrganismos não-alvo.
- Produtos de frango prontos para cozinhar
- Adquira produtos de frango prontos para cozinhar na seção de alimentos congelados dos varejistas locais. Dividir assepticamente em amostras de 25 g.
- Colete amostras do centro das peças individuais para garantir que todos os ingredientes (por exemplo, empanados e queijo) sejam incluídos.
- Carne fresca
- Preparação de mídia
- Prepare a Água Peptonada Tamponada (BPW) dissolvendo 25 g de pó BPW em 1 L de H2O nanopuro.
- Prepare placas de infusão cérebro-coração (BHI). Para fazer isso, dissolva 37 g de pó de BHI em 1 L de H2O nanopuro e adicione 15 g de ágar na solução de BHI. Esterilize todos os meios em autoclavagem a 121 °C durante 15 min. Despeje 20 a 25 mL de mídia em placas de Petri com tampa transparente (100 mm x 15 mm).
NOTA: É melhor colocar as placas dentro de um gabinete de segurança biológica para manter as condições assépticas.
2. Cultura celular
- Preparar a cultura inicial com uma colocação em linha de Salmonella enterica sorovar Typhimurium ATCC 14028 numa placa de ágar BHI e incubar a 37 °C durante a noite.
- Prepare culturas durante a noite inoculando 25 mL de caldo BHI com uma colônia de Salmonella recém-cultivada. Cultive culturas aeróbicas durante a noite a 37 °C com agitação a 100 rpm.
3. Inoculação de amostras de aves de capoeira
- Diluição de cultura e plaqueamento
- Preparar uma série de diluições de 10 vezes da cultura durante a noite em BPW para atingir concentrações finais de aproximadamente 1 x 108 a 1 x 101 UFC / mL. Suponha que a concentração da cultura noturna de Salmonella seja de 1 x 109 UFC / mL.
- Transfira 0,5 mL da cultura durante a noite para 4,5 mL de BPW, misture e, em seguida, transfira 0,5 mL da diluição para 4,5 mL de BPW para cada diluição adicional.
- Espalhar 10 μL da diluição de 1 x 103 UFC/ml numa placa de ágar BHI em triplicado para enumeração celular para calcular a concentração da cultura durante a noite.
- Inoculação de amostras de carne
- Transfira assepticamente 25 g de frango moído irradiado para um saco estéril em duplicado. A bolsa tem 7,5 x 12 polegadas e é rotulada como 1,63 L. O saco contém uma divisória de filtro com um diâmetro de furo de 330 μm e 285 por cm quadrado.
- Inocular cada amostra com 1 ml da diluição da concentração alvo. Por exemplo, adicione 1 mL de cultura da diluição de 1 x 10³ UFC/mL para atingir um nível de contaminação de aproximadamente 1.000 células/25 g de frango. Distribuir suavemente o inóculo líquido sobre a superfície das amostras de frango utilizando um espalhador de células esterilizado e deixá-lo repousar durante 1 h a 4 °C.
- Preparar amostras de controlo negativo adicionando 1 ml de BPW estéril.
4. Processamento de amostras
- Adicione 225 mL de BPW a cada amostra. A proporção de volume para mídia foi selecionada para se alinhar com o FSIS MLG 4.154.
- Homogeneizar as amostras utilizando o Stomacher7 à velocidade normal e com uma duração de 120 s.
- Centrifugação e ressuspensão
- Remova cuidadosamente o líquido do lado filtrado do saco usando uma pipeta de 50 mL. Divida o líquido em dois frascos de centrífuga estéreis.
- Equilibre os frascos com BPW estéril para garantir pesos iguais. Centrifugue as amostras a 10.000 x g por 10 min. Descarte o sobrenadante. Ressuspenda o pellet celular em 3 mL de caldo BPW com uma espátula estéril.
- Adicione mais 27 mL de caldo BPW e misture bem, mexendo com uma espátula. Combine o conteúdo de ambos os frascos de centrífuga em um frasco para cada amostra.
5. Configuração de bloco MPN
NOTA: A Tabela 1 mostra um esquema das diluições em um bloco de 48 poços.
- Adicionar 3 ml da amostra ressuspensa a cada alvéolo na coluna 1 do bloco de 48 poços (8 réplicas).
- Prepare uma série de diluições de 10 vezes nas colunas 1-6 dentro do bloco usando uma pipeta de oito canais.
- Adicione 0,3 mL de amostra em 2,7 mL de pipeta BPW para misturar. Repita para cada diluição. Incubar os blocos durante a noite (~18 h) a 37 °C com agitação a ~100 rpm.
6. Chapeamento e enumeração
- Enumeração de placa suspensa modificada
- Placa 7 μL de amostra cultivada durante a noite de cada diluição em uma grade 4 x 6 em uma placa de ágar usando uma pipeta multicanal (Figura 2). O uso de uma grade 4 x 6 em duas placas acomoda melhor 8 amostras, em oposição à grade típica de 6 x 6 de gotículas8.
- Deixe as placas secarem ao ar por 10 minutos antes da incubação. Incubar as placas de ágar-ágar durante a noite (~18-24 h) a 37 °C. Após a incubação, conte o número de colônias em cada placa.
7. Detecção de qPCR de Salmonella
- Extração de DNA usando um kit comercial
- Misture culturas no bloco de 48 poços pipetando para cima e para baixo várias vezes. Pipete 200 μL de cada cultura em uma placa de PCR de 96 poços.
- Sele e centrifugue a placa a 6.600 x g por 10 min. Remova o sobrenadante e adicione 20 μL do reagente do kit ao pellet.
- Ressuspenda o pellet pipetando para cima e para baixo. Sele e aqueça a placa a 99 °C por 10 min, seguido de resfriamento a 20 °C.
- Centrifugue novamente a 6.600 x g por 10 min. Use 2 μL do sobrenadante para análise de qPCR.
- Configuração da placa
- Preparar a mistura de reação qPCR de acordo com o protocoloestabelecido 9 da seguinte forma: 10 μL de 2x Master Mix; 0,4 μL de cada primer e sonda (solução de trabalho de 10 μM): invA direto: 5'-GTTGAGGATGTTATTCGCAAAGG-3', invA reverso: 5'-GGAGGCTTCCGGGTCAAG-3', sonda invA: 5'-CCGTCAGACCTCTGGCAGTACCTTCCTC-3' marcado com o corante de fluorenos Cal Fluor Orange 560; 0,2 μL de modelo de controle de amplificação interna (IAC) (6 x 104 cópias/μL)9; 0,4 μL de cada primer e sonda IAC (10 μM): IAC direto: 5'-GGCGCGCCTAACACATCT-3', IAC reverso: 5'-TGGAAGCAATGCCAAATGTGTA-3', sonda IAC: 5'-TTACAACGGGAGAAGACAATGCCACCA-3' marcado com corante TAMRA. Ajuste o volume com ddH2O para 20 μL no total.
- Realize PCR em tempo real com as seguintes condições de ciclagem9: 95 °C por 10 min (desnaturação inicial do DNA e ativação da polimerase de partida a quente), 40 ciclos de 95 °C por 15 s e 60 °C por 1 min, use as configurações padrão de Ct para exportar os resultados para análise.
8. Detecção usando o ensaio 3M MDS
- Siga o protocolo do kit de Salmonella do ensaio de detecção molecular. Misture culturas no bloco de 48 poços pipetando para cima e para baixo várias vezes. Pipete 20 μL de cada amostra no tubo de lise fornecido pelo kit.
- Aquecer as amostras a 100 °C durante 15 min. A solução mudará de rosa para amarelo. Incube as amostras por 10 min em temperatura ambiente. A solução mudará de amarelo para rosa.
- Transfira 20 μL de lisado para um tubo de reagente e carregue os tubos de reagente no suporte.
- Adicione o suporte ao instrumento MDS e configure o software para comunicar as informações sobre o kit e a amostra. O instrumento exige que cada poço seja rotulado com o número do lote do ensaio e um nome de amostra. Execute o software MDS e exporte o relatório.
9. Análise dos dados
- Classificação de resultados positivos e negativos.
- Para placas de gota 4 x 6, avalie as manchas nas placas de ágar com pelo menos 1 colônia como positivas e as manchas nas placas de ágar sem crescimento como negativas.
- Para qPCR, avalie os poços que têm um Ct menor ou igual a 30 como positivo e os poços que têm um Ct maior que 30 como negativo.
- Para SMD, use os resultados do sistema MDS, relatados como positivos ou negativos.
- Cálculo do MPN
- Analise positivos e negativos anotados usando o método de resolução de probabilidade máxima simples (SMPR) descrito anteriormente6 ou calculadoras MPN verificadas alternativas. 10
Resultados
Carne irradiada
Na análise de regressão, uma inclinação de 1 indica que para cada aumento de unidade na variável independente (eixo x), a variável dependente (eixo y) aumenta exatamente 1 unidade. Isso sugere uma relação proporcional entre as duas variáveis, o que significa que a mudança na variável dependente reflete a mudança na variável independente. Uma interceptação de 0 significa que quando a variável independente é 0, a variável dependente também é 0. Isso sugere que não há deslocamento fixo ou viés na relação entre as duas variáveis. Juntos, uma inclinação de 1 e um intercepto de 0 sugerem concordância entre as variáveis11,12. Esse cenário ideal não indica nenhum erro ou viés sistemático e seria o resultado esperado em um sistema bem calibrado, onde as medições se alinham perfeitamente com as previsões.
A Figura 3A apresenta o ensaio MPN-LAMP (eixo y) plotado em relação ao inóculo teórico verificado pela análise da placa de espalhamento discutida na nota da etapa 3.1.1. (eixo x). A equação da reta de regressão, y = -72,36 + 0,161x, com um valor de R² de 0,978, indica uma forte correlação linear entre os resultados do MPN-LAMP e os níveis bacterianos inoculados. O alto valor de R² sugere que o ensaio MPN-LAMP é altamente confiável na previsão quantitativa de contaminação bacteriana. No entanto, a inclinação de 0,161 desvia-se significativamente da inclinação ideal de 1, indicando que o método MPN-LAMP subestima o inóculo, prevendo apenas 16,1% da concentração bacteriana no inóculo teórico. Além disso, a interceptação negativa de -72,36 sugere um viés negativo em concentrações mais baixas, desviando-se ainda mais do ideal.
A Figura 3B ilustra o método MPN-qPCR (eixo y) plotado em relação ao inóculo teórico (eixo x). A reta de regressão, y = -46,84 + 0,0395x, com um valor de R² de 0,978, demonstra uma forte relação linear entre o ensaio MPN-qPCR e o inóculo teórico. Embora o alto R² indique uma correlação robusta, a inclinação de 0,0395 é ainda menor do que a dos resultados do MPN-LAMP, sugerindo um maior grau de subestimação na contaminação bacteriana por MPN-qPCR. A interceptação negativa de -46,84 indica ainda um potencial viés de queda em concentrações mais baixas.
A Figura 3C apresenta os resultados do MPN-plating (eixo y) plotados em relação ao inóculo teórico (eixo x), com uma equação de regressão de y = -330,4 + 0,3138x e um valor de R² de 0,944. Embora esse valor de R² indique uma forte relação linear, ele é ligeiramente inferior aos resultados de MPN-LAMP e MPN-qPCR, sugerindo maior variabilidade na quantificação de colônias. A inclinação de 0,3138, embora maior do que as inclinações observadas nos outros métodos, ainda indica uma subestimação da concentração bacteriana em relação ao inóculo teórico. Além disso, a interceptação negativa de -330,4 aponta para um viés considerável, especialmente em concentrações mais baixas, o que pode indicar possíveis imprecisões nas técnicas de plaqueamento ou manuseio de amostras. Essa ligeira redução no R² e a variabilidade observada podem indicar que o processo tradicional de plaqueamento é mais variável do que os métodos moleculares.
Métodos de comparação
Todos os três métodos (MPN-LAMP, MPN-plating e MPN-qPCR) mostraram uma tendência a subestimar os níveis bacterianos em relação à inoculação teórica. Essa subestimação pode ser atribuída a problemas de recuperação celular, onde a Salmonella pode não se desprender totalmente da superfície do frango ou do material particulado durante o processamento, reduzindo sua transferência para o enxágue ou ensaio. Como a Salmonella foi inoculada diretamente no frango, uma parte das células bacterianas pode se ligar fortemente à superfície, limitando a recuperação na etapa de enxágue. Além disso, algumas células podem permanecer presas ao material particulado no pellet após a centrifugação, diminuindo ainda mais a recuperação. Abordagens como tratamentos enzimáticos, processamento mecânico ou protocolos otimizados de estômago podem aumentar o descolamento bacteriano. Essas estratégias são discutidas mais adiante na seção de discussão, enfatizando seu potencial para melhorar as taxas de recuperação
A Figura 4A apresenta a recuperação no eixo y em relação a três métodos MPN no eixo x. A porcentagem de recuperação, calculada como a razão entre o valor do NMP e o nível teórico de inoculação, produziu os seguintes resultados: o método MPN-LAMP teve uma recuperação média de 15,19% com um desvio padrão de 10,04%; o método de MPN-plating teve uma recuperação média de 13,13% com um desvio padrão de 11,45%; e o método MPN-qPCR teve uma recuperação média de 6,67% com um desvio padrão de 3,19%. Um teste de Tukey-Kramer HSD não identificou uma diferença estatisticamente significativa na recuperação entre os métodos (P > 0,1969), sugerindo que todos os três métodos tiveram desempenho semelhante em termos de recuperação ao processar frango inoculado com Salmonella.
Apesar do tratamento idêntico entre os métodos de separação de Salmonella da matriz de frango, foram observadas variações na detecção entre MPN-LAMP, MPN-qPCR e MPN-plating. Isso sugere que fatores além da separação física das bactérias da amostra podem afetar a detecção. A presença de inibidores na matriz da amostra pode interferir nos métodos moleculares - qPCR e LAMP - afetando sua sensibilidade e desempenho geral. Além disso, a viabilidade bacteriana pode ter impactado a capacidade de Salmonella de crescer em placas de ágar sólido (revestimento MPN) versus caldo líquido (qPCR, LAMP), levando a variação na recuperação entre os métodos.
Para explorar ainda mais a sensibilidade dos métodos, primeiro examinamos o número de respostas positivas em cada diluição para MPN-LAMP, MPN-qPCR e MPN-plating em sete ensaios independentes (Tabela 2). Em diluições mais baixas (1 x 10-5 e 1 x 10-4), o MPN-LAMP demonstrou um maior número de respostas positivas em comparação com o MPN-plating e o MPN-qPCR, sugerindo que o método LAMP é mais sensível a baixas concentrações de Salmonella. Quando as amostras estavam mais concentradas (diluições inferiores a 1 x 10-3), todos os três métodos mostraram um aumento nas detecções positivas, com a convergência ocorrendo em torno da diluição 1 x 10-2 , onde todos os métodos detectaram um número semelhante de amostras positivas.
A Figura 4B apresenta a porcentagem de medições positivas em cada diluição nos três métodos, fornecendo uma comparação direta de sua sensibilidade na detecção de Salmonella. A porcentagem de respostas positivas oferece uma visualização mais clara do desempenho do método em toda a série de diluição, e as barras de erro que representam ± intervalos de confiança (IC) de Clopper-Pearson de 95% fornecem informações sobre a variabilidade em cada nível. Em diluições mais baixas, os intervalos de confiança mais amplos indicam maior variabilidade no desempenho de detecção de cada método. Consistente com o número bruto de detecções positivas, o MPN-LAMP detecta uma porcentagem maior de amostras positivas nas concentrações mais diluídas (1 x 10-5), enquanto o MPN-qPCR e o MPN-plating detectam menos positivos. À medida que a concentração bacteriana aumenta, os métodos convergem, com todas as três abordagens mostrando quase 100% de detecção em concentrações mais altas (por exemplo, 1 x 10-2 e acima). O estreitamento dos intervalos de confiança nessas concentrações mais altas indica maior consistência em todos os métodos, enfatizando seu desempenho comparável quando os níveis bacterianos são mais altos.
Os resultados na Figura 3 sugerem que a subprevisão consistente (quantitativamente) de todos os três métodos pode ser impulsionada pela separação incompleta de Salmonella da matriz da amostra. No entanto, inibidores moleculares ou fatores de viabilidade bacteriana podem estar impulsionando a variação observada na detecção em diferentes diluições (qualitativamente) e em diferentes métodos (qualitativa e quantitativamente). Os intervalos de confiança mais apertados em concentrações mais altas na Figura 4B indicam menos variabilidade e desempenho qualitativo mais consistente entre os métodos, enquanto os intervalos mais amplos em concentrações mais baixas apontam para maior incerteza na detecção qualitativa.
Detecção em frango pronto para cozinhar
Selecionamos o método LAMP para a detecção de Salmonella em frango pronto para cozinhar, pois apresentou um número maior de amostras positivas (14,3%) do que outros métodos testados na menor diluição de Salmonella . Na diluição mais baixa de Salmonella (1 x 10-5), nenhuma das amostras foi detectada como positiva pelo método interno de qPCR. Por outro lado, alguns positivos foram detectados por plaqueamento (7,1%, metade do número detectado pelo LAMP). No entanto, os resultados desse método baseado em cultura podem ser afetados ao passar para produtos mais complexos, como frango pronto para cozinhar, onde a presença de microflora de fundo pode distorcer os resultados porque as amostras não são irradiadas. Por esses motivos, decidimos investigar o método MPN-LAMP para quantificação de Salmonella de frango pronto para cozinhar.
A Figura 5 mostra os resultados do ensaio MPN-LAMP usado para quantificar Salmonella em amostras de cordon bleu de frango inoculadas, congeladas e prontas para cozinhar. O nível teórico de inoculação (UFC/g) foi plotado no eixo x, enquanto as medidas correspondentes do MPN-LAMP foram plotadas no eixo y. A análise de regressão resultou na equação y = -15,04 + 0,3869x, com coeficiente de determinação (R²) de 0,933, indicando uma forte correlação linear entre o inóculo teórico e os resultados do MPN-LAMP. Este alto valor de R² sugere que o método MPN-LAMP quantifica de forma confiável a Salmonella nessas amostras, embora a inclinação de 0,3869 indique que o método subestima o nível real de inóculo. A área sombreada ao redor da linha de regressão representa o intervalo de confiança de 95%.
Para ilustrar melhor o desempenho do MPN-LAMP em níveis de inoculação mais baixos, o gráfico inserido na Figura 5 amplia os pontos de dados abaixo de 300 UFC/g. Com base na equação de regressão, o limite inferior de detecção pode ser estimado em aproximadamente 40 UFC/g (38,9 UFC/g). No entanto, o intervalo de confiança de 95% sugere que o limite inferior prático de detecção está mais próximo de 325 UFC/g, pois quaisquer valores negativos de MPN-LAMP devem ser interpretados como zero. A linha vermelha na inserção representa zero e, embora nenhum ponto de dados tenha caído abaixo dessa linha, o potencial de os valores caírem abaixo de zero, particularmente em níveis mais baixos de inóculo, introduz a possibilidade de resultados falso-negativos. Essa tendência destaca as dificuldades de quantificar com precisão as Salmonella em baixas concentrações, e dados adicionais seriam necessários para estabelecer definitivamente o limite inferior de detecção.

Figura 1: Diagrama de blocos do ensaio MPN modificado para detecção de salmonela. Uma visão geral esquemática do ensaio modificado do Número Mais Provável (MPN). Ele descreve as principais etapas do processo, começando com a inoculação de amostras de frango moído irradiadas, seguidas pela homogeneização da amostra, centrifugação, distribuição de alíquotas e incubação sob condições especificadas. A figura inclui fotografias do bloco de 48 poços utilizado para diluição e crescimento replicado, juntamente com três técnicas usadas para produzir resultados positivos e negativos para o cálculo da Resolução de Probabilidade Máxima Simples (SMPR). Clique aqui para ver uma versão maior desta figura.

Figura 2: Esquema do ensaio da placa de gota. A configuração do ensaio de placa de queda para uma diluição específica (por exemplo, não diluída, 10-1, 10-2, etc.) do bloco de 48 poços. Cada amostra é plaqueada em um formato de grade 4 x 6 em uma placa de ágar usando uma pipeta multicanal. O layout foi modificado da grade tradicional 6 x 6 para duas grades 4 x 6 para garantir que seis réplicas de oito diluições pudessem ser acomodadas. Clique aqui para ver uma versão maior desta figura.
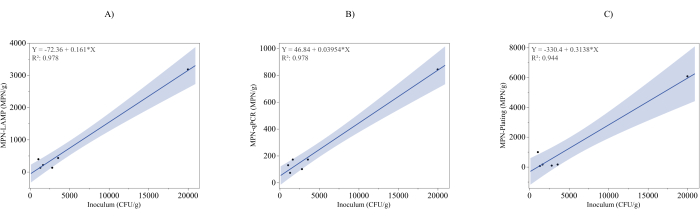
Figura 3: Comparação do inóculo com os métodos MPN. (A) Um gráfico da concentração MPN-LAMP (eixo y) em relação ao inóculo teórico verificado por plaqueamento (eixo x). Uma reta de regressão ajustada (y = -72,36 + 0,161x) com coeficiente de determinação (R² = 0,978) demonstra uma forte relação linear. (B) Um gráfico da concentração de MPN-qPCR (eixo y) contra o inóculo teórico (eixo x), com uma linha de regressão ajustada (y = -46,84 + 0,0395x) e R² = 0,978, mostrando uma correlação igualmente forte. (C) Um gráfico da concentração de placas MPN (eixo y) em relação ao inóculo teórico (eixo x), com uma linha de regressão (y = -330,4 + 0,3138x) e R² = 0,944, indicando uma correlação forte, mas ligeiramente menor do que LAMP e qPCR. A equação de regressão e o coeficiente de determinação para cada figura são apresentados como um encarte no canto superior esquerdo dos gráficos. Clique aqui para ver uma versão maior desta figura.

Figura 4: Comparação dos métodos MPN. (A) A figura apresenta a porcentagem de recuperação, calculada como a razão entre o valor do MPN e o nível teórico de inoculação para os métodos MPN-LAMP, MPN-plating e MPN-qPCR. O método MPN-LAMP teve uma recuperação média de 15,19% (±10,04%), o método MPN-plating teve uma recuperação média de 13,13% (± 11,45%) e o método MPN-qPCR teve uma recuperação média de 6,67% (± 3,19%). Os gráficos de caixa destacam a distribuição dos dados de recuperação, com valores discrepantes indicados. As barras de erro representam o intervalo de dados dentro de 1,5 vezes o intervalo interquartil (IQR). (B) Porcentagem de detecções positivas de Salmonella em cada diluição em três métodos: MPN-LAMP (azul), MPN-plating (vermelho) e MPN-qPCR (verde). O eixo x representa os níveis de diluição (10-6 a 10) e o eixo y mostra a percentagem de medições positivas. As barras de erro indicam os intervalos de confiança de Clopper-Pearson de ± 95%, que refletem a variabilidade na detecção de cada método. O MPN-LAMP mostra porcentagens mais altas de detecções do que outros métodos em diluições mais baixas, enquanto todos os métodos convergem para perto de 100% de detecção em concentrações mais altas. Clique aqui para ver uma versão maior desta figura.
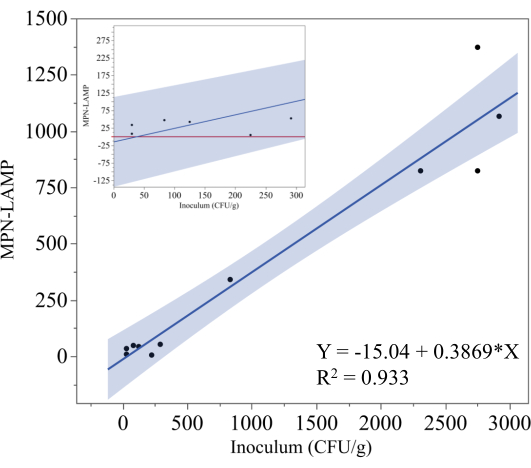
Figura 5: Método MPN para frango pronto para cozinhar. Análise de regressão dos resultados do MPN-LAMP em relação ao nível teórico de inoculação de Salmonella em amostras de cordon bleu de frango congeladas e prontas para cozinhar. A reta de regressão é representada por y = -15,04 + 0,3869x com um R² de 0,933, indicando uma forte relação linear. A região sombreada representa o intervalo de confiança de 95%. A inserção se concentra nas medições MPN-LAMP em níveis de inoculação mais baixos. Clique aqui para ver uma versão maior desta figura.
| 1 | 2 | 3 | 4 | 5 | 6 | |
| Um | n=1 | n=1 | n=1 | n=1 | n=1 | n=1 |
| B | 2 | 2 | 2 | 2 | 2 | 2 |
| C | 3 | 3 | 3 | 3 | 3 | 3 |
| D | 4 | 4 | 4 | 4 | 4 | 4 |
| E | 5 | 5 | 5 | 5 | 5 | 5 |
| F | 6 | 6 | 6 | 6 | 6 | 6 |
| G | 7 | 7 | 7 | 7 | 7 | 7 |
| H | 8 | 8 | 8 | 8 | 8 | 8 |
| 100 | 10-1 | 10-2 | 10-3 | 10-4 | 10-5 |
Tabela 1: Esquema de réplicas e diluições em um bloco de 48 poços. Esta figura ilustra o arranjo de réplicas e diluições seriais de 10 vezes dentro do bloco de 48 poços. Cada coluna representa um factor de diluição específico, que varia entre 100 e 10-5 , com replicações técnicas para cada diluição distribuídas pelas oito linhas.
| Diluição | ||||||
| 10-5 | 10-4 | 10-3 | 10-2 | 10-1 | 100 | |
| MPN-LÂMPADA | 8 | 16 | 31 | 3 | 56 | 56 |
| MPN-chapeamento | 4 | 11 | 32 | 55 | 56 | 56 |
| MPN-qPCR | 0 | 7 | 26 | 55 | 56 | 56 |
Tabela 2: Número de eventos positivos. A tabela exibe o número de reações positivas (mais de 56 réplicas no total). Os resultados são organizados em uma tabela onde as colunas representam a diluição e as linhas são representativas do método.
Discussão
Significado do protocolo
A salmonela continua sendo uma grande preocupação na segurança alimentar, particularmente em produtos avícolas, que são frequentemente implicados em surtos de doenças transmitidas por alimentos13,14. Como uma das principais causas de doenças bacterianas transmitidas por alimentos nos Estados Unidos, métodos confiáveis para detectar Salmonella em produtos avícolas frescos e prontos para cozinhar são essenciais para garantir a segurança alimentar15. A capacidade de quantificar a Salmonella em níveis baixos é vital para identificar a contaminação que, de outra forma, poderia passar despercebida. A sensibilidade do método é particularmente importante para a conformidade regulatória e a saúde pública, pois mesmo baixas doses de Salmonella podem resultar em doenças, especialmente em populações vulneráveis.
O ensaio MPN-LAMP utiliza o sistema MDS, que atualmente é usado pelo FSIS no MLG 4.15, para determinar a prevalência de contaminação4. O ensaio aqui apresentado fornece uma extensão do método FSIS atual para quantificar Salmonella viável, mesmo em níveis baixos, fornecendo dados mais precisos sobre o nível de contaminação. Os resultados deste estudo demonstraram uma forte correlação entre o método MPN-LAMP e os valores teóricos de inoculação, com um R² de 0,933 (Figura 5). Isso sugere que o método é confiável para quantificar Salmonella em alimentos, embora tenha ocorrido alguma subestimação em níveis mais baixos de inoculação.
Etapas críticas
O protocolo para quantificação de Salmonella requer precisão na preparação da amostra, inoculação e análise molecular. A técnica asséptica adequada, diluições precisas e aderência às condições de incubação são essenciais para a detecção e enumeração confiáveis de Salmonella. Garantir condições estéreis durante a transferência de amostras e meios de aves é essencial para evitar a contaminação.
Igualmente importante é a homogeneização das amostras durante a estomacalização, seguida de uma ressuspensão cuidadosa do pellet após a centrifugação para separar as bactérias da matriz alimentar. O manuseio adequado durante essas etapas garante que as células viáveis sejam recuperadas para análise posterior. A pipetagem precisa durante o processo de diluição em série para a configuração do MPN, juntamente com condições de incubação consistentes, é crucial para a obtenção de resultados reprodutíveis.
Finalmente, os métodos de detecção molecular, como qPCR, requerem precisão e reprodutibilidade na extração de DNA e ciclagem de qPCR para evitar falsos negativos. Embora a extração de DNA seja inerentemente variável, reagentes e procedimentos padronizados garantem a repetibilidade, minimizando a interferência do inibidor e melhorando a recuperação do DNA. Essas etapas são fundamentais para confirmar a presença de Salmonella e para direcionar marcadores genéticos específicos que podem ajudar a superar as limitações nos métodos tradicionais de plaqueamento e enriquecimento.
Possíveis armadilhas e limitações
Viabilidade bacteriana
Um dos principais desafios da implementação de um ensaio baseado em crescimento, como o MPN-plating, é a viabilidade bacteriana, e isso é especialmente relevante para amostras complexas de frango prontas para cozinhar. Produtos de frango congelados, empanados e recheados, como frango cordon bleu, são particularmente propensos a flutuações de temperatura e períodos prolongados de armazenamento, os quais podem estressar as células de Salmonella e tornar o patógeno mais difícil de detectar em ensaios baseados em crescimento. Além disso, a presença de ingredientes não derivados de frango (por exemplo, queijo, empanado, presunto, especiarias, manteiga) introduz mais complexidade na matriz alimentar e pode inibir o crescimento de Salmonella durante as fases de enriquecimento, complicando a quantificação precisa. Além disso, a Salmonella deve competir contra micróbios de fundo em métodos baseados em cultura16, o que é especialmente relevante para amostras prontas para cozinhar com vários ingredientes, o que adiciona diversidade à microbiota em relação ao frango cru. Finalmente, os estressores de manuseio e processamento, como antimicrobianos, podem influenciar a recuperação de Salmonella 17.
A viabilidade bacteriana pode ter impactado o desempenho dos métodos testados neste estudo. Todos os métodos subestimaram as concentrações de Salmonella em níveis baixos e tiveram maior variabilidade de detecção em concentrações mais baixas de patógenos (Figura 4A, B). No entanto, o método MPN-LAMP de base molecular superou o MPN-plating e o MPN-qPCR na detecção qualitativa em níveis de inoculação mais baixos. Os resultados enfatizam a necessidade de refinamento em baixas concentrações de patógenos.
Heterogeneidade da amostra
Outra limitação importante envolve a heterogeneidade das amostras de alimentos, o que pode introduzir variabilidade nos resultados de detecção de Salmonella . O protocolo foi projetado para dispersar 30 mL de amostra homogeneizada, permitindo a distribuição de 8 repetições como alíquotas de 3 mL (24 mL total, 6 mL em excesso) através do bloco. Embora essa abordagem conservadora (resultando em uma amostra extra) seja responsável por perdas potenciais, ela introduz subamostragem, pois todo o volume não é testado.
A heterogeneidade da amostra de alimento pode afetar a precisão da medição ao transferir amostras da mistura redispersa para o bloco e realizar diluições18,19. De acordo com a Teoria da Amostragem (TOS), os erros fundamentais de amostragem podem ser reduzidos aumentando a massa da amostra ou reduzindo a heterogeneidade composicional por meio da redução do tamanho das partículas20. No entanto, embora alguns estudos indiquem que a quebra da matriz por estômago não afeta significativamente a recuperação bacteriana da contaminação da superfície, a redução do tamanho das partículas da matriz alimentar demonstrou diminuir a recuperação bacteriana em alguns casos 21,22,23. Neste estudo, o tamanho das partículas foi reduzido pelo estômago antes do enriquecimento; Embora isso possa reduzir o erro de amostragem, reduzindo a heterogeneidade composicional nas amostras, o estômago e a centrifugação podem afetar a recuperação bacteriana.
Finalmente, a composição e morfologia heterogêneas do alimento, bem como se a contaminação é interna ou superficial, podem levar à variabilidade na recuperação do patógeno. Trabalhos anteriores 18,22 demonstraram que a forte ligação bacteriana às superfícies dos tecidos pode reduzir as taxas de recuperação. Estudos em que enzimas como tripsina, colagenase e endopeptidase foram usadas para liberar bactérias de matrizes de carnes e aves24,25 e pectinase e celulase em folhas verdes19 demonstram o potencial de melhorar a recuperação de Salmonella de matrizes alimentares.
Sugestões para solução de problemas
Para evitar problemas, certifique-se de que todos os meios e reagentes estejam preparados e estéreis. Materiais vencidos ou esterilizados incorretamente podem levar à contaminação ou afetar o crescimento de Salmonella. Se for observada contaminação, verifique se a técnica asséptica foi seguida de forma consistente durante todo o protocolo, particularmente durante a inoculação, diluição, alíquota e plaqueamento da amostra.
Quando as contagens de colônias são menores do que o esperado, verificar a precisão das diluições seriadas e das concentrações de inóculo pode ajudar a identificar erros. As condições de incubação, incluindo temperatura, tempo e agitação, devem ser cuidadosamente monitoradas para apoiar o crescimento ideal de Salmonella . Em casos de baixo ou nenhum crescimento, estômago inadequado ou vazamento de bolsas podem contribuir para a distribuição desigual da bactéria.
Se a detecção molecular produzir resultados inconsistentes, verificar os procedimentos de extração de DNA e verificar a qualidade dos primers e sondas são etapas fundamentais. Além disso, a variabilidade entre matrizes alimentares pode apresentar diferentes níveis de inibição, que podem ser monitorados por meio do controle de amplificação interna (IAC). O armazenamento e manuseio adequados de reagentes de extração são cruciais para evitar falsos negativos devido à degradação ou contaminação. Métodos de extração de DNA mais eficientes também podem ser necessários.
A incorporação de tratamentos como o sistema GentleMACS26, que emprega ações mecânicas, térmicas e enzimáticas, pode otimizar ainda mais a recuperação de bactérias dos alimentos. Este sistema pode reduzir a heterogeneidade da matriz alimentar, facilitando a transferência de amostras uniformes para o bloco e a realização de diluições seriadas precisas.
Refinamento e expansão potenciais
Existem várias oportunidades para refinar e expandir este protocolo. A exploração de formulações alternativas de meios de enriquecimento pode melhorar a recuperação de células de Salmonella estressadas ou subletalmente feridas, particularmente de matrizes alimentares complexas, como o cordon bleu de frango. Embora o ágar seletivo seja comumente usado para atingir a Salmonella, as células feridas ou estressadas devido às condições de processamento e armazenamento podem levar a falsos negativos nesses meios. Para superar esse desafio, otimizar o equilíbrio entre pressão seletiva e recuperação microbiana pode melhorar a detecção de sorovares de Salmonella que são menos competitivos em condições de enriquecimento padrão. Esse refinamento pode melhorar a sensibilidade geral do ensaio, particularmente em produtos alimentícios desafiadores onde o estresse microbiano é comum. Encurtar os tempos de incubação pode acelerar o tempo de obtenção de resultados, embora isso possa reduzir a sensibilidade e limitar a detecção. Trabalhos futuros devem explorar os avanços nos meios de enriquecimento para apoiar a rápida replicação bacteriana, garantindo uma quantificação precisa e atendendo aos requisitos das partes interessadas.
Além disso, investigar diferentes temperaturas e durações de incubação para o ensaio MPN pode otimizar a recuperação de Salmonella de vários produtos avícolas. Isso poderia expandir a aplicabilidade do protocolo a outros patógenos transmitidos por alimentos que requerem condições de crescimento distintas, ampliando assim sua utilidade em testes de segurança alimentar. A automação pode aumentar a escalabilidade desse protocolo integrando manipuladores robóticos de líquidos para etapas de alíquota e diluição. Centrífugas de fluxo contínuo ou sistemas de centrífuga de alta capacidade também podem abordar as limitações de processamento em lote, permitindo aplicações de alto rendimento em ambientes industriais.
Expandir o aspecto de detecção molecular para incluir PCR multiplex pode permitir a detecção simultânea de vários patógenos, melhorando o rendimento em ambientes de vigilância e teste. Além disso, a incorporação do sequenciamento de próxima geração (NGS) pode fornecer informações valiosas de sorotipo e virulência diretamente das culturas de enriquecimento do MPN, facilitando as investigações epidemiológicas e os esforços de rastreamento de fontes.
Por fim, o limite de quantificação/detecção pode ser ampliado aumentando a quantidade de amostras utilizadas no NMP. Por exemplo, se o peso da amostra de carne for aumentado de 25 g para 325 g, o limite de detecção poderá ser extrapolado para 4 UFC/g usando a equação de regressão linear. No entanto, assumindo o mesmo intervalo de confiança relativo de 95%, o limite inferior prático de quantificação é >30 UFC/g.
Conclusão
O desenvolvimento do ensaio MPN-LAMP neste estudo oferece uma ferramenta valiosa e prática para quantificar a contaminação por Salmonella em produtos avícolas. Com uma forte correlação com os níveis teóricos de inoculação (R² = 0,933), o método detecta de forma confiável Salmonella em níveis baixos, o que é crítico para a segurança alimentar e a conformidade regulatória. Esse recurso é especialmente importante na detecção de contaminação que, de outra forma, poderia passar despercebida, permitindo que os produtores abordassem proativamente os riscos potenciais. O ensaio MPN-LAMP fornece uma alternativa quantitativa aos métodos tradicionais baseados em prevalência, oferecendo um nível de contaminação por Salmonella em uma variedade de produtos avícolas, incluindo itens prontos para cozinhar mais complexos.
Um dos principais pontos fortes desse método reside em sua adaptabilidade a diferentes matrizes alimentares, particularmente produtos congelados e processados, como frango cordon bleu, onde os métodos tradicionais podem ter dificuldades devido à presença de ingredientes adicionais, como empanados e queijo. A integração de técnicas moleculares, como LAMP e qPCR, com o ensaio MPN garante uma abordagem abrangente para a detecção, superando os desafios impostos pelos micróbios de fundo e pelas condições de processamento. Esses métodos moleculares aumentam a sensibilidade do protocolo, tornando-o uma ferramenta versátil em testes de segurança alimentar de rotina e vigilância de contaminação, com potencial para reduzir o risco de surtos relacionados à Salmonella.
Embora nenhum método de detecção único seja isento de limitações, o ensaio MPN-LAMP demonstra desempenho consistente em uma variedade de níveis de inóculo e matrizes alimentares. A variabilidade em níveis mais baixos de inoculação pode ser abordada por meio de refinamentos futuros, como otimizar a preparação de amostras e formulações de meios para maximizar a recuperação, mas isso não diminui a robustez geral do protocolo. Ao fornecer detecção precisa de células de Salmonella , mesmo sob condições desafiadoras, o método MPN-LAMP equipa a indústria alimentícia com uma ferramenta inestimável para proteger a saúde pública.
Olhando para o futuro, há espaço para melhorias adicionais, como expandir o ensaio para incluir mais sorovares de Salmonella ou incorporar sequenciamento de última geração para maior caracterização de patógenos. No entanto, este protocolo já representa um avanço significativo na detecção e quantificação de Salmonella , fornecendo um método prático, eficiente e confiável que atende às crescentes demandas de testes de segurança alimentar.
Divulgações
Todos os autores declaram que não há conflito de interesses.
Agradecimentos
Esta pesquisa foi apoiada pelo Departamento de Agricultura dos EUA, Serviço de Pesquisa Agrícola (USDA-ARS), Programa Nacional 108, Sistema de Informação de Pesquisa Atual números 8072-42000-093-000-D e 8072-42000-094-000-D. A menção de nomes comerciais ou produtos comerciais neste artigo é exclusivamente para fins de fornecer informações específicas e não implica recomendação ou endosso do Departamento de Agricultura dos EUA. O USDA é um provedor e empregador de oportunidades iguais.
Materiais
| Name | Company | Catalog Number | Comments |
| 48 deep well block 4.6ml | Fisher Scientific International, Inc | NC1964628 | |
| Agar - Solidifying Agent (Difco) | Becton, Dickinson and Company (BD) | 281230 | |
| Analytical Balance | Mettler Toledo | JL602-G/L | Equipment |
| Analytical Balance | Mettler Toledo | AB54-S | Equipment |
| Autoclave - Amsco Lab250, Laboratory Steam Sterilizer | Steris plc | LV-250 | Equipment |
| Biological Safety Cabinet, Type A2, Purifier Logic+ | Labconco Corporation | 302411101 | Equipment |
| Brain Heart Infusion (BHI) Broth | Becton, Dickinson and Company (BD) | 237500 | |
| Buffered Peptone Water | Bio-Rad Laboratories Inc. | 3564684 | |
| Cell Spreader - L-shaped | VWR | 76208-438 | |
| Centrifuge Microcentrifuge 5424 | Eppendorf | 5424 | Equipment |
| Centrifuge, Avanti J-25 | Beckman Coulter, Inc. | Equipment | |
| DNA Extraction - PreMan Ultra Sample Preparation Reagent | Thermo Fisher Scientific Inc. | 4318930 | |
| Ground Chicken | Local retailers | ||
| IAC forward primer: 5'-GGCGCGCCTAACACATCT-3' | Integrated DNA Technologies | ||
| IAC probe: 5'-TTACAACGGGAGAAGACAATGC CACCA-3' labeled with 5' TAMRA/3' BHQ-2 | Biosearch Technologies | ||
| IAC reverse primer: 5'-TGGAAGCAATGCCAAATGTGTA-3' | Integrated DNA Technologies | ||
| Incubator - Inova 4230 incubator shaker | New Brunswick Scientific | 4230 | Equipment |
| Inoculating Loop - Combi Loop 10µL and 1µL | Fisher Scientific International, Inc | 22-363-602 | |
| invA forward primer: 5'-GTTGAGGATGTTATTCGCAAAG G-3' | Integrated DNA Technologies | ||
| invA probe: 5'-CCGTCAGACCTCTGGCAGTAC CTTCCTC-3' labeled with 5' Cal Fluor Orange 560/3' BHQ-1 | Biosearch Technologies | ||
| invA reverse primer: 5'-GGAGGCTTCCGGGTCAAG-3' | Integrated DNA Technologies | ||
| Irradiation Treatment | Texas A&M Agrilife Research National Center for Electron Beam Research | Service | |
| Luria Bertani (LB) Broth | Becton, Dickinson and Company (BD) | 244620 | |
| Manual pipette Pipet-Lite LTS Pipette L-1000XLS+ | Mettler Toledo | 17014382 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-100XLS+ | Mettler Toledo | 17014384 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-10XLS+ | Mettler Toledo | 17014388 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-200XLS+ | Mettler Toledo | 17014391 | Equipment |
| Manual pipette Pipet-Lite LTS Pipette L-20XLS+ | Mettler Toledo | 17014392 | Equipment |
| Manual pipette Pipet-Lite Multi Pipette L8-200XLS+ | Mettler Toledo | 17013805 | Equipment |
| Manual pipette Pipet-Lite Multi Pipette L8-20XLS+ | Mettler Toledo | 17013803 | Equipment |
| Media Storage Bottle -PYREX 1L Square Glass Bottle, with GL45 Screw Cap | Corning Inc. | 1396-1L | Equipment |
| Media Storage Bottle -PYREX 2L Round Wide Mouth Bottle, with GLS80 Screw Cap | Corning Inc. | 1397-2L | Equipment |
| Microtiter plate, 96 well plate, flat bottom, polystyrene, 0.34cm2, sterile, 108/cs | MilliporeSigma | Z707902 | |
| Mixer - Vortex Genie 2 | Scientific Industries Inc. | SI-0236 | Equipment |
| Molecular Detection Assay 2-Salmonella kit | Neogen | MDA2SAL96 | |
| Molecular Detection Instrument | Neogen | MDS100 | Equipment |
| Motorized pipette controller, PIPETBOY2 | INTEGRA Biosciences Corp. | 155019 | Equipment |
| PCR Mastermix 2× TaqMan Gene Expression | Thermo Fisher Scientific Inc. | 4369542 | |
| Petri Dish Rotator - bioWORLD Inoculation Turntable | Fisher Scientific International, Inc | 3489E20 | Equipment |
| Petri Dishes with Clear Lid (100 mm x 15mm) | Fisher Scientific International, Inc | FB0875713 | |
| Pipette Tips GP LTS 1000µL S 768A/8 | Mettler Toledo | 30389273 | |
| Pipette Tips GP LTS 20µL 960A/10 | Mettler Toledo | 30389270 | |
| Pipette Tips GP LTS 200µL F 960A/10 | Mettler Toledo | 30389276 | |
| Ready to cook chicken products | Local retailers | ||
| Reagent Reservoir, 25 mL sterile reservoir used with multichannel pipettors | Thermo Fisher Scientific Inc. | 8093-11 | |
| Realtime PCR - 7500 Real-Time PCR system | (Applied Biosystems, Foster City, CA) | 2750036476 | Equipment |
| Serological Pipettes, Nunc Serological Pipettes (10 mL) | Thermo Fisher Scientific Inc. | 170356N | |
| Serological Pipettes, Nunc Serological Pipettes (2 mL) | Thermo Fisher Scientific Inc. | 170372N | |
| Serological Pipettes, Nunc Serological Pipettes (25 mL) | Thermo Fisher Scientific Inc. | 170357N | |
| Serological Pipettes, Nunc Serological Pipettes (50 mL) | Thermo Fisher Scientific Inc. | 170376N | |
| Spreader - Fisherbrand L-Shaped Cell Spreaders | Fisher Scientific International, Inc | 14-665-230 | |
| Stomacher bag, Nasco Whirl-Pak Write-On Homogenizer Blender Filter Bags | Thermo Fisher Scientific Inc. | 01-812 | |
| Stomacher 80 Biomaster Lab Blender | Seward | 30010019 | Equipment |
| Thermocycler (GeneAmp PCR system 9700) | Applied Biosystems | 535028293 | Equipment |
| Water Filtration - Elga Veolia Purelab Flex | Elga LabWater | PF2XXXXM1-US | Equipment |
| Whirlpak bags 1.63L | VWR | 11216-777 |
Referências
- Batz, M., Hoffmann, S., Morris, J. G. Disease-outcome trees, eq-5d scores, and estimated annual losses of quality-adjusted life years (qalys) for 14 foodborne pathogens in the united states. Foodborne Pathogens and Disease. 11 (5), 395-402 (2014).
- The Grand Challenge: Salmonella. , United States Department of Agriculture. https://tellus.ars.usda.gov/stories/articles/the-grand-challenge-salmonella (2024).
- National Advisory Committee on Microbiological Criteria in Foods (NACMCF). Response to questions posed by the food safety and inspection service: Enhancing Salmonella control in poultry products. J Food Prot. 82 (4), 645-668 (2019).
- Food Safety and Inspection Service. 4.15 Isolation and identification of Salmonella from meat, poultry, pasteurized egg, siluriformes (Fish) products and carcass and environmental sponges. , (2024).
- Irwin, P., Reed, S., Brewster, J., Nguyen, L., He, Y. P. Non-stochastic sampling error in quantal analyses for campylobacter species on poultry products. Analytical and Bioanalytical Chemistry. 405 (7), 2353-2369 (2013).
- Irwin, P., Tu, S., Damert, W., Phillips, J. A modified gauss-newton algorithm and ninety-six well micro-technique for calculating mpn using excel spreadsheets. Journal of Rapid Methods & Automation in Microbiology. 8 (3), 171-191 (2000).
- Ravishankar, S. Food microbiology: A laboratory manual. Food Microbiology. Ahmed, E. Y., Carlstrom, C. 21, wiley-interscience. singapore. 489(2004).
- Chen, C. Y., Nace, G. W., Irwin, P. L. A 6 x 6 drop plate method for simultaneous colony counting and mpn enumeration of campylobacter jejuni, listeria monocytogenes, and escherichia coli. J Microbiol Methods. 55 (2), 475-479 (2003).
- Suo, B., He, Y., Tu, S. I., Shi, X. A multiplex real-time polymerase chain reaction for simultaneous detection of salmonella spp., escherichia coli o157, and listeria monocytogenes in meat products. Foodborne Pathogens and Disease. 7 (6), 619-628 (2010).
- Jarvis, B., Wilrich, C., Wilrich, P. T. Reconsideration of the derivation of most probable numbers, their standard deviations, confidence bounds and rarity values. J Appl Microbiol. 109 (5), 1660-1667 (2010).
- Stevens, R., Poppe, K. Validation of clinical prediction models: What does the "calibration slope" really measure. Journal of Clinical Epidemiology. 118, (2019).
- Miller, M. E., Hui, S. L., Tierney, W. M. Validation techniques for logistic regression models. Statistics in Medicine. 10 (8), 1213-1226 (1991).
- Galán-Relaño, Á, et al. Salmonella and salmonellosis: An update on public health implications and control strategies. Animals. 13 (23), 3666(2023).
- Gorski, L., et al. Growth assessment of salmonella enterica multi-serovar populations in poultry rinsates with commonly used enrichment and plating media. Food Microbiology. 119, 104431(2024).
- Schmidt, J. W., et al. Evaluation of methods for identifying poultry wing rinses with salmonella concentrations greater than or equal to 10 cfu/ml. J Food Prot. 87 (11), 100362(2024).
- Gorski, A., Liang, L. S. Effect of enrichment medium on real-time detection of salmonella enterica from lettuce and tomato enrichment cultures. Journal of Food Protection. 73 (6), 1047-1056 (2010).
- Guillén, S., Nadal, L., Álvarez, I., Mañas, P., Cebrián, G. Impact of the resistance responses to stress conditions encountered in food and food processing environments on the virulence and growth fitness of non-typhoidal salmonellae. Foods. 10 (3), 617(2021).
- Rohde, A., Hammerl, J. A., Appel, B., Dieckmann, R., Al Dahouk, S. Sampling and homogenization strategies significantly influence the detection of foodborne pathogens in meat. BioMed Research International. 2015, (2015).
- Wang, D., Wang, Z., He, F., Kinchla, A. J., Nugen, S. R. Enzymatic digestion for improved bacteria separation from leafy green vegetables. Journal of Food Protection. 79 (8), 1378-1386 (2016).
- Pitard, F. F. Theory of sampling and sampling practice. , Chapman and Hall/CRC. (2019).
- Sharpe, A. in Detecting pathogens in food. , Elsevier. 52-68 (2003).
- Hannah, J., et al. Effect of stomaching on numbers of bacteria recovered from chicken skin. Poultry Science. 90 (2), 491-493 (2011).
- Mcmeekin, T., Thomas, C. Retention of bacteria on chicken skin after immersion in bacterial suspensions. Journal of Applied Bacteriology. 45 (3), 383-387 (1978).
- Rodrigues-Szulc, U., Ventoura, G., Mackey, B., Payne, M. Rapid physicochemical detachment, separation and concentration of bacteria from beef surfaces. Journal of Applied Bacteriology. 80 (6), 673-681 (1996).
- Vibbert, H. B., et al. Accelerating sample preparation through enzyme-assisted microfiltration of salmonella in chicken extract. Biotechnol Prog. 31 (6), 1551-1562 (2015).
- Armstrong, C. M., et al. Use of a commercial tissue dissociation system to detect salmonella-contaminated poultry products. Analytical and Bioanalytical Chemistry. 416 (3), 621-626 (2024).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados