Method Article
Modelos volumétricos simplificados como estratégia eficaz para segmentar redes de actina em tomogramas crioeletrônicos
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para colocar modelos volumétricos simplificados em volumes 3D tomográficos complexos e ruidosos. Isso permite a segmentação rápida das densidades dos filamentos de actina, a detecção de dobras sistemáticas dos filamentos e de lacunas nos filamentos dos feixes de cabelo, bem como a quantificação conveniente das propriedades do modelo volumétrico, como distâncias.
Resumo
Métodos eficientes para a extração de características de interesse continuam sendo um dos maiores desafios para a interpretação de tomogramas crioeletrônicos. Várias abordagens automatizadas foram propostas, muitas das quais funcionam bem para conjuntos de dados de alto contraste, onde os recursos de interesse podem ser facilmente detectados e estão claramente separados uns dos outros. Nossos conjuntos de dados tomográficos crioeletrônicos de estereocílios do ouvido interno são caracterizados por uma densa matriz de filamentos de actina hexagonalmente compactados que são frequentemente conectados de forma cruzada. Esses recursos tornam a segmentação automatizada muito desafiadora, agravada ainda mais pelo ambiente de alto ruído dos tomogramas crioeletrônicos e pela alta complexidade dos recursos densamente compactados. Usando conhecimento prévio sobre a organização do feixe de actina, colocamos camadas de um modelo de actina de bola e bastão altamente simplificado para primeiro obter um ajuste global ao mapa de densidade, seguido por ajustes regionais e locais do modelo. Mostramos que a construção de modelos volumétricos não apenas nos permite lidar com a alta complexidade, mas também fornece medições e estatísticas precisas sobre o feixe de actina. Os modelos volumétricos também servem como pontos de ancoragem para segmentação local, como no caso dos conectores cruzados actina-actina. A construção de modelos volumétricos, particularmente quando aumentada por abordagens de ajuste automatizadas baseadas em computador, pode ser uma alternativa poderosa quando as abordagens convencionais de segmentação automatizada não são bem-sucedidas.
Introdução
A tomografia crioeletrônica permite que organelas inteiras ou partes de células e tecidos sejam visualizadas em resoluções nanométricas em seu estado quase nativo 1,2,3 usando congelamento por imersão4 ou congelamento de alta pressãoultrarrápido Como apenas uma dose limitada de elétrons pode ser tolerada pela amostra criopreservada, não corada e hidratada congelada, os dados tomográficos 3D são muito ruidosos. Esse ruído geralmente pode ser significativamente reduzido por uma variedade de algoritmos de filtragem de ruído 6,7, incluindo difusão anisotrópica não linear8, filtragem bilateral9 e filtragem mediana recursiva10.
Além disso, as limitações de inclinação do estágio do microscópio, resultando em uma cunha de informações ausentes, e o fato de que a espessura da amostra aumenta em altos ângulos de inclinação, levam a reconstruções 3D com resolução anisotrópica. Isso significa que a densidade é manchada na terceira dimensão devido à menor resolução na direção Z. Como resultado, a forma das macromoléculas parece distorcida (ou seja, menos bem definida e alongada na terceira dimensão).
Entre os maiores desafios na interpretação dos dados tomográficos está a extração automatizada das características relevantes, também conhecida como segmentação11. Com características de forma únicas suficientes e baixo ruído, as máquinas macromoleculares em volumes 3D complexos podem ser identificadas pela correspondência de modelos 12,13,14; No entanto, o sucesso da correspondência de modelos depende da resolução do tomograma, de um modelo de pesquisa adequado, bem como das características de tamanho e forma dos volumes de recursos. Se as características de interesse estiverem suficientemente espaçadas e motivos repetidos (como grandes máquinas macromoleculares) puderem ser prontamente identificados, os subvolumes do tomograma podem ser combinados para aumentar a relação sinal-ruído e calcular a média das distorções individuais da forma das partículas. A segmentação automatizada de uma rede de filamentos de actina em tomogramas eletrônicos da borda fina de células de Dictyostelium discoideum hidratadas congeladas por correspondência de modelos foi relatada15.
No entanto, se as características de interesse estiverem bem espaçadas, a anisotropia da resolução de dados pode levar a uma mancha nas densidades do mapa na direção Z (ao longo da direção do feixe de elétrons), resultando em uma aparente fusão do envelope de densidade de máquinas macromoleculares ou complexos supramoleculares espaçados. Nesses casos, abordagens automatizadas para segmentação, como bacia hidrográfica16, segmentação de limite17 ou uma variedade de abordagens de classificação baseadas em aprendizado de máquina18,19, podem não ser capazes de reconhecer as características de interesse ou estabelecer um limite correto em torno de um objeto de interesse. Muitas vezes, acaba-se com algumas peças muito grandes ou com um volume fortemente segmentado, onde é necessário muito esforço para mesclar muitas peças pequenas até que a característica de interesse seja percebida como completa. Essa curadoria manual dos resultados da segmentação pode ser muito trabalhosa e pode até falhar completamente quando a estrutura de interesse é uma matriz de filamentos espaçados que são interconectados por meio de ligantes curtos. Nessa gigantesca rede de estruturas filamentosas, pode ser difícil se orientar. Isso ocorre porque, devido à anisotropia de resolução, as densidades parecem se misturar, apresentando um desafio formidável tanto para abordagens de segmentação automatizadas quanto para abordagens de segmentação manual interativa. Como consequência, pode-se facilmente "pular" entre os filamentos quando apenas inspecionando visualmente pequenas regiões.
Felizmente, no caso do feixe de actina nos estereocílios das células ciliadas da orelha interna, temos conhecimento sobre a organização geral do feixe de actina e a direcionalidade dos filamentos de actina20,21. O feixe de actina consiste em centenas de filamentos de actina hexagonalmente densamente compactados de 6 a 8 nm de diâmetro, espaçados cerca de 12 a 13 nm um do outro22.
Isso nos permitiu adotar uma abordagem bastante diferente para a segmentação, baseada em modelos simplificados de bola e bastão para representar filamentos de actina. A estratégia envolveu a colocação simultânea de uma matriz regular idealizada de modelos de filamentos em placas dos mapas de densidade de tomografia crioeletrônica para construir um modelo 3D do feixe de actina camada por camada. Garantimos que o modelo tivesse um ajuste geral geral ao mapa de densidade antes de fazer ajustes locais em modelos de filamentos individuais ou grupos de modelos de filamentos para corresponder ao mapa de densidade. Por meio da codificação automática por cores do valor da densidade do mapa no local do modelo do filamento, fomos capazes de detectar facilmente lacunas aparentes no feixe de actina. Os modelos volumétricos permitem uma análise quantitativa das propriedades volumétricas, como distâncias entre filamentos de actina, e também levam a uma exibição simplificada da organização geral da rede filamentosa 3D.
Além disso, os modelos também podem servir como estruturas de ancoragem para a segmentação de recursos adicionais, como ligantes actina-actina, pois (partes de) modelos de filamentos individuais podem ser selecionados, em torno dos quais zonas de densidade de mapa de raio apropriadas podem ser geradas para inspeção e segmentação adicional.
Acreditamos que nossa abordagem de segmentação baseada em modelo volumétrico é particularmente útil para grandes redes de estrutura filamentosa que podem conter lacunas e conexões cruzadas entre filamentos. Os algoritmos de segmentação tendem a operar localmente, enquanto o cérebro humano leva em consideração áreas maiores e, portanto, é superior aos computadores quando se trata de reconhecer estruturas de filamentos, mesmo em um ambiente complexo e de alto ruído.
Protocolo
O protocolo segue as diretrizes do comitê de ética em pesquisa humana da Southeast University.
1. Fonte de dados de tomografia crioeletrônica para construção de modelos volumétricos
NOTA: As reconstruções tomográficas crioeletrônicas dos estereocílios utilizadas para a construção de um modelo volumétrico foram publicadasanteriormente22,23 e foram obtidas conforme descrito por Metlagel et al.22.
Os scripts python UCSF Chimera para modelagem de estereocílios são fornecidos no Arquivo Suplementar 1, Arquivo de Codificação Suplementar 1, Arquivo de Codificação Suplementar 2, Arquivo de Codificação Suplementar 3, Arquivo de Codificação Suplementar 4 e Arquivo de Codificação Suplementar 5.
- Resumidamente, seque os estereocílios da superfície apical do epitélio sensorial do utrículo do camundongo no filme de suporte de carbono rendado de uma grade de microscópio eletrônico (EM). Em seguida, realizar a vitrificação ultrarrápida por congelamento por imersão e a coleta de dados tomográficos crioeletrônicos de eixo único em uma microscopia eletrônica de transmissão criogênica (crio-TEM), operada a 300 kV com desfocagem nominal de 3,5-4,5 μm, usando uma câmera do tipo CMOS em modo de integração, com tamanho de pixel de 0,47 a 0,59 nm (ver Tabela de Materiais).
- Uma dose típica para coleta de dados de eixo único é tipicamente 80-100 elétrons / Å2. Reconstruir os volumes 3D do tomograma usando o pacote de software IMOD24, seja por retroprojeção ponderada ou pelo método SIRT 25,26.
- Reduza o ruído dos tomogramas usando filtragem recursiva mediana ou bilateral no Priism27 ou a opção de difusão anisotrópica não linear no IMOD.
NOTA: O principal pacote de software usado para visualização interativa, construção de modelos volumétricos e análise quantitativa foi o software UCSF Chimera28,29. Todos os pacotes de software usados estão listados na Tabela de Materiais.
2. Preparação de dados de tomografia crioeletrônica para construção de modelos volumétricos
- Rotação do tomograma
NOTA: O objetivo desta etapa é encontrar os "ângulos ideais" nos quais o núcleo de actina está alinhado em dois dos três eixos (x e z), para que a colocação dos modelos em 3D possa ser feita com apenas um eixo para se preocupar. As etapas a seguir são executadas no pacote de software IMOD.- Abra o 3dmod (IMOD) e abra o arquivo de pilha de imagens 3D no formato de arquivo .mrc. Certifique-se de que a pilha 3D esteja no modo de escala de cinza de 16 bits e exiba as dimensões X, Y, Z corretas no cabeçalho do arquivo de imagem. Se necessário, faça correções usando o comando alterheader : alterheader -d (x_pixelspacing),(y_pixelspacing),(z_pixelspacing) inputfilename.mrc no prompt de comando. Se a pilha 3D estiver no formato de arquivo TIFF, use o comando tif2mrc inputfilename.tif outputfilename.mrc no prompt de comando para criar um arquivo .mrc.
- Inspecione visualmente o tomograma e abra o "fatiador" (pressionando a barra invertida "\") para encontrar os melhores ângulos de rotação nas dimensões X, Y e Z para alinhamento do plano do filamento de actina com o plano Z. Manipule a rotação X, rotação Y, barra de rotação Z para descobrir o ângulo ideal no qual os filamentos de actina estão alinhados nos planos X e Z. Observe que alterar a espessura média para a exibição do tomograma (Grosso: Img) pode ajudar a encontrar uma quantidade ideal de média da densidade no plano Z e, portanto, aumentar o contraste. Aumente e diminua o tomografia utilizando as teclas + e -, respectivamente, no teclado.
- Uma vez identificados os ângulos ideais de rotação, gire o tomograma digitando o comando IMOD rotatevol -a (Ângulos de rotação Z),(Ângulos de rotação Y),(Ângulos de rotação X) -s (x-width),(y-height),(z-depth) inputfilename.mrc outputfilename.mrc no prompt de comando. Certifique-se de dar ao mapa bastante espaço para acomodar a rotação para evitar cortar partes do mapa durante a rotação.
NOTA: Para obter mais informações sobre como usar o comando rotatevol, use a opção no menu de ajuda ou visite o seguinte URL (https://bio3d.colorado.edu/imod/doc/man/rotatevol.html). Este comando (rotatevol) não altera o mapa atualmente mostrado na janela IMOD, mas cria um novo arquivo de mapa com um mapa de densidade sendo girado. - Usando a janela de segmentação, identifique as coordenadas X, Y e Z de dois cantos opostos de um mapa para identificar a região de corte que contém o stereocílio usando o comando trimvol .
- Use o comando IMOD Trimvol para cortar o mapa nas coordenadas obtidas anteriormente, digitando trimvol -x (x-coordiante 1),( x-coordiante 2) -y (y-coordiante 1),( y-coordiante 2) -z (z-coordiante 1),( z-coordiante 2) inputfilename.mrc outputfilename.mrc no prompt de comando. Agora, o mapa é muito menor e, portanto, mais fácil e rápido de trabalhar a partir deste ponto.
NOTA: Para obter mais informações sobre como usar o comando trimvol, use a opção no menu de ajuda ou visite o seguinte URL (https://bio3d.colorado.edu/imod/doc/man/trimvol.html). Essas etapas criam um arquivo .mrc que foi girado e cortado.
- Filtragem de tomograma
NOTA: Esta etapa usa um filtro de difusão anisotrópica não linear (NAD) fornecido pelo IMOD para redução de ruído. Usamos como valor inicial os recomendados na página de ajuda do IMOD para filtragem de difusão anisotrópica não linear, conforme listado na URL a seguir (https://bio3d.colorado.edu/imod/doc/NADexample.html), e foi usado como os valores iniciais.- Digite etomo no prompt de comando para iniciar a interface gráfica do usuário etomo do IMOD.
- Selecione a opção Difusão anisotrópica não linear no menu etomo e selecione o arquivo de mapa (Escolha um volume).
- Para encontrar um valor K e o número de iterações que fornecem os melhores resultados de filtragem, conforme julgado por inspeção visual, aplique o filtro NAD a um volume de teste menor para extrair o volume de teste do filtro NAD especificado. Clique na ferramenta Rubberband na parte superior da interface do 3dmod, clique com o botão esquerdo do mouse para selecionar o volume de teste e clique nos botões Hi e Lo para indicar que a fatia Z é o limite do volume de teste (Obter intervalo de volume de teste do 3dmod). Extraia o volume de teste clicando em Extrair Volume de Teste.
- Filtre o volume de teste usando vários valores K (Localizar um valor K para volume de teste > Lista de valores K), como 0,1, 1,5, 10, 15, 25, 30, 50 e 75. Execute a filtragem NAD com cada um dos valores K especificados (Executar com diferentes valores K > Exibir diferentes resultados do teste de valores K). Use a janela de segmentação para avaliar o desempenho do filtro NAD e selecione um valor K que será usado para filtrar todo o volume.
- Use o valor K identificado na etapa 2.2.4 e teste diferentes números de iterações, como 2, 5, 8, 11, 15 e 21 (Localizar um número de iteração para o volume de teste > Lista de iterações). Avalie o desempenho do filtro NAD para diferentes iterações clicando em Exibir resultados de teste de iteração diferentes ou usando uma ferramenta de segmentação. Escolha um valor para um número diferente de iterações.
- Use o valor K identificado e o número de iterações (Filtrar Volume Completo > Valor K e Iterações) e filtre todo o volume (Filtrar Volume Completo). Um volume recém-filtrado aparecerá no mesmo diretório em que o volume completo carregado está localizado com uma extensão de arquivo .nad anexada ao arquivo .mrc.
NOTA: Essas etapas criam um arquivo .mrc que foi filtrado com um filtro NAD e exibe um ruído menor e, portanto, uma relação sinal-ruído aprimorada.
3. Construção de modelos volumétricos
- Preparação de posicionamento de modelo 3D
NOTA: O objetivo desta etapa é preparar e analisar o mapa para colocar modelos volumétricos 3D no mapa de densidade girado e sem ruído usando o programa UCSF Chimera.- Usando a janela do fatiador, obtenha uma vista transversal (laje de 30 fatias/28,4 nm) para identificar as coordenadas Z do centro da laje de densidade, na qual o modelo de filamento de actina deve ser colocado. Clique no centro da densidade do filamento e observe as coordenadas Z na janela principal do IMOD.
- Anote as coordenadas identificadas para a futura colocação do modelo no UCSF Chimera.
NOTA: As coordenadas Z observadas nesta etapa são usadas durante a etapa 4 para encontrar e colocar facilmente modelos de filamentos de actina pré-fabricados nas alturas Z corretas, tornando o posicionamento do modelo mais rápido.
- Posicionamento do modelo 3D
NOTA: O objetivo desta etapa é colocar um modelo volumétrico 3D no mapa de densidade preparado usando o UCSF Chimera.- No UCSF Chimera, abra o mapa filtrado e girado (Arquivo > Abrir)
- Verifique se os parâmetros do mapa estão definidos corretamente selecionando Feições > Planos e Coordenadas de > de Feição no Visualizador de Volume para abrir a interface de feição e coordenadas. Verifique o tamanho do voxel para ver se o espaçamento correto do voxel está definido usando o comando header no prompt de comando (no IMOD). Caso contrário, corrija a interface Voxel Size para o espaçamento correto do voxel. Centralize o mapa para colocar a câmera no centro da janela de visualização (Índice de Origem > Centro).
- Abra a janela Controle da câmera (Ferramentas > Controle de visualização > Câmera). Em seguida, defina a visualização da câmera para a visualização ortogonal (projeção > ortográfica)
- Carregue o modelo de script python pré-fabricado (Arquivo > Abrir > ActinFilamentPlane.py) e abra tantos scripts de plano de filamento de actina quantos forem os planos de filamento de actina no mapa.
- Realoque os modelos carregados ativando o Modo de Movimento do Mouse (Ferramentas > Movimento > Modo de Movimento do Mouse). Ative a opção de movimento do mouse (Definir modo de movimento do mouse > Seleção de movimento). Use Ctrl + Arrastar para selecionar vários modelos presentes na sessão e clique com o botão direito do mouse (ou qualquer outra tecla especificada em Preferências > Mouse) para mover os modelos na direção desejada.
NOTA: Os modelos foram criados com bolas e palitos com certa espessura (raio de ActinFilamentPlane.py script) e com certos espaçamentos (Lattice_Spacing de ActinFilamentPlane.py script). - Para visualizar apenas o modelo em que se está interessado, abra o Model Panel (Ferramentas > Controles Gerais > Model Panel). Clique nas caixas de seleção em S (Mostrar) para ativar ou desativar a visibilidade de um determinado modelo.
- Ative o painel da linha de comando (Ferramentas > Controles Gerais > Linha de Comando).
- Depois de colocar cada modelo de plano de filamento de actina em sua altura Z adequada, exclua qualquer excesso de filamentos de actina em cada modelo de plano de filamento de actina selecionando a parte excedente dos modelos e digitando del sel (Excluir selecionado) na linha de comando na parte inferior da janela de visualização principal do UCSF Chimera.
- Visualize apenas um plano de filamento de actina por vez, acessando a janela do Painel de Modelo e selecionando e ocultando todos os outros modelos de filamento de actina (Ctrl + Clique > Ocultar).
- Para visualizar partes da densidade do mapa que estão nas proximidades imediatas do modelo de plano de filamento de actina único de interesse, selecione o modelo (Painel Modelo > selecionar) e abra o painel de controle de zona no Visualizador de Volume (Visualizador de Volume > Recursos > Zona). Defina o raio da zona para 100 Å e, em seguida, clique em Zona para zonear o mapa em um raio de 100 Å (= 10 nm) do modelo selecionado. Observe que a dimensão em Quimera é definida em Angstrøm (1 Å = 0,1 nm).
- Se necessário, ajuste o modelo para se ajustar ao mapa de densidade ativando o Modo de Movimento do Rato. Selecione vários modelos que não estejam na posição correta para mover os modelos a serem colocados corretamente dentro do mapa de densidade zoneada.
- Para superar um bug de programação no ActinFilamentPlane.py, execute o script FixingMarkerID.py , que corrige o MarkerID ausente dos "átomos" (bolas no modelo bola e bastão). Selecione todo o modelo e digite runscript FixingMarkerID.py na linha de comando (o script deve ser especificado pelo diretório correto, como C: \ directory \ Script.py).
- Se o mapa de densidade indicar que o filamento de actina é curvo, certifique-se de dividir o filamento de actina reto em várias partes para acomodar a curvatura, selecionando os modelos desejados e digitando runscript dividelinks.py número. Marcadores adicionais (especificados pelo número no comando) são colocados dentro do modelo de actina equidistantes uns dos outros, formando "juntas" onde o modelo pode ser dobrado.
- Ajuste as juntas para refletir a curvatura movendo "juntas" de marcadores adicionais ao longo do mapa zoneado usando o Modo de Movimento do Mouse. Adicione marcadores adicionais ao modelo conforme necessário usando o script dividelinks.py .
- Para verificar se todos os filamentos de actina estão colocados corretamente dentro das densidades de actina, use barras de recorte clicando em Ferramentas > ViewControle > Câmera no menu suspenso UCSF Chimera para abrir a janela Controle da câmera e, em seguida, selecione Side View para ver as duas barras de recorte (planos de recorte próximos e distantes) da câmera. Arraste duas barras amarelas para uma fenda estreita clicando com o botão esquerdo do mouse e arraste a parte de recorte usando o botão do meio do mouse para inspecionar os modelos um pequeno bit de cada vez.
NOTA: O modelo de núcleo de actina de estereocílio está agora completo.
- Segmentação de membrana
NOTA: O objetivo desta etapa é criar um modelo de superfície da membrana dos estereocílios.- A segmentação de superfícies utiliza a ferramenta Traçador de volume (Ferramentas > Dados de volume > Rastreador de volume).
- Na janela Rastreador de volume , clique em Mouse e marque apenas as opções Colocar marcadores em planos de dados e Vincular novo marcador ao marcador selecionado. Isso permite colocar com precisão os marcadores no mesmo plano Z (quando um único plano Z é mostrado). Verifique também Marcadores de lugar usando o botão do meio do mouse na janela Rastreador de volume .
- Crie um novo conjunto de marcadores (Arquivo > Novo conjunto de marcadores). O conjunto de marcadores 1 deve ser criado e selecionado na janela Rastreador de volume .
- Use o Visualizador de volume para exibir planos Z únicos na janela de visualização principal (Eixo para Z > Um.)
- Usando o botão do meio do mouse, trace a membrana de uma extremidade da membrana visível a outra. Todos os marcadores devem ser conectados por links de uma extremidade à outra.
- Desmarque o marcador colocado por último pressionando Ctrl + Clique no plano de fundo onde nenhum item selecionável está localizado. Isso garante que o próximo marcador a ser colocado esteja conectado com links no novo plano, e não com um traço de membrana anterior de outro plano Z.
- Repetir o traçado da membrana para cada 10 nm na mesma direcção do traçado de uma extremidade da membrana visível para o outro lado.
- Quando muitas bandas paralelas de membranas forem segmentadas, vá para a janela Rastreador de volume e clique em Recursos > superfícies. Isso ativa a interface de superfície na janela Rastreador de Volume . Clique em Criar ao lado da interface Superfícies para criar superfícies entre bandas de segmentação de membrana. Isso cria uma aba fina do modelo de superfície que preenche os traços de membrana espaçados.
NOTA: A criação do modelo de superfície da membrana do estereocílio está concluída.
- Modelagem de reticulador
NOTA: O objetivo desta etapa é modelar os reticuladores entre os filamentos de actina.- Os reticuladores também podem ser colocados utilizando a ferramenta Rastreador de volume .
- Na janela Traçador de volume , clique com o botão esquerdo do mouse para marcar somente Colocar marcadores em superfícies e Vincular novo marcador ao marcador selecionado. Em seguida, marque Colocar marcadores usando o botão do meio do mouse na janela Rastreador de volume .
- Visualize apenas as densidades em torno do modelo de plano de filamento de actina único de interesse usando a função de zona. Defina o raio da zona para 100 Å para visualizar o mapa dentro de um raio de 100 Å do modelo selecionado.
- Coloque marcadores nas densidades visíveis dos reticuladores clicando no botão do meio do mouse. Depois que dois marcadores forem colocados (e, portanto, um único reticulador for modelado), certifique-se de desmarcar o segundo marcador para evitar que os links sejam conectados continuamente ao próximo modelo de reticulador.
- Repita as etapas acima para cada plano de filamento de actina e repita todo o processo acima para todas as três direções principais nas quais os reticuladores podem estar localizados. Ao zonear os planos do filamento de actina na direção diagonal, selecione manualmente os modelos de filamento de actina individualmente.
NOTA: Para cada um dos modelos de reticulador de actina, podemos registrar a posição nos filamentos de actina aos quais os reticuladores putativos se conectam. Essas informações poderiam, em princípio, ser extraídas para uma análise de acompanhamento usando a média do subtomograma, o que está além do escopo deste estudo.
4. Análise quantitativa do modelo 3D
- Análise de lacunas
NOTA: O objetivo desta etapa é detectar lacunas nos filamentos de actina.- Os itens selecionados podem ser analisados. Especifique seus parâmetros, como sua cor, usando a ferramenta Inspecionar seleção (Ação > Inspecionar).
- A cor do item selecionado pode ser especificada selecionando qualquer item selecionável no UCSF Chimera e alterando a cor ao lado da seção Cor da janela Inspecionar seleção .
- Especifique o tamanho dos marcadores e vínculos selecionando quaisquer marcadores e vínculos no UCSF Chimera e alterando os valores ao lado da seção raio da janela Inspecionar seleção , onde as unidades estão em Angstrøm.
- Realize a análise de lacunas por detecção automática de lacunas de densidade, utilizando a função Valores nas Posições dos Átomos (Ferramentas > Dados de Volume > Valores nas Posições dos Átomos).
- Selecione o mapa no qual os valores serão lidos selecionando o mapa desejado ao lado da seção Dados de Volume da janela Valores em Posições Atômicas . Selecione o modelo no qual todos os marcadores receberão um parâmetro, selecionando o modelo desejado ao lado da seção Molécula .
- Com o botão Histograma na parte inferior da janela Valores nas posições atômicas , um histograma é criado em uma nova janela denominada Renderizar/Selecionar por atributo. Ctrl + Clique no histograma adicionará ou removerá pontos de corte, para os quais cores diferentes podem ser selecionadas. Desmarque Manter opaco para aplicar uma cor transparente à cena. Marcadores com uma densidade abaixo de um determinado limite, que representa lacunas, podem não apenas ser coloridos, mas também podem ser selecionados e, assim, nos permitem contar os marcadores numéricos na região da lacuna. Para selecionar, clique em Selecionar > > value_mapname Atributo.
- Determinando distâncias entre actina
- Para medir distâncias entre actinas, coloque marcadores em um único plano XZ (referido no UCSF Chimera como um plano Y) usando a janela Rastreador de volume , clicando em Mouse e marcando apenas Colocar marcadores em planos de dados e desmarcando Vincular novo marcador ao marcador selecionado. Isso permite colocar com precisão os marcadores no mesmo plano Y (quando um único plano Z é mostrado). Verifique também Colocar marcadores usando o botão do meio do mouse na janela Rastreador de volume e coloque os marcadores em um plano Y de destino de interesse para medição de distância entre actinas.
- Uma vez que as posições de actina do plano Y único sejam marcadas com sucesso, selecione todos os marcadores e digite o comando findclash #model-spec test self overlapCutoff -200 hbondAllowance 0 log true Linewidth 10 pbColor preto na linha de comando na parte inferior do plano do filamento de actina. Isso localiza todos os conflitos entre marcadores dentro de uma distância de 200 Å (overlapCutoff -200) e cria linhas pretas (pbColor black) com uma espessura de 10 Å (largura de linha 10) entre os marcadores.
- Como existem marcadores sobrepostos dentro do intervalo de 200 Å, remova as medições redundantes que não sejam links apenas entre os marcadores adjacentes mais próximos. Faça isso usando o script RemoveCross.py fornecido no site do UCSF Chimera, que remove todos os links sobrepostos que não são o link mais curto com seus marcadores adjacentes.
- Para medir a distância das ligações restantes, selecione todas as pseudoligações (links criados por findclash) e use o script python pblengths.py digitando runscript pblengths.py. Veja os valores medidos no Registro de Respostas (Ferramentas > Utilitários > Registro de Respostas). Em seguida, copie os valores obtidos do Log de Respostas e transfira para uma planilha ou qualquer outra ferramenta adequada para criar um histograma.
Resultados
Usando tomografia crioeletrônica de estereocílios individuais não corados e hidratados congelados embebidos em gelo vítreo, obtivemos mapas de densidade do feixe de actina com seus filamentos de actina dispostos hexagonalmente, ligados por proteínas de conector cruzado23. A dimensão de um voxel individual foi de 0,947 nm. A inspeção visual no programa IMOD slicer de uma renderização de volume de todo o tomograma (400 fatias/379 nm) indicou a presença de estruturas filamentosas alinhadas com o eixo longitudinal dos estereocílios, como pode ser visto em vistas longitudinais (plano XY; Figura 1A-C, painéis superiores), bem como em vistas transversais (plano XZ; Figura 1A-C, painéis inferiores). Observamos que a visão de projeção através da rede de filamentos de 400 fatias / 379 nm tornou-se mais clara ao girar o volume reconstruído original em -6 ° ao redor do eixo X, -13,5 ° ao redor do eixo Y e 5 ° ao redor do eixo Z. Nesse ângulo, todos os filamentos estão alinhados uns sobre os outros e, portanto, o contraste é máximo, como pode ser apreciado nas vistas transversais (Figura 1B). Como as fatias transversais únicas não têm sinal suficiente para distinguir inequivocamente os filamentos de actina, optamos por renderizar em volume uma placa de 30 fatias / 28,4 nm, que mostra claramente um padrão hexagonal na visualização da seção transversal. As linhas azuis na Figura 1C (painéis superiores) indicam a posição do meio das 30 fatias/28,4 nm correspondentes
Pequenos desvios desse ângulo de visão ideal, em apenas ±2 °, reduziram significativamente a ordem percebida da rede de filamentos de actina (Figura 1A, C), o que é uma indicação de como é fácil se perder no volume 3D do tomograma.
Para ilustrar o desafio de usar abordagens de segmentação automatizadas, como segmentação de bacias hidrográficas, escolhemos um pequeno subvolume (representado como ouro) para segmentação de bacias hidrográficas, conforme implementado no pacote de software UCSF Chimera (Ferramentas > Dados de Volume > Segger > Segment). A posição do subvolume em relação a todo o mapa de estereocílios é indicada pela pequena inserção na Figura 1B.
A Figura 1D-F mostra o subvolume escolhido em diferentes orientações, com a Figura 1D,E mostrando a direção de visualização longitudinal e a Figura 1F mostrando uma direção de visualização transversal. As setas no lado esquerdo da Figura 1D-F indicam a direção dos filamentos de actina.
A Figura 1D-F (painéis à direita) mostra os resultados da segmentação da bacia hidrográfica. O subvolume está sendo codificado por cores por identidade de objeto, com cores sendo atribuídas aleatoriamente aos diferentes objetos. Cores diferentes indicam uma identidade de objeto diferente, portanto, torna-se aparente na Figura 1D-F que as densidades do mapa para filamentos foram fragmentadas ao longo do eixo do filamento, enquanto a mesma cor e, portanto, a identidade do objeto foram dadas para mapear densidades que conectavam filamentos vizinhos. Em outras palavras, o algoritmo de segmentação de bacias hidrográficas não foi capaz de seguir o mapa de densidade dos filamentos de actina por um período prolongado e, em vez disso, levou à conexão de densidades de filamentos vizinhos. Embora seja possível selecionar manualmente a seleção (por exemplo, excluindo ou mesclando objetos), essa abordagem é bastante trabalhosa e, portanto, demorada.
Embora não seja absolutamente necessário que nossa estratégia de construção de modelo volumétrico funcione, ela ajudou a reorientar (girar) o mapa 3D para que o eixo da rede do filamento de actina se alinhe com o eixo Y e os planos do modelo de filamento de actina alinhados com o plano XY do tomograma. Referimo-nos a essa orientação como a orientação padrão para exibição tomográfica de estereocílios.
Decidimos, portanto, explorar uma estratégia diferente para segmentação de imagens, explorando o fato de que os filamentos de actina exibiam uma organização regular geral (empacotamento hexagonal), com espaçamento regular e orientação geral definida do feixe. Nossa estratégia foi encontrar um ajuste geral dos modelos de um feixe de actina, como uma matriz de filamentos, seguido de ajustes regionais e locais da posição do modelo para se ajustar ao mapa de densidade experimental. Ao colocar um modelo geral em primeiro lugar, podemos superar as ambiguidades do mapa local e detectar tendências regionais de desvios do modelo de sua organização original, como flexão de filamentos.
Para colocar o modelo, exibimos placas de densidade (10 fatias / 9,47 nm) na orientação padrão que correspondia a uma espessura de uma única camada dos filamentos de actina, à qual uma camada de modelos de filamentos de actina retos e espaçados regularmente foi encaixada. É claro que isso é uma simplificação excessiva dos filamentos de actina, cada um dos quais consiste em uma matriz linear de monômeros de actina com simetria helicoidal. A Figura 2A-C mostra três camadas representativas em diferentes alturas Z, com os bastonetes de cor vermelha representando os filamentos de actina. Os painéis superiores, representando seções transversais de ~ 30 fatias / 28,4 nm de espessura, mostram em qual altura Z uma camada de modelo de actina individual de 19 hastes foi colocada, enquanto os painéis inferiores mostram uma orientação longitudinal (embora mostrada em perspectiva). A Figura 2D mostra o modelo simplificado completo, tanto na vista de corte transversal (painel superior) quanto na vista de perspectiva longitudinal (painel inferior). A orientação da seção transversal nos permitiu posicionar os filamentos com boa confiança. Aqui, nosso movimento original de reorientar todo o volume para coincidir com os eixos principais dos tomogramas provou ser útil, pois significava que a orientação de nosso modelo em nossa direção de visualização padrão também era paralela aos eixos principais. No entanto, estritamente falando, nossa abordagem também teria funcionado sem a reorientação do tomograma, apenas a colocação do modelo na densidade teria sido mais desafiadora.
Após uma inspeção cuidadosa das lajes individuais do mapa de densidade, notamos que um modelo de actina perfeitamente reto não se encaixava no mapa de densidade observado movendo-se da extremidade proximal para a extremidade distal (ou seja, em direção à ponta) dos estereocílios ( Figura 3A-C ). Perto da ponta dos estereocílios, a densidade do mapa para filamentos foi deslocada em mais de 13 nm (espaçamento actina-actina), o que pudemos compensar ajustando o modelo enquanto nos movemos da porção proximal para a distal do mapa de densidade dos estereocílios, introduzindo assim uma curvatura gradual pequena, mas distinguível, em nosso modelo de actina. A Figura 3D mostra uma única placa da densidade do mapa do filamento de actina, com um modelo volumétrico ajustado ao mapa de densidade. Uma comparação entre o modelo reto (vermelho) e o curvo (amarelo) é mostrada na Figura 3E. Essa curvatura é melhor apreciada inclinando uma placa do mapa de densidade com o modelo colocado em 80° em torno do eixo X, o que permite ter uma visão em perspectiva ao longo da direção dos filamentos de actina (Figura 3D, E).
O desvio dos dois modelos, com a posição do modelo de actina perto da ponta sendo deslocada aproximadamente na mesma distância que o espaçamento dos filamentos de actina, poderia ter causado muita confusão se não tivéssemos procedido da maneira que fizemos. Esse posicionamento "global" de uma camada do modelo de filamento de actina, seguido de ajuste "regional", permitiu detectar essa curvatura, que é quase imperceptível na visão longitudinal ou transversal. No entanto, a sobreposição dos dois modelos, conforme mostrado na Figura 3E, revela a diferença sutil.
Repetir essa abordagem para várias camadas permite obter um modelo 3D completo (Figura 3F), limitado apenas pela incerteza dos dados na parte superior e inferior dos estereocílios, quando vistos na orientação da seção transversal (Figura 3G). Essa falta de densidade é causada pela falta de cunha na coleta de dados tomográficos (eixo único) e pela anisotropia de resolução de dados correspondente, e seu efeito é indicado pela ausência de uma densidade de mapa bem definida para as membranas dos estereocílios.
Uma vez que tínhamos um modelo 3D, codificamos por cores cada local do modelo volumétrico de acordo com o valor da densidade do mapa naquela posição. As regiões do modelo com densidade de mapa fraca subjacente foram coloridas em vermelho, enquanto as regiões do modelo com sinal de densidade de mapa forte foram coloridas em amarelo (Figura 4A). Interpretamos essas regiões de cor vermelha, que podem se estender por dezenas de nanômetros, como lacunas nas estruturas do filamento de actina que, devido à sua extensão, não podem ser atribuídas a variações de densidade frequentemente encontradas no ambiente de alto ruído de um mapa crio-EM. O ruído tende a afetar voxels individuais ou pequenos grupos de voxels, mas é improvável que seja a fonte de volumes que consistem em centenas de voxels, para os quais a densidade do filamento está faltando. Em vez disso, essas lacunas provavelmente são uma característica real da malha de actina dos estereocílios e podem constituir locais de renovação da actina. A Figura 4A tem dois valores diferentes de densidade de mapa, mostrados em azul claro e azul escuro. Deve-se notar explicitamente que nossa abordagem de construção de modelo volumétrico, combinada com um código de cores automatizado de nosso modelo em regiões de densidade fraca, é uma maneira rápida e conveniente de detectar e visualizar a distribuição de tais lacunas no modelo de filamento de actina, o que teria sido muito difícil de outra forma.
Conforme mostrado na Figura 4B, partes do modelo volumétrico em locais com uma densidade comparativamente fraca podem ser facilmente ocultadas com base nos resultados obtidos na Figura 4A. Isso resulta em um modelo mais fragmentado que pode retratar de forma mais realista o modelo de actina nos estereocílios. A alternativa de construir pequenos trechos de filamentos de actina teria sido muito trabalhosa e pode ter falhado completamente, devido aos problemas discutidos ao descrever a Figura 1.
Além disso, o modelo volumétrico nos permite modelar facilmente os conectores cruzados simplesmente colocando uma conexão (mostrada em vermelho) entre as posições dos pontos do modelo de filamento de actina em ambos os lados da conexão cruzada (Figura 4C). Em nossa abordagem simplificada, não precisamos fazer nenhuma suposição sobre a identidade exata de cada proteína de conexão cruzada, o que exigiria maior resolução e / ou abordagens sofisticadas de rotulagem. Em vez disso, tudo o que precisamos determinar é se existe uma densidade que une os filamentos de actina adjacentes; Se houver, podemos colocar uma conexão curta de um filamento à sua contraparte adjacente. Na Figura 4D, é mostrado um modelo de cinco filamentos de actina com seus conectores cruzados, o que dá uma impressão da distribuição dos conectores cruzados ao longo do eixo do filamento de actina.
Outra vantagem de construir um modelo volumétrico do feixe de actina é que se pode determinar rapidamente o espaçamento entre os filamentos de actina adjacentes (Figura 4E-H). A Figura 4E, F mostra uma visão transversal do mapa de densidade sem e com um modelo ajustado à rede hexagonal da densidade do mapa, respectivamente. A Figura 4G mostra o modelo com conexões entre as bolas vizinhas mais próximas. O UCSF Chimera permite o cálculo automático da distância dos centros vizinhos mais próximos, cujo resultado pode ser plotado como uma distribuição de distância (Figura 4H). A construção de modelos para dois conjuntos de dados adicionais é mostrada na Figura Suplementar 1 e na Figura Suplementar 2.
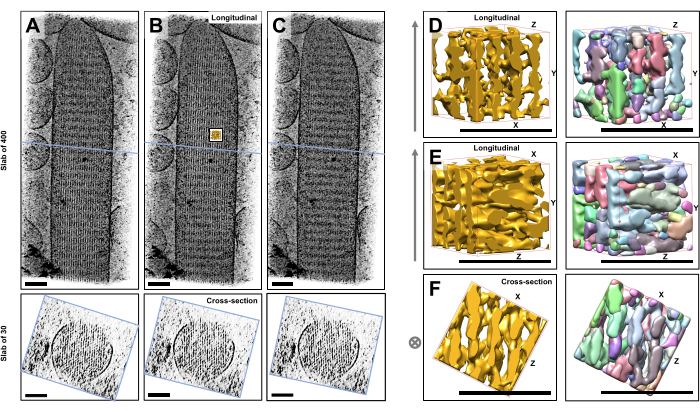
Figura 1: Os desafios encontrados pela segmentação de bacias hidrográficas de tomogramas de estereocílios de células ciliadas. (A-C) Projeções longitudinais (400 fatias/379 nm) através do mapa tomográfico 3D no plano XY (painéis superiores) e vistas transversais (30 fatias/28,4 nm) no plano XZ (painéis inferiores). (A) O mapa tomográfico girou -2° ao longo do eixo Y a partir de sua orientação ideal. (B) Mapa tomográfico na orientação ótima, determinado pelo ajuste dos ângulos de rotação dos eixos X, Y e Z (X = -6°, Y = -13,5° e Z= 5°) e revelando um alto grau de ordem no mapa de densidade, sugerindo uma rede de filamentos de actina altamente ordenada. (C) Mapa tomográfico girado +2° ao longo do eixo Y a partir de sua orientação ideal; a rotação de apenas 2° em torno do eixo Y para longe da direção de visualização ideal compromete severamente a regularidade percebida do mapa de densidade. Os painéis inferiores revelam a regularidade da matriz de filamentos de actina quando vistos na direção da seção transversal. A linha azul em A-C indica a posição da laje da seção transversal. (D-F) Um cubo de 50 nm x 50 nm x 50 nm visto de três direções diferentes antes (painéis esquerdos) e depois (painéis direitos) da segmentação da bacia hidrográfica. Observe que a segmentação da bacia hidrográfica não consegue detectar a densidade contínua do filamento de actina, enquanto os filamentos de actina adjacentes e sua conexão cruzada compartilham a mesma identidade de objeto, sugerindo que a segmentação da bacia hidrográfica não é uma abordagem adequada para a segmentação do tomograma. Nos painéis D-F, o mapa de densidade no Chimera é mostrado como estilo de mapa "Superfície". (AC) Barras de escala = 100 nm. (D-F) Barras de escala = 50 nm. Clique aqui para ver uma versão maior desta figura.

Figura 2: Construção de um modelo volumétrico de filamento de actina em forma de bola e bastão. (A-C) Superior: vistas transversais de 30 fatias / 28,4 nm do mapa de densidade com um modelo de filamento de actina simplificado de camada única colocado em uma altura Z diferente. Inferior: uma única fatia de 10 fatias/9,47 nm do mapa de densidade com um modelo de filamento de actina simplificado de camada única. (D) Modelo completo de filamento de actina reto em uma visão transversal de 30 fatias / 28,4 nm (superior) e vista em perspectiva (inferior); Barras de escala = 100 nm. Clique aqui para ver uma versão maior desta figura.
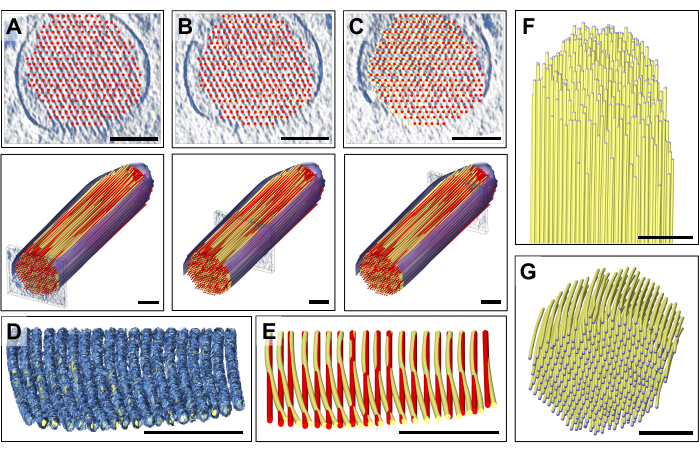
Figura 3: Ajuste do modelo para corrigir a curvatura detectada da densidade do filamento de actina. (A-C) Uma inspeção visual cuidadosa do modelo, seja na vista transversal (painéis superiores) ou longitudinal (painéis inferiores), revelou um bom ajuste do modelo com o mapa de densidade na extremidade proximal dos estereocílios. No entanto, à medida que se avança em direção à extremidade distal dos estereocílios, o ajuste torna-se cada vez pior para todos os modelos de filamentos de actina. Isso pode ser corrigido movendo as bolas do modelo de bola e bastão para a posição correta do mapa de densidade, o que resulta em um modelo de filamento de actina ligeiramente curvo. O modelo reto é mostrado em vermelho e o modelo dobrado corrigido é mostrado em amarelo. (D) Laje única do mapa de densidade com o modelo curvo ajustado a ela, revelando a curvatura da densidade de actina em direção à ponta dos estereocílios. O modelo de actina foi girado 80° em torno do eixo X para mostrar melhor essa curvatura sutil, mas significativa, dos filamentos de actina. (E) Comparação entre o modelo de actina reto e não corrigido mostrado em vermelho e o modelo de actina curvo e corrigido mostrado em amarelo. Por razões de clareza, apenas uma camada do modelo de filamento de actina é mostrada. (F-G) Modelo de feixe de actina com filamentos de actina curvos e corrigidos mostrados na orientação longitudinal (F) e transversal (G). A membrana segmentada é mostrada em azul. No painel D, o mapa de densidade no Chimera é mostrado como estilo de mapa "Malha". Barras de escala = 100 nm. Clique aqui para ver uma versão maior desta figura.

Figura 4: Detecção de lacunas no feixe de actina usando a construção de modelos volumétricos. Quantificação de propriedades volumétricas usando modelos de bola e bastão. (AC) Uma pequena região de uma laje longitudinal de ~ 10 fatias / 9,47 nm de espessura do mapa de densidade de estereocílios é mostrada em azul, junto com o modelo de filamento de actina curvo e corrigido. (A) O mapa de densidade é mostrado em um limite de densidade mais baixo (azul claro) e um limite de densidade mais alto (azul escuro). Existem regiões do modelo de filamento de actina para as quais não há densidade correspondente. Nesses locais, o modelo foi codificado por cores vermelhas para representar a falta de densidade. Interpretamos esses locais como lacunas nos filamentos de actina. (B) Modelo dos filamentos de actina que parecem fragmentados, refletindo a falta de densidade de filamentos de actina em tais locais de lacuna. (C) Modelo dos filamentos de actina com elos de conexão cruzada (mostrados em vermelho) adicionados em locais onde uma forte densidade foi encontrada para unir os filamentos de actina adjacentes. (D) Três modelos selecionados de filamentos de actina são mostrados com conectores cruzados para filamentos de actina adjacentes (que não são mostrados para maior clareza). Observe que muitas, mas não todas, as possíveis posições de filamentos inter-actina estão sendo ocupadas por proteínas conectoras; (E) Uma seção transversal de 30 fatias / 28,4 nm da densidade dos estereocílios. (F) Modelo de filamentos de actina ajustados à laje de densidade de estereocílios de seção transversal de 30 fatias / 28,4 nm. (G) Modelo de filamentos de actina sem a densidade do mapa subjacente. Detecção automática das distâncias do filamento de actina vizinho mais próximo, conforme indicado por conexões finas entre as bolas do modelo de filamento de actina. (H) Histograma das distâncias do modelo de filamento de actina. Nos painéis A-C, o mapa de densidade no Chimera é mostrado como estilo de mapa "Malha". (AC) Barras de escala = 50 nm. (D) Barra de escala = 25 nm. (E-G) Barras de escala = 100 nm. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Construção de modelo para o primeiro de dois conjuntos de dados adicionais de estereocílios. (AC) Uma pequena região de uma laje longitudinal de ~ 10 fatias / 9,47 nm de espessura do mapa de densidade de estereocílios é mostrada em azul usando a exibição do modo de malha. O modelo inicialmente colocado é mostrado em vermelho e o modelo corrigido mostrado em amarelo. (A) Apenas densidade do mapa. (B) Modelo inicial colocado no mapa de densidade. (C) Modelo corrigido colocado no mapa de densidade. (D-E) Região dos estereocílios maior sem (D) e com (E) o modelo corrigido ajustado a uma laje longitudinal de ~ 10 fatias / 9,47 nm de espessura do mapa de densidade dos estereocílios. (F-G) Toda a região do tomograma dos estereocílios é mostrada. (F) Apenas mapa. (G) Mapa com o modelo corrigido. (H) Sobreposição dos modelos inicial e corrigido. Barras de escala = 100 nm Clique aqui para baixar este arquivo.
Figura suplementar 2: Construção do modelo para o segundo de dois conjuntos de dados adicionais de estereocílios. (AC) Uma pequena região de uma laje longitudinal de ~ 10 fatias / 9,47 nm de espessura do mapa de densidade de estereocílios é mostrada em azul usando a exibição do modo de malha. O modelo inicialmente colocado é mostrado em vermelho e o modelo corrigido mostrado em amarelo; (A) Apenas densidade do mapa. (B) Modelo inicial colocado no mapa de densidade. (C) Modelo corrigido colocado no mapa de densidade. (D-E) Região dos estereocílios maior sem (D) e com (E) o modelo corrigido ajustado a uma laje longitudinal de ~ 10 fatias / 9,47 nm de espessura do mapa de densidade dos estereocílios. (F-G) Toda a região do tomograma dos estereocílios é mostrada. (F) Apenas mapa. (G) Mapa com o modelo corrigido. (H) Sobreposição do modelo inicial e corrigido. Barras de escala = 100 nm. Clique aqui para baixar este arquivo.
Arquivo Suplementar 1: Scripts python UCSF Chimera para modelagem de estereocílios. Clique aqui para baixar este arquivo.
Arquivo de codificação suplementar 1: pblengths.py. Clique aqui para baixar este arquivo.
Arquivo de codificação suplementar 2: RemoveCross.py. Clique aqui para baixar este arquivo.
Arquivo de codificação suplementar 3: ActinFilamentPlane.py. Clique aqui para baixar este arquivo.
Arquivo de codificação suplementar 4: dividelinks.py. Clique aqui para baixar este arquivo.
Arquivo de codificação suplementar 5: FixingMarkerID.py. Clique aqui para baixar este arquivo.
Discussão
Mostramos que abordagens automatizadas para segmentação, como segmentação de bacias hidrográficas, podem falhar no ambiente de alto ruído e alta complexidade de tomogramas crioeletrônicos de estereocílios de células ciliadas. Distinguir qual parte dessa rede filamentosa representa filamentos de actina e o que constitui ligações cruzadas em um nível de ambiente local parece desafiador, na melhor das hipóteses, quando apenas inspecionamos pequenos subvolumes tomográficos. A abordagem de construção de modelo usada neste estudo se beneficia do conhecimento prévio da ordem em larga escala do feixe de actina, o que ajuda no desenvolvimento de uma expectativa sobre a orientação dos filamentos de actina e as densidades do reticulador. Talvez ainda mais significativo seja que um cérebro humano pode facilmente encontrar padrões considerando o contexto maior além da distribuição de densidade local, enquanto um algoritmo de computador só funciona para uma região relativamente pequena que é considerada pelo algoritmo; portanto, tendências de maior escala não podem ser facilmente levadas em consideração. Ao ajustar um modelo globalmente a uma camada de densidade, evitamos a confusão que pode ocorrer ao tentar criar um modelo para pequenas porções de filamentos de actina simples de cada vez. É claro que esse ajuste global assume uma ordem que se estende por grandes distâncias. No entanto, como tivemos uma curvatura gradual inesperada, pequena, mas significativa, dos filamentos de actina, o ajuste global foi apenas uma aproximação inicial e exigiu ajustes locais do modelo para ajustar o mapa de densidade. Como o modelo inicial era um bom ponto de partida, os ajustes podiam ser feitos com alta confiança. Uma grande vantagem de nossa abordagem foi que pudemos optar por exibir apenas uma zona definida de densidade, o que ajudou a reduzir a complexidade do cenário. Além disso, a visualização da laje de densidade do mapa ao longo do eixo do modelo de filamento ajudou a identificar a curvatura inesperada, que provavelmente teríamos perdido ao simplesmente exibir subvolumes menores. A colocação do modelo inicial também facilitou o zoom rápido para dentro e para fora, para alternar entre uma visão geral da respectiva camada de filamentos de actina e as visualizações detalhadas para fazer ajustes no modelo.
As etapas críticas dentro do protocolo incluíram a rotação do mapa após a inspeção visual, a criação e colocação do modelo no mapa de densidade, bem como a divisão do modelo de filamento em segmentos menores. A posição do átomo dos segmentos pode então ser ajustada espacialmente para se ajustar ao mapa de densidade e / ou codificada por cores para detectar lacunas.
Essa abordagem de construção de modelos de actina também pode ser modificada colocando um conjunto de "átomos" (ou seja, as bolas do modelo de bola e bastão) em densidades de filamento usando uma visão transversal de uma placa média de densidade de 10-30 fatias / 9,47-28,4 nm, que então pode ser conectada por ligações (ou seja, as varas do modelo de bola e bastão). Usamos essa abordagem, que é uma modificação do protocolo descrito aqui em detalhes, para a construção do modelo volumétrico na região cônica dos estereocílios das células ciliadas23. Além disso, como descrevemos aqui, nossa abordagem de construção de modelos volumétricos também é adequada para a segmentação e construção de modelos de membranas.
Embora a construção de modelos volumétricos possa ser aplicada a qualquer mapa de densidade que mostre características filamentosas, a técnica que descrevemos aqui é mais eficiente quando temos uma matriz de filamentos regularmente espaçados, para os quais um ajuste global de um modelo volumétrico pode ser obtido. Também depende das características filamentosas alterar sua direcionalidade de maneira gradual. Se houver dobras repentinas e curvas acentuadas nas estruturas filamentosas, nossa abordagem pode não ser particularmente útil para a segmentação.
Enquanto isso, nossos colaboradores desenvolveram uma abordagem automatizada para rastreamento automatizado de filamentos que segue um conceito semelhante usado aqui para segmentação manual30,31. No futuro, a melhor abordagem pode muito bem ser um híbrido de identificação manual e colocação de um modelo esparso inicial (mesmo que apenas algumas bolas) na densidade como ponto de partida e, em seguida, deixar um algoritmo de pesquisa e ajuste terminar o rastreamento dos filamentos.
Modelos volumétricos simplificados reduzem a complexidade de um sistema e permitem que certos padrões, como a flexão do filamento de actina perto da ponta, sejam melhor apreciados. Além disso, o modelo volumétrico pode ser usado como uma "âncora" para exibir uma zona de densidade em torno do modelo de ancoragem de bola e bastão escolhido, o que permite a detecção e visualização de densidades de reticulador entre filamentos de actina adjacentes. A capacidade de selecionar filamentos individuais e definir raios apropriados como uma zona na qual a densidade é exibida novamente permite que a complexidade esmagadora do cenário seja reduzida a um nível gerenciável.
Uma vantagem dessa abordagem de construção de modelo volumétrico de ajuste global, seguida de ajustes locais, foi que fomos capazes de identificar regiões onde os filamentos de actina pareciam ser interrompidos, e lacunas significativas nos filamentos de actina foram indicadas pela ausência de densidade do mapa. Como havíamos colocado um modelo volumétrico de bola e bastão, poderíamos fazer uso de uma rotina no pacote de software UCSF Chimera que codifica por cores cada posição da bola do modelo de acordo com o valor da densidade do mapa naquele local. Essa abordagem permitiu uma rápida detecção e visualização de lacunas de filamentos de actina no feixe de actina, que é uma característica biologicamente significativa que encontramos em nosso tomograma crioeletrônico e que teria sido muito difícil de detectar e visualizar com abordagens tradicionais de segmentação. Outra vantagem do nosso modelo volumétrico é que as propriedades volumétricas, incluindo comprimentos e distâncias, podem ser facilmente obtidas, o que permite que números reais e, portanto, uma análise estatística seja realizada.
Em resumo, a colocação manual interativa do ponto do modelo, possivelmente aumentada pelos recursos subsequentes de ajuste local automatizado e rastreamento de filamentos, é uma abordagem bastante promissora para a visualização e análise quantitativa dos volumes subcelulares tomográficos eletrônicos. Isso ocorre porque ele usa o poder do cérebro humano para reconhecimento de padrões e o poder da ciência da computação para otimização de modelos.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes ou outros conflitos de interesse.
Agradecimentos
Gostaríamos de agradecer ao Dr. Peter Barr-Gillespie e sua equipe por seu papel na preparação de amostras e aos ex-membros do laboratório Auer e do laboratório Dr. Dorit Hanein por seu papel na coleta de dados tomográficos. Também gostaríamos de agradecer a Tom Goddard, da UCSF Resource for Biocomputing, Visualization, and Informatics (RBVI), por fornecer vários scripts do UCSF Chimera.
Materiais
| Name | Company | Catalog Number | Comments |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/download.html | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=win64/chimera-1.16-win64.exe | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=mac64/chimera-1.16-mac64.dmg | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=linux_x86_64/chimera-1.16-linux_x86_64.bin | |
| Excel | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Falcon II | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/accessories-em/falcon-detector.html | |
| IMOD | University of Colorado | Version 4.11.1 https://bio3d.colorado.edu/imod/download.html | |
| PC Desktop | Intel | Windows 10, ver. 22H2 | |
| PC Laptop | Gigabyte | Windows 10, ver. 22H2 | |
| Powerpoint | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Titan Krios Electron Microscope | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/transmission-electron-microscopes/krios-g4-cryo-tem.html | |
| Word | Microsoft | Version 2211 https://www.office.com/?auth=1 |
Referências
- Downing, K. H., Sui, H., Auer, M. Electron tomography: A 3D view of the subcellular world. Analytical Chemistry. 79 (21), 7949-7957 (2007).
- Koning, R. I., Koster, A. J. Cellular nanoimaging by cryo electron tomography. Methods in Molecular Biology. 950, 227-251 (2013).
- Asano, S., Engel, B. D., Baumeister, W. In situ cryo-electron tomography: A post-reductionist approach to structural biology. Journal of Molecular Biology. 428 (2), 332-343 (2016).
- Serwas, D., Davies, K. M. Getting started with in situ cryo-electron tomography. Methods in Molecular Biology. 2215, 3-23 (2021).
- McDonald, K. L., Auer, M. High-pressure freezing, cellular tomography, and structural cell biology. Biotechniques. 41 (2), 137-143 (2006).
- Narasimha, R., et al. Evaluation of denoising algorithms for biological electron tomography. Journal of Structural Biology. 164 (1), 7-17 (2008).
- Frangakis, A. S. It's noisy out there! A review of denoising techniques in cryo-electron tomography. Journal of Structural Biology. 213 (4), 107804 (2021).
- Frangakis, A. S., Hegerl, R. Noise reduction in electron tomographic reconstructions using nonlinear anisotropic diffusion. Journal of Structural Biology. 135 (3), 239-250 (2001).
- Jiang, W., Baker, M. L., Wu, Q., Bajaj, C., Chiu, W. Applications of a bilateral denoising filter in biological electron microscopy. Journal of Structural Biology. 144 (1-2), 114-122 (2003).
- vander Heide, P., Xu, X. -. P., Marsh, B. J., Hanein, D., Volkmann, N. Efficient automatic noise reduction of electron tomographic reconstructions based on iterative median filtering. Journal of Structural Biology. 158 (2), 196-204 (2007).
- Volkmann, N. Methods for segmentation and interpretation of electron tomographic reconstructions. Methods in Enzymology. 483, 31-46 (2010).
- Böhm, J., et al. Toward detecting and identifying macromolecules in a cellular context: template matching applied to electron tomograms. Proceedings of the National Academy of Sciences. 97 (26), 14245-14250 (2000).
- Frangakis, A. S., et al. Identification of macromolecular complexes in cryoelectron tomograms of phantom cells. Proceedings of the National Academy of Sciences. 99 (22), 14153-14158 (2002).
- Lebbink, M. N., et al. Template matching as a tool for annotation of tomograms of stained biological structures. Journal of Structural Biology. 158 (3), 327-335 (2007).
- Rigort, A., et al. Automated segmentation of electron tomograms for a quantitative description of actin filament networks. Journal of Structural Biology. 177 (1), 135-144 (2012).
- Volkmann, N. A novel three-dimensional variant of the watershed transform for segmentation of electron density maps. Journal of Structural Biology. 138 (1-2), 123-129 (2002).
- Bajaj, C., Yu, Z., Auer, M. Volumetric feature extraction and visualization of tomographic molecular imaging. Journal of Structural Biology. 144 (1-2), 132-143 (2003).
- Yu, Z., Frangakis, A. S. Classification of electron sub-tomograms with neural networks and its application to template-matching. Journal of Structural Biology. 174 (3), 494-504 (2011).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Tilney, L. G., Derosier, D. J., Mulroy, M. J. The organization of actin filaments in the stereocilia of cochlear hair cells. The Journal of Cell Biology. 86 (1), 244-259 (1980).
- Tilney, L. G., Tilney, M. S., DeRosier, D. J. Actin filaments, stereocilia, and hair cells: how cells count and measure. Annual Review of Cell Biology. 8, 257-274 (1992).
- Metlagel, Z., et al. Electron cryo-tomography of vestibular hair-cell stereocilia. Journal of Structural Biology. 206 (2), 149-155 (2019).
- Song, J., et al. A cryo-tomography-based volumetric model of the actin core of mouse vestibular hair cell stereocilia lacking plastin 1. Journal of Structural Biology. 210 (1), 107461 (2020).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Agulleiro, J. I., Fernandez, J. J. Fast tomographic reconstruction on multicore computers. Bioinformatics. 27 (4), 582-583 (2011).
- Fernandez, J. J. Computational methods for electron tomography. Micron. 43 (10), 1010-1030 (2012).
- Chen, H., Clyborne, W. K., Sedat, J. W., Agard, D. A. Priism: an integrated system for display and analysis of 3-D microscope images. Biomedical Image Processing and Three-Dimensional Microscopy. 1660, 784-790 (1992).
- Pettersen, E. F., et al. UCSF Chimera-a visualization system for exploratory research and analysis. Journal of Computational Chemistry. 25 (13), 1605-1612 (2004).
- Goddard, T. D., Huang, C. C., Ferrin, T. E. Visualizing density maps with UCSF Chimera. Journal of Structural Biology. 157 (1), 281-287 (2007).
- Sazzed, S., et al. Tracing actin filament bundles in three-dimensional electron tomography density maps of hair cell stereocilia. Molecules. 23 (4), 882 (2018).
- Sazzed, S., Scheible, P., He, P., Wriggers, J. Spaghetti tracer: A framework for tracing semiregular filamentous densities in 3D tomograms. Biomolecules. 12 (8), 1022 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados
Usamos cookies para melhorar sua experiência em nosso site.
Ao continuar usando nosso site ou clicando em 'continuar', você concorda em aceitar nossos cookies.