Method Article
Avaliação do Processo e Cinética da Expressão da Quitina Desacetilase Recombinante em Células de E. coli Rosetta pLysS Usando uma Técnica Estatística
Neste Artigo
Resumo
No protocolo atual, uma técnica estatística, planejamento composto central (CCD), foi aplicada para otimizar as condições de processo para a expressão da quitina desacetilase bacteriana recombinante (BaCDA) em células de E. coli Rosetta pLysS. O emprego de CCD resultou em um aumento de ~ 2,39 vezes na expressão e atividade de BaCDA.
Resumo
Nos últimos anos, a rota mais ecológica da desacetilação da quitina para quitosana usando a enzima quitina desacetilase ganhou importância. A quitosana convertida enzimaticamente com características emuladoras tem uma ampla gama de aplicações, particularmente no campo biomédico. Várias quitina desacetilases recombinantes de várias fontes ambientais foram relatadas, mas não há estudos sobre otimização de processos para a produção dessas quitina desacetilases recombinantes. O presente estudo utilizou a metodologia de planejamento composto central de superfície de resposta para maximizar a produção de quitina desacetilase bacteriana recombinante (BaCDA) em E. coli Rosetta pLysS. As condições otimizadas do processo foram concentração de glicose de 0,061%, concentração de lactose de 1%, temperatura de incubação de 22 °C, velocidade de agitação de 128 rpm e 30 h de fermentação. Em condições otimizadas, a expressão devido à indução de lactose foi iniciada após 16 h de fermentação. A expressão máxima, biomassa e atividade de BaCDA foram registradas 14 h após a indução. Na condição otimizada, a atividade BaCDA do BaCDA expresso foi aumentada ~ 2,39 vezes. A otimização do processo reduziu o ciclo total de fermentação em 22 h e o tempo de expressão em 10 h pós-indução. Este é o primeiro estudo a relatar a otimização do processo de expressão de quitina desacetilase recombinante usando um projeto composto central e seu perfil cinético. A adaptação dessas condições ideais de crescimento pode resultar em uma produção econômica e em larga escala da moneran desacetilase menos explorada, embarcando em uma rota mais ecológica para a produção de quitosana de grau biomédico.
Introdução
A quitina, um polímero natural ligado a 1-4 glicosídicos de β estrutural, é o segundo polissacarídeo mais abundante na natureza depois da celulose. Apesar disso, a quitina tem aplicações industriais limitadas devido à sua insolubilidade1. Esse gargalo é resolvido submetendo a quitina à N-desacetilação, que confere uma carga positiva e aumenta a solubilidade do polímero resultante, a quitosana1. A quitina pode ser modificada para quitosana por duas vias diferentes: química e enzimática. A aplicação biomédica da quitosana requer desacetilação controlada e definida, que é restrita em vias químicas 2,3. Essa limitação pode ser abordada usando quitina desacetilases (CDAs), uma abordagem enzimática verde para o processo de desacetilação 4,5.
A quitina desacetilase pertence à família carboidrato esterase 4 (CE-4), definida no banco de dados de enzimas ativas de carboidratos (CAZY). As enzimas da família CE-4 compartilham a homologia NodB ou domínio polissacarídeo desacetilase como região conservada. O design composto central (CCD), uma ferramenta estatística, é usado para a otimização de várias enzimas modificadoras de quitina do tipo selvagem 6,7,8,9. No entanto, as etapas a jusante no uso de organismos do tipo selvagem tornam-se tediosas, daí a mudança para enzimas recombinantes 10,11,12,13,14,15,16,17. Nos últimos anos, os CDA recombinantes halofílicos de origem marinha ganharam importância devido à sua facilidade na aplicação industrial e produção de quitosana de grau biomédico18,19.
A produção de enzimas recombinantes em E. coli tem uma limitação no processo, e a otimização do meio é necessária, pois sua expressão em E. coli varia dependendo do gene e do plasmídeo usado20. Assim, a triagem de um processo adequado e parâmetros de nutrientes torna-se importante. Um fator de cada vez (OFAT), o método de otimização comumente empregado, requer enormes recursos e tempo para realizar experimentos passo a passo. Esse método sofre de falta de informações estatísticas sobre a interação entre os parâmetros20,21,22,23. Portanto, a metodologia CCD de superfície de resposta (RSM) foi adotada para estudar o rendimento da expressão da quitina desacetilase bacteriana halofílica (BaCDA) e a atividade de BaCDA em E. coli Rosetta pLysS. Os parâmetros considerados para otimização da expressão no hospedeiro de E. coli foram concentração de lactose, concentração de glicose, temperatura de incubação, taxa de agitação e tempo de incubação. Na maioria dos estudos de expressão de E. coli, o meio Luria Bertani (LB) com isopropil β-d-1-tiogalactopiranosídeo (IPTG) foi usado como indutor. Essa adição de IPTG exigiu monitoramento regular do crescimento24. Essas mediações recorrentes durante o processo fermentativo também abrem caminhos para contaminação. Assim, os grupos de pesquisa mudaram para o caldo fantástico (TB) com lactose como indutor. A inclusão de lactose na mídia em vez de IPTG aborda essa preocupação; E. coli consome essa lactose e produz alo-lactose como subproduto, resultando em uma condição de auto-indução. Este meio auto-indutor inclui glicerol, que exibiu melhores rendimentos de proteína recombinante25. Essa superexpressão de proteínas recombinantes em meio TB foi melhorada ainda mais com a otimização dos parâmetros do processo. No presente estudo, um delineamento composto central foi aplicado para otimizar a expressão heteróloga de BaCDA halofílico em células de E. coli Rosetta pLysS. Os parâmetros de processo escolhidos foram temperatura de incubação, taxa de agitação e tempo de incubação, e os parâmetros nutricionais avaliados foram concentração de glicose e lactose. A expressão halofílica de BaCDA foi avaliada com a condição otimizada prevista e validada cruzada usando SDS-PAGE.
Protocolo
1. Meios de expressão e condição de cultura
- Transformar o vector pET-22b que contém o gene BaCDA em células competentes para E. coli Rosetta pLysS, utilizando o método do choque térmico, tal como descrito no ponto15.
NOTA: Deve-se ter cuidado ao trabalhar com microrganismos. Todo o trabalho microbiológico deve ser realizado dentro de uma coifa de gabinete de biossegurança para evitar contaminação. - Realizar o estudo preliminar de expressão em meio TB contendo 0,05% (p/v) de glicose e 0,2% (p/v) de lactose a 16 °C e 180 rpm. Cultive 6,792 x 107 células E. coli Rosetta pLysS em 100 mL de meio contendo plasmídeo clonado BaCDA até atingir a fase estacionária. Nas condições de crescimento mencionadas, as células de E. coli Rosetta pLysS atingem uma densidade óptica 600 (OD600) de 10,85 ± 0,21.
NOTA: Composição do meio TB (% p/v): triptona, 1,2; extrato de levedura, 2,4; glicerol, 0,6; e 1x TB de sal (17 mM KH2PO4 e 74 mM K2HPO4)17.
2. Otimização e desenho experimental
- Projete os experimentos e ajuste o modelo polinomial de segunda ordem no projeto composto central usando o software da ferramenta estatística. O software estatístico utilizado no presente estudo foi o MINITAB 17.0 (versão de teste).
- Para fazer isso, abra o software e clique nos seguintes botões: Stat > DoE > Superfície de resposta > Criar superfície de resposta > Response design > Central composite. A saída aparece como uma caixa de diálogo.
- Alimente os cinco parâmetros, temperatura de incubação, taxa de agitação, tempo de incubação, concentração de glicose e concentração de lactose, em cinco níveis (-2, -1, 0, +1, +2) na caixa de diálogo.
- Para fazer isso, insira os parâmetros com os níveis na caixa de diálogo, insira os detalhes de todos os parâmetros e pressione OK. Isso gera a matriz de planejamento experimental com seis repetições no ponto central, exigindo 32 ensaios experimentais. O software gera uma tabela contendo os parâmetros e seus níveis em termos codificados e não codificados, utilizados na otimização do processo utilizando CCD (Tabela 1).
- Realize os 32 experimentos com as condições geradas por software. Alimente os resultados experimentais na matriz de planejamento experimental (gerada pelo software) contendo níveis e parâmetros em termos de unidades codificadas e não codificadas (Tabela 2).
- Analisar a matriz de planejamento experimental (Tabela 2) utilizando o software. Para fazer isso, abra o software e alimente a resposta na planilha de dados. Selecione a coluna Resposta > Estatística > DoE > Superfície de resposta > Analisar o design da superfície de resposta > OK. A saída é analisada quanto à significância estatística e um modelo é previsto pelo software.
Tabela 1: Os parâmetros e seus níveis em termos codificados e não codificados usados no projeto experimental para estimar a expressão da quitina desacetilase recombinante em células pLysS de E. coli Rosetta. Clique aqui para baixar esta tabela.
Tabela 2: Matriz de projeto experimental com atividade experimental e prevista de BaCDA (expressão) de quitina desacetilase recombinante em células de E. coli Rosetta pLysS. Clique aqui para baixar esta tabela.
3. Validação do modelo
- Valide o modelo projetado usando a ferramenta otimizada de resposta disponível no software.
- Repita a fermentação em condições ótimas previstas pelo software e compare o valor experimental com o valor previsto pelo software.
- Para fazer isso, abra o software e selecione a coluna Resposta > Estatística > DoE > superfície de resposta > Otimizador de resposta > Maximizar > OK. A saída são as condições ideais do processo e os valores previstos pelo software. Repita a fermentação nas condições ideais previstas e compare os valores.
4. Métodos analíticos
- Expressão, biomassa e quantificação de proteínas
- Realizar a fermentação em 32 condições dadas pelo software na matriz de planejamento experimental (Tabela 2). Obter o pellet após cada ensaio, centrifugando a cultura a 5,405 x g durante 10 min a 4 °C. Pesar o pellet celular para determinar o rendimento da biomassa.
NOTA: Todo o manuseio microbiano deve ser feito em condições assépticas (ou seja, dentro de cabines de biossegurança). - Lise o pellet celular para obter proteína periplasmática por sonicação. Adicione 5 mL de tampão de lise (50 mM Tris-HCl, 300 mM NaCl, 10 mM Imidazol) a cada grama de pellet celular e interrompa sonicando por 10 ciclos com um pulso de 10 s ligado e 10 s desligado a 60% de amplitude. Recolher o lisado centrifugando a 5,405 x g durante 10 min a 4 °C. Quantificar a concentração de proteínas pelo ensaio de Bradford26.
NOTA: Para evitar a desnaturação das proteínas, a sonicação deve ser feita em condições frias. Coloque as amostras no gelo durante a sonicação. - Analise a expressão BaCDA usando SDS-PAGE seguido pelo software ImageJ. Carregue 10 μL de lisado de células solúveis (proteína periplasmática) em gel de bis-acrilamida a 12% e passe sob 1x tampão tris-glicina-SDS (TGS) a uma corrente de 100 V por 2 h. Após a conclusão da corrida, pinte o gel com azul Coomassie e destrate para remover o fundo27.
- Capture uma imagem usando uma unidade de documentação de gel. Determine a expressão BaCDA comparando a intensidade da banda na forma dos valores de pixel usando o software ImageJ. Considere a execução experimental com a menor atividade de BaCDA e biomassa como o pixel de referência 28.
- Para fazer isso, abra o software e abra o arquivo de imagem e selecione cada pista usando a caixa retangular. Vá para Analisar e defina cada pista como 100% e, em seguida, selecione a banda superexpressa em cada pista usando a caixa retangular. Vá para Analisar e determine a intensidade da banda, repita o processo em cada pista e exporte o resultado como .xls arquivo. Considere a intensidade da banda da corrida experimental com a menor atividade de BaCDA como intensidade de referência e analise a expressão de cada pista.
- Realizar a fermentação em 32 condições dadas pelo software na matriz de planejamento experimental (Tabela 2). Obter o pellet após cada ensaio, centrifugando a cultura a 5,405 x g durante 10 min a 4 °C. Pesar o pellet celular para determinar o rendimento da biomassa.
- Ensaio de atividade enzimática
- Determinar a atividade de BaCDA usando um kit de ensaio de acetato15, usando quitina de etilenoglicol (1 mg/mL; EGC) como substrato29.
- Deixar reagir 20 μL de BaCDA com 40 μl do substrato na presença de 40 μl de tampão Tris-HCl (pH 7) a 50 mM durante 1 h a 30 °C, agitando a 800 rpm.
- Após 1 h, centrifugue o reagente de 100 μL através de uma coluna de 3 kDa a 2,111 x g por 15 min a 4 °C. Rejeitar o retentado constituído por BaCDA e recolher o filtrado contendo acetato libertado durante a reacção. Utilizar o filtrado para o ensaio de acetato para determinar a actividade de BaCDA.
NOTA: Uma unidade da enzima é definida como a atividade que libera 1 μM de acetato do substrato por microlitro de enzima por minuto. O ensaio de atividade enzimática foi realizado em triplicatas, e a respectiva atividade enzimática foi calculada de acordo.
- Cinética de fermentação da indução de lactose
- Investigue o perfil de atividade para determinar o ponto de indução da lactose e o ponto de início da expressão. O perfil de atividade é feito estimando a biomassa e a atividade de BaCDA com as condições de fermentação otimizadas.
- Determine a concentração de glicose no meio usando um kit de estimativa de glicose. Plote o perfil de atividade usando biomassa, concentração de glicose e atividade de BaCDA em relação ao tempo de fermentação.
Resultados
Otimização do processo de expressão da enzima quitina desacetilase recombinante periplasmática em E. coli usando planejamento composto central (CCD)
A construção pET22b-Ba CDA, quando cultivada em condições não otimizadas, proporcionou um rendimento máximo de biomassa e atividade de BaCDA de 22,26 ± 0,98 g/L e 84,67 ± 0,56 U/L, respectivamente15. No presente estudo, uma abordagem estatística CCD foi adotada para encontrar as condições ideais de processo para expressar a enzima quitina desacetilase recombinante periplasmática em células de E. coli Rosetta pLysS. Os cinco parâmetros do processo, temperatura de incubação (A), taxa de agitação (B), tempo de incubação (C), concentração de glicose (D) e concentração de lactose (E), foram selecionados com base em nosso trabalho preliminar (Tabela 1). A expressão da enzima recombinante periplasmática quitina desacetilase foi quantificada e representada como atividade de BaCDA (U/L) da quitina desacetilase recombinante (Tabela 2). Assim, os parâmetros A, B, C, D e E foram tomados como parâmetros exógenos, enquanto a atividade BaCDA da expressão da quitina desacetilase recombinante periplasmática foi escolhida como parâmetro endógeno (resposta). A matriz de planejamento experimental mostrou que a atividade máxima de BaCDA foi de 199,72 ± 1,14 U/L (corrida 26), e a atividade mínima de BaCDA foi de 1,27 ± 0,34 U/L (corrida 17), obtida a partir dos rendimentos de biomassa de 13,04 ± 0,48 g/L e 6,60 ± 0,70 g/L, respectivamente (Tabela 2 e Tabela 3). Os resultados do rendimento de biomassa, expressão de BaCDA, teor de proteína total e atividade específica em todos os 32 ensaios experimentais são mostrados na Tabela 3. A expressão máxima de BaCDA foi de 2,945 (corrida 26) e a expressão mínima foi de 1,00 (execução 17; Tabela 3 e Figura 1, respectivamente). A atividade específica correspondente ao máximo e mínimo foi de 0,079 U/mg e 0,001 U/mg, respectivamente (Tabela 3).
Tabela 3: Os resultados do rendimento de biomassa, expressão de BaCDA halofílico, teor de proteína total e a atividade específica de todos os ensaios experimentais fornecidos pelo planejamento composto central. Clique aqui para baixar esta tabela.
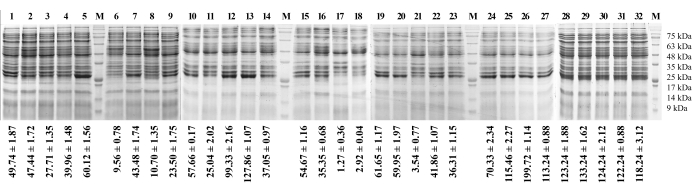
Figura 1: Resultados da análise SDS-PAGE. A figura mostra a análise SDS-PAGE sobre a expressão da enzima recombinante periplasmática quitina desacetilase em E. coli Rosetta pLysS para todas as execuções experimentais fornecidas pelo projeto composto central. A expressão de BaCDA halofílico foi observada em 29 kDa. Os números na parte superior representam a execução experimental e o valor na parte inferior representa a atividade do BaCDA na condição específica. Clique aqui para ver uma versão maior desta figura.
Inicialmente, o modelo completo de regressão de segunda ordem obtido para a atividade BaCDA da quitina desacetilase recombinante foi significativo, com um alto coeficiente de determinação (R2) de 0,9595, enquanto o R2 previsto foi de 0%, indicando a falta de previsibilidade de um modelo, e a equação de regressão é dada abaixo:
Z1 (atividade de BACDA, U/L) = -1844 + 99,7 A + 2,64 B + 36,33 C + 808 D + 55 E - 1,842 A2 - 0,00925 B2 - 0,3801 C2 - 26676 D2 + 234,9 E2 + 0,0562 A x B - 0,609 A x C + 44,8 A x D - 10,32 A x E - 0,0203 B x C + 0,83 B x D - 0,979 B x E + 28,2 C x D + 3,10 C x E + 432 D x E (Eq. 1)
em que A = temperatura de incubação, °C; B = taxa de agitação, rpm; C = tempo de incubação, h; D = concentração de glicose, % (p/v); e E = concentração de lactose, % (p/v).
Os resultados da análise de variância (ANOVA) do modelo completo (Equação 1) são apresentados na Tabela 4. Além disso, para aumentar a previsibilidade do modelo, os parâmetros mais insignificantes (A, B, C, AD, BC, BD, CD, CE e DE; [valor de p > 0,05]) foram removidos da Equação 1, e o modelo de regressão modificado final foi obtido para a atividade de BaCDA da quitina desacetilase recombinante, mostrada abaixo como:
Z2 (atividade de BACDA, U/L) = 121,81 + 9,99 D + 8,19 E - 29,48 A2 - 14,80 B2 - 24,33 C2 - 16,67 D2 + 9,40 E2 + 9,00 A x B - 19,49 A x C - 8,26 A x E - 7,83 B x E (Eq. 2)
Tabela 4: Análise de variância (ANOVA) para o modelo completo. Clique aqui para baixar esta tabela.
Tabela 5: Análise de variância (ANOVA) para o modelo reduzido e teste de significância para a expressão de quitina desacetilase recombinante em células de E. coli Rosetta pLysS. Clique aqui para baixar esta tabela.
Os valores previstos da atividade de BaCDA foram dados pelo software com base em um modelo de regressão modificado (Equação 2) e estão representados na Tabela 5. Além disso, a suposição de normalidade é cumprida à medida que os resíduos são normalmente distribuídos (ou seja, os pontos de dados estão mais próximos da linha reta), conforme mostrado no gráfico de probabilidade normal (Figura 2), indicando a capacidade do modelo de otimizar a expressão da quitina desacetilase recombinante. Portanto, a Equação 2 modificada deve ser aplicada para descobrir os níveis ideais e seu espaço de design para o processo. O impacto das interações entre os parâmetros independentes foi visualizado pelos gráficos de contorno bidimensionais para a expressão da quitina desacetilase recombinante (Figura 3).

Figura 2: Gráfico de probabilidade normal. A figura mostra o gráfico de probabilidade normal entre a atividade residual (eixo X) versus a porcentagem de probabilidade cumulativa dos dados (eixo Y). O gráfico indica a capacidade do modelo de otimizar a expressão da quitina desacetilase recombinante. Clique aqui para ver uma versão maior desta figura.
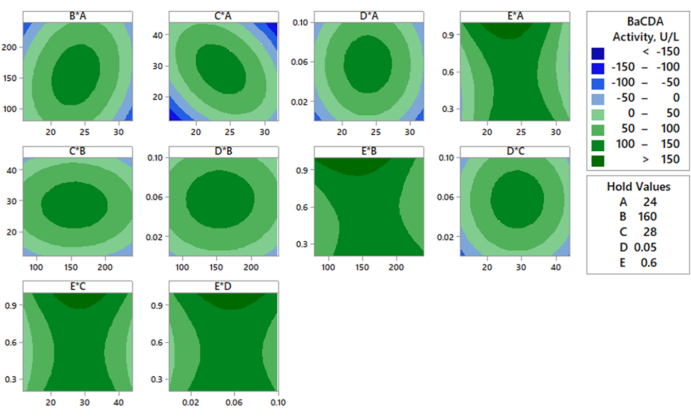
Figura 3: Gráficos de contorno. Os gráficos de contorno de todas as combinações de parâmetros. A: temperatura de incubação em °C; B: taxa de agitação em rpm; C: tempo de incubação em h; D: concentração de glicose em % (p/v); e E: concentração de lactose em % (p/v). Clique aqui para ver uma versão maior desta figura.
Validação do modelo
A ferramenta de otimização de resposta no software foi usada para resolver o modelo de regressão reduzida (Equação 2) e para encontrar as condições ideais para o aumento da atividade de BaCDA da enzima recombinante quitina desacetilase e sua expressão em E. coli Rosetta pLysS. O modelo foi validado nas condições ótimas de processo (ou seja, temperatura de incubação de 22 °C; taxa de agitação de 128 rpm; tempo de incubação de 30 h; concentração de glicose de 0,058% [p/v]; e concentração de lactose de 1% [p/v]) (Tabela 6 e Figura 4). Os valores ótimos de todos os cinco parâmetros foram colocados dentro dos níveis selecionados, exceto o parâmetro E (concentração de lactose), e a atividade predita e experimental de BaCDA da quitina desacetilase recombinante nessas condições ótimas foi de 190,85 U/L e 202,39 ± 0,31, respectivamente (Tabela 6).
Tabela 6: As condições ideais de processo para o aumento da atividade de BaCDA da enzima recombinante quitina desacetilase e sua expressão em E. coli Rosetta pLysS. Clique aqui para baixar esta tabela.
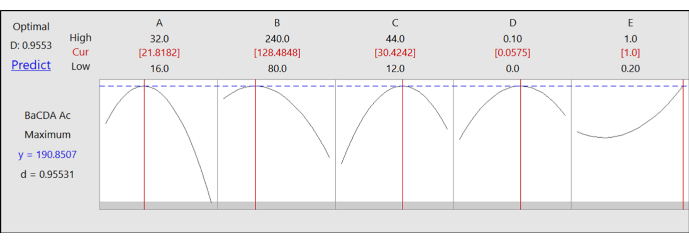
Figura 4: Gráfico de otimização. O gráfico de otimização apresenta valores ótimos para o aumento da atividade de BaCDA da enzima quitina desacetilase recombinante e sua expressão em E. coli Rosetta pLysS. A: temperatura de incubação em °C; B: taxa de agitação em rpm; C: tempo de incubação em h; D: concentração de glicose em % (p/v); e E: concentração de lactose em % (p/v). O eixo X representa os níveis dos parâmetros e o eixo Y representa a atividade BaCDA. Clique aqui para ver uma versão maior desta figura.
O valor ótimo da concentração de lactose usando a ferramenta de otimização de resposta foi de 1% no nível +2 no modelo. Portanto, foi estudado o impacto de concentrações mais altas de lactose (1-2,5% [p/v]) na expressão e na atividade de BaCDA. A atividade de BaCDA foi de 201.840 ± 1,92 U/L, 201.900 ± 1,95 U/L, 202.186 ± 1,59 U/L e 202.173 ± 2,09 U/L com 1%, 1,5%, 2% e 2,5% de lactose, respectivamente. Não houve diferença significativa na atividade de BaCDA, e a análise de SDS-PAGE também mostrou o mesmo nível de expressão em todas as concentrações de lactose, conforme mostrado na Figura 5.
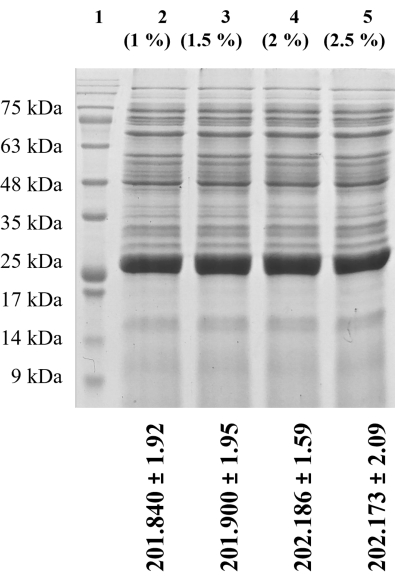
Figura 5: Resultados da análise SDS-PAGE. Análise SDS-PAGE na expressão da enzima quitina desacetilase recombinante periplasmática em E. coli Rosetta pLysS com diferentes concentrações de lactose em uma condição de processo otimizada. Pista 1: marcador; Faixa 2: concentração de lactose a 1% (p/v); Faixa 3: concentração de lactose a 1,5% (p/v); Faixa 4: concentração de lactose a 2% (p/v); e Pista 5: concentração de lactose de 2,5% (p/v). Clique aqui para ver uma versão maior desta figura.
Cinética de fermentação da indução de lactose
Este estudo mostra a cinética de produção de biomassa, concentração de glicose e atividade de BaCDA nas condições otimizadas. O crescimento de E. coli apresentou um padrão diauxico devido à inclusão de glicose (0,061%) e lactose (1%) na mídia. A primeira fase logarítmica foi observada até 12 h devido ao esgotamento completo da concentração de glicose. O rendimento de biomassa correspondente foi de 7,89 ± 0,18 g/L. A segunda fase logarítmica começou às 16 h com o consumo de lactose e iniciou a expressão halofílica de BaCDA. A atividade máxima de BaCDA e a produção de biomassa foram de 202,39 ± 0,31 U e 17,53 ± 0,07 g/L, respectivamente, às 32 h de fermentação (Figura 6).

Figura 6: Cinética do parâmetro de rendimento. Cinética de rendimento de biomassa, concentração de glicose e atividade de BaCDA expressando enzima recombinante periplasmática quitina desacetilase em E. coli Rosetta pLysS nas condições de processo otimizadas. Todas as corridas foram realizadas em triplicatas, e o valor foi representado como uma média com desvio padrão. Clique aqui para ver uma versão maior desta figura.
Discussão
A quitina desacetilada, quitosana, tem muitas aplicações, especialmente na área biomédica30. No entanto, a reprodutibilidade da quitosana em relação ao seu grau de acetilação (DA) e padrão de acetilação (PA) é uma grande preocupação, além de outras apreensões ambientais. A rota mais verde, usando enzimas, foi assim explorada. A matriz de CDAs pode ser empregada para criar quitosana com um padrão único de desacetilação, o que aumentaria suas aplicações biomédicas 4,5. Em nosso trabalho anterior, o gene BaCDA clonado foi expresso em células pLysS de E. coli Rosetta15.
O BaCDA foi expresso como corpos de inclusão a 37 °C na triagem primária. Com base no levantamento da literatura, uma temperatura de expressão mais baixa (ou seja, 16 °C) foi usada para obter BaCDA solúvel em meio de autoindução de TB31. No entanto, a atividade máxima de 84,67 ± 0,56 U/L foi obtida após 52 h de fermentação a 16 °C. A redução da temperatura de expressão resultou em um aumento do tempo de fermentação20. Assim, uma abordagem estatística, CCD, foi adotada para otimizar os parâmetros do processo para reduzir o tempo de fermentação e aumentar a atividade de BaCDA; vários estudos sobre a produção de CDA do tipo selvagem foram relatados 6,7,8. Até onde sabemos, este é o primeiro relatório sobre a otimização do processo de produção de BaCDA recombinante halofílico.
A escolha correta de parâmetros e níveis foi uma etapa crucial para a otimização baseada em CCD, e era necessária experiência. A temperatura de indução foi crucial, pois as variações levam à expressão da enzima na forma solúvel ou como corpos de inclusão20 , 31 , 32 . No estudo preliminar, a temperatura de fermentação a 37 °C resultou na expressão de BaCDA como corpos de inclusão, enquanto que a 16 °C, resultou em BaCDA solúvel. Portanto, a faixa de temperatura foi selecionada entre 16 °C a 32 °C para evitar a formação de corpos de inclusão. Previu-se que a faixa selecionada expressaria BaCDA como uma fração solúvel em contraste com a taxa de crescimento rápido. No estudo preliminar, o tempo de incubação necessário para que o crescimento da cultura atingisse a fase estacionária foi de 52 h a 16 °C. Um aumento na temperatura levou a um crescimento mais rápido, portanto, para a faixa de temperatura de incubação selecionada, o tempo de incubação foi escolhido entre 12 h a 44 h. Uma velocidade de agitação mais alta melhora a transferência de oxigênio e o crescimento de E. coli. A taxa de agitação entre 160 a 250 rpm tem sido estudada33; Portanto, no presente estudo, a taxa de agitação foi selecionada entre 80 a 240 rpm. A inclusão de IPTG induziu a proteína heteróloga; no entanto, grupos de pesquisa têm optado pela lactose como indutor para criar condições de autoindução e reduzir o custo de produção, pois a lactose é um substituto mais barato do IPTG, proporcionando resultados iguais ou melhores34,35. Assim, a expressão das enzimas recombinantes no sistema E. coli dependeu do crescimento diauxico glicose-lactose36. A maioria dos pesquisadores utilizou glicose a 0,05% e lactose a 0,2% para autoindução37,38. Em diferentes estudos de otimização estatística, a concentração de lactose foi otimizada na faixa entre 0% a 12,5%. No presente estudo, foi selecionada uma concentração de glicose de 0,05% no nível 0, e a faixa foi selecionada entre 0% e 0,1%. A lactose na concentração de 0,2% foi selecionada no nível -2, e a faixa foi selecionada entre 0,2% a 1%. Com base na literatura 34,35,36,38,39 e em nossos experimentos preliminares, os parâmetros do processo, temperatura de incubação, taxa de agitação, tempo de incubação, concentração de glicose e concentração de lactose foram selecionados para a expressão halofílica de BaCDA em E. coli com cinco níveis diferentes, usando um delineamento composto central (Tabela 1). A atividade de BaCDA variou de 1,27 a 199,72 U/L, indicando a influência dos parâmetros e seus níveis na expressão de BaCDA halofílico em células de E. coli Rosetta pLysS (Tabela 2 e Figura 1). Observou-se uma variação de apenas 9,71% na atividade de BaCDA entre os valores experimentais e previstos (Tabela 2), indicando a acurácia da experimentação. Da mesma forma, a Tabela 3 também está em boa concordância com a Tabela 2.
A adequação do modelo e a adequação foram avaliadas por meio da análise de variância (ANOVA) para o delineamento experimental utilizado (Tabela 4). O alto valor de R2 sugere uma maior significância do modelo40. A baixa diferença observada de 0,431 entre o valor ajustado de R2 (0,8785) e o valor de R2 (0,9216) confirma a precisão dos dados. A equação 1 foi altamente significativa, com um valor de F de 21,37, conforme mostrado pelo teste F de Fisher, juntamente com um valor de probabilidade muito baixo (modelo P > F = 0,000), que foi significativo em um intervalo de confiança de 95%. O valor F do modelo foi calculado usando a fórmula:

Os parâmetros são considerados significativos apenas se o valor da probabilidade da estatística F for menor que 0,05. Para o modelo proposto (Equação 1), os termos D, E, A2, B2, C2, D2, E2, AB e AC foram significativos com 95% de confiança (p < 0,05; Tabela 5). O valor F da Falta de Ajuste foi de 8,24, o que indica sua insignificância, e tem 14% de chance de ser significativo (Tabela 5). O insignificante valor F da falta de ajuste representa a adequação dos dados experimentais ao modelo (Figura 2). Portanto, com base nas estatísticas acima, pode-se concluir que a concentração de glicose (D) e a concentração de lactose (E) são vitais na expressão da quitina desacetilase recombinante em E. coli. Neste estudo, as condições físicas ambientais, temperatura de incubação (A), taxa de agitação (B) e tempo de incubação (C) não mostraram significância na expressão da quitina desacetilase recombinante (Tabela 5), enquanto a interação entre A e B e A e C foi muito significativa (Tabela 5 e Figura 3). Os resultados estão em boa concordância com os fatos gerais de valores F mais altos do modelo do que o valor F de falta de ajuste, e valores R2 mais altos (>0,70) especificam que o modelo se ajusta melhor aos dados. Além disso, para validar o modelo de regressão polinomial (Equação 2), foram realizados experimentos nas condições ótimas em triplicatas (Tabela 6 e Figura 4), e o valor experimental foi de 202,39 ± 0,31 U/L. A excelente correlação entre os valores de atividade de BaCDA previstos e experimentais indica um bom ajuste e, portanto, valida a competência do modelo. A concentração ótima de lactose foi estimada e, acima de 1%, não houve aumento na atividade de BaCDA obtida (Figura 5). Muitos grupos de pesquisa têm trabalhado na otimização do processo de produção de metabólitos recombinantes 41,42,43,44,45; na mesma linha, esta técnica de otimização estatística reduziu a duração da fermentação de 52 h para 30 h e aumentou a atividade de BaCDA de 84,67 ± 0,56 U/L (antes da otimização) para 202,39 ± 0,31 U/L (na condição otimizada), um aumento de ~ 2,39 vezes. A vantagem da otimização baseada em CCD foi que o tempo e o custo de otimização foram reduzidos com um conjunto limitado de experimentos em comparação com outras técnicas de otimização, como OFAT. Essa técnica estatística pode otimizar apenas 10 parâmetros por vez com 160 execuções experimentais. O modelo matemático não prevê com precisão a resposta além dos níveis selecionados para os parâmetros no experimento.
Em uma nota semelhante, lipase recombinante, ativina A, sacarase ramificada α-(1→2), proteína estafilocócica A (SpA) e expressão de ficina foram otimizadas usando CCD. Em todos esses exemplos, o vetor pET foi usado para expressão, e E. coli fez usado como expressão. Em todos esses estudos, a mídia LB foi utilizada como mídia de escolha. No presente estudo, o rendimento de biomassa foi aumentado usando meios de TB. Para a lipase, 12,5% de lactose a 24 °C por 15 h de fermentação aumentou a expressão em 2,1 vezes42. A expressão da ativina A foi melhorada com IPTG 1,5 mM a OD600 0,6, com uma temperatura de fermentação de 30 °C por 10 h44. No caso da sacarase, a lactose a 1% a 23 ° C por 26,5 h de fermentação aumentou a expressão em 165 vezes42. A expressão da proteína estafilocócica A (EpA) foi aumentada cinco vezes com a inclusão de lactose a 10%. A fermentação foi realizada a 33 °C com uma duração total de 11 h45. Um experimento semelhante realizado com ficina levou a um aumento de três vezes com lactose em 10%. O tempo de fermentação foi de 11 h a 24 °C43.
A cinética do perfil de expressão foi realizada com as condições otimizadas do processo. A concentração de glicose, o rendimento de biomassa e a atividade de BaCDA foram avaliados. Como a glicose é a forma mais simples de carbono, para o crescimento inicial, a E. coli utiliza a glicose primeiro. Uma vez que a glicose se esgota na mídia, a E. coli começa a utilizar lactose46. Neste estudo, a glicose (0,061%) foi esgotada em 16 h de fermentação, levando ao início do deslocamento diauxico. Houve uma segunda fase de defasagem durante essa transição de utilização de açúcar com duração de 4 h. A expressão do halofílico BaCDA aumentou fortemente durante esse deslocamento diaúxico. A cultura atingiu sua saturação em 32 h, registrando máximos na atividade de BaCDA (202,39 ± 0,31 U/L) e biomassa (17,53 ± 0,07 g/L; Figura 6).
No presente estudo, a adição de lactose e glicose no meio resultou em um crescimento diaúxico com maior atividade de BaCDA. A lactose, quando usada como autoindutor, reduz a mediação regular e as chances de contaminação46. A indução a baixas temperaturas e baixa agitação resultou na produção controlada de BaCDA halofílico como enzima solúvel. O emprego da ferramenta estatística CCD aumentou a atividade de BaCDA e reduziu o tempo de fermentação. Esta técnica pode ser aplicada posteriormente para estudos de aumento de escala para produção econômica de BaCDA. A quitosana derivatizada enzimaticamente usando BaCDA pode encontrar aplicações no setor biomédico.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Os autores gostariam de agradecer à Manipal Academy for Higher Education (MAHE) pelo fundo MAHE UNSW, e os autores gostariam de agradecer ao Conselho de Pesquisa Científica e Industrial - Grupo de Desenvolvimento de Recursos Humanos (CSIR-MHRD), Govt. da Índia por uma bolsa de pesquisa sênior, carta de premiação número 09/1165 (0007) 2K19 EMR-I datada de 31.3.2019.
Materiais
| Name | Company | Catalog Number | Comments |
| Kits | |||
| Acetate assay kit | Megazyme, Ireland | K-ACETAK | The protocol has been slightly modified and optimized to perform the assay in 96 well plate |
| Glucose estimation kit | Agappe diagnosis Ltd., India | 12018013 | The protocol has been slightly modified and optimized to perform the assay in 96 well plate |
| Chemicals | |||
| Acetic acid | Hi-media, India | AS001 | Used for preparing SDS-PAGE staining and destaing solution |
| Acrylamide | Hi-media, India | MB068 | Used for preparing SDS-PAGE gel |
| Ammonium pursulphate | Hi-media, India | MB003 | Used for preparing SDS-PAGE gel |
| Bis-acrylamide | Hi-media, India | MB005 | Used for preparing SDS-PAGE gel |
| Coomassie briliiant blue G-250 | Hi-media, India | MB092 | Used for preparing SDS-PAGE staining and destaing solution |
| Coomassie briliiant blue R-250 | Hi-media, India | MB153 | Used for preparing Bardford's assay |
| Ethylene glycol chitosan | Sigma-aldrich, USA | E1502 | Used to prepare Ethylene glycol chitin and Ethylene glycol chitin was used as substrate for enzymatic reaction |
| D-glucose | Hi-media, India | MB037 | Used as an media component. |
| Imidazole | Hi-media, India | GRM1864 | Used in lysis buffer |
| Lactose | Hi-media, India | GRM017 | Used as an media component. |
| Methanol | Finar, India | 30930LC250 | Used for preparing SDS-PAGE staining and destaing solution |
| Sodium chloride (NaCl) | Hi-media, India | MB023 | Used in lysis buffer |
| Phosphoric acid | Hi-media, India | MB157 | Used for preparing Bardford's assay |
| sodium dodecyl sulfate (SDS) | Hi-media, India | GRM6218 | Used for preparing SDS-PAGE gel |
| Sodium phosphate dibasic anhydrous | Hi-media, India | MB024 | Used to prepare TB sald for media and buffer for enzymatic reaction. |
| Sodium phosphate monobasic anhydrous | Hi-media, India | GRM3964 | Used to prepare TB sald for media and buffer for enzymatic reaction. |
| Tetramethylethylenediamine (TEMED) | Hi-media, India | MB026 | Used for preparing SDS-PAGE gel |
| Tris base | Hi-media, India | MB029 | Used for preparing SDS-PAGE gel |
| Tryptone | Hi-media, India | RM7707 | Used as an media component. |
| Yeast extract | Hi-media, India | RM027 | Used as an media component. |
| Equipment | |||
| AlphaImager HP gel documentation unit | ProteinSimple, USA | 92-13823-00 | Used to capture SDS-PAGE photographs |
| Benchtop mixer | Eppendorf, Germany | 9.776 660 | Used to keep for enzymatic reaction with 2 mL adaptor |
| Bioincubator shaker | Trishul instruments, India | 13410622 | Used to incubate bacterial culture at different temparature and RPM |
| Biospectrophotometer | Eppendorf, Germany | 6135000009 | Used to take all spectroscopic readings |
| Cooling centrifuge | Eppendorf, Germany | 5805000017 | Used to centrifuge culture, lysate and all other centrifuging protocols |
| Dry bath | Labnet International, USA | S81522039 | Used to denature protein sample for SDS-PAGE |
| Micropipettes | Eppendorf, Germany | 3123000900 | Used throghout the protocol for volume measurements |
| Rocker shaker | Trishul instruments, India | 11770719 | Used to shake SDS-PAGE gel for staining and destaining |
| SDS-PAGE unit | Bio-Rad, USA | 1658001FC | Used to cast and run SDS-PAGE gel |
| Ultra sonicator | Sonics & Materials, Inc., USA | VCX 130 | Used to lyse the bacterial cell by ultra sonication |
| Weighing balance | Sartorius, Germany | BSA124 S | Used to measure weight throughout the protocol |
| Devices | |||
| Nanosep Centrifugal Devices with Omega Membrane (3 kDa) | PALL life sciences, USA | OD003C33 | Used to separate enzyme after substrate treatment |
| Softwares | Version | Developed at | |
| MINITAB | 17.0 (Trial version) | The Pennsylvania State University | Used to design the experimental model and analyse the data |
| ImageJ | 1.53o | National Institutes of Health (NIH) | Used to analyse the expression level using SDS-PAGE image |
| Plasmid | |||
| pET22b (+) DNA—Novagen | Merck- Millipore, USA | 69744 | Stored at − 20 °C |
| Cells | |||
| E. coli Rosetta pLysS—Novagen | Merck- Millipore, USA | 70956 | Maintained in Luria–Bertani (LB) broth containing 25% glycerol at − 80 °C |
Referências
- Yadav, M., et al. Seafood waste: a source for preparation of commercially employable chitin/chitosan materials. Bioresources and Bioprocessing. 6 (1), 1-20 (2019).
- Anil, S. Potential medical applications of chitooligosaccharides. Polymers. 14 (17), 3558 (2022).
- Amiri, H., et al. Chitin and chitosan derived from crustacean waste valorization streams can support food systems and the UN Sustainable Development Goals. Nature Food. 3 (10), 822-828 (2022).
- Wattjes, J., et al. Patterns matter part 1: Chitosan polymers with non-random patterns of acetylation. Reactive and Functional Polymers. 151, 104583 (2020).
- Cord-Landwehr, S., et al. Patterns matter part 2: Chitosan oligomers with defined patterns of acetylation. Reactive and Functional Polymers. 151, 104577 (2020).
- He, Y., Xu, J., Wang, S., Zhou, G., Liu, J. Optimization of medium components for production of chitin deacetylase by Bacillus amyloliquefaciens Z7, using response surface methodology. Biotechnology and Biotechnological Equipment. 28 (2), 242-247 (2014).
- Pareek, N., Vivekanand, V., Saroj, S., Sharma, A. K., Singh, R. P. Purification and characterization of chitin deacetylase from Penicillium oxalicum SAE M-51. Carbohydrate Polymers. 87 (2), 1091-1097 (2012).
- Yonis, R. W., Luti, K. J. K., Aziz, G. M. Statistical optimization of chitin bioconversion to produce an effective chitosan in solid state fermentation by Asperigellus flavus. Iraqi Journal of Agricultural Sciences. 50 (3), 916-927 (2019).
- Deeba, F., Shakir, H. A., Irfan, M., Khan, M., Qazi, J. I. Utilization of agro-industrial waste for chitinase production by locally isolated bacillus subtilis using response surface methodology. Pakistan Journal of Zoology. 54 (2), 551-560 (2022).
- Pandey, A., Tripathi, A. H., Tripathi, P. H. Production, purification, and application of the microbial enzymes. Industrial Applications of Microbial Enzymes. , 19-39 (2022).
- Bhat, P., et al. Expression of Bacillus licheniformis chitin deacetylase in E. coli pLysS: Sustainable production, purification and characterisation. International Journal of Biological Macromolecules. 131, 1008-1013 (2019).
- Hamer, S. N., et al. Enzymatic production of defined chitosan oligomers with a specific pattern of acetylation using a combination of chitin oligosaccharide deacetylases. Scientific Reports. 5, 8716 (2015).
- Kadokura, K., et al. Production of a recombinant chitin oligosaccharide deacetylase from Vibrio parahaemolyticus in the culture medium of Escherichia coli cells. Biotechnology Letters. 29 (8), 1209-1215 (2007).
- Liu, J., et al. Identification and characterization of a chitin deacetylase from a metagenomic library of deep-sea sediments of the Arctic Ocean. Gene. 590 (1), 79-84 (2016).
- Pawaskar, G. M., Raval, K., Rohit, P., Shenoy, R. P., Raval, R. Cloning, expression, purification and characterization of chitin deacetylase extremozyme from halophilic Bacillus aryabhattai B8W22. Biotech. 11 (12), 515 (2021).
- Raval, R., Simsa, R., Raval, K. Expression studies of Bacillus licheniformis chitin deacetylase in E. coli Rosetta cells. International Journal of Biological Macromolecules. 104, 1692-1696 (2017).
- Rosano, G. L., Ceccarelli, E. A. Recombinant protein expression in Escherichia coli: Advances and challenges. Frontiers in Microbiology. 5, 172 (2014).
- Yang, G., et al. Enzymatic modification of native chitin and chitin oligosaccharides by an alkaline chitin deacetylase from Microbacterium esteraromaticum MCDA02. International Journal of Biological Macromolecules. 203, 671-678 (2022).
- Amer, O. A., et al. Exploring new marine bacterial species, Alcaligenes faecalis Alca F2018 valued for bioconversion of shrimp chitin to chitosan for concomitant biotechnological applications. International Journal of Biological Macromolecules. 196, 35-45 (2022).
- Packiam, K. A. R., Ramanan, R. N., Ooi, C. W., Krishnaswamy, L., Tey, B. T. Stepwise optimization of recombinant protein production in Escherichia coli utilizing computational and experimental approaches. Applied Microbiology and Biotechnology. 104 (8), 3253-3266 (2020).
- Jayakumar, D., Sachith, S. K., Nathan, V. K., Rishad, K. S. M. Statistical optimization of thermostable alkaline protease from Bacillus cereus KM 05 using response surface methodology. Biotechnology Letters. 43 (10), 2053-2065 (2021).
- Balakrishnan, R., Mohan, N., Sivaprakasam, S. Application of design of experiments in bioprocessing: process analysis, optimization, and reliability. Current Developments in Biotechnology and Bioengineering. , 289-319 (2022).
- Papaneophytou, C. P., Kontopidis, G. Statistical approaches to maximize recombinant protein expression in Escherichia coli: A general review. Protein Expression and Purification. 94, 22-32 (2014).
- Tang, S. R., Somasundaram, B., Lua, L. H. L. Protein expression optimization strategies in E. coli: a tailored approach in strain selection and parallelizing expression conditions. Methods in Molecular Biology. 2406, 93-111 (2022).
- Nag, N., Khan, H., Tripathi, T. Strategies to improve the expression and solubility of recombinant proteins in E. coli. Advances in Protein Molecular and Structural Biology Methods. , 1-12 (2022).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Kielkopf, C. L., Bauer, W., Urbatsch, I. L. Variations of staining sodium dodecyl sulfate-polyacrylamide gels with coomassie brilliant blue. Cold Spring Harbor Protocols. 2021 (12), (2021).
- Etemadzadeh, M. H., Arashkia, A., Roohvand, F., Norouzian, D., Azadmanesh, K. Isolation, cloning, and expression of E. coli BirA gene for biotinylation applications. Advanced Biomedical Research. 4, 149 (2015).
- Yin, L., Wang, Q., Sun, J., Mao, X. Expression and molecular modification of chitin deacetylase from Streptomyces bacillaris. Molecules. 28 (1), 113 (2023).
- Hamedi, H., Moradi, S., Hudson, S. M., Tonelli, A. E., King, M. W. Chitosan based bioadhesives for biomedical applications: A review. Carbohydrate Polymers. 282, 119100 (2022).
- Falak, S., Sajed, M., Rashid, N. Strategies to enhance soluble production of heterologous proteins in Escherichia coli. Biologia. 77 (3), 893-905 (2022).
- Shahzadi, I., et al. Scale-up fermentation of Escherichia coli for the production of recombinant endoglucanase from Clostridium thermocellum. Scientific Reports. 11 (1), 7145 (2021).
- Ganjave, S. D., Dodia, H., Sunder, A. V., Madhu, S., Wangikar, P. P. High cell density cultivation of E. coli in shake flasks for the production of recombinant proteins. Biotechnology Reports. 33, 00694 (2022).
- Wurm, D. J., et al. The E. coli pET expression system revisited-mechanistic correlation between glucose and lactose uptake. Applied Microbiology and Biotechnology. 100 (20), 8721-8729 (2016).
- Gennari, A., et al. Recombinant production in Escherichia coli of a β-galactosidase fused to a cellulose-binding domain using low-cost inducers in fed-batch cultivation. Process Biochemistry. 124, 290-298 (2023).
- Mostovenko, E., Deelder, A. M., Palmblad, M. Protein expression dynamics during Escherichia coli glucose-lactose diauxie. BMC Microbiology. 11, 126 (2011).
- Studier, F. W. Stable expression clones and auto-induction for protein production in E. coli. Methods in Molecular Biology. 1091, 17-32 (2014).
- Studier, F. W. Stable cultures and auto-induction for inducible protein production in E. coli. Methods in Molecular Biology. 1091, 17-22 (2014).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. Journal of Molecular Biology. 189 (1), 113-130 (1986).
- Selvaraj, S., Natarajan, K., Nowak, A., Murty, V. R. Mathematical modeling and simulation of newly isolated bacillus cereus M1GT for tannase production through semi-solid state fermentation with agriculture residue triphala. South African Journal of Chemical Engineering. 35 (1), 89-97 (2021).
- Ghahremanifard, P., Rezaeinezhad, N., Rigi, G., Ramezani, F., Ahmadian, G. Designing a novel signal sequence for efficient secretion of Candida antarctica lipase B in E. coli: The molecular dynamic simulation, codon optimization and statistical analysis approach. International Journal of Biological Macromolecules. 119, 291-305 (2018).
- Vuillemin, M., Malbert, Y., Laguerre, S., Remaud-Siméon, M., Moulis, C. Optimizing the production of an α-(1→2) branching sucrase in Escherichia coli using statistical design. Applied Microbiology and Biotechnology. 98 (12), 5173-5184 (2014).
- Sattari, F., Rigi, G., Ghaedmohammadi, S. The first report on molecular cloning, functional expression, purification, and statistical optimization of Escherichia coli-derived recombinant Ficin from Iranian fig tree (Ficus carica cv.Sabz). International Journal of Biological Macromolecules. 165, 2126-2135 (2020).
- Hajihassan, Z., Biroonro, N. Enhanced expression of recombinant activin A in Escherichia coli by optimization of induction parameters. Journal of Sciences, Islamic Republic of Iran. 29 (2), 105-111 (2018).
- Rigi, G., Mohammadi, S. G., Arjomand, M. R., Ahmadian, G., Noghabi, K. A. Optimization of extracellular truncated staphylococcal protein A expression in Escherichia coli BL21 (DE3). Biotechnology and Applied Biochemistry. 61 (2), 217-225 (2014).
- Faust, G., Stand, A., Weuster-Botz, D. IPTG can replace lactose in auto-induction media to enhance protein expression in batch-cultured Escherichia coli. Engineering in Life Sciences. 15 (8), 824-829 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
ISSN 2578-2614
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados
Usamos cookies para melhorar sua experiência em nosso site.
Ao continuar usando nosso site ou clicando em 'continuar', você concorda em aceitar nossos cookies.