Method Article
Quantificação da Competição Interbacteriana usando imagens de fluorescência unicelular
Neste Artigo
Resumo
Este manuscrito descreve um método para o uso de microscopia de fluorescência unicelular para visualizar e quantificar a competição bacteriana na cocultura.
Resumo
A competição interbacteriana pode impactar diretamente a estrutura e a função dos microbiomas. Este trabalho descreve uma abordagem de microscopia de fluorescência que pode ser usada para visualizar e quantificar interações competitivas entre diferentes cepas bacterianas no nível unicelular. O protocolo descrito aqui fornece métodos para abordagens avançadas na preparação de slides em microscópios de epifluorescência vertical e invertido, técnicas de imagem de células vivas e de lapso de tempo e análise quantitativa de imagens usando o software de código aberto FIJI. A abordagem neste manuscrito descreve a quantificação das interações competitivas entre as populações simbióticas vibrio fischeri medindo a mudança de área ao longo do tempo para duas cepas coincubatizadas que estão expressando diferentes proteínas fluorescentes de plasmídeos estáveis. Métodos alternativos são descritos para otimizar este protocolo em sistemas de modelos bacterianos que requerem diferentes condições de crescimento. Embora o ensaio descrito aqui use condições otimizadas para V. fischeri,essa abordagem é altamente reprodutível e pode ser facilmente adaptada para estudar a concorrência entre isolados culturais de diversos microbiomas.
Introdução
Este artigo descreve um método para quantificar a competição bacteriana no nível unicelular usando microscopia de fluorescência. A estrutura e função das comunidades microbianas é muitas vezes moldada por interações competitivas entre micróbios, e em muitos casos caracterizar essas interações requer observar diferentes cepas bacterianas na coincubação1,2,3,4,5,6,7,8 . Tradicionalmente, a competição bacteriana é quantificada a nível populacional por meio da contagem de unidades formadoras de colônias (UFC) de inibidores e cepas alvo antes e depois de um período de coincubação2,9. Os mecanismos de competição microbiana são amplamente distribuídos entre as bactérias e podem contar com difusão ou contato celular para inibir células-alvo10,11,12,13,14,15,16,17,18,19.
Embora as cepas bacterianas sejam frequentemente observadas na coincubação a nível populacional, este manuscrito descreve um ensaio para quantificação unicelular da competição bacteriana. Além disso, este trabalho inclui sugestões para adaptar o protocolo para o uso com outras espécies bacterianas. Embora as técnicas específicas deste artigo sejam utilizadas para estudar a competição intraespecífica dependente de contato entre cepas da bactéria simbiótica Vibrio fischeri20,21,22, elas podem ser adaptadas para a competição entre muitos organismos. Este artigo fornece instruções para a configuração de slides em microscópios eretos e invertidos, e toda a análise é descrita usando o software de código aberto FIJI23 para que o método possa ser usado pelos pesquisadores com acesso a diferentes configurações de imagem e programas de análise. Dada a importância de estudar a concorrência microbiana tanto no nível populacional quanto em células únicas, esse método será um recurso valioso para os pesquisadores quantificarem as interações competitivas, particularmente aquelas que não têm acesso a softwares de análise proprietários.
Protocolo
1. Otimização de cepas bacterianas
- Escolha duas cepas bacterianas para ensaios de competição bacteriana unicelular. Aqui, são utilizadas duas cepas de V. fischeri: uma cepa alvo (ES11424) e uma cepa inibidora (MJ1125) que é conhecida por matar a cepa alvo usando o sistema de secreção tipo VI no cromossomo II (T6SS2)1, que é um mecanismo de morte dependente de contato.
- Determine os controles apropriados para o experimento. Neste exemplo, o controle apropriado é incubar tanto o tipo selvagem quanto o inibidor mutante T6SS com a cepa alvo para quantificar o efeito da matança mediada por T6SS.
NOTA: Controles adicionais podem incluir uma cepa alvo que expressa o gene(s) de imunidade necessário para evitar a morte dependente de T6SS ou uma cepa mutante inibidora expressando cópias do tipo selvagem dos genes mutantes em trans para restaurar a atividade T6SS1. - Quando possível, transforme cepas com plasmídeos estáveis codificando genes para diferentes proteínas fluorescentes (por exemplo, GFP ou RFP) para distinguir visualmente os tipos de tensão no microscópio. Aqui, a cepa inibidora é marcada com um plasmídeo de codificação GFP (pVSV102), e a cepa de destino é marcada com um plasmídeo de codificação dsRed (pVSV208)26.
NOTA: Se não for possível usar plasmídeos estáveis, as etiquetas fluorescentes podem ser introduzidas no cromossomo bacteriano para visualização27,28. - Durante o período inicial de otimização, as culturas clonais de imagem das cepas marcadas sob cada um dos filtros fluorescentes que serão usados durante o experimento para garantir que as células sejam visíveis apenas no canal pretendido. Por exemplo, certifique-se de que uma cepa marcada por GFP só esteja visível no canal FITC.
2. Preparação do bloco de agarose
- Prepare a solução de almofada agarose dissolvendo 2% de agarose de baixo derretimento (w/v) em mPBS. Aqueça a solução brevemente no micro-ondas e vórtice até que a agarose esteja completamente dissolvida. Mantenha esta solução aquecida colocando-a em um banho de água de 55°C até estar pronto para usar. Consulte a seção Discussão para obter mais informações sobre a preparação de almofadas agaroses.
NOTA: Aqui, o mPBS foi preparado adicionando 20 g/L NaCl ao PBS padrão 1x. - Enrole um pedaço de fita de laboratório em torno de um slide de vidro cinco vezes. Repita este processo uma segunda vez no mesmo slide para que a distância entre as duas peças de fita seja ligeiramente menor do que a largura de um deslizamento(Figura 1A). Por exemplo, se usar tampas de 25 mm2, as peças de fita devem ser espaçadas com aproximadamente 20 mm de distância.
NOTA: Embora o número de vezes que a fita é enrolada ao redor do slide possa ser modificado para ajustar a espessura da almofada de agarose, é importante que as camadas de fita sejam da mesma altura em ambos os lados do slide para que a almofada agarose permaneça plana. - Pipeta quente agarose solução entre as duas peças de fita e imediatamente cubra com um tapa-tampa para que repouse sobre as peças de fita. Isso garantirá que a superfície da almofada agarose permaneça plana. O volume da solução agarose insustada nesta etapa deve ser suficiente para que o deslizamento de cobertura faça contato com o líquido e empurre para fora quaisquer bolhas na solução agarose. Para esta configuração em particular, 200 μL de agarose quente é suficiente.
- Deixe a almofada de agarose solidificar à temperatura ambiente por pelo menos 1 h antes do ensaio de coincubação. O passo 2.2 produzirá uma almofada de ágarose de aproximadamente 20 mm2.
- Corte esta almofada de agarose com uma lâmina de barbear em quatro, 5 mm2 almofadas para serem usadas para imagens.
NOTA: As almofadas de agarose podem ser feitas até uma semana antes do experimento e armazenadas a 4°C em uma placa de Petri vazia e estéril selada com parafilme para evitar a secagem.
3. Prepare cepas para co-incubação
- Retire cada cepa a ser usada no ensaio de coincubação de -80 °C em placas de ágar LBS complementadas com os antibióticos apropriados e incubar durante a noite a 24°C. Para este exemplo, três cepas são usadas: a cepa inibidora do tipo selvagem, o mutante do sistema de secreção tipo VI e a cepa alvo.
- No dia seguinte, inicie culturas durante a noite em duplicata biológica, escolhendo duas colônias de cada cepa e reutilizando-as em meio LBS complementada com os antibióticos apropriados e incubar durante a noite a 24°C com agitação a 200 rpm.
- Na manhã seguinte, a subcultura cada biológico replica 1:1000 em um meio LBS fresco sem antibióticos e incuba a 24°C com agitação por 4-5 h ou até que as células atinjam um OD600 de ~1,5.
NOTA: O tempo das etapas 3.1, 3.2 e 3.3 pode precisar ser otimizado para diferentes espécies bacterianas, pois sua taxa de crescimento pode variar substancialmente. Para este ensaio, as células foram destinadas a estar em fase de registro médio no início do ensaio de coincubação.
4. Cepas bacterianas coincubate
- Começando com culturas de registro médio a partir da etapa 3.3, meça e registre a densidade óptica em 600 nm (OD600) para todas as amostras.
- Normalize cada amostra para um OD600 = 1,0, o que corresponde a aproximadamente 109 UFC/mL para V. fischeri,diluindo a cultura com meio LBS.
- Misture as duas cepas concorrentes em uma razão de 1:1 com base no volume adicionando 30 μL de cada cepa normalizada a um tubo de 1,5 mL rotulado. Vórtice a cultura de tensão mista para 1-2 s.
NOTA: Em alguns casos, pode ser apropriado misturar coculturas em diferentes proporções. Por exemplo, quando uma cepa cresce muito mais rápido que a outra, pode ser necessário iniciar a tensão de crescimento mais lento a uma vantagem numérica para observar a competição. A otimização também pode ser necessária se o OD600 não corresponder a CFU/mL semelhante para ambas as cepas. - Repita o passo 4.3 para cada réplica biológica e tratamento. No exemplo aqui mostrado, isso resultará em um total de quatro tubos de cepa mista: duas réplicas biológicas com a cepa inibidora do tipo selvagem misturada com a cepa alvo e duas réplicas biológicas com a cepa mutante do sistema de secreção tipo VI misturada com a cepa alvo.
- Para garantir que as células concorrentes sejam suficientemente densas para matar dependente de contato na cunhagem no ágar pad, concentre cada cultura mista 3 vezes por centrifugação da cultura mista em um tubo de centrífugas padrão de 1,5 mL por 1 min a 21.130 x g,descartando o supernatante, e resusentando cada pelota em 20 μL LBS médio. Repita para cada amostra.
NOTA: Algumas células bacterianas são sensíveis a danos por centrifugação em alto rcf; nesses casos, a cultura mista pode ser centrifuada por 3 min a 4600 x g29. Além disso, ao quantificar a concorrência dependente de contato, é importante garantir densidade celular suficiente no slide para observar a matança. Neste artigo, os tratamentos "lotados", onde o assassinato é observado, tinham aproximadamente 10 células/20 μm2; consulte a seção Discussão para obter mais informações.
5. Configuração de slides
- Ao usar um microscópio vertical, coloque uma almofada de agarose de~5 mm 2 em um slide de vidro padrão de 1 mm. Local 2 μL de uma cultura mista sobre a almofada de agarose e coloque um deslizamento de cobertura #1,5 (25 mm2) sobre o local. Consulte a Figura 1B para obter um exemplo.
- Ao usar um microscópio invertido, local 2 μL de uma cultura mista no fundo de cobertura #1.5 de uma placa de Petri de 35 mm e coloque uma almofada de 5 mm2 sobre o ponto de agação. Coloque uma tampa de vidro circular de 12 mm sobre a almofada de agarose. Consulte a Figura 1C para obter um exemplo.
- Repita o passo 5.1 ou 5.2, dependendo da configuração do microscópio utilizado, para as três culturas mistas restantes, resultando em quatro slides ou pratos a serem imageados.
- Deixe que os slides se sentem no banco por aproximadamente 5 minutos antes de prosseguir para a etapa 6. Isso permite que as células se instalem no ágar pad e elimina o movimento durante o processo de imagem.
6. Microscopia de fluorescência
- Comece focando nas células usando luz branca (contraste de fase ou DIC) para minimizar os efeitos do branqueamento fotográfico. Com base no tamanho médio de uma única célula bacteriana, use um objetivo de óleo de 60x ou 100x.
- Ajuste o tempo de exposição e as configurações de aquisição de cada canal para que as células sejam visíveis no canal apropriado com detecção mínima de fundo.
NOTA: É apropriado usar diferentes tempos de exposição para diferentes canais, mas o mesmo tempo de exposição deve ser usado em todas as réplicas biológicas e tratamentos para um determinado canal. - Para cada amostra, selecione pelo menos cinco campos de exibição (FOV) e adquira imagens em cada canal apropriado utilizando as configurações de aquisição da etapa 6.2 (Veja exemplos na Figura 2). Salve os pontos XY de cada FOV para que o mesmo FOV possa ser imagedo durante o ponto de tempo final. A imagem do mesmo FOV em cada ponto de tempo é necessária para determinar a proporção de área ocupada por células alvo ou inibidoras durante as etapas de análise.
NOTA: Neste exemplo, a fluorescência do GFP é detectada usando um filtro com comprimento de onda de excitação de 467 - 498 nm e um filtro de emissão de 513 - 556 nm e é verde de cor falsa. A fluorescência de dsRed é detectada usando um filtro com um comprimento de onda de excitação de 542 - 582 nm e um filtro de emissão de 603 - 678 nm e é magenta de cor falsa. - Após 2h, repita o passo 6.3 para cada amostra usando os pontos XY previamente salvos(Fig 2).
NOTA: O tempo das imagens subsequentes pode precisar ser otimizado para organismos com diferentes taxas de crescimento ou mecanismos competitivos.
7. Análise de imagem em FIJI
- Baixe e instale o software de processamento de imagens FIJI usando as instruções encontradas aqui: https://imagej.net/Fiji/Downloads
- Abra FIJI e importe arquivos de imagem para análise.
NOTA: Na maioria dos casos . Os arquivos TIFF serão usados para análise de imagens, embora algum software de aquisição de imagens seja exportado usando tipos de arquivos proprietários. O FIJI pode reconhecer que a maioria dos tipos de arquivos e imagens proprietários podem ser importadas e analisadas da seguinte forma. - Para cada imagem adquirida nas etapas 6.3 e 6.4, converta a imagem em escala de cinza, separe os canais e comece por limiar (Ctrl + Shift + T) e crie uma máscara binária da imagem pré-processada(Figura 3A,B).
NOTA: Aqui, as configurações de limiar padrão em FIJI são usadas. Em alguns casos, pode ser necessário alterar essas configurações, nesse caso as mesmas configurações devem ser usadas para todas as imagens desse experimento. - Escala de conjunto na imagem (Analisar | Escala definida) utilizando os valores apropriados para a configuração de microscopia23.
- Medidas de conjunto (Analisar | Definir medidas) e selecionar Área.
NOTA: Outras medidas podem ser adicionadas se forem apropriadas para o experimento. Apenas a medição da área do objeto é necessária para a análise de exemplo mostrada aqui. - Analisar partículas (Analisar | Analisar partículas) usando as configurações padrão(Figura 3C). Se houver detritos na amostra, pode ser necessário ajustar o tamanho ou a circularidade para filtrar partículas não celulares. Selecione Mostrar | Contornos para que a saída desta análise inclua um contorno numerado de todas as partículas analisadas(Figura 3D).
NOTA: Comparar o contorno na Figura 3D com a imagem inicial é especialmente importante na etapa de otimização para garantir que (1) todas as células estejam sendo analisadas e (2) que quaisquer detritos sejam excluídos da análise. - Exporte as medidas a partir da etapa 7.4 (Figura 3E) em um software de planilha para análise e grafia posterior.
- Repita os passos 7.1 - 7.5 para todos os canais e imagens adquiridos durante o experimento.
8. Calcular a porcentagem da área alvo inicial ao longo do tempo
- Para cada campo de visão analisado na seção 7, certifique-se de que o arquivo exportado contenha uma medição individual da área para cada partícula analisada. Começando pelo canal de fluorescência da cepa alvo, calcule a área de partícula da soma para cada campo de visão individual. Para duas réplicas biológicas com cinco FOV cada, isso deve resultar em dez áreas de soma por tratamento em cada ponto de tempo.
- Calcule a porcentagem da área alvo inicial ao longo do tempo para cada FOV usando a seguinte equação: (
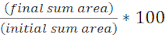 )
) - Repita este cálculo para todos os tratamentos e gráfico o percentual da área alvo inicial (resultado da equação a partir da etapa 8.2) para cada tratamento(Figura 4A).
- Determinar se há aumento líquido da população-alvo (indicando crescimento), uma diminuição líquida da população-alvo (indicando morte) ou nenhuma mudança (não indicando crescimento ou morte) para cada tratamento.
NOTA: Por cento da área alvo inicial com valores superiores a 100 indicam crescimento de alvos líquidos, e valores inferiores a 100 indicam morte de alvo líquido. Por cento dos valores-alvo iniciais que permanecem em 100 indicam nenhuma mudança líquida na população-alvo. Veja a discussão para experimentos de acompanhamento sugeridos.
9. Calcular a porcentagem da área inibidora inicial ao longo do tempo
- Repetir as etapas 8.1 a 8.3, desta vez utilizando as medidas coletadas do canal de fluorescência da cepa inibidora na seção 7 (Figura 4B).
- Determinar se houve aumento líquido da população inibidora (crescimento); uma diminuição líquida da população inibidora (morte), ou nenhuma mudança para cada tratamento. Valores superiores a 100 indicam crescimento líquido do inibidor, e valores inferiores a 100 indicam morte do inibidor líquido.
Resultados
Para visualizar e quantificar interações competitivas entre bactérias no nível unicelular, um protocolo foi desenvolvido e otimizado para V. fischeri modificando nosso bem estabelecido ensaio baseado em CFU1,2. Este método utiliza plasmídeos estáveis de codificação GFP e dsRed para distinguir visualmente diferentes cepas de V. fischeri. O resultado competitivo dessas interações pode ser quantificado analisando as imagens adquiridas a partir deste ensaio usando o software de código aberto FIJI. Como exemplo, o experimento a seguir foi realizado utilizando-se isoladores V. fischeri. Uma cepa inibidora abrigava um plasmídeo que codifica gfp, e uma cepa alvo abrigava um plasmídeo que codifica dsRed. Dado que o T6SS2 codificado pelo inibidor é um mecanismo de morte dependente de contato, foram incluídos tratamentos onde as células estavam lotadas (contato com células altas) ou dispersam (contato com células baixas) em um slide para destacar o impacto da configuração experimental nos resultados finais deste ensaio. Nos dados da amostra, as cepas concorrentes foram misturadas em uma razão de 1:1 e incubadas em uma almofada de agarose por 2h, e ambas as imagens iniciais e finais (2h) foram tiradas. Como controle, uma cepa mutante T6SS2 também foi cunhada com a tensão alvo em condições de aglomeração e dispersão. Culturas de cada cepa foram preparadas e cunhadas como descrito acima e slides foram preparados como mostrado na Figura 1.
A Figura 2 mostra imagens representativas de microscopia de fluorescência de cada tratamento experimental com o mesmo campo de visão visualizado em um ponto de tempo inicial e final. Para cada tratamento, um inibidor do tipo selvagem ou uma cepa mutante T6SS que abriga um plasmídeo codificador de GFP foi misturado a uma proporção de 1:1 com a cepa alvo abrigando um plasmídeo de codificação dsRed. Durante um período de coincubação de 2 h com esta configuração experimental, as células V. fischeri em crescimento podem passar por 1-2 divisões(Figura 2; setas cinza). Na Figura 2A,o contato celular-célula foi forçado entre o alvo e o inibidor concentrando a cultura mista antes de detectar no slide. Várias células-alvo são observadas para se tornarem arredondadas e/ou desaparecerem ao longo de 2h, consistentes com as células-alvo sendo eliminadas pelo inibidor(Figura 2; setas brancas). Consulte a seção Discussão para obter mais informações sobre como interpretar células-alvo de arredondamento ou de lise. Na Figura 2B,a mesma cunhagem foi vista em um slide, desta vez sem concentrar a cultura mista para que as células permanecessem dispersas e houvesse contato mínimo entre as cepas no slide. Aqui, não são observadas células-alvo para desaparecer ou arredondar, sugerindo que a cepa alvo não foi inibida neste tratamento. Figura 2C e Figura 2D mostram os mesmos tratamentos lotados e dispersos descritos acima, desta vez usando um mutante T6SS como a cepa inibidora. As células-alvo não foram observadas para desaparecer ou arredondar quando coincubated com um mutante T6SS em condições lotadas ou dispersas, novamente sugerindo que o alvo não foi inibido em nenhum dos tratamentos.
A Figura 3 mostra o fluxo de trabalho de análise FIJI usado para quantificar a concorrência neste protocolo. Uma imagem representativa do canal alvo foi selecionada(Figura 3A) e uma máscara binária foi criada usando as configurações de limiar padrão em FIJI (Figura 3B). A escala de imagem foi definida apropriadamente para esta configuração de microscopia. As partículas foram analisadas utilizando-se o parâmetro de tamanho = 0 - infinito, parâmetro de circularidade = 0,00 - 1,00, e foram selecionados Contornos de Show (Figura 3C). Os resultados desta análise de partículas são mostrados tanto como um contorno numerado de cada partícula (Figura 3D), quanto como uma tabela com colunas para o número de partículas, nome do arquivo (rótulo) e área de partículas em μm2 (área)(Figura 3E).
Na Figura 4,os dados obtidos da Figura 3E são gráficos e analisados. Na Figura 4A,o percentual da área alvo inicial no ponto de tempo final é apresentado para cada tratamento de acordo com a etapa 8.2. Se o percentual da área alvo inicial for superior a 100, isso representa aumento líquido da meta (ou seja, crescimento) e é observado em condições em que a população-alvo não é significativamente inibida. No entanto, se o percentual da área alvo inicial for inferior a 100, esse resultado indica uma diminuição líquida da meta (ou seja, morte) e é observado em condições em que a população-alvo é significativamente inibida. Quando o alvo foi cunhado com um inibidor do tipo selvagem em condições lotadas, os dados mostram uma diminuição líquida na área alvo. Em contraste, quando o alvo foi cunhado com um inibidor do tipo selvagem em condições de dispersão ou um mutante T6SS em condições de aglomeração ou dispersão, os dados mostram um aumento líquido na área alvo. O percentual da área alvo inicial quando o alvo foi cunhado com um inibidor do tipo selvagem em condições lotadas foi inferior a 100 e significativamente menor do que todos os outros tratamentos de acordo com um ANOVA unidirecional seguido de um teste de comparações múltiplas de um Tukey em todos os tratamentos(p < 0,0001). Esses dados indicam que a morte celular alvo depende de um T6SS funcional no inibidor e ressalta a importância de uma configuração experimental que permite contato celular-célula suficiente, a fim de detectar a morte celular de um mecanismo de morte dependente de contato.
A Figura 4B apresenta a porcentagem da área inibidora inicial no ponto de tempo final para cada tratamento. Neste exemplo, observou-se crescimento líquido da cepa inibidora em todos os tratamentos. No entanto, o percentual da área inibidora inicial foi significativamente maior quando um inibidor do tipo selvagem foi cunhado com o alvo em condições lotadas em comparação com todos os outros tratamentos de acordo com um ANOVA unidirecional seguido de um teste de comparações múltiplas de um Tukey em todos os tratamentos(p < 0,0001). Inicialmente, consideramos que o aumento líquido da área inibidora pode ser impulsionado pelo aumento do espaço disponível para crescer à medida que as células-alvo são eliminadas. No entanto, esse mesmo aumento no crescimento inibidor não foi observado em tratamentos de dispersão, onde as células inibidoras tiveram espaço para crescer desde o início da coincubação. Alternativamente, esse resultado poderia sugerir que os nutrientes liberados das células-alvo permitem um aumento maior da população inibidora. Juntos, esses resultados sugerem que a cepa inibidora elimina o alvo de forma dependente de T6SS apenas quando o contato de células altas é forçado por células de aglomeração no slide.

Figura 1: Preparação do bloco de agarose e configuração de slides para ensaios de coincubação. (A) Configuração para fazer 2% de almofadas de agarose. Cinco camadas de fita de laboratório (verde) são enroladas em torno de um deslizamento de cobertura em dois pontos aproximadamente 20 mm de distância. Em seguida, o calor de 2% de agarose em mPBS (amarelo) é esboçado entre os pedaços de fita e imediatamente coberto com um deslizamento de cobertura de 25 mm2 e permitido solidificar por pelo menos 1h à temperatura ambiente. Use uma lâmina de barbear para cortar a almofada de agarose em ~5 mm2 peças e use pinças para transferir a almofada para um novo slide para imagem. (B) Ao fotografar um microscópio vertical, coloque a almofada de 5 mm2 diretamente sobre o slide, siga a cultura mista (azul) e um deslizamento de cobertura circular de 12 mm #1.5. (C) Ao fotografar um microscópio invertido, localhe a cultura mista diretamente no deslizamento de tampa de vidro #1,5 de uma placa de Petri de 35 mm, e coloque uma almofada de agarose em cima da cultura seguida de um segundo deslizamento de cobertura circular de 12 mm para achatar a almofada de ágarose. Clique aqui para ver uma versão maior desta figura.

Figura 2: Imagens de lapso de tempo de pontos de cunhagem em condições lotadas ou dispersas. (A) Imagens representativas em pontos de tempo iniciais e finais onde uma cultura mista de alvo e inibidor do tipo selvagem estava concentrada 3x antes de detectar no slide para forçar o contato celular entre cepas. Setas brancas no canal TRITC indicam exemplos de células-alvo que rondam ou lise ao longo do experimento. (B) Imagens representativas onde uma cultura mista de alvo e inibidor do tipo selvagem foi detectada sem concentração para que as células se dispersem e haja um mínimo contato celular-célula entre cepas. Setas cinza nos canais FITC e TRITC indicam exemplos de divisão celular ao longo do experimento. (C) Imagens representativas onde a cultura mista do alvo e do T6SS- mutante estava concentrada 3x antes de detectar no slide para forçar o contato celular entre cepas. (D) Imagens representativas onde a cultura mista do alvo e do T6SS- mutante foi detectado sem concentração para que as células se dispersem e haja um contato mínimo celular-célula entre cepas. Barras de escala = 5 μm e são consistentes em todas as imagens; O canal TRITC é magenta de cor falsa, o canal FITC é verde de cor falsa. A desconvolução foi realizada em todas as imagens; fundo foi subtraído, e brilho/contraste ajustado uniformemente em todas as imagens. Clique aqui para ver uma versão maior desta figura.
Figura 3: Fluxo de trabalho de análise FIJI. (A) Imagem representativa para análise. Este fluxo de trabalho é repetido para ambos os canais em todos os campos de visão e amostras. Barras de escala = 5 μm e são consistentes em todas as imagens; O canal TRITC é magenta de cor falsa, o canal FITC é verde de cor falsa. (B) Máscara binária criada pelo limiar da imagem usando as configurações padrão em FIJI. (C) Exemplo de configurações para análise de partículas utilizadas neste manuscrito. Faixa de tamanho = 0 - infinito μm2; circularidade = 0,00 - 1,00; mostrar = contornos. (D) Contorno de partículas criado como saída de análise de partículas em (C). O contorno de partículas em(D)deve ser comparado com a imagem original (A) para garantir que todas as células foram capturadas na análise de partículas. (E) Tabela de resultados criada como saída a partir da análise de partículas em (C). O número do objeto (coluna 1) corresponde a partículas individuais (uma ou mais células) descritas e rotuladas em vermelho no painel(D). Rótulo = nome do arquivo da imagem analisada; Área = área total de partículas em μm2. Clique aqui para ver uma versão maior desta figura.
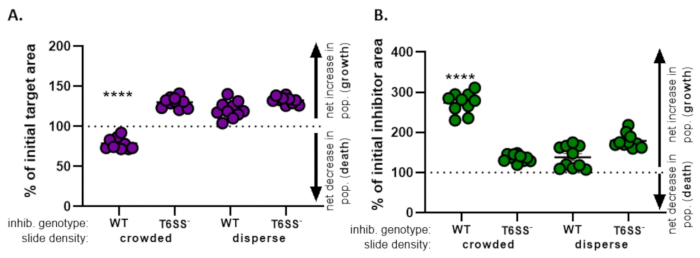
Figura 4: Dados amostrais para avaliar se a cepa alvo é inibida. A porcentagem da área inicial nos pontos de tempo final para a cepa alvo (A) e a cepa inibidora(B),em diferentes densidades celulares iniciais. A densidade do slide indica uma densidade celular inicial que está lotada (alto contato celular entre cepas) ou mais dispersão (baixo contato celular entre cepas) conforme descrito na Figura 2. O genótipo inibidor indica que uma cepa de tipo selvagem ou mutante T6SS (T6SS- )foi cunhado com a cepa alvo. Os asteriscos indicam uma diferença significativa na % de mudança comparando todos os tratamentos (ANOVA unidirecional seguido de um teste de comparações múltiplas de um Tukey comparando todos os tratamentos; (p < 0,0001). A linha tracejada não indica nenhuma alteração líquida na área de tensão entre o ponto de tempo inicial e final; a % mudança > 100 indica aumento líquido (ou seja, crescimento) e % mudança < 100 indica queda líquida (ou seja, morte celular). Clique aqui para ver uma versão maior desta figura.
Discussão
O protocolo descrito acima fornece uma poderosa ferramenta para quantificar e caracterizar a competição interbacteriana no nível de célula única. Este ensaio, que foi desenvolvido modificando nosso ensaio de competição baseado em CFU em placasde ágar 1,2, permitiu a visualização da competição unicelular entre isolados e sugestões de V. fischeri para otimizar o método para uma ampla gama de sistemas e configurações de microscopia. Embora o método descrito aqui tenha sido otimizado para o simbionte de órgãos leves V. fischeri,ele pode ser facilmente modificado para acomodar muitos micróbios diversos e culturais. É importante notar que mecanismos competitivos podem ser regulados por qualquer número de variáveis ambientais, incluindo temperatura, salinidade e viscosidade30,31,32,33,34. Trabalhos anteriores confirmaram que V. fischeri compete utilizando um sistema de secreção tipo VI dependente de contato que está ativo nas superfícies30, tornando as condições descritas neste ensaio adequadas para o estudo da competição entre as cepas de exemplo. Também é importante considerar a densidade inicial das células no slide ao quantificar a competição bacteriana. Dado que o contato entre células alvo e inibidoras é frequentemente necessário para que a matança ocorra, a cultura mista deve ser concentrada de tal forma que o contato celular-célula é maximizado e as células permanecem em um único plano na lâmina. As culturas celulares devem ser cultivadas para uma densidade óptica semelhante (fase de tronco médio) e, em seguida, concentradas para forçar o contato em vez de simplesmente cultivar culturas para uma densidade óptica maior devido às mudanças fisiológicas das células em diferentes fases de crescimento. Em outros sistemas, as condições de cultura e a configuração experimental podem precisar ser modificadas para garantir que o mecanismo competitivo esteja ativo e possa ser detectado na condição de coincubação.
As almofadas de agarose usadas neste ensaio proporcionam vários benefícios: elas fornecem estabilização para que as células não se movam livremente, e impedem que a cultura seque ao longo do experimento. Além disso, se os indutores químicos, como isopropílico-β-D-thiogalactoside (IPTG), são necessários para o experimento, eles podem ser facilmente adicionados à solução agarose. No entanto, é importante notar que a preparação de agarose provavelmente precisará ser ajustada para diferentes sistemas. No exemplo descrito acima, a almofada agarose foi preparada dissolvendo 2% de agarose (w/v) em 20 psu mPBS, que é a salinidade padrão usada no meio de crescimento V. fischeri. Além disso, em alguns casos, uma fonte de carbono pode precisar ser adicionada à almofada de agarose para que as células cresçam e concorram por experimentos mais longos. Nesse caso, o mPBS em almofadas de agarose pode ser substituído por qualquer meio de crescimento, embora os nutrientes no meio de crescimento possam vir com a troca de fluorescência de fundo adicional.
Sem um software proprietário de análise de imagem, pode ser muito difícil obter contagem individual de células quando o contato celular é alto, o que, como mostramos aqui, é necessário observar mortes dependentes de contato. Este ensaio foi projetado para fornecer um método alternativo de quantificação que não depende de contagem de células individuais. Em vez disso, a área celular total para cada canal de fluorescência é usada para quantificar a extensão da matança entre cepas coincubatizadas. Como este método se baseia em área em vez de contagens de células individuais, as configurações de limiar padrão são tipicamente suficientes para delinear a área total da célula. A precisão do limiar pode ser verificada dividindo a área total do objeto para um campo de visão representativo pelo tamanho médio da célula para o organismo modelo e comparando este número celular estimado com uma contagem manual de células para a mesma imagem.
Em co-empréstimos entre um inibidor e um alvo (não-assassino), prevê-se o crescimento líquido do inibidor. Como visto na Figura 4,o crescimento do inibidor pode ser significativamente maior nos tratamentos onde a matança é observada, em comparação com os tratamentos onde a matança não é observada, talvez porque os nutrientes liberados pelas células-alvo permitem que a cepa inibidora cresça mais rapidamente. No exemplo aqui mostrado, a morte por alvo líquido é observada porque a concorrência mediada pelo T6SS resulta em lise celular alvo onde o alvo é fisicamente eliminado. No entanto, é importante notar que nem todos os mecanismos competitivos resultam na eliminação física das células-alvo. Se uma meta é incapacitada por uma toxina que causa inibição do crescimento, o protocolo aqui descrito pode fazer com que a população-alvo visível se adeque estável ao longo do tempo, pois as células-alvo não crescem mais, mas também não crescem. Nesse caso, seria apropriado comparar os resultados deste ensaio com testes de acompanhamento para viabilidade celular alvo, como revestimento para unidades formadoras de colônias (UFC) ou por meio da realização de ensaios mortos vivos por manchas com iodeto de propídio ou SYTOX verde35,36.
Comparado aos ensaios de coincubação que dependem das contagens da UFC, este ensaio permite observar e quantificar a estrutura espacial da competição entre cepas e rastrear mudanças na morfologia celular alvo ao longo do tempo. Por exemplo, as células inibidoras que matam usando um T6SS são conhecidas por codificar proteínas de domínio lysM que degradam a parede celular alvo, resultando em arredondamento inicial celular e, em seguida, lise13, que observamos no exemplo mostrado na Figura 2A. Além disso, este protocolo pode ser usado para acompanhar a concorrência em alta resolução em escalas de tempo muito curtas. No exemplo aqui mostrado, observa-se uma diminuição significativa na área alvo após apenas duas horas em que as células estão lotadas e o contato celular-célula é forçado entre as cepas(Figura 4). A análise de imagem aqui descrita também poderia ser realizada por meio de microscopia confocal, o que permitiria estudar a concorrência bacteriana in vivo ou em biofilmes complexos, sem interromper a distribuição espacial de cepas coincubatadas.
Em resumo, o ensaio aqui descrito visa fornecer uma abordagem acessível e facilmente modificada para visualizar e quantificar a competição bacteriana no nível unicelular usando microscopia de fluorescência. Este método pode ser aplicado a diversos isolados bacterianos e pode ser usado para visualizar a competição bacteriana mesmo em ambientes complexos, como dentro de uma matriz hospedeira ou biofilm.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
A A.N.S foi apoiada pela bolsa NIGMS R35 GM137886 e a S.N.S foi apoiada pelo National Defense Science and Engineering Graduate Fellowship Program.
Materiais
| Name | Company | Catalog Number | Comments |
| 1.5 mL Microcentrifuge tube | Fisher | 05-408-129 | |
| 10 uL single channel pipette | |||
| 1000 uL single channel pipette | |||
| 20 uL single channel pipette | |||
| 200 uL single channel pipette | |||
| Agarose | Fisher | BP165-25 | Low melting agarose |
| Calculator | |||
| Cellvis 35 mm Dish | Fisher | NC0409658 | #1.5 cover glass bottom |
| Chloramphenicol | Sigma | C0378 | stock (20 mg/mL in Ethanol); final concentration in media (2 μg /mL LBS) |
| DAPI Nucleic Acid Stain | Fisher | EN62248 | optional (if not using stable plasmids) |
| FIJI image analysis sofware | ImageJ | https://imagej.net/Fiji/Downloads | open-source software |
| Fisherbrand Cover Glasses: Circles | Fisher | 12-545-81P | #1.5 cover glass; 12 mm diameter |
| Kanamycin Sulfate | Fisher | BP906-5 | stock (100 mg/mL in water, filter sterilize); final concentration in media (1 μg/mL LBS) |
| Lens Cleaning Tissue Paper | Fisher | S24530 | |
| Parafilm | Fisher | 13-374-12 | |
| Petri Plates | Fisher | FB0875713 | sterile with lid |
| Razor Blades | Fisher | S65921 | |
| Semi-micro Cuvettes | VWR | 97000-586 | |
| Spectrophotometer | |||
| SYBR Green Nucleic Acid Stain | Fisher | S7563 | optional (if not using stable plasmids) |
| Thermo Scientific Gold Seal Plain Microscope Slides | Fisher | 12-518-100B | |
| Thermo Scientific Richard-Allan Scientific Cover Glass | Fisher | 22-050-235 | #1.5 cover glass, 25 mm2 |
| Type F Immersion Oil | Fisher | NC0297589 | |
| Upright or inverted fluorescence microscope with camera and imaging software | Images in this article were acquired on a Nikon TI-2 inverted fluorescent microscope outfitted with an ORCA-Fusion Digital CMOS camera using NIS-Elements software. | ||
| Vortex | |||
| Water bath | Used to keep agarose warm prior to pipetting | ||
| LBS media | |||
| 1M Tris Buffer (pH ~7.5) | 50 mL 1 M stock buffer (62 mL HCl, 938 mL DI water, 121 g Trizma Base) | ||
| Agar Technical | Fisher | DF0812-17-9 | 15 g (Add only for plates) |
| DI water | 950 mL | ||
| Sodium Chloride | Fisher | S640-3 | 20 g |
| Tryptone | Fisher | BP97265 | 10 g |
| Yeast Extract | Fisher | BP9727-2 | 5 g |
| mPBS (marine PBS) | Phosphate buffered saline with marine salts added; used for making agarose pad | ||
| 10X PBS | Fisher | ICN1960454 | |
| Instant Ocean Sea Salt | Instant Ocean | SS1-160P | Adjust concentration to appropriate salinity; 20 psu used here |
| Sterile Vacuum Filter Units | Fisher | SCGVU01RE | Used to filter-sterilize mPBS |
| Vacuum pump | Used to filter-sterilize mPBS |
Referências
- Speare, L., et al. Bacterial symbionts use a type VI secretion system to eliminate competitors in their natural host. Proceedings of the National Academy of Sciences. 115 (36), 8528-8537 (2018).
- Speare, L., Septer, A. N. Coincubation assay for quantifying competitive interactions between Vibrio fischeri isolates. Journal of Visualized Experiments. (149), e59759 (2019).
- Frost, I., et al. Cooperation, competition and antibiotic resistance in bacterial colonies. The ISME journal. 12 (6), 1582-1593 (2018).
- Stubbendieck, R. M., Vargas-Bautista, C., Straight, P. D. Bacterial communities: interactions to scale. Frontiers in Microbiology. 7, 1234 (2016).
- Souza, D. P., et al. Bacterial killing via a type IV secretion system. Nature Communications. 6 (1), 1-9 (2015).
- Anderson, M. C., Vonaesch, P., Saffarian, A., Marteyn, B. S., Sansonetti, P. J. Shigella sonnei encodes a functional T6SS used for interbacterial competition and niche occupancy. Cell Host and Microbe. 21 (6), 769-776 (2017).
- Basler, M., Ho, B., Mekalanos, J. Tit-for-tat: Type VI secretion system counterattack during bacterial cell-cell interactions. Cell. 152 (4), 884-894 (2013).
- Guillemette, R., Ushijima, B., Jalan, M., Häse, C. C., Azam, F. Insight into the resilience and susceptibility of marine bacteria to T6SS attack by Vibrio cholerae and Vibrio coralliilyticus. PloS One. 15 (1), 0227864 (2020).
- Hachani, A., Lossi, N. S., Filloux, A. A visual assay to monitor T6SS-mediated bacterial competition. Journal of Visualized Experiments. (73), e50103 (2013).
- Hibbing, M. E., Fuqua, C., Parsek, M. R., Peterson, S. B. Bacterial competition: surviving and thriving in the microbial jungle. Nature Reviews Microbiology. 8 (1), 15-25 (2010).
- Ruhe, Z. C., Low, D. A., Hayes, C. S. Bacterial contact-dependent growth inhibition. Trends in Microbiology. 21 (5), 230-237 (2013).
- Wood, D. W., Pierson, L. S. The phzI gene of Pseudomonas aureofaciens 30-84 is responsible for the production of a diffusible signal required for phenazine antibiotic production. Gene. 168 (1), 49-53 (1996).
- Smith, W. P., et al. The evolution of the type VI secretion system as a disintegration weapon. PLoS Biology. 18 (5), 3000720 (2020).
- Chen, L., Zou, Y., She, P., Wu, Y. Composition, function, and regulation of T6SS in Pseudomonas aeruginosa. Microbiological Research. 172, 19-25 (2015).
- Sana, T. G., Lugo, K. A., Monack, D. M. T6SS: The bacterial "fight club" in the host gut. PLoS Pathogens. 13 (6), 1006325 (2017).
- Basler, M. Type VI secretion system: secretion by a contractile nanomachine. Philosophical Transactions of the Royal Society B: Biological Sciences. 370 (1679), 20150021 (2015).
- Joshi, A., et al. Rules of engagement: the type VI secretion system in Vibrio cholerae. Trends in Microbiology. 25 (4), 267-279 (2017).
- Nadell, C. D., Drescher, K., Foster, K. R. Spatial structure, cooperation and competition in biofilms. Nature Reviews Microbiology. 14 (9), 589-600 (2016).
- Stubbendieck, R. M., Straight, P. D. Multifaceted interfaces of bacterial competition. Journal of Bacteriology. 198 (16), 2145-2155 (2016).
- Septer, A. N. The Vibrio-squid symbiosis as a model for studying interbacterial competition. Msystems. 4 (3), (2019).
- Tischler, A. H., Hodge-Hanson, K. M., Visick, K. L. Vibrio fischeri-squid symbiosis. eLS. , 1-9 (2019).
- Mandel, M. J., Dunn, A. K. Impact and influence of the natural Vibrio-squid symbiosis in understanding bacterial-animal interactions. Frontiers in Microbiology. 7, 1982 (2016).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Boettcher, K., Ruby, E. Depressed light emission by symbiotic Vibrio fischeri of the sepiolid squid Euprymna Scolopes. Journal of Bacteriology. 172 (7), 3701-3706 (1990).
- Doino, J. A., McFall-Ngai, M. J. A transient exposure to symbiosis-competent bacteria induces light organ morphogenesis in the host squid. The Biological Bulletin. 189 (3), 347-355 (1995).
- Dunn, A. K., Millikan, D. S., Adin, D. M., Bose, J. L., Stabb, E. V. New rfp-and pES213-derived tools for analyzing symbiotic Vibrio fischeri reveal patterns of infection and lux expression in situ. Applied and Environmental Microbiology. 72 (1), 802-810 (2006).
- Lambertsen, L., Sternberg, C., Molin, S. Mini-Tn7 transposons for site-specific tagging of bacteria with fluorescent proteins. Environmental Microbiology. 6 (7), 726-732 (2004).
- Koch, B., Jensen, L. E., Nybroe, O. A panel of Tn7-based vectors for insertion of the gfp marker gene or for delivery of cloned DNA into Gram-negative bacteria at a neutral chromosomal site. Journal of Microbiological Methods. 45 (3), 187-195 (2001).
- Peterson, B. W., Sharma, P. K., Van Der Mei, H. C., Busscher, H. J. Bacterial cell surface damage due to centrifugal compaction. Applied and Environmental Microbiology. 78 (1), 120-125 (2012).
- Speare, L., Smith, S., Salvato, F., Kleiner, M., Septer, A. N. Environmental viscosity modulates interbacterial killing during habitat transition. MBio. 11 (1), (2020).
- Salomon, D., Gonzalez, H., Updegraff, B. L., Orth, K. Vibrio parahaemolyticus type VI secretion system 1 is activated in marine conditions to target bacteria, and is differentially regulated from system 2. PloS One. 8 (4), 61086 (2013).
- Sana, T. G., et al. Salmonella Typhimurium utilizes a T6SS-mediated antibacterial weapon to establish in the host gut. Proceedings of the National Academy of Sciences. 113 (34), 5044-5051 (2016).
- Bachmann, V., et al. Bile salts modulate the mucin-activated type VI secretion system of pandemic Vibrio cholerae. PLoS Neglected Tropical Diseases. 9 (8), 0004031 (2015).
- Ishikawa, T., et al. Pathoadaptive conditional regulation of the type VI secretion system in Vibrio cholerae O1 strains. Infection and Immunity. 80 (2), 575-584 (2012).
- Johnson, M. B., Criss, A. K. Fluorescence microscopy methods for determining the viability of bacteria in association with mammalian cells. Journal of Visualized Experiments. (79), e50729 (2013).
- Stiefel, P., Schmidt-Emrich, S., Maniura-Weber, K., Ren, Q. Critical aspects of using bacterial cell viability assays with the fluorophores SYTO9 and propidium iodide. BMC Microbiology. 15 (1), 36 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados