Method Article
In Vitro Evolução dirigida de uma endonuclease de restrição com especificidade mais rigorosa
Neste Artigo
Resumo
Endonucleases de restrição com nova especificidade de seqüência podem ser desenvolvidas a partir de enzimas que reconhecem uma seqüência parcialmente degenerada. Aqui fornecemos um protocolo detalhado que usamos com sucesso para alterar a especificidade da seqüência da enzima NlaIV. Os ingredientes-chave do protocolo são a compartimentação in vitro da reação de transcrição/tradução e a seleção de variantes com novas especificidades de seqüência.
Resumo
A engenharia de especificidade de endonuclease de restrição (REase) é extremamente difícil. Aqui descrevemos um protocolo multipasso que ajuda a produzir variantes REase que têm especificidade mais rigorosa do que a enzima parental. O protocolo requer a criação de uma biblioteca de de seleção de expressão (ESCs) para variantes do REase, idealmente com variabilidade em posições susceptíveis de afetar a ligação de DNA. O ESC é flanqueado de um lado por uma seqüência para a atividade do local de restrição desejada e uma etiqueta de biotina e do outro lado por um local de restrição para a atividade indesejada e um local de recozimento de primer. Os ESCs são transcritos e traduzidos em uma emulsão de água no óleo, em condições que tornam improvável a presença de mais de uma molécula de DNA por gota. Portanto, o DNA em cada molécula é submetido apenas à atividade da enzima traduzida e codificada. As variantes REase da especificidade desejada removem a etiqueta de biotina, mas não o local de recozimento do primer. Após a quebra da emulsão, as moléculas de DNA são submetidas a uma retração de biotina, e apenas as do sobrenadante são retidas. Esta etapa garante que apenas ESCs para variantes que não perderam a atividade desejada sejam mantidos. Essas moléculas de DNA são então submetidas a uma primeira reação de PCR. O decote na seqüência indesejada corta o local de ligação do primer para um dos primers. Portanto, o PCR amplifica apenas ESCs de gotículas sem a atividade indesejada. Uma segunda reação de PCR é então realizada para reintroduzir o local de restrição para a especificidade desejada e a tag de biotina, para que a etapa de seleção possa ser reiterada. Quadros de leitura abertos selecionados podem ser superexpressos em células bacterianas que também expressam a metiltransferência de cognato do REase parental, porque o REase recém-evoluído tem como alvo apenas um subconjunto dos locais-alvo da metiltransferase.
Introdução
A engenharia de especificidade de seqüência é extremamente desafiadora para rEases classe II. Nesta classe de endonucleases, o reconhecimento de seqüências e a catálise estão intimamente entrelaçados, provavelmente como uma salvaguarda evolutiva contra a criação de uma endonuclease de especificidade mais ampla do que sua metiltransferência de cognato, o que danificaria o DNA do hospedeiro. A evolução dirigida de novas especificidades nas células é ainda mais complicada pela necessidade de proteger o DNA do hospedeiro contra a atividade de endonuclease recém-projetada. Portanto, há apenas algumas tentativas bem sucedidas de engenharia REase relatadas e todas elas exploram as características únicas de uma enzima particular1,,2,,3,4,5,6,7.
Aqui fornecemos um protocolo detalhado para engenharia de especificidade que pode ser usado para gerar variantes de endonuclease que têm especificidade mais estreita do que uma enzima parental que se baseia em nossa engenharia bem sucedida de endonuclease NlaIV8. Para qualquer enzima com uma seqüência de reconhecimento arbitrário, uma especificidade extra pode ser introduzida para bases nos flancos. Para enzimas parentais que reconhecem sequências parcialmente degeneradas (como NlaIV com seu alvo GGNNCC), uma especificidade adicional também pode ser introduzida dentro da seqüência de reconhecimento. Como a especificidade extra provavelmente exigirá contatos de Proteína-DNA, as bases recém-reconhecidas devem estar dentro da pegada da endonuclease parental no DNA. Em princípio, os esquemas de seleção podem ser configurados para qualquer especialização desejada da seqüência de reconhecimento. No entanto, a maioria dos REases que reconhecem sequências de alvo palindômicas e quase palindômicas são dimers funcionais que reconhecem apenas um meio-local do palíndromo. Assim, é improvável que a seleção de novas especificidades que violem a simetria das interações nucleicas proteicas funcione. Para o NlaIV dimérico, por exemplo, a seqüência GGNNCC pode teoricamente ser reduzida ao GGATCC, mas reduzir a especificidade para GGAACC é esperado ser mais difícil. Nosso esquema envolve seleção positiva e negativa.
O processo é mais eficiente quando a seleção negativa também é usada para remover as especificidades capazes de cirizar todas as seqüências que não a especificidade mais estreita preferida. Por exemplo, a seleção para GGATCC pode ser combinada com antisseleção contra GGBVCC (onde B é qualquer base diferente de A, e V é qualquer base diferente de T). Quando algumas das possíveis seqüências de alvo não são cobertas, o resultado do experimento de seleção depende da eficácia da seleção positiva e negativa. Em nosso trabalho nlaIV, selecionamos para GGATCC, e contra GGSSCC (onde S é G ou C), e obtivemos uma especificidade que, ignorando metas de quebra de simetria, poderia ser descrita como GGWWCC (onde W é A ou T), sugerindo que neste caso específico, a seleção negativa era mais importante do que a seleção positiva.
Nossa abordagem começa com a criação de um de seleção de expressão (ESC). O ESC está estruturado em seções. Na seção interna do núcleo, há variantes do quadro de leitura aberta (ORF) do REase, o controle do promotor T7. Esta seção central do ESC não pode conter qualquer local cognato para o REase projetado. O núcleo é sanduíche entre dois locais cognatos para o tipo selvagem REase: um site de decote para a atividade indesejada (seqüência de contador selecionado, GGSCCneste exemplo) e um site de decote para a atividade desejada (seqüência selecionada, GGATCC no exemplo). A etapa final da preparação do ESC em PCR adiciona biotina próxima à atividade desejada no final de 5' e cria uma variedade de seqüências de contra-selecionados (GGSSCC no exemplo). A estratégia de seleção baseia-se no uso de primers cuidadosamente projetados no protocolo de reamplificação ESC após um protocolo de transcrição/tradução/seleção in vitro(Figura 1A). A biblioteca ESC é expressa em uma transcrição compartimentalizada in vitro tradução água-em-óleo emulsão9,10,11. Dentro de cada gotícula, a especificidade da enzima expressa afeta o estado da ESC (Figura 1B, passo I). Para o arranjo descrito, a atividade desejada de decote da proteína traduzida remove a etiqueta de biotina do DNA, mas não afeta a outra extremidade ESC com a seqüência do contador selecionado. Quando a emulsão é quebrada, fragmentos biotinylados são removidos por streptavidin affinity pulldown, de modo que apenas fragmentos de gotículas com a atividade desejada permanecem (Figura 1B, passo II). Esta etapa remove variantes REase inativas. A fração sobrenadante do passo pull-down é então amplificada por PCR. Nas primeiras revezas pcr são utilizados os primers F2 e R1 (Figura 1A,B, passo III). O primer F2 liga-se à seção ESC entre a seqüência do contador selecionada e a extremidade da molécula. Portanto, os ESCs que expressam variantes capazes de afiar a seqüência de contador selecionado (e, portanto, separar os locais de ligação para primers F2 e R1 em duas moléculas de DNA diferentes) não são amplificados e são assim removidos da biblioteca. O primer R1 se liga entre o local selecionado e o núcleo do ESC para que ele não seja afetado pelo status do decote do local selecionado e restaure o local do decote para a atividade desejada (GGATCC). O ciclo é fechado por um segundo PCR (com primers F1 e R2) que adiciona biotina na extremidade de 5' perto do local selecionado e restaura a variação projetada no balcão selecionado local próximo à extremidade oposta do ESC (Figura 1B, passo IV). A mistura de DNA resultante está pronta para outra rodada de seleção.
O sucesso do protocolo de seleção depende fortemente da escolha adequada da nova e mais rigorosa seqüência de reconhecimento de alvo e do design cuidadoso da estratégia de mutagênese e sua implementação eficaz. Como é muito mais fácil melhorar as preferências preexistentes do REase do que superá-las, recomendamos começar com um estudo cinético de quaisquer preferências pré-existentes. A necessidade de um design de mutagênese cuidadoso resulta do tamanho limitado de uma biblioteca mutante que pode ser processada pelo protocolo apresentado (109 clones em um único experimento). Portanto, todas as 20 possíveis substituições de aminoácidos podem ser efetivamente testadas em apenas algumas posições (ver Discussão). Mutagêneses aleatórias, como pcr propenso a erros (EP-PCR) apresentados como um método alternativo, levarão a uma profunda subamostragem da complexidade existente. Se alguma informação sobre potenciais posições de aminoácidos envolvidas em contatos com DNA (ou mesmo localizada em uma proximidade com os nucleotídeos degenerados em uma seqüência de cognato) certamente deve ser usada para selecionar alguns aminoácidos para saturação guiada por oligonucleotídeos mutagênese (passos de protocolo 1.6-3.10).
Protocolo
1. Preparação de ESCs
- Clone metiltransferase do sistema de modificação de restrição a ser projetado em um número de cópia baixa plasmid (por exemplo, pACYC184 ou pACYC174 ou seus derivados).
NOTA: A cepa de hospedeiro bacteriano deve ser capaz de tolerar a metilação introduzida pela enzima clonada e fornecer expressão induzida de polimerase T7 RNA. Recomenda-se o uso da cepa ER2566 (portadora de mutações McRA, McRBC e Mrr). - Confirme que o DNA plasmídeo recombinante é protegido contra o decote pela endonuclease cognato, tratando 0,5 μg de DNA plasmático com 10 unidades de REase cognato em tampão e temperatura recomendada pelo fornecedor da enzima para 2 h.
- Prepare células competentes desta cepa.
NOTA: Qualquer método pode ser usado. O projeto de engenharia NlaIV utilizou um método simples de cloreto de cálcio12. - Construa plasmídeo recombinante com o ORF para o REase controle do promotor T7 de um grupo de exclusão diferente e com um marcador de seleção diferente daquele que contém o gene metiltrasferase na etapa 1.1. Podem ser utilizados vetores pET28 e pET30.
- Remova todos os locais de reconhecimento da enzima projetada da seção do plasmídeo recombinante entre o promotor T7 e o códon de parada da enzima ORF introduzindo mutações silenciosas(Figura 2, Tabela 1A).
NOTA: Se mais de um desses sites for necessário, serão necessárias várias rodadas de mutação (etapas 1.5.1-1.5.7).- Use uma reação PCR de dentro para fora que amplifica o plasmídeo de comprimento completo com variações projetadas introduzidas nas extremidades de 5' dos primers(Tabela 2A).
- Remova o DNA do modelo adicionando 10 U de endonuclease DpnI aos 50 μL da reação PCR e incubar por 2 h a 37 °C.
- Resolver os produtos por eletroforese em gel de agarose. Corte a banda correspondente ao plasmídeo de comprimento completo e purifique-a com um kit comercial.
- Adicione tampão de ligadura de 10x (a uma concentração de 1x) e complemente com ATP (até 1 mM). Adicione 10 U de T4 polinucleotídeo quinase e incubar por 20 min a 37 °C. Inativar a enzima aquecendo a 70 °C por 10 min.
- Adicione PEG 4000 a 5%, suplemente novamente com ATP (a 1 mM), e adicione 5 U de Ligase de DNA T4. Incubar por 2 h à temperatura ambiente (RT).
- Transforme-se em uma cepa bacteriana competente que transporta metiltransferase cognato (passo 1.1).
- Isole o DNA plasmídeo em pequena escala e confirme a introdução de alterações de seqüência por sequenciamento de dideoxy.
- Introduzir locais de restrição exclusivos próximos às seqüências(s) visadas pela mutagênese guiada por oligonucleotídeo (Figura 2, Tabela 1B). Siga as etapas 1.5.1-1.5.7 para cada site.
NOTA: Esta etapa é realizada somente quando uma mutagênese direcionada é usada. Se estiver fazendo mutagênese aleatória, pule as etapas 2-3 e prossiga para a seção 3. No exemplo apresentado, todos os locais foram introduzidos a montante das regiões-alvo, mas também podem ser introduzidos a jusante. - Primers de design para a amplificação do ESC (Tabela 1C).
- Projete uma ligação de primer reversa a jusante do ORF endonuclease que introduzirá o local de reconhecimento selecionado (R1) e sua versão mais curta (R2) que se liga fora da seqüência NlaIV selecionada e contém biotina na extremidade de 5' (ver Figura 1).
- Projete uma nupcial de frente (F1) ligando-se ao esc a montante do promotor T7. Este primer também deve introduzir variantes contra-selecionadas da seqüência de reconhecimento original (ou seja, o máximo de variações de seqüência reconhecidas pela enzima original, com exceção da seqüência reversa selecionada).
NOTA: Uma versão mais curta deste primer (F2) que cobre a seqüência distal à seqüência contra-selecionada será usada mais tarde no PCR seletivo (etapa 5.9).
2. Síntese dividida e mistura de primers mutagênicos
NOTA: Esta etapa é usada apenas para projetos que requerem mutagênese de subsaturação em mais de um local. É necessário um sintetizador com múltiplas colunas de síntese. Atribua colunas para síntese de trigêmeos de codon NNS randomizados e trigêmeos codon selvagens de acordo com as freqüências de mutagênese. Por exemplo, se sete colunas de síntese de volume iguais estiverem disponíveis, e uma taxa de mutagênese de 0,3 for desejável em um determinado local, adicione códons NNS randomizados em ~0,3 x 7 ou duas colunas, e codons do tipo selvagem em ~0,7 x 7 ou cinco colunas(Figura 3).
- Decida sobre os locais para a mutaçãodesubsatura. Escolha as freqüências de mutagênese de acordo com a importância hipotética dos sites (ou seja, quanto mais importante o site, maior a frequência), mantendo os limites da complexidade geral da biblioteca em mente (ver Discussão).
- Sintetizar oligonucleotídeos em todas as colunas, até o trigêmeo imediatamente precedendo o segundo local de mutaçãogênese de subsaturação contando a partir do 3'-end. Não remova o grupo protetor de 5'-trityl no último ciclo de síntese (use a opção trityl-on no sintetizador). O grupo protetor será removido no início do próximo ciclo de síntese (etapa 1 na Figura 3).
- Abra as colunas de síntese. Coletar suporte de síntese de vidro poroso controlado (CPG) em um tubo seco de 1,5 mL e misturar por vórtice. Repartição da resina CPG mista em novas colunas de síntese. Evite introduzir umidade, pois diminuirá o rendimento total (etapas 2 e 4 na Figura 3).
- Continuar a síntese, a partir da subsaturação do sítio mutagênese trigêmeo. Atribua colunas a trigêmeos NNS randomizados ou trigêmeos do tipo selvagem de acordo com a freqüência de mutagênese desejada (veja nota acima). Se houver locais adicionais de subsaturação, prossiga apenas para o trigêmeo que precede o próximo local de mutaçãode subsaturação. Novamente, deixe um grupo de 5'-tritil no final (opção 5'-trityl-on no sintetizador) (passo 3 na Figura 3). Em seguida, continue com o passo 2.3.
- Se não houver mais locais de subsaturação a jusante, complete a síntese, deixando um grupo de 5'-trityl no final (opção 5'-trityl-on no sintetizador) (passo 5 na Figura 3).
- Desproteger e purificar a biblioteca de oligonucleotídeos de acordo com as instruções do fabricante do cartucho de purificação.
NOTA: Os oligonucleotídeos liberados pela desproteção do CPG também podem ser purificados na fase inversa cromatografia líquida de alto desempenho (HPLC) com tritil-on seguido de uma remoção manual do grupo trityl (tratamento 1h com ácido acético 80% no RT) e uma segunda purificação hplc. - Verifique a qualidade da biblioteca oligonucleotídeo em um gel de ureia-PAGE.
3. Gerando bibliotecas variantes
NOTA: Use o plasmídeo recombinante do passo 1.6.
-
Gerar as bibliotecas por oligonucleotídeo dirigido mutagênese.
NOTA: Alternativamente, utilize o protocolo EP-PCR (etapa 3.2).- Amplie uma seção do promotor T7 para o local de enzima de restrição única flanqueando a seqüência direcionada com mutagênese (no caso de NlaIV: SalI, EcoRI ou Eco52I) (Tabela 1B-C, Tabela 2B, Figura 4). Amplie a segunda parte do local da enzima de restrição única até o final de 3'' do ESC.
- Misture separadamente 5 μL das reações de PCR (a partir do passo 3.1.1) com 8 μL de água, 1,5 μL de tampão enzimático de restrição de 10x e 5 unidades das enzimas de restrição apropriadas (SalI, EcoRI ou Eco52I) e incubar na temperatura apropriada por 2 h.
- Resolver os produtos de ambas as reações usando eletroforese em gel de agarose. Corte as faixas de tamanho esperadas e purifique com um kit comercial.
- Corra até 1/3 dos produtos purificados em um gel de agarose e meça a concentração de cada banda purificada por densitometria.
- Configure a ligadura de duas partes dos ESCs em uma razão molar de 1:1 com tampão de ligase 1x e 1 U de Ligase de DNA T4 e incubar por 2 h no RT.
- Resolva os produtos de reação no gel de agarose. Corte os produtos de tamanho esperado e purifique com um kit comercial.
- Amplie os produtos de ligadura purificados em uma reação PCR com os primers F1 e R2 (Tabela 1C e Tabela 2A). Não execute mais de 20 ciclos de amplificação.
- Fracione as reações do PCR em um gel de agarose. Corte os produtos e purifique com um kit comercial.
- Execute uma alíquota de 5 μL da biblioteca purificada da etapa anterior no gel de agarose e meça a concentração por densitometria.
- Clonar uma pequena amostra da biblioteca (até 5 μL) e seqüência >15 clones para verificar a frequência e distribuição de mutação(Tabela 3). Prossiga para o passo 4.
NOTA: Alternativamente, pode-se utilizar o sequenciamento de alto throughput da pequena amostra dos ESCs.
-
Executar EP-PCR.
- Amplie o ESC a partir do plasmídeo obtido na etapa 1.5.7 com primers F1 e R1. Executar 20 ciclos com Polimerase Taq I(Tabela 1B).
- Gel purificar o produto PCR.
- Configure o EP-PCR com 2 ng de produto PCR purificado da etapa anterior e execute 15 ciclos de EP-PCR (Tabela 1C)com primers F1 e R1.
- O gel purifica o produto e quantifica-o por densitometria de gel.
NOTA: Devido à baixa concentração do produto EP-PCR purificado, utilize cerca de 1/3 para quantificação. - Clonar uma pequena amostra da biblioteca (até 1/5) e seqüenciar >15 clones para verificar a frequência e distribuição de mutação(Tabela 4).
NOTA: Alternativamente, realize o sequenciamento de alto throughput da pequena amostra dos ESCs.
4. Realizar reação de tradução de transcrição in vitro compartimentalizada
-
Teste a expressão de endonuclease e a atividade enzimática na tradução de transcrição in vitro.
- Prepare um substrato curto (200-500 bp) com um único local de reconhecimento para a endonuclease localizada perto do centro da molécula para que a reação do decote possa ser facilmente detectada.
NOTA: A maneira mais fácil de preparar o substrato é pela amplificação do PCR de um fragmento apropriado de qualquer molécula de DNA. O substrato pode ser rotulado por rádio ou fluorescentemente para simplificar a detecção do decote. - Configure 50 μL de uma reação de tradução de transcrição com 0,5 μg de ESC tipo selvagem de acordo com as recomendações do fabricante. Adicione sal de magnésio (MgCl2, MgSO4, e acetato de magnésio pode ser testado) a 1,5 mM e a quantidade apropriada de substrato da etapa anterior (pelo menos 0,5 μg em caso de DNA não rotulado).
NOTA: Qualquer kit de transcrição/tradução que não contenha nuclease ativado por magnésio pode ser usado. Alguns fornecedores de kits usam nucleases para remover a contaminação do DNA durante a produção e, em seguida, adicionar queladores como inibidores de nuclease. Tais kits não são compatíveis com este método. - Incubar a reação de tradução de transcrição de acordo com as instruções do fabricante. Em seguida, transfira a mistura de reação para a temperatura ideal para a enzima de restrição por 2 h.
- Analisar o decote do substrato em gel de agarose seguido de detecção adequada (por exemplo, coloração de DNA, visualização de fluorescência ou autoradiografia) (Figura 5).
NOTA: Pelo menos o decote parcial do substrato é necessário antes de prosseguir com a compartimentação. Se isso não for alcançado, é necessária uma otimização adicional da forma química de magnésio ou sua concentração.
- Prepare um substrato curto (200-500 bp) com um único local de reconhecimento para a endonuclease localizada perto do centro da molécula para que a reação do decote possa ser facilmente detectada.
- Prepare uma mistura de óleo-surfactante adicionando 225 μL de Extensão 80 e 25 μL de Tween 80 a 5 mL de óleo mineral em um tubo cônico de 15 mL. Misture bem invertendo suavemente o tubo 15x.
- Para cada biblioteca, transfira 950 μL da mistura óleo-surfactante para um frasco criogênico de fundo redondo de 2 mL, etiqueta com um nome de biblioteca e transferência para gelo. Coloque uma pequena barra de agitação cilíndrica (5 x 2 mm2) em cada frasco.
- Prepare uma mistura de reação de tradução de transcrição in vitro (50 μL para cada biblioteca) de acordo com as sugestões do fabricante. Suplementar a mistura com cloreto de magnésio para uma concentração final de 1,5 mM (ver passo 4.1.4).
- Distribua as alíquotas de 50 μL em tubos de 1,5 mL no gelo.
- Adicione 1,7 fmole da biblioteca (da seção 3) à mistura de reação no gelo.
NOTA: Não use uma biblioteca de expressão maior para obter eficiência de seleção. É crucial minimizar a frequência de gotículas aquosas contendo mais de uma molécula de DNA. -
Prepare a emulsão água-em-óleo consecutivamente para cada biblioteca.
- Coloque um béquer pequeno (ou copo de garrafa grande) cheio de gelo em um agitador magnético com a velocidade de agitação definida a 1.150 rpm.
- Transfira um frasco criogênico com 950 μL de mistura de óleo-surfactante e uma pequena barra de agitação do passo 4.3 para um copo gelado no agitador magnético. Verifique se a barra de agitação está girando.
- Adicione cinco alíquotas de 10 μL da mistura in vitro biblioteca-transcrição-tradução durante um período de 2 minutos em intervalos de 30 s e continue mexendo por um minuto adicional. Transfira o frasco com a emulsão para um recipiente de gelo. Prossiga com a próxima biblioteca começando com o passo 4.7.2.
- Depois que todas as bibliotecas são processadas iniciam a incubação de todas as bibliotecas de acordo com as recomendações do fabricante do kit.
- Transfira os frascos para a temperatura ideal para a endonuclease projetada por mais 2 h e, em seguida, coloque-os no gelo por pelo menos 10 min.
5. Processamento contínuo de bibliotecas e seleção
- Transfira as emulsões dos frascos criogênicos para tubos frios de 1,5 mL, adicione 1 μL de EDTA de 0,5 M e centrífuga-as a 13.000 x g por 5 min à temperatura ambiente.
- Remova a parte superior do óleo com uma pipeta. Se uma interfase óleo-água não for visível, incubar o tubo por pelo menos 5 min a -20 °C para congelar a fase aquosa e, em seguida, tubo imediatamente para fora da fase do óleo líquido.
- Adicionar 100 μL de 10 mM Tris HCl pH 8.0 e realizar imediatamente a extração com 150 μL de fenol:clorofórmio (1v/v) por vticização curta seguida de separação de fase por centrifugação de 30 s a 13.000 x g. Colete a fase superior aquosa.
- Precipitar o DNA adicionando 0,1 vol (15 μL) de 3 M de acetato de sódio (pH = 5,2), 2,5-5 μg de glicogênio e 2,5 vol (375 μL) de etanol. Incubar a -20 °C por 1h e centrífuga por 15 min a 13.000 x g, 4 °C. Descarte o sobrenadante e lave brevemente a pelota com 1 mL de etanol frio de 70%.
- Seque a pelota de DNA/glicogênio em velocidade ou ar seco por >5 min.
- Dissolver a pelota em 50 μL de 10 mM Tris-HCl (pH = 7,5). Adicione 5 μL de contas magnéticas de streptavidin as preparadas de acordo com as instruções do fabricante e misture por 1 h no RT, de preferência em um misturador de carrossel ou por vórtices suaves.
- Separe as contas em um suporte magnético e colete o líquido enriquecido em DNA sem biotina.
- Concentre o DNA por precipitação de etanol (passos 5.4-5.5).
- Dissolver o DNA concentrado da etapa anterior em 5 μL de água e usar como modelo em uma reação pcr com primers F2 e R1(Tabela 1A).
NOTA: Para evitar problemas com a contaminação do modelo e minimizar os artefatos PCR use taq polymerase (não Pfu ou Phusion) e execute ciclos de 18 a 20 com o tempo de extensão proporcional ao tamanho do modelo (1 kb = 1 min) (ver Tabela 2B). - Fracione o produto PCR em um gel de agarose e corte o produto de tamanho esperado. Algumas manchas indicam que existem produtos de diferentes tamanhos (ver Figura 6). Purifique o DNA da laje de gel com um kit comercial.
- Execute uma segunda reação pcr com até 50 ng de DNA da etapa 5.10 e primers F1 e R2 usando o mesmo protocolo da etapa 5.9. Proceder à purificação do produto conforme descrito em 5.10. DNA purificado após quantificação por densitometria de gel de agarose (não espectroscopia UV) pode ser usado na próxima rodada de seleção in vitro (passo 4.6).
6. Variantes de tela para especificidade de seqüência alterada
- Clone variantes selecionadas.
- Digerir o produto da etapa 5.10 para 2 h com 10 U de enzimas de restrição apropriadas para clonagem do ORF no vetor de expressão (para NlaIV: NcoI e XhoI) na temperatura e tampão recomendados pelo fornecedor de enzimas. Resolva os produtos com eletroforese em gel de agarose e isole o fragmento de tamanho esperado.
- Prepare o vetor plasmático (por exemplo, pET28) por decote duplo com as mesmas enzimas da etapa 6.1.1 e purifique o produto com um kit comercial de purificação de gel de DNA.
- Estimar as concentrações de vetor e inserção por densitometria com eletroforese de gel de agarose.
- Configure uma ligadura com 1-5 U de Ligase de DNA T4 e vetor: inserir a razão molar 1:3-1:5 em tampão de ligase 1x recomendado pelo fornecedor de enzimas. Incubar por 2 h no RT e introduzir em bactérias hospedeiras apropriadas (a partir do passo 1.3) por transformação ou eletroporação12.
- Selecione os extransformantes em placas LB contendo o antibiótico apropriado (50 μg/mL de kanamicina para vetores pET28 ou pET30) e 1% de glicose.
- Variantes de proteína expressa.
- Inocular colônias individuais da transformação (até 24 clones podem ser facilmente processados em uma única corrida) em 2 mL de LB com kanamicina (50 μg/mL) e 1% de glicose e crescer durante a noite a 37 °C com agitação.
- Inocula15 mL de lb quente (37 °C) contendo 100 μg de kanamicina e sem glicose com 0,75 mL da cultura noturna e incuba a 37 °C com agitação vigorosa.
NOTA: Podem ser usados tubos de centrífuga de 50 mL ou frascos erlenmayer de 100 mL. - Adicionar 176 μL de glicerol a 1 mL de cultura noturna (concentração final de glicerol = 15%) misture bem e congele a -70 °C.
- Após 2-3 h suplemento 15 mL da cultura (da etapa 6.2.1) com IPTG a 1 mM e cultura por mais 5 h.
- Coletar a pelota bacteriana por centrifugação (10.000 x g, 4 °C, 10 min) e congelar a -70 °C.
- Purifique as variantes proteicas.
- Transfira 20 μL de suspensão de resina de afinidade de níquel para 200 μL de tampão B1 em um tubo de 1,5 mL com uma ponta de pipeta de furo largo, misture suavemente e centrífuga (5.000 x g,30 s, 4 °C). Remova o sobrenadante por pipetting e deixe o tubo no gelo.
- Resuspender a pelota bacteriana do passo 6.2.5 em 300 μL de B1 por vórtice vigoroso. Transfira a suspensão para um tubo de 1,5 mL.
- Adicione 3 μL de coquetel inibidor de protease de 100x e solução sossômica em B1 (concentração final de 1 mg/mL). Desintegre as células por sonofrização com uma sonda de ponta equipada. Use seis rajadas de 10 s por amostra com o tempo de resfriamento da ponta de >15 s no gelo no meio. Mantenha as suspensões das células no gelo o tempo todo.
- Detritos de células de pelotas por centrifugação (2 min, 12.000 x g, 4 °C) e transferência de 250 μL de sobrenadante para a alíquota de resina do passo 6.3.1.
- Misture por 15 min em uma sala fria, de preferência em um misturador de carrossel ou por vórtice suave.
- Centrífuga (5.000 x g, 30 s, 4 °C) e aspirar o sobrenadante com uma pipeta.
- Adicione 500 μL de tampão W e resuspenda suavemente a resina. Centrífuga (5.000 x g, 30 s, 4 °C) e aspirar o sobrenadante com uma pipeta.
- Repita o passo 6.3.7.
- Adicione 20 μL de tampão E, resuspenda suavemente a resina e deixe a amostra no gelo por 2-5 min. Centrifuge (5.000 x g, 30 s, 4 °C) e colete o sobrenadante.
- Repita o passo 6.3.9. Supernatantes da piscina.
- Analisar amostras de proteínas por SDS-PAGE (5-10 μL) (Figura 7).
- Tela para variantes com a especificidade alterada.
- Atividade de decote de ensaio no DNA bacteriophage lambda. A amostra de proteínas pode constituir até 10% do volume de reação final. Um total de 2 μL de amostra de proteína por 0,5 μg de DNA e 2 h de tempo de reação é um bom ponto de partida.
- Analisar os produtos de reação por eletroforese em gel de agarose junto com os produtos gerados pela enzima do tipo selvagem. Selecione os clones que geram padrões de decote claramente distinguíveis daquele gerado pela enzima do tipo selvagem para análise posterior(Figura 8).
Resultados
Este protocolo é apenas uma ferramenta para aumentar a freqüência de variantes desejadas de um REase projetado, esgotando (mas não eliminando) duas classes indesejadas: enzimas inativas e endonucleases com especificidade de seqüência de tipo selvagem inalterada. Por outro lado, como mudar a especificidade do REase é extremamente difícil, encontrar até mesmo uma dessas variantes produzindo um padrão de decote diferente da enzima do tipo selvagem em uma única triagem de 24 clones deve ser considerado um sucesso. Em nossas mãos, as melhores telas poderiam identificar até 20% das variantes promissoras(Figura 8A).
O resultado positivo depende fortemente de uma qualidade de biblioteca (ou seja, frequência limitada de substituições e sua distribuição aleatória) e captura eficiente da população biotinylated de membros da biblioteca (passos 3.6-3.7). Ambos os problemas podem ser detectados. A qualidade da biblioteca deve ser verificada antes da seleção sequenciando o maior número possível de clones (>15) ou por sequenciamento direto da biblioteca por sequenciamento de alto throughput (etapa 3.10, Tabela 3). Se a maioria dos clones selecionados não estiver ativa, esta é uma clara indicação de falha na seleção de captura de streptavidin. Um efeito semelhante é observado no caso de bibliotecas que passam por muitos ciclos de seleção, pois essas bibliotecas são provavelmente dominadas por variantes inativas que escaparam da etapa de seleção de captura de streptavidin(Figura 8B). Portanto, é aconselhável executar a triagem após cada ciclo de seleção e desenvolver variantes promissoras selecionadas manualmente em vez de depender da iteração de seleção.
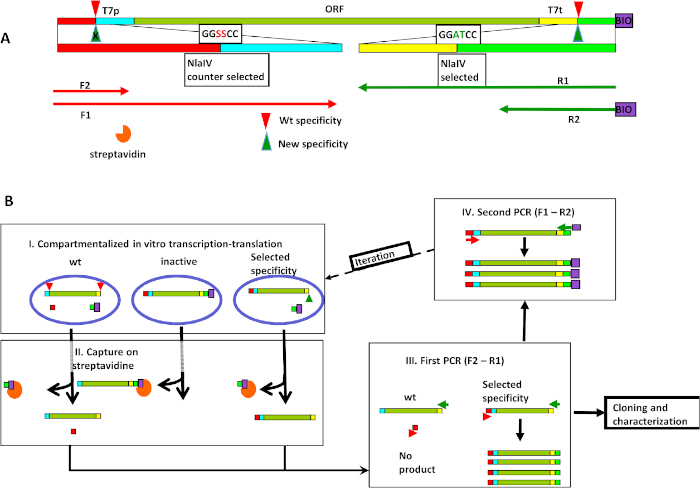
Figura 1: Seleção in vitro de uma nova especificidade de seqüência baseada na engenharia NlaIV. (A) A organização do de expressão/seleção (ESC) inclui dois locais de reconhecimento para REase, 1) a seqüência selecionada (GGATCC) perto da extremidade direita e 2) a seqüência de contador selecionada (GGSSCC) perto da extremidade esquerda, bem como o promotor T7p e T7t-T7 e o exterminador T7t-T7. Os locais de ligação do primer são mostrados abaixo. O decote por tipo selvagem e as variantes NlaIV selecionadas são mostrados como triângulos vermelhoe verde, respectivamente. (B) Etapas do ciclo de seleção: I) Emulsificação das misturas de reação transcrição-tradução-decote com a biblioteca ESC; II) Todo DNA biotinylado é capturado em partículas magnéticas revestidas com streptavidin e removidas, removendo assim variantes inativas codificadoras; III) As ESCs que codificam REases com atividade do tipo selvagem (ou seja, aquelas capazes de cira da seqüência GGSSCC) são eliminadas porque o decote da seqüência separa os locais de ligação para os primers para frente e para trás. Portanto, não ocorre a ampliação dessas ESCs; IV) A entrada para a próxima rodada de seleção é criada pela adição de biotina na extremidade direita e reintrodução da variação das seqüências do contador selecionado na extremidade esquerda. Reimpresso de Czapinska et al.8 com permissão da Elsevier. Clique aqui para ver uma versão maior desta figura.
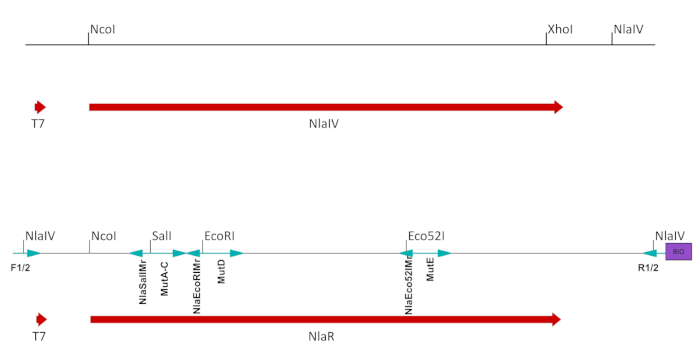
Figura 2: Preparação de ESC. Fragmento derivado do construto original em um vetor de expressão contendo NlaIV ORF sob controle do promotor T7 foi modificado para ser adequado para expressão/seleção. O site NlaIV a jusante do ORF NlaIV foi removido e locais únicos (SalI, EcoRI e Eco52I) que foram usados para mutagenizar posições selecionadas foram introduzidos no ORF NlaIV como mutações silenciosas. A construção final foi amplificada com primers de flanqueamento que introduziram dois locais de NlaIV flanqueando: a seqüência de contador selecionado (GGSSCC) à esquerda e seqüência selecionada (GGATCC) à direita. O primer reverso também introduziu a biotina. Os primers usados na criação de ECS mutados são mostrados como setas azuis e rotulados abaixo (ver Tabela 1B,C). Clique aqui para ver uma versão maior desta figura.
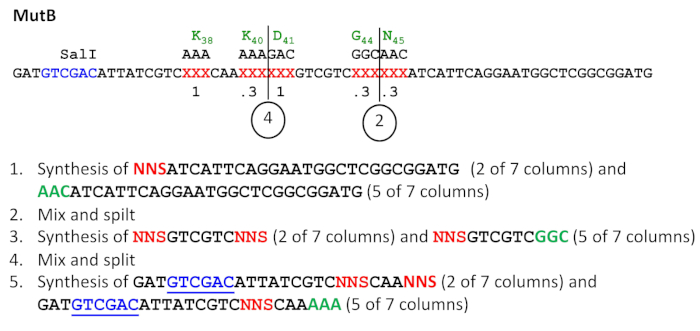
Figura 3: Esquema de síntese de divisão e mistura. O exemplo refere-se à síntese de primer MutB onde uma seqüência NNS foi introduzida em freqüência de 0,8 em quatro posições (ver também Tabela 3). Note que a síntese química é realizada de 3' a 5', mas todas as seqüências são mostradas na orientação canônica de 5'-3' (ou seja, prossegue da direita para a esquerda neste esquema). Sequências de tipo selvagem em posições mutagenizadas são mostradas em verde, enquanto seqüências mutagênicas NNS estão em vermelho. O site de reconhecimento SalI que mais tarde é usado para introduzir mutações em ESCs é sublinhado. São indicados pontos de mistura e divisão (2 e 4). Clique aqui para ver uma versão maior desta figura.
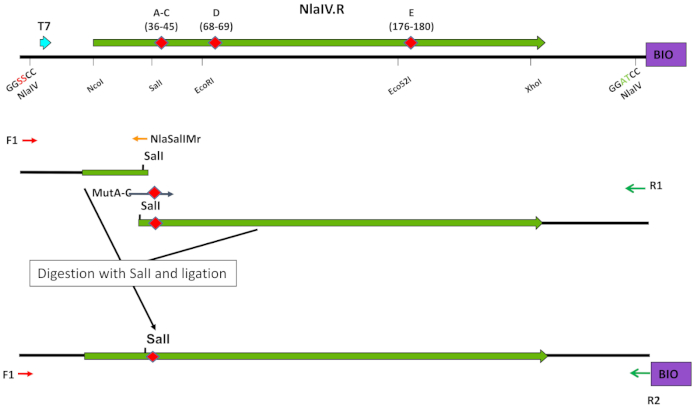
Figura 4: Uso de locais de enzimas de restrição únicas em mutagêneses direcionadas ao oligonucleotídeo. A estratégia de introdução de mutação é mostrada em um exemplo da construção de bibliotecas A-C (ver passos 3.1-3.7). Reimpresso de Czapinska et al.8 com permissão da Elsevier. Clique aqui para ver uma versão maior desta figura.
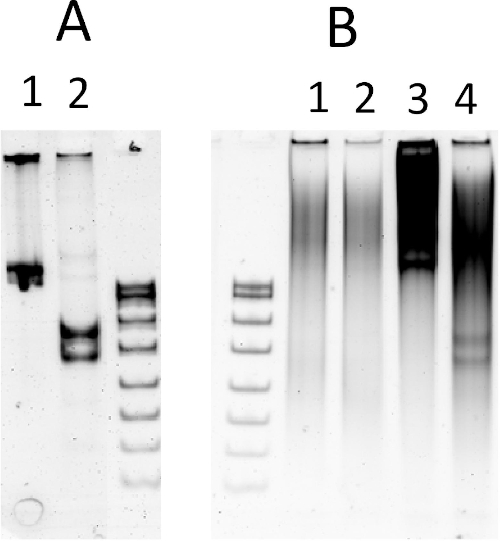
Figura 5: Decote endonucleolítico em tradução de transcrição in vitro. (A) Decote de um substrato de teste no buffer REase ideal: 1) Substrato, produto PCR de 612 bp com um único local de reconhecimento NlaIV; 2) Produtos de decote, 355 bp e 257 bp.(B) Decote em uma reação in vitro de tradução de transcrição (contendo 0,5 μg de ESC): 1-2) alíquotas de 15 μL de tradução in vitro de transcrição sem substrato (linha 2: reação complementada com 1,5 mM MgCl2); 3-4) alíquotas de 15 μL de tradução de transcrição in vitro com 1 μg de substrato de ensaio; (linha 4: reação complementada com 1,5 mM MgCl2). Marcador de tamanho de S-DNA (pBR322 digerido com MspI). As amostras foram resolvidas em 6% de PAGE nativo. DNA foi manchado com brometo de etídio. Clique aqui para ver uma versão maior desta figura.
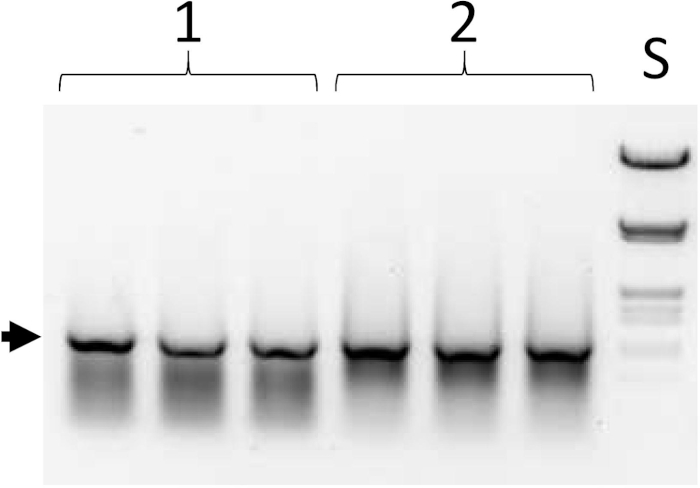
Figura 6: Produtos do primeiro PCR no ciclo de seleção. Ver Figura 1B, etapa III; passo de protocolo 5.10. Os conjuntos de colunas 1 e 2 são alíquotas de duas bibliotecas diferentes carregadas em triplicado. Padrão de tamanho de S-DNA (DNA lambda digerido com HindIII e EcoRI). A seta indica a posição do ESC de comprimento completo (1.050 bp). Clique aqui para ver uma versão maior desta figura.
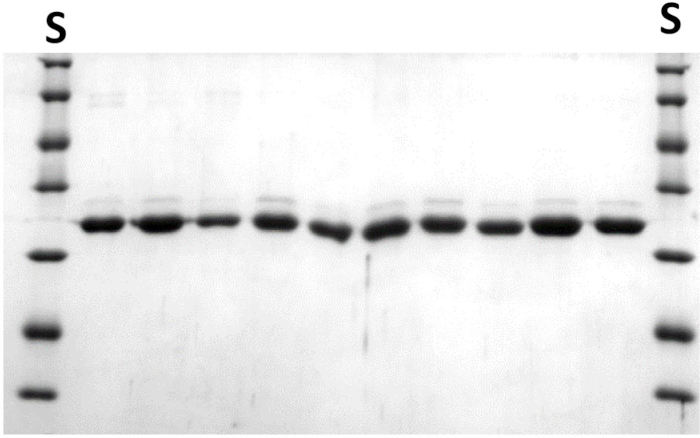
Figura 7: Variantes NlaIV purificadas para posterior triagem em mini escala. Veja o passo 6.3.11. Cada linha contém uma alíquota de 10 μL de uma variante diferente. Padrão de peso molecular de proteína S. A massa molecular da subunidade NaIV REase é de 29,9 kDa. Clique aqui para ver uma versão maior desta figura.
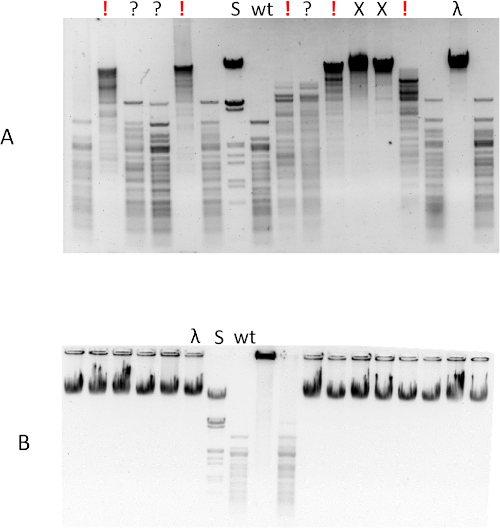
Figura 8: Exemplos de triagem de variantes NlaIV para alteração de especificidade da seqüência. Veja o passo 6.4.2. (A)Triagem bem sucedida com alta frequência de variantes promissoras. S = Marcador de tamanho de DNA, DNA lambda com HindIII e EcoRI; tipo selvagem (wt) = DNA lambda com nlaIV tipo selvagem; λ = substrato de DNA lambda, não cortado; outras colunas=variantes com atividade muito baixa. Variantes são rotuladas ! = variantes promissoras que produzem um padrão de decote distinto da enzima do tipo selvagem; ? = variantes que também podem ter alterado a preferência de seqüência. (B) Triagem mal sucedida, com uma maioria de variantes inativas e uma variante com padrão de decote aparentemente inalterado. Clique aqui para ver uma versão maior desta figura.
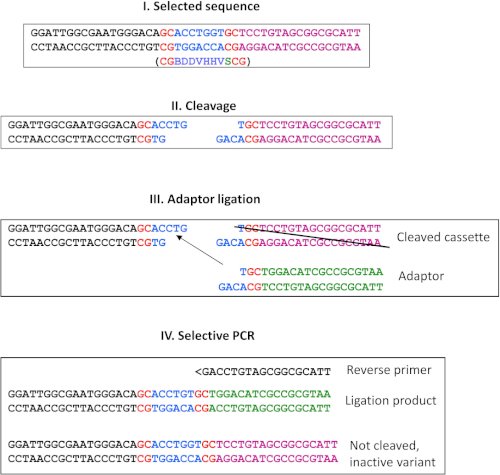
Figura 9: Seleção alternativa por ligadura. Esta alternativa pode ser usada para todos os REases gerando extremidades pegajosas. Aqui apresentamos um protocolo de exemplo para um esquema de seleção de enzima SéI (inédito). I) Seqüência selecionada (localizada na extremidade direita do ESC) com resíduos definidos mostrados em vermelho e variação selecionada da seqüência cognato mostrada em azul. Entre parênteses abaixo, é mostrada a seqüência de contador selecionada a ser colocada na extremidade esquerda do ESC; II) Produto do decote MwoI; III) Após o término da transcrição/tradução in vitro, os produtos são purificados e a ligadura é realizada com adaptador em excesso. Somente os produtos de decote que foram cortados na seqüência selecionada podem participar da ligadura. Portanto, as variantes inativas são eliminadas, e a etapa de pulldown é desnecessária. O produto de decote na seqüência selecionada do contador (na extremidade esquerda do ESC, não mostrado) não pode participar desta ligadura porque a extremidade saliente do adaptador não é complementar à seqüência do contador selecionado; IV) O PCR seletivo usa a mesma estratégia do protocolo principal para eliminar variantes com a especificidade da seqüência de degeneração do tipo selvagem (f1 primer binding distal ao site selecionado do contador), enquanto as variantes inativas são eliminadas pelo primer reverso seletivo que não pode se ligar ao final direito impuro (e, portanto, não modificado pela ligadura do adaptador). No próximo ciclo, o processo pode ser iterado usando um adaptador idêntico ao produto de decote da etapa anterior (ou seja, o de celado no painel III), e um primer reverso seletivo apropriado. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Primers usados na engenharia NlaIV. As seqüências dos locais de restrição mencionados nos comentários são sublinhadas. Letras pequenas indicam sequências que não possuem complementos nos modelos de DNA. Clique aqui para ver esta tabela (Clique com o botão direito do mouse para baixar).
Tabela 2: Condições das reações de PCR a serem utilizadas no protocolo. Tm = temperatura de fusão do primer (se Tm for diferente para os primers, o Tm inferior deve ser usado). Clique aqui para ver esta tabela (Clique com o botão direito do mouse para baixar).
Tabela 3: Resultados da verificação de qualidade de dois primers mutagênicos sintetizados com estratégia split-and-mix. Códons mutagenizados são indicados com [XXX]. Um número de índice mais baixo indica a posição de um aminoácido codificado. Adaptado de Czapinska et al.8 com permissão da Elsevier. Clique aqui para ver esta tabela (Clique com o botão direito do mouse para baixar).
Tabela 4: Resultados do EP-PCR. Principais parâmetros derivados da análise seqüencial de 22 clones de ECS. Clique aqui para ver esta tabela (Clique com o botão direito do mouse para baixar).
Discussão
O protocolo de seleção descrito aqui foi testado para NlaIV8, uma seqüência de reconhecimento de dobra pd-(D/E)XK dimérica que reconhece um local de destino palindômico com bases nn centrais e catalisa um corte de extremidade sem corte entre as bases NN. NlaIV foi escolhido porque o decote entre as bases NN sugere que essas bases estão próximas da proteína no complexo. Em princípio, o protocolo pode ser usado para qualquer seqüência específica de restrição endonuclease, monomérica ou dimeric, de qualquer grupo dobrável, catalisando quebras duplas de fios de qualquer cambalhota, independentemente de os domínios catalíticos e de especificidade coincidirem (como no exemplo NlaIV) ou são separados (por exemplo, FokI). Além disso, o protocolo em princípio é útil não apenas para a geração de novas especificidades enzimáticas mais estreitas, mas também poderia ser usado para eliminar atividades estelares, ou para criar endonucleases de alta fidelidade. No entanto, tudo isso ainda não foi testado. Em particular, a eliminação direcionada da atividade estelar pode ser complicada, porque os mesmos resíduos de aminoácidos podem estar envolvidos na ligação às bases desejadas e indesejadas. As etapas in vitro descritas neste protocolo não se limitam à seleção de especificidades reduzidas, mas também podem ser usadas para selecionar especificidades alteradas de outra forma. No entanto, há então um problema com endonucleases variantes: se o espectro de substratos inclui novos alvos não cortados pela endonuclease parental, não há, em geral, uma boa maneira de proteger as células dos efeitos nocivos dessa atividade. Em contraste, se a especificidade da endonuclease for reduzida apenas, os alvos são um subconjunto dos alvos do tipo selvagem e, portanto, o cognato metiltransferase já disponível deve ser totalmente protetor.
Nosso protocolo difere em vários aspectos de muitos protocolos de evolução direcionados. A diversidade de quadros de leitura aberta é gerada uma vez no início do experimento, não em todas as iterações. Além disso, é criado por síntese split-and-mix, em vez de por EP-PCR. Para substituições nns de códons, como usado neste trabalho, há (4 x 4 x 2)6 ~ 1,07 x 109 combinações para seis posições. Portanto, qualquer variante está presente em média uma vez em 1,7 fmoles de ESC. Essa capacidade pode ser aumentada para sete posições usando a síntese com uma mistura de 20 precursores trinucleotídeos que é oferecida pela Glen Research ou diminuindo a frequência de mutação em posições menos promissoras com síntese de oligonucleotídeos split-and-mix. Se possível, recomenda-se limitar a extensão da variação a seis posições. Obviamente, tal alvo de mutagênese requer algum conhecimento preexistente sobre pelo menos as regiões da REase envolvidas na ligação do substrato. O protocolo split-and-mix para gerar diversidade tem claras vantagens em comparação com o EP-PCR. Utilizando EP-PCR, obtivemos variantes e seqüências inalteradas que carregam oito substituições para ESCs NlaIV no mesmo EP-PCR (Tabela 4). A biblioteca do EP-PCR contém uma fração substancial de clones que devem ser evitados (seqüências de tipo selvagem, múltiplas substituições, frameshift e mutações sem sentido, e mutações em lugares improváveis de afetar a especificidade da seqüência).
Nosso protocolo também difere de muitos outros protocolos de evolução direcionados pela presença de duas etapas de seleção sequencial. A seleção positiva garante que a atividade desejada seja retida, caso contrário, a etiqueta de biotina não será removida e a seqüência de codificação pode ser removida por pull-down. É tecnicamente possível que o surgimento fortuito de uma especificidade nova e não sobreposta (por exemplo, GCATGC) possa levar ao severção da etiqueta de biotina também, se um local de decote adequado estiver presente perto do decote desejado, mas não em outro lugar. No entanto, isso deve ser altamente improvável. A seleção negativa remove quadros de leitura abertos que codificam enzimas que ainda têm a atividade indesejada. Esta etapa não é estritamente obrigatória, pois o protocolo ainda enriquecerá a biblioteca de saída com variantes que são capazes de cortar a seqüência de seleção, mas não são capazes de cortar em outros lugares do ESC, tornando-a inadequada para amplificação pcr. No entanto, espera-se que a eficácia da seleção seja menor porque as enzimas com a especificidade da seqüência original não serão removidas da saída e superarão as variantes promissoras com especificidade alterada, mas também diminuírão a atividade enzimática. Note que, no nível populacional, tanto as seqüências de alvo desejadas quanto indesejadas podem, mas não precisam ser, degeneradas. No exemplo nlaiv, o anti-alvo era degenerado e o alvo não degenerado. Mesmo quando há degeneração no nível populacional, em uma única gotícula apenas um alvo (não degenerado) ou anti-alvo está presente. Em nosso protocolo, seqüências de alvo e anti-alvo são reintroduzidas a cada repetição das etapas de seleção. Portanto, um quadro de leitura aberto deve codificar uma enzima capaz de cachar todos os alvos possíveis, e incapaz de cachar qualquer um dos anti-alvos, para sobreviver a múltiplas rodadas de seleção. Observe que a necessidade de reintroduzir a meta de antisseleção em cada iteração do protocolo impõe dois PCRs seqüenciais. O primeiro PCR usa um primer que anneals fora do anti-alvo, de modo que o decote do anti-alvo previne a reação pcr. O segundo PCR requer um primer que ultrapasse o anti-alvo, e reintroduz o anti-alvo, para garantir que durante várias rodadas de seleção, cada quadro de leitura aberto seja testado contra todas as variantes do anti-alvo.
Para enzimas que geram extremidades pegajosas, um protocolo alternativo relacionado baseado em um método previamente descrito para o isolamento do REase ORF10 pode ser usado. O esgotamento das variantes inativas pela captura de biotina que é usada em nossos experimentos é substituído no protocolo alternativo pela ligadura do adaptador compatível com uma seqüência que é usada como um local de ligação de primer em um PCR seletivo(Figura 9). Apenas AS ESCs que produzem enzimas com a especificidade selecionada geram extremidades capazes de ligadura e, portanto, serão selecionadas. A seqüência da extremidade pegajosa da seqüência eselecionada do contador deve ser projetada de tal forma que não possa participar da ligadura com adaptadores. A iteração do processo de seleção pode ser facilmente alcançada alternando entre dois adaptadores diferentes e, consequentemente, dois primers reversos diferentes em PCR seletivo.
Mesmo com novos protocolos, a tarefa de criar novas especificidades in vitro ainda é muito desafiadora. Para as reases do tipo II típicas, a especificidade da seqüência e a atividade endonucleolítica dependem das mesmas regiões proteicas. É, portanto, difícil alterar um sem afetar o outro. O sucesso é mais provável por uma estratégia que leva em conta a pegada da enzima, respeita a simetria das interações proteína-DNA e se baseia em preferências enzimáticas pré-existentes, que devem ser determinadas antecipadamente em experimentos bioquímicos, como foi feito para o exemplo NlaIV8.
Divulgações
Os autores não têm nada para revelar.
Agradecimentos
Este trabalho foi apoiado pelas bolsas do Ministério da Ciência e Ensino Superior (0295/B/PO1/2008/34 ao MB e N301 100 31/3043 à KS), do Centro Nacional de Ciência da Polônia (NCN) (UMO-2011/02/A/NZ1/00052, UMO-2014/13/B/NZ1/03991 e UMO-2014/14/M/NZ5/00558 à MB) e por bolsa EMBO de curto prazo à KS (ATSF 277.00-05).
Materiais
| Name | Company | Catalog Number | Comments |
| 1000Å CPG Support (dA, dT, dC, dG) | Biosset | 45-1000-050 | Other vendors can be used as well |
| ASM-800 DNA/RNA | Biosset | 800-001-000 | |
| GeneJET Gel Extraction Kit | Thermo Scientific | K0691 | Any other kit can be used |
| Glen-Pak DNA purification cartridge | Glen Research | 60-5200 | |
| HIS-Select Nickel Affinity Gel | Sigma | P6611 | |
| pET 28a vector | Any other vector with T7 promoter upstream of plycloning site can be used instead | ||
| Phusion High-Fidelity DNA Polymerase | Thermo Scientific | F530S | Any other high fidelity and highly processive thermophilic polymearse can be used instead |
| Porous steel foil | Biosset | 40-063 | |
| Rapid Translation System RTS 100, E.coli HY Kit | Roche | 3 186 148 | |
| Restriction endonucleases | Thermo Scientific | Obviously other vendors, enzymes can be used | |
| Streptavidin Magnetic Beads | New England Biolabs | S1420S | Other vendors can be used as well. We have positively tested beds form Sigma |
| Synthesis chemicals including phosphoramidities | Carl Roth | Other vendors can be used as well | |
| Synthesis columns (different sizes) | Biosset | ||
| T4 DNA ligase | Thermo Scientific | EL0011 | Any other ligase can be used |
Referências
- Schöttler, S., Wenz, C., Lanio, T., Jeltsch, A., Pingoud, A. Protein engineering of the restriction endonuclease EcoRV--structure-guided design of enzyme variants that recognize the base pairs flanking the recognition site. European Journal of Biochemistry. 258 (1), 184-191 (1998).
- Wenz, C., Hahn, M., Pingoud, A. Engineering of variants of the restriction endonuclease EcoRV that depend in their cleavage activity on the flexibility of sequences flanking the recognition site. Biochemistry. 37 (8), 2234-2242 (1998).
- Samuelson, J. C., Xu, S. Y. Directed evolution of restriction endonuclease BstYI to achieve increased substrate specificity. Journal of Molecular Biology. 319 (3), 673-683 (2002).
- Samuelson, J. C., et al. Engineering a rare-cutting restriction enzyme: genetic screening and selection of NotI variants. Nucleic Acids Research. 34 (3), 796-805 (2006).
- Rimseliene, R., Maneliene, Z., Lubys, A., Janulaitis, A. Engineering of restriction endonucleases: using methylation activity of the bifunctional endonuclease Eco57I to select the mutant with a novel sequence specificity. Journal of Molecular Biology. 327 (2), 383-391 (2003).
- Morgan, R. D., Luyten, Y. A. Rational engineering of type II restriction endonuclease DNA binding and cleavage specificity. Nucleic Acids Research. 37 (15), 5222-5233 (2009).
- Skowronek, K., Boniecki, M. J., Kluge, B., Bujnicki, J. M. Rational engineering of sequence specificity in R.MwoI restriction endonuclease. Nucleic Acids Research. 40 (17), 8579-8592 (2012).
- Czapinska, H., et al. Crystal Structure and Directed Evolution of Specificity of NlaIV Restriction Endonuclease. Journal of Molecular Biology. 431 (11), 2082-2094 (2019).
- Miller, O. J., et al. Directed evolution by in vitro compartmentalization. Nature Methods. 3 (7), 561-570 (2006).
- Zheng, Y., Roberts, R. J. Selection of restriction endonucleases using artificial cells. Nucleic Acids Research. 35 (11), e83 (2007).
- Takeuchi, R., Choi, M., Stoddard, B. L. Redesign of extensive protein-DNA interfaces of meganucleases using iterative cycles of in vitro compartmentalization. Proceedings of the National Academy of Science U.S.A. 111 (11), 4061-4066 (2014).
- Howland, J. L., Ausubel, F., Brent, R., Kingston, R. E., Moore, D. D., Seidman, J. G., Smith, J. A., Struhl, K. . Short Protocols in Molecular Biology. , (1995).
- Wilson, D. S., Keefe, A. D. Chapter 8 Unit 8.3: Random mutagenesis by PCR. Current Protocols in Molecular Biology. , (2001).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados