Method Article
Geração de mutantes de deleção de genes no quadro em Pseudomonas aeruginosa e testes para atenuação de virulência em um modelo simples de infecção por camundongos
Neste Artigo
Resumo
Aqui, descrevemos um protocolo simples e reproduzível do modelo de infecção do rato para avaliar a atenuação das cepas geneticamente modificadas de Pseudomonas aeruginosa em comparação com os Estados Unidos Food and Drug Administration (FDA) aprovado Escherichia coli para aplicações comerciais.
Resumo
Os microrganismos são geneticamente versáteis e diversificados e tornaram-se uma importante fonte de muitos produtos comerciais e biofármacos. Embora alguns desses produtos sejam produzidos naturalmente pelos organismos, outros produtos exigem engenharia genética do organismo para aumentar os rendimentos da produção. Cepas virulentas de Escherichia coli têm sido tradicionalmente as espécies bacterianas preferidas para a produção de biofármacos; no entanto, alguns produtos são difíceis para E. coli para produzir. Assim, cepas virulentas de outras espécies bacterianas poderiam fornecer alternativas úteis para a produção de alguns produtos comerciais. Pseudomonas aeruginosa é uma bactéria gram-negativa comum e bem estudada que poderia fornecer uma alternativa adequada para E. coli. No entanto, P. aeruginosa é um patógeno humano oportunista. Aqui, detalhamos um procedimento que pode ser usado para gerar cepas não patogênicas de P. aeruginosa através de exclusões genômicas sequenciais usando o plasmídeo pEX100T-NotI. A principal vantagem deste método é produzir uma cepa livre de marcadores. Este método pode ser usado para gerar cepas p. aeruginosa altamente atenuadas para a produção de produtos comerciais, ou para projetar cepas para outros usos específicos. Também descrevemos um modelo de camundongo simples e reprodutível de infecção sistêmica bacteriana por meio de injeção intraperitoneal de cepas de teste validadas para testar a atenuação da cepa geneticamente modificada em comparação com a cepa BL21 aprovada pela FDA de E. coli.
Introdução
Pseudomonas aeruginosa é um patógeno bacteriano oportunista que pode causar doenças fatais em seres humanos, especialmente no imunocomprometido. A patogenicidade de P. aeruginosa é devido à expressão de muitos fatores de virulência, incluindo proteases e lipopolissacarídeos, bem como a sua capacidade de formar um biofilme protetor1. Devido à sua capacidade de produzir fatores de virulência e causar doenças em seres humanos, usando P. aeruginosa para fazer produtos comerciais apresenta preocupações de segurança. Cepas não patogênicas de E. coli têm sido tradicionalmente usadas para bioengenharia de produtos médicos e comerciais para uso humano. No entanto, alguns produtos são difíceis para E. coli fazer, e muitos são embalados em corpos de inclusão, tornando a extração trabalhosa. Cepas bacterianas projetadas com a capacidade de fazer e secretar produtos específicos é altamente desejável, como secreção provavelmente aumentaria o rendimento e facilitar os processos de purificação. Assim, cepas não patogênicas de outras espécies de bactérias (por exemplo, espécies que utilizam mais vias de secreção) podem fornecer alternativas úteis para E. coli. Recentemente relatou o desenvolvimento de uma cepa de P. aeruginosa, PGN5, em que a patogenicidade e toxicidade do organismo é altamente atenuada2. Importante, esta cepa ainda produz grandes quantidades do alginato polissacarídeo, um componente comercialmente interessante do biofilme P. aeruginosa.
A cepa PGN5 foi gerada usando um procedimento de troca alocílica de duas etapas com o plasmid pEX100T-NotI para excluir sequencialmente cinco genes(toxA, plcH, phzM, wapR, aroA), conhecido por contribuir para a patogenicidade do organismo. pEX100T-NotI foi gerado mudando o SmaI para um local de reconhecimento de enzimas de restrição NotI dentro do local de clonagem múltipla do plasmídeo pEX100T, que foi desenvolvido no laboratório3,4de Herbert Schweizer. O local de reconhecimento para a enzima de restrição NotI é uma sequência de DNA mais rara em comparação com smai e menos propensos a estar presente em seqüências sendo clonadas, portanto, é mais conveniente para fins de clonagem. O plasmídeo carrega genes que permitem a seleção, incluindo o gene bla, que codifica ß-lactamase e confere resistência à carbenicilina, e o gene B. subtilissacB, que confere sensibilidade à sacarose (Figura 1A). O plasmídeo também carrega uma origem de replicação (ori) compatível com E. coli, e uma origem de transferência(oriT)que permite a transferência plasmídea de E. coli para espécies pseudomonas via conjugação. No entanto, o plasmídeo carece de uma origem de replicação compatível com Pseudomonas,e, portanto, não pode replicar dentro da espécie Pseudomonas (ou seja, é um vetor de suicídio Pseudomonas). Estas características fazem pEX100T-NotI ideal para alvejar deleções genéticas do cromossoma de Pseudomonas. As etapas de clonagem de Plasmid são realizadas usando E. coli e o plasmídeo resultante é transferido para Pseudomonas por transformação ou conjugação. Então, através de eventos de recombinação homóloga e etapas seletivas, a exclusão direcionada no quadro é gerada, sem marcadores. Este método de apagar sequencialmente regiões genômicas do cromossomo de P. aeruginosa poderia ser usado para gerar cepas pseudomonas altamente atenuadas, como PGN5, ou para projetar cepas para outros usos específicos (por exemplo, cepas deficientes em endonucleases para propagação plasminável ou cepas deficientes em proteases para a produção de proteínas de interesse).
A virulência geral de cepas de bactérias é afetada por condições e fases de crescimento, durante as quais as mutações ocorrem com freqüência. Portanto, medir a segurança das cepas geneticamente modificadas pode ser um desafio. Para avaliar isolados bacterianos para a virulência sistêmica, adaptamos um protocolo de infecção previamente publicado por injeção intraperitoneal de camundongos C57BL/65. Nós modificamos este procedimento para usar estoques bacterianos congelados para a injeção, que permitiu a dosagem precisa e a validação fácil das tensões usadas. Neste modelo, a cepa E. coli BL21, aprovada pela FDA para produção de biofármacos, foi utilizada como padrão de segurança de controle para determinar a patogênese relativa da cepa6,7,8. A principal vantagem para o uso deste método é que ele é reproduzível e minimiza as fontes de variação, pois as cepas infectantes são validadas para o número de células bacterianas, fenótipo e marcadores genéticos antes e depois da infecção. Com estas etapas controladas, o número de animais necessários é reduzido. Neste modelo, cepas de P. aeruginosa que resultam em taxas de mortalidade de urina C57BL/6 iguais ou inferiores a E. coli BL21 quando injetadas intraperitoneally podem ser consideradas atenuadas. Este modelo simples do rato da infecção pode igualmente ser usado para avaliar o pathogenicity atenuado de tensões genetically projetadas de outras espécies usando a tensão FDA-aprovada do E. coli como a referência. Os passos 1-7 detalham a geração de exclusões genômicas sequenciais em P. aeruginosa (Figura 1) e passos 8-12 detalham o uso de um modelo de mouse para testar a patogenicidade das cepas p. aeruginosa.
Protocolo
Antes do início das experiências com animais, o protocolo a ser utilizado deve ser aprovado pelo Institutional Animal Care and Use Committee (IACUC). A aprovação do protocolo descrito foi obtida através da IACUC na Universidade Marshall (Huntington, WV, EUA).
1. Projeto Plasmid
- Para gerar uma exclusão genética usando o plasmid pEX100T-NotI, clonem as regiões do DNA flanqueando a seqüência de exclusão desejada e inseram no local de restrição noti do plasmídeo. A inserção plasmídea deve conter cerca de 500 nucleotídeos a montante da sequência alvo diretamente adjacente a cerca de 500 nucleotídeos a jusante da sequência de exclusão alvo. Além disso, a inserção deve conter a sequência de reconhecimento NotI (GCGGCCGC) em suas extremidades de 5 e 3'(Figura 1B).
2. Preparação Plasmid
- Opção 1: Utilize os procedimentos tradicionais de clonagem. Use PCR para amplificar regiões genômicas a montante e a jusante do gene de interesse, seguido pelo crossover PCR9,10 para se juntar aos fragmentos gerados, restrição endonuclease digestão do produto PCR e plasmasia, e ligadura11 ( Figura1B,C).
- Opção 2: Depois de projetar a sequência de exclusão em silico, contraia uma empresa que de novo sintetiza para inserir no plasmídeo pEX100T-NotI. Muitas empresas simplificaram o processo de clonagem para gerar de forma rápida e eficiente o plasmídeo de interesse. Além disso, a sequência verifica os plasmídeos para serem livres de mutação antes do parto.
3. E. coli Transformação
- Transforme e. coli eletrocompetente com o plasmídeo de acordo com as recomendações do fabricante. Usando um laço inoculador estéril, raia 10 μL da reação de transformação para colônias isoladas em uma placa de ágar pré-aquecida Luria Broth (LB) complementada com 100 μg/mL de carbenicilina e incubada durante a noite em 37 °C.
NOTA: Todos os equipamentos e meios usados para bactérias da cultura devem ser tratados de acordo com as diretrizes de segurança da instituição. - Passagem duas vezes.
- Retire a placa da incubadora e identifique uma colônia isolada. Usando um laço inoculante estéril, pegue a colônia e estria uma placa de ágar LB pré-aquecido complementado com 100 μg/mL de carbenicilina para colônias isoladas. Incubar durante a noite em 37 °C. Repita este passo mais uma vez para gerar uma cultura pura.
- Usando um laço inoculador estéril, inocular 5 mL de LB com uma única colônia da placa de ágar final. Coloque a cultura em uma incubadora tremendo em 37 °C durante a noite. No dia seguinte, misture 1 mL desta cultura com 1 mL de 5% em um criovial e armazenar a -80 °C para gerar um estoque congelado da cepa.
4. Preparação da tensão bacteriana e conjugação triparental
- Use uma única colônia isolada de placas de ágar das seguintes cepas para inocular culturas de caldo e coloque em uma incubadora tremer durante a noite a 37 °C.
- Adicionar E. coli pEX100T-NotI em 5 mL de LB complementado com 100 μg/mL de carbenicilina.
- Adicionar P. aeruginosa cepa PAO1 em 5 mL de Pseudomonas Isolamento Caldo (PIB).
- Adicione E. coli prk2013 em 5 mL de LB complementado com 50 μg/mL de kanamicina.
NOTA: O plasmídeo prk2013 é um plasmóso auxiliar que se replica em E. coli, mas não P. aeruginosa; transporta os genes de transferência de ação transquenais que mobilizam o plasmid pEX100T-NotI do doador E. coli para o receptor P. aeruginosa 12. P. aeruginosa é um patógeno de Nível 2 de Biossegurança (BSL-2). Por favor, siga as diretrizes da instituição para a segurança ao trabalhar com organismos BSL-2.
- No dia seguinte, retire as culturas durante a noite da incubadora e adicione 0,5 mL de cada cultura a um tubo de microcentrírífuga de 1,5 mL. Centrífuga a 6.000 x g por 5 min. Descarte o supernatant e suspenda a pelota celular em 50-100 μL de LB.
- Pipete a suspensão celular inteira em uma gota em uma placa de ágar LB pré-aquecido. Deixe a gota secar. Em seguida, inverna a placa e incubar a 37 °C para 4-6 h.
- Após a incubação, use um laço estéril da inoculação para coletar as pilhas em 1 mL do LB em um tubo do microcentrífuga. Pipeette para cima e para baixo para misturar as células.
- Usando um propagador de células, células com raia uniformemente em uma placa seca pré-aquecida Pseudomonas Isolation Agar (PIA) complementada com 300 μg/mL de carbenicilina. Placas múltiplas da raia com volumes crescentes da mistura da pilha (por exemplo, 10 μL, 100 μL, 500 μL). Incubar durante a noite em 37 °C.
5. Detecção de recombinantes de crossover único de P. aeruginosa
- Retire as placas da incubadora e inspecione colônias resistentes à carbenicina isolada. Porque o plasmid pEX100T-NotI não pode replicar em P. aeruginosa, colônias que cresceram em placas carbenicilina-complementadas devem ter surgido a partir de células em que o plasmídeo foi integrado no cromossomo.
- Escolha pelo menos 4 dessas colônias e raia para isolamento em placas pré-aquecidas de PIA complementadas com 300 μg/mL de carbenicilina. Incubate placas durante a noite em 37 °C.
- Retire as placas da incubadora e inspecione para crescimento. Colônias resistentes à carbenicina devem ser recombinantes de cruzamento único (ou seja, eles incorporaram o plasmídeo no cromossomo através de um evento de recombinação entre uma região homóloga da inserção plasmítica e o cromossomo de P. aeruginosa).
- Patch 8 ou mais colônias com palitos estéreis em placas pré-aquecidas de: 1) PIA complementada com 300 μg/mL de carbenicilina e 2) PIA complementada com 300 μg/mL de carbenicilina e 10% de sacarose (sem glicerol).
- Se nenhum crescimento da colônia foi obtido a partir do passo 5.1, repita a conjugação e aumente o volume da mistura celular listrada na etapa 4.5. Se ocorrer demasiado crescimento, repita a conjugação e diminua o volume estriado.
- Se a conjugação falhar repetidamente, prepare células eletrocompetentes da cepa P. aeruginosa e transforme-se diretamente com o plasmídeo pEX100T-NotI. Protocolos detalhados para a preparação do eletrocompetente P. aeruginosa e transformação estão disponíveis em outros lugares13,14.
- Incubar placas a 37 °C durante a noite.
- Retire as placas da incubadora e inspecione para crescimento. Verdadeiros recombinantes de crossover único serão resistentes à carbenicina e sensíveis à sacarose (ou seja, colônias que cresceram na PIA complementadas com carbenicilina, mas não crescerão na PIA complementadas com carbenicilina e sacarose).
- Escolha 4 ou mais verdadeiros recombinantes de crossover único e inocular cada um em 5 mL de LB sem seleção. Incubar em uma incubadora tremendo em 37 °C durante a noite.
- Se não foram detectados recombinantes de cruzamento único, repita a conjugação.
6. Detecção de recombinantes de crossover duplo de P. aeruginosa
- Para cada cultura de caldo, inocular 10 μL de cultura em um prato pré-aquecido de PIA complementado com 10% de sacarose (sem glicerol) e raia para colônias isoladas. Incubate placas durante a noite em 37 °C.
- No dia seguinte, retire as placas da incubadora e inspecione-as para o crescimento. Colônias resistentes à sacarose devem ser recombinantes de cruzamento duplo (ou seja, removeram o plasmídeo do cromossomo através de um evento de recombinação entre a outra região homóloga da inserção plasmítica e o cromossomo P. aeruginosa).
- Patch pelo menos 20 colônias com palitos estéreis em placas pré-aquecidas de: 1) PIA, 2) PIA complementada com 10% de sacarose (sem glicerol), e 3) PIA complementada com 300 μg/mL de carbenicilina.
- Incubate placas durante a noite em 37 °C.
- Retire as placas da incubadora e examine-as para o crescimento. Verdadeiros recombinantes de cruzamento duplo serão sensíveis à carbenicilina e resistentes à sacarose (ou seja, colônias que cresceram na PIA e PIA complementadas com sacarose, mas não cresceram na PIA complementadas com carbenicilina).
7. Confirmação da deleção do gene através da colônia PCR
- Prepare 10-20 colônias para uma tela de exclusão com PCR.
- Pegue o crescimento de um suspeito de dupla cruze o recombinante com um palito estéril e suspenda as células em 50 μL de soro soro para baixo teor de fosfato 1x (PBS). Ferva a suspensão a 100 °C por 10 min, centrífuga por 3 min a 13.000 x g,e depois coloque no gelo.
- Executar PCR para selecionar colônias para a exclusão direcionada.
- Use 1 μL do supernatant como o modelo em uma reação de 25 μL PCR para confirmar a exclusão do gene do interesse.
- Use primers específicos do gene que amplificam a região da exclusão genômica. Use primers que amplificam a região da exclusão genômica mais 100-200 bp de flanqueamento de sequências a montante e a jusante.
- Prepare uma reação de PCR de controle separado com a cepa do pai (por exemplo, PAO1).
NOTA: As condições do biromórciclo variarão dependendo da temperatura de annealing ideal para pares de primer, o coquetel de polimerase usado e o comprimento da região a ser amplificado.
- Realize eletroforese de gel agarose nos produtos PCR. Colônias em que a região de interesse foi excluído produzir produtos menores de amplificação do que colônias que não têm a exclusão (Figura 2).
- Escolha uma ou mais colônias com a exclusão confirmada pelo PCR. Raia para colônias isoladas em uma placa pre-aquecida de PIA (s) e incubam em 37 °C durante a noite.
- Passagem pelo menos mais uma vez. Retire a placa (s) da incubadora e identifique uma colônia isolada. Usando um laço inoculante estéril, pegue a colônia e estria uma placa de ágar PIA pré-aquecida para colônias isoladas. Incubar durante a noite em 37 °C.
- Escolha uma colônia de cada placa final e use para inocular 5 mL de PIB. Coloque em uma incubadora tremendo a 37 °C durante a noite.
- Misture 1 mL desta cultura com 1 mL de 5% de leite desnatado em um criovial e armazenar a -80 °C para gerar um estoque da cepa.
- Usando essa cultura, prepare dna genômico da cepa (por exemplo, usando um kit de purificação de DNA). Amplifique a região de exclusão genômica usando PCR e primers específicos da região de interesse.
- Purifique esses produtos PCR (por exemplo, com um kit de purificação de DNA, ou extração de cloroforme fenol) e sequenciar diretamente com os primers específicos do gene ou ligado em um vetor para sequenciamento com primers plasmídeos específicos.
- Depois que a supressão do gene é confirmada através do sequenciamento, repita este procedimento com a nova cepa de exclusão para gerar sequencialmente numerosas exclusões genômicas sem marcadores. Quando a cepa desejada é gerada, use o sequenciamento do genoma inteiro para verificar as exclusões direcionadas e para detectar outras alterações no genoma (em comparação com a cepa de referência, por exemplo, PAO1) que ocorreu durante todo o processo. Depois de anotar os genes, deposite a sequência no GenBank e recorde de números de adesão.
8. Preparação da tensão bacteriana para o teste animal
- Para testar a patogépcidade atenuada das cepas p. aeruginosa, primeiro prepare culturas e estoques validados. Prepare as cepas de interesse p. aeruginosa, uma cepa selvagem de P. aeruginosa (virulenta) e uma cepa aprovada pela FDA de E. coli (por exemplo, BL21) para servir como um controle de segurança não patogênico.
- Estria as cepas das bactérias que estão sendo testadas em ágar seletivo de estoques congelados sequenciados e validados. Incubar a 37 °C durante a noite.
- Com um laço inoculante estéril, pegue uma única colônia de cada cepa e raia para colônias isoladas em mídia seletiva novamente. Incubar a 37 °C durante a noite.
- Retire as placas da incubadora. Para cada cepa, escolha uma única colônia e raia para o isolamento em placas LB.
- Após 24 h de crescimento a 37 °C, inocular um frasco de 500 mL contendo 250 mL de LB com uma única colônia isolar de cada cepa.
- Etapa de validação: usando os restos da mesma colônia, valide a cepa usando PCR e primers específicos de tensão e/ou cartilha para verificar a presença de modificações genéticas feitas à cepa. Use as cartilha abaixo para verificação de cepas no exemplo apresentado:
E. coli BL21:
T7 polymerase F:TGGCTATCGCTAATGGTCTTACG
T7 polimerase R:TTACGCGAACGAAGTCC
VE2 e PGN5:
aroa aroa F: GCGAACGCCAACAGCCGATAAAGC
aroa aroa R: ATCTGGCTCGCGATGCCGGTCC
- Etapa de validação: usando os restos da mesma colônia, valide a cepa usando PCR e primers específicos de tensão e/ou cartilha para verificar a presença de modificações genéticas feitas à cepa. Use as cartilha abaixo para verificação de cepas no exemplo apresentado:
- Incubar as culturas em uma incubadora tremendo em 160 rpm e 37 °C até que alcancem o crescimento da fase do registro (isto é, medida de OD600 de 0.4-0.6 em um espectrômetro).
- Usando o valor OD600 obtido quando a fase de registro foi alcançada, calcule o volume de caldo necessário para produzir 2,5 x 109 unidades formadoras de colônias (CFU) por mL. Pelotas o volume de caldo calculado em tubos de 50 mL em 4.500 x g por 10 min.
- Descarte o supernatant e resuspenda a pelota em um tubo usando 50 mL de 1x PBS para lavar as pilhas. Centrífuga novamente em 4.500 x g por 10 min.
- Descarte o supernatant e resuspenda a pelota em 25 mL de 5% de leite desnatado em 1x PBS.
- Etapa de validação: Use uma amostra da resuspensão de 25 mL para realizar contagens de placas viáveis para determinar o número de CFU/mL.
- Aliquot a 25 mL resuspensão da cultura do leite desnatado em 2 estoques de cultura mL em 2 mL crioviais. Flash congelamento em nitrogênio líquido e armazenar a -80 °C, pelo menos durante a noite antes do uso.
9. Validação de crescimento e tensão de estoques armazenados para testes em animais.
- Para que cada cepa seja testada, retire pelo menos 3 crioviais de estoques congelados de -80 °C de armazenamento e descongele a 4 °C para 2-4 h. Se algum estoque congelado permanecer, brevemente quente a 37 °C.
- Tome amostras pequenas de cada criovial para validar cada tensão.
- Realizar contagens de placas viáveis para determinar o número de CFU/mL. É normal ter menos CFU/mL após o congelamento, devido à morte de algumas células bacterianas.
- Use PCR e primers específicos de tensão para validar cada cepa.
- Raia cada tensão em mídia seletiva para verificar o fenótipo.
- Depois de confirmar que as cepas são do genótipo correto e fenótipo, e validando CFU /mL, proceder ao teste em animais.
10. Inoculação de animais com cepas bacterianas por injeção
- Na manhã das injeções, remova os crioviais das tensões bacterianas que estão sendo testadas e descongele em 4 °C para 3-4 h. Degelo 0.5 mL para cada rato que será injetado. Mantenha frascos no gelo após o descongelamento e injetar ratos dentro de 2 h.
- Após o descongelamento, transfira cada criovial para um novo tubo de 2 mL e centrífuga a 4.500 x g por 10 min, descarte o supernatant e resuspenda a pelota celular em 1 mL de 1x PBS.
- Centrífuga novamente em 4.500 x g para 10 m. Descarte supernatant e resuspender pelota em 1x PBS para uma concentração final 2,5 x 109 CFU/mL. Para determinar a quantidade de 1x PBS necessária para a resuspensão, use os dados cfu/mL obtidos a partir de contagens de placas viáveis em estoques congelados na etapa 2.2.1. A quantidade exata de 1x PBS usado variará ligeiramente entre tensões.
- Tome 3 amostras da suspensão final de cada cepa para validar CFU/mL, genótipo e fenótipo, conforme descrito acima.
- Para cada cepa, alíquota 1,5 mL de PBS / suspensão celular em um tubo de 2 mL por 5 ratos para limitar o número de vezes que o tubo é inserido. Além disso, preparar tubos de 1x PBS para injeções de controle.
- Reúna camundongos (10 machos e 10 c57BL/6 femininos por grupo) e materiais necessários para injeções (seringas, agulhas, recipientes afiados, marcadores, caneta e papel,etc.). Mude-se para a sala cirúrgica animal estéril. Limpe todas as superfícies com lenços de sanitização.
- Para eliminar a aflição e o risco de ferimento ao experimentador, traga somente um grupo do sexo e experimental dos ratos ao quarto cirúrgico de cada vez (por exemplo, um grupo de 10 ratos masculinos a ser contaminados com uma tensão particular). Use dois pares de luvas de látex para eliminar a punção de luvas se mordido. Use jaleco, óculos de segurança e máscara facial para evitar a contaminação.
- Comece as injeções do grupo controle com 1x PBS. Isto irá verificar se quaisquer efeitos adversos resultam apenas da injeção.
- Retire um rato da gaiola. Basta remover um rato de cada vez.
- Pese o mouse e marque sua cauda com marcador permanente para rastrear a perda de peso pós-injeção.
- Abra uma nova seringa de 1 mL e 27 G agulha (use uma nova seringa e agulha para cada rato para eliminar a contaminação) e injetar 200 μL de estéril 1x PBS.
- Pegue o rato atrás de suas orelhas usando o polegar e o indicador. Pitada para criar dobra da pele na nuca para segurar - uma dobra mais apertada reduz o movimento do pescoço e risco de ser mordido durante a injeção. Mantenha a cauda na palma da mão usando o mindinho para segurar o mouse plana com pouco movimento.
- Vire o mouse e insira agulha no ângulo de 30° na cavidade peritoneal para o lado esquerdo ou direito da linha média. Levante a agulha ligeiramente uma vez inserida para garantir que ela fosse inserida na área intraperitoneal e não em órgãos. Lentamente injetar o PBS e, em seguida, retirar a agulha.
- Coloque a agulha usada em um recipiente designado de pontas de risco biológico. Não reutilize a seringa ou a agulha. Um bóus no local da injeção é típico.
- Após a injeção, mova o rato para uma gaiola separada.
- Repita o procedimento com o próximo mouse. Depois que todos os ratos de uma gaiola são injetados, revide-os a sua gaiola original imediatamente.
- Após injetar o grupo controle, comece a injetar suspensões de cepas a serem testadas após o mesmo procedimento.
- Injetar 200 μL da suspensão celular. Ao começar com suspensões celulares de 2,5 x 109 CFU/mL, cada mouse recebe 5 x 108 CFU.
NOTA: Essas concentrações foram otimizadas com pesquisas preliminares em animais para determinar doses letais de cada cepa. A dosagem pode precisar ser ajustada para outras cepas/espécies.
- Injetar 200 μL da suspensão celular. Ao começar com suspensões celulares de 2,5 x 109 CFU/mL, cada mouse recebe 5 x 108 CFU.
- Uma vez que todas as injeções estão completas, retornar ratos para a sala de habitação para aliviar a angústia. Limpar a área de trabalho com lenços de sanitização.
- Monitore os animais para a mortalidade após a injeção, verificando gaiolas para ratos mortos a cada 3 h para 72 h e a cada 12 h por 10 dias. Registre o peso dos ratos a cada 12 h para determinar a perda de peso devido à doença.
- Registre o comportamento adverso nas horas seguintes à injeção, como a diferença de postura, a falta de aliciamento ou escavação, imobilidade ou alterações na respiração. Camundongos que morrem de lesão associada à injeção exibirão comportamento adverso e morrerão rapidamente após a injeção. Por outro lado, camundongos que cessam de infecção não vai começar a apresentar comportamento adverso ou morte até depois de 18 h. Todos os camundongos que apresentam comportamento adverso ou extensos sinais de morbidade, incluindo respiração rápida ou trabalhada, imobilidade, postura curvada, olhos afundados, desidratação grave ou outros devem ser sacrificados para reduzir o sofrimento desnecessário (conforme protocolos aprovados pelo IACUC). No entanto, em nossos experimentos, a eutanásia não foi necessária para nenhum camundongo durante o experimento. Camundongos sobreviventes foram sacrificados 10 dias após a conclusão dos testes.
- Após o período de monitoramento de 10 dias, permitir que os animais que permanecem se recuperem totalmente e fiquem livres de qualquer infecção administrada durante o teste. Eutanásia animais após o procedimento iacuc.
11. Análise Estatística da Mortalidade Animal
- Realizar análise estatística usando software de grafógrafo. Qualquer software capaz de produzir gráficos e realizar análiseestatística é adequado.
- Para traçar os dados de mortalidade, use a coluna X para representar o tempo (h) e a coluna Y para representar os grupos testados.
- Represente cada mouse ou sujeito no estudo usando código = 0 (zero) ou código = 1, indicando sobrevivência ou morte, respectivamente.
- Para cada animal que morre, coloque um 1 na coluna Y desse grupo no momento da morte na coluna X. Se houver várias mortes em um único momento dentro de um grupo, uma cópia desse ponto de tempo pode ser colocada na coluna X. Por exemplo, se três indivíduos dentro de um grupo morrerem às 3 h, o ponto de tempo de 3 h aparecerá três vezes na coluna X.
- Para todos os animais sobreviventes dentro de um grupo, coloque um zero na coluna Y no ponto de tempo final medido. Por exemplo, se quatro camundongos sobreviverem, coloque quatro pontos de tempo de término na coluna X marcados com quatro zeros na coluna Y.
- Depois que os dados animais são inseridos para todos os grupos, use um modelo gráfico de sobrevivência para produzir um gráfico de sobrevivência.
- Deixe a codificação padrão como 0 e 1.
- Defina os parâmetros para o gráfico como porcentagem.
- Uma vez que os parâmetros para a curva de sobrevivência são selecionados, realize a análise estatística usando um teste de Mantel-Cox (rank de registro).
- Formato Kaplan-Meier parcelas usando dados estatísticos.
NOTA: Cepas que apresentam taxas de mortalidade que são menores ou iguais à cepa dos pais e a cepa aprovada pela FDA (por exemplo, E. coli BL21) podem ser consideradas atenuadas.
12. Visualização da Infecção com Bioluminescência
- Para visualizar o progresso da infecção, insira um operon bioluminescente cromossômico(luxCDABE)na cepa PGN5 e VE2 testada. Os plasmídeos e protocolo usado para rotular essas cepas foram desenvolvidos no laboratório schweizer e pode não ser compatível com todas as espécies / cepas de bactérias13. É importante ressaltar que a visualização da infecção é opcional; assim, a inserção genômica deste operon não é necessária para realizar o estudo de mortalidade descrito acima.
- Prepare e valide cepas usando o método descrito acima. Além disso, verifique se há bioluminescência em cepas rotuladas em cada etapa de validação.
- Depois que as cepas são preparadas, injete os animais em grupos de 10 com as cepas bioluminescentes seguindo os degraus acima.
- Imagem dos animais a cada 3 h para 24 h usando um sistema de imagem animal capaz de bioluminescência.
- Primeiro prepare o imager, definindo os parâmetros da câmera e aquecendo o palco para os animais. Também definir o fluxo de oxigênio para 1,5 L por min (ou seguindo as recomendações do fabricante).
- Depois que o imager e o estágio forem estabilizados, coloc um rato na câmara da anestesia imediatamente depois da injeção e administra 3.5% isoflurane na câmara com o fluxoo 2 por aproximadamente 4 min. Os métodos de anestesia podem variar dependendo da câmara e/ou do agente anestésico utilizado; siga as recomendações do fabricante. Determinar a anestesia adequada através do teste reflexo de retirada.
- Mova o mouse para o estágio estabilizado de temperatura. Posicione o mouse de costas com os braços estendidos e ajuste o mouse com um cone de nariz para administração de 2,5% isoflurane durante todo o procedimento de imagem.
- Feche a porta e tire imagens bioluminescentes e raios-X do rato.
- Quando a imagem estiver completa, devolva o mouse à sua gaiola e monitore-a. O rato deve recuperar a consciência dentro de 3-5 min.
- Continue a imagem ratos a cada 3 h para 24 h, cada vez usando um mouse diferente de cada grupo. Não reimage um mouse dentro de 24 h devido à possibilidade de efeitos adversos devido à reexposição à anestesia. Um único rato deve receber somente uma dose da anestesia cada 24-36 h. Limpe a plataforma da imagem latente depois que cada rato é imaged. Desligue o imager entre os pontos de tempo de imagem.
NOTA: A bioluminescência desaparecerá, independentemente da cepa injetada. A intensidade e longevidade da bioluminescência variarão dependendo de muitos fatores, incluindo o número de bactérias injetadas, tensão do rato, tensão bacteriana, etc.
Resultados
Como mostrado na Figura 2,a exclusão genômica direcionada pode ser confirmada usando a colônia PCR com cartilhas específicas que amplificam a região de interesse. Colônias que carregam uma exclusão genômica produzirão um tamanho mais curto da banda PCR em comparação com colônias de tipos selvagens. Uma tela PCR de 10-12 colônias geralmente é suficiente para detectar pelo menos uma colônia que carrega a exclusão direcionada. Se não houver exclusões após várias rodadas de telas, repita o procedimento começando com a conjugação. Se a exclusão ainda falhar, a inserção plasmídea pode precisar ser confirmada por meio de sequenciamento, redesenhado ou a exclusão pode ser letal. Após a verificação de uma exclusão gênica via PCR, confirme a exclusão por meio de sequenciamento. A cepa resultante pode ser submetida ao procedimento repetidamente para gerar modificações genômicas sequenciais.
Como mostrado na Figura 3,a mortalidade associada à injeção intraperitoneal da cepa atenuada de P. aeruginosa PGN5 (+mucE) foi de 0%, o que equivaleà mortalidade observada com E. coli BL21. Por outro lado, a injeção intraperitoneal da cepa do pai (VE2) foi fatal para 80% dos camundongos. Estes resultados foram obtidos com etapas extensivas validar as tensões injetadas. Quando a causa exata da morte nestes ratos for desconhecida, pode pelo menos na parte ser atribuída à expressão de fatores da virulência na tensão do pai que foram suprimidas da tensão atenuada de PGN5. As diferenças na progressão da infecção foram controladas usando o pai bioluminescence-marcado e as tensões atenuadas. A cepa atenuada permaneceu localizada no local da injeção até que a bioluminescência desaparecia (Figura 4). O afastamento da infecção coincidiu muito provavelmente com o desvanecimento da bioluminescência. A bioluminescência não foi detectada 24 h após a injeção e os camundongos viveram por semanas após a injeção até serem sacrificados, sem efeitos adversos observados.

Figura 1: Geração de apagamentos genéticos em P. aeruginosa com pEX100T-NotI. (A)Mapa do plasmid pEX100T-NotI. (B) Geração de uma construção composta por regiões diretamente a montante (amarelo) e rio abaixo (azul) da região de interesse (ROI), ladeada com sites de reconhecimento de enzimas de restrição NotI. Primeiro, as regiões a montante e a jusante amplificam pcr de forma independente com cartilhas específicas que adicionam locais de digestão noti 5' (por exemplo,NotI-aroA F CGCGGGCGCTGAAGGTCCTGGGCTCCTATCCGAAGCTGCTCT e NotI-aroA R GCGGCCGCAGTTGGGTTGTTCTGCGATGGCGCGCGCGTGGGATGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGG CCAGGCA) e 3' regiões homologous sobrepostas como mostrado (por exemplo, aroA-crossover F CTCCAGGCGCTGGGCAAGGTGGTGGCGGCGGACTGACTTCACCGCGCGCGCGGTGTGTGGAGA ACA e aroA-crossover R TGTTCTCCACGGCGCGCGCGTGACCTCAGTGCGCCAGCACCTTGCCCAGCCTGGAG. Em seguida, use primers contendo PCR com NotI para se juntar aos produtos a montante e a jusante gerados na primeira reação de PCR. (C)O plasmid pEX100T-NotI, armado e pronto. Ligate o produto de PCR cross-over digerido pelo NotI no plasmídeo digerido pelo NotI. (D)Diagrama de fluxo do processo para excluir regiões genômicas do cromossomo P. aeruginosa usando o plasmídeo pEX100T-NotI. Depois que a supressão desejada foi confirmada e purificada, a tensão resultante pode ser tomada através do procedimento repetidamente para suprimir outras regiões genômicas do cromossomo. Quando a cepa desejada é obtida, sequenciar todo o genoma para confirmar exclusões e outras alterações ao cromossomo. A patogépcidade da cepa pode então ser testada em camundongos usando o procedimento descrito na Parte II do Protocolo. Clique aqui para ver uma versão maior deste número.

Figura 2: Eletroforese gel de produtos PCR colônia de uma tela para a exclusão aroA para gerar a cepa P. aeruginosa atenuada, PGN5. Os produtos do PCR da colônia funcionam nas pistas 2-5 e 8-11 indicam colônias com aroAdo selvagem-tipo. Os produtos colony PCR funcionam nas pistas 6 e 7 carregam a exclusão do gene aroA, indicada pelo produto menor do PCR (asteriscos amarelos). Os primers usaram especificamente a região genômica contendo o gene aroA: aroA-F: GCGAACGAACAGCCGATAAAGC, e aroA-R: ATCTGGCTCGCGCGCGCGCGGTCC. O tamanho esperado do produto PCR em colônias do tipo selvagem foi de 2.548 nucleotídeos (nt). O tamanho esperado do produto pcr em colônias com exclusão de aroa foi de 307 nt. Uma escada do ADN foi funcionada nas pistas 1 e 12. Clique aqui para ver uma versão maior deste número.
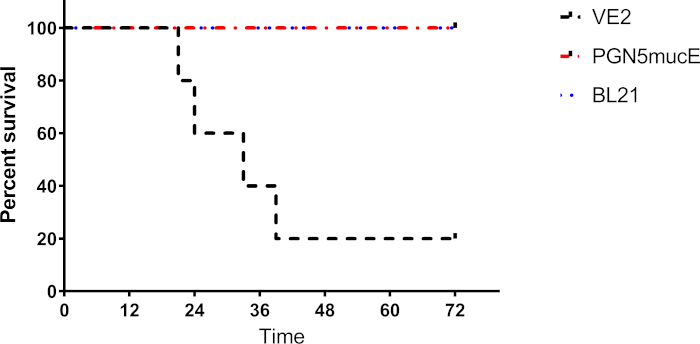
Figura 3: Mortalidade geral de camundongos injetados com cepa p. aeruginosa patogênica (VE2), cepa p. aeruginosa atenuada (PGN5+mucE) e a cepa de E. coli de controle da FDA (BL21). Somente os ratos injetados com a tensão patogênica do pai exibiram a mortalidade em 80%. Cepa atenuada de P. aeruginosa e cepa de E. coli controle da FDA apresentaram mortalidade de 0%. Clique aqui para ver uma versão maior deste número.
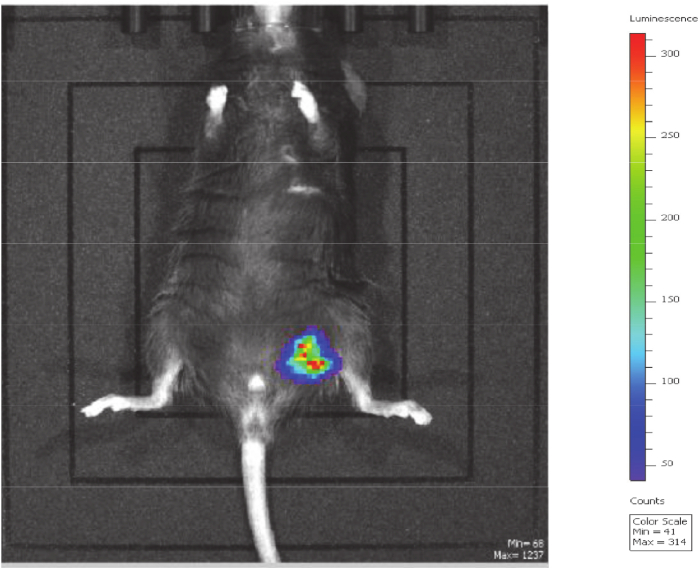
Figura 4: Imagem do rato 3 h pós-injeção de cepa atenuada de P. aeruginosa PGN5+mucE carregando um marcador bioluminescente. As bactérias bioluminescentes foram detectáveis até 18-24 h após a injeção. Durante este período, a bioluminescência permaneceu no local da injeção indicando que as bactérias permaneceram localizadas no local da injeção. Este rato recuperou inteiramente sem efeitos adversos. Clique aqui para ver uma versão maior deste número.
Discussão
O plasmídeo pEX100T-Not1 é um mediador eficiente de exclusões genômicas sequenciais que são livres de marcadores e no quadro. Quando a engenharia de cepas bacterianas para a vidulência atenuada, a exclusão de sequências genéticas inteiras em vez de gerar mutações pontuais diminui a probabilidade de reversão a um fenótipo virulento. Além disso, cada aupressão gênica de patogenicidade atenua ainda mais o patógeno, reforçando ainda mais a estabilidade da atenuação.
Este método também pode ser usado para gerar modificações genômicas além de exclusões, como mutações pontuais e inserções, simplesmente modificando o design da inserção plasmínica. Estes tipos de modificações podem ser mais úteis do que deleções genéticas inteiras para bactérias de engenharia com metabolismo modificado, por exemplo. A modificação genômica sequencial tem um potencial significativo para gerar cepas bacterianas de designer para atender a propósitos específicos em pesquisa e indústria. Outros métodos de geração de modificações genômicas desejadas sem marcadores em bactérias foram descritos15,16,17,18. Tal como acontece com todos os métodos de edição do genoma, as tentativas de modificação em regiões genômicas essenciais podem ser letais e, portanto, mal sucedidas. Nestes casos, a identificação de modificações genéticas diferentes ou de outros genes do candidato é exigida gerar a tensão bacteriana do interesse.
Dado os inúmeros eventos de replicação e passagens de cada colônia ao longo deste protocolo, alterações não intencionais ao genoma ocorrerão na cepa gerada. As alterações genômicas exatas podem ser identificadas através do sequenciamento do genoma inteiro. No entanto, o impacto dessas mudanças é mais difícil de determinar. Quando as bactérias de engenharia para um propósito específico, as mudanças genômicas que não afetam negativamente o crescimento do organismo ou a via direcionada (s) são toleráveis. Dependendo da cepa que está sendo gerada, pode ser possível identificar uma "leitura" para garantir que a cepa ainda é útil para o propósito pretendido. Por exemplo, com o PGN5, o objetivo era criar uma cepa atenuada que mantivesse a capacidade de produzir grandes quantidades de alginato. Após a supressão de cinco genes de patogénio, a quantidade e composição do alginato produzidas pelo PGN5 foram medidas e determinadas como comparáveis a outras cepas produtoras de alginato. Assim, a produção de alginato não foi afetada pelas cinco deleções gênicas, nem pelas alterações genômicas não intencionais que ocorreram durante o desenvolvimento do PGN5.
Um modelo de injeção intraperitoneal do rato foi usado para determinar se uma tensão projetada foi atenuada comparada à tensão do pai e e. coli BL21, uma tensão aprovada pelo FDA para a produção dos biopharmaceuticals. As medidas mais importantes tomadas durante este procedimento de teste em animais foram a preparação e validação de estoques bacterianos congelados. Preparação e uso de culturas bacterianas congeladas para injetar ratos é preferível ao uso da cultura contínua, pois reduz o número de mutações que ocorrem naturalmente em populações bacterianas19. Além disso, as culturas congeladas devem permanecer viáveis por anos. As contagens viáveis da placa não mostraram nenhuma diferença significativa entre o CFU/mL diretamente depois que os estoques foram preparados e três meses após a preparação. O uso de múltiplas etapas de validação ao longo deste procedimento garantiu que o método fosse reproduzível, e os resultados não foram distorcidos pela contaminação de bactérias. Além disso, com o número de medidas de precaução tomadas para garantir a reprodutibilidade, menos animais eram necessários. Usando uma cepa bacteriana que é aprovada pela FDA para a produção biofarmacêutica como o controle (como a cepa de E. coli BL21), este método poderia ser usado para testar a atenuação de outras cepas geneticamente modificadas de P. aeruginosa, ou outras espécies de bactérias.
O uso de bioluminescência como marcador fornece validação adicional das cepas bacterianas injetadas, pois o marcador pode ser visualizado no local da injeção. A inserção do marcador de bioluminescência no cromossomo bacteriano é necessária para imagens de bioluminescência, mas pode não ser possível se trabalhar com cepas/espécies incompatíveis. No entanto, a marcação de cepas com bioluminescência não é necessária para testar a atenuação. As cepas testadas neste estudo foram marcadas com bioluminescência, o que permitiu a visualização das diferenças de localização entre cepas ao longo do curso da infecção. Observamos que a cepa patogênica disseminada através do corpo do camundongo, mas a cepa não patogênica permaneceu no local da injeção. Enquanto este experimento só testou duas cepas muito intimamente relacionadas de P. aeruginosa, sugere que a disseminação bacteriana está ligada à virulência, pelo menos em P. aeruginosa. Assim, este procedimento de rotulagem com bioluminescência para visualizar a progressão da infecção poderia ser usado no futuro para avaliar rapidamente a atenuação de cepas projetadas de bactérias.
Divulgações
O autor Hongwei D. Yu é o Diretor de Ciência e co-fundador da Progenesis Technologies, LLC.
Agradecimentos
Este trabalho foi apoiado pelos Institutos Nacionais de Saúde (NIH) concede R44GM113545 e P20GM103434.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.2 mL tubes with flat caps | ThermoScientific | AB-0620 | via Fisher Scientific |
| 1 mL Syringe | BD | 22-253-260 | via Fisher Scientific |
| 1.5 mL disposable polystyrene cuvette | Fisher Scientific | 14955127 | |
| 1.5 mL Microcentrifuge Tubes | Fisher Scientific | 05-408-129 | |
| 2.0 mL Cryogenic Vials | Corning | 430659 | via Fisher Scientific |
| 27G needle | BD | 14-821-13B | via Fisher Scientific |
| 50 mL tubes | Fisher Scientific | 05-539-13 | via Fisher Scientific |
| Accu block Digital Dry Bath | Labnet | NC0205808 | via Fisher Scientific |
| Benchtop Centrifuge 5804R | Eppendorf | 04-987-372 | via Fisher Scientific |
| Benchtop Microcentrifuge | Sorvall | 75-003-287 | via Fisher Scientific |
| Cabinet Incubator | VWR | 1540 | |
| Carbenicillin disodium salt | Fisher Scientific | BP2648250 | |
| Culture Test Tube, Polystyrene | Fisher Scientific | 14-956-6D | via Fisher Scientific |
| Diposable Inoculation Loops | Fisher Scientific | 22-363-597 | |
| Dneasy UltraClean Microbial Kit (50) | Qiagen | 12224-50 | or preferred method/vendor |
| E.Z.N.A. Cycle Pure Kit (50) | Omega bio-tek | D6493-01 | or preferred method/vendor |
| EcoRI-HF, restriction endonuclease | New England BioLabs | R3101L | |
| Electroporation Cuvettes | Bulldog Bio | NC0492929 | via Fisher Scientific |
| FastLink II DNA Ligation Kit | Epicentre Technologies | LK6201H | via Fisher Scientific |
| Gentamycin Sulfate | Fisher Scientific | BP918-1 | |
| Glycerol | Fisher Scientific | BP229-4 | |
| GoTaq G2 Colorless Master Mix | Promega | M7833 | via Fisher Scientific |
| Isothesia Isoflurane | Henry Schein Animal Health | 29405 | |
| IVIS Lumina XRMS Series III In Vivo Imaging System | Perkins and Elmer | CLS136340 | |
| Kanamycin monosulfate | Fisher Scientific | BP906-5 | |
| LE agarose | Genemate | 3120-500 | via Fisher Scientific |
| Luria Broth | Difco | 240230 | via Fisher Scientific |
| MicroPulser Electroporator | BioRad | 1652100 | |
| Noble agar, ultrapure | Affymetris/USB | AAJ10907A1 | via Fisher Scientific |
| NotI-HF, restriction endonuclease | New England BioLabs | R3189 | |
| One Shot TOP10 Electrocomp E. coli | Invitrogen | C404052 | via Fisher Scientific |
| Phosphate buffered saline powder | Sigma | P3813-10PAK | Sigma-Aldrich |
| Prism 7 | GraphPad | https://www.graphpad.com/scientific-software/prism/ | |
| Pseudomonas isolation agar | Difco | 292710 | via Fisher Scientific |
| Pseudomonas isolation broth | Alpha Biosciences | P16-115 | Custom made batch |
| QIAprep Spin Miniprep Kit (250) | Qiagen | 27106 | or preferred method/vendor |
| Shaking Incubator | New Brunswick Scientific | Innova 4080 | shake at 200 rpm |
| SimpliAmp Thermal Cycler | Applied Biosystems | A24811 | |
| Skim Milk | Difco | DF0032-17-3 | via Fisher Scientific |
| Small Plates (100 O.D. x 10 mm) | Fisher Scientific | FB0875713 | |
| SmartSpec Plus Spectrophotometer | Bio-Rad | 170-2525 | or preferred method/vendor |
| Sucrose | Fisher Scientific | S5-500 | |
| Toothpicks, round | Diamond | Any brand of toothpicks, autoclaved | |
| TOPO TA Cloning Kit, for seqeuncing | Invitrogen | 45-0030 | |
| XAF-8 Anesthesia System Filters | Perkins and Elmer | 118999 | |
| XGI 8 Gas Anesthesia System | Caliper Life Sciences/Xenogen |
Referências
- Gellatly, S. L., Hancock, R. E. Pseudomonas aeruginosa: new insights into pathogenesis and host defenses. Pathogens and Disease. 67 (3), 159-173 (2013).
- Valentine, M. E., et al. Generation of a highly attenuated strain of Pseudomonas aeruginosa for commercial production of alginate. Microbial Biotechnology. , (2019).
- Schweizer, H. P., Hoang, T. T. An improved system for gene replacement and xylE fusion analysis in Pseudomonas aeruginosa. Gene. 158 (1), 15-22 (1995).
- Damron, F. H., Qiu, D., Yu, H. D. The Pseudomonas aeruginosa sensor kinase KinB negatively controls alginate production through AlgW-dependent MucA proteolysis. Journal of Bacteriology. 191 (7), 2285-2295 (2009).
- Yu, H., Boucher, J. C., Hibler, N. S., Deretic, V. Virulence properties of Pseudomonas aeruginosa lacking the extreme-stress sigma factor AlgU (sigmaE). Infection and Immunity. 64 (7), 2774-2781 (1996).
- Baeshen, M. N., et al. Production of Biopharmaceuticals in E. coli: Current Scenario and Future Perspectives. Journal of Microbiology and Biotechnology. 25 (7), 953-962 (2015).
- Marisch, K., Bayer, K., Cserjan-Puschmann, M., Luchner, M., Striedner, G. Evaluation of three industrial Escherichia coli strains in fed-batch cultivations during high-level SOD protein production. Microbial Cell Factories. 12, 58 (2013).
- Ferrer-Miralles, N., Domingo-Espin, J., Corchero, J. L., Vazquez, E., Villaverde, A. Microbial factories for recombinant pharmaceuticals. Microbial Cell Factories. 8, 17 (2009).
- Horton, R. M., Cai, Z. L., Ho, S. N., Pease, L. R. Gene splicing by overlap extension: tailor-made genes using the polymerase chain reaction. Biotechniques. 8 (5), 528-535 (1990).
- Horton, R. M., Hunt, H. D., Ho, S. N., Pullen, J. K., Pease, L. R. Engineering hybrid genes without the use of restriction enzymes: gene splicing by overlap extension. Gene. 77 (1), 61-68 (1989).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular cloning: a laboratory manual. , (1989).
- Figurski, D. H., Helinski, D. R. Replication of an origin-containing derivative of plasmid RK2 dependent on a plasmid function provided in trans. Proceedings of the National Academy of Sciences of the United States of America. 76 (4), 1648-1652 (1979).
- Choi, K. H., Schweizer, H. P. mini-Tn7 insertion in bacteria with single attTn7 sites: example Pseudomonas aeruginosa. Nature Protocols. 1 (1), 153-161 (2006).
- Choi, K. H., Kumar, A., Schweizer, H. P. A 10-min method for preparation of highly electrocompetent Pseudomonas aeruginosa cells: application for DNA fragment transfer between chromosomes and plasmid transformation. Journal of Microbiological Methods. 64 (3), 391-397 (2006).
- Liang, R., Liu, J. Scarless and sequential gene modification in Pseudomonas using PCR product flanked by short homology regions. BMC Microbiology. 10, 209 (2010).
- Martinez-Garcia, E., de Lorenzo, V. Engineering multiple genomic deletions in Gram-negative bacteria: analysis of the multi-resistant antibiotic profile of Pseudomonas putida KT2440. Environmental Microbiology. 13 (10), 2702-2716 (2011).
- Song, C. W., Lee, S. Y. Rapid one-step inactivation of single or multiple genes in Escherichia coli. Biotechnology Journal. 8 (7), 776-784 (2013).
- Yan, M. Y., et al. CRISPR-Cas12a-Assisted Recombineering in Bacteria. Applied and Environmental Microbiology. 83 (17), (2017).
- Prakash, O., Nimonkar, Y., Shouche, Y. S. Practice and prospects of microbial preservation. FEMS Microbiology Letters. 339 (1), 1-9 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados