Method Article
Polysome Fracionamento e Análise de mamíferos Translatomes em uma escala genômica
Neste Artigo
Resumo
Ribossomas desempenham um papel central na síntese de proteínas. Polyribosome (polysome) fracionamento por centrifugação em gradiente de densidade de sacarose permite a determinação direta da eficiência de tradução de mRNAs individuais em uma escala de todo o genoma. Além disso, este método pode ser usado para a análise bioquímica de ribossomas e factores associados--polissomas como acompanhantes e moléculas de sinalização.
Resumo
tradução do mRNA desempenha um papel central na regulação da expressão do gene e representa o maior consumo de energia no processo de células de mamíferos. Consequentemente, a desregulação da tradução do mRNA é considerada ter um papel importante numa variedade de estados patológicos, incluindo o cancro. Os ribossomos também acolher acompanhantes, que facilitam a dobradura de polipeptídeos nascentes, modulando assim a função e estabilidade de polipeptídeos recém-sintetizados. Além disso, os dados indicam que os ribossomas emergentes servir como uma plataforma para um repertório de moléculas de sinalização, que estão implicados numa variedade de modificações pós-translacionais de polipéptidos sintetizados de novo à medida que emergem a partir do ribossoma, e / ou componentes da maquinaria de tradução. Nesse caso, um método bem estabelecido de fraccionamento ribossoma utilizando gradiente de densidade de sacarose de centrifugação é descrito. Em conjunto com o algoritmo desenvolvido "anota" in-house este método permite a determinação direta de diferentradução rential de mRNAs individuais em uma escala de todo o genoma. Além disso, este protocolo versátil, pode ser usado para uma variedade de estudos bioquímicos destinadas para dissecar a função de complexos de proteínas associadas ao ribossoma, incluindo aqueles que desempenham um papel central na dobragem e degradação de polipéptidos sintetizados de novo.
Introdução
As redes reguladoras que controlam a expressão de genes têm sido extensivamente estudados nas duas últimas décadas. A grande maioria desses esforços de pesquisa voltadas para a regulação da transcrição, em que alterações nos níveis de mRNA de estado estacionário em uma escala do genoma foram usados para determinar os chamados "expressão gênica" perfis. Estudos recentes revelaram que os níveis de mRNA de estado estacionário correspondendo apenas vagamente à composição do proteoma de 1,2, indicando assim que os mecanismos de pós-transcrição, incluindo a tradução do mRNA, desempenham um papel importante na regulação da expressão do gene de 3,4. De facto, estima-se que aproximadamente 50% dos níveis de proteína são determinadas ao nível da tradução do mRNA em fibroblastos de rato imortalizadas 5.
tradução do mRNA é um processo altamente regulado durante o qual mRNAs são traduzidos em proteínas via ação orquestrada de ribossomos, RNAs de transferência (tRNAs) e os fatores de acessórios commonly referidos como fatores de conversão (TFS) 6. A síntese da proteína é o mais processo que consome energia em células de mamíferos e, portanto, as taxas de 7 de tradução do mRNA globais são ajustados para acomodar a disponibilidade de nutrientes e as taxas de proliferação celular 8. Além de alterações nas taxas globais de tradução do mRNA, vários estímulos extracelulares (por exemplo, hormônios e fatores de crescimento), sinais intracelulares (por exemplo, os níveis de aminoácidos) e diferentes tipos de estresse (por exemplo, ER-stress) induzem alterações seletivas nas piscinas de mRNAs que são sendo traduzida (translatome) 6. Dependendo do tipo de estímulo, a actividade de tradução de alguns, mas não todos os mRNAs são drasticamente afectadas, resultando assim em alterações no proteoma que são necessárias para montar uma resposta celular rápida 6. Essas mudanças qualitativas e quantitativas no translatome são pensados para ser mediada pela interação entre fatores trans-atuação (por exemplo, componentes da maquinaria de tradução, fatores auxiliares ou miRNAs) e cis-elementos (elementos de RNA ou recursos) presentes em subconjuntos selecionados de mRNAs 6,9. Por exemplo, mudanças nos níveis e / ou atividade de iniciação da tradução limitante fatores eIF4E e eIF2 modular seletivamente a tradução de transcritos com base nas características específicas 5'UTR 6. eIF4E e eIF2 são necessários para o recrutamento de mRNA e iniciador tRNA ao ribossomo, respectivamente 6. Um aumento na actividade eIF4E reforça selectivamente a tradução de mRNAs que albergam 5'UTRs longos e altamente estruturadas, incluindo os que codificam para a proliferação e sobrevivência estimulando as proteínas, incluindo as ciclinas, c-myc e Bcl-xL, 10. Por sua vez, a inativação de eIF2 leva a uma síntese global de proteínas shut-down, enquanto seletivamente até regulação da tradução de mRNAs contendo inibidores de upstream quadros de leitura aberta curtas (uORFs) em suas 5'UTRs, como os mestre codificação treguladores ranscriptional da resposta desdobrada da proteína (por exemplo ATF4). Em resposta a vários estímulos, incluindo nutrientes, factores de crescimento e hormonas, o alvo mecanicista / mamífero de rapamicina (mTOR) via estimula a actividade de eIF4E por inactivação da proteína de ligação de 4E (4E-BP) família de supressores de tradução, enquanto que a via da MAPK fosforila directamente eIF4E 6,11. Por sua vez, quinases eIF2α (ie PERK, PKR, HRI e GCN2) inibem eIF2 por fosforilação sua subunidade reguladora eIF2α em resposta à privação de nutrientes, ER-stress e infecção pelo vírus 6,12. Alterações na atividade e / ou expressão de TFs, outros componentes da maquinaria de tradução e fatores regulatórios, incluindo miRNAs têm sido observadas em vários estados patológicos, incluindo o câncer, síndromes metabólicas, distúrbios neurológicos e psiquiátricos e doenças renais e cardiovasculares 13-19. Coletivamente, estes dados indicam que me Translationalchanisms desempenham um papel fundamental na manutenção da homeostase celular e que a sua anulação desempenha um papel central na etiologia de diversas doenças humanas.
Nisto, um protocolo para o fraccionamento de polissomas por centrifugação em gradiente de densidade de sacarose em células de mamíferos, o qual é usado para separar a partir de polissomas monosomes, subunidades ribossomais e partículas mensageiro ribonucleoproteínas (mRNPs) é descrita. Isso permite discriminação entre traduzido de forma eficiente (associado com polissomos pesados) de mal traduzidas (associado com polissomos luz) mRNAs. Neste ensaio, os ribossomas são imobilizadas sobre a tradução do mRNA usando inibidores de alongamento, tais como ciclo-heximida 20 e extractos citosólicos são separados em 5-50% de sacarose linear gradientes de densidade por ultracentrifugação. Fraccionamento posterior dos gradientes de sacarose permite o isolamento de ARNm de acordo com o número de ribossomas que se ligam. ARN extraído de cada fracção pode ser então utilizada para determinarmudanças nas distribuições de ARNm em todo o gradiente entre diferentes condições, pelo que aumenta a eficiência da tradução a partir do topo para o fundo do gradiente. Northern Blot ou transcrição reversa da polimerase de reacção em cadeia quantitativa (qRT-PCR), são utilizados para determinar os níveis de ARNm em cada fracção.
Alternativamente, fracções contendo polissomos pesados (normalmente mais de 3 ribossomos) são reunidos e os níveis de mRNA-associados polissomas do genoma são determinados utilizando microarray ou deep-seqüenciamento. É de extrema importância ressaltar que os níveis de mRNAs associados-polissomas são, além de tradução, afetado por mecanismos de transcrição e pós-transcricional que influenciam os níveis de mRNAs citossólicas 21. Portanto, para determinar diferenças na tradução usando dados do genoma de mRNA-associados polysome é necessário para corrigir os efeitos de passos na expressão gênica caminho que estão a montante da traduçãoção 21. Para permitir que uma tal correcção, ARN citosólica é preparada em paralelo com o RNA-associados polissoma a partir de cada amostra e os níveis de mRNA de estado estacionário de todo o genoma são determinadas 21. Atualmente, a chamada "tradução-eficiência" (TE) pontos (ou seja, o log-ratio entre os dados de mRNA-associados polissomas e dados de mRNA citosólicas) são frequentemente utilizados para corrigir os efeitos de mudanças nos níveis de mRNA citosólicas na eficiência da tradução de um dado mRNA 22. No entanto, usando pontuação TE identificar tradução diferencial está associada a um número substancial de resultados negativos falsos positivos e falsos devido a uma propriedade aritmética das pontuações TE comumente referido como correlação espúria 27. Na verdade uma pesquisa de conjuntos de dados de vários laboratórios indicaram que tais correlações espúrias parece ser inevitável quando se analisa mudanças nas translatomes 23. Isso levou o desenvolvimento da "análise de diferencial tr anslation "(anota) algoritmo, que não sofre das limitações acima mencionadas 23. Durante anota-análise de um modelo de regressão é usada para derivar as medidas de atividade de translação independentes de níveis de RNA citosólicas. Tais medidas são então comparadas entre as condições e uma estatística é calculada. O usuário tem a opção de aplicar um método de encolhimento variância, o que melhora o poder estatístico e reduz a ocorrência de resultados falso-positivos em estudos com poucas repetições 24. Significativamente, foi recentemente demonstrado que as perturbações no translatome capturado por anota, mas os escores não TE, correlacionar com as mudanças no proteoma 25. Portanto, é altamente recomendável aplicar a análise anota para identificação de alterações na tradução em uma escala de todo o genoma. Embora os fundamentos teóricos do algoritmo anota foram discutidos em detalhe anteriormente 21,23,26, aqui o foco está em como aplicá-la na prática.
ve_content "> Além de estudar alterações na actividade de tradução de ARNm na célula, este protocolo de fraccionamento ribossoma pode ser usado para isolar e caracterizar bioquimicamente e funcionalmente ribossoma-e complexos de proteína associada à polissomas. Esta abordagem foi implementada com sucesso no passado para identificar complexos que regulam a estabilidade de polipéptidos sintetizados de novo 27 e / ou estão envolvidas na fosforilação de componentes da maquinaria de tradução. Esta aplicação do método de fraccionamento polissoma também irá ser discutido brevemente.Protocolo
1. Sacarose Gradiente Preparação
- Preparar 100 ml de 60% (w / v) de uma solução de sacarose em ddH 2 O. A solução deve ser filtrada através de um filtro de 0,22 mM para evitar o entupimento do tubo, durante o passo de fraccionamento (passo 3.5).
- Prepare 5 ml de 10x tampão de gradiente de sacarose: HEPES 200 mM (pH 7,6), 1 M de KCl, 50 mM de MgCl2, 100 ug / ml de cicloheximida, 1x cocktail inibidor de protease (EDTA livre), 100 unidades / ml de inibidor de RNase.
- Usando a solução a 60% preparada como descrito no passo 1.1, fazer 40 ml de 5% e 50% de soluções de sacarose em tampão de gradiente de sacarose 1x.
- Use o bloco marcador fornecido com o fabricante de gradiente para marcar o ponto pela metade em cada tubo de ultracentrifugação polialómero para camadas (6 tubos de ultracentrifugação no total). Utilizando o dispositivo de camadas fornecida com o formador de gradiente, adicionar solução de sacarose a 5%, até atingir o ponto de meio-cheio. Em seguida, adicione solução de sacarose 50% a partir do fundo até que o interface entre as duas soluções atinge o ponto de meio-cheio. Um cuidado especial deve ser tomado durante este passo para que os gradientes são tão semelhantes quanto possível, o que é fundamental para a obtenção de alta reprodutibilidade (ver abaixo).
- Selar tubos com taxa tampas zonais fornecidos com o fabricante gradiente e transferir os tubos selados para o titular do tubo sobre o fabricante de gradiente.
- Executar o fabricante de gradiente para obter um gradiente linear de 5% a 50%. 6 gradientes lineares será formada em poucos minutos de rotação no ângulo de inclinação elevada.
Nota: gradientes de sacarose pode ser utilizada imediatamente ou armazenada a -80 ° C durante 6 meses.
2. Isolamento e sedimentação de polissomas
- Dependendo do tipo de célula, propagar células em 1-5 15 cm placas de Petri (15 x 10 ~ 6 células), pelo menos, um dia antes da lise. No dia das experiências as células devem ser ~ 80% confluentes. Nota: confluência ideal é crítico, porque as taxas de conversão e de proliferação são diretamenteproporcionais 8, e, portanto, devem ser analisados polissomos em células em proliferação ativa (ou seja, as taxas de conversão vai cair significativamente em mais células confluentes enquanto baixo confluência de células não pode fornecer material suficiente para uma análise mais aprofundada). Excepções a esta regra pode ser feito se, por exemplo, o pesquisador está interessado em estudar o efeito de inibição de contato em tradução. No entanto, as células não devem ser mantidos sob condições mais confluentes por muito tempo, pois isso pode ter efeitos involuntários no translatome.
Se é necessária a transfecção, a maior parte dos kits disponíveis comercialmente não afectam os níveis de polissomas. Porque no dia da transfecção as células devem ser de 80-90% confluentes, recomenda-se propagar células 24 h pós-transfecção e isolar polissomas 48 horas pós-transfecção de células de ~ 80% confluentes. - Incubar as células com ciclo-heximida, a uma concentração final de 100 ug / ml em meio de crescimento de 5 min a 37 ° C e 5% De CO 2; e lavar as células duas vezes com 10 ml de gelo-frio 1x PBS contendo 100 ug / ml de cicloheximida. Cicloheximida é um inibidor da tradução alongamento que "congela" os ribossomos no mRNA, impedindo assim ribossomo correr 20.
- Raspar as células suavemente, em 5 ml de gelo-frio 1x PBS contendo 100 ug / ml de cicloheximida e recolhê-las em um tubo de 50 ml. É importante que esta etapa é realizada razoavelmente rápido deixando as células sobre gelo durante um período prolongado de tempo vai diminuir drasticamente a qualidade da preparação de polissoma.
- Recolher as células por centrifugação a 200 xg durante 5 min a 4 ° C.
- Rejeitar sobrenadante e ressuspender as células em 425 ul de tampão hipotónico [(5 mM Tris-HCl (pH 7,5), MgCl2 2,5 mM, KCl 1,5 mM e 1x cocktail inibidor de protease (EDTA livre)], adicionam-se 5 ul de 10 mg / CHX ml, 1 ul de DTT 1 M, 100 unidades de inibidor da ARNase e vortex durante 5 segundos, seguido por adição de 25 ul de 10% Triton X-100 (concentração final 0,5%) e 25 ul de 10% de desoxicolato de sódio (concentração final 0,5%) e agitar em vortex durante 5 segundos. Devido à expansão da célula, o tampão hipotónico irá romper a membrana plasmática, enquanto que as condições de detergentes suaves são usados para solubilizar citosólica e ribossomas associados endoplasmático-retículo sem interromper o envelope nuclear.
- Lisados de centrifugar a 16.000 x g durante 7 minutos a 4 ° C e a transferência de sobrenadante (~ 500 mL) para um novo tubo de pré-arrefecida de 1,5 ml. Medir OD a 260 nm para cada amostra utilizando um espectrofotómetro e manter 10% do lisado como entrada que vai ser utilizado para determinar os níveis de mRNA citosólicos de estado estacionário. Diluir a entrada para 750 mL de RNAse livre H 2 O, adicionar 750 mL de Trizol e congelamento de flash em nitrogênio líquido.
- Transferir os tubos ultracentrífuga contendo gradientes de sacarose em baldes de rotor pré-refrigerados. Remover 500 ul a partir do topo de gradientes de sacarose. Ajustar lisados de modo que contenham o mesmo OD (10-20 OD a 260 nm) em 500 ul de tampão de lise (descrito no passo 2.5) e carregá-los em cada gradiente de sacarose.
Nota: É importante a carga imediatamente lisados nos gradientes, pois isso irá melhorar de forma crítica a qualidade das preparações de polissomas. - Pesar e equilibrar cada gradiente antes da ultra-centrifugação.
- Centrifugar a 222.228 x g (36.000 rpm), durante 2 horas a 4 ° C utilizando SW41Ti rotor.
Nota: A fim de não perturbar gradientes de sacarose, a opção de freio "baixa" deve ser selecionada.
3. Polissoma Fracionamento e Extração de RNA
- Prepare o coletor de fração por limpá-lo com água quente RNAse livre contendo RNase ZAP (alguns sprays). Repita este passo com RNAse livre apenas água morna. Interruptor da luz UV e esperar a luz verde para aparecer.
- Cuidadosamente remover tubos do rotor e colocá-los em gelo. Ligue computador, bomba, detector UV e coletor de fração. Definir bomba em 3 ml / min e fill do tubo com a solução de perseguição [60% (w / v) de sacarose contendo 0,02% (w / v) de azul de bromofenol] até que ele atinja a agulha. Certifique-se de ver, pelo menos, uma gota que sai da agulha, e verificar que há bolhas são introduzidas na seringa bomba ou tubulação.
- Coloque 2 ml tubos em um coletor de fração. Lançamento de programa de análise e definir a sensibilidade de + / - 10 MeV. Defina "base de tempo" para 100 seg.
- Posicione cada tubo de ultracentrifugação para o detector UV, enquanto certificando-se de que o tubo está na posição ortogonal. Furar o tubo com a agulha rodando o botão abaixo titular do tubo.
- Definir a bomba de 1,5 ml / minuto, e recolher as fracções de ajuste do tempo de 30 seg sobre o colector de fracções (isto irá resultar em ~ 750 uL de cada fracção). Coloque a bomba na posição "remota", inicie o coletor bomba e fração, e ao mesmo tempo iniciar a gravação usando o traçador DAQ. Isto irá iniciar um deslocamento para cima da gra sacarosediente e uma detecção simultânea de absorção de UV a 254 nm. Pare recolha assim que a primeira gota de solução de perseguição cai em um tubo de recolha de 2 ml.
- Salve o rastreamento no formato CSV e (uma vez passo 3.7 está concluída), em um aplicativo de planilha, faça o seguinte para identificar o posicionamento de cada fração no perfil de absorção UV.:
- Escolha da coluna contendo os valores correspondentes para a absorvância a 254 nm (canal 0).
- Criar um gráfico de linha de dispersão suave das absorbâncias.
- Definir a unidade principal do eixo X para 300 (o valor obtido dividindo a quantidade de 5% -50% de gradiente de sacarose (~ 12 ml) pelo tempo de recolha de cada fracção (30 seg).
- Adicionar 750 mL de Trizol em cada fração e flash congelar as frações em nitrogênio líquido.
- Isolar RNA associada à polysome e citosólica (de 2,6) usando o protocolo Trizol acordo com as instruções do fabricante. Para aumentar a produção de RNA, prossiga com uma precisão de RNApitation a -80 ° C durante 30 min ou a -20 ° C durante a noite.
Observação: Uma vez que a quantidade de ARN precipitado a partir de fracções polissomal não pode ser claramente visível é recomendável adicionar transportadora. Adicionar 1 ml de transportador para o tubo antes do passo de precipitação do RNA durante o protocolo de Trizol. - Determine quais frações correspondem a mRNA associado com> 3 ribossomo (usando abordagem descrita 3.6) e juntar essas frações. Use o kit de limpeza de RNA para executar a limpeza de RNA reunidos e citosólica associada à polysome (de 2,6). Enviar amostras para uma instalação para determinar os níveis de mRNA do genoma usando microarrays ou deep-seqüenciamento.
Se a tradução de mRNAs específicos devem ser monitorizados por RT-qPCR, recomenda-se a utilização de 500 ng de ARN (a partir das fracções reunidas contendo> 3 ribossomas ou de ARN total) para sintetizar cDNA.
Nota: mRNAs associados com> 3 ribossomas está definido arbitrariamente para representar ARNm traduzido de forma eficiente e contêm mais de 80% dos recém-sintetizadaspolipeptídeos de tamanho 28,29. Incluindo as fracções correspondentes à luz (1-2), meio (2-3) e polissomas pesados (> 3), seria vantajoso, mas isso irá aumentar o número das amostras de 2 vezes (4 vs 2 por condição) e, assim, o custo do experimento. Não obstante que prevê-se que a diminuição do custo de deep-seqüenciamento e análise de microarray, no futuro, deve favorecer a última abordagem, é aconselhável que durante a validação, distribuição de mRNAs individuais é monitorado em cada fração.
4. Genome-wide Análise do ARNm Tradução
- Instale R, bioconductor eo pacote anota:
- Instale R (download de r-project.org) e iniciar uma sessão R.
Nota: R está disponível para todos os sistemas operacionais e anota será executado em todos eles. - Instale bioconductor (bioconductor.org). R código é interpretado pelo (por exemplo, o console do terminal R-R no Windows - que é lançado por fazeruble clicando no atalho R). Bioconductor está instalado digitando (no terminal-R):
fonte ("http://bioconductor.org/biocLite.R")
biocLite () - Instale o pacote bioconductor anota (eo pacote qvalue que anota requer) digitando (no terminal R-):
fonte ("http://bioconductor.org/biocLite.R")
biocLite (c ("anota", "qvalue")) - Teste se o pacote foi instalado e abrir o manual anota digitando (no terminal R-) com sucesso:
biblioteca ("anota")
vinheta ("anota") - Nota: A ajuda adicional para todas as funções que são usados no pacote anota podem ser encontrados tanto na página da web anota (http://www.bioconductor.org/packages/release/bioc/html/anota.html) ou usando a função de ajuda dentro de R. Por exemplo, para obter ajuda sobre a função anotaPerformQc ir para o R-terminal e tipo (note que isso só funciona quando o pacote anota foi carregado):
biblioteca ("anota") # carrega o pacote anota
? AnotaPerformQc # abre a ajuda para esta função
- Instale R (download de r-project.org) e iniciar uma sessão R.
- Importar dados para R:
- Preparar os dados de expressão para a análise. Os dados devem ser pré-processados (por exemplo, normalizada e qualidade controlada) no que diz respeito à técnica que foi usada para medir os níveis de mRNA. Valores de entrada deve ser log transformado, o mais comum é log2. Compilar uma tabela com dados de todas as amostras de RNA associada à polissomas e um para todas as amostras de RNA citosólicas. A primeira coluna deve conter os-identificadores de genes seguido por uma coluna de dados por amostra; a primeira linha deve incluir os nomes das amostras. É fundamental que as tabelas têm a ordem da amostra ea ordem idêntica gene idêntico. Salve os arquivos como arquivos de texto delimitados por tabulação chamados "myPolysomeData.txt" e "myCytosolicData.txt".
- Neste exemplo, duas classes de amostra são utilizados (de controlo ou doente), com 3 repetições por classe, gerando, assim, seis amostras com este fim: C1, C2, C3, D1, D2, D3 em ambos os ficheiros myCytosolicData.txt e myPolysomeData.txt. Anota requer pelo menos três réplicas por condição, quando existem duas classes de amostra. Estes devem ser repetições biológicas independentes.
- Crie um diretório que contém os arquivos de entrada de dados (myPolysomeData.txt e myCytosolicData.txt). Abrir R desse diretório. No Windows / Mac isso pode ser feito copiando um atalho-R para este diretório e lançamento de R usando este atalho (o trabalho direto também pode ser alterado usando os menus drop-down).
- Crie um arquivo chamado myCode.R dentro do diretório recém-criado e abri-lo usando um software de edição de texto. Escreva o código neste arquivo.
- Para carregar os dados em R escrever o seguinte código para o arquivo myCode.R (lembre-se de voltar a salvar o arquivo depois de cada adição de código). Abaixo está um s passo-a-tep explicação do código, mas todo o código também pode ser escrito inicialmente ea função da fonte (que executa o código, veja abaixo) usado apenas uma vez:
# # Este carrega a biblioteca anota
biblioteca (anota)
# # Este carrega dados de entrada em R
dataCyto <- as.matrix (read.table ("myCytosolicData.txt", header = TRUE, row.names = 1, sep = " t"))
dataPoly <- as.matrix (read.table ("myPolysomeData.txt", header = TRUE, row.names = 1, sep = " t")) - Execute o código em R digitando diretamente no terminal R:
fonte ("myCode.R") - Verifique se os dados foram carregados com sucesso digitando (no terminal R-):
cabeça (dataCyto) # mostrará topo da tabela de dataCyto
cabeça (dataPoly)
- Identificação de genes diferencialmente traduzidos:
- Gerar um vetor que descreveas categorias de amostra (um vetor é um tipo de objeto em R). Este vetor deve refletir a ordem da amostra no myPolysomeData.txt e myCytosolicData.txt para que todos os três objetos têm uma ordem idêntica das amostras. O vector vai ser utilizado quando anota instruindo sobre o qual as amostras de comparação. Adicione o seguinte ao arquivo myCode.R:
# # Note que as amostras idênticas têm aula de amostra idêntico
# # Descrições (ou seja, "C" ou "D") neste vetor.
myPhenotypes <- c ("C", "C", "C", "D", "D", "D") - Realizar o controle do conjunto de dados de qualidade. Há um certo número de medidas de qualidade que necessitam de ser avaliada antes anota pode ser utilizado para a análise. Estes são discutidos em detalhe no manual anota. Adicione este código ao myCode.R arquivo e salvar:
anotaQcOut <- anotaPerformQc (datat = dataCyto, dataP = dataPoly,phenoVec = myPhenotypes)
anotaResidOut <- anotaResidOutlierTest (anotaQcObj = anotaQcOut) - Execute o código, digitando em R-terminal:
fonte ("myCode.R")
Isso irá gerar um número de arquivos de saída, que pode ser examinado, conforme as instruções no manual anota. - Identificar genes diferencialmente traduzidos. As amostras serão comparadas com base na ordem alfabética dos nomes fornecidos para o parâmetro "phenoVec" (ou seja, o "myPhenotypes" vector) a menos que eles são especificados pelo usuário (usando o "contrastes" parâmetro). Adicione o seguinte código para myCode.R arquivo e acabamento, usando o comando "fonte" dentro do descrito acima do terminal R-:
anotaSigOut <- anotaGetSigGenes (datat = dataCyto, dataP = dataPoly, phenoVec = myPhenotypes, anotaQcObj = anotaQcOut) - Use a ajuda para anotaGetSigGenes para entender como o output está formatado e veja como escolher categorias de amostra para comparar digitando (no terminal R-):
? anotaGetSigGenes - Filtrar e enredo genes que são diferencialmente traduzidos. Existem vários limiares que podem ser aplicadas dentro da função anotaPlotSigGenes e usando a ajuda irá simplificar uma combinação personalizada de tais limites. Para selecionar os genes analisados de forma confiável aplicar o minSlope (-0,5), maxSlope (1,5) e (0,01) slopeP configurações.
Nota: Além disso, as configurações que se aplicam a RVM 23,26,30 taxa de falsa descoberta (FDR) (0,15) e prega mudanças (log2 [1.5]) pode por exemplo ser usado para identificar genes diferencialmente traduzidos. Outros filtros tais como "selDeltaPT" pode ser útil para uma filtragem mais rigorosas (por exemplo, criando a selDeltaPT log2 [1,5]). É também necessário para especificar qual a comparação deve ser filtrada (utilizando o argumento selCont). Aplicar as configurações descritas, adicionando a seguinte linha ao myCode.Rarquivo:
anotaSigFiltered <- anotaPlotSigGenes (anotaSigObj = anotaSigOut, selContr = 1, minSlope = (-0,5), maxSlope = 1,5, slopeP = 0,01, maxRvmPAdj = 0,15, minEff = log2 (1,5), selDeltaPT = log2 (1,5)) - Realize a análise usando a função de origem da como descrito acima do terminal R-. Isso também vai gerar uma saída gráfica. Examinando essa saída é um bom ponto de partida para avaliar o desempenho da análise e identificar as alterações necessárias nas configurações.
- Gerar uma tabela de saída. Adicione o seguinte código para o arquivo myCode.R:
write.table (anotaSigFiltered $ selectedRvmData, file = "MySignificantGenes.txt", sep = " t") - Execute o código usando a função de fonte no terminal R. As colunas da tabela resultante são explicados na ajuda para a função anotaPlotSigGenes.
- Gerar um vetor que descreveas categorias de amostra (um vetor é um tipo de objeto em R). Este vetor deve refletir a ordem da amostra no myPolysomeData.txt e myCytosolicData.txt para que todos os três objetos têm uma ordem idêntica das amostras. O vector vai ser utilizado quando anota instruindo sobre o qual as amostras de comparação. Adicione o seguinte ao arquivo myCode.R:
Resultados
mTOR é um grande nó da rede celular, que coordena as taxas de síntese de proteínas globais com a disponibilidade de nutrientes 19. tradução do mRNA é regulada principalmente no início passo limitante da taxa de 6. A proporção de ribossomos envolvidos em polissomos correlaciona-se positivamente com as taxas de iniciação da tradução 28. Um exemplo de aplicação do método de polissoma fraccionamento para investigar o papel da sinalização de mTOR na mediação dos efeitos da insulina na tradução do mRNA é apresentado. Para este fim, células de cancro da mama humano MCF-7 foram mantidas em baixo soro e em seguida estimuladas com insulina por si só ou em combinação com o local activo inibidor de mTOR Torin1. As células não-estimuladas que foram continuamente mantidas em baixo soro, serviu como um controlo. mRNPs, monosome (80S) e frações polissomas foram separados utilizando o método polysome fracionamento. Em relação às células de controlo, a insulina provocou um aumento na absorvância em fracções de gradiente correspondentesde polissomas, acompanhada por uma diminuição concomitante na absorvância na fracção monosome (Figura 1). Estes resultados mostram que a proporção de ribossomas envolvidas em polissomas é aumentada em tratados com insulina, em comparação com células de controlo, por conseguinte, indicando que, tal como esperado, a insulina estimula as taxas globais de iniciação da tradução. Torin1 reverteu os efeitos da insulina sobre os perfis de absorvância (Figura 1), corroborando os resultados de que a sinalização de mTOR desempenha um papel importante na mediação dos efeitos da insulina sobre a maquinaria de tradução 19.
Com base num princípio que as inconsistências na preparação gradiente não pode ser evitada, foram levantadas questões sobre a reprodutibilidade dos dados obtidos usando o método de polissoma fraccionamento 22. Para testar empiricamente esta questão potencialmente deletéria, citosólica e RNA associada à polysome pesado (mRNA associado com 4 e mais ribossomos) foram isolados a partir de células MCF-7 tratadas com insulina por si só ou em combinação com insulina Torin1 de 4 réplicas biológicas independentes (Figura 2). Os efeitos da insulina e Torin1 sobre a composição da citosólica e pesado mRNA associado-polysome em cada repetição foi determinada em uma escala do genoma utilizando uma abordagem de microarray. Para determinar a reprodutibilidade do método polysome fracionamento da análise de componentes principais (PCA) foi aplicada. Tal análise revelou que as amostras referentes a cada condição foram firmemente posicionados dentro dos primeiros dois componentes, enquanto as diferentes condições foram bem separadas (Figura 2). Estes resultados mostram que o método polysome fracionamento, conforme descrito aqui é altamente reprodutível e, portanto, adequado para estudar mudanças quantitativas e qualitativas na tradução a nível de todo o genoma.
ad/51455/51455fig1highres.jpg "src =" / files/ftp_upload/51455/51455fig1.jpg "/>
Perfis Figura 1. Polissomal que mostram os efeitos de privação de soro, insulina e mTOR sinalização na conversão global em células MCF-7. Células MCF7 foram privadas de nutrientes (mantidas em 0,1% de FBS) durante 16 horas e tratou-se com 5 nM de insulina (Ins) sozinho ou em combinação com 250 nM Torin1 (+ Ins Torin1) durante 4 horas. Células não tratadas que foram privadas de nutrientes continuamente (0,1% de FBS) foram utilizadas como um controlo. Os correspondentes extractos citossólicos foram sedimentadas por centrifugação em gradientes de 5-50% de sacarose. Subunidades ribossomais Grátis (40S e 60S), monosomes (80S) e número de ribossomos nas frações polissomas são indicados.
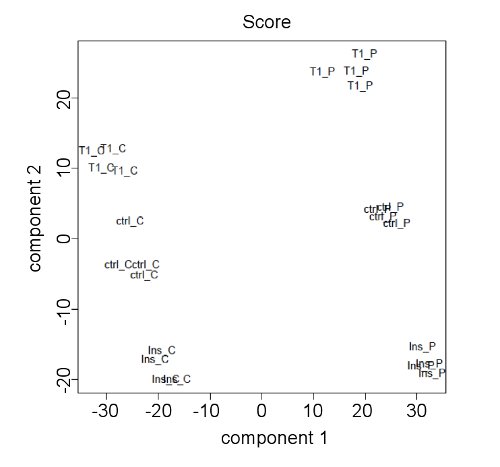
Figura 2. Dados à escala do genoma obtidos utilizando-se polysome perfilamento é altamente reprodutível. células MCF-7 foram tratados como na Figura 1. citosólico e RNA associadas a polissoma (> 3 ribossomas) foi extraído a partir de 4 repetições biológicos independentes e os seus níveis de ARNm de todo o genoma foram determinados utilizando matrizes GeneTitan (Affymetrix). APC foi usado para avaliar a reprodutibilidade dos dados resultantes. São mostrados os dois primeiros componentes da PCA para todos os tratamentos (T1 = Torin 1; ctrl = controle; Ins = insulina), origem de RNA (C = P = citosólica; associada à polysome) e replica. As amostras de mesma condição e origem RNA estão intimamente posicionado indicando alta reprodutibilidade.
Discussão
Este artigo descreve um protocolo polysome fracionamento bem estabelecido seguido por um método de análise desenvolvido in-house para a captura de alterações qualitativas e quantitativas na atividade de translação em uma escala do genoma em células de mamíferos. Para a conclusão deste protocolo especial atenção deve ser dada a 1) confluência celular (como as taxas de proliferação e disponibilidade de nutrientes correlacionam com a atividade da tradução do mRNA e pode afetar a composição da translatome, confluência celular deve ser consistente em repetições e condições experimentais), 2) lise celular rápida (Não obstante a presença de cicloheximida e inibidores de RNAse em tampões, lise deve ser rápida e extractos celulares devem ser sobrepostos em gradientes de sacarose imediatamente após a lise para evitar a degradação do RNA e dissociação de polissomas), 3) a preparação de gradiente (para assegurar alta reprodutibilidade de dados, gradientes de sacarose deve ser preparada com a fabricante de gradiente e SPEcial cuidado deve ser tomado ao manuseá-los).
Além de estudar as alterações na actividade de translação, este protocolo pode ser utilizado para isolar os complexos de proteína associada ao ribossoma e estabelecer as suas funções fisiológicas. Para este fim, e os níveis de estado de fosforilação de várias proteínas associadas ao ribossoma pode ser determinada por precipitação com TCA, seguido por transferência de Western, enquanto que os complexos de proteína de ribossoma-associado pode ser imunoprecipitada a partir de fracções do gradiente e analisadas por transferência de Western ou de espectrometria de massa. A desvantagem desta técnica é que o método de fração inclinação lenta pode resultar em dissociação de proteínas ainda fortemente ligadas cuja cinética de ligação estão em rápida associação / dissociação. Factores associados a polipéptidos sintetizados de novo são exemplos típicos. Polipéptidos sintetizados de novo emergentes formar os ribossomas podem ser imobilizadas em complexos de proteínas associados a ribossomas utilizando reticuladores químicos, tais como 3, 3'-ditiobis [sulfosuccinimidylpropionate] (DTSSP) 31. Esta abordagem revelou que o receptor para a Quinase C activada 1 (RACK1) / c-Jun, cinase N-terminal (JNK) / factor de tradução eucariótica alongamento 1A2 complexo (eEF1A2) regula a degradação de polipéptidos sintetizados de novo, em resposta ao stress, 27. Além disso, a metodologia semelhante foi utilizada em estudos que mostram que a proteína quinase C bII (PKCbII) é recrutado para os ribossomas por RACK1 32 e que a activação do complexo mTOR 2 (mTORC2) ocorre sobre os ribossomas, onde fosforila AKT polipéptidos sintetizados de novo e regula a sua estabilidade 33,34.
As principais limitações do método de polissoma fraccionamento seguido por análise anota são: 1) necessidade de um número relativamente alto de células (15 x 10 ~ 6 células), 2) a falta de informação sobre a localização posicionai de ribossoma de uma determinada molécula de ARNm, e 3 ) Questões relacionadas com a hetero celular e moleculargeneidade de tecidos normais e tumorais. Questões relacionadas com o número de células necessário pode ser resolvido através do alinhamento picos dos espectros de absorvência correspondentes aos monosomes (80S) de células cujas quantidades são limitantes com os obtidos a partir de células de elevada abundância (por exemplo, células HeLa) 35. Técnica de perfilamento ribossoma (ver abaixo) pode ser utilizado para determinar a posição exacta do ribossoma de uma determinada molécula de ARNm, enquanto que os problemas relacionados com efeitos de confusão de heterogeneidade do tecido sobre a interpretação de alterações na expressão de genes obtidos a partir de sistemas complexos, tais como os tecidos humanos são discutidos em detalhes em uma publicação por Leek e Storey 36.
Recentemente, um novo ribossomo profiling técnica foi desenvolvida, na qual fragmentos ribossomo protegido (RPFS) são gerados por RNAse I tratamento e analisadas por deep-seqüenciamento 22. Esta técnica permite a determinação da posição do ribossomo em um único nucleotídeo resolução, proporcionando assim até então UNPRpercepções ecedented em biologia ribossomo. Por exemplo, o perfil do ribossoma pode ser utilizado para a determinação da densidade de ribossoma em uma determinada molécula de ARNm, ou a identificação dos elementos que as taxas de iniciação de tradução, tais como a influência dos sítios de iniciação alternativos, nos codões de iniciação não agosto e elementos reguladores, tais como uORFs. No entanto, existem várias limitações metodológicas que restringem a capacidade de criação de perfis ribossomo para estimar com precisão a eficiência da tradução do mRNA. Estes incluem preconceitos independentes introduzidas pela fragmentação aleatória e RNAse eu digestão, preconceitos introduzidas por inibidores de tradução (por exemplo, inibidores de alongamento, como emetine e cicloheximida são susceptíveis de induzir o acúmulo ribossomo em locais de iniciação de tradução), um grande número de resultados falso-positivos e falso-negativos associados com pontuação TE, bem como a sua imprecisão na previsão das leituras que se originam a partir de mRNAs de codificação de proteínas 37. Talvez o mais importante, ao passo queperfilamento ribossoma permite a identificação directa de posição ribossoma numa dada molécula de mRNA, o número de ribossomas que estão associados com um determinado mRNA é indirectamente estimada, normalizando as frequências de leituras em RPFS (mRNA associada ao ribossoma) sobre os observados em mRNAs fragmentadas aleatoriamente (total ARNm). Por exemplo, em um ambiente simples, onde quatro moléculas de mRNA "B" (BA ou BB, BC e BD) são ocupados por quatro ribossomos nas posições 1, 2, 3 e 4, uma falha inerente à técnica de perfis ribossomo não permitirá uma distinção entre um cenário onde todos os 4 ribossomos associar apenas com um mRNA Ba nas posições 1, 2, 3 e 4 e um cenário onde Ba, Bb, Bc e Bd mRNA são cada ocupada por um único ribossomo na posição 1, 2 , 3, e 4, respectivamente. Em contraste, durante o fraccionamento polissoma, polissoma integridade é preservada, permitindo assim o isolamento de grupos de ARNm associado a um número definido de ribossomas (Figura 1). Esta importaçãodistinção entre formiga fraccionamento polissoma e perfilamento ribossoma sugere que, enquanto o primeiro método pode ser utilizado para comparar directamente mRNAs mRNP, leves e pesados polissomas fracções, o último método provavelmente não conseguem capturar mudanças na translatome que são causadas por mRNAs que transição acender a polissomos pesados e superestima a contribuição daqueles que mudam a partir da fração mRNP para polissomos pesados. O significado biológico destes discrepâncias entre os métodos acima referidos é sublinhada por um grande corpo de dados que mostram que a activação da tradução de um subconjunto dos ARNm, tais como aqueles que são sensíveis ao eIF4E, a transição de luz para polissomas pesadas, ao passo que outros, tais como aqueles que alberga elementos 5'TOP, são recrutados para polissomos pesados diretamente de poças de mRNA sem ribossomo 6. Curiosamente, a mTOR tem sido mostrado para modular concomitantemente tradução de "eIF4E-sensíveis" e 5'TOP mRNAs 11. Portanto, diferenças metodológicas que são discutidos acima pode explicar a aparente discordância da conclusão entre estudos utilizando ribossomo perfis 38,39 e polysome fracionamento 24 para avaliar os efeitos da inibição do mTOR no translatome.
Em conclusão, o fracionamento polysome e profiling ribossomo são métodos complementares que fornecem principalmente informações sobre o número de ribossomos associados ao mRNA e posição do ribossomo em mRNA, respectivamente. É importante ressaltar que apesar das deficiências e vantagens destes métodos, é essencial que os dados do genoma obtidos por ambos os procedimentos são adequadamente analisados e funcionalmente e bioquimicamente validado.
Divulgações
Os autores declaram não haver interesses financeiros concorrentes.
Agradecimentos
Esta pesquisa foi apoiada pelos Institutos Canadenses de Pesquisa em Saúde (concessão CIHR MOP-115195) e FRQ-S a ele, que também é um receptor de CIHR Young Investigator Award; e do Conselho de Pesquisa sueco eo sueco Cancer Society para OL
Materiais
| Name | Company | Catalog Number | Comments |
| HEPES | Biobasic | HB0264 | |
| MgCl 2 | Sigma | M8266 | |
| KCl | VWR | CABDH4532 | |
| Dithiothreitol (DTT) | Biobasic | DB0058 | |
| Tris | Biobasic | TB0196 | |
| Glycoblue | Ambion | AM9515 | RNA precipitation carrier |
| Sodium Deoxycholate | Sigma | D6750 | |
| Triton X-100 | Sigma | X100 | |
| Cycloheximide | Sigma | C1988 | |
| Protease inhibitor cocktail | Roche | 04693132001 | Tablets (EDTA-free) |
| RNase inhibitor | Promega | N2515 | |
| 0.45 µm Filter System 150 ml | Corning | 431155 | |
| Sucrose | Sigma | S0389 | |
| RNeasy MinElute Cleanup kit | Qiagen | 74204 | RNA cleanup kit |
| Thin wall polyallomer ultracentrifuge tubes | Beckman Coulter | 331372 | |
| Equipments | |||
| SW41Ti Rotor Package with accessories | Beckman Coulter | 331336 | |
| Optima L100 XP ultra centrifuge | Beckman Coulter | DS-9340A | |
| ISCO fraction collector | Brandel | FC-176-R1 | |
| Type II Detector with Chart Recorder | Brandel | UA-6 | UV detector |
| Tube Piercer w/Flow Cell and Syringe Pump | Brandel | BR-A86-5 | |
| UA-6 Detector Cable | Brandel | 60-1020-211 | |
| FC-176 Communication Cable | Brandel | FCC-176 | |
| Gradient Master Base Unit | Biocomp | 108-1 | Gradient Maker |
| Gradient Forming Attachments | Biocomp | 105-914A-1R | Gradient Maker attachment |
| Measurement Computing 8-channel 50kHz Data Acquisition Device | MicroDAQ | USB-1208FS | Analysis software attachment |
| Measurement Computing Data Acquisition Software | MicroDAQ | TracerDAQ Pro | Analysis software (Download only) |
| Buffers | |||
| 10X buffer for sucrose gradients: | |||
| 200 mM HEPES (pH7.6) | |||
| 1 M KCl | |||
| 50 mM MgCl2 | |||
| 100 µg/ml cycloheximide | |||
| 1x protease inhibitor cocktail (EDTA-free) | |||
| 100 units/ml RNase inhibitor | |||
| Lysis buffer | |||
| 5 mM Tris-HCl (pH7.5) | |||
| 2.5 mM MgCl2 | |||
| 1.5 mM KCl | |||
| 1x protease inhibitor cocktail (EDTA-free)] | |||
| Then add cycloheximide (CHX), DTT, RNAse inhibitor, Triton and Sodium Deoxycholate as indicated in the main text |
Referências
- Maier, T., Guell, M., Serrano, L. Correlation of mRNA and protein in complex biological samples. FEBS Lett. 583, 3966-3973 (2009).
- de Sousa Abreu, R., Penalva, L. O., Marcotte, E. M., Vogel, C. Global signatures of protein and mRNA expression levels. Mol Biosyst. 5, 1512-1526 (2009).
- Keene, J. D. RNA regulons: coordination of post-transcriptional events. Nat Rev Genet. 8, 533-543 (2007).
- Komili, S., Silver, P. A. Coupling and coordination in gene expression processes: a systems biology view. Nat Rev Genet. 9, 38-48 (2008).
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473, 337-342 (2011).
- Sonenberg, N., Hinnebusch, A. G. Regulation of translation initiation in eukaryotes: mechanisms and biological targets. Cell. 136, 731-745 (2009).
- Rolfe, D. F., Brown, G. C. Cellular energy utilization and molecular origin of standard metabolic rate in mammals. Physiol Rev. 77, 731-758 (1997).
- Johnson, L. F., Levis, R., Abelson, H. T., Green, H., Penman, S. Changes in RNA in relation to growth of the fibroblast. IV. Alterations in theproduction and processing of mRNA and rRNA in resting and growing cells. J Cell Biol. 71, 933-938 (1976).
- Fabian, M. R., Sonenberg, N., Filipowicz, W. Regulation of mRNA translation and stability by microRNAs. Annu Rev Biochem. 79, 351-379 (2010).
- De Benedetti, A., Graff, J. R. eIF-4E expression and its role in malignancies and metastases. Oncogene. 23, 3189-3199 (2004).
- Roux, P. P., Topisirovic, I. Regulation of mRNA translation by signaling pathways. Cold Spring Harb Perspect Biol. 4, (2012).
- Ron, D., Harding, H. P. Protein-folding homeostasis in the endoplasmic reticulum and nutritional regulation. Cold Spring Harb Perspect Biol. 4, (2012).
- Silvera, D., Formenti, S. C., Schneider, R. J. Translational control in cancer. Nat Rev Cancer. 10, 254-266 (2010).
- Proud, C. G. mTOR Signalling in Health and Disease. Biochem Soc Trans. 39, 431-436 (2011).
- Topisirovic, I., Sonenberg, N. mRNA Translation and Energy Metabolism in Cancer: The Role of the MAPK and mTORC1 Pathways. Cold Spring Harb Symp Quant Biol. , (2011).
- Pavitt, G. D., Proud, C. G. Protein synthesis and its control in neuronal cells with a focus on vanishing white matter disease. Biochem Soc Trans. 37, 1298-1310 (2009).
- Abe, M., Bonini, N. M. MicroRNAs and neurodegeneration: role and impact. Trends Cell Biol. 23, 30-36 (2013).
- Kasinath, B. S., et al. Regulation of mRNA translation in renal physiology and disease. Am J Physiol Renal Physiol. 297, 1153-1165 (2009).
- Zoncu, R., Efeyan, A., Sabatini, D. M. mTOR: from growth signal integration to cancer, diabetes and ageing. Nat Rev Mol Cell Biol. 12, 21-35 (2011).
- Schneider-Poetsch, T., et al. Inhibition of eukaryotic translation elongation by cycloheximide and lactimidomycin. Nat Chem Biol. 6, 209-217 (2010).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harb Perspect Biol. 5, (2013).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324, 218-223 (2009).
- Larsson, O., Sonenberg, N., Nadon, R. Identification of differential translation in genome wide studies. Proc Natl Acad Sci U S A. , (2010).
- Larsson, O., et al. Distinct perturbation of the translatome by the antidiabetic drug metformin. Proc Natl Acad Sci U S A. 109, 8977-8982 (2012).
- Colman, H., et al. Genome-wide analysis of host mRNA translation during hepatitis C virus infection. J Virol. 87, 6668-6677 (2013).
- Larsson, O., Sonenberg, N., Nadon, R. anota: Analysis of differential translation in genome-wide studies. Bioinformatics. 27, 1440-1441 (2011).
- Gandin, V., et al. Degradation of Newly Synthesized Polypeptides by Ribosome-Associated RACK1/c-Jun N-Terminal Kinase/Eukaryotic Elongation Factor 1A2. Complex. Mol Cell Biol. 33, 2510-2526 (2013).
- Warner, J. R., Knopf, P. M., Rich, A. A multiple ribosomal structure in protein synthesis. Proc Natl Acad Sci U S A. 49, 122-129 (1963).
- Gierer, A. Function of aggregated reticulocyte ribosomes in protein synthesis. J Mol Biol. 6, 148-157 (1963).
- Wright, G. W., Simon, R. M. A random variance model for detection of differential gene expression in small microarray experiments. Bioinformatics. 19, 2448-2455 (2003).
- Eggers, D. K., Welch, W. J., Hansen, W. J. Complexes between nascent polypeptides and their molecular chaperones in the cytosol of mammalian cells. Mol Biol Cell. 8, 1559-1573 (1997).
- Grosso, S., et al. PKCbetaII modulates translation independently from mTOR and through RACK1. Biochem J. 415, 77-85 (2008).
- Oh, W. J., et al. mTORC2 can associate with ribosomes to promote cotranslational phosphorylation and stability of nascent Akt polypeptide. EMBO J. 29, 3939-3951 (2010).
- Zinzalla, V., Stracka, D., Oppliger, W., Hall, M. N. Activation of mTORC2 by association with the ribosome. Cell. 144, 757-768 (2011).
- Bjur, E., et al. Distinct translational control in CD4+ T cell subsets. PLoS Genet. 9, e13 (2013).
- Leek, J. T., Storey, J. D. Capturing heterogeneity in gene expression studies by surrogate variable analysis. PLoS Genet. 3, 1724-1735 (2007).
- Guttman, M., Russell, P., Ingolia, N. T., Weissman, J. S., Lander, E. S. Ribosome Profiling Provides Evidence that Large Noncoding RNAs Do Not Encode Proteins. Cell. 154, 240-251 (2013).
- Hsieh, A. C., et al. The translational landscape of mTOR signalling steers cancer initiation and metastasis. Nature. 485, 55-61 (2012).
- Thoreen, C. C., et al. A unifying model for mTORC1-mediated regulation of mRNA translation. Nature. 485, 109-113 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados