Method Article
고효율 플라스미드 구조를 위한 In Vivo Assembly 사용
요약
In vivo assembly는 박테리아의 고유 DNA 복구 효소에 의존하여 상동 재조합에 의한 DNA 단편을 조립하는 결찰 독립적 클로닝 방법입니다. 이 프로토콜은 시약이 거의 필요하지 않고 클로닝 효율이 99%에 달하기 때문에 시간과 비용 면에서 모두 효율적입니다.
초록
IVA(In vivo Assembly)는 박테리아에 존재하는 고유 효소를 사용하여 DNA 단편의 분자간 재조합을 촉진하여 플라스미드를 조립하는 분자 클로닝 방법입니다. 이 방법은 상동성 영역이 15-50bp인 DNA 단편을 일반적으로 사용되는 실험실 대장균 균주로 변형시키는 방식으로 기능하며, 박테리아는 RecA 독립 복구 경로를 사용하여 DNA 단편을 플라스미드로 조립합니다. 이 방법은 E. coli 균주로 전환되기 전에 플라스미드의 체외 조립에 의존하는 많은 분자 클로닝 방법보다 빠르고 비용 효율적입니다. 이는 in vitro 방법을 사용하려면 특수 효소를 구입하고 배양이 필요한 순차적 효소 반응을 수행해야 하기 때문입니다. 그러나 in vitro 방법과 달리 IVA는 선형 플라스미드를 조립하는 것으로 실험적으로 입증되지 않았습니다. 여기에서는 복제 기원과 항생제 내성 마커가 다른 플라스미드 간에 플라스미드 및 서브클론 DNA 단편을 신속하게 조립하기 위해 실험실에서 사용하는 IVA 프로토콜을 공유합니다.
서문
분자 클로닝은 특정 재조합 DNA1을 포함하는 플라스미드를 생산하는 데 필요한 일련의 실험실 기술을 포함합니다. 이러한 유비쿼터스 기술은 종종 실험 워크플로우2에서 병목 현상으로 작용합니다. 많은 분자 클로닝 기술은 증폭을 위해 숙주 균주(예: 대장균 DH5a)로 형질전환하기 전에 일련의 효소 반응을 사용하여 시험관에서 DNA 단편의 조립에 의존합니다 3,4,5,6,7. 체외 플라스미드 조립 방법은 효소에 의존하기 때문에 효소를 구매하거나 정제하는 것은 비용과 시간이 많이 소요될 수 있습니다.
생체 내 조립(IVA)은 적절한 숙주에서 DNA 단편의 분자간 재조합에 의존하는 분자 클로닝 방법입니다 8,9,10,11,12. 이 방법의 기초는 일반적으로 사용되는 E. coli의 recA-cloning 균주가 제한 효소13에 의해 절단된 플라스미드와 단일 가닥 프라이머의 분자간 재조합을 매개할 수 있다는 관찰이었습니다. 대장균에 있는 플라스미드 집합을 위한 상동 DNA 끝을 생성하는 PCR의 사용은 1990 년대에서 기술되고 이 방법은 재조합 PCR 3,14이라고 지명되었다. 재조합 PCR의 효율은 약 50%14로 보고되었습니다. 그러나 이 방법은 프라이머의 높은 비용과 큰 DNA 단편을 증폭하기 위해 저충실도 중합효소를 사용하여 원치 않는 돌연변이를 도입할 위험이 있기 때문에 널리 채택되지 않았습니다. 이러한 단점은 1990년대에 심각했으며 제한 효소 매개 복제와 같은 체외 복제 방법을 사용하여 피할 수 있었습니다.
최근 몇 년 동안 프라이머의 비용이 감소했으며 새로운 상업용 중합효소가 PCR 증폭의 충실도를 높였습니다. 그 결과, 재조합 PCR은 빠르고 비용 효율적인 분자 클로닝 기술로 재검토되었으며 IVA 2,9,15,16으로 브랜드가 변경되었습니다. 타당성이 높아짐에 따라 IVA에 대한 연구가 더욱 심화되었으며, 이 방법은 최대 99%16의 클로닝 효율에 도달하도록 최적화되었습니다. 최적화를 통해 염기쌍의 수 및 용융 온도와 같은 상동 DNA의 특징이 생체 내 재결합 효율을 극대화하는 것으로 확인되었습니다 2,16. 추가 분석은 150 bp에서 7 kbp에 이르는 최대 6개의 DNA 단편이 IVA15에 의해 효율적으로 조립될 수 있음을 보여주었습니다. 또한, 우리 그룹은 최근 IVA를 사용하여 다양한 항생제 내성 카세트와 복제 기원을 가진 플라스미드 시리즈를 조립했습니다17. 이 연구에서 IVA 클로닝은 각 플라스미드(n = 2)에 대해 소수의 클론을 테스트하여 매우 효율적(71-100%)이었습니다17. 여기에서는 본 실험실에서 사용하는 IVA 프로토콜에 대해 설명합니다.
프로토콜
1. 상동 말단을 가진 디자인 뇌관 또는 DNA 파편
- 워드 프로세싱 소프트웨어 또는 특수 DNA 분석 소프트웨어를 사용하여 원하는 플라스미드를 인위적으로 조립합니다.
참고: 다양한 출처의 DNA 단편을 색상으로 구분하고 다운스트림 조립 단계에서 사용할 상동 영역을 강조하는 것이 종종 도움이 됩니다. 또한 방향성 조립을 보장하기 위해 상동 DNA를 색상으로 구분하는 것도 도움이 됩니다. - 각 DNA 단편에 결합하고 상동 DNA 서열 (그림 1) 2,15의 15-50 bp를 포함하는 설계 프라이머.
- IVA 효율을 높이려면 상동 DNA 단편의 용융 온도가 47-52°C2인지 확인하십시오.
참고: 용융 온도는 온라인 소프트웨어18 또는 공식 : 용융 온도 = 64.9 + 41 * (nG + nC - 16.4) / (nA + nT + nG + nC), 여기서 n = 시퀀스 6,19의 G, C, A 또는 T의 수. - HIS-태그를 인코딩하는 CATCATCATCATCATCAT과 같이 매우 반복적인 서열을 가진 프라이머는 불안정성을 유발할 수 있으므로 피하십시오20. poly-HIS 태그 삽입과 같은 응용 분야에 최종 반복 아미노산 서열이 필요한 경우 CATCACCATCATCACCAC과 같은 대체 코돈을 사용하여 핵산 서열 반복을 줄입니다.
참고: 프라이머는 선호하는 공급업체에서 주문할 수 있습니다. DNA 단편이 합성되려면 상동 말단이 합성된 DNA 단편에 통합되어야 합니다.
- IVA 효율을 높이려면 상동 DNA 단편의 용융 온도가 47-52°C2인지 확인하십시오.
2. 상동 말단을 포함하는 DNA 산물의 증폭
- 시판되는 표준 DNA 분리 키트 또는 알칼리 용해를 사용하여 플라스미드 DNA 또는 게놈 DNA 템플릿을 분리하십시오 6,17.
- 시판되는 high-fidelity polymerase를 사용한 PCR 반응에서 template DNA(단계 2.1)와 상동 영역(단계 1.2)을 포함하는 프라이머를 사용합니다. 다운스트림 반응에서 DNA template carryover를 제한하려면 각 반응에서 적은 양의 template DNA(20pg-1ng)를 사용하십시오. 효소 특이적 PCR 반응 혼합물 성분 및 권장 PCR 사이클링 조건에 대한 제조업체의 권장 사항을 참조하십시오. PCR 반응을 진행한다.
- PCR 반응(50 μL)을 설정하여 그림 2 에 표시된 DNA 단편을 100 μM의 dNTP, 0.2 μM의 각 DNA 프라이머, 템플릿 DNA(50 pg의 pSU19 및 1 ng의 pSU18mCherry), Mg2+ (2 mM 최종 농도) 및 1 U의 high-fidelity DNA 중합효소를 포함하는 1x PCR 반응 완충액 혼합물을 증폭합니다.
- touchdown PCR cycling 프로그램을 사용하여 pSU19 및 mCherry를 증폭합니다(그림 2). 초기 변성 단계를 95°C에서 5분 동안 설정한 다음 95°C에서 15초, 55°C-0.5°C에서 15초, 72°C에서 1분 동안 1회 설정한 다음 95°C에서 15초, 50°C에서 15초, 72°C에서 1분 동안 20회 추가 사이클을 설정합니다. 72°C에서 10분 동안 최종 확장 단계로 PCR 사이클을 완료합니다.
참고: 플라스미드 골격을 증폭해야 하는 경우, 분자량에 따라 DNA 템플릿의 수량을 조정합니다: PCR 반응 50μL당 ~20pg/1kbp의 플라스미드 DNA(pSU19(그림 참조)의 경우 DNA 50pg). 또한, 증폭 중에 도입되는 돌연변이의 수를 최소화하기 위해 PCR 사이클을 조정하십시오: 플라스미드 골격의 증폭을 위해서는 20-25 PCR 사이클 정도면 충분합니다.
- PCR 반응이 완료되면 PCR 반응의 부분 표본(2 μL)을 제거하고, 로딩 염료(1x 농도까지)와 결합하고, 아가로즈 겔 전기영동21로 DNA 단편을 분리합니다. 자외선(UV) 또는 LED 투과광기를 사용하여 아가로스 겔에서 이동한 DNA 단편을 시각화합니다. PCR 산물이 고유한지 확인하고 제품을 DNA 래더와 비교하여 예상 분자량인지 확인합니다(그림 2). 올바른 분자량의 PCR 산물이 시각화되지 않는 경우, PCR 반응 최적화를 위한 제조업체의 권장 사항을 참조하십시오.
참고: PCR 산물이 고유하지 않지만 예상 분자량에 해당하는 풍부한 DNA 단편이 있는 경우 추가 PCR 반응을 피할 수 있습니다. - 아가로스 겔에 여러 DNA 단편이 존재하는 경우, 아가로스 겔에서 예상 분자량에 해당하는 DNA 단편을 절제하고 아가로스 겔 추출 키트를 사용하여 DNA 단편을 회수합니다.
- PCR 반응에서 메틸화된 템플릿 DNA를 제거하려면 1μL의 DpnI을 반응 튜브에 직접 첨가합니다. 37°C에서 15분에서 하룻밤 동안 반응을 배양합니다.
참고: 이 선택적 단계는 다운스트림 반응에서 운반되는 template DNA의 양을 제한하는 데 적극 권장됩니다. 그러나 PCR 반응에서 매우 낮은 농도의 템플릿 DNA가 사용되는 경우 이 단계를 건너뛸 수 있습니다. - 핵산 정제 키트를 사용하여 효소 분석으로 인한 잔류 효소, 염, 프라이머 이량체 및 기타 원치 않는 저분자 DNA 산물을 제거합니다.
참고: DNA 단편이 합성되면 2.1-2.6단계를 건너뛸 수 있습니다. - 분광 광도법 또는 겔 추정을 통해 정제된 DNA 단편의 DNA 농도를 정량화합니다.
3. IVA(그림 3)
- 각 IVA 반응에 필요한 DNA의 양을 계산합니다(25-50ng의 플라스미드 DNA 권장). 분자량이 더 높은 플라스미드의 경우 더 많은 양의 플라스미드 DNA를 사용합니다(예: 2.3 kbp의 플라스미드의 경우 25 ng의 플라스미드를 사용하지만 플라스미드가 9 kbp인 경우 각 반응에서 50 ng을 사용). 반응에 필요한 삽입 DNA의 부피를 계산합니다. 1:3 또는 1:5 몰 비율의 plasmid:insert DNA를 사용합니다.
참고: 온라인 바이오 계산기 또는 아래 제공된 공식을 사용하여 이중 가닥 DNA 단편의 어금니 수량을 계산하고 각 DNA 단편의 몰 비율을 결정할 수 있습니다.
이중 가닥 DNA 단편의 어금니 수량을 계산하기 위해 다음 공식을 사용합니다: pmol = (ng의 무게) × 1,000 / (염기쌍 × 650 달톤). 또한, 몰비는 질량 인서트 (g) = 원하는 인서트 / 플라스미드 몰 비율 × 플라스미드 질량 (g) × 플라스미드 길이 인서트의 비율로 계산할수 있습니다. - 계산된 부피의 플라스미드를 결합하고 DNA를 미리 냉각된 1.5mL 마이크로 원심분리 튜브에 삽입하고 얼음 위에 보관합니다.
- 플라스미드를 옮기고 3.2단계의 DNA 혼합물을 해동된 recA- 화학적으로 적극적인 대장 균(예: 대장균 DH5a;22).
참고: 사용되는 유능 세포의 부피는 플라스미드 및 삽입 DNA 혼합물의 부피에 따라 다릅니다. 이 혼합물은 유능 세포의 10% 부피를 초과해서는 안 됩니다. - 열 충격 변환을 진행하십시오23.
- 화학적으로 유능한 대장균 과 DNA의 혼합물을 얼음 위에서 30분 동안 배양합니다.
- 42°C 수조에 1분 동안 넣은 다음 튜브를 얼음에 2분 동안 넣습니다.
- LB를 무균 방식으로 튜브로 옮겨 부피를 1mL로 만듭니다.
- 1mL 부피를 유리 배양 튜브로 옮기고 37°C, 220rpm 또는 박테리아 균주 및/또는 플라스미드가 회복될 수 있도록 권장 온도(일반적으로 25°C-37°C)로 설정된 진탕 인큐베이터에 넣고 플라스미드 인코딩 항생제 선택 마커를 생산할 수 있도록 합니다.
- 세포가 30분에서 1시간 동안 회복된 후 형질전환 반응의 분취액(1,000μL 중 100μL)을 고체 선택 배지에 증착하고 멸균 세포 스프레더를 사용하여 한천 배양 플레이트에 분취액을 분산시킵니다. 원심분리(1분 동안 13,000 × g )로 형질전환된 세포의 나머지 부분을 수집하고, 상등액을 버리고, 세포 펠렛을 100μL의 멸균 배지에 재현탁하고, 위와 같이 고체 선택 배지에 증착합니다. 액체가 고체 매체에 흡수되도록 30분 동안 하고 배양 플레이트를 뒤집습니다. 그리고 박테리아 균주 및/또는 플라스미드에 권장되는 온도로 밤새 인큐베이터에 넣습니다.
4. 올바른 플라스미드 조립을 위한 스크리닝
- 배양 후(3.5단계), 인큐베이터에서 배양 플레이트를 제거하고 각 배양 플레이트에 여러 개의 분리된 박테리아 콜로니가 있는 경우 직접 계수하여 콜로니를 열거합니다. 스크리닝을 위해 여러 콜로니(1-10)를 선택하여 원하는 조립된 플라스미드를 함유하고 있는지 확인합니다.
- 적절한 항생제가 보충된 멸균 성장 배지를 멸균 배양 튜브(유리 또는 플라스틱)로 옮깁니다. 멸균 전달 바늘 또는 루프로 1개의 콜로니를 터치하여 배양 배지를 접종하고 전달 바늘로 세포를 배양관 배지로 이식합니다. 배양 튜브를 진탕 인큐베이터에 넣고 박테리아 균주 및/또는 플라스미드에 권장되는 온도(일반적으로 25°C에서 37°C)에 권장되는 온도에서 16-18시간 동안 배양합니다.
- 다음 날, 알칼리 용해6 또는 제조업체의 지침에 따라 시판되는 플라스미드 분리 키트를 사용하여 박테리아 배양에서 플라스미드 DNA를 분리합니다.
- 플라스미드가 올바르게 조립되었는지 확인합니다.
- 분광광도법 또는 겔 추정을 통해 분리된 플라스미드의 DNA 농도를 정량화합니다.
- 진단용 제한 분해 반응(10 μL 최종 부피)을 위해 부분 표본(50-150 ng)의 플라스미드 DNA를 사용합니다. 조립된 플라스미드 DNA를 절단할 것으로 예상되는 위치에 따라 제한 효소를 선택합니다.
참고: 두 가지 개별 반응을 권장합니다: (1) 단일 부위에서 플라스미드를 절단할 것으로 예상되는 제한 효소로 플라스미드 DNA를 처리하고, (2) 삽입된 DNA 단편의 전체 또는 일부를 절단할 것으로 예상되는 두 개의 제한 효소로 플라스미드 DNA를 처리합니다. 제한 효소는 시판되고 있으며 제조업체의 지침에 따라 사용해야 합니다. - 제한 효소 반응이 완료되면, 로딩 염료(1x 농도까지)와 결합하고, 전체 혼합물을 아가로스 겔에 로드하고, 아가로즈 겔 전기영동21로 DNA 단편을 분리합니다. UV 또는 LED transilluminator를 사용하여 아가로스 겔에서 이동한 DNA 단편을 시각화합니다. 제한된 DNA 단편을 분자량 래더와 비교하여 예상 분자량인지 확인합니다(그림 4). 예상되는 효소 제한 패턴( positive clones이라고 함)을 가진 플라스미드를 주목하십시오. 예상되는 제한 패턴이 없는 플라스미드가 포함된 튜브( negative clone이라고 함)를 폐기합니다.
- positive clone이 예상되는 nucleotide sequence를 갖도록 하려면 염기서열분석을 위해 positive plasmid clone의 부분 표본을 보냅니다.
참고: 전체 플라스미드 염기서열분석(예: 나노포어 염기서열분석)이 권장됩니다.
결과
이 원고에서는 IVA 사용의 예로, 제공된 IVA 워크플로우를 따라 mCherry open reading frame을 plasmid pSU19의 다중 클로닝 부위에 재복제하여 pSU19mCherry와 동일한 plasmid를 생성했습니다(그림 3)17. IVA에 적합한 프라이머는 프로토콜 단계 1.1 및 1.2를 기반으로 설계되었습니다. 그런 다음 플라스미드 분리 키트를 사용하여 pSU19 및 pSU18mCherry를 분리했으며, 이는 각각 플라스미드 골격을 증폭하고 DNA를 삽입하기 위한 PCR 반응의 템플릿 역할을 했습니다(프로토콜 단계 2.1). pSU19 플라스미드 골격은 PCR-증폭되었고(프라이머 쌍: pSU19_F: TAGGGTACCGAGCTCGAATTC 및 pSU19_R CCCGGGGATCCTCTAGAGTC), mCherry 오픈 리딩 프레임은 pSU18mCherry(프라이머 쌍 mCherry_F: ctctagaggatccccgggATGGTATCAAAAGGAGAGGAAG 및 mCherry_R: gaattcgagctctcggtaccctaTTATCATTACTTGTACAGTTC; mCherry 프라이머의 소문자 뉴클레오티드 서열은 pSU19 백본을 증폭하는 데 사용되는 프라이머와 상동적인 뉴클레오티드를 나타냅니다; 프로토콜 단계 2.2). 특이적 PCR 증폭은 아가로즈 겔 전기영동에 의해 검증되었다(프로토콜 단계 2.3; 그림 2) PCR 산물은 나머지 Day 1 워크플로우(프로토콜 단계 2.5-3.5; 그림 3).
다음 날, 형질전환판의 콜로니(colonies)가 열거되었습니다(프로토콜 단계 4.1; 그림 3, 2일차). 이 실험에서는 6개의 군체가 보였다. 콜로니의 수는 회수된 배양액의 더 큰 부분 표본을 도금하거나 유능 세포로 형질전환되는 DNA의 양을 증가시킴으로써 증가할 수 있습니다. 형질전환 플레이트에 콜로니가 없는 경우, 먼저 유능한 세포가 높은 형질전환 효율을 갖는지 확인하는 것이 중요합니다. 당사는 형질전환된 pUC19 DNA의 107 - 109 콜로니 형성 단위/μg 범위의 형질전환 효율을 가진 수용 세포를 사용하여 성공을 거두었습니다. 형질전환 효율이 문제가 되지 않는 경우, 더 높은 분자량 플라스미드로 IVA를 수행하거나 많은 DNA 단편을 조립할 때 수용 세포로 형질전환된 DNA의 양을 늘리거나 수용 세포의 부피를 증가시켜야 할 수 있습니다.
올바른 플라스미드 조립을 위해 스크리닝하기 위해 두 개의 콜로니를 선택했습니다(프로토콜 단계 4.1-4.3.3). 다음 날에는 3일차의 워크플로를 따랐습니다(그림 3). 플라스미드 분리 후, 올바르게 조립된 플라스미드를 한 번 또는 두 번 절단할 것으로 예상되는 XbaI 또는 XbaI 및 EcoRI로 각 플라스미드를 처리합니다(그림 4). 그림 4에서 볼 수 있듯이, 두 효소 반응 모두 플라스미드 클론 1에 대해 2.3 kbp에 해당하는 단일 DNA 산물을 생성했으며, 이는 이것이 pSU19 단독임을 나타냅니다. plasmid clone 2의 효소 처리는 XbaI으로 처리한 후 단일 ~3 kbp DNA 산물과 XbaI 및 EcoRI로 처리한 후 두 개의 DNA 산물(2.3 kbp 및 770 bp)을 생성했습니다. 그런 다음 전체 플라스미드 염기서열분석 (프로토콜 단계 4.4; 그림 3, 3일차). 이 예에서 올바른 플라스미드 조립 효율은 50%였습니다. 올바른 플라스미드 조립을 위해 두 개 이상의 클론을 스크리닝하는 경우는 거의 없으며 일반적으로 50-100%의 플라스미드 조립 효율을 가지고 있습니다. 경험에 비추어 볼 때, 음성 클론은 일반적으로 (i) 재순환된 template DNA에서 전달된 plasmid backbone 또는 (ii) hybrid plasmid로 재결합된 template DNA로 구성된 hybrid DNA product를 포함합니다. 증권 시세 표시기I 처리(프로토콜 단계 2.5)는 음성 클론의 수를 제한합니다. 이렇게 해도 IVA 효율이 개선되지 않으면 PCR primer design을 검증하고 plasmid backbone 및 PCR 산물을 재증폭하는 것이 좋습니다.

그림 1: IVA 프라이머 설계 전략. IVA 프라이머 설계의 첫 번째 단계(1단계)는 소프트웨어를 사용하여 원하는 조립된 플라스미드 in silico의 시퀀스 파일을 구축하는 것입니다. 두 번째 단계(Step 2)는 플라스미드와 삽입된 DNA 염기서열의 접합부 근처에서 플라스미드 골격을 증폭하는 프라이머를 설계하는 것입니다. 삽입 염기서열(insert sequence)을 위한 프라이머는 원하는 DNA 산물을 특이적으로 결합 및 증폭할 수 있는 충분한 뉴클레오티드를 가져야 하며, 상동 재조합을 위해 인접한 플라스미드 골격(plasmid backbone)의 15-50 bp를 함유해야 합니다. plasmid design의 homology 영역은 색상으로 구분됩니다. 약어 : IVA = in vivo assembly. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
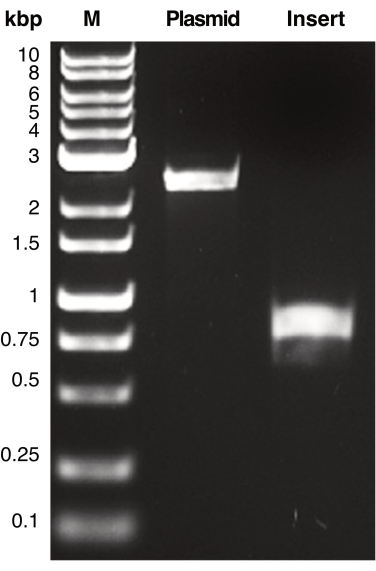
그림 2: 플라스미드 및 삽입 DNA의 PCR 증폭. 표시된 것은 플라스미드에 해당하는 PCR 증폭 DNA 단편의 대표적인 이미지이며 상동성 영역을 포함하는 DNA를 삽입합니다. DNA 단편은 겔 전기영동으로 분리하고 UV 투과조명으로 시각화했습니다. DNA 산물을 분자량 마커(M)와 비교하여 각 PCR 산물의 겉보기 분자량을 측정했습니다. 플라스미드(pSU19) 및 삽입 DNA(mCherry)의 예상 분자량은 각각 2.3kbp 및 770bp입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: in vivo assembly workflow의 개략도. DNA는 PCR 증폭되어 5' 및 3' 상동 영역을 생성합니다. 증권 시세 표시기I 처리는 주형 DNA를 절단하는 데 사용됩니다. 뉴클레오티드 정제 키트는 PCR 증폭 DNA에서 염과 효소를 제거하는 데 사용됩니다. UV 분광 광도계는 DNA 농도를 측정하는 데 사용되며, 3:1 인서트 대 플라스미드 몰 비율이 사전 냉각된 마이크로분리 튜브에 결합됩니다. 결합된 DNA는 해동된 E. coli DH5α 수용 세포의 분취액을 포함하는 얼음처럼 차가운 마이크로분리 튜브로 전달됩니다. DNA를 열 충격에 의해 E. coli DH5α로 형질전환하고, 고체 선택 배지에 스프레드 플레이트를 적용하고, 37°C에서 18시간 동안 배양합니다. 배양 후, 플라스미드 클론을 포함하는 단일 콜로니를 멸균 액체 배지와 항생제를 함유한 배양 튜브로 옮긴 다음 37°C에서 18시간 동안 교반하면서 배양합니다. 다음 날, 세포에서 플라스미드 DNA를 분리하고 제한 효소 처리를 사용하여 양성 클론을 스크리닝합니다. 그런 다음 플라스미드의 DNA 염기서열을 확인하기 위해 염기서열분석 분석을 위해 양성 클론을 보냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: 양성 플라스미드 클론을 스크리닝하기 위한 겔 전기영동. 2개의 상이한 플라스미드 클론을 분리하고 제한 효소로 처리하였는데, 한 번은 양성 클론을 절단하여 플라스미드를 선형화하거나 두 번은 삽입된 DNA를 방출할 것으로 예상되었습니다. 제한 효소 처리 후, 샘플을 겔 전기영동으로 분리하고 UV 투과조명으로 시각화했습니다. 분자량은 분자량 마커와 비교하여 추정되었습니다. 선형화된 양성 클론(플라스미드 + 삽입)의 예상 분자량은 3.07kbp였고, 양성 클론을 두 번 절단한 후 DNA 단편의 예상 분자량은 2.3kbp(플라스미드 백본) 및 770bp(삽입)였습니다. 플라스미드 클론 1은 음성(빈 플라스미드)이고 클론 2는 양성 클론입니다. 약어: SC = 단일 분열; DC = 이중 분열; M = 분자량 마커. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
성공적인 IVA 클로닝을 위한 가장 중요한 단계는 DNA 단편과 프라이머 설계입니다. IVA 효율은 상동 단편이 약 47-52°C의 용융 온도에서 길이가 15bp 이상으로 설계될 때 크게 향상됩니다. IVA 단편 설계의 최적화를 탐구하는 상세한 연구가 발표되었다16. 높은 클로닝 효율을 위한 또 다른 중요한 단계는 PCR 증폭 단계에서 가능한 한 적은 템플릿 DNA를 사용하는 것입니다. template DNA의 carryover를 더욱 줄이기 위해 DpnI으로 postcycle PCR 반응을 처리하여 methylated template DNA를 절단합니다. 이는 양성 클론을 분리하는 데 필요한 스크리닝의 양을 크게 줄일 수 있습니다. 우리의 경험에 비추어 볼 때, DpnI 처리의 경우 형질전환당 1-2개의 집락만 스크리닝하면 됩니다. DpnI 처리가 없으면 형질전환당 5-10개의 콜로니를 스크리닝해야 합니다.
우리와 다른 사람들이 IVA 클로닝이 많은 콜로니 형성 단위16,17을 산출한다고 보고했지만, 형질전환 플레이트에 콜로니가 존재하지 않을 가능성이 있습니다. 우리의 경험에 비추어 볼 때, 수용 세포로 형질전환된 DNA의 양을 늘리거나(특히 큰 플라스미드 골격으로 작업할 때) 사용되는 수용 세포의 부피를 늘리면 형질전환 효율을 향상시킬 수 있습니다. DNA 또는 유능 세포의 양을 조정한 후에도 콜로니가 보이지 않는 경우, 유능 세포 스톡의 형질전환 효율을 실험적으로 확인하고 선형 DNA 단편을 염기서열 분석하여 원하는 상동 영역을 포함하는지 확인합니다. 이러한 문제 해결 단계를 따르면 일반적으로 IVA 효율성이 향상됩니다.
IVA 클로닝 방법에는 몇 가지 제한 사항이 있습니다. 이는 특정 DNA 단편을 증폭하기 위해 PCR을 사용하는 것과 관련이 있습니다. 예를 들어, 플라스미드 백본은 크기가 다양할 수 있으며, 일부는 길이가 10kbp에 근접하거나 이를 수 있습니다(예: pMMB67HE24, pFUSE25). 원하는 플라스미드 골격이 너무 크거나 충실도가 높은 교정 중합효소로 증폭하기 어려운 경우, 제한 효소(예: SmaI 또는 EcoRV)로 플라스미드를 선형화하고 IVA 반응에 PCR 산물 대신 선형화된 플라스미드를 사용할 수 있는 옵션이 여전히 있습니다16. 우리의 경험에 비추어 볼 때, 제한 효소 처리로 100%에 가까운 플라스미드 선형화가 달성되지 않으면 IVA의 효율성이 크게 감소합니다.
IVA의 또 다른 잠재적 한계는 각각의 고유한 DNA 단편을 클로닝하기 위해 상동 DNA 돌출부를 포함하는 새로운 프라이머를 구입하는 것과 관련된 비용입니다. 실험실에서 제한된 수의 플라스미드 백본을 사용하고 범용 프라이머를 사용하여 향후 IVA 클로닝을 위해 대량의 증폭된 플라스미드 백본을 생성할 수 있는 경우 이 비용을 부분적으로 완화할 수 있습니다. 또한 실험실에서 사용되는 모든 플라스미드가 동일한 여러 클로닝 부위를 가지고 있는 경우 모든 플라스미드에 대해 동일한 범용 프라이머를 사용할 수 있습니다. IVA 방법의 또 다른 한계는 우리가 아는 한, 반복적이거나 불안정한 DNA26을 클로닝하는 데 일반적으로 사용되는 선형 플라스미드의 조립에 대해 테스트되지 않았다는 것입니다.
IVA는 체외 DNA 절편 조립에 의존하는 다른 분자 클로닝 방법에 비해 몇 가지 장점이 있는 시간 및 비용 효율적인 클로닝 방법입니다. 플라스미드 조립은 DNA 4,5,27을 조립하기 위해 상업적으로 구입된 효소 또는 완충액에 의존하지 않기 때문에 일반적으로 비용 효율적입니다. 이를 통해 일반적인 효소 반응 단계를 피할 수 있기 때문에 시간과 비용을 절약할 수 있으며, 이를 통해 최대 72시간의 실험실 조작을 절약할 수 있습니다 2,15. 또한 IVA에 필요한 장비 및 시약은 일반적으로 분자 클로닝을 일상적으로 수행하는 실험실에 이미 존재하거나 사용할 수 있습니다.
우리 그룹은 최근 IVA를 사용하여 동일한 다중 클로닝 부위를 가진 일련의 24개의 플라스미드, 플라스미드 복제 수에 대한 3가지 옵션, 항생제 선택 마커에 대한 5가지 옵션을 생성했습니다17. 우리는 범용 프라이머를 사용하여 각 플라스미드17에 유전자를 복제하는 방법을 보여주었습니다. 이 연구에서 클로닝 효율은 71-100%였는데, 이는 양성 클론을 식별하기 위해 스크리닝된 콜로니가 매우 적었기 때문입니다(5개의 양성 클론에 대해 7개의 스크리닝)17. 또 다른 연구에서는 IVA 클로닝 효율이 최대 97% ± 1.9%에 이른다고 보고했으며, 이러한 높은 효율은 저자가 올바른 플라스미드 어셈블리를 위해 많은 수의 클론을 체계적으로 테스트함으로써 설명할 수 있습니다16. IVA는 또한 부위 지정 돌연변이 유발 또는 태그 서열을 포함하는 프라이머의 통합과 같은 응용 분야에도 사용할 수 있습니다27. IVA는 실험실의 많은 일상적인 분자 클로닝 응용 분야에 적용할 수 있는 다재다능하고 효율적인 클로닝 기술입니다.
공개
저자는 선언할 이해 상충이 없습니다.
감사의 말
HGB는 캐나다 자연 과학 및 공학 연구 위원회(NSERC)의 캐나다 대학원 장학금 - 석사 프로그램과 학장 박사상(서스캐처원 대학교)의 자금 지원을 받습니다. 이 작업은 NSERC 디스커버리 그랜트(RGPIN-2021-03066), 서스캐처원 대학교 및 캐나다 혁신 재단 John R. Evans Leaders Fund(JLT에 대한 보조금 번호 42269)의 창업 기금(JLT로)의 지원을 받았습니다. 저자들은 자외선 분광법(UV spectroscopy)에 의한 DNA 정량화 사진을 제공해 준 에릭 툼즈(Eric Toombs)에게 감사를 표한다.
자료
| Name | Company | Catalog Number | Comments |
| 1 kb Plus DNA ladder | FroggaBio | DM015-R500 | DNA molecular weight marker |
| AccuReady M Single Channel 0.5-10 µL | Bulldog Bio | BPP020 | Pipettes for transferring liquid |
| AccuReady M Single Channel Pipettor Kit | Bulldog Bio | BPK100 | Pipettes for transferring liquid |
| Agar | BioShop Canada | AGR003.1 | Solidifying agent for growth medium |
| Agarose | Biobasic | D0012 | Make agarose gels for DNA electrophoresis |
| Biometra TOne | Analytikjena | 846-2-070-301 | Thermocycler |
| Caps for glass culture tubes | Fisher Scientific | 14-957-91 | Reusable caps for glass culture tubes |
| DNA primers | Integrated DNA Technologies | N/A | Bind and amplify template DNA in PCR reaction |
| DpnI | New England Biolabs | R0176S | Cleave methylated template DNA following PCR amplification |
| EcoRI | New England Biolabs | R3101L | Restriction enzyme, comes with appropriate buffer |
| Eppendorf microtubes 1.5 mL | Sarstedt | 72.690.300 | Microtube, 1.5 mL, conical base, PP, attached flat cap, molded graduations and frosted writing space |
| Ethidium bromide | Fisher Scientific | AAL0748203 | DNA visualization/intercalating agent, toxic |
| EZ-10 Spin Column Plasmid DNA Miniprep Kit | Biobasic | BS614 | Isolate plasmid DNA from overnight bacterial cultures |
| GelDoc Go-Gel system | Bio-Rad | 12009077 | Imaging DNA gels |
| Glass culture tubes | Fisher Scientific | 14-925E | Glass culture tubes |
| myGel Mini Electrophoresis System | Sigma-Aldrich | Z742288 | System used for gel electrophoresis |
| NanoDrop One | Thermo Fisher | ND-ONE-W | Determine DNA concentration |
| PCR Clean Up for DNA Sequencing | Biobasic | BT5100 | Purify PCR products |
| Petri dish | Sarstedt | 82.1473.011 | Petri dish 92 x 16 mm, PS, transparent, with ventilation cams |
| Pipette tip, 1000 µL | Sarstedt | 70.305 | Pipette tips for 100-1000 µL |
| Pipette tip, 2.5 µL | Sarstedt | 70.3010.265 | Pipette tips for up to 2.5 µL |
| Pipette tip, 20 µL | Sarstedt | 70.3020.200 | Pipette tips for up to 20 µL |
| Pipette tip, 200 µL | Sarstedt | 70.3030.020 | Pipette tips for 1-200 µL |
| Salt (NaCl) | Fisher Scientific | S271-10 | Component of bacterial growth medium (10 g/L) |
| Single 0.2 mL PCR tubes with flat cap | FroggaBio | TF-1000 | PCR tubes |
| Tryptone (Bacteriological) | BioShop Canada | TRP402.5 | Component of bacterial growth medium (10 g/L) |
| VeriFi DNA polymerase | PCR Biosystems | PB10.42-01 | High fidelity polymerase, the PCR buffer containing Mg2+ and dNTPs is provided with purchase |
| Xba I | New England Biolabs | R0145S | Restriction enzyme, comes with appropriate buffer |
| Yeast extract | BioShop Canada | YEX401.205 | Component of bacterial growth medium (5 g/L) |
참고문헌
- Bertero, A., Brown, S., Vallier, L. Methods of cloning. Basic Science Methods for Clinical Researchers. Chapter 2, 19-39 (2017).
- Garcia-Nafria, J., Watson, J. F., Greger, I. H. IVA cloning: A single-tube universal cloning system exploiting bacterial in vivo assembly. Sci Rep. 6, 27459 (2016).
- Bubeck, P., Winkler, M., Bautsch, W. Rapid cloning by homologous recombination in vivo. Nucleic Acids Res. 21 (15), 3601-3602 (1993).
- Avilan, L. Assembling multiple fragments: the Gibson assembly. Methods Mol Biol. 2633, 45-53 (2023).
- Stukenberg, D., et al. The Marburg Collection: A Golden Gate DNA assembly framework for synthetic biology applications in Vibrio natriegens. ACS Synth Biol. 10 (8), 1904-1919 (2021).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular cloning: a laboratory manual. , (1989).
- Engler, C., Marillonnet, S. Golden Gate cloning. Methods Mol Biol. 1116, 119-131 (2014).
- Ma, H., Kunes, S., Schatz, P. J., Botstein, D. Plasmid construction by homologous recombination in yeast. Gene. 58 (2-3), 201-216 (1987).
- Cao, P., Wang, L., Zhou, G., Wang, Y., Chen, Y. Rapid assembly of multiple DNA fragments through direct transformation of PCR products into E. coli and Lactobacillus. Plasmid. 76, 40-46 (2014).
- Conley, E. C., Saunders, V. A., Saunders, J. R. Deletion and rearrangement of plasmid DNA during transformation of Escherichia coli with linear plasmid molecules. Nucleic Acids Res. 14 (22), 8905-8917 (1986).
- Conley, E. C., Saunders, V. A., Jackson, V., Saunders, J. R. Mechanism of intramolecular recyclization and deletion formation following transformation of Escherichia coli with linearized plasmid DNA. Nucleic Acids Res. 14 (22), 8919-8932 (1986).
- Sung, W. L., Zahab, D. M. Site-specific recombination directed by single-stranded crossover linkers: specific deletion of the amino-terminal region of the beta-galactosidase gene in pUC plasmids. DNA. 6 (4), 373-379 (1987).
- Mandecki, W. Oligonucleotide-directed double-strand break repair in plasmids of Escherichia coli: a method for site-specific mutagenesis. Proc Natl Acad Sci USA. 83 (19), 7177-7181 (1986).
- Jones, D. H., Howard, B. H. A rapid method for recombination and site-specific mutagenesis by placing homologous ends on DNA using polymerase chain reaction. Biotechniques. 10 (1), 62-66 (1991).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5alpha-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), e0137466 (2015).
- Chen, F., et al. Simplified plasmid cloning with a universal MCS design and bacterial in vivo assembly. BMC Biotechnol. 21 (1), 24 (2021).
- Braun, H. G., Kanwal, N., Rivera Lopez, L. F., Thomassin, J. L. Generation of a plasmid series for rapid sub-cloning and use in various Enterobacteriaceae. J Biosci Bioeng. 138 (6), 478-487 (2024).
- Kibbe, W. A. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids Res. 35 (Web Server issue), W43-W46 (2007).
- Wallace, R. B., et al. Hybridization of synthetic oligodeoxyribonucleotides to phi chi 174 DNA: the effect of single base pair mismatch. Nucleic Acids Res. 6 (11), 3543-3557 (1979).
- Bzymek, M., Lovett, S. T. Instability of repetitive DNA sequences: The role of replication in multiple mechanisms. Proc Natl Acad Sci USA. 98 (15), 8319-8325 (2001).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), e3923 (2012).
- Sambrook, J., Russell, D. W. The inoue method for preparation and transformation of competent E. coli: "ultra-competent" cells. CSH Protoc. 2006 (1), (2006).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. J Vis Exp. (6), e253 (2007).
- Fürste, J. P., et al. Molecular cloning of the plasmid RP4 primase region in a multi-host-range tacP expression vector. Gene. 48 (1), 119-131 (1986).
- Bäumler, A. J., et al. Identification of a new iron regulated locus of Salmonella typhi. Gene. 183 (1-2), 207-213 (1996).
- Godiska, R., et al. Linear plasmid vector for cloning of repetitive or unstable sequences in Escherichia coli. Nucleic Acids Res. 38 (6), e88 (2010).
- Watson, J. F., Garcia-Nafria, J. In vivo DNA assembly using common laboratory bacteria: A re-emerging tool to simplify molecular cloning. J Biol Chem. 294 (42), 15271-15281 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
ISSN 2578-2614
Copyright © 2025 MyJoVE Corporation. 판권 소유
당사 웹 사이트에서는 사용자의 경험을 향상시키기 위해 쿠키를 사용합니다.
당사 웹 사이트를 계속 사용하거나 '계속'을 클릭하는 것은 당사 쿠키 수락에 동의하는 것을 의미합니다.
