Method Article
S. cerevisiae細胞から精製された双方向キネシン-5 Cin8の一分子およびクラスターの運動性
要約
双方向有糸分裂キネシン-5 Cin8は、運動性の間に分裂して融合するクラスターに蓄積する。クラスターへの蓄積は、Cin8の速度と方向性も変化させます。ここでは、精製Cin8−GFPを用いた運動性アッセイおよびCin8の単一分子およびクラスターの運動性特性の分析のためのプロトコルが記載されている。
要約
有糸分裂バイポーラキネシン-5モーターは、スピンドルダイナミクスにおいて重要な機能を果たします。これらのモーターは、活性複合体の両端に位置する2対の触媒モータードメインを有するホモ四量体構造を示す。このユニークなアーキテクチャにより、キネシン-5モータは、逆平行スピンドル微小管(MT)を架橋してスライドさせることができ、スピンドルポールを分離する外向きの力を提供します。以前は、キネシン-5モーターはもっぱらプラスエンド指向であると考えられていました。しかし、最近の研究では、いくつかの真菌キネシン-5モーターがマイナス末端が単一分子レベルに向けられており、さまざまな実験条件下で方向性を切り替えることができることが明らかになりました。 サッカロミセス・セレビシエ・ キネシン−5 Cin8は、そのような双方向モータータンパク質の一例である:高イオン強度条件下では、Cin8の単一分子はMTのマイナス末端方向に移動する。また、Cin8は主にMTのマイナス端で運動性クラスターを形成し、そのようなクラスタリングはCin8が方向性を切り替え、ゆっくりとしたプラスエンド指向性を経験することを可能にすることも示された。この記事では、 S. cerevisiae 細胞におけるタンパク質過剰発現およびその精製から in vitro 単一分子運動性アッセイまで、GFPタグ付きキネシン-5 Cin8を使用するすべてのステップについて詳細なプロトコルを提供します。ここで説明する新しく開発された方法は、蛍光強度に基づいてCin8の単一分子とクラスターを区別するのに役立ちます。この方法は、Cin8の単一分子およびクラスターの運動性の別々の分析を可能にし、したがって、Cin8の運動性のクラスターサイズに対する依存性の特性評価を提供する。
概要
真核細胞内の多数の運動性事象は、分子運動タンパク質の機能によって媒介される。これらのモーターは、細胞骨格フィラメント、アクチンフィラメント、および微小管(MT)に沿って移動し、ATP加水分解の化学エネルギーを細胞内の生物学的運動性を駆動するために必要な運動学的および機械的力に変換する。MTベースのS. cerevisiae Cin8は、双極性のホモ四量体キネシン-5モータータンパク質であり、スピンドルMTを架橋してスライドさせる1。Cin8は、有糸分裂中、紡錘体アセンブリ2,3,4および分裂後期5,6,7中の紡錘体伸長において、本質的な機能を果たす。これまで、Cin8は双方向モータであり、異なる実験条件下で方向性を切り替えることが実証されていました。例えば、高いイオン強度条件下では、単一のCin8モータはMTのマイナス端に向かって移動するが、クラスタでは、マルチモータMTグライダーアッセイでは、および反並列MT間では、Cin8モータは主にMTのプラス端に向かって移動する8,9,10,11,12 .これらの発見は、いくつかの理由により非常に予想外であった。第一に、Cin8はアミノ末端に触媒モータードメインを持ち、そのようなモーターは以前はもっぱらプラスエンド指向性であると考えられていたが、Cin8は単一分子レベルでのマイナス末端指向性であることが示された。第二に、キネシンモーターはマイナス端またはプラス端のいずれかの単方向であると考えられていたが、Cin8は実験条件に応じて双方向であることが示された。最後に、有糸分裂スピンドルにおけるMT配向のために、スピンドルアセンブリ中のスピンドル極と分裂後期Bの分離におけるキネシン-5モータの古典的な役割は、それらが架橋するMT上のプラスエンド指向性によってのみ説明できた1,13。Cin8の双方向性に関する最初の報告に続いて、他のいくつかのキネシンモーターが双方向であることが実証された14,15,16、キネシンモーターの双方向運動性は、以前に信じられていたよりも一般的である可能性があることを示している。
細胞内では、Cin8も双方向で移動することが以前に報告されており8、いくつかのキネシン5モーターの双方向運動性が細胞内機能にとって重要であるという考えを支持している。また、双方向であることが報告された3つのキネシン-5モーターは真菌細胞由来であることから、このような細胞10においてキネシン-5モーターの双方向性に対する役割の可能性が最近提案されている。このモデルによると、有糸分裂中に核エンベロープが破壊されない真菌細胞の閉鎖有糸分裂では、キネシン-5モーターは、紡錘体組み立ての前に紡錘極を分離する初期力を提供する。このタスクを実行するために、主軸極分離の前に、キネシン-5モータは、単一の核MT上のマイナス端指向運動性によって、主軸極の近くに局在する。この位置に着くと、キネシン-5モーターはクラスタリングし、方向を切り替え、キャプチャし、隣接するスピンドルポールからMTをクロスリンクします。続いて、キネシン-5モータは、それらが架橋するMT上のプラスエンド指向性運動性によって極の初期分離を提供する。このモデルでは、真菌キネシン-5モータがスピンドルアセンブリでその役割を果たすために、単一MT上のマイナス端指向性運動性と、逆平行摺動中の架橋MT上のプラスエンド指向性運動性の両方が必要である1,13。
記載された方法の全体的な目標は、高純度の真菌GFPタグ付きキネシン-5 Cin8を取得し、単一分子およびCin8のクラスターの運動性を別々に分析しながら、単一分子運動性アッセイ(図1)を行うことである。単一分子とクラスターの間の分離は、Cin8の方向性に影響を与えることが実証された要因の1つがMTs10,12上のクラスターへの蓄積であるため、重要である。MT表面滑空およびMT摺動アッセイなどの代替運動性アッセイは、単一モータータンパク質の活性に関する情報を提供しない17、18。ここで説明する堅牢な単一分子運動性アッセイおよび分析方法は、キネシン-5モーター、Cin8およびKip1 10、11、12、14、19、20のさまざまな側面を特徴付けるために首尾よく適用されています。
ここでは、Cin8の過剰発現および精製、MTの重合、および単一分子運動性アッセイについて、詳細なプロトコールを提示する。さらに、Cin8の単一分子とクラスターを区別し、平均変位(MD)および平均二乗変位(MSD)分析によって単一モーター速度およびクラスター速度を決定するための分析も記載されている。このプロトコルは、研究者が手順のすべてのステップを視覚化し、このタイプのアッセイのトラブルシューティングを支援することを目的としています。

図1:一分子運動性アッセイの概略図。 ビオチン化蛍光MTはガラス表面に付着し、表面結合ビオチン化BSAと相互作用するアビジンでコーティングされている。緑色の矢印は、高イオン強度条件下での単一のCin8分子の移動方向を表す。+/- MTの極性を表します 。この図の拡大版を表示するには、ここをクリックしてください。
プロトコル
1. 緩衝液および試薬の調製
- バッファー
- -Leu aaドロップアウトミックス:アデニン、ウラシル、トリプトファン、ヒスチジン、リジン、メチオニンの各2gを混合し、室温で保存する。
- 酵母選択培地とラフィノース(1L):酵母窒素ベース6.7g(硫酸アンモニウムを含む)、2gの-Leu aaドロップアウトミックス、および20gのラフィノースを二重蒸留水に混合し、完全に溶解するまで(加熱せずに)撹拌する。0.22 μm フィルターを使用して、溶液を滅菌ボトルにろ過します。
- 溶解緩衝液:50 mM トリス、30 mM パイプ、500 mM KCl、10% グリセロール、1.5 mM β-メルカプトエタノール、1 mM MgCl 2、0.1 mM ATP、および 0.1% Triton X-100 からなるトリプル蒸留水 (TDW)に 25 mL の溶液を調製します。6 M HClを使用してpHを8に調整します。
- 溶出緩衝液:50 mM トリス、30 mM パイプ、500 mM KCl、350 mM イミダゾール、10% グリセロール、1.5 mM β-メルカプトエタノール、1 mM MgCl2、0.1 mM ATP、および 0.1% Triton X-100 からなる TDW 中の溶液 10 mL を調製します。6 M HClを使用してpHを7.2に調整します。
- P12バッファー:12 mM パイプ、1 mM EGTA、および 2 mM MgCl 2 からなる TDW 中の溶液 10 mL を調製します。10 M NaOHを使用してpHを6.9に調整します。
- BRB80バッファー:超純水中に80 mM パイプ、1 mM EGTAおよび2 mM MgCl2 からなる溶液50 mLを調製する。10 M NaOHを使用してpHを6.9に調整します。
- 一般チューブリンバッファー(GTB):超純水中で80 mM パイプ、0.5 mM EGTA、および 2 mM MgCl2 からなる溶液 50 mL を調製します。10 M NaOHを使用してpHを6.9に調整します。
- トリスパイプス溶液:TDWで6.055gのトリスと9.07gのパイプを混合して40mLの1Mトリス-0.6Mパイプス溶液を調製し、6M HClを使用してpHを7.2に調整します。
注:P12、BRB80、およびTris-Pipesバッファーは、運動性アッセイ用のストック溶液の調製に使用されます。これらの緩衝液は、大量に調製し、1.5mLチューブに小分けし、スナップ凍結し、-20°Cで保存することができる。
- 運動性アッセイのためのストッリューション
- チューブリン(10 mg mL-1):凍結乾燥チューブリン1 mgを100 μLの冷たい(4°C)一般チューブリン緩衝液(GTB)に溶解する。1 μLのアリコートをスナップフリーズし、-80°Cで保存します。
- ビオチン化チューブリン(1mgmL-1):凍結乾燥チューブリン20μgを20μLの低温GTBに溶解する。1 μLのアリコートをスナップフリーズし、-80°Cで保存します。
- ローダミン標識チューブリン(1mg mL-1):凍結乾燥チューブリン20 μgを20 μLの低温GTBに溶解する。0.5 μLのアリコートをスナップフリーズし、-80 °Cで保存します。
- GMPCPP(10 μM):GMPCPPは、100 μL水溶液として供給業者から得られ、-80°Cで保存される。 氷の上でGMPCPPでバイアルを解凍する。1μLのアリコートを調製し、スナップフリーズし、-80°Cで保存する。
- ATP:0.5 M トリス緩衝液(pH 8)中の100 mM ATPの500 μL溶液を調製する。2 μLのアリコートをスナップフリーズし、-20°Cで保存します。
- MgCl2:P12緩衝液中の200mMMgCl2の1mL溶液を調製する。5 μL アリコートを -20 °C で保存します。
- カゼイン:BRB 80緩衝液中の5mgmL-1カゼインの1mL溶液を調製する。10 μLアリコートをスナップフリーズし、-20 °Cで保存します。
- D-グルコース:P12緩衝液中の1 M D-グルコースの1 mL溶液を調製する。10 μL アリコートを -20 °C で保存します。
- グルコースオキシダーゼ:P12緩衝液中の10mgmL−1 グルコースオキシダーゼの1mL溶液を調製する。2 μLのアリコートをスナップフリーズし、-20°Cで保存します。
- カタラーゼ:P12緩衝液中の0.8 mg-1 カタラーゼの1 mL溶液を調製する。2 μLのアリコートをスナップ凍結し、-20°Cで保存する。
- ジチオスレイトール(DTT):ヒュームフード内のP12緩衝液中の1 M DTTの1mL溶液を調製する。10 μLアリコートをスナップフリーズし、-20 °Cで保存します。
- クレアチンリン酸:P12緩衝液中の1 Mクレアチンリン酸の1 mL溶液を調製する。2 μLのアリコートをスナップフリーズし、-20°Cで保存します。
- クレアチンホスホキナーゼ:0.25Mグリシルグリシン中の5mgmL-1クレアチンホスホキナーゼの1mL 溶液を調製し、pH7.4。2 μLのアリコートをスナップフリーズし、-20°Cで保存します。
- EGTA:超純水中に100mM EGTA溶液を調製し、室温で保存する。
- KCl:超純水中に1M KCl溶液を調製し、室温で保存する。
- 運動性緩衝液および反応混合物
- 運動性緩衝液(MB)と145 mM KCl、2xストック:100 μLの既製のTris-Pipes溶液、20 μLの100 mM EGTA、290 μLのKCl、および590 μLのTDWを混合して、運動性緩衝液の2 mLを1 mL調製する。バッファーを氷の上に置きます。
- 運動性反応混合物: 表1 に従って運動性反応混合物を調製し、氷上に保存する。
| 容積 | 株式 | 試薬名 |
| 50 μL | 2倍速 | MB (ステップ 1.3.1 から) |
| 40 μL | - | ティッカー |
| 1 μL | 100ミリアンペア月間 | ティッカー |
| 1 μL | 200ミリアンペア時 | マグネシウム2 |
| 2 μL | 5ミリグラム/ミリリットル | カゼイン |
| 1 μL | 1 M | グルコース |
| 1 μL | 1 M | ティッカー |
| 1 μL | 10 ミリグラム/ミリリットル | グルコースオキシダーゼ |
| 1 μL | 8ミリグラム/ミリリットル | カタラーゼ |
| 1 μL | 1 M | ホスホクレアチン |
| 1 μL | 5ミリグラム/ミリリットル | クレアチンホスホキナーゼ |
| 100 μL | トータル |
表 1.
2. セレビシエ菌細胞からのCin8の過剰発現と精製
- Cin8-GFP-6Hisの過剰発現のためのプラスミドを含むセレビシエ菌細胞を指数関数的増殖期(OD600=0.6-0.8)に2%ラフィノースを添加した1Lの酵母選択培地(工程1.1.2参照)中で28°Cで増殖させる12。
- 2%ガラクトースの添加によりCin8-GFP-6His過剰発現を誘導する。600nmにおける吸光度を測定することによって酵母培養増殖をモニターする。
- ガラクトース添加後5時間後、4,000 x g で4°Cで15分間遠心分離して細胞を回収し、溶解バッファーに細胞を懸濁し、液体N2で凍結した。
注:凍結細胞は、さらなる使用のために-80°Cで保存することも、液体N2で直ちに粉砕することもできます。 - 凍結細胞を冷却乳鉢および乳棒を用いて液体N2 で粉砕する。粉砕中に液体N2 を加え、抽出物を凍結状態に保つ。典型的には、液体N2を4〜5回添加する必要がある。
- 位相差またはDIC顕微鏡下での観察により細胞溶解をモニターする。
- 粉砕した細胞を解凍し、21,000 x g で4°Cで30分間遠心分離機で遠心分離する。 上清を2mLのNi-NTAで満たされた重力フローカラムにロードし、溶解緩衝液で予め平衡化します。上清をカラムから流出させます。
- カラムを 5 カラム容量の溶解バッファーで洗浄し、次に 5 カラム容量の溶解バッファーで 25 mM イミダゾールを添加して洗浄します。
- 溶出緩衝液を用いてCin8-GFP-6Hisを溶出する(ステップ1.1.4参照)。
- 溶出したサンプルをSDS−PAGE分画によって分析し、続いてクマシーブルー染色およびウェスタンブロット分析をα−GFP抗体19でプローブした。
- Cin8-GFP-6Hisを含むフラクションをプールする(ステップ2.8および2.9)。さらに、流速0.5 mL min-1 およびカラム圧力限界1.5 MPaでサイズ排除クロマトグラフィー(SEC)によってそれらを精製し、280 nmでの吸光度と488 nmでの励起によるGFPフローネッセンス発光を同時にモニタリングします(図2A)。
- Cin8-GFP四量体に対応する画分を収集し、SDS-PAGEおよびウェスタンブロッティング(ステップ2.9参照)によって分析する(図2B)。
- 分光光度法またはブラッドフォードアッセイ、BCAアッセイなどの生化学的アッセイを使用してタンパク質濃度を推定します。
- 選択した画分をアリコートし、液体N2 中でスナップフリーズし、-80°Cで使用時まで保存した。 これらの精製タンパク質サンプルは、6ヶ月間使用することができる。
注:Cin8-GFPは、ガラクトース誘導性プロモーターからのCin8-GFP-6His過剰発現のための2μmプラスミドを含むプロテアーゼ欠損セレビシエ株から過剰発現および精製され、LGY 4093:MATα、leu2-3,112、reg-1-501、ura3-52、pep4-3、prb1-1122、gal1、pOS7(2μ、LEU2、P GAL1-CIN8-GFP-6HIS)である。 酵母株およびプラスミドは、要求に応じて入手可能である。

図2:Cin8-GFPの精製。 (A)Ni-NTA精製Cin8-GFPのサイズ排除クロマトグラムで、約510nmでの488nm励起および発光による連続GFP蛍光検出。Cin8-GFP 四量体は、SEC カラム (矢印でマーク) から約 10 mL で溶出します。レーン内のサンプルは以下の通りである:M-分子量マーカー、Ni2+-Ni-NTA精製α Cin8-GFA精製Cin8-GFPサンプルのSECカラムにロードされた、GF画分:GF画分:パネル Aに記されたCin8-GFP溶出に対応する画分。右側の矢印は、Cin8-GFPモノマーのサイズを示します(SDS-PAGEで予想されます)。 この図の拡大版を表示するには、ここをクリックしてください。
精製Cin8による1分子運動性アッセイ
- ビオチンおよびローダミン標識MTsの重合を、GMPCPPで安定化する。
- 1.5 mL チューブで次の成分を混合して MT 重合を開始します: 1 μL の 10 mg/mL チューブリンタンパク質、1 μL の 1 mg/mL ビオチン化チューブリン、0.5 μL の 1 mg/mL ローダミン標識チューブリン、1 μL の 10 mM GMPCPP、および 6.5 μL の一般チューブリンバッファー (GTB)。混合物を37°Cで1時間インキュベートする。
- MT重合に続いて、80 μLの温かい(37°C)GTBを加え、慎重に混合し、16,500 x g で20分間遠心分離する。
- 上清を捨て、50 μLの温かいGTBで上下にピしてペレットを慎重に再懸濁します。懸濁液を28°Cで保存する。
- 647nmのローダミンチャネルを用いた蛍光顕微鏡でMTsを調べた(図3A)。
注:ビオチン化蛍光標識MTを得るために、重合反応は、非標識チューブリン、ならびにビオチン化および蛍光標識チューブリンを含有する。このプロトコールでは、ローダミン標識チューブリンが使用されるが、他の蛍光コンジュゲートも同様に利用され得る。
- フローチャンバーアセンブリ
- 4 本の両面テープ (約 4 cm x ~ 3 mm) を高度な粘着ガラス スライド (長い端と平行、約 3 ~ 4 mm 離して) の上に置き、テープ ストリップ間に 3 つの「レーン」を作成して、フローチャンバーを組み立てます。テープストリップから保護紙を取り出し、テープストリップ上にシラン処理されたカバースリップ10 を配置して、容積約10μLの3つのフローチャンバを作成します。
- アビジンコート表面へのMT固定化(図1)
- マイクロピペットを使用して、1 mg/mL のビオチン化ウシ血清アルブミン (b-BSA、GTB に溶解) 15 μL をフローチャンバーに灌流して、シラン化カバースリップをコーティングします。5分後、チャンバーを80μLのGTBで洗浄する。
- 続いて、ステップ3.3.1と同様に、b-BSAに結合する1mg/mLアビジン(GTBに溶解)を15μLのフローチャンバに挿入する。5分後、チャンバーを80μLのGTBで洗浄する。
- 20 μLの1%ポロクサマーを使用してシラン処理されたカバースリップ表面を不動態化します。3分後、80μLのGTBで洗浄する。
- ビオチン化MTをb-BSA-アビジンコーティングされたカバースリップに付着させるには、GTBで通常1:20に希釈した20μLのMTを挿入します。スライドを反転位置、すなわち、カバースリップを下向きにして、暗湿度チャンバー(例えば、湿ったティッシュペーパーを含むペトリ皿)中で、室温で5分間インキュベートする。その後、200μLのGTBで洗浄する。
- 30 μLの運動性反応混合物(ステップ1.3.2を参照)をフローチャンバーに塗布する。
- Cin8-GFPモーター(ステップ2.13)を20μLの運動性反応ミックス( 表1参照)で希釈する(典型的には最終濃度5〜10μM)。それらをフローチャンバに適用し、MTに沿ったモーターの動きをすぐに画像化します。
- 運動運動性イメージング
注:MT結合およびモーターの運動性は、水銀アーク灯、100x/1.4開口数対物レンズ、および波長647nm(ローダミン用)および波長488nm(GFP用)の2つの蛍光バンドパスフィルターセットを備えた落射蛍光倒立顕微鏡を用いてモニターした。- 顕微鏡対物レンズに液浸油を一滴置きます。フローチャンバーを蛍光顕微鏡ステージに置き、カバースリップを対物レンズに向けます。
- ローダミンチャンネルをオンにしてカバースリップ表面に取り付けられたMTに焦点を合わせ、Micro-Manager ImageJ-Fijiソフトウェア21を使用して20ms露光で画像を取得します。
- GFPチャンネルをオンにし、1秒間隔、800ミリ秒の露光で90枚のタイムラプス画像を取得し、Cin8-GFPの運動性を分析します。

図3:MTとMTがCin8-GFPに結合した(A)MTの2つのフィールド(左と右)からの画像は、ステップ3.1で説明したプロトコルに従って重合され、ステップ3.4で説明したように100倍の対物レンズで画像化されました。(B) 上のパネルに示されているMTに取り付けられたCin8-GFP(下のパネル、矢印でマークされた)の2つのフィールド(左右)からの画像。スケール バー: 4 μm。この図の拡大版を表示するには、ここをクリックしてください。
4. 運動性解析
メモ:ImageJ-Fijiソフトウェアを使用して、すべての画像解析を実行し、キモグラフを生成します。
- キモグラフ生成
- タイムラプスムービーと対応するMTフィールド画像を開きます。これら 2 つのウィンドウを同期するには、「 >の分析」>「ウィンドウの同期」を選択します。
- セグメント 化された線 オプションを使用して 1 つの MT を強調表示し 、[マルチキモグラフ>分析 ] タブを使用してキモグラフを取得します。
- Cin8-GFPのクラスターサイズの決定(すなわち、クラスター内のCin8分子の数)
- 背景の減算と不均一な照明の補正を実行するには、[ 背景を処理]オプションを使用> ます。 「ローリングボールの半径」 を 100 ピクセルに設定し、「 スライド放物面」 オプションをオンにします。
- ImageJ-FijiソフトウェアのTrackMateプラグインを使用して、特定の非運動性Cin8-GFPモーターの平均蛍光強度(図3B)を半径4ピクセルの円内の時間の関数として追跡するには、次のオプションを選択します: プラグイン>トラッキング>TrackMate>LoG検出器>シンプルラップトラッカー。
- 異なるCin8-GFPモータに対してステップ4.2.2を繰り返します。異なるCin8-GFPモータの蛍光強度を時間の関数としてプロットします。
注:クラスターサイズ、すなわちクラスター内のCin8分子の数を測定するための実験戦略は、Cin8クラスタリング関連の運動性の分析の基礎を確立する。Cin8に結合したGFPのフォトブリーチングは、Cin8クラスターの総強度に対する単一のGFP分子の寄与を決定するために用いられる。例えば、蛍光強度は〜50の任意の単位(a.u.)のステップで減少し、おそらく1つのステップごとに1つのGFP分子のフォトブリーチングを表す(図4A)。Cin8はホモ四量体モータータンパク質であるため、4つのGFP分子を含んでいる。したがって、a.u.≤強度を有する全てのCin8モーターは、単一の四量体Cin8分子である可能性が高い。この方法に続いて、Cin8モーター蛍光の強度範囲は、単一のCin8分子、Cin8分子の対(Cin8四量体の二量体)、およびCin8オリゴマーについて、それぞれ12に対して<200、200〜400、および>400として割り当てられる。
- Cin8-GFPモータの強度分布解析
- ステップ4.2.2で説明されているように、ImageJ-FijiのTrackMateプラグインを使用して、タイムラプスシーケンスの最初のフレーム内のすべての蛍光Cin8-GFPモーターの平均蛍光強度を測定します。
- ビンサイズが20 a.u.のCin8-GFPの平均強度のヒストグラムをプロットし、ヒストグラムの主要なピークをガウス曲線に合わせます(図4B)。
注:強度分布分析は、フォトブリーチング実験からのCin8-GFPモータのクラスターサイズ決定を補完するものです。Cin8-GFP集団の強度分布ヒストグラムに適合したガウス曲線は、約125a.u.でピークに達し、これは、1つ、2つ、3つ、または4つの蛍光(非漂白)GFP分子のいずれかを含む単一の4量体Cin8分子の平均強度と一致し、各蛍光GFP分子は〜50a.uに寄与する。したがって、この強度分布法を用いて、1つのGFP分子の寄与を計算することもでき、これはCin8−GFP分子のクラスターサイズを割り当てるためにさらに利用することができる。

図4:Cin8-GFP漂白プロファイルと強度分布。 (A)4つの異なるCin8-GFPモータにおけるGFPのフォトブリーチング。単一のフォトブリーチングステップは、それぞれが1つのGFPのフォトブリーチングを表す可能性が高く、(B)タイムラプスシーケンスの最初のフレームにおけるCin8−GFPモータの強度分布(差し込み図)〜50a.u.の蛍光強度の低下をもたらす。〜125 a.uを中心とするガウスピーク(青色)は、単一のCin8−GFP分子を表す。このピークは、1つ、2つ、3つ、または4つの蛍光GFP分子を有する単一のCin8四量体の平均強度を示し、各GFP分子は総強度に〜50a.u.寄与する(すなわち、(50+100+150+200)/4=125)。 この図の拡大版を表示するには、ここをクリックしてください。
- MTトラックに沿ったCin8-GFP分子の運動性の追跡
- 記録されたフレームのタイムラプスシーケンスで分析するMTをトリミングするには、 長方形ツールを使用してMT を強調表示し、[ 画像]>[トリミング]を選択します。
- 分析のために蛍光Cin8-GFP粒子を選択してください。タイムラプスシーケンスの各フレーム(タイムポイント)のパーティクル座標を 、[ポイントツール] オプションと[ メジャー] オプションを使用して記録します。タイムラプス配列中の他の蛍光粒子についても同様の座標の記録を行う。
- ステップ4.2で説明されているように、外観の最初のフレームで検査されたすべてのCin8-GFP粒子にクラスターサイズを割り当てます。
- 平均変位(MD)および平均二乗変位(MSD)解析
- ステップ4.4で決定したCin8-GFPの動きの座標から、所定の座標を持つ2点間の距離を計算するための式を使用して、初期座標に対する各時点におけるCin8-GFPの変位を計算します。

ここで、dtは時刻tにおけるCin8-GFPの変位であり、xtおよびytは時刻tにおけるそれぞれの座標であり、x0およびy0はt=0におけるCin8-GFPのそれぞれの座標である。 - これらの変位値から、特定のCin8-GFP粒子について可能なすべての時間間隔の変位を計算します。検査したすべてのCin8-GFP粒子についてこの手順を繰り返します。
- 調べたすべてのCin8-GFP粒子の平均変位(MD)を時間間隔に対してプロットし、線形適合(MD = v x t + c)を被験者とする。この適合の傾き(v)は、運動性Cin8−GFP粒子の平均速度を表す。
注:このようにして、各クラスターサイズに属するすべてのCin8-GFP分子の平均速度を別々に計算して、異なるクラスターサイズの運動性を特徴付けることができる。MD解析に加えて、平均二乗変位(MSD)解析は、ステップ4.5.1および4.5.2で計算された変位値を2乗することによっても実行できます。MSD値は時間間隔に対してプロットされ、多項式曲線MSD = v 2 x t2 +2D x t + cに適合され、Cin8-GFP移動の拡散係数である追加パラメータDが与えられます。MD分析は、MTs8,10と記された極性に対して行うべきであるが、MSD分析のためにMT極性の知識は必要ない。
- ステップ4.4で決定したCin8-GFPの動きの座標から、所定の座標を持つ2点間の距離を計算するための式を使用して、初期座標に対する各時点におけるCin8-GFPの変位を計算します。
結果
この実験は、単一MT上の異なるクラスターサイズの双方向モータータンパク質Cin8の運動性特性を調べることを目的としています。Cin8-GFPの代表的な運動性は、経時的なモーターの空間的位置が示されている 図5Aのキモグラフからも明らかです。
Cin8-GFPの運動特性の分析のために、まず、クラスターサイズを各MT付着運動性Cin8-GFP粒子に割り当て(ステップ4.3)、次いで、検査されたCin8粒子の位置を時間の関数として追跡する(ステップ4.4)。クラスターサイズカテゴリごとに、個々のCin8-GFPの>40の軌跡が記録から抽出されました(図5B)。追跡解析から得られた座標を用いて、MDおよびMSD解析は、クラスタサイズ母集団ごとに別々に実行される。速度は、 図5Cに示すように、MDへの線形適合から得られます。単一のCin8-GFP分子が一方向、マイナス端指向性で高速で移動するのに対し、Cin8クラスターは双方向運動性の高い傾向でかなり低い速度を示すことがわかりました(図5B、C)。
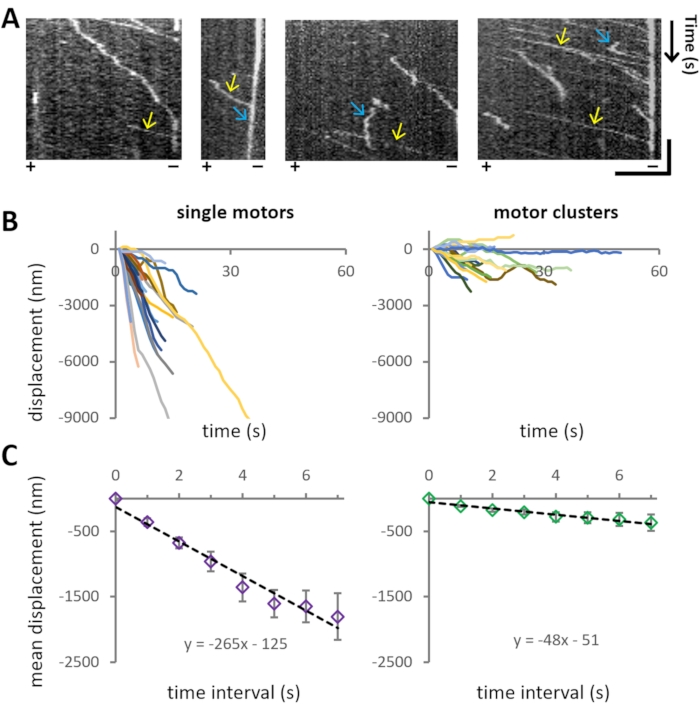
図5:Cin8-GFPの運動性。 (A) MT上のCin8-GFPモータの運動性を表すキモグラフ。X軸とY軸は、それぞれMT格子と時間を表す。黄色の矢印は、MTのマイナス端方向に向かう単一のCin8-GFP粒子の速い運動性を示し、青い矢印は、MTのプラス端方向におけるCin8クラスターの遅い運動性を示す。MTの極性は、各キモグラフの下部(+/-)に表示されます。水平バー:4μm、垂直バー:20秒(B)Cin8-GFPモータの単一モータ(左)とクラスタ(右)の変位トレース。変位トレースは、ステップ4.4で説明したように、個々のCin8-GFPモータを追跡した後に得られた座標を使用してプロットされました。変位の負の値と正の値は、それぞれMTのマイナス方向とプラス端方向の動きを示します。同じアッセイ下では、Cin8クラスターの運動性は、Cin8の単一分子と比較して遅く、双方向であることに留意されたい。(C)平均変位(MD)±SEM、Cin8モーターの単一分子(左)とクラスター(右)を時間間隔の関数としてプロットします。黒い線はプロットの線形適合値を表します(MD = v xt + c、vは平均速度、tは時間間隔、cは切片を表します)。フィッティングから、Cin8の単一モーターとクラスターの平均速度は、それぞれ-265 ± 20 nm/s、-48 ± 5 nm/sであることは明らかです。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
この研究では、双方向キネシン-5 Cin8を用いた単一分子運動性アッセイおよび運動性分析のためのプロトコルが提示される。C末端における天然核局在化シグナル(NLS)を含む全長Cin8 18 は、天然宿主 S. cerevisiaeから精製されている。Cin8は核モータータンパク質であるため、液体窒素下で セレビシエ 菌細胞を粉砕することが、細胞溶解のための最も効率的な方法であることが見出されている。溶解後、金属親和性およびサイズ排除クロマトグラフィーを組み合わせることにより、高純度のCin8が得られ、これは単一分子運動性アッセイにとって重要である。粗抽出物と精製試料8におけるCin8の運動特性との間には相違があることが以前に報告されている。さらに、モータータンパク質および非モータータンパク質によるMTクラウディングが双方向キネシン-5 Cut722の方向性に影響を与えることも報告されている。したがって、高純度のモーターは、野生型および変異型の運動挙動に関する信頼性の高い運動性分析および結論のために必要とされる。ここで説明する技術は、適切なバッファー調整で酵母から他の核タンパク質を精製するために容易に適合させることができる。
ここでは、GFPタグ付きCin8を用いた非常に堅牢で高感度な1分子運動性アッセイについて説明します。このアッセイの成功は、適切なMT重合および表面への固定化に大きく依存する。強力なアビジン - ビオチン相互作用は、MTを不可逆的に付着させる疎水性ガラス表面にMTを固定化するために利用される。GFP標識Cin8を用いたこれらの固定化MT上で、Cin8運動性を確実に追跡することができる11、12、19。
Cin8は、複数の四量体モーター10,12を含むクラスターを形成することが報告されており、これらのクラスターの運動性は単一のCin8分子の運動性とは異なる。Cin8の運動性をそのサイズの関数として正確に特徴付けるために、各Cin8粒子12のクラスターサイズを識別するために蛍光強度ベースの方法が開発されている。このサイズ分類に基づいて、運動性は各サイズカテゴリで別々に分析される。このサイズベースの分析に続いて、洞察に満ちた詳細が提供され、同じ分子11、12、19のオリゴマーの異なる挙動を理解するために利用することができる。ここで説明するクラスターサイズ決定手順は、種々の蛍光標識分子のサイズを決定するために適用することができる。蛍光ベースのサイズ決定を行う際には、大きなクラスターがフォトブリーチング後に小さなクラスターとして現れる可能性があるため、漂白の影響を避けるために、最初の出現フレームでCin8-GFP粒子のクラスターサイズを決定するように注意する必要があります。
運動性特性評価は、MDおよび/またはMSD分析によって行われる。モータ速度のみを求めることが対象となる場合は、MD解析で十分です。しかし、運動運動性が能動的成分と受動成分の両方を含み、拡散係数の決定も必要な場合は、MSD分析20、23、24、25を実行する必要があります。MD解析とMSD解析の両方で、各時点のモータの座標を決定する必要があります。効率的なトラッキングのためには、モーターの集中力を最適に保つことが重要です。MTはモーターで混雑しすぎてはいけません。理想的には、約10μmのMT上に一度に3〜4個のCin8-GFPモータ/パーティクルが存在する必要があります。ImageJ-Fijiの「KymoButler」や「TrackMate」プラグインなどの自動化されたツールも、運動性モーターを追跡するために使用できます26,27。これらの自動化ツールは時間と作業を節約しますが、いくつかの制限があります。たとえば、一部の粒子の運動性が非常に遅い場合、これらのツールはそれらを非運動性粒子として読み取ることができます。さらに、これらのツールは、低強度分子を認識するのに限界があります。したがって、それらは高強度バイアスを示すことができる。一方、手動追跡は(時間がかかりますが)追跡エラーの影響を受けにくくなります。
要約すると、このプロトコールは、 セレビシエ菌において過剰発現されたCin8の精製から始まり、この双方向キネシン−5の単一分子運動性アッセイおよびその後の運動性分析を包括的に説明する。このプロトコルは、Cin8などの運動タンパク質の運動性を精製および特徴付けるために容易に従うことができる。さらに、プロトコルの異なる部分は、酵母からタンパク質を精製したり、異なる運動タンパク質およびそれらの運動性特性評価のための単一分子運動性アッセイを開発するために適合させることができる。
開示事項
著者らは、開示する利益相反はありません。
謝辞
この研究は、L.G.に授与されたイスラエル科学財団助成金(ISF-386/18)とイスラエル二国間科学財団助成金(BSF-2019008)によって部分的に支援されました。
資料
| Name | Company | Catalog Number | Comments |
| Adenine | FORMEDIUM | DOC0230 | |
| ATP | Sigma | A7699 | |
| Biotinylated-BSA | Sigma | A8549 | |
| Casein | Sigma | C7078 | |
| Catalase (C40) | Sigma | C40 | |
| Creatine-Kinase | Sigma | C3755 | |
| Dithiothreitol (DTT) | Sigma | D0632 | |
| EDTA | Sigma | E5134 | |
| EGTA | Sigma | E4378 | |
| Fluorescence filter set for GFP | Chroma | 49002: ET-EGFP (FITC/Cy2) | |
| Fluorescence filter set for Rhodamine | Chroma | 49004: ET-CY3/TRITC | |
| Fluorescence inverted microscope | Zeiss | Axiovert 200M | |
| Galactose | Tivan Biotech | GAL02 | |
| Glucose | Sigma | G8270 | |
| Glucose Oxidase | Sigma | G7141 | |
| Glycerol | Sigma | G5516 | |
| GlycylGlycine | Merck | G0674 | |
| GMPCPP | Jana Bioscience | Nu-405L | |
| GTB | Cytoskeleton | BST01-010 | |
| GTP | Sigma | G8877 | |
| Histidine | Duchefa Biochemie | H0710.0100 | |
| ImageJ-FIJI software | https://imagej.net/plugins/trackmate/ | version 2.1.0/1.53c; Java 1.8.0_172 [64-bit] for Windows 10 | |
| Imidazole | Sigma | I0125 | |
| InstantBlue Coomassie Protein Stain | Abcam | ab119211 | |
| Lens | Zeiss | 100x/1.4 oil DIC objective | |
| Lysine | FORMEDIUM | DOC0161 | |
| Magnesium Chloride | Sigma | M8266 | |
| Methionine | Duchefa Biochemie | M0715.0100 | |
| Neo | Andor Technologies | sCMOS camera | |
| NeutraAvidin | Life | A2666 | |
| Ni-NTA Agarose | Invitrogen | R901-15 | |
| Phospho-Creatine | Sigma | P1937 | |
| Pipes | Sigma | P1851 | |
| Pluronic acid F-127 (poloxamer) | Sigma | P2443 | |
| Potassium Chloride | Sigma | P9541 | |
| Raffinose | Tivan Biotech | RAF01 | |
| Size Exclusion chromatography instument | GE Healthcare | AKTA Pure | |
| Spectrophotometer | ThermoFisher Scientific | NanoDrop | |
| Superose-6 10/300 GL | GE Healthcare | 17-5172-01 | |
| Tris | Roshe | 10708976001 | |
| Triton X-100 | Sigma | T8787 | |
| Tryptophan | Duchefa Biochemie | T0720.0100 | |
| Tubulin protein | Cytoskeleton | T240 | |
| Tubulin, biotinylated | Cytoskeleton | T333P | |
| Tubulin, TRITC Rhodamine | Cytoskeleton | TL530M | |
| Uracil | Sigma | U0750-100G | |
| Yeast nitrogen base | FORMEDIUM | CYN0401S | |
| α-GFP antibody | Santa Cruz Biotechnology | SC8036 | |
| β-mercaptoethanol | Sigma | M3148 |
参考文献
- Singh, S. K., Pandey, H., Al-Bassam, J., Gheber, L. Bidirectional motility of kinesin-5 motor proteins: structural determinants, cumulative functions and physiological roles. Cellular and Molecular Life Sciences. 75 (10), 1757-1771 (2018).
- Hoyt, M. A., He, L., Totis, L., Saunders, W. S. Loss of function of Saccharomyces cerevisiae kinesin-related CIN8 and KIP1 is suppressed by KAR3 motor domain mutations. Genetics. 135 (1), 35-44 (1993).
- Saunders, W. S., Hoyt, M. A. Kinesin-related proteins required for structural integrity of the mitotic spindle. Cell. 70 (3), 451-458 (1992).
- Hoyt, M. A., He, L., Loo, K. K., Saunders, W. S. Two Saccharomyces cerevisiae kinesin-related gene products required for mitotic spindle assembly. Journal of Cell Biology. 118 (1), 109-120 (1992).
- Gerson-Gurwitz, A., et al. Mid-anaphase arrest in S. cerevisiae cells eliminated for the function of Cin8 and dynein. Cellular and Molecular Life Sciences. 66 (2), 301-313 (2009).
- Fridman, V., Gerson-Gurwitz, A., Movshovich, N., Kupiec, M., Gheber, L. Midzone organization restricts interpolar microtubule plus-end dynamics during spindle elongation. EMBO Reports. 10 (4), 387-393 (2009).
- Movshovich, N., et al. Slk19-dependent mid-anaphase pause in kinesin-5-mutated cells. Journal of Cell Science. 121 (15), 2529-2539 (2008).
- Gerson-Gurwitz, A., et al. Directionality of individual kinesin-5 Cin8 motors is modulated by loop 8, ionic strength and microtubule geometry. Embo Journal. 30 (24), 4942-4954 (2011).
- Roostalu, J., et al. Directional switching of the kinesin Cin8 through motor coupling. Science. 332 (6025), 94-99 (2011).
- Shapira, O., Goldstein, A., Al-Bassam, J., Gheber, L. A potential physiological role for bi-directional motility and motor clustering of mitotic kinesin-5 Cin8 in yeast mitosis. Journal of Cell Science. 130 (4), 725-734 (2017).
- Goldstein-Levitin, A., Pandey, H., Allhuzaeel, K., Kass, I., Gheber, L. Intracellular functions and motile properties of bi-directional kinesin-5 Cin8 are regulated by neck linker docking. eLife. 10, 71036 (2021).
- Pandey, H., et al. Drag-induced directionality switching of kinesin-5 Cin8 revealed by cluster-motility analysis. Science Advances. 7 (6), 1687 (2021).
- Pandey, H., Popov, M., Goldstein-Levitin, A., Gheber, L. Mechanisms by which kinesin-5 motors perform their multiple intracellular functions. International Journal of Molecular Sciences. 22 (12), 6420 (2021).
- Fridman, V., et al. Kinesin-5 Kip1 is a bi-directional motor that stabilizes microtubules and tracks their plus-ends in vivo. Journal of Cell Science. 126, 4147-4159 (2013).
- Edamatsu, M. Bidirectional motility of the fission yeast kinesin-5, Cut7. Biochemical and Biophysical Research Communications. 446 (1), 231-234 (2014).
- Popchock, A. R., et al. The mitotic kinesin-14 KlpA contains a context-dependent directionality switch. Nature Communications. 8, 13999 (2017).
- Bodrug, T., et al. The kinesin-5 tail domain directly modulates the mechanochemical cycle of the motor domain for anti-parallel microtubule sliding. eLife. 9 (9), 51131 (2020).
- Gheber, L., Kuo, S. C., Hoyt, M. A. Motile properties of the kinesin-related Cin8p spindle motor extracted from Saccharomyces cerevisiae cells. Journal of Biological Chemistry. 274 (14), 9564-9572 (1999).
- Pandey, H., et al. Flexible microtubule anchoring modulates the bi-directional motility of the kinesin-5 Cin8. Cellular and Molecular Life Sciences. 78 (16), 6051-6068 (2021).
- Shapira, O., Gheber, L. Motile properties of the bi-directional kinesin-5 Cin8 are affected by phosphorylation in its motor domain. Scientific Reports. 6, 25597 (2016).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Britto, M., et al. Schizosaccharomyces pombe kinesin-5 switches direction using a steric blocking mechanism. Proceedings of the National Academy of Sciences of the United States of America. 113 (47), 7483-7489 (2016).
- Kapitein, L. C., et al. Microtubule cross-linking triggers the directional motility of kinesin-5. Journal of Cell Biology. 182 (3), 421-428 (2008).
- Furuta, K., Edamatsu, M., Maeda, Y., Toyoshima, Y. Y. Diffusion and directed movement in vitro motile properties of fission yeast kinesin-14 Pkl1. Journal of Biological Chemistry. 283 (52), 36465-36473 (2008).
- Katrukha, E. A., et al. Probing cytoskeletal modulation of passive and active intracellular dynamics using nanobody-functionalized quantum dots. Nature Communications. 8, 14772 (2017).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Jakobs, M. A. H., Dimitracopoulos, A., Franze, K. KymoBulter, a deep learning software for automated kymograph analysis. eLife. 8, 42288 (2019).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved