Method Article
ヒト神経変性におけるヌクレオポリン変化を調べるための核単離と超解像構造化照明顕微鏡
要約
このプロトコルは、人工多能性幹細胞由来ニューロンおよび死後ヒト組織の核質およびNPC内の個々のヌクレオポリンを評価するための、核単離および超解像構造化照明顕微鏡法のための最適化されたワークフローを説明しています。
要約
核孔複合体(NPC)は、~30種類のヌクレオポリンタンパク質(Nup)の複数のコピーからなる複雑な高分子構造です。これらのNupは、ゲノム組織化、遺伝子発現、核細胞質輸送(NCT)を調節する機能を果たしています。近年、筋萎縮性側索硬化症(ALS)、アルツハイマー病(AD)/前頭側頭型認知症(FTD)、ハンチントン病(HD)など、複数の神経変性疾患において、NCTの欠損や特定のNupsの変化が早期に顕著な病態として特定されています。光学顕微鏡と電子顕微鏡の両方の進歩により、NPCとそのNup成分を含む細胞内構造を、より高い精度と分解能で徹底的に調べることが可能になりました。一般的に使用される技術のうち、超解像構造化照明顕微鏡(SIM)は、従来の抗体ベースの標識戦略を使用して個々のNupの局在と発現を研究する比類のない機会を提供します。SIMの前に核を単離することで、NPCおよび核質内の個々のNupタンパク質を完全かつ正確に再構築された3D空間で可視化することができます。このプロトコルは、ヒトiPS細胞由来CNS細胞および死後組織におけるNupの発現と分布を評価するための核単離およびSIMの手順を説明しています。
概要
加齢に伴う神経変性疾患の有病率は、人口の高齢化とともに増加しています1.遺伝的基盤と病理学的特徴は十分に特徴付けられていますが、ニューロンの損傷につながる正確な分子イベントは十分に理解されていません2,3,4,5,6,7,8,9,10,11,12.最近では、G4C2 C9orf72遺伝子の第1イントロンにおけるヘキサヌクレオチド反復拡大は、関連する神経変性疾患である筋萎縮性側索硬化症(ALS)および前頭側頭型認知症(FTD)の最も一般的な遺伝的原因として同定されました13,14.現在、いくつかの研究は、核孔複合体(NPC)や核輸送受容体(NTR、カリオフヘリン)などの核輸送機構の混乱がC9orf72 ALSの原因であるとして中心的な役割を果たしていることを支持しています15,16.ラットの脳内の非分裂細胞では、足場ヌクレオポリン(Nups)は非常に長寿命です。その結果、老化に伴うNPCやNCTの変化が報告されています17,18,19,20.さらに、一部のヌクレオポリンまたはトランスポーチンは、変異すると、特定の神経疾患に関連しています21,22.例えば、Nup62の変異は、尾状核と被殻に影響を与える神経疾患である乳児両側性線条体壊死(IBSN)に関連しています23;Gle1の変異は、胎児運動ニューロン疾患であるヒト致死性先天性拘縮症候群-1(LCCS1)に関与しています24;アラジンの突然変異はトリプルA症候群の原因です25.機能的NCTの変化は、ALS、ハンチントン病(HD)、アルツハイマー病(AD)などの加齢性神経変性疾患で悪化します16,26,27,28,29,30,31.さらに、特定のNupおよびNTRは、C9orf72を介した毒性の修飾因子として報告されています。 Drosophila 目28 または、FUSやタウなどの疾患関連タンパク質の凝集状態を生化学的に修飾します27,32,33,34.まとめると、これらの初期の研究は、NCTの変化がALSおよびFTDの主要および初期の病理学的特徴である可能性があることを示唆しています。過剰発現に基づくモデルシステムに関する研究では、特定のNupsおよびカリオフェリンの誤った局在化がNCTに影響を与える可能性があることが示唆されています16,35,36,37,38.しかし、これらの病理学的研究は、NPCタンパク質の細胞質蓄積をNPCの構造や機能の欠陥に実際には関連付けていません。例えば、この病理は、NPCの組成および機能にほとんど影響を与えない、Nupタンパク質の細胞質プールの調節不全を単に反映している可能性があります。対照的に、超解像構造化照明顕微鏡(SIM)を用いた最近の研究では、核質内の特定のNupレベルの低下を特徴とするNPC自体への重大な損傷の出現を示しています ヒトC9orf72 ALS/FTDニューロンのNPCは、最終的にNPC機能の変化につながります 病原性疾患カスケードの早期開始イベントとして15.
核と細胞質の間の高分子の通過は、核孔複合体(NPC)によって支配されています。NPCは、30のヌクレオポリンタンパク質(Nups)39,40,41の複数のコピーからなる核膜に埋め込まれた大きな高分子複合体である。Nut化学量論は細胞タイプによって異なるが、NCT、ゲノム組織化、および全体的な細胞生存率39,41,45,46にとって、全体的なNPC組成の維持は重要である。その結果、NPC組成の変化とそれに続く機能輸送の欠陥は、無数の下流の細胞機能に影響を与える可能性があります。NPCのNup成分は、細胞質リングとフィラメント、中央チャネル、外輪、内輪、膜貫通リング、核バスケットなど、複数のサブコンプレックスに高度に組織化されています。全体として、内輪、外輪、および膜貫通輪の足場Nupsは、核膜内にNPCを固定し、細胞質リング、中央チャネル、および核バスケットのNupsのアンカーポイントを提供します。低分子(<40-60 kD)はNPCを介して受動的に拡散することができますが、より大きな貨物の能動的な輸送は、核輸送受容体(NTR、カリオフヘリン)と細胞質フィラメント、中央チャネル、および核バスケット39,40,41,45のFG Nupsとの間の相互作用によって促進されます。また、一握りのNupsは、NPCの外側、核質内でさらに機能し、遺伝子発現を調節することができる46,47。
単一のヒトNPCの横方向の寸法が約100〜120nmであることを考えると40、標準的な広視野顕微鏡または共焦点顕微鏡法は、個々のNPC48を解決するには不十分である。TEMやSEMなどの電子顕微鏡(EM)技術は、NPCの全体構造を評価するためによく使用されます39,40。NPCの超微細構造を分離するためのこれらの技術の利点にもかかわらず、NPC内の個々のNupタンパク質の存在を評価するためにあまり一般的に使用されていません。抗体またはタグベースの標識をこれらの最先端技術であるTEMおよびSEMと組み合わせることの技術的な制限により、NPCまたは核質内の個々のNup自体を正確かつ確実に評価できるとは限りません。さらに、これらの手法は技術的に困難な場合があり、すべての研究者が広く利用できるわけではありません。しかし、近年の光学顕微鏡や蛍光顕微鏡の進歩により、超解像イメージング技術の利用可能性が高まっています。具体的には、SIMは、1人の人間のNPCの横方向の寸法40,48,49,50,51に近い解像度で個々のNupを画像化する比類のない機会を提供します。確率的光学再構成顕微鏡法(STORM)や誘導放出減少(STED)などの他の超解像アプローチとは対照的に、SIMは従来の抗体ベースの免疫染色と互換性がある49。したがって、SIMは、特定のNup抗体が利用可能なすべてのNupの包括的な分析を可能にします。同じ調製物で複数の異なるNupをサンプリングおよびイメージングする能力は、NPCを構成する多くのタンパク質を調査する際に、他のイメージング方法に大きな利点をもたらします。次の手順では、人工多能性幹細胞(iPSC)由来ニューロン(iPSN)および死後ヒト中枢神経系(CNS)組織から単離された核を使用して、NPCの個々のNupコンポーネントを評価するための最適化されたプロトコルについて詳しく説明します。
プロトコル
iPS細胞の作製および剖検組織採取のためのすべての血液サンプルは、ジョンズ・ホプキンス大学の倫理監督のもと、ジョンズ・ホプキンス大学IRBによって承認されています。すべての患者情報はHIPPAに準拠しています。以下のプロトコルは、ジョンズ・ホプキンス大学のすべてのバイオセーフティー手順に準拠しています。
1. 免疫染色およびイメージング用スライドの調製
- 正に帯電したガラス顕微鏡スライドを空のサイトファンネルに配置し、疎水性バリアペンで円を描き、核を堆積させる領域の輪郭を描きます。
- ステップ1.1で描いた円の中心に1 mg/mLのコラーゲン溶液50 μL(1x PBSで希釈)を加え、室温で5分間インキュベートします。
- コラーゲン溶液を吸引し、スライドを室温で約1時間風乾させます。
2. 溶解緩衝液およびスクロースグラジエントの調製
- 核分離キットのプロトコルに従って、溶解バッファーとショ糖勾配溶液を調製します。
- 各サンプルについて、付属の溶解バッファー11 mL、付属の10% Triton X-100溶液110 μL、および調製または解凍したばかりの1 M DTT(ジチオスレイトール)11 μLを組み合わせて、溶解バッファーの50 mLコニカルチューブを調製します。
- 各サンプルについて、50 mLのコニカルチューブを使用して、付属の2 Mショ糖クッション溶液27.75 mL、付属のショ糖クッションバッファー2.25 mL、および新たに調製または解凍した1 M DTT30 μLを組み合わせて、1.85 Mショ糖クッション溶液を調製します。
注:1.85 Mショ糖クッション溶液は、iPSNおよび死後ヒトCNS組織に最適ですが、他の細胞タイプや組織サンプルに合わせて調整してください。HEK293細胞、iPS細胞由来アストロサイト、および死後CNS組織からのオリゴデンドロサイト核の濃縮に関する追加の詳細が最近発表されました15。
- 溶解バッファーとショ糖クッション溶液を円錐形のチューブを反転させて穏やかに混合し、氷上に保存します。
3. iPSNおよび死後ヒトCNS組織の溶解
- 溶解を進める前に、超遠心分離機のローター(サンプルホルダーなし)を超遠心分離機に入れ、4°Cに予冷します。
- iPSNの溶解。
注:溶解プロトコルは、iPSNと死後のヒトCNS組織で異なります。以下に指定されているサンプルタイプ(ステップ3.2:iPSNs、ステップ3.3:死後ヒトCNS組織)のプロトコルに従ってください。- インキュベーターからiPSNを取り出し、培地を吸引します。1x PBSで短時間すすぎ、溶解バッファーをウェルまたはプレートに直接加えます。
注:iPSNは、破片や死んだ細胞を取り除くために、十分な量の1x PBSで適切にすすぐように注意してください。iPSNプレートに添加する溶解バッファーの量は異なります。 表1を参照してください。出発物質が>250万iPSNであることを確認してください。 - 細胞スクレーパーでiPSNを掻き取り、50 mLコニカルチューブ内の残りの溶解バッファーに移します。
- 各コニカルチューブにキャップをし、サンプルを20秒間ボルテックスして、iPSN溶解を促進します。
- 先に進む前に、サンプルを氷の上に1〜2分間置いてください。
- インキュベーターからiPSNを取り出し、培地を吸引します。1x PBSで短時間すすぎ、溶解バッファーをウェルまたはプレートに直接加えます。
- 死後ヒトCNS組織の溶解。
- ドライアイスのベッドに置いたシャーレでカミソリの刃で切断することにより、凍結死後のヒトCNS組織の~100 mgを秤量します。局所的な組織汚染を避けるために、必ず使い捨てのパッドで囲まれた表面で作業してください。組織解剖時に見える特定の灰白質と白質の解剖(例:皮質マントルピースと下にある白質)に注意してください。
注:凍結死後のヒト組織サンプルを取り扱うときは、目の保護具や手袋など、適切なPPEを着用してください。このステップは、-80°Cで保存された組織アリコートを事前に完了することもでき、50 mgは核の単離に十分な量です。しかし、100 mgを超える組織では、一部の核の周囲に無傷の細胞質が存在することからも明らかなように、核の分離が不完全になることがよくあります。 - Dounceホモジナイザーは、蒸留水で洗浄し、十分にすすいで調製します。ホモジナイザーを氷の上に置いて冷やします。
- 新たに洗浄し、すすぎ、冷やした(氷上)2 mLの調製した溶解バッファーを含むDounceホモジナイザーに100 mgのティッシュを加えます。
- Dounce ホモジナイザーで、氷上での標準的な手順を使用して均質化します。
注:通常、乳棒あたり10〜20ストロークで、抵抗なくサンプル内を移動するのに十分です。 - 2 mLの死後ヒト脳ホモジネートを、コニカルチューブ内の残りの9 mLの溶解バッファーに移します。
- 30秒間激しく渦巻き、溶解を促進するために氷の上に5分間置いてください。
- ドライアイスのベッドに置いたシャーレでカミソリの刃で切断することにより、凍結死後のヒトCNS組織の~100 mgを秤量します。局所的な組織汚染を避けるために、必ず使い捨てのパッドで囲まれた表面で作業してください。組織解剖時に見える特定の灰白質と白質の解剖(例:皮質マントルピースと下にある白質)に注意してください。
4. iPSNおよび死後ヒトCNS組織からの核の単離
- 1.85 Mショ糖クッション溶液(ステップ2.1.2で作製)10 mLを超遠心チューブの底に重ね、各ライセートに1.85 Mショ糖クッション溶液18 mLを加えます。
- 50 mLのコニカルチューブを反転させて、ライセート/ショ糖クッション溶液(ステップ4.1で組み合わせたもの)を穏やかに混合します。
- 超遠心チューブ内の1.85 Mショ糖クッション溶液10 mLの上部に、28 mLのライセート/ショ糖クッション溶液ミックスをゆっくりと加えます(ステップ4.1から)。
注:50mLのコニカルチューブには、合計29mLのライセート/ショ糖クッション溶液混合物があります(ステップ4.1-4.2)。このライセート/スクロースクッション溶液ミックス1mLを50mLのコニカルチューブに残します。これにより、ショ糖クッション溶液の粘度により、すべてのサンプル間で正確で一貫したピペッティングが保証されます。 - 超遠心チューブをホルダーに入れ、必要に応じて1.85 Mショ糖クッション溶液(ステップ2.1.2で作成)を追加して、サンプルのバランスを取ります。
- サンプルホルダーを冷却した超遠心分離機のローターに入れ、30,000 x g、4°Cで45分間回転させます。
- サンプルホルダーから超遠心チューブを取り外し、上清を捨てます。核は、超遠心チューブの底面にペレットとして表示されます。
- ボルテックスにより、供給した核保存バッファー1 mLに核ペレットを再懸濁し、微量遠心チューブに移します。
- 微量遠心チューブを2,500 x g、4°Cで5分間遠心分離します。
- 上清を除去し、供給した新しい1 mLの核保存バッファーでボルテックスして核を再懸濁します。
- 免疫染色を続行するか、核を-80°Cで保存します。
注:核は-80°Cで最大6か月間保存でき、構造的完全性が損なわれる前に2回の凍結/融解サイクルにかけることができます。
5. 単離された核の免疫染色
- 核懸濁液の10 μLアリコートを採取し、血球計算盤または自動セルカウンターを使用してカウントします。
- 調製したスライド(ステップ1.3)をサイトファンネルに組み立てます。
- 各サイトファンネルに、200 μL の新鮮な核保存バッファーと ~100,000 核を層状にします。
- サイトファンネルをサイトスピンに入れ、100 x gで3分間遠心分離することにより、核をスライド上に穏やかに回転させます。
- サイトファンネルを抜き、直ちに~100 μLの4% PFA(パラホルムアルデヒド)をスライドに加え、室温で15分間インキュベートします。
注:核はメタノールで固定することもできます。各抗体に適した固定方法を使用してください。メタノールを使用する場合は、透過化ステップ(5.7)をスキップしてください。サイトファンネルは使い捨てです。この時点でそれらを破棄します。 - 核を1x PBSで5分間3回洗浄します。
- 0.1% Triton X-100 を 1x PBS 中で室温で 10 分間、核を透過処理します。
- ブロック溶液(1x PBS中の10%ヤギまたはロバの血清)で核を室温で30分間ブロックします。
- ブロック溶液で希釈した一次抗体と核をインキュベートし、4°Cで一晩保存します。
- 核を1x PBSで5分間3回洗浄します。
- ブロック溶液で希釈した二次抗体と核を室温で1時間インキュベートします。
注:Alexa Fluor 488、568、および647二次抗体が推奨されます。 - 核を1x PBSで5分間3回洗浄します。
- オプション:DAPIまたはHoechstで核を5分間インキュベートした後、さらに1x PBSで5分間を2回洗浄します。
- 核を含む円の端に糸くずの出ないワイプを持って、最後のPBS洗浄を完全に取り除きます。
- 各スライドに1滴(~10 μL)のハードマウント退色防止封入剤(DAPIなし)を追加し、各スライドに高許容範囲の18 mm x 18 mm角のカバースリップをそっと置きます。
注意:ハードマウントメディアを使用する場合、スライドが適切な時間枠内で画像化されていれば、スライドシーリングは必要ありません。Nupの免疫反応性は、通常、4°Cで~6ヶ月間保存した未密封スライド上で安定しています。ただし、ウェットマウントメディアを使用する場合や長期保管する場合は、カバースリップの端をマニキュアの薄い層で密封できます。 - スライドを光から保護し、室温で一晩硬化させます。
- 原子核を超解像構造化照明顕微鏡(SIM)でイメージングできます。
注:Zeiss、Nikon、GE HealthcareはすべてSIM顕微鏡を製造しています。画像の取得と処理については、システム固有のプロトコルに従ってください。各Nupおよび蛍光色素に適したイメージングパラメータ(レーザー出力、フィルターセット、露光時間)を使用してください。イメージングするときは、これらの核は通常、遠心分離ステップ(5.4)の結果として平らにされ、広がるため、円の端(ステップ1.1から)を避けてください。完全にデコンボリューションおよび処理された画像は、FIJIまたはImaris画像分析ソフトウェアの標準3D分析モジュールを使用したスポット検出や体積測定など、さまざまな分析を行うことができます。分析方法に関する追加の詳細は最近発表されました15。
結果
ヒト神経核におけるPOM121のNPCおよび核質分布および発現を調べるために、コントロールおよびC9orf72 iPSNを前述の通りに分化させた15。死後、ヒト運動皮質および32日目のiPSNを溶解し、上記のように核単離および免疫染色を行った。NeuN正の単離核を、超解像構造化照明顕微鏡(Zeiss)を用いた超解像構造化照明顕微鏡(SIM)でイメージングし、前述した15 のデフォルトの構造化照明デコンボリューションパラメータを用いて処理した(ワークフローについては 図1 も参照)。画像は、63倍の油浸対物レンズ、1倍の倍率、1%の出力のHRダイオード448レーザー、100msの露光時間、BP495-550 / LP750発光フィルター、厚さ110 nmのzセクション、および5つのグリッド回転を使用して取得されました。 図2の3D最大強度予測に示されているように、個々のPOM121スポットはiPSNと死後ニューロン核の両方で分離されました。これらの画像は、その後、前述のように、FIJIまたはImarisの3Dスイートプラグインを使用してスポット検出にかけられました15。先行解析15と同様に、この方法論は、対照と比較して、C9orf72 iPSNおよび死後運動皮質核におけるPOM121スポット数の大幅な減少を検出するのに十分であった(図2)。

図1:核単離、免疫蛍光染色、超解像イメージング、および解析ワークフローの概略図。この図の拡大版を見るには、ここをクリックしてください。
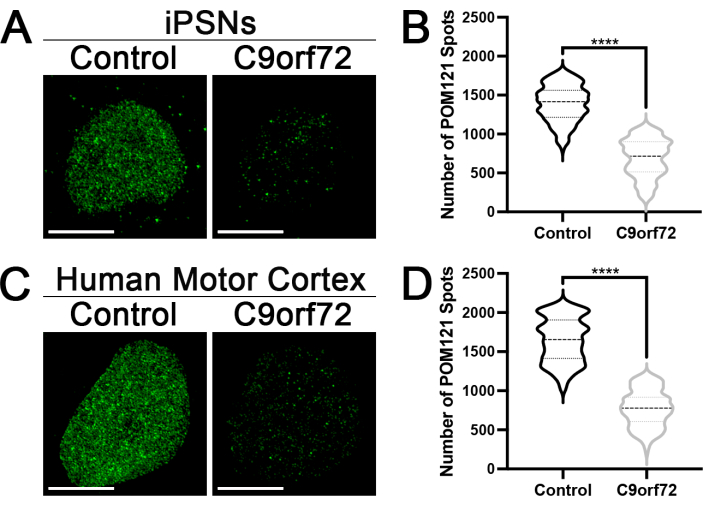
図2:代表的な構造化照明顕微鏡(SIM)の画像と定量。 (A-B)コントロールおよびC9orf72 iPSNから分離されたNeuN+核のPOM121スポットの最大強度投影SIM画像とPOM121スポットの定量化。n = 3コントロールおよび3つのC9orf72 iPS細胞系統、1ラインあたり50核。スチューデントのt検定を使用して統計的有意性を計算しました。p < 0.0001 です。(C-D)コントロールおよびC9orf72死後運動皮質組織から分離されたNeuN+核のPOM121スポットの最大強度投影SIM画像と定量化。n = 3コントロールおよび3つのC9orf72患者ケース、ケースあたり50核。スチューデントのt検定を使用して統計的有意性を計算しました。p < 0.0001 です。スケールバー = 5 μm。統計解析を行い、iPS細胞株当たり50核の平均スポット数を用いて、前述の15の統計的有意性を算出した。この図の拡大版を表示するには、ここをクリックしてください。
| 培養容器 | Lysis Bufferのボリューム |
| 12ウェルプレート | ウェルあたり0.5 mL |
| 6ウェルプレート | ウェルあたり1 mL |
| 10cm皿 | 3ミリリットル |
| T25フラスコ | 2ミリリットル |
表1:溶解バッファーのボリューム。この表には、iPSNsまたは異なる培養容器で増殖した培養細胞の溶解に使用する溶解バッファーの量の情報が含まれています。
ディスカッション
NCT欠損が複数の神経変性疾患16,27,28,30,31における初期の顕著な現象として最近特定されたことを考えると、この病状が発生するメカニズムを徹底的に調査する重要な必要性が存在します。NPCとその個々のNupタンパク質は機能的なNCT39,41を決定的に制御しているため、神経変性疾患モデルにおけるNPC組成の調査は不可欠です。しかし、標準的な広視野顕微鏡および共焦点顕微鏡法は、個々のNPC40,48,49,51を解明するには不十分である。したがって、今日まで、単一のNPCの解像度でのNupsの分析は、日常的な実験的アプローチとして採用されていませんでした。
近年、SIMなどの超解像イメージング技術が身近なものになってきています。これらの方法論は、従来の免疫蛍光染色と高度な画像処理およびデコンボリューションを組み合わせて、個々のNPC40,48,49,51を分離するのに十分な高い分解能を提供します。標準的な免疫染色プロトコルと互換性があるため、研究者は特定の抗Nup抗体が利用可能な任意のNupを高解像度48,49,50で検出することができますが、以下に概説するように、SIMには技術的な課題や制限がないわけではありません。
超解像イメージング実験の成功は、サンプルの品質に大きく依存します。具体的には、SIMの場合、不完全な核の単離やサンプルの過剰溶解は、細胞質や細胞膜の保持、または核の断片化または崩壊して縮むことにつながる可能性があります。この問題を解決するために、それに応じて溶解時間を調整する必要があります。上記のプロトコルは、iPSNおよび死後ヒトCNS組織に最適化されていますが、さまざまな細胞タイプや組織から高品質の核を作製するために簡単に修正できます。これは、ショ糖グラジエントの濃度および/またはサンプル溶解時間と方法を調整することによって達成されます。HEK293細胞、iPS細胞由来アストロサイト、および死後ヒトCNS組織からのオリゴデンドロサイト核の濃縮に関するショ糖勾配調整に関する追加の詳細が最近発表されました15。
核単離のプロセス自体が、NPCと一過性に結合する不安定なNupまたはNTRの関連を混乱させる可能性があります。その結果、単離された核のSIMイメージングから得られた結果を検証するために、研究者は、以前に説明したように、免疫染色や無傷の細胞または組織の共焦点イメージングなどの追加の技術を採用することを検討すべきである15。しかし、Nupのルーチン共焦点イメージングは、個々のNPCスポット48 を分離するための分解能を提供しず、したがって、全体的なNup強度に関する半定量的な情報しか提供できないことに留意されたい。細胞質プール52、53、54、55内にも通常存在するNupについては、全細胞ライセートからのウェスタンブロット分析は、細胞質に局在する可能性のあるNupからの汚染のために、NPC組成の検証方法として推奨されない。しかし、単離された核サンプルからのウェスタンブロット分析は、イメージング技術から得られたデータを検証するために採用されてきました15。
SIM画像の解像度は、特定のイメージングシステム51の個々の点像分布関数に応じて、蛍光色素間で異なり得る。これは、先に進む前に、各サンプルタイプについてユーザーが実験的にテストする必要があります。SIMイメージングは、厚さが10〜15μm48,51を超えるサンプルには理想的ではありません。その結果、核の単離により、ヒト核の全厚のイメージングが可能になり、Nupスポットとボリュームの最も正確な3D再構成と定量が可能になります。ただし、核の厚さが>15μmのままの場合は、Airy Scanイメージングを代わりに使用できます。分解能は低下しますが、Airy Scanイメージングはサンプルの厚さ56によって制限されません。
また、図2に示す画像は、SIMによって画像化された個々の原子核の3D再構成後の最大強度投影を表していることも考慮する必要があります。したがって、このような画像では、核質内のスポットとは対照的に、NPCに真に関連するスポットを正確に区別することは困難です。そのため、3D画像を解析する際には、各zセクションまたはzセクションの小さなシリーズを個別に解析してセグメント化し、核質ではなく核膜に関連するスポットを区別することが重要です。図2に示す定量は、分析された核の外側の面に沿って検出されたスポットのみを表しています。言い換えれば、代表定量は、核膜に関連するPOM121スポットの数を示します。
最後に、核の不均一性と変動性15,42を考慮すると、採用する超解像イメージング方法に関係なく、サンプルごとに多数の核をイメージングおよび評価することが重要です。
バルクプロテオミクスやウェスタンブロットアッセイとは対照的に、イメージング法は、特定の細胞タイプまたは核マーカー(NeuN、Olig2など)によって同定されたヒトCNS細胞のサブセット内のNupの分布と発現を調べる比類のない機会を提供します。SIMは、NPCとその個々のNup成分を研究するために採用することができる多くの超解像イメージング技術の1つである50,57,58。各方法論(SIM、STORM、EM)は、NPCの構造と組成について独自の洞察をもたらします。SIMは、抗Nup抗体が利用可能な各Nupを単一のNPCの分解能で評価することができるのに対し、STORMは個々のNPCのオクテット構造を分離することができ、EM技術は、NPC全体の構造を高解像度で詳細に把握することができる50。NPC内の個々のNupを評価するためのSTORMおよびImmuno EMアプローチは、技術的に困難です。具体的には、STORMは、従来の抗体ベースの染色による立体障害を克服するために、内因性蛍光タグの使用を必要とすることがよくあります。今日まで、この技術によって画像化された非ニューロン細胞株のNupはほんの一握りです。さらに、従来、STORMは原子核の単一表面上にNPCを画像化していたため、空間分布とNPCの不均一性を評価するための全原子核の完全な3D再構成の機会が排除されていた50。その結果、SIMは、NPCおよび核質内のNupを高分解能で堅牢に評価するための好ましい光学顕微鏡法です。最近の研究では、SEMとSIMの両方の技術を使用して、全体的なNPC構造は損なわれていないが、特定のNupsはC9orf72 ALS / FTDニューロン核15のNPCと核質から減少したと結論付けました。この研究は、複数の高解像度イメージングアプローチを組み合わせて、NPCの組成と構造、および23の個々のヒトNupsを調べるためのSIMの強度に関する新しい洞察をもたらすことの決定的な重要性を強調しています。
開示事項
著者は、競合する金銭的利益を宣言しません。
謝辞
死後ヒトCNS組織は、ジョンズホプキンスALS剖検バンクおよびTarget ALS Postmortem Tissue Coreによって提供されました。この研究は、ALSA Milton Safenowitz Postdoctoral Fellowship(ANC)の支援を受けたほか、NIH-NINDS、国防総省、ALS協会、筋ジストロフィー協会、F Prime、Robert Packard Center for ALS Research Answer ALS Program、Chan Zuckerberg Initiativeからの資金提供を受けました。
資料
| Name | Company | Catalog Number | Comments |
| 50 mL conical tubes | Fisher Scientific | 14-959-49A | |
| Beckman Ultracentrifuge | Beckman Coulter | ||
| Cell Scrapers | Sarstedt | 83.183 | |
| Collagen | Advanced Biomatrix | 5005 | |
| Coverslips | MatTek | PCS-170-1818 | |
| Cytofunnel | Thermo Fisher Scientific | A78710020 | |
| Cytospin 4 | Fisher Scientific | A78300003 | |
| Dounce Homogenizers | DWK Life Sciences | 357542 | |
| DTT | Sigma Aldrich | D0632 | |
| Eppendorf tubes | Fisher Scientific | 05-408-129 | |
| Goat Anti-Chicken Alexa 647 | Thermo Fisher Scientific | A-21449 | |
| Goat Anti-Mouse Alexa 488 | Thermo Fisher Scientific | A-11029 | |
| Goat Anti-Mouse Alexa 568 | Thermo Fisher Scientific | A-11031 | |
| Goat Anti-Mouse Alexa 647 | Thermo Fisher Scientific | A-21236 | |
| Goat Anti-Rabbit Alexa 488 | Thermo Fisher Scientific | A-11034 | |
| Goat Anti-Rabbit Alexa 568 | Thermo Fisher Scientific | A-11036 | |
| Goat Anti-Rabbit Alexa 647 | Thermo Fisher Scientific | A-21245 | |
| Goat Anti-Rat Alexa 488 | Thermo Fisher Scientific | A-11006 | |
| Goat Anti-Rat Alexa 568 | Thermo Fisher Scientific | A-11077 | |
| Goat Anti-Rat Alexa 647 | Thermo Fisher Scientific | A-21247 | |
| Hemacytometer | Fisher Scientific | 267110 | |
| Microscope Slides | Fisher Scientific | 12-550-15 | |
| Normal Goat Serum | Vector Labs | S-1000 | |
| Nuclei PURE Prep Nuclei Isolation Kit | Sigma Aldrich | NUC201 | Contains Lysis Buffer, 10% Triton X-100, 2 M Sucrose Gradient, Sucrose Cushion Solution, and Nuclei Storage Buffer; Referenced in protocol as "nuclei isolation kit" |
| PBS | Thermo Fisher Scientific | 10010023 | |
| PFA | Electron Microscopy Sciences | 15714-S | |
| Prolong Gold Antifade | Invitrogen | P36930 | Referenced in protocol as "hard mount antifade mounting media" |
| SW 32 Ti Ultracentrifuge Rotor | Beckman Coulter | 369694 | Referenced in protocol as "ultracentrifuge rotor" |
| Triton X-100 | Sigma Aldrich | T9284 | |
| Trypan Blue | Thermo Fisher Scientific | 15-250-061 | |
| Ultracentrifuge Tubes | Beckman Coulter | 344058 | |
| Nucleoporin Primary Antibodies | Primary antibodies suitable for immunofluorescent detection of invidual nucleoporins are available from multiple companies |
参考文献
- Hou, Y., et al. Ageing as a risk factor for neurodegenerative disease. Nature Reviews. Neurology. 15 (10), 565-581 (2019).
- Kim, G., Gautier, O., Tassoni-Tsuchida, E., Ma, X. R., Gitler, A. D. ALS genetics: Gains, losses, and implications for future therapies. Neuron. 108 (5), 822-842 (2020).
- Bang, J., Spina, S., Miller, B. L. Frontotemporal dementia. Lancet. 386 (10004), 1672-1682 (2015).
- Blauwendraat, C., Nalls, M. A., Singleton, A. B. The genetic architecture of Parkinson's disease. The Lancet Neurology. 19 (2), 170-178 (2020).
- Cacace, R., Sleegers, K., Van Broeckhoven, C. Molecular genetics of early-onset Alzheimer's disease revisited. Alzheimer's & Dementia. 12 (6), 733-748 (2016).
- Di Resta, C., Ferrari, M. New molecular approaches to Alzheimer's disease. Clinical Biochemistry. 72, 81-86 (2019).
- Karch, C. M., Cruchaga, C., Goate, A. M. Alzheimer's disease genetics: from the bench to the clinic. Neuron. 83 (1), 11-26 (2014).
- Kovacs, G. G. Molecular pathology of neurodegenerative diseases: principles and practice. Journal of Clinical Pathology. 72 (11), 725-735 (2019).
- McColgan, P., Tabrizi, S. J. Huntington's disease: a clinical review. European Journal of Neurology. 25 (1), 24-34 (2018).
- Ross, C. A., Tabrizi, S. J. Huntington's disease: from molecular pathogenesis to clinical treatment. The Lancet. Neurology. 10 (1), 83-98 (2011).
- Dugger, B. N., Dickson, D. W. Pathology of neurodegenerative diseases. Cold Spring Harbor Perspectives in Biology. 9 (7), 028035(2017).
- Ross, C. A., Poirier, M. A. Protein aggregation and neurodegenerative disease. Nature Medicine. 10, Suppl 10-17 (2004).
- DeJesus-Hernandez, M., et al. Expanded GGGGCC hexanucleotide repeat in noncoding region of C9ORF72 causes chromosome 9p-linked FTD and ALS. Neuron. 72 (2), 245-256 (2011).
- Renton, A. E., et al. A hexanucleotide repeat expansion in C9ORF72 is the cause of chromosome 9p21-linked ALS-FTD. Neuron. 72 (2), 257-268 (2011).
- Coyne, A. N., et al. G(4)C(2) repeat RNA initiates a POM121-mediated reduction in specific nucleoporins in C9orf72 ALS/FTD. Neuron. 107 (4), 1124-1140 (2020).
- Zhang, K., et al. The C9orf72 repeat expansion disrupts nucleocytoplasmic transport. Nature. 525 (7567), 56-61 (2015).
- D'Angelo, M. A., Raices, M., Panowski, S. H., Hetzer, M. W. Age-dependent deterioration of nuclear pore complexes causes a loss of nuclear integrity in postmitotic cells. Cell. 136 (2), 284-295 (2009).
- Hetzer, M. W. The role of the nuclear pore complex in aging of post-mitotic cells. Aging (Albany NY). 2 (2), 74-75 (2010).
- Savas, J. N., Toyama, B. H., Xu, T., Yates, J. R., Hetzer, M. W. Extremely long-lived nuclear pore proteins in the rat brain. Science. 335 (6071), 942(2012).
- Toyama, B. H., et al. Identification of long-lived proteins reveals exceptional stability of essential cellular structures. Cell. 154 (5), 971-982 (2013).
- Nofrini, V., Di Giacomo, D., Mecucci, C. Nucleoporin genes in human diseases. European Journal of Human Genetics. 24 (10), 1388-1395 (2016).
- Sakuma, S., D'Angelo, M. A. The roles of the nuclear pore complex in cellular dysfunction, aging and disease. Seminars in Cell & Developmental Biology. 68, 72-84 (2017).
- Basel-Vanagaite, L., et al. Mutated nup62 causes autosomal recessive infantile bilateral striatal necrosis. Annals of Neurology. 60 (2), 214-222 (2006).
- Nousiainen, H. O., et al. Mutations in mRNA export mediator GLE1 result in a fetal motoneuron disease. Nature Genetics. 40 (2), 155-157 (2008).
- Cronshaw, J. M., Matunis, M. J. The nuclear pore complex: disease associations and functional correlations. Trends in Endocrinology and Metabolism. 15 (1), 34-39 (2004).
- Chou, C. C., et al. TDP-43 pathology disrupts nuclear pore complexes and nucleocytoplasmic transport in ALS/FTD. Nature Neuroscience. 21 (2), 228-239 (2018).
- Eftekharzadeh, B., et al. Tau protein disrupts nucleocytoplasmic transport in Alzheimer's disease. Neuron. 99 (5), 925-940 (2018).
- Freibaum, B. D., et al. GGGGCC repeat expansion in C9orf72 compromises nucleocytoplasmic transport. Nature. 525 (7567), 129-133 (2015).
- Gasset-Rosa, F., et al. Polyglutamine-expanded huntingtin exacerbates age-related disruption of nuclear integrity and nucleocytoplasmic transport. Neuron. 94 (1), 48-57 (2017).
- Grima, J. C., et al. Mutant huntingtin disrupts the nuclear pore complex. Neuron. 94 (1), 93-107 (2017).
- Jovicic, A., et al. Modifiers of C9orf72 dipeptide repeat toxicity connect nucleocytoplasmic transport defects to FTD/ALS. Nature Neuroscience. 18 (9), 1226-1229 (2015).
- Guo, L., et al. Nuclear-import receptors reverse aberrant phase transitions of RNA-binding proteins with prion-like domains. Cell. 173 (3), 677-692 (2018).
- Hofweber, M., et al. Phase Separation of FUS Is Suppressed by Its Nuclear Import Receptor and Arginine Methylation. Cell. 173 (3), 706-719 (2018).
- Yoshizawa, T., et al. Nuclear Import Receptor Inhibits Phase Separation of FUS through Binding to Multiple Sites. Cell. 173 (3), 693-705 (2018).
- Chew, J., et al. Aberrant deposition of stress granule-resident proteins linked to C9orf72-associated TDP-43 proteinopathy. Molecular Neurodegeneration. 14 (1), 9(2019).
- Zhang, Y. J., et al. Poly(GR) impairs protein translation and stress granule dynamics in C9orf72-associated frontotemporal dementia and amyotrophic lateral sclerosis. Nat Medicine. 24 (8), 1136-1142 (2018).
- Zhang, Y. J., et al. C9ORF72 poly(GA) aggregates sequester and impair HR23 and nucleocytoplasmic transport proteins. Nature Neuroscience. 19 (5), 668-677 (2016).
- Zhang, Y. J., et al. Heterochromatin anomalies and double-stranded RNA accumulation underlie C9orf72 poly(PR) toxicity. Science. 363 (6428), (2019).
- Beck, M., Hurt, E. The nuclear pore complex: understanding its function through structural insight. Nature Reviews. Molecular Cell Biology. 18 (2), 73-89 (2017).
- Lin, D. H., Hoelz, A. The structure of the nuclear pore complex (an update). Annual Review of Biochemistry. 88, 725-783 (2019).
- Raices, M., D'Angelo, M. A. Nuclear pore complex composition: a new regulator of tissue-specific and developmental functions. Nature Reviews. Molecular Cell Biology. 13 (11), 687-699 (2012).
- Kinoshita, Y., Kalir, T., Dottino, P., Kohtz, D. S. Nuclear distributions of NUP62 and NUP214 suggest architectural diversity and spatial patterning among nuclear pore complexes. PLoS One. 7 (4), 36137(2012).
- Ori, A., et al. Cell type-specific nuclear pores: a case in point for context-dependent stoichiometry of molecular machines. Molecular Systems Biology. 9, 648(2013).
- Rajoo, S., Vallotton, P., Onischenko, E., Weis, K. Stoichiometry and compositional plasticity of the yeast nuclear pore complex revealed by quantitative fluorescence microscopy. Proceedings of the National Academy of Sciences of the United States of America. 115 (17), 3969-3977 (2018).
- Li, C., Goryaynov, A., Yang, W. The selective permeability barrier in the nuclear pore complex. Nucleus. 7 (5), 430-446 (2016).
- Raices, M., D'Angelo, M. A. Nuclear pore complexes and regulation of gene expression. Current Opinion in Cell Biology. 46, 26-32 (2017).
- Pascual-Garcia, P., Capelson, M. Nuclear pores in genome architecture and enhancer function. Current Opinion in Cell Biology. 58, 126-133 (2019).
- Schermelleh, L., et al. Subdiffraction multicolor imaging of the nuclear periphery with 3D structured illumination microscopy. Science. 320 (5881), 1332-1336 (2008).
- Maglione, M., Sigrist, S. J. Seeing the forest tree by tree: super-resolution light microscopy meets the neurosciences. Nature Neuroscience. 16 (7), 790-797 (2013).
- Thevathasan, J. V., et al. Nuclear pores as versatile reference standards for quantitative superresolution microscopy. Nature Methods. 16 (10), 1045-1053 (2019).
- Wu, Y., Shroff, H. Faster, sharper, and deeper: structured illumination microscopy for biological imaging. Nature Methods. 15 (12), 1011-1019 (2018).
- Hampoelz, B., et al. Pre-assembled nuclear pores insert into the nuclear envelope during early development. Cell. 166 (3), 664-678 (2016).
- Hampoelz, B., et al. Nuclear pores assemble from nucleoporin condensates during oogenesis. Cell. 179 (3), 671-686 (2019).
- Agote-Aran, A., et al. Spatial control of nucleoporin condensation by fragile X-related proteins. The EMBO Journal. 39 (20), 104467(2020).
- Colombi, P., Webster, B. M., Fröhlich, F., Lusk, C. P. The transmission of nuclear pore complexes to daughter cells requires a cytoplasmic pool of Nsp1. The Journal of Cell Biology. 203 (2), 215-232 (2013).
- Sivaguru, M., et al. Comparative performance of airyscan and structured illumination superresolution microscopy in the study of the surface texture and 3D shape of pollen. Microscopy Research and Technique. 81 (2), 101-114 (2018).
- Löschberger, A., Franke, C., Krohne, G., van de Linde, S., Sauer, M. Correlative super-resolution fluorescence and electron microscopy of the nuclear pore complex with molecular resolution. Journal of Cell Science. 127, Pt 20 4351-4355 (2014).
- Löschberger, A., et al. Super-resolution imaging visualizes the eightfold symmetry of gp210 proteins around the nuclear pore complex and resolves the central channel with nanometer resolution. Journal of Cell Science. 125, Pt 3 570-575 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
ISSN 1940-087X
Copyright © 2023 MyJoVE Corporation. All rights reserved
当社はcookieを使用しています。
「続行」をクリックすることで、当社のcookieへの同意となります。