Method Article
変形性関節症に対するオートファジーモジュレーターの同定のためのフラックスベースのアッセイ
要約
この論文では、細胞ベースのイメージングスクリーニングにより、オートファジーフラックスをモニタリングして新しい分子を同定する方法について詳しく説明します。
要約
オートファジーは、ホメオスタシスを調節する中心的なメカニズムです。オートファジーの変化は、老化関連疾患の一因となります。オートファジーの調節因子を同定する表現型法は、新規治療法の同定に利用できる可能性があります。この記事では、ヒト軟骨細胞のオートファジーフラックス(mCherry-EGFP-LC3B)のレポーターとしてLC3を使用してオートファジーフラックスをモニターするために開発された細胞ベースのイメージングスクリーニングワークフローについて説明します。データ取得は、自動化されたハイコンテントイメージングスクリーニングシステム顕微鏡を使用して行われます。アルゴリズムベースの自動画像解析プロトコルが開発され、オートファジーフラックスを活性化する分子を特定するために検証されました。重要な手順、説明文、および現在のオートファジーモニタリングプロトコルに対する改善点が報告されています。老化の特徴を標的とする生理学的に関連性のある表現型スクリーニングアプローチは、加齢に伴う筋骨格系疾患に対するより効果的な創薬戦略を促進することができます。
概要
多くの慢性疾患は、オートファジーの欠陥など、老化の特徴と関連しています1。変形性関節症(OA)は最も蔓延している関節疾患であり、高齢化社会の日常生活を制限する上で大きな影響を及ぼしますが、予防策も疾患修飾治療もまだ利用できません2。
関節の老化と変形性関節症は、オートファジーの欠陥や老化など、軟骨変性の進行を定義する特徴と関連しています3,4。筋骨格系組織のオートファジーを標的とすることで、リウマチ性疾患の革新的な治療法を見つけることができます5,6。オートファジーの薬理学的調節は、前臨床疾患モデル7への介入に有望で関連性のあるメカニズムです。OAでは、オートファジーの活性化が関節機能障害の予防に用いられています8。定量分析を可能にする堅牢で再現性のあるプロトコルに基づいてオートファジーをモニタリングする方法は、新規薬剤を同定し、老化の疾患関連特性の薬理学的標的化を促進するために使用できます。
オートファジーフラックスは分解活性を反映しており、オートファジー9を活性化する新しい分子を特定するための適切な測定値です。本研究では、ヒト軟骨細胞のオートファジーレポーター細胞株(TC28a2)を用いてオートファジー分解活性を測定することにより、オートファジーフラックスを測定するために開発された方法について述べています。mCherry-EGFP-LC3-一過性発現は、生細胞のリソソーム10におけるGFPシグナルとmCherry LC3シグナルとの間のpH感度の違いを定量化することにより、オートリソソームの形成および分解イベントの同時モニタリングを可能にする。
報告されたこのフローサイトメトリー法を生軟骨細胞の安定発現イメージングモニタリングシステムに適応させることで、軟骨生物学の文脈でオートファジーフラックスを活性化する分子を同定できる可能性があります。
プロトコル
1. 不死化ヒト軟骨細胞のオートファジーレポーター細胞株の作製
注:フローサイトメトリーの定量的読み出しを確立することにより、pBABE-mCherry-GFP-LC3の安定トランスフェクションによるオートファジーレポーター細胞株の作製は、以前に説明した11。レトロウイルストランスフェクションでは、コトランスフェクションプロセス中のHEK 293-T17と感染ステップのT/C28a2の2つの細胞株を使用しました。
- コトランスフェクションプロセス
- HEK 293-T17細胞用の増殖培地を調製します:500 mLのEagle's minimum essential medium(EMEM)、10%ウシ胎児血清(FBS)、および1%ペニシリン-ストレプトマイシン(P/S)。増殖培地は4°Cで保存し、37°Cまで温めてから使用してください。
- 1ウェルあたり1 x 106 HEK 293-T17細胞を100 mm細胞培養プレートに播種し、37°Cおよび5%CO2でインキュベートし、それらが~75%のコンフルエンスにあることを確認してから、コトランスフェクションプロセスを開始します。
- 培地を取り出し、ハンク平衡塩溶液(HBSS)ですすぎ、2%FBSと1%P/Sを含む8mLのEMEMを加えます。
- コニカルチューブに、10 μg、3 μg、7 μgのpBABE-puro mCherry-EGFP-LC3B、VSVを加えます。GプラスミドとpCL-Ecoプラスミド。次に、200 μLの1倍還元血清培地(材料表)を加えます。トランスフェクション試薬(材料表)を脂質の非リポソーム混合物として添加し、1:3の比率(1 μg DNA:3 μgトランスフェクション試薬、最終容量= 60 μL)にします。
注:トランスフェクションする10 cmの細胞プレートごとに、合計20 μgの混合物を調製します。 - 混合物を室温で20分間インキュベートします。トランスフェクションミックスを滴下してHEK 293-T17パッケージングセルに慎重に移します。細胞を37°Cおよび5%CO2 で少なくとも48時間インキュベートしてから、蛍光顕微鏡で緑色および赤色のタンパク質発現を確認します。
注:これらの条件下では、細胞の75%以上が蛍光陽性であるはずです。トランスフェクション効率が高いことを確認します(例:細胞の75%以上が陽性です)。高品質のプラスミドDNA(例:主にスーパーコイル状でゲノムDNA、RNA、タンパク質を含まない、高濃度、エンドトキシンや塩を含まない)および高品質の細胞培養(例:均質、低継代、単分子膜、指数関数的増殖期にトランスフェクションされた、マイコプラズマを含まない)を使用してください。
- コトランスフェクションの同日に、T/C28a2軟骨細胞を調製します。
- T/C28a2細胞増殖培地を調製する:500 mLのダルベッコ改変イーグル培地(DMEM)、10%ウシ胎児血清(FCS)、および1%P/Sを調製します。増殖培地を4°Cで保存し、37°Cまで温めてから使用してください。
- 2つの6ウェルマルチプレートに2 x 105 T/C28a2軟骨細胞を播種し、37°Cおよび5%CO2で48時間インキュベートします。
- レトロウイルス感染プロセス
- ステップ1.1.5で得られたHEK 293-T17レトロウイルスパッケージング細胞からの培地を吸引します。4mLのHBSSで1回すすぎ、HBSSを吸引します。
- 2mLのトリプシンを加え、37°Cで2分間待ちます。 6 mLのHEK 293-T17培地を加えてトリプシンを中和し、細胞と培地を15 mLのコニカルチューブに移します。
- 細胞を400 x g で5分間遠心分離します。上清を0.45 μmのメンブレンを介して滅菌フィルターに移します。
- 6ウェルマルチプレートからT/C28a2軟骨細胞培養培地を吸引します。T/C28a2細胞に感染させるには、ウェルあたり2 mLのウイルス懸濁液(ステップ1.3.3)を6ウェルマルチプレートに添加します。コントロール用のウェル1つ(すなわち、トランスフェクションされていない細胞)を使用し、ウイルス懸濁液の代わりに2 mLのT/C28a2細胞増殖培地を添加します。
- 37°Cおよび5%CO2で48時間インキュベートします。
- 選択プロセスを実行するには、トランスフェクションされた細胞に抗生物質選択を使用して、トランスフェクションされていない細胞を排除し、均質な細胞集団を取得します。
注:pBABE-puro mCherry-EGFP-LC3Bレトロウイルスベクターは、ピューロマイシンに対する哺乳類の抗生物質耐性を提供し、ウイルストランスフェクション後の安定した細胞培養の選択を可能にします。T/C28a2軟骨細胞の最適なピューロマイシン濃度のキルカーブを事前に設定して、高発現レベルの導入遺伝子を持つ細胞クローンを選択するための適切な条件を確立する必要があります。- T/C28a2-pBABE-puro mCherry-EGFP-LC3B 安定細胞株増殖培地を調製します: 500 mL の DMEM、10% FCS、1% P/S、および 2.5 μL/mL のピューロマイシン。培地は4°Cで保存し、使用する前に37°Cまで温めてください。
- 媒体を変更します。6ウェルマルチプレートごとに、培地を吸引し、2mLのHBSSで1回すすぎ、吸引します。2 mLの培地を加え、37°Cおよび5%CO2でインキュベートします。これが選考プロセスの始まりです。
- 2日ごとに培地を交換し、死細胞とピューロマイシン耐性細胞集団を観察します。結果をトランスフェクションしていないコントロール細胞と比較します。コントロール細胞はピューロマイシンに耐性がなく、死にます。
- クローン選択を開始するには、モノクローナル集団が増加したことを確認します。各6ウェルマルチプレートについて:
- ウェルあたり200 μLの培地を96ウェルマルチプレートに加えます。
注:6ウェルマルチプレートから選択された各シングルセルは、96ウェルマルチプレートの個々のウェルに播種されます。 - 顕微鏡を使用して、6ウェルマルチプレートの各モノクローナル集団を同定します。単一細胞から形成されたコロニーを観察することができます。
- 6ウェルマルチプレートから培地を吸引し、2 mLのHBSSで1回すすぎ、HBSSを吸引します。
- 各モノクローナル集団に10 μLのトリプシンを添加して、96ウェルマルチプレートの各ウェルに単一細胞を移設します。37°Cおよび5%CO2で48時間インキュベートします。
- ウェルあたり200 μLの培地を96ウェルマルチプレートに加えます。
- 48時間後、培地をウェルあたり100 μLの新鮮な培地と交換します。単一細胞の拡大によりコロニーが形成されるまで、この手順を繰り返します。
- 1つの細胞がウェルに増殖したら、各モノクローナル集団を24ウェル、6ウェル、および100 mmの細胞培養プレートの個々のウェルにプレート化します。
- シングルセルソーティング法を用いて、安定したモノクローナル細胞株を作製します。
- 100 mm細胞培養プレートから培地を吸引します。HBSSを8mLで1回すすぎ、吸引します。
- 各プレートに2 mLのトリプシンを加え、37°Cで2分間インキュベートします。 8 mLの培地を加えてトリプシンを中和します。
- 細胞を252 x g で5分間遠心分離し、上清を捨てます。各細胞ペレットに4 mLのHBSSを添加し、慎重に再懸濁し、サイトメトリーチューブに移します。
- 各細胞サンプルをセルソーティングテクノロジーを備えたフローサイトメーター(Table of Materials)に注入し、緑色と赤色の蛍光を多く使用した細胞を分離します。
- 選択した細胞を15 mLのコニカルチューブに移し、細胞を252 x g で5分間遠心分離します。細胞ペレットを9 mLの安定細胞株増殖培地を入れたT-25フラスコに加えます。37°Cおよび5%CO2でインキュベートします。
注:細胞選別後、3つのモノクローナル集団(表1)を取得する必要があります。
- 24時間後、培地を取り出し、8mLの新鮮な培地を追加します。この手順を2日ごとに繰り返し、細胞がコンフルエントに達するまで繰り返します。
注:オートファジーフラックスの研究には、高オートファジーフラックスシグナルリファレンスとしてラパマイシン、低オートファジーフラックスシグナルリファレンスとしてクロロキンの2つのコントロールを使用することができます。ジメチルスルホキシド(DMSO)中の10 mMラパマイシン原液とH2O中の30 mMクロロキン原液を調製し、-20°Cで保存してください。 発現mCherryおよびGFPを試験して最適なモノクローナル集団を選択するには、フローサイトメトリー(ステップ1.10)またはハイコンテント蛍光画像解析(ステップ1.11)の2つの方法を使用することができます。 - フローサイトメトリー
- 各モノクローナル集団のウェルあたり2.5 x 105 細胞を12ウェルプレートに播種し、処理の少なくとも24時間前に1 mLの安定細胞株増殖培地でインキュベートし、37°Cおよび5%CO2でインキュベートします。
- 治療当日は、培地を取り出し、ウェルあたり2% FCSを添加した安定な細胞株増殖培地1 mLを加えます。10 μMラパマイシンと30 μMクロロキンを2% FCSを含む安定細胞株増殖培地に添加し、37 °Cおよび5% CO2で16時間インキュベートします。
- 培地を取り出し、ウェルあたり1 mLのHBSSですすぎ、吸引します。
- 各ウェルに500 μLのトリプシンを加え、37°Cで1分間インキュベートし、500 μLの培地を加えて反応を中和します。細胞をこすり落とし、円錐形のチューブに移します。
- 細胞を252 x g で5分間遠心分離し、上清を吸引します。細胞ペレットを500 μLのリン酸緩衝生理食塩水(PBS)ですすいでください。252 x g で5分間遠心分離し、上清を捨てます。
- 500 μLのPBSを添加し、細胞をサイトメトリーチューブに移し、各サンプルをサイトメトリー装置に注入します。条件ごとに10,000個のイベントを収集します。
- フローサイトメトリーでモノクローナル集団を選択するには、488 nmレーザーを使用してEGFPを励起し、633 nmレーザーをmCherryに励起します。次に、mCherry蛍光とEGFP蛍光の比率を確立して、オートファジーフラックスが高い細胞集団と低い細胞集団を選択します。
- ハイコンテント蛍光画像解析
- 10% FCS、1% P/S、および2.5 μL/mLピューロマイシンを添加した増殖培地中の384ウェルプレート(黒壁/透明底)に、ウェルあたり各モノクローナル集団の4 x 103 細胞を播種します。処理前に、37°Cおよび5%CO2 で24時間インキュベートしてください。
- 培地を取り出し、ウェルあたり2% FCSを添加した50 μLの安定細胞株増殖培地に10 μMのラパマイシンと30 μMのクロロキンを加えます。37°Cおよび5%CO2で16時間インキュベートします。
- 培地を取り出し、ウェルあたり50 μLのHBSSですすぎ、吸引します。細胞を4%パラホルムアルデヒド(PFA)で37°Cで10分間固定します。 PFAを除去し、ウェルあたり50μLのHBSSですすいでください。
- 細胞核を同定するには、Hoechst 33342(2.5 μg/mL)で37°Cで10分間染色します。 Hoechst 33342色素を吸引し、ウェルあたり50 μLのHBSSですすぎ、ウェルあたり50 μLのPBSを添加します。
- セクション3で説明したように、HTSプラットフォームによって得られたオートファジーフラックスのデータ取得と分析を行います。
注:モノクローナル集団を選択する基準は、オートファジーフラックスがラパマイシンまたはクロロキンでそれぞれ増加したか減少したかに基づいています(表1)。mCherryおよびGFPの発現量が高いクローンは、オートファジーフラックスの正確なハイコンテント蛍光画像解析定量に適しています。
2. 生軟骨細胞における画像ベースのオートファジーフラックスアッセイ
注:クローンを選択した後、アッセイを開始してオートファジーフラックスを定量します。
- ハンドヘルド電動384チャンネルピペットを使用して、選択したクローン(ステップ1.11)の4 x 103 細胞をウェルあたり384ウェルプレート(黒壁/透明底)に播種し、ウェルあたり10% FCS、1% P/S、および2.5 μL/mLのピューロマイシンを添加した50 μLの安定細胞株増殖培地に入れます。処理前に、37°Cおよび5%CO2 で24時間インキュベートしてください。
- 実験当日は、化合物添加用のインターバルプレートを用意します。
- 音響リキッドハンドリング技術と電子式384チャンネルピペットを使用して、各化合物/コントロールと安定細胞株増殖培地の容量をインターバルプレートにそれぞれ追加し、インターバル濃度を取得します。
注:このアッセイのコントロールには、2%FCS、30μMクロロキン、および10μMラパマイシンを補充した安定細胞株増殖培地が含まれていました。間隔濃度は、目的の最終濃度によって異なります。例えば、このアッセイでは、クロロキンとラパマイシンをウェルあたり50μLの安定な細胞株増殖培地に添加して、それぞれ300μMと100μMのインターバル濃度を得ました。 - マイクロプレートウォッシャーロボットを使用して、細胞プレートから培地を自動的に吸引し、ウェルあたり2% FCSを添加した45 μLの安定した細胞株増殖培地を追加します。
- 次に、ウェルあたり5 μLをインターバルプレートからアッセイプレートに移し、他の自動リキッドハンドラーワークステーションを使用して最終濃度を取得します。
- 音響リキッドハンドリング技術と電子式384チャンネルピペットを使用して、各化合物/コントロールと安定細胞株増殖培地の容量をインターバルプレートにそれぞれ追加し、インターバル濃度を取得します。
- 37°Cおよび5%CO2 で16時間インキュベートします。マイクロプレートウォッシャーロボットを使用して、培地を吸引します。
- ウェルあたり50 μLの4% PFAで細胞を1ウェルあたり10分間、37°Cで固定します。 ウェルあたり50μLのHBSSで洗浄します。
- Hoechst 33342(2.5 μg/mL)で核を37°Cで10分間染色します。 Hoechst 33342を吸引し、ウェルあたり50 μLのHBSSですすいでください。
- ウェルあたり50 μLのPBSを添加します。これで、プレートの読み取り、データ収集、およびその後の分析の準備が整いました。
注:このプロトコルは、ハイスループットスクリーニング(HTS)について説明しています。ただし、使用する化合物が少ない場合は、自動装置を使用せずに手動で追加できます。キャリブレーション済みのリキッドハンドリング機器や電動ピペットを使用した細胞や上清の操作を最小限に抑えることで、ハイコンテント蛍光画像解析によるオートファジーフラックスの定量のための細胞培養単分子膜の調製が容易になります。
3. オートファジーフラックスのデータ取得と解析
- データ取得には、ハイコンテントスクリーニングシステム(HCS)と自動顕微鏡を使用し、画像処理アルゴリズムによる自動画像取得と分析を可能にします。
- 取得プロトコルを開発するには、いくつかのチャンネルと露光時間(表2)を選択して、オートファジーフラックスイベント中の核とオートファゴソームおよびオートリソソームの形成を同定します。
- 各ウェルについて、ウェル表面を覆う4つのフィールドから、8μmから0.2μmの作動距離で開始する4つのスタックで画像を収集します。60倍対物レンズを使用して画像をキャプチャすると、詳細な細胞イメージングが可能になります。
- HCS顕微鏡が提供する画像解析ソフトウェアを使用してデータを解析します。オートファジーフラックスモニタリングのための堅牢な解析プロトコルアルゴリズムを開発するには、自動細胞画像セグメンテーションと、LC3活性化に対応するスペックルを特異的に検出するための最適な方法を選択します。最終的な解析シーケンスを実行するには、以下に説明するように、メソッド、細胞セグメンテーションステップ、および定量手順を追加します。
- [Find Nuclei] ビルディング ブロックをクリックし、核色素チャネル [Hoechst 33342] を選択します。[Method] を選択し、出力母集団 [Nuclei] を選択します。
- 「Find Cytoplasm」ビルディングブロックをクリックし、細胞質色素チャネル「Fluorescein」を選択します。選択方法 |個々のしきい値。
- 「Find Cytoplasm (2)」ビルディングブロックをクリックし、細胞質色素チャネル RFP を選択します。選択方法 |個々のしきい値。
- 「母集団」構成要素をクリックし、調査母集団の Nuclei を選択します。方法「共通フィルター」(境界線オブジェクトの削除)を選択し、出力母集団「境界線内の核」を選択します。
- [スポットの検索] ビルディングブロックをクリックし、特定のマーカーチャネル [フルオレセイン] を選択します。研究集団の Nuclei Inside 境界を選択し、研究領域 Cytoplasm を選択します。[方法] を選択し、出力母集団 [Spots Inside Borders] を選択します。
- [スポットの検索] (2) ビルディングブロックをクリックし、特定のマーカーチャネル RFP を選択します。研究母集団の Nuclei Inside 境界を選択し、研究領域 Cytoplasm (2) を選択します。[方法] を選択し、出力母集団 [Spots (2) Inside Borders] を選択します。
- [強度プロパティの計算] ビルディング ブロックをクリックし、特定のマーカー チャネル [フルオレセイン] を選択します。研究母集団 Nuclei Inside Borders を選択し、分析領域 Spot を選択します。「方法」を選択し、出力プロパティ「Intensity Spot Fluorescein Inside Borders」を選択します。
- [Calculate Intensity Properties] ビルディングブロックをクリックし、特定のマーカーチャネル RFP を選択します。境界内の研究母集団 Nuclei を選択し、分析領域 Spot (2) を選択します。[方法] を選択し、出力プロパティの [強度スポット (2) RFP 境界内] を選択します。
- [ Define Results ] ビルディングブロックをクリックし、[ Population Nuclei Inside Borders] を選択し、[ Number of Objects of Nuclei (Mean)] を選択し、[ Relative Intensity Spots Fluorescein – mean per well] を選択し、[ Relative Intensity Spots (2) – RFP – mean per well] を選択します。
- オートファジーフラックスを定量するには、前述のフローサイトメトリー解析11と同じ方法を使用します。相対強度スポットのmCherry/EGFP比を設定します。
注:フラックスが高い細胞は緑色が少なくなり、細胞内のmCherry/EGFP比が増加します。細胞内のオートファジーフラックスイベントから得られる関連性のあるシグナルの数を特定し、正確に定量することは、統計的に有意な差を確立するために非常に重要です。
結果
オートファジーフラックスは、薬理学的調節または細胞ストレス応答によって細胞内でモニターできます。不死化ヒト軟骨細胞(T/C28a2)を用いて、LC3をオートファジー用の蛍光レポーター(mCherry-EGFP-LC3B)として用いたオートファジーレポーター細胞株を開発しました。 図1 は、ヒト軟骨細胞のオートファジーレポーター細胞株(mCherry-EGFP-LC3-T/C28a2)の開発から始まり、薬理学的調節によるオートファジーフラックスの誘導または阻害、そしてハイコンテントイメージング顕微鏡によるデータ解析までのスクリーニングアッセイの概略的なワークフローを示しています。 図2 は、オートファジーフラックスをモニタリングしてオートファジーモジュレーターを同定するセルベースイメージングアッセイの概略図を示しています。このシステムの基礎は、mCherryに比べてオートリソソームの酸性環境に対するEGFP蛍光の感度が高いことにあります。 図3 は、細胞ベースの画像からオートファジーフラックスを定量化するための解析のシーケンスを示しています。 図4 は、クロロキンとラパマイシンのmCherry/GFPの比率で表される低オートファジーフラックスと高オートファジーフラックスの代表的な画像を示しています。

図1:細胞ベースのイメージングアッセイによるオートファジーフラックスの測定に使用したワークフローの概略図。 オートファジーレポーター細胞株を作製するために、レトロウイルストランスフェクションでは、コトランスフェクションプロセスではHEK 293-T17細胞株を、感染ステップではT/C28a2不死化ヒト軟骨細胞の2つの細胞株を使用しました。pBABE-puro-mCherry-EGFP-LC3Bプラスミドを前述の10 のように細胞に導入し、ピューロマイシンを使用してウイルストランスフェクション後の安定した細胞培養を選択しました。細胞を増殖培地中の384ウェルプレート(1ウェルあたり4 x 103 細胞)に播種し、処理前に37°Cおよび5%CO2 で24時間インキュベートしました。次に、培地を取り出し、増殖培地に 2% FCS と 1% P/S、および 2.5 μL/mL ピューロマイシンを添加しました。細胞を30 μMクロロキンおよび10 μMラパマイシンで処理しました。データ取得には、ハイコンテントスクリーニングシステムを使用しました。読み取りプロトコルを開発するために、いくつかのチャネルと露光時間を選択しました。データは画像解析ソフトウェアを使用して解析し、解析プロトコルを選択しました。最後に、相対強度スポットのmCherry/EGFP比を確認しました。 この図の拡大版を表示するには、ここをクリックしてください。
図2:オートファジーモジュレーターを同定するための細胞ベースのイメージングアッセイの表現。 pBABE-puro-mCherry-EGFP-LC3Bプラスミドを細胞内に導入しました。オートファジーが活性化されると、貨物はファゴフォアに飲み込まれ、オートファゴソームが生成されました。LC3は脂質化され、LC3がカーゴ受容体と相互作用できるオートファゴソーム膜に取り込まれました。オートファゴソームとEGFP pKa ≥ 6.0の塩基性環境により、緑色蛍光が発せられました。オートファジーフラックスが続くと、オートファゴソームがリソソームと融合し、オートリソソームが作られ、pHが低下し、赤橙色の蛍光が発せられました。 この図の拡大版を表示するには、ここをクリックしてください。
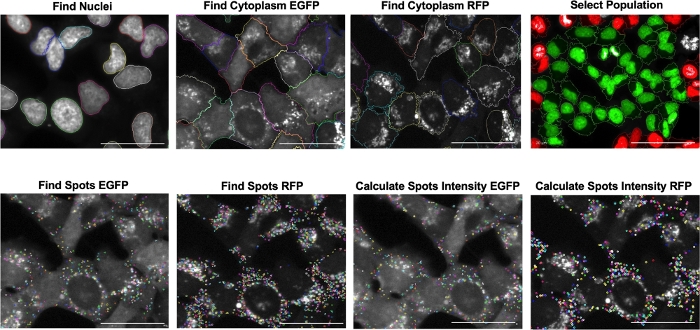
図3:さまざまなビルディングブロックを使用して細胞画像からオートファジーフラックスを定量化し、プロトコルを作成するための解析のシーケンス。 核は、ヘキストチャネルを使用して同定されました。次に、フルオレセイン(EGFP)とRFPの2つのチャネルを使用して細胞質を同定しました。次に、細胞集団を選択するために境界オブジェクトを削除しました。ソフトウェアは、セル全体を緑色で、不完全なセルを赤色で識別します。同定された各細胞質では、斑点(スポットと呼ばれる)の蓄積が見られました。最後に、各細胞質からの相対強度スポットを計算し、LC3活性化に関連するmCherry/EGFPの比率に基づいてオートファジーフラックスを定量しました。スケールバー = 50 μm。 この図の拡大版を表示するには、ここをクリックしてください。

図4:クロロキンとラパマイシンで処理したmCherry-EGFP-LC3 T/C28a2軟骨細胞のイメージングによる定量。 2% FCS を添加した培地をコントロールとして使用しました。クロロキンはオートファジーフラックス阻害剤として使用されました。オートファジーフラックスが活性化されると、オートファゴソームが作られましたが、オートファゴソームとリソソームの融合は起こりませんでした。そのため、オートファゴソームが蓄積し、緑色蛍光が発せられ、mCherry/EGFPの比率が減少しました。さらに、ラパマイシンはオートファジーフラックス活性化剤として使用されました。オートファゴソームがリソソームと融合すると、pHが低下し、赤橙色の蛍光が発せられ、mCherry/EGFPの比率が上昇しました。スケールバー = 50 μm。 この図の拡大版を表示するには、ここをクリックしてください。
| モノクローナル集団 | 条件 | レシオ mCherry/EGFP フローサイトメトリー | レシオ mCherry/EGFP HTS |
| クローンB | DMEM 2% FCSの | 1 | 1 |
| ラパマイシン 5 μM | 1.5 | 1.13 | |
| クロロキン 30 μM | 0.96 | 0.76 | |
| クローンD | DMEM 2% FCSの | 1 | 1 |
| ラパマイシン 5 μM | 1.32 | 1.22 | |
| クロロキン 30 μM | 1.05 | 0.76 | |
| クロンH | DMEM 2% FCSの | 1 | 1 |
| ラパマイシン 5 μM | 1.64 | 1.12 | |
| クロロキン 30 μM | 0.88 | 0.87 |
表1:フローサイトメトリーとHTSによって得られたデータで、オートファジーフラックスを定量し、最適なモノクローナル集団を選択します。
| チャンネル | エミッション (nm) | 励起(nm) | 露出 (ミリ秒) |
| 明視野 | 652−760 | 感染 | 20 |
| ヘキスト | 410−480 | 360−400 | 100 |
| フルオレセイン | 500−550 | 460−490 | 700 |
| RFPの | 560−630 | 520−550 | 1000 |
表2:オートファジーフラックスプロセスのハイコンテントスクリーニング解析のための画像取得設定。各チャンネルの発光波長と励起波長、露光時間。
ディスカッション
オートファジーの欠陥は、加齢に伴う関節変性の重要な特徴ですが、オートファジーを標的とした予防的治療も疾患修飾的治療も、軟骨変性に対する治療法はまだありません2。オートファジーは、その関連性と臨床的意義から、創薬と開発の関心対象となっていますが、この重要な恒常性メカニズムの変化を直接モニタリングする方法は困難であることが証明されています。
ここでは、生きたヒト軟骨細胞におけるオートファジーフラックスを決定するための細胞ベースの表現型アッセイについて説明します。オートファジー活性化のデュアルレポーターであるmCherry-EGFP-LC3を含むプラスミドは、フローサイトメトリー11により、生細胞におけるGFPシグナルとmCherryシグナルのpH感度の違いによるオートリソソームの形成と分解イベントを同時にモニターするために使用されます。mCherry-EGFP-LC3Bを安定して高発現するヒト軟骨細胞は、オートファゴソームフラックスのダイナミクスを正確にモニターするために使用できます。軟骨細胞を用いた細胞ベースのイメージングアッセイは、オートリソソーム形成による動的なpH変化をリアルタイムで観察できるため、オートファジーモジュレーターの同定に使用することができます。オートファジーが活性化すると、ファゴフォアとオートファゴソームが生成され、使い捨ての貨物を捕まえます。LC3の脂質化、オートファゴソーム膜への包含、およびカーゴ受容体との相互作用は、緑色蛍光の発光に対応するpKa ≥ 6.0の基本環境を提供します。オートファジーフラックスが持続すると、オートファゴソームはリソソームと融合してオートリソソームを生成し、pHが低下して赤オレンジ色の蛍光を発します。
プロトコールを設定するための重要なステップには、高品質の細胞培養とDNAを使用してウイルス粒子を生成することにより高いトランスフェクション効率を得ること、最適な抗生物質処理とフローサイトロメトリーソーティングによる高レベルの導入遺伝子を持つ細胞クローンの効率的な選択、高品質の画像がオートファジーフラックスの定量分析を可能にするための細胞単層の穏やかな操作が含まれます。 また、オートファジーフラックスイベントから得られる有意な数の関連シグナルを正確に同定することもできます。
現在の方法はフローサイトメトリーに大きく依存しており、細胞を懸濁状態にする必要があるため、細胞間コミュニケーションや細胞内イベントに関する情報を取得するのが困難です。精度が高く、小さな亜集団や複雑な亜集団の精製には適していますが、ソーティングが遅すぎる場合があり、表面受容体密度の平均に関するデータしか提供されません。したがって、細胞ベースのイメージングは、静的分析によるマーカーの検出によるオートファジーのモニタリングと測定に利点があり、例えば、アップレギュレーションと分解阻害を区別することはできません12。また、画像取得の高感度化と、画像のキャプチャと分析を可能にする自動化された方法は、ハイコンテントスクリーニングシステムを使用することによってのみ可能です。自動定量分析では、軟骨細胞をクロロキンに曝露することで得られるオートファジーシグナルは一貫して低く(緑色)、軟骨細胞をラパマイシン(赤橙色)に曝露することで高いオートファジー活性化シグナルが得られることが示されています。これらの逆の効果により、大規模な実験でオートファジーを調節する薬剤(低分子のライブラリー、ゲノムの機能喪失または機能獲得スクリーニングライブラリーなど)を体系的に同定することができます。実際、この細胞ベースのアッセイを薬物転用アプローチで使用することにより、脂質低下薬として使用されたPPARαフィブラートは、変形性関節症の疾患修飾治療薬候補として特定されました13。
この方法は、フローサイトメトリーを生軟骨細胞の安定発現イメージングモニタリングシステムに適応させます。このプロトコールは、軟骨生物学の文脈でオートファジーフラックスを活性化する分子の同定を可能にするだけでなく、これらのメカニズムに影響を与える薬剤をスクリーニングする方法を可能にする可能性があります。
開示事項
著者は何も開示していません。
謝辞
この研究は、Instituto de Salud Carlos III- Ministerio de Ciencia, Innovación y Universidades, Spain, Plan Estatal 2013-2016 and Fondo Europeo de Desarrollo Regional (FEDER), "Una manera de hacer Europa", PI14/01324 and PI17/02059 の支援を受けました。 Innopharma Pharmacogenomics プラットフォームは、標的の検証と前臨床段階への薬剤候補の発見に適用されました。また、リウマチ学研究財団(FOREUM)のご支援にも感謝いたします。
資料
| Name | Company | Catalog Number | Comments |
| 100 mm cell culture plate | Corning | 430167 | Cell culture plate |
| 12-well multiplate | Corning | 353043 | Cell culture plate |
| 15 mL Centrifuge conical tube | Falcon-Corning | 352095 | Centrifuge conical tube |
| 24-well multiplate | Corning | 351147 | Cell culture plate |
| 25 cm2 Cell Culture Flask | Falcon-Corning | 353014 | Cell culture flask |
| 384-well multiplate Cell Carrier | Perkin Elmer | 6007550 | Cell culture plate |
| 6-well multiplate | Corning | 351146 | Cell culture plate |
| 96-well multiplate | Corning | 353077 | Cell culture plate |
| Acoustic liquid handling technology | Labcyte | − | https://www.labcyte.com |
| Chloroquine | Sigma-Aldrich | C6628 | autophagic flux inhibitor |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich, St. Louis, MO | D2650 | Disolvent |
| Dulbecco's Modified Eagle's Medium (DMEM) | Lonza, Basel, Switzerland | BE-604F | T/C28a2 growth medium |
| Eagle's Minimum Essential Medium (EMEM) | ATCC | 30–2003 | HEK 293-T17 growth medium |
| FACScalibur cytometer | Becton Dickinson, CA | − | https://www.bdbiosciences.com/en-eu |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich, St. Louis, MO | F9665 | HEK 293-T17 serum |
| Fetal Calf Serum (FCS) | Gibco by Life Technologies, CA | 26010–074 | Serum |
| FuGene | Promega | E2691 | A nonliposomal mixture of lipids as a plasmid delivery method to create autophagy reported cell line |
| Handheld electronic 384 channel pipette | Integra | − | https://www.integra-biosciences.com/united-states/en/electronic-pipettes/viaflo-96384#downloads |
| Hank's Balanced Salt Solution (HBSS) | Sigma-Aldrich | H6648 | Buffer |
| HEK 293-T17 | ATCC | CRL-11268 | Kidney cell line. Cells were used to facilitate retroviral packaging |
| High Content Screening System | Perkin Elmer | − | https://www.perkinelmer.com/es/product/operetta-cls-system-hh16000000 |
| Hoechst 33342 | Thermo Fisher Scientific | 62249 | DNA staining |
| Image Analysis Software | Perkin Elmer | − | https://www.perkinelmer.com/es/product/harmony-4-9-office-license-hh17000010 |
| Liquid Handler workstation | Perkin Elmer | − | https://www.perkinelmer.com/es/category/janus-liquid-handler-workstations |
| Microplate washer robot | Biotek | − | https://go.biotek.com/405tradein |
| Opti-MEM® (1x) | Thermo Fisher Scientific | 11058 | Transfection medium |
| Paraformaldehyde (PFA) | Sigma-Aldrich | 158127 | Fixer |
| pBABE-puro mCherry-EGFP-LC3B | Addgene, Cambridge, MA | 22418 | Plasmid |
| pCL-Eco | Addgene, Cambridge, MA | 12371 | Plasmid |
| Penicillin-Streptomycin (P/S) | Sigma-Aldrich | P0781 | Antibiotic |
| Phosphate-buffered saline (PBS) | MP Biomedicals | 2810305 | Buffer |
| Puromycin | Sigma-Aldrich, St. Louis, MO | P8833 | Antibiotic |
| Rapamycin | Calbiochem, Germany | 5053210 | autophagic flux activator |
| Software CellQuestPro | Becton Dickinson | − | https://www.bdbiosciences.com/en-eu |
| Syringe filters 0.45 μm | Corning | CLS431220 | Sterile filter |
| T/C28a2 | − | − | human chondrocytes cell line |
| Trypsin | Gibco by Life Technologies, CA | 15400054 | Trypsin used with T/C28a2 cells |
| Trypsin | Sigma-Aldrich, St. Louis, MO | SM-2002-C | Trypsin used with HEK 293-T17 cells |
| VSV.G | Addgene, Cambridge, MA | 14888 | Plasmid |
参考文献
- Lopez-Otin, C., Blasco, M. A., Partridge, L., Serrano, M., Kroemer, G. The hallmarks of aging. Cell. 153 (6), 1194-1217 (2013).
- Lotz, M. K., Carames, B. Autophagy and cartilage homeostasis mechanisms in joint health, aging and OA. Nature Reviews Rheumatology. 7 (10), 579-587 (2011).
- Carames, B., Taniguchi, N., Otsuki, S., Blanco, F. J., Lotz, M. Autophagy is a protective mechanism in normal cartilage, and its aging-related loss is linked with cell death and osteoarthritis. Arthritis & Rheumatology. 62 (3), 791-801 (2010).
- Carames, B., Olmer, M., Kiosses, W. B., Lotz, M. K. The relationship of autophagy defects to cartilage damage during joint aging in a mouse model. Arthritis & Rheumatology. 67 (6), 1568-1576 (2015).
- Kroemer, G. Autophagy: a druggable process that is deregulated in aging and human disease. The Journal of Clinical Investigation. 125 (1), 1-4 (2015).
- Leidal, A. M., Levine, B., Debnath, J. Autophagy and the cell biology of age-related disease. Nature Cell Biology. 20 (12), 1338-1348 (2018).
- Maiuri, M. C., Kroemer, G. Therapeutic modulation of autophagy: which disease comes first. Cell Death & Differentiation. 26 (4), 680-689 (2019).
- Vinatier, C., Dominguez, E., Guicheux, J., Carames, B. Role of the inflammation- autophagy-senescence integrative network in osteoarthritis. Frontiers in Physiology. 9, 706(2018).
- du Toit, A., Hofmeyr, J. S., Gniadek, T. J., Loos, B. Measuring autophagosome flux. Autophagy. 14 (6), 1060-1071 (2018).
- N'Diaye, E. N., et al. PLIC proteins or ubiquilins regulate autophagy-dependent cell survival during nutrient starvation. EMBO Reports. 10 (2), 173-179 (2009).
- Gump, J. M., Thorburn, A. Sorting cells for basal and induced autophagic flux by quantitative ratiometric flow cytometry. Autophagy. 10 (7), 1327-1334 (2014).
- Yoshii, S. R., Mizushima, N. Monitoring and Measuring Autophagy. International Journal of Molecular Sciences. 18 (9), 1865(2017).
- Nogueira-Recalde, U., et al. Fibrates as drugs with senolytic and autophagic activity for osteoarthritis therapy. EBioMedicine. 45, 588-605 (2019).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved