Method Article
Sonda del metabolismo e della viscosità delle cellule tumorali mediante microscopia per imaging a fluorescenza
In questo articolo
Riepilogo
Qui, dimostriamo l'uso della microscopia a fluorescenza (FLIM) per visualizzare in sequenza il metabolismo cellulare e la viscosità della membrana plasmatica in colture di cellule tumorali vive. Le valutazioni metaboliche vengono eseguite rilevando la fluorescenza endogena. La viscosità viene misurata utilizzando un rotore molecolare fluorescente.
Abstract
La viscosità è un'importante proprietà fisica di una membrana biologica, in quanto è uno dei parametri chiave per la regolazione dello stato morfologico e fisiologico delle cellule viventi. È noto che le membrane plasmatiche delle cellule tumorali presentano alterazioni significative nella composizione, nella struttura e nelle caratteristiche funzionali. Insieme al metabolismo disregolato del glucosio e dei lipidi, queste specifiche proprietà della membrana aiutano le cellule tumorali ad adattarsi al microambiente ostile e a sviluppare resistenza alle terapie farmacologiche. Qui, dimostriamo l'uso della microscopia a fluorescenza (FLIM) per visualizzare in sequenza il metabolismo cellulare e la viscosità della membrana plasmatica in colture di cellule tumorali vive. Le valutazioni metaboliche vengono eseguite rilevando la fluorescenza di cofattori metabolici endogeni, come la nicotinammide adenina dinucleotide ridotta NAD(P)H e le flavine ossidate. La viscosità viene misurata utilizzando un rotore molecolare fluorescente, un colorante sintetico sensibile alla viscosità, con una forte dipendenza dalla durata della fluorescenza dalla viscosità dell'ambiente circostante. In combinazione, queste tecniche ci consentono di comprendere meglio i legami tra lo stato della membrana e il profilo metabolico delle cellule tumorali e di visualizzare i cambiamenti indotti dalla chemioterapia.
Introduzione
La trasformazione maligna delle cellule è accompagnata da molteplici alterazioni del loro stato morfologico e fisiologico. La crescita rapida e incontrollata delle cellule tumorali richiede una riorganizzazione fondamentale delle vie biochimiche responsabili della produzione di energia e della biosintesi. I segni distintivi caratteristici del metabolismo del cancro sono l'aumento del tasso di glicolisi, anche sotto le normali concentrazioni di ossigeno (effetto Warburg), l'uso di aminoacidi, acidi grassi e lattato come combustibili alternativi, l'elevata produzione di ROS in presenza di alti livelli di antiossidanti e l'aumento della biosintesi degli acidi grassi 1,2. È ormai noto che il metabolismo delle cellule tumorali è altamente flessibile, il che consente loro di adattarsi all'ambiente sfavorevole ed eterogeneo e fornisce un ulteriore vantaggio di sopravvivenza3.
L'alterato metabolismo supporta l'organizzazione e la composizione specifica delle membrane delle cellule tumorali. Il profilo lipidico della membrana plasmatica nelle cellule tumorali differisce quantitativamente dalle cellule non cancerose. I principali cambiamenti nel lipidoma sono l'aumento del livello di fosfolipidi tra cui fosfatidilinositolo, fosfatidilserina, fosfatidiletanolammina e fosfatidilcolina, la diminuzione del livello di sfingomielina, l'aumento della quantità di colesterolo e un minor grado di insaturazione degli acidi grassi, per citarne alcuni 4,5,6. Pertanto, le proprietà fisiche della membrana, come la viscosità della membrana, l'inverso della fluidità, cambiano inevitabilmente. Poiché la viscosità determina la permeabilità delle membrane biologiche e controlla l'attività delle proteine associate alla membrana (enzimi, trasportatori, recettori), la sua regolazione omeostatica è vitale per il funzionamento cellulare. Allo stesso tempo, la modifica della viscosità attraverso l'aggiustamento del profilo lipidico di membrana è importante per la migrazione/invasione cellulare e la sopravvivenza in caso di cambiamenti condizionali.
La microscopia a fluorescenza con imaging a vita (FLIM) si è affermata come un potente approccio per la valutazione non invasiva di più parametri nelle cellule viventi, utilizzando la fluorescenza endogena o le sonde esogene7. FLIM è comunemente realizzato su un microscopio a scansione laser multifotone, che fornisce una risoluzione (sub)cellulare. Essendo dotato del modulo di conteggio di singoli fotoni correlato al tempo (TCSPC), consente misurazioni della fluorescenza risolte nel tempo con elevata precisione8.
L'indagine sul metabolismo cellulare mediante FLIM si basa sulla misurazione della fluorescenza di cofattori endogeni delle deidrogenasi, della nicotinammide adenina dinucleotide (fosfato) NAD(P)H ridotta e delle flavine ossidate - flavina adenina dinucleotide FAD e flavina mononucleotide FMN, che agiscono come trasportatori di elettroni in una serie di reazioni biochimiche 7,9,10. La fluorescenza rilevata di NAD(P)H proviene dal NADH e dalla sua forma fosforilata, NADPH, poiché sono spettralmente quasi identici. Tipicamente, i decadimenti di fluorescenza del NAD(P)H e delle flavine si adattano a una funzione bi-esponenziale. Nel caso del NAD(P)H, il primo componente (~0,3-0,5 ns, ~70%-80%) è attribuito al suo stato libero, associato alla glicolisi, e il secondo componente (~1,2-2,5 ns, ~20%-30%) al suo stato legato alle proteine, associato alla respirazione mitocondriale. Nel caso delle flavine, la componente corta (~0,3-0,4 ns, ~75%-85%) può essere assegnata allo stato spento del FAD e la componente lunga (~2,5-2,8 ns, ~15%-25%) al FAD non estinto, alla FMN e alla riboflavina. Alterazioni nei livelli relativi di glicolisi, glutamminolisi, fosforilazione ossidativa e sintesi degli acidi grassi provocano cambiamenti nelle frazioni di breve e lunga vita dei cofattori. Inoltre, il rapporto di intensità della fluorescenza di questi fluorofori (il rapporto redox) riflette lo stato redox cellulare ed è anche usato come indicatore metabolico. Sebbene il rapporto redox presenti una metrica più semplice, rispetto al tempo di vita della fluorescenza, in termini di acquisizione dei dati, FLIM è vantaggioso per stimare NAD(P)H e FAD, perché il tempo di vita della fluorescenza è una caratteristica intrinseca del fluoroforo e quasi non è influenzato da fattori come il potere di eccitazione, il fotosbiancamento, la messa a fuoco, la diffusione e l'assorbimento della luce, specialmente nei tessuti, a differenza dell'intensità dell'emissione.
Uno dei modi convenienti per mappare la viscosità nelle cellule e nei tessuti viventi a livello microscopico si basa sull'uso di rotori molecolari fluorescenti, piccoli coloranti sintetici sensibili alla viscosità, in cui i parametri di fluorescenza dipendono fortemente dalla viscosità locale11,12. In un mezzo viscoso, la durata di fluorescenza del rotore aumenta a causa del rallentamento della torsione o rotazione intramolecolare. Tra i rotori molecolari, i derivati del boro dipirromete (BODIPY) sono adatti per rilevare la viscosità nei sistemi biologici in quanto hanno un buon intervallo dinamico di tempi di vita della fluorescenza in un intervallo fisiologico di viscosità, indipendenza dalla temperatura, decadimenti di fluorescenza monoesponenziali che consentono una semplice interpretazione dei dati, sufficiente solubilità in acqua e bassa citotossicità13,14. Valutazioni quantitative della microviscosità utilizzando rotori a base di BODIPY e FLIM sono state precedentemente dimostrate su cellule tumorali in vitro, sferoidi tumorali multicellulari e tumori di topo in vivo15,16.
Qui, presentiamo una descrizione dettagliata delle metodologie di sondaggio sequenziale per lo studio del metabolismo cellulare e della viscosità della membrana plasmatica nelle cellule tumorali in vitro mediante FLIM. Per evitare la contaminazione della fluorescenza endogena relativamente debole con la fluorescenza del rotore basato su BODIPY, l'imaging dello stesso strato di cellule viene eseguito in sequenza con la fluorescenza di NAD(P)H e FAD prima visualizzata. La durata di fluorescenza dei cofattori viene misurata nel citoplasma e la durata di fluorescenza del rotore viene misurata nelle membrane plasmatiche delle cellule mediante la selezione manuale delle zone corrispondenti come regioni di interesse. Il protocollo è stato applicato per correlare lo stato metabolico e la viscosità per diverse linee cellulari tumorali e per valutare i cambiamenti dopo la chemioterapia.
Il protocollo per la preparazione dei campioni FLIM non differisce da quello per la microscopia a fluorescenza confocale. Una volta acquisiti i dati, il compito principale è quello di estrarre il tempo di vita della fluorescenza dai dati grezzi. Le prestazioni del protocollo sono dimostrate utilizzando cellule HCT116 (carcinoma colorettale umano), CT26 (carcinoma del colon murino), HeLa (carcinoma cervicale umano) e huFB (fibroblasti della pelle umana).
Protocollo
1. Descrizione della configurazione minima per eseguire FLIM
- Per eseguire questo esperimento, assicurarsi che sia disponibile la configurazione richiesta: un microscopio confocale invertito, un laser pulsato, tipicamente un ps o fs, con il segnale di sincronizzazione, un rivelatore di conteggio dei fotoni veloce (risposta temporale 150 ps) e un'elettronica di conteggio dei fotoni, porte di uscita e di ingresso disponibili per il rivelatore e il laser, rispettivamente, sul microscopio, gli impulsi dell'orologio di scansione dal controller di scansione del microscopio, la testa di scansione del microscopio con i combinatori di raggi laser e i principali dicroici beam splitter adatti alla lunghezza d'onda del laser utilizzato per FLIM.
- Se per FLIM si utilizza l'eccitazione a due fotoni, assicurarsi che il microscopio contenga la porta NDD.
- Per gli studi sulle cellule di mammifero, in particolare per gli esperimenti a lungo termine, assicurarsi di mantenere un incubatore di CO2 alla temperatura desiderata.
NOTA: Per il sistema utilizzato in questo esperimento, vedere Tabella dei materiali.
2. Preparazione delle cellule per la microscopia
- Far crescere le cellule regolarmente in un incubatore a 37 °C con il 5% di CO2 e un'atmosfera umida.
- Per la microscopia, preparare la sospensione cellulare in un terreno di coltura completo alla concentrazione di 1 x 106 cellule/mL.
NOTA: Le concentrazioni delle celle e le condizioni dei mezzi dipendono dalle cellule. Il numero di cellule utilizzate per la semina e il tempo di incubazione devono essere adattati per ottenere una confluenza del 70%-80% nel piatto microscopico. - Seminare le cellule su piastre per colture cellulari da 35 mm con fondo di vetro (1 x 10,5 cellule in 100 μl per piastra) utilizzando una pipetta automatica da 200 μl.
NOTA: Utilizzare piastre con fondo di vetro a griglia per la semina delle cellule per monitorare le cellule negli stessi campi microscopici visivi in dinamica.- Durante la semina manuale, assicurarsi che la punta della pipetta non graffi il fondo o i lati della piastra per evitare di danneggiare il fondo.
- Posizionare la capsula nell'incubatore di CO2 (37 °C, 5% CO2, atmosfera umida) e incubare le cellule per 24 ore.
- Dopo 24 ore, rimuovere la capsula dall'incubatrice e controllare la morfologia e la confluenza delle cellule al microscopio ottico. Se le cellule non hanno raggiunto circa l'80% di confluenza, incubare per altre 24 ore.
- Rimuovere delicatamente il vecchio terreno dalla piastra utilizzando una pipetta automatica da 1.000 μl e aggiungere 2 mL di terreno DMEM senza rosso fenolo (ad esempio, DMEM Life o FluoroBrite).
NOTA: Per l'imaging è possibile utilizzare diversi terreni di coltura. Evitare il rosso fenolo nel terreno quando si utilizzano le cellule per la microscopia. - Mettere il piatto nell'incubatrice per 60-120 minuti per consentire l'adattamento delle cellule.
3. FLIM dei cofattori metabolici
- Posizionare il piatto con fondo di vetro con le cellule (dal passaggio 2.7) sul tavolino del microscopio.
- Fare clic sulla scheda Localizza nel software del microscopio a scansione laser (ad esempio, ZEN - ZEISS Efficient Navigation), quindi fare clic su Luce di trasmissione (TL) per accendere la luce.
- Trova il piano focale del campione osservandolo attraverso l'oculare a livello della fetta centrale delle celle, dove un quadrato è occupato al massimo dalle celle (con ingrandimento 40x).
- Fare clic sul pulsante OFF per spegnere la luce.
- Apri la scheda Acquisizione . Per ottenere immagini di intensità di trasmissione e autofluorescenza di NAD(P)H endogeno, inserire le seguenti impostazioni: Lunghezza d'onda di eccitazione: modalità a due fotoni 750 nm, Intervallo di registrazione: 450-490 nm, Potenza laser: 5% (~6 mW), Dimensioni dell'immagine: 1024 x 1024 pixel.
NOTA: La scelta della lunghezza d'onda di eccitazione e dell'intervallo di registrazione si basa sulle caratteristiche spettrali (massima eccitazione e massima emissione) di NAD(P)H17.- Per l'acquisizione dell'immagine utilizzare un obiettivo a immersione in olio С Plan-Apochromat 40x/1,3 NA.
- Premere il pulsante Snap e salvare l'immagine in formato ZEN.
- Per ottenere le immagini di trasmissione e intensità di autofluorescenza del FAD, modificare la lunghezza d'onda di eccitazione a 900 nm. Impostare l'intervallo di registrazione: 500-550 nm, la potenza del laser: 9% (~6 mW) e la dimensione dell'immagine: 1024 x 1024 pixel.
NOTA: La scelta della lunghezza d'onda di eccitazione e dell'intervallo di registrazione si basa sulle caratteristiche spettrali (massima eccitazione e massima emissione) del FAD18.- Per l'acquisizione delle immagini utilizzare un obiettivo a immersione in olio С Plan-Apochromat 40x/1,3 NA.
- Premere il pulsante Snap e salvare l'immagine in formato ZEN.
- Per NAD(P)H, impostare i parametri come descritto al punto 3.5 nel software del microscopio a scansione laser. Modificare la dimensione dell'immagine a 256 x 256 pixel.
- Inserire i seguenti parametri nel menu del software operativo SPCM (Single Photon Counting Modules) del modulo FLIM: Tempo di raccolta: 60 s; Gamma TAC: 5.00E-8; Limite CFD basso: -29.41; Risoluzione ADC: 256, Dimensione immagine: 256 x 256 pixel.
- Scansiona il campione per 60 s, interrompi la scansione e salva l'immagine FLIM ottenuta di NAD(P)H.
- Controllare i dati FLIM ottenuti. Per questo, apri i dati grezzi nel software di immagine, seleziona un pixel nel citoplasma della cellula posizionando il cursore su di esso e analizza il decadimento della fluorescenza in questo pixel. L'intensità dei pixel dovrebbe essere di ≥3.000 fotoni per curva di decadimento al binning 1.
NOTA: Se il numero di fotoni è inferiore a 3.000, aumentare la potenza del laser o il tempo di raccolta delle immagini, controllando la morfologia delle celle e la velocità di conteggio dei fotoni. In genere, se il calo del tasso di conteggio supera il 10% del valore iniziale, si verifica il fotosbiancamento. - Per FAD, impostare i parametri come descritto al punto 3.7 nel software del microscopio a scansione laser. Cambia le dimensioni dell'immagine a 256 x 256 pixel.
- Inserire i seguenti parametri nel menu del software operativo SPCM (Single Photon Counting Modules) del modulo FLIM: Tempo di raccolta: 60 s; Gamma TAC: 5.00E-8; Limite CFD basso: -29.41; Risoluzione ADC: 256, Dimensione immagine: 256 x 256 pixel.
- Scansiona il campione per 60 s. Interrompere la scansione e salvare l'immagine FLIM ottenuta di FAD.
NOTA: I parametri indicati nei passaggi 3.10 e 3.15 sono specifici per l'elettronica e il rivelatore utilizzato. - Controllare i dati ottenuti come descritto nel passaggio 3.12.
- Ripetere i passaggi 3.5-3.16 per registrare immagini FLIM da diversi campi visivi.
4. Colorazione delle cellule con il rotore molecolare fluorescente
NOTA: Le celle vengono visualizzate nella soluzione fluorescente del rotore molecolare senza lavaggio a temperatura ambiente (~20 °C) per rallentare l'internalizzazione del rotore. La viscosità della membrana dipende dalla temperatura, come dimostrato nei nostri precedenti lavori19,20. Il tavolino a temperatura controllata del microscopio deve essere spento in anticipo, cioè prima che il rotore venga aggiunto alle celle. Per la nostra configurazione, il raffreddamento del palco richiede circa 10 minuti.
- Preparare una soluzione madre generale del rotore molecolare fluorescente BODIPY 2 (Stock 1, 25,7 mM).
- Aprire BODIPY 2 in un ambiente sterile e pesare circa 2 mg, utilizzando una bilancia accurata. Posizionarlo con cura in una provetta da microcentrifuga.
- Utilizzare una pipetta automatica da 20 μl per aggiungere 3 μl di un solvente adatto (ad es. DMSO).
- Una volta che il campione si è completamente sciolto nel DMSO, aggiungere 297 μL di PBS sterile e mescolare accuratamente utilizzando una pipetta automatica da 200 μL.
NOTA: Conservare la soluzione madre in frigorifero a +4 °C in confezione scura. Una volta risospeso, può essere conservato in frigorifero per diversi mesi.
- Preparare uno Stock 2 (8,9 mM) aggiungendo 25 μL della soluzione madre generica (Stock 1) a una provetta per microcentrifuga, seguiti da 48 μL di PBS sterile. Miscelare delicatamente utilizzando una pipetta automatica da 200 μl.
NOTA: Utilizzare il materiale di produzione 2 per preparare il materiale di colorazione finale, che viene applicato per la colorazione cellulare poiché è richiesta la concentrazione micromolare. - Riposizionare delicatamente il terreno di coltura nella piastra (dal passaggio 3.1) con una soluzione di Hank ghiacciata senza Ca2+/Mg2+ e incubare le cellule a +4 °C per 3 minuti.
NOTA: L'uso di una soluzione ghiacciata e l'incubazione a +4 °C rallenta l'internalizzazione del rotore molecolare e la colorazione locale della membrana persiste per 20-30 minuti. - Preparare la soluzione colorante finale contenente 4,5 μM di BODIPY 2 aggiungendo 1 μL di Stock 2 a 999 μL di soluzione di Hank ghiacciata o PBS.
NOTA: La concentrazione di BODIPY 2 nella soluzione di colorazione finale può essere aumentata a ~10 μM senza effetti tossici sulle cellule, il che si traduce in una colorazione più efficiente e in un maggior numero di fotoni raccolti. A concentrazioni più elevate, può verificarsi un sovraccarico del rivelatore FLIM. - Aspirare la soluzione di Hank dalla piastra di coltura cellulare e sostituirla con una soluzione ghiacciata da 4,5 μM di BODIPY 2. Le cellule vengono visualizzate in soluzione BODIPY 2 senza lavaggio.
5. FLIM del rotore molecolare fluorescente nelle cellule
NOTA: Eseguire sempre la FLIM del rotore molecolare fluorescente dopo la FLIM dei cofattori metabolici perché lo spettro di fluorescenza di BODIPY 2 è sovrapposto all'emissione di cofattori endogeni NAD(Р)H e FAD 12,17,18.
- Trasferire la piastra con le cellule colorate sul tavolino del microscopio (~20 °C) per l'imaging.
- Impostare i seguenti parametri per la modalità a un fotone nel software del microscopio a scansione laser: Eccitazione alla lunghezza d'onda di 488 nm con un laser a ioni argon, Potenza laser 1%-2%, intervallo di registrazione 500-550 nm lunghezza d'onda.
- Per l'acquisizione delle immagini utilizzare un obiettivo a immersione in olio С Plan-Apochromat 40x/1,3 NA.
- Premere il pulsante Live . Avvia la scansione e utilizza il posizionamento XY e Z tramite un tavolino motorizzato integrato, regola la messa a fuoco e ottieni immagini di trasmissione e intensità di fluorescenza delle cellule in una finestra di anteprima. Salvare le immagini ottenute, se necessario.
- Controllare l'immagine di trasmissione e fluorescenza sovrapposta per vedere se la fluorescenza del rotore proviene dalla posizione prevista (membrana plasmatica della cellula).
- Inserire i seguenti parametri nel menu del software SPCM del modulo FLIM: Tempo di raccolta: 60 s; Gamma TAC: 5.00E-8; Limite CFD basso: -29.41; Risoluzione ADC: 256, Dimensione immagine: 256 x 256 pixel.
NOTA: A seconda della configurazione del sistema e dei rilevatori utilizzati per FLIM, i parametri di acquisizione delle immagini possono variare. - Regolare il laser Ti:Sapphire del microscopio su una lunghezza d'onda di 850 nm e la potenza del laser su 1%-2% per FLIM a due fotoni.
- Selezionare la scheda Continua nel software del microscopio a scansione laser, quindi premere Avvia nel software SPCM. Scansiona il campione per 60 s, interrompi la scansione e salva l'immagine FLIM ottenuta.
- Controllare i dati FLIM ottenuti. A tal fine, caricare i dati grezzi nel software SPCImage per l'analisi dei dati FLIM, selezionare un pixel nella membrana della cella posizionando il cursore su di esso e analizzare il decadimento della fluorescenza in questo pixel. L'intensità dei pixel dovrebbe essere di ≥5.000 per decadimento (possibilmente includendo il binning) a un tempo di raccolta ragionevole (60-120 s).
- Ripetere i passaggi 5.4-5.8 per registrare le immagini FLIM delle celle da diversi campi visivi.
NOTA: Le misurazioni FLIM di cellule vive colorate con BODIPY 2 devono essere limitate a ~30 minuti dopo l'aggiunta di BODIPY 2.
6. Analisi dei dati
- Analisi dell'intensità della fluorescenza: rapporto redox
- Apri le immagini dell'intensità di fluorescenza di NAD(P)H e FAD utilizzando ImageJ.
- Evidenziate un'area priva di celle nell'immagine NAD(P)H utilizzando un'opzione circolare o quadrata. Fare clic su Misura, quindi fare clic su Sottrai (selezionare Elabora nel pannello principale, quindi Matematica e sottrai) per sottrarre il valore ottenuto del segnale di fondo.
- Ripetere il passaggio 6.1.2 per l'immagine FAD.
- Ottenere l'immagine del rapporto redox dividendo l'intensità di fluorescenza FAD per l'intensità di fluorescenza NAD(P)H. Fallo selezionando Elabora nel pannello principale, quindi seleziona Calcolatrice immagini e Dividi; selezionare la casella Crea nuova finestra, quindi premere OK.
- Salva l'immagine in formato TIFF.
- Per calcolare il rapporto redox, selezionare la regione del citoplasma nella cellula specifica sull'immagine TIFF e premere il tasto M . Ripetere l'operazione per tutte le celle di interesse.
- Importa la misurazione in un foglio di calcolo.
NOTA: In alternativa, le intensità di fluorescenza di NAD(P)H e FAD nelle cellule possono essere misurate utilizzando il software standard del microscopio e il rapporto redox può essere ottenuto dividendo questi valori nel software del foglio di calcolo.
- Analisi dei dati FLIM: metabolismo
- Importa l'immagine FLIM di NAD(P)H nel software SPCImage.
- Applica un adattamento di decadimento bi-esponenziale all'immagine inserendo 2 nella sezione Componenti .
- Correggi il parametro Offset selezionando la casella corrispondente nel software SPCImage.
- Vai su Opzioni e seleziona Modello. Utilizzare il modello di fitting multiesponenziale incompleto e il metodo di adattamento MLE.
- Regola il binning per ottenere intensità di pixel di ≥5000 fotoni per curva di decadimento.
- Controllare il valore χ2. Il χ2≤ 1,20 indica che il modello utilizzato fornisce un adattamento ragionevole.
- Calcola l'istogramma del tempo di vita della fluorescenza in ogni immagine cliccando sul menu in alto Calcola, e poi su Matrice di decadimento.
- Selezionare l'area nel citoplasma della cellula specifica come regione di interesse.
- Analizza i componenti di durata breve e lunga (τ1 e τ2, rispettivamente) e le ampiezze relative dei componenti di durata (a1 e a2, dove a1 + a2 = 100%) utilizzando l'opzione Colore .
- Esporta le misurazioni in un software per fogli di calcolo.
- Ripetere i passaggi 6.2.8-6.2.10 per ogni cella di interesse.
- Ripetere i passaggi 6.2.1-6.2.11 per l'immagine FAD.
- Analisi dei dati FLIM: viscosità
- Importa l'immagine FLIM nel software SPCImage per l'analisi dei dati FLIM.
- Rimuovere il segno nella casella Scatter .
- Metti 1 nella sezione Componenti , poiché il decadimento della fluorescenza del rotore dovrebbe adattarsi a un modello monoesponenziale.
- Regola il binning per ottenere un'intensità di pixel di ≥5000 fotoni per curva di decadimento.
- Controllare il valore χ2 nella membrana plasmatica. Un valore di χ2≤ 1,20 indica che il modello utilizzato fornisce un adattamento ragionevole. Nel caso di χ2≥ 1,20, l'approssimazione monoesponenziale non è applicabile, tali dati possono indicare l'aggregazione del colorante e devono essere scartati. L'aggregazione rende impossibile l'utilizzo delle curve di calibrazione e porta a stime di viscosità errate.
NOTA: I decadimenti biesponenziali possono essere indicativi di aggregazione. Su un microscopio con modulo FLIM con filtri variabili, questo può essere rilevato testando gli intervalli di lunghezze d'onda di emissione specifici del monomero e dell'aggregato, 500-550 nm e 580-650 nm, come descritto nel riferimento21. - Genera l'istogramma del tempo di vita della fluorescenza τ per ogni immagine cliccando sul menu in alto Calcola, e poi su Matrice di decadimento.
- Selezionare la regione della membrana plasmatica di una singola cellula con decadimento monoesponenziale, χ2≤ 1,20, utilizzando l'opzione ROI.
- Esporta il valore della durata della fluorescenza in un software per fogli di calcolo.
- Ripetere i passaggi 6.3.7-6.3.8 per ogni cella di interesse.
- Convertire i tempi di vita misurati sperimentalmente di BODIPY 2 (in ns) in valori di viscosità (in cP) utilizzando la seguente equazione (precedentemente ottenuta sulla base del grafico di calibrazione di BODIPY in miscele di metanolo/glicerolo):
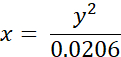
dove x - viscosità (in cP), y - durata di vita della fluorescenza τ (in ns).
NOTA: IRF (Instrument Response Function) è una parte importante del fitting FLIM. In SPCImage l'IRF viene calcolato automaticamente dal fronte di salita delle curve di decadimento della fluorescenza. Nel frattempo, l'IRF può essere registrato utilizzando un campione non fluorescente, ad esempio ceramiche, o un campione che produce un segnale SHG (Second Harmonic Generation), ad esempio collagene, cristalli di urea o zucchero. L'uso degli IRF registrati non è raccomandato se nel software è disponibile un'opzione per calcolarli.
Risultati
Utilizzando il protocollo qui descritto, abbiamo visualizzato i cofattori metabolici e la viscosità microscopica della membrana in cellule vive in coltura utilizzando FLIM. Le misurazioni sono state effettuate in diverse linee cellulari tumorali: carcinoma colorettale umano HCT116, carcinoma del colon murino CT26, carcinoma cervicale umano HeLa Kyoto e fibroblasti cutanei umani huFB.
Il rapporto redox basato sull'intensità della fluorescenza FAD/NAD(P)H e la durata di vita della fluorescenza di NAD(P)H e FAD consentono di valutare lo stato metabolico cellulare (Figura 1, Figura 2). NAD(P)H e FAD localizzati principalmente nel citoplasma delle cellule. Diversi tipi di cellule nel nostro studio hanno mostrato tempi di fluorescenza simili di NAD(P)H e flavine e contributi delle frazioni a breve e lunga vita, il che probabilmente indicava che avevano uno stato metabolico simile in condizioni in vitro . I tempi di vita della fluorescenza e i contributi relativi del NAD(P)H libero (τ1, a1) e legato alle proteine (τ2, a2) sono stati misurati rispettivamente a ~0,54 ns, ~75% e ~2,5 ns, ~25%. Per il FAD quenched (τ1, a1) e non quenched (τ2, a2) i tempi di fluorescenza e i relativi contributi sono stati rispettivamente di ~0,35 ns, ~85% e ~2,0 ns, ~15% (Tabella 1).
Utilizzando il protocollo sviluppato, abbiamo studiato la risposta metabolica delle cellule HCT116 alla chemioterapia con 5-fluorouracile. Il 5-fluorouracile è stato utilizzato alla dose di 4 μM (la concentrazione inibitoria semimassima IC50). Le cellule sono state incubate con il farmaco per 1 ora o 24 ore. Le cellule non trattate fungevano da controllo. FLIM di NAD(P)H nelle cellule di controllo e nelle cellule trattate con 5-fluorouracile ha mostrato che il contributo relativo di NAD(P)H (a1) libero è diminuito da 77,82 ± 1,69% a 66,34 ± 1,71% (p = 0,0001) in 24 ore (Figura 3).
Abbiamo dimostrato la possibilità di utilizzare il protocollo presentato per le misure di viscosità della membrana. BODIPY 2 ha colorato efficacemente la membrana plasmatica delle cellule tumorali (Figura 4). Le linee cellulari tumorali studiate hanno mostrato diversi tempi di fluorescenza del rotore, il che significa che avevano una viscosità della membrana plasmatica diversa. Il tempo di vita più alto della fluorescenza del rotore è stato registrato nelle membrane delle cellule HCT116, 3,19 ± 0,14 ns, che corrispondeva a un valore di viscosità di 492 ± 33 cP. Le membrane delle cellule CT26 erano leggermente meno viscose: la durata della fluorescenza era di 2,98 ± 0,16 ns, la viscosità era di 432 ± 39 cP. Le membrane più fluide di questa serie erano possedute dalle celle HeLa Kyoto - 2,65 ± 0,15 ns, che corrispondevano a 344 ± 36 cP.
La valutazione della viscosità della membrana nei fibroblasti della pelle umana è risultata problematica, in primo luogo, perché BODIPY 2 è entrato nelle cellule e si è diffuso diffusamente in tutta la cellula molto rapidamente, e in secondo luogo, perché la sua fluorescenza nella membrana dei fibroblasti aveva un decadimento bi-esponenziale (Figura 4E).
La colorazione chiara della membrana plasmatica di tutte le cellule tumorali studiate si osserva nel periodo di tempo da 5 a 30 minuti dopo l'aggiunta di BODIPY 2 alle cellule (Figura 5). Entro questo periodo, il decadimento della fluorescenza di BODIPY 2 si adatta a un modello monoesponenziale. Un'ulteriore incubazione con BODIPY 2 porta all'internalizzazione del colorante e alla colorazione del citoplasma cellulare insieme alla membrana plasmatica. L'analisi delle curve di decadimento di BODIPY 2 localizzato nel citoplasma ha mostrato decadimenti bi-esponenziali, presumibilmente dovuti alla presenza di aggregati BODIPY 2.
Parallelamente alle valutazioni metaboliche, abbiamo studiato se il 5-fluorouracile inducesse cambiamenti nella viscosità della membrana plasmatica nelle cellule HCT116 (Figura 6). È importante ricordare che la viscosità della membrana è stata misurata solo nelle cellule vitali, poiché nelle cellule arrotondate con morfologia anormale (presumibilmente, cellule morte) il rotore non ha colorato le membrane plasmatiche, ma si è accumulato all'interno delle cellule, dove la sua fluorescenza è decaduta in modo biesponenziale, indicativo dell'aggregazione del rotore22. Il tempo di vita in fluorescenza di BODIPY 2 prima dell'aggiunta di 5-fluorouracile era di 3,27 ± 0,14 ns, che corrisponde a un valore di viscosità di 511 ± 43 cP. Dopo 1 ora di incubazione con 5-fluorouracile, il tempo di vita della fluorescenza è aumentato a 3,75 ± 0,24 ns e la viscosità delle membrane era di 697 ± 80 cP (p < 0,0001). Dopo 24 ore di incubazione con il farmaco, il tempo di vita della fluorescenza è tornato a un livello di controllo di 3,18 ± 0,23 ns e la viscosità era di 516 ± 68 cP. Dalle immagini si può vedere che il numero di cellule nel campo visivo è diminuito significativamente dopo il trattamento. Ciò è dovuto al distacco di cellule alterate nel corso della procedura di colorazione.
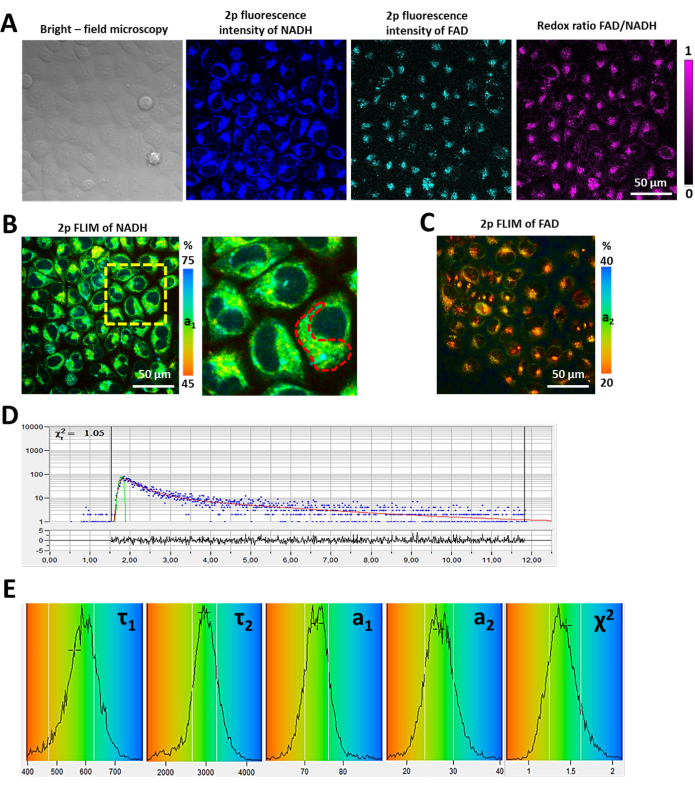
Figura 1: Imaging metabolico delle cellule HeLa utilizzando FLIM. (A) Microscopia a campo chiaro, immagini di intensità di fluorescenza eccitata a due fotoni di NAD(P)H e FAD, e il rapporto redox FAD/NAD(P)H. (B) FLIM A DUE FOTONI DI NAD(P)H.A 1 è il contributo relativo di NAD(P)H libero. L'area ingrandita è mostrata dal quadrato tratteggiato giallo. Nell'immagine ingrandita, la linea rossa tratteggiata indica l'area del citoplasma selezionata per l'analisi. (C) FLIM a due fotoni di FAD. a2 è il contributo relativo del FAD non estinto. (D) Curva di decadimento tipica della fluorescenza di NAD(P)H ottenuta nel software SPCImage: i dati sperimentali (punti blu), adattamento bi-esponenziale (curva rossa), funzione di risposta dello strumento (curva verde). La barra è di 50 μm, applicabile a tutte le immagini. (E) Distribuzioni rappresentative dei parametri del tempo di vita della fluorescenza (τ1, τ2, a1, a2, χ2) nelle cellule HeLa. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: FLIM di NAD(P)H in cellule HCT116, CT26 e huFB. La barra è di 50 μm, applicabile a tutte le immagini. Le immagini codificate a colori mostrano il contributo relativo del NAD(P)H libero (a1, %). Clicca qui per visualizzare una versione più grande di questa figura.
| NADH | ||||
| Linea cellulare | τ1, ns | τ2, ns | a1, % | a2, % |
| HCT116 | 0,55±0,06 | 2,55±0,11 | 74.74±1.83 | 25.26±1.83 |
| Motore CT26 | 0,53±0,03 | 2.51±0.09 | 77.18±2.06 | 22.82±2.06 |
| HeLa Kyoto | 0,52±0,05 | 2,61±0,21 | 76.41±1.85 | 23.59±1.85 |
| huFB | 0,51±0,05 | 2,47±0,38 | 78.36±4.17 | 21.64±4.17 |
| MODA | ||||
| HCT116 | 0,38±0,03 | 1,97±0,14 | 80.86±4.19 | 19.14±4.19 |
| Motore CT26 | 0,36±0,03 | 2.01±0.08 | 85.01±1.65 | 14.99±1.65 |
| HeLa Kyoto | 0,39±0,04 | 2.01±0.09 | 86.01±2.14 | 13.99±2.14 |
| huFB | 0,37±0,03 | 2.11±0.19 | 81.82±3.51 | 18.18±3.51 |
Tabella 1: Parametri del tempo di vita della fluorescenza di NAD(P)H e FAD in varie linee cellulari. Media ± SD, n = 25-50 cellule.
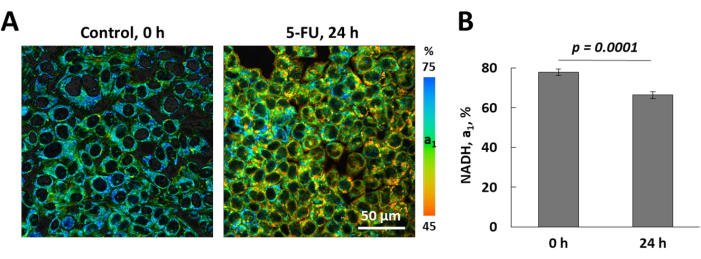
Figura 3: FLIM di NAD(P)H in cellule HCT116 dopo chemioterapia. (A) Il contributo relativo del NADH libero (a1,%) prima (controllo) e nelle 24 ore successive alla chemioterapia con 5-fluorouracile. La barra è di 50 μm. (B) Quantificazione di a1 NAD(P)H nelle cellule di controllo e trattate. Media ± SD, n = 25-50 cellule. Clicca qui per visualizzare una versione più grande di questa figura.
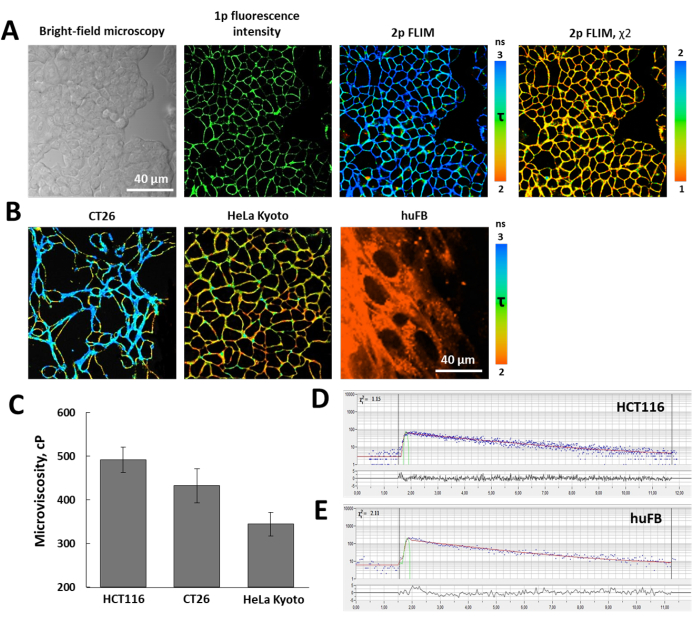
Figura 4: Mappatura della viscosità della membrana plasmatica di cellule tumorali in coltura utilizzando rotore molecolare fluorescente BODIPY 2 e FLIM. (A) Microscopia a campo chiaro, immagine dell'intensità di fluorescenza eccitata a un fotone di BODIPY 2, FLIM eccitata a due fotoni e distribuzione del valore χ2 . (B) Immagini FLIM di cellule CT26, HeLa e huFB colorate con BODIPY 2. Le immagini codificate a colori mostrano il tempo di fluorescenza di BODIPY 2 (τ, ns). La barra è di 40 μm, applicabile a tutte le immagini. (C) Quantificazione della microviscosità della membrana plasmatica nelle cellule tumorali. Media ± SD, 30-50 cellule. (D) Curva di decadimento della fluorescenza tipica di BODIPY 2 ottenuta da cellule tumorali HCT116 nel software SPCImage: i dati sperimentali (punti blu), adattamento bi-esponenziale (curva rossa), funzione di risposta dello strumento (curva verde). (E) Curva di decadimento della fluorescenza di BODIPY 2 ottenuta dai fibroblasti huFb. Questo adattamento è inaccettabile a causa dello scarso χ2. Clicca qui per visualizzare una versione più grande di questa figura.
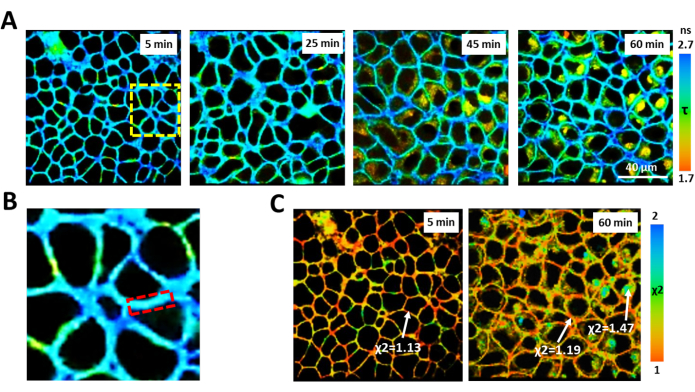
Figura 5: Monitoraggio della durata di fluorescenza del rotore molecolare BODIPY 2 nelle cellule HeLa. (A) FLIM delle cellule nei punti temporali indicati dopo l'aggiunta di BODIPY 2. (B) Area ingrandita mostrata dal quadrato tratteggiato in giallo in A (5 min). La linea rossa tratteggiata indica l'area della membrana plasmatica selezionata per l'analisi. (C) Distribuzione del valore χ2 dopo 5 minuti e 60 minuti di incubazione con BODIPY 2. BODIPY 2 localizzato nella membrana plasmatica mostra decadimento monoesponenziale con χ2≤ 1.2. La BODIPY 2 localizzata nel citoplasma ha una scarsa χ2 con fitting monoesponenziale. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6. Mappatura della viscosità della membrana plasmatica in cellule e cellule HCT116 non trattate dopo esposizione di 1 e 24 ore al 5-fluorouracile. (A) Immagini rappresentative del tempo di vita della fluorescenza delle cellule HCT116 di controllo e trattate (B) Quantificazione della microviscosità della membrana plasmatica nelle cellule. Media ± SD, 20-30 cellule. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Questo protocollo illustra le possibilità di FLIM per l'analisi multiparametrica, funzionale e biofisica delle cellule tumorali. La combinazione dell'imaging metabolico ottico basato sulla fluorescenza endogena e delle misure della viscosità della membrana plasmatica mediante marcatura esogena con rotore molecolare fluorescente ci consente di caratterizzare le interconnessioni tra questi due parametri in cellule tumorali vive in una coltura cellulare e di seguire i cambiamenti in risposta alla chemioterapia.
La microscopia per imaging a fluorescenza eccitata a due fotoni (FLIM) è una tecnica promettente per una valutazione non invasiva, sensibile, quantitativa e ad alta risoluzione di uno stato funzionale di cellule e tessuti viventi. Attualmente sono disponibili sensori chimici fluorescenti e con codifica genetica basati sul rilevamento del tempo di vita per visualizzare vari parametri fisiologici e fisico-chimici, tra cui concentrazioni di ioni, pH, attività enzimatica, ipossia, ciclo cellulare, viscosità, potenziale di membrana e altri. Inoltre, FLIM può catturare le caratteristiche metaboliche delle cellule utilizzando la fluorescenza intrinseca dei cofattori NAD(P)H e flavina. A differenza dell'intensità della fluorescenza, la durata della fluorescenza consente di superare le difficoltà associate a una concentrazione di fluorofori sconosciuta e/o instabile, al fotosbiancamento, alle configurazioni dello strumento (ad esempio, intensità di eccitazione, illuminazione non uniforme e lunghezza del percorso ottico), agli eventi di assorbimento e diffusione23. Grazie alla capacità di misurare più parametri da un singolo campione, FLIM diventa una modalità sempre più popolare nelle applicazioni biomediche.
A seconda del microscopio e del laser, è possibile utilizzare diversi obiettivi e potenze laser. Per l'eccitazione a due fotoni, sono preferibili obiettivi a immersione in olio o in acqua con un'elevata apertura numerica (>1). La potenza del laser viene selezionata in base all'obiettivo disponibile e deve essere regolata per evitare problemi di fotosbiancamento e saturazione. La microscopia multifotone viene normalmente eseguita con una potenza laser inferiore a 10 mW, che è considerata sicura per le cellule vive. La dimensione dell'immagine viene selezionata in base alle preferenze personali.
Uno degli aspetti più utili della tecnica FLIM è la capacità di esaminare in modo non invasivo i processi metabolici su base label-free, basandosi sulla fluorescenza endogena dei coenzimi, NAD(P)H e FAD/FMN. La capacità di sondare il metabolismo a livello di singola cellula, dinamicamente nel tempo, in cellule vive utilizzando fonti di contrasto interamente endogene offre vantaggi significativi rispetto ai saggi metabolici convenzionali, come il rilevamento di metaboliti (ad esempio, glucosio, glutammina, lattato, ATP) o l'attività di geni o enzimi correlati al metabolismo. La FLIM metabolica ha dimostrato di essere un approccio altamente sensibile per differenziare le cellule cancerose e normali 24,25,26,27 e rilevare le risposte alle terapie antitumorali in vitro e in vivo28,29,30,31. In precedenza, questa metodologia è stata utilizzata nel nostro laboratorio per analizzare lo stato metabolico delle cellule tumorali sottoposte ad apoptosi32, per seguire la risposta metabolica agli agenti chemioterapici 30,33,34 e i cambiamenti metabolici che accompagnano le interazioni tra cellule tumorali e fibroblasti 35.
I decadimenti di fluorescenza di NAD(P)H e delle flavine sono comunemente descritti da una funzione bi-esponenziale. Per un adeguato adattamento bi-esponenziale, le intensità dei pixel dovrebbero essere di ≥5.000 fotoni per curva di decadimento, possibilmente con binning. Sul sistema utilizzato, 5.000 fotoni per curva di decadimento (binning 1) vengono tipicamente raccolti alla velocità di conteggio dei fotoni 1-2 x 105 fotoni/s.
Nel presente lavoro, abbiamo ottenuto le metriche metaboliche ottiche per diverse linee cellulari tumorali e fibroblasti della pelle umana. Tutti i tempi di vita misurati in fluorescenza di NAD(P)H e flavine sono correlati con quelli riportati in letteratura36,37. A causa delle specifiche del metabolismo, l'intensità della fluorescenza delle flavine nelle linee cellulari tumorali è tipicamente inferiore rispetto alle cellule normali (ad esempio, fibroblasti o cellule staminali mesenchimali). Pertanto, l'analisi del tempo di vita della fluorescenza nelle cellule tumorali richiede l'applicazione del pixel binning per regolare il numero di fotoni a ≥5.000. In particolare, l'opzione di binning viene utilizzata se l'aumento della potenza del laser o del tempo di raccolta delle immagini è impossibile a causa di alterazioni morfologiche (un indicatore di un danno cellulare) o del fotosbiancamento. Nel presente protocollo, un fattore di binning di 3-4 viene utilizzato per le flavine nelle cellule tumorali e 2-3 nei fibroblasti. Un fattore di binning 1-2 viene utilizzato per NAD(P)H nelle celle con le impostazioni indicate. Tieni presente che un binning di grandi dimensioni riduce la risoluzione dell'immagine.
Ciascuno dei valori estratti dal decadimento bi-esponenziale (τ1, a1, τ2, a2) contiene informazioni su diversi parametri dell'ambiente molecolare. In particolare, il componente τ2 a lungo tempo di vita dipende dall'insieme delle proteine leganti il NAD(P)H nel caso del NAD(P)H e dal contributo della FMN nel caso delle favine. Le ampiezze a1 e a2 riflettono le quantità relative di due frazioni dei fluorofori: NAD(P)H libero e legato, flavine temprate e non estinte. Tra gli altri, i valori a1 e a2 sono le metriche più variabili in base alle perturbazioni metaboliche38. Se i valori di τ1 e/o τ2 variano per qualche motivo, anche la durata media della fluorescenza (τm = (a1· τ1 + a2· τ2) ∕ (a1+a2)) può essere una metrica rilevante. Tenendo conto del fatto che la fluorescenza delle flavine nelle cellule tumorali è tipicamente debole, i contributi relativi del NAD(P)H libero e legato (a1, a2 o il loro rapporto a1/a2) sono più frequentemente utilizzati come biomarcatore dei cambiamenti metabolici.
L'interpretazione dei dati NAD(P)H FLIM dal punto di vista biochimico è piuttosto semplice, poiché il NADH è principalmente coinvolto nel metabolismo energetico. L'unico fluoroforo che può complicare l'analisi dei dati è la forma fosforilata legata alle proteine di NADH, NADPH (τ 4.4 ns)39. Tuttavia, è presente nelle cellule tumorali in concentrazioni inferiori rispetto al NADH40. I tempi di vita della fluorescenza dei cofattori flavinici sono più difficili da interpretare, poiché partecipano a una serie di processi, oltre alla produzione di energia, e il contributo della FMN (τ ~4,7 ns) può essere essenziale.
Nel presente protocollo, utilizziamo il metodo dei minimi quadrati ponderati dell'adattamento del decadimento della fluorescenza come gold standard. Nel frattempo, gli approcci non fitting, cioè non parametrici, all'analisi dei dati FLIM sono sempre più sviluppati 41,42,43,44.
Oltre alle misure del tempo di vita della fluorescenza, il nostro protocollo fornisce una metodologia per la valutazione del rapporto redox ottico mediante microscopia a scansione laser. Il rapporto redox ottico, introdotto per la prima volta da Britton Chance, come rapporto tra le intensità di fluorescenza dei cofattori ridotti e ossidati, rappresenta una semplice metrica dello stato redox cellulare. Diverse equazioni possono essere utilizzate per calcolare il rapporto redox ottico, ad esempio NAD(P)H/FAD29,31 o FAD/(FAD+NAD(P)H)38,45. La scelta dell'equazione dipende dal compito e dalle caratteristiche del segnale. È stato riportato che il rapporto redox ottico è ben correlato con il rapporto redox biochimico di NAD+/(NADH+NAD+)45 e il tasso di consumo di ossigeno46. Tuttavia, l'interpretazione del rapporto redox ottico non è sempre semplice perché l'intensità della fluorescenza è influenzata da molti fattori oltre alla concentrazione dei fluorofori (vedi sopra). Pertanto, nel corso dell'acquisizione dei dati, è necessario prestare maggiore attenzione agli effetti del fotobleaching nelle cellule visualizzate. Poiché la potenza di eccitazione influisce sull'intensità dell'emissione, la potenza del laser alla lunghezza d'onda specifica (750 nm o 900 nm) dovrebbe essere la stessa per tutte le immagini raccolte per le misure del rapporto redox.
Abbiamo utilizzato la FLIM metabolica per lo studio delle alterazioni metaboliche nelle cellule di cancro colorettale umano HCT116 indotte dal 5-fluorouracile, un farmaco chemioterapico standard della classe degli antimetaboliti (analogo della pirimidina). Il trattamento con 5-fluorouracile ha determinato un aumento della frazione di NAD(P)H legata dopo 24 ore, mentre non sono stati osservati cambiamenti metabolici dopo 1 ora di incubazione (dati non mostrati). Nei nostri studi precedenti su colture cellulari e modelli tumorali murini, abbiamo dimostrato che cambiamenti simili nel tempo di vita della fluorescenza del NAD(P)H si sono sviluppati in risposta a farmaci con diversi meccanismi d'azione, ad esempio cisplatino, paclitaxel, irinotecan, e hanno associato questo all'inibizione della proliferazione cellulare30,33. Ipotizziamo che i cambiamenti osservati nella fluorescenza del NAD(P)H potrebbero essere causati da uno spostamento del metabolismo energetico a un livello più ossidativo dovuto all'attivazione di OXPHOS e/o alla diminuzione del tasso di glicolisi. Sebbene la FLIM da sola non consenta di specificare esattamente i cambiamenti nelle vie metaboliche, identifica i biomarcatori intrinseci della risposta cellulare alla terapia.
Sebbene sia stato utilizzato questo protocollo per visualizzare i cambiamenti metabolici solo nei punti temporali selezionati (0, 1 e 24 ore), in linea di principio può essere utilizzato per l'imaging dinamico di cellule vive. Negli esperimenti prolungati destinati all'imaging ripetuto delle stesse cellule, è necessario considerare l'equilibrio ottimale tra la quantità di dati acquisiti e il potenziale di danno cellulare e fotobleaching causato da un'eccessiva esposizione al laser.
Nell'attuale protocollo, l'imaging del metabolismo e della viscosità è stato eseguito in sequenza (il metabolismo è stato visualizzato per primo) perché la fluorescenza della sonda di viscosità BODIPY 2 è sovrapposta a NAD(P)H (leggermente) e flavine (significativamente). Nel caso di NAD(P)H, questo problema può essere risolto utilizzando filtri molto stretti per una separazione più accurata dei segnali. Nel caso del FAD, questo è problematico, perché il suo spettro di emissione corrisponde strettamente allo spettro di emissione di BODIPY 2. Poiché il rotore molecolare entra nella cellula molto rapidamente, pur conservando una concentrazione sufficiente nella membrana plasmatica, la sua fluorescenza nel citoplasma distorcerà i decadimenti di fluorescenza dei cofattori metabolici. L'imaging simultaneo dei due parametri potrebbe essere possibile con l'uso di sonde di viscosità che emettono rosso. Diverse sonde di viscosità fluorescenti rosse sono state sviluppate di recente e utilizzate per monitorare la viscosità mitocondriale e lisosomiale nelle cellule viventi 47,48,49,50.
Poiché il rotore BODIPY 2 è un colorante esogeno che viene trattenuto nella membrana plasmatica solo entro ~30 minuti, il monitoraggio a lungo termine della viscosità nelle stesse cellule non è possibile con questa tecnica e devono essere preparati piatti di coltura separati per diversi punti temporali dell'esperimento. A tempi di incubazione più lunghi (>30 minuti), può verificarsi l'endocitosi di BODIPY 2 e la sua aggregazione all'interno delle cellule, che può distorcere i decadimenti della fluorescenza registrati dalla membrana plasmatica, poiché un certo volume del citoplasma viene inevitabilmente catturato durante l'elaborazione dei dati FLIM (tramite binning). Inoltre, la concentrazione del rotore nella membrana plasmatica diminuisce dopo ~30-60 minuti, il che si traduce in un tasso di conteggio dei fotoni inferiore.
Come mostrato in questo studio, la viscosità della membrana plasmatica delle cellule tumorali si trova nell'intervallo ~340-490 cP e differisce per le diverse linee di cellule tumorali. Nei fibroblasti, non siamo riusciti a misurare la viscosità della membrana perché il rotore BODIPY 2 mostrava un decadimento della fluorescenza bi-esponenziale nella membrana e si internalizzava nelle cellule molto rapidamente. Supponiamo che ciò sia associato a specifiche proprietà biofisiche della membrana plasmatica dei fibroblasti. La membrana dei fibroblasti normali ha dimostrato di essere più fluida (meno viscosa) rispetto alla membrana delle cellule tumorali51. È noto che l'aumento della fluidità della membrana plasmatica favorisce la motilità cellulare, insita nelle cellule del fenotipo mesenchimale, come i fibroblasti.
Dopo 1 ora di esposizione al 5-fluorouracile, abbiamo osservato un aumento statisticamente significativo della viscosità della membrana plasmatica delle cellule tumorali HCT116 in coltura. A differenza del 5-fluorouracile, il cisplatino ha causato una diminuzione non significativa della viscosità della membrana delle cellule tumorali in questo puntotemporale 22, il che indicava che il tipo e la dinamica dei cambiamenti erano specifici del farmaco.
Nel complesso, questi risultati mostrano che la microviscosità della membrana plasmatica non è direttamente correlata con l'attività metabolica delle cellule. In conclusione, si prega di notare che mentre abbiamo utilizzato il microscopio Carl Zeiss LSM 880 e il modulo Becker&Hickl FLIM, con le impostazioni appropriate, è possibile utilizzare con successo anche altri sistemi di microscopia confocale e/o FLIM. Con l'avanzamento dei metodi di analisi dei dati FLIM, sarà possibile la segmentazione e l'elaborazione automatica delle immagini. I protocolli qui dimostrati per FLIM del metabolismo e della microviscosità nelle cellule tumorali in coltura possono essere utilizzati in modo indipendente e facilmente applicati ad altri tipi di cellule e a diversi interventi.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Lo sviluppo del protocollo di imaging metabolico è stato supportato dal Ministero della Salute della Federazione Russa (Incarico governativo, registrazione n. АААА-А0-120022590098-0). Lo studio della viscosità è stato sostenuto dalla Russian Science Foundation (Progetto n. 20-14-00111). Gli autori sono grati ad Anton Plekhanov (PRMU) per il suo aiuto nella produzione video.
Materiali
| Name | Company | Catalog Number | Comments |
| Item/Device | |||
| Cell culture incubator | Sanyo | 37°C, 5% CO2, humidified atmosphere | |
| Centrifuge 5702 R | Eppendorf | 5703000010 | |
| imageJ 1.53c | Wayne Rasband (NIH) | ||
| FLIM module Simple Tau 152 TCSPC (in LSM 880) | Becker & Hickl GmbH | ||
| Laminar flow hood | ThermoFisher Scientific | ||
| Leica microscope DFC290 | Leica Microsystems | ||
| LSM 880 confocal microscope | Carl Zeiss | ||
| Ti:Sapphire femtosecond laser Mai Tai | Spectra Physics | ||
| Microscope incubator XLmulti S DARK LS | PeCon GmbH | 273-800 050 | |
| Mechanical pipettor | Sartorius mLINE | volume 0.5-10 μL; 20-200 μL; 100-1000 μL | |
| Oil-immersion objective C-Apochromat 40×/1.2 NA W Korr (in LSM 880) | Carl Zeiss | 421767-9971-790 | |
| Power-Tau 152 module with the detector HPM-100-40 | Becker&Hickl GmbH | ||
| SPCImage software | Becker & Hickl GmbH | SPC 9.8; SPCImage 8.3 | |
| ZEN software | Carl Zeiss | ZEN 2.1 SP3 (black), Version 14.0.0.201 | |
| Reagent/Material | |||
| 5-fluorouracil | Medac GmbH | 3728044 | |
| DMEM | Gibco, Life Technologies | 31885023 | |
| DMSO | PanEco | F135 | |
| FBS | Hyclone | A3160801 | |
| FluoroBright DMEM | Gibco, Life Technologies | A1896701 | |
| Hank’s solution without Ca2+/Mg2+ | Gibco, Life Technologies | 14175 | |
| l-Glutamine | PanEco | F032 | |
| Mammalian cells | HCT116, CT26, HeLa Kyoto, huFB | ||
| Molecular rotor BODIPY 2 | Synthesized and Supplied by Marina Kuimova Group, Imperial College London | ||
| Penicillin/streptomycin | PanEco | A065 | |
| Tissue culture dish with cover glass-bottom FluoroDishes | World Precision Instruments, Inc | ||
| Trypsin- EDTA 0.25% | PanEco | P034 | |
| Versen buffer | PanEco | R080p |
Riferimenti
- Vazquez, A., et al. Cancer metabolism at a glance. Journal of Cell Science. 129 (18), 3367-3373 (2016).
- Li, Z., Zhang, H. Reprogramming of glucose, fatty acid, and amino acid metabolism for cancer progression. Cellular and Molecular Life Sciences. 73 (2), 377-392 (2015).
- Strickaert, A., et al. Cancer heterogeneity is not compatible with one unique cancer cell metabolic map. Oncogene. 36 (19), 2637-2642 (2016).
- Szlasa, W., Zendran, I., Zalesińska, A., Tarek, M., Kulbacka, J. Lipid composition of the cancer cell membrane. Journal of Bioenergetics and Biomembranes. 52, 321-342 (2020).
- Preta, G. New insights into targeting membrane lipids for cancer therapy. Frontiers in Cell and Developmental Biology. 8, 571237(2020).
- Kojima, K. Molecular aspects of the plasma membrane in tumor cells. Nagoya Journal of Medical Science. 56, 1-18 (1993).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 071203(2020).
- Becker, W. Advanced time-correlated single photon counting applications. Springer Series in Chemical Physics. , Springer. (2015).
- Shirmanova, M. V., Shcheslavskiy, V. I., Lukina, M. M., Becker, W., Zagaynova, E. V. Exploring tumor metabolism with time-resolved fluorescence methods: From single cells to a whole tumor. Multimodal Optical Diagnostics of Cancer. 3, 133-155 (2020).
- Kalinina, S., Rück, A. FLIM and PLIM in biomedical research. An innovative way to combine autofluorescence and oxygen measurements. Photonics & Lasers in Medicine. 5 (4), 257-266 (2016).
- Kuimova, M. K. Molecular rotors image intracellular viscosity. Chimia. 66 (4), 159-165 (2012).
- Kuimova, M. K. Mapping viscosity in cells using molecular rotors. Physical Chemistry Chemical Physics. 14 (37), 12671(2012).
- Haidekker, M. A., Theodorakis, E. A. Molecular rotors-fluorescent biosensors for viscosity and flow. Organic & Biomolecular Chemistry. 5, 1669-1678 (2007).
- Liu, X., et al. Molecular mechanism of viscosity sensitivity in BODIPY rotors and application to motion-based fluorescent sensors. ACS Sensors. 5 (3), 731-739 (2020).
- Shirmanova, M. V., Shimolina, L. E., Lukina, M. M., Zagaynova, E. V., Kuimova, M. K. Live cell imaging of viscosity in 3D tumour cell models. Multi-Parametric Live Cell Microscopy of 3D Tissue Models. Advances in Experimental Medicine and Biology. Dmitriev, R. 1035, 143-153 (2017).
- Shimolina, L. E., et al. Imaging tumor microscopic viscosity in vivo using molecular rotors. Scientific Reports. 7, 41097(2017).
- Scott, T. G., Spencer, R. D., Leonard, N. J., Weber, G. Synthetic spectroscopic models related to coenzymes and base pairs. V. Emission properties of NADH. Studies of fluorescence lifetimes and quantum efficiencies of NADH, AcPyADH, [reduced acetylpyridineadenine dinucleotide] and simplified synthetic models. Journal of the American Chemical Society. 92 (3), 687-695 (1970).
- Harvey, R. A., Damle, S. A fluorescent modification of flavin adenine dinucleotide. FEBS Letters. 26 (1-2), 341-343 (1972).
- Kubánková, M., Summers, P., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Microscopic viscosity of neuronal plasma membranes measured using fluorescent molecular rotors: Effects of oxidative stress and neuroprotection. ACS Applied Materials and Interfaces. 11, 36307-36315 (2019).
- Kubánková, M., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Molecular rotors report on changes in live cell plasma membrane microviscosity upon interaction with beta-amyloid aggregates. Soft Matter. 14, 9466-9474 (2018).
- Wu, Y., et al. Molecular rheometry: Direct determination of viscosity in Lo and Ld lipid phases via fluorescence lifetime imaging. Physical Chemistry Chemical Physics. 15 (36), 14986(2013).
- Shimolina, L. E., et al. Mapping cisplatin-induced viscosity alterations in cancer cells using molecular rotor and fluorescence lifetime imaging microscopy. Journal of Biomedical Optics. 25 (12), 126004(2020).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014(2007).
- Rück, A., Hauser, C., Mosch, S., Kalinina, S. Spectrally resolved fluorescence lifetime imaging to investigate cell metabolism in malignant and nonmalignant oral mucosa cells. Journal of Biomedical Optics. 19 (9), 096005(2014).
- Lee, D. -H., Li, X., Ma, N., Digman, M. A., Lee, A. P. Rapid and label-free identification of single leukemia cells from blood in a high-density microfluidic trapping array by fluorescence lifetime imaging microscopy. Lab on a Chip. 18 (9), 1349-1358 (2018).
- Lukina, M. M., et al. Interrogation of tumor metabolism in tissue samples ex vivo using fluorescence lifetime imaging of NAD(P)H. Methods and Applications in Fluorescence. 8 (1), 014002(2019).
- Alam, S. R., et al. Investigation of mitochondrial metabolic response to doxorubicin in prostate cancer cells: An NADH, FAD and Tryptophan FLIM assay. Scientific Reports. 7 (1), 10451(2017).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Lukina, M. M., et al. In vivo metabolic and SHG imaging for monitoring of tumor response to chemotherapy. Cytometry Part A. 95 (1), 47-55 (2019).
- Shah, A. T., et al. Optical metabolic imaging of treatment response in human head and neck squamous cell carcinoma. PLoS One. 9 (3), 90746(2014).
- Sergeeva, T. F., et al. Relationship between intracellular pH, metabolic co-factors, and caspase-3 activation in cancer cells during apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1864 (3), 604-611 (2017).
- Shirmanova, M. V., et al. Chemotherapy with cisplatin: insights into intracellular pH and metabolic landscape of cancer cells in vitro and in vivo. Scientific Reports. 7, 8911(2017).
- Lukina, M. M., et al. Metabolic cofactors NAD(P)H and FAD as potential indicators of cancer cell response to chemotherapy with paclitaxel. Biochimica et Biophysica Acta (BBA) - General Subjects. 1862 (8), 1693-1700 (2018).
- Druzhkova, I. N., et al. The metabolic interaction of cancer cells and fibroblasts - coupling between NAD(P)H and FAD, intracellular pH and hydrogen peroxide. Cell Cycle. 15 (9), 1257-1266 (2016).
- Lakowicz, J., Szmacinski, H., Nowaczyk, K., Johnson, M. Fluorescence lifetime imaging of free and protein bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Vanden Berg, P. A. W., Feenstra, K. A., Mark, A. E., Berendsen, H. J. C., Visser, A. J. W. G. Dynamic conformations of flavin adenine dinucleotide: Simulated molecular dynamics of the flavin cofactor related to the time-resolved fluorescence characteristics. The Journal of Physical Chemistry B. 106 (34), 8858-8869 (2002).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5, 3936(2014).
- Lu, W., Wang, L., Chen, L., Hui, S., Rabinowitz, J. D. Extraction and quantitation of nicotinamide adenine dinucleotide redox cofactors. Antioxidants and Redox Signaling. 28 (3), 167-179 (2018).
- Ranjit, S., Malacrida, L., Jameson, D. M., Gratton, E. Fit-free analysis of fluorescence lifetime imaging data using the phasor approach. Nature Protocols. 13 (9), 1979-2004 (2018).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Smith, J. T., et al. Fast fit-free analysis of fluorescence lifetime imaging via deep learning. Proceedings of the National Academy of Sciences of the United States of America. 116 (48), 24019-24030 (2019).
- Wang, S., Chacko, J. V., Sagar, A. K., Eliceiri, K. W., Yuan, M. Nonparametric empirical Bayesian framework for fluorescence-lifetime imaging microscopy. Biomedical Optics Express. 10 (11), 5497-5517 (2019).
- Quinn, K. P., et al. Quantitative metabolic imaging using endogenous fluorescence to detect stem cell differentiation. Scientific Reports. 3 (1), 3432(2013).
- Hou, J., et al. Correlating two-photon excited fluorescence imaging of breast cancer cellular redox state with seahorse flux analysis of normalized cellular oxygen consumption. Journal of Biomedical Optics. 21 (6), 060503(2016).
- Wang, H., et al. Red-emitting fluorescence probe for sensing viscosity in living cells. Chemical Papers. 74, 1071-1078 (2020).
- Chen, B., et al. Sensing and imaging of mitochondrial viscosity in living cells by a red photoluminescent probe with long lifetime. Chemical Communications. 55, 7410(2019).
- Shen, B., Wang, L. F., Zhi, X., Qian, Y. Construction of a red emission BODIPY-based probe for tracing lysosomal viscosity changes in culture cells. Sensors and Actuators B: Chemical. 304, 127271(2019).
- Chen, T., Chen, Z., Liu, R., Zheng, S. NIR fluorescent probe for detection of viscosity and lysosome imaging in live cells. Organic and Biomolecular Chemistry. 17, 6398(2019).
- Angelucci, C., et al. Epithelial-stromal interactions in human breast cancer: effects on adhesion, plasma membrane fluidity and migration speed and directness. PLoS One. 7 (12), 50804(2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon