Method Article
Microdissezione a cattura laser su tessuti chirurgici per identificare l'espressione genica aberrante nella guarigione delle ferite compromessa nel diabete di tipo 2
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questa tecnica fornisce una guida per infliggere ferite ex vivo , eseguire la microdissezione a cattura laser e quantificare i cambiamenti nell'espressione genica correlati a scarsi processi di guarigione delle ferite nel diabete utilizzando tessuto umano clinicamente rilevante.
Abstract
La prevalenza globale del diabete mellito di tipo 2 (T2DM) sta aumentando a un ritmo rapido. I pazienti con diabete di tipo 2 soffrono di una moltitudine di complicanze e una di queste è la compromissione della guarigione delle ferite. Questo può portare allo sviluppo di piaghe o ulcere del piede che non guariscono e, infine, all'amputazione. Negli individui sani, la guarigione delle ferite segue una sequenza controllata e sovrapposta di eventi che comprende infiammazione, proliferazione e rimodellamento. Nel diabete di tipo 2, uno o più di questi passaggi diventano disfunzionali. Gli attuali modelli per studiare la guarigione delle ferite compromessa nel diabete di tipo 2 includono saggi in vitro su ferite da graffio, equivalenti cutanei o modelli animali per esaminare i meccanismi molecolari alla base della guarigione delle ferite e/o potenziali opzioni terapeutiche. Tuttavia, questi non ricapitolano completamente il complesso processo di guarigione delle ferite nei pazienti con diabete di tipo 2 e i test cutanei umani ex vivo sono problematici a causa dell'etica di prelevare biopsie da pazienti in cui è noto che guariranno male. Qui viene descritta una tecnica in base alla quale i profili di espressione delle cellule specifiche coinvolte nella risposta (dis)funzionale alla guarigione delle ferite nei pazienti con diabete di tipo 2 possono essere esaminati utilizzando il tessuto in eccesso scartato dopo l'amputazione o la chirurgia estetica elettiva. In questo protocollo vengono raccolti campioni di pelle donata, feriti, coltivati ex vivo nell'interfaccia aria-liquido, fissati in diversi punti temporali e sezionati. I tipi cellulari specifici coinvolti nella guarigione delle ferite (ad esempio, cheratinociti epidermici, fibroblasti dermici (papillari e reticolari), il sistema vascolare) vengono isolati mediante microdissezione a cattura laser e le differenze nell'espressione genica vengono analizzate mediante sequenziamento o microarray, con geni di interesse ulteriormente convalidati mediante qPCR. Questo protocollo può essere utilizzato per identificare le differenze intrinseche nell'espressione genica tra pelle poco cicatrizzante e intatta, in pazienti con o senza diabete, utilizzando tessuto normalmente scartato dopo l'intervento chirurgico. Produrrà una maggiore comprensione dei meccanismi molecolari che contribuiscono alle ferite croniche del diabete di tipo 2 e alla perdita degli arti inferiori.
Introduzione
L'incidenza del diabete di tipo 2 è in crescita a livello globale, a causa di un'epidemia di obesità e inattività fisica. In questi pazienti è comune una scarsa guarigione delle ferite e fino al 25% dei pazienti svilupperà una ferita cronica che non guarisce. I meccanismi alla base di questo sono complessi e non completamente compresi, limitando il tasso di scoperta di nuove terapie. Uno dei fattori che contribuiscono a questo è la mancanza di un modello adatto per studiare la guarigione delle ferite nei pazienti con diabete di tipo 2. Pertanto, lo scopo di questo metodo è quello di fornire un modello ex vivo fisiologicamente rilevante per esaminare la guarigione delle ferite in coloro che sono a rischio di ferite croniche, consentendo l'analisi trascrittomica per progredire nell'identificazione di nuovi bersagli terapeutici.
Ci sono diversi modelli attualmente disponibili per studiare la guarigione delle ferite e ognuno ha i suoi punti di forza e di debolezza. I modelli animali in vivo, come il topo db/db2 o i ratti diabetici indotti da streptozotocina3 sono ampiamente utilizzati; Tuttavia, le ferite nei modelli di roditori guariscono per contrazione, che è molto diversa dal meccanismo impiegato nel corpo umano, e hanno un successo limitato nella traduzione negli studi clinici 4,5. I benefici dell'uso di tessuti umani sono ben noti4, ma sono complicati dall'etica di infliggere ferite sperimentali a individui che sono già noti per sostenere una risposta di guarigione delle ferite compromessa. Di conseguenza, gli studi su soggetti umani con diabete sono più comunemente diretti verso la risposta infiammatoria piuttosto che verso la sperimentazione su tessuto asportato6. Sono disponibili anche i modelli Organ-on-a-chip7 e pelle artificiale8. Questi hanno il vantaggio di essere in grado di analizzare i contributi delle cellule umane, ma danno poche indicazioni sulla variabilità tra i pazienti. Pertanto, modelli clinicamente rilevanti per studiare i progressi della guarigione delle ferite in popolazioni di pazienti vulnerabili potrebbero accelerare la comprensione meccanicistica e la scoperta di farmaci in questo settore.
Il protocollo di ferimento ex vivo descritto di seguito è adattato da Stojadinovic e Tomic-Canic, 20139. È adatto per l'esame dei dati trascrizionali provenienti da ferite controllate di campioni di tessuto umano ex vivo e può essere applicato a campioni clinici di pazienti con scarsa guarigione delle ferite (ad esempio, diabete di tipo 2, individui anziani), al fine di far progredire le conoscenze su come la guarigione delle ferite è influenzata in queste condizioni e potenzialmente su come può essere ripristinata.
Protocollo
Questo protocollo si basa sulla fornitura di tessuto chirurgico umano. L'approvazione etica e il consenso informato del paziente sono stati ottenuti prima della sperimentazione e lo studio è conforme ai principi delineati nella Dichiarazione di Helsinki.
1. Raccolta di tessuti e ferite ex vivo
- Raccogliere il tessuto chirurgico dopo l'amputazione/intervento chirurgico dell'arto in un contenitore sterile contenente il Medium Modified Eagle Medium (DMEM) di Dulbecco con il 5% di penicillina-streptomicina-fungizone, 2 mM di L-glutammina e il 10% di siero fetale bovino (FBS; terreno di crescita completo).
NOTA: Il tessuto raccolto in questo modo può essere conservato a 4 °C per un massimo di 24 ore prima della sperimentazione. Per garantire la vitalità del tessuto per gli esperimenti ex vivo , si raccomanda di processare il tessuto il prima possibile o di mantenere un intervallo di tempo fisso tra la raccolta e la ferita per standardizzare gli esperimenti su più donazioni di tessuti. Se il tessuto deve essere conservato per un certo periodo di tempo, è necessario aggiungere una soluzione di stabilizzazione dell'RNA al campione per mantenere l'integrità dell'RNA. - Utilizzando una pinza sterile, trasferire il tessuto in una piastra di Petri da 60 mm riempita con soluzione salina tamponata con fosfato sterile (PBS) integrata con il 5% di penicillina-streptomicina-fungino. Tagliare il grasso dal derma usando un bisturi sterile e/o forbici chirurgiche e gettarlo. Trasferire il fazzoletto in una capsula di Petri fresca riempita con PBS sterile.
- Fissare due lame sterili insieme in modo che ci sia uno spazio di 1 mm tra le due lame. Tracciare due linee lineari parallele, distanti ~1 mm l'una dall'altra, lungo la lunghezza dell'epidermide e la parte superiore del derma papillare10,11.
- Rimuovere l'epidermide tra le linee di incisione utilizzando una pinza sterile e forbici chirurgiche per creare una ferita lineare.
- Utilizzare un bisturi per tagliare il tessuto in piccoli rettangoli (dimensione massima di 1 cm2) che avvolgano la ferita con l'epidermide intatta su entrambi i lati.
NOTA: Per mantenere l'integrità del tessuto e del successivo RNA, i passaggi 1.2-1.5 devono essere eseguiti il più rapidamente possibile. - Scatta un'immagine del tessuto ferito utilizzando un microscopio per microdissezione con ingrandimento 4x, assicurandoti che il campo visivo catturi l'intera ferita e il tessuto epidermico intatto circostante.
- Utilizzando una pinza sterile, posizionare i rettangoli di tessuto su un inserto di coltura tissutale in una piastra a 6 pozzetti e pipettare delicatamente 2 mL di terreno di crescita completo nel pozzetto. Ciò garantirà che il campione venga coltivato all'interfaccia liquido-aria.
- Incubare a 37 °C in 5% di CO2 in aria per un massimo di 120 ore.
NOTA: Al termine dell'incubazione, il tessuto deve essere ripreso con un microscopio di microdissezione allo stesso ingrandimento del passaggio 1.6. Se necessario, le immagini possono essere scattate anche ogni 24 ore.
2. Fissazione tissutale e criosezione
- Snap congelare il tessuto in azoto liquido. Posizionare una piccola quantità di mezzo da taglio compatibile con criostato su un mandrino per criostato e incorporare il tessuto al suo interno. Orientare il tessuto perpendicolarmente al mandrino in modo che la faccia di taglio tagli tutti gli strati della pelle, compresa l'area ferita. Conservare a -80 °C.
NOTA: Il protocollo può essere messo in pausa qui. - Pulire il criostato a temperatura ambiente con etanolo al 70% e spray decontaminante a base di RNasi. Impostare la temperatura del criostato a -28 °C.
- Verificare che l'orientamento del mandrino e della lama del criostato possa produrre sezioni che racchiudono l'intero spessore del tessuto, compreso il tessuto ferito e il derma sottostante.
- Utilizzando il criostato, tagliare fino a dieci sezioni da 7 μm da ciascuna ferita e posizionarle su vetrini di microdissezione privi di RNA.
- Conservare i vetrini nella scatola originale in cui sono stati forniti i vetrini di microdissezione a -80 °C per garantire un ambiente privo di RNasi.
NOTA: Il protocollo può essere messo in pausa qui, ma tieni presente che la degradazione dell'RNA aumenterà quanto più lungo è il tempo di conservazione.
3. Microdissezione a cattura laser
- Posizionare i vetrini di microdissezione con la sezione di tessuto nel ghiaccio secco e passare alla sala del microscopio di microdissezione a cattura laser.
- Asciugare all'ariail 1° vetrino e procedere rapidamente alla colorazione delle sezioni con ematossilina utilizzando un kit di colorazione con ematossilina ed eosina senza RNasi immediatamente prima di eseguire la microdissezione a cattura laser.
- Visualizza il tessuto ferito utilizzando un microscopio di microdissezione a cattura laser con ingrandimento 10x e scatta una foto dell'area di interesse che verrà catturata al laser.
- Tracciare intorno a quell'area (ad esempio, lingua epiteliale, tessuto di granulazione, microvasi) e raccogliere in microdissezione i cappucci isolanti del diffusore da 0,5 ml utilizzando le istruzioni del software per il particolare microscopio in uso.
- Rivisualizza e visualizza l'area di interesse catturata al laser utilizzando il microscopio di microdissezione a cattura laser con ingrandimento 10x per dimostrare la posizione e l'escissione completa del tessuto sezionato.
NOTA: Eseguire l'acquisizione laser per ogni campione (fino a 10 sezioni) per un tempo massimo di 1 ora per ridurre al minimo la degradazione dell'RNA. - Aggiungere il tampone di conservazione dell'RNA alla provetta contenente il tessuto microsezionato secondo le istruzioni del produttore e conservare in ghiaccio secco fino a quando non può essere trasferito in un congelatore a -80°C.
NOTA: il protocollo può essere temporaneamente messo in pausa qui.
4. Quantificazione dell'espressione genica differenziale
- Centrifugare le provette del campione alla massima velocità per 1 minuto.
- Isolare l'RNA dal tessuto microsezionato seguendo le istruzioni del produttore.
- Eluire l'RNA in un volume finale di 12 μL di acqua priva di RNasi e amplificare l'RNA utilizzando un kit di amplificazione dell'RNA e un termociclatore, secondo le istruzioni del produttore. Si raccomandano due cicli di amplificazione per garantire una quantità sufficiente di RNA per ulteriori analisi. Utilizzare le condizioni di amplificazione nella Tabella 1.
| Primo round di amplificazione | Secondo round di amplificazione | ||||
| Sintesi del primo filamento | Sintesi del primo filamento | ||||
| Passo | Temperatura | Ore | Passo | Temperatura | Ore |
| 1 | 65 °C | 5 minuti | 1 | 65 °C | 5 minuti |
| 2 | 4 °C | tenere | 2 | 4 °C | tenere |
| 3 | 42 °C | 45 minuti | 3 | 25 °C | 10 minuti |
| 4 | 4 °C | tenere | 4 | 37 °C | 45 minuti |
| 5 | 37 °C | 20 minuti | 5 | 4 °C | tenere |
| 6 | 95 °C | 5 minuti | |||
| 7 | 4 °C | tenere | Sintesi del secondo filone | ||
| Passo | Temperatura | Ore | |||
| Sintesi del secondo filone | 1 | 95 °C | 2 minuti | ||
| Passo | Temperatura | Ore | 2 | 4 °C | tenere |
| 1 | 95 °C | 2 minuti | 3 | 37 °C | 15 |
| 2 | 4 °C | tenere | 4 | 70 °C | 5 minuti |
| 3 | 25 °C | 5 minuti | 5 | 4 °C | tenere |
| 4 | 37 °C | 10 minuti | |||
| 5 | 70 °C | 5 minuti | Trascrizione in vitro | ||
| 6 | 4 °C | tenere | Passo | Temperatura | Ore |
| 1 | 42 °C | 6 h | |||
| Trascrizione in vitro | 2 | 4 °C | tenere | ||
| Passo | Temperatura | Ore | 3 | 37 °C | 15 minuti |
| 1 | 42 °C | 3 h | 4 | 4 °C | tenere |
| 2 | 4 °C | tenere | |||
| 3 | 37 °C | 15 minuti | |||
| 4 | 4 °C | tenere | |||
Tabella 1: Condizioni di amplificazione.
- Quantificare la concentrazione e la purezza dell'RNA amplificato. I rapporti di assorbanza A260/230 (purezza) e A260/280 (contaminanti) > 1,8 sono adatti per ulteriori analisi.
NOTA: L'RNA amplificato purificato può essere utilizzato per studi mirati sull'espressione genica (passaggi 4.5-4.6) o può essere inviato per l'analisi di microarray. - Sintetizzare il cDNA utilizzando il kit di trascrizione inversa del cDNA ad alta capacità secondo le istruzioni del produttore. Le condizioni rappresentative sono le seguenti:
- 25 °C per 10 min
- 37 °C per 2 h
- 85 °C per 5 min
- Mantenimento a 4 °C
- Eseguire la PCR quantitativa utilizzando 0,5 μL di cDNA e 1 μM di primer (del gene di interesse) secondo le istruzioni del produttore. Le condizioni rappresentative e le sequenze di primer sono le seguenti.
- Utilizzare le seguenti condizioni per le condizioni di PCR quantitativa: 95 °C per 10 minuti; 40 cicli a 95°C per 15 s e 62°C per 1 min; 95 °C per 5 min; e mantenimento a 4°C.
- Utilizzare le seguenti sequenze di base:
GAPDH
Avanti 5'-TATAAATTGAGCCCGCAGCC-3'
Inverso 5'-CGACCAAATCCGTTGACTCC-3'
KRT17
Avanti 5'-AGGGAGAGGATGCCCACCTG-3'
Inverso 5'-GCGGGAGGAGATGACCTTGC-3'
5. Interpretazione dei dati
- Calcolare il tasso di guarigione delle ferite utilizzando le immagini del tessuto ex vivo generate nei passaggi 1.6-1.8 utilizzando ImageJ. Ci sono diverse pubblicazioni recenti che evidenziano diverse variazioni su come analizzare automaticamente le aree della ferita in ImageJ 12,13,14.
- Quantificare l'espressione genica utilizzando il metodo ΔCT , confrontando i cicli soglia per il gene di interesse rispetto a quello della governante (ad esempio, gliceraldeide-3-fosfato; GAPDH). Per fare ciò, annota il valore CT per la governante e per il gene di interesse.
- Calcola la differenza tra i due valori di CT per normalizzare la quantità di mRNA presente e controllare le differenze nelle concentrazioni di estrazione dell'RNA tra i diversi isolamenti:
ΔCT = CT (gene di interesse) - CT (governante) - Calcolare l'entità della differenza tra i valori CT per ottenere la quantificazione relativa del gene di interesse, espressa in percentuale della governante:
Quantificazione relativa = (2-ΔCT) x 100 - Esaminare le differenze tra i diversi donatori di pazienti/stati patologici confrontando le quantificazioni relative dei geni di interesse per ciascun campione.
NOTA: Uno schema dell'intera tecnica può essere trovato nella Figura 1.
- Calcola la differenza tra i due valori di CT per normalizzare la quantità di mRNA presente e controllare le differenze nelle concentrazioni di estrazione dell'RNA tra i diversi isolamenti:
Risultati
Seguendo il protocollo, è stato scelto un punto temporale di 48 ore per generare risultati rappresentativi. La creazione della ferita iniziale nel tessuto in eccesso dalla chirurgia estetica elettiva può essere vista nella Figura 2A dove la ferita asportata è chiaramente visibile. La colorazione con ematossilina ed eosina conferma che questo ha generato una ferita a tutto spessore (Figura 2B). Dopo 48 ore, la chiusura parziale della ferita è visibile al microscopio ottico (Figura 2C). La colorazione istologica rivela la lingua epiteliale che sta progredendo per guarire la ferita (Figura 2D), dimostrando che il modello di guarigione delle ferite ex vivo è un valido proxy per la guarigione delle ferite in vivo .
Dopo il sezionamento e la colorazione con ematossilina ed eosina, la ferita in via di guarigione è stata visualizzata sul sistema di microdissezione a cattura laser e l'area della ferita è stata selezionata (Figura 3A). Quest'area è stata completamente asportata con questo metodo, come si può vedere nella Figura 3B. La qualità e la purezza dell'RNA erano ragionevoli (analizzate in base al numero di integrità dell'RNA (RIN); Figura 3C) - L'RNA amplificato di scarsa qualità avrebbe molti picchi e cali che indicano più prodotti di degradazione. L'isolamento di piccole sezioni di tessuto può produrre RNA di una concentrazione molto bassa che rende difficile un'interpretazione valida della qPCR. La Figura 3D mostra la variazione della concentrazione di RNA che ci si aspetta utilizzando questa tecnica, con un intervallo da 2,00 a 6,15 ng/mL. È importante sottolineare che anche i campioni diluiti sono stati in grado di fornire robusti valori di CT sia per la governante (GAPDH) che per i geni specifici della pelle di interesse (cheratina 17; KRT17; Figura 3E), a conferma dell'idoneità della tecnica per studi di trascrittomica comparativa.
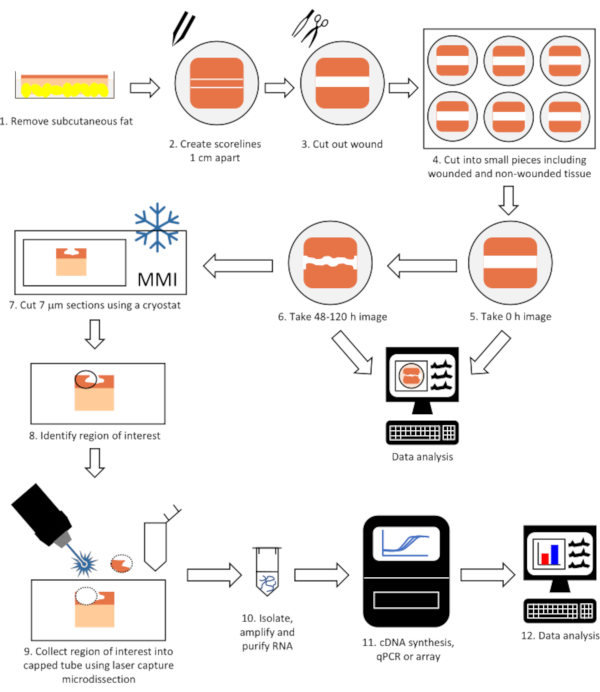
Figura 1: Diagramma di flusso della tecnica completa per eseguire l'analisi dell'espressione genica su tessuto microsezionato con laser da pelle ferita. Il tessuto viene ferito, lasciato guarire in un incubatore di tessuto e sottoposto a imaging (fasi 1-6) prima di essere tagliato in sezioni di 7 μm utilizzando un criostato (fase 7). La regione di interesse (ad es. lingua epiteliale) viene identificata e raccolta mediante microdissezione a cattura laser (fasi 8-9) e isolata con RNA, purificata e determinata l'espressione genica (fasi 10-12). Clicca qui per visualizzare una versione più grande di questa figura.
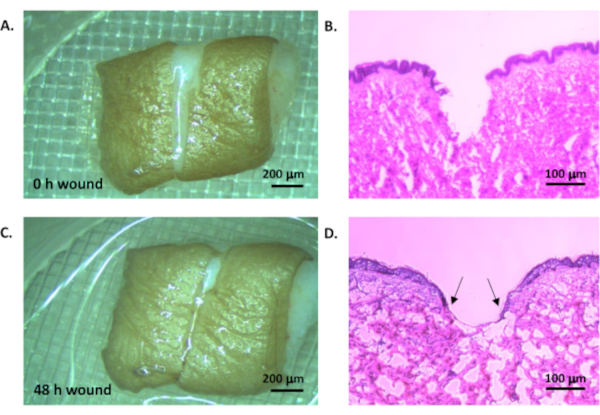
Figura 2: Modello di ferita ex vivo. Il tessuto umano è stato ferito creando due tagli paralleli e rimuovendo il tessuto in mezzo per lasciare un'area ferita uniforme (A, microscopio ottico, barra graduata = 200 μm; B, colorazione con emoatossilina ed eosina, barra graduata = 100 μm). Il tessuto è stato coltivato in un incubatore standard per colture tissutali a 37°C in CO2 al 5% in aria per 48 ore (C, microscopio ottico, barra graduata = 200 μm; D, colorazione con ematossilina ed eosina, barra graduata = 100 μm). Le punte delle frecce indicano la lingua epiteliale. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Microdissezione a cattura laser ed espressione genica. A. La regione di interesse (in questo caso, tessuto guarito) è stata identificata utilizzando la colorazione con ematossilina e raccolta utilizzando la microdissezione a cattura laser. B. La stessa regione ripresa dopo la microdissezione. Barre di scala = 50 μm. C. Elettroferogramma rappresentativo dell'RNA che è stato isolato, amplificato e quantificato dal tessuto microsezionato al laser. D. Concentrazione di RNA dal tessuto raccolto (n=24 campioni). E. Rilevamento riproducibile dell'espressione di GAPDH e KRT17 mediante qPCR (n=13 campioni, media ± SEM). Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Con l'aumento dell'incidenza di malattie croniche come il diabete di tipo 2 a livello globale, la necessità di tecniche che possano facilitare studi fisiopatologicamente rilevanti diventa più urgente. Il protocollo sopra descritto fornisce un metodo standardizzato per l'esame dei dati trascrittomici da ferite in guarigione ex vivo utilizzando tessuto umano.
Questo protocollo dipende dalla fornitura di tessuto clinico in eccedenza per il quale è stata concessa l'autorizzazione etica dall'autorità competente e dai pazienti che hanno dato il consenso informato. Comunemente questo sarà da pazienti sottoposti ad amputazione o interventi di chirurgia estetica elettiva. I dati demografici clinici dei donatori devono essere attentamente considerati, tuttavia uno dei punti di forza di questo metodo è l'opportunità di eseguire studi su tessuti di più donatori pazienti da individui sani di controllo (ad esempio, coloro che stanno subendo amputazione a seguito di un incidente o che si stanno sottoponendo a procedure cosmetiche elettive) e quelli con malattie croniche come il diabete di tipo 2. Inoltre, poiché il metodo può essere messo in pausa in numerosi punti (vedi protocollo), le ferite ex vivo possono essere preparate man mano che si svolgono gli interventi chirurgici e quindi conservate per l'analisi dell'RNA fianco a fianco una volta che sono stati assicurati campioni di tessuto sufficienti.
La fase più critica di questo protocollo è la microdissezione a cattura laser (sezione 3 del protocollo). Sebbene esistano molte pubblicazioni che utilizzano questa tecnica per l'analisi trascrittomica, il presente rapporto è il primo a fornire un protocollo sperimentale completo per applicarlo agli studi su tessuto umano di pazienti con una risposta di guarigione delle ferite compromessa. È essenziale che l'acquisizione laser venga eseguita il più rapidamente possibile e per un'ora al massimo assoluto. Questo serve a ridurre al minimo la degradazione dell'RNA quando le sezioni congelate tornano a temperatura ambiente e possono essere monitorate durante il controllo di qualità dell'RNA amplificato nella sezione 4 del protocollo.
Sebbene questa tecnica aggiunga valore nel consentire di considerare la variabilità interpaziente nei meccanismi di guarigione delle ferite umane, presenta alcune limitazioni. L'impatto delle cellule infiammatorie non può essere valutato in quanto il modello ex vivo non ha una circolazione funzionante. Se questo è il fulcro di uno studio, allora i modelli in vivo (sia animali 2,3 che umani6) sarebbero di maggiore beneficio. Inoltre, la fornitura di tessuto clinicamente rilevante può essere un fattore limitante. In questi casi, i modelli Artificial Skin8 o Skin-on-a-Chip7 potrebbero essere più accessibili. Indipendentemente da ciò, uno dei principali punti di forza del protocollo è che può studiare eventi a livello trascrizionale da tessuto umano ferito, da più donatori e da diversi gruppi di pazienti. Normalmente, studi di questo tipo sarebbero condotti utilizzando modelli in vitro che, pur fornendo informazioni preziose, non replicano le complessità del tessuto ferito15. Nel complesso, questa tecnica è un'utile aggiunta alla suite attualmente esistente di modelli in vitro, in vivo ed ex vivo per la guarigione delle ferite nel diabete.
In sintesi, questo flusso di lavoro può essere utilizzato per studiare i meccanismi di guarigione delle ferite non solo nel diabete di tipo 2, ma anche in altri gruppi clinicamente vulnerabili, ad esempio gli anziani o quelli con disturbi del tessuto connettivo. Il continuo sviluppo di saggi proteomici e metabolomici con una maggiore sensibilità 16,17,18 amplierà ulteriormente le applicazioni di questa tecnica.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
ICP è stato sostenuto dal 7° Programma Quadro per la Ricerca e lo Sviluppo Tecnico della Commissione Europea - Marie Curie Innovative Training Networks (ITN), Grant agreement n.: 607886. RW è stata supportata da Aveda, Hair Innovation & Technology, USA. RB, SS sono stati supportati dal Centre of Skin Sciences dell'Università di Bradford.
Materiali
| Name | Company | Catalog Number | Comments |
| Arcturus RiboAmp PLUS kit | ThermoFisher Scientific | KIT0521 | RNA amplification kit |
| Diffuser Caps 0.5mL | MMI | K10028161 | Laser capture microdissection caps; 50 pack |
| Dulbecco’s Modified Eagle Medium (DMEM) | Sigma-Aldrich | D6046 | With 1000 mg/L glucose, L-glutamine, and sodium bicarbonate, liquid, sterile-filtered, suitable for cell culture |
| Foetal Bovine Serum | Thermo Fisher Scientific | 10270106 | Cell culture supplement |
| H&E Staining Kit Plus | MMI | K10028305 | Rnase-free haematoxylin and eosin staining kit |
| High capacity cDNA reverse transcription kit | Applied Biosystems | 4368814 | Reverse transcription kit |
| L-glutamine | Thermo Fisher Scientific | 25030149 | Cell culture supplement |
| MembraneSlides | MMI | K10028153 | Laer capture microdissection slides; 5 per box |
| Netwell Mesh Insert | Corning | 3479 | Cell culture insert |
| Penicillin-Streptomycin-Fungizone | Thermo Fisher Scientific | 15070-063 | Cell culture supplement |
| 15290-026 | |||
| OCT | Tissue-Tek Sakura | 4583 | Cryostat-compatible cutting medium |
| PBS | Thermo Fisher Scientific | 10209252 | Five tablets per 100ml sterile water and then autoclaved for cell culture use |
| RNeasy Micro Kit | Qiagen | 74004 | RNA extraction kit |
| RNase Away | Sigma-Aldrich | 83931 | RNase spray |
| Sterile blades | Scientific Laboratory Supplies | INS4974 | Tissue dissection implements |
| Support Slide | MMI | K10028159 | Laser capture microdissection support slide, RNase-free |
| Surgical scissors | Scientific Laboratory Supplies | INS4860 | Tissue dissection implements |
| Surgical forceps | Scientific Laboratory Supplies | INS2026 | Tissue dissection implements |
| SYBR Green Supermix | Applied Biosystems | 4344463 | Quantitative PCR mastermix |
Riferimenti
- Gianino, E., Miller, C., Gilmore, J. Smart Wound Dressings for Diabetic Chronic Wounds. Bioengineering. 5 (3), (2018).
- Song, M., et al. Cryptotanshinone enhances wound healing in type 2 diabetes with modulatory effects on inflammation, angiogenesis and extracellular matrix remodelling. Pharmaceutical Biology. 58 (1), 845-853 (2020).
- Liu, J., et al. Involvement of miRNA203 in the proliferation of epidermal stem cells during the process of DM chronic wound healing through Wnt signal pathways. Stem Cell Research and Therapy. 11 (1), 348 (2020).
- Pastar, I., Wong, L. L., Egger, A. N., Tomic-Canic, M. Descriptive vs mechanistic scientific approach to study wound healing and its inhibition: Is there a value of translational research involving human subjects. Experimental Dermatology. 27 (5), 551-562 (2018).
- Zomer, H. D., Trentin, A. G. Skin wound healing in humans and mice: Challenges in translational research. Journal of Dermatological Science. 90 (1), 3-12 (2018).
- Mirza, R. E., Fang, M. M., Weinheimer-Haus, E. M., Ennis, W. J., Koh, T. J. Sustained inflammasome activity in macrophages impairs wound healing in type 2 diabetic humans and mice. Diabetes. 63 (3), 1103-1114 (2014).
- Ataç, B., et al. Skin and hair on-a-chip: in vitro skin models versus ex vivo tissue maintenance with dynamic perfusion. Lab Chip. 13 (18), 3555-3561 (2013).
- Yun, Y., Jung, Y. J., Choi, Y. J., Choi, J. S., Cho, Y. W. Artificial skin models for animal-free testing. Journal of Pharmaceutical Investigation. 48, 215-223 (2018).
- Stojadinovic, O., Tomic-Canic, M. Human ex vivo wound healing model. Methods in Molecular Biology. 1037, 255-264 (2013).
- Castellano-Pellicena, I., et al. Does blue light restore human epidermal barrier function via activation of Opsin during cutaneous wound healing. Lasers in Surgery and Medicine. 51 (4), 370-382 (2019).
- Rizzo, A. E., Beckett, L. A., Baier, B. S., Isseroff, R. R. The linear excisional wound: an improved model for human ex vivo wound epithelialization studies. Skin Research and Technology. 18 (1), 125-132 (2012).
- Castellano-Pellicena, I., Thornton, M. J. Isolation of epidermal keratinocytes from human skin: The scratch-wound assay for assessment of epidermal keratinocyte migration. Methods in Molecular Biology. 2154, 1-12 (2020).
- Suarez-Arnedo, A., et al. An imaje J plugin for the high throughput image analysis of in vitro scratch wound healing assays. PLoS ONE. 15 (7), 0232565 (2020).
- Venter, C., Niesler, C. U. Rapid quantification of cellular proliferation and migration using ImageJ. Biotechniques. 66 (2), (2019).
- Al-Rikabi, A. H. A., Riches-Suman, K., Tobin, D. J., Thornton, M. J. A proinflammatory environment induces changes in the diabetic phenotype of human dermal fibroblasts derived from diabetic and nondiabetic donors: Implications for wound healing. Scientific Reports. , (2020).
- Chen, A., et al. Towards single-cell ionomics: a novel micro-scaled method for multi-element analysis of nanogram-sized biological samples. Plant Methods. 16, 31 (2020).
- Lutz, B. M., Peng, J. Deep profiling of the aggregated proteome in Alzheimer's Disease: from pathology to disease mechanisms. Proteomes. 6 (4), 46 (2018).
- Rinschen, M. M. Single glomerular proteomics: A novel tool for translational glomerular cell biology. Methods in Cell Biology. 154, 1-14 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon
Utilizziamo i cookies per migliorare la tua esperienza sul nostro sito web.
Continuando a utilizzare il nostro sito web o cliccando “Continua”, accetti l'utilizzo dei cookies.