Method Article
Discriminazione e mappatura delle trascrizioni primarie ed elaborate nel mitocondrio di mais utilizzando una strategia circolare basata su RT-PCR
In questo articolo
Riepilogo
Vi presentiamo una strategia circolare basata su RT-PCR combinando RT-PCR circolare, RT-PCR quantitativo, trattamento polifosculo DI RNA 5 e macchia settentrionale. Questo protocollo include una fase di normalizzazione per ridurre al minimo l'influenza del tripofatore instabile 5', ed è adatto per discriminare e mappare le trascrizioni primarie ed elaborate accumulate stabilmente nel mitocondrio di mais.
Abstract
Nei mitocondri delle piante, alcune trascrizioni in stato costante hanno 5' triposfato derivato dall'initiazione della trascrizione (trascrizioni primaria), mentre le altre contengono 5' monofofafato generato post-transcriptionally (trascrizioni elaborate). Per discriminare tra i due tipi di trascrizioni, sono state sviluppate diverse strategie e la maggior parte di esse dipende dalla presenza/assenza di 7' triposfato. Tuttavia, il triplofato al termine primario 5' è instabile, e ostacola una chiara discriminazione dei due tipi di trascrizioni. Per differenziare e mappare sistematicamente le trascrizioni primarie ed elaborate accumulate stabilmente nel mitocondrio di mais, abbiamo sviluppato una strategia circolare basata su RT-PCR (cRT-PCR) combinando cRT-PCR, trattamento quantitativo dei polifoshpatasi di RNA 5', RT-PCR (RT-qPCR) e Northern blot. Come miglioramento, questa strategia include un passo di normalizzazione dell'RNA per ridurre al minimo l'influenza del tripfosfato unstable 5'.
In questo protocollo, l'RNA mitocondriale arricchito è pre-trattato da RNA 5' polifosforasi, che converte il triphsofato da 5' in monofagofato. Dopo la circolarizzazione e la trascrizione inversa, i due cDNA derivati da 5' RNA trattati con polifofofosi e non trattati sono normalizzati dal mais 26S rRNA maturo, che ha una fine elaborata 5' ed è insensibile a 5' polifosfolasi. Dopo la normalizzazione, le trascrizioni primarie e trasformate vengono discriminate confrontando i prodotti cRT-PCR e RT-qPCR ottenuti dagli RNA trattati e non trattati. La prova di trascrizione è determinata dalla clonazione e dal sequenziamento dei prodotti cRT-PCR e quindi verificata da Northern blot.
Utilizzando questa strategia, sono state determinate la maggior parte delle trascrizioni a stato costante nel mitocondrio di mais. A causa del complicato schema di trascrizione di alcuni geni mitocondriali, alcune trascrizioni a stato costante non sono state differenziate e/o mappate, anche se sono state rilevate in una macchia settentrionale. Non siamo sicuri se questa strategia sia adatta a discriminare e mappare le trascrizioni dello stato costante in altri mitocondri vegetali o in plastidi.
Introduzione
Nei mitocondri vegetali, molti RNA maturi e precursori vengono accumulati come isoformi multipli, e le trascrizioni allo stato costante possono essere divise in due gruppi in base alla differenza alle loro 5 ' estremità1,2,3 4. Le trascrizioni primarie hanno 5' estremità triposphate, che derivano dall'avvio della trascrizione. Al contrario, le trascrizioni elaborate hanno 5' monofafato generato dall'elaborazione post-trascrizione. La discriminazione e la mappatura dei due tipi di trascrizioni sono importanti per svelare i meccanismi molecolari alla base della trascrizione e della maturazione finale della trascrizione.
Per distinguere tra le trascrizioni primarie ed elaborate nel mitocondrio vegetale, sono state sviluppate quattro strategie principali. La prima strategia consiste nel pretrattare gli RNA mitocondriali con il pirofosofasi acido del tabacco (TAP), che converte il tripfosfato 5' in monofagofato e consente alle trascrizioni primarie di essere circolati dalla ligase dell'RNA. L'abbondanza di trascrizioni di campioni di RNA trattati con TAP e non trattati viene quindi confrontata con una rapida amplificazione delle estremità cDNA (RACE) o RT-PCR circolari (cRT-PCR)2,3,4. Nella seconda strategia, le trascrizioni elaborate vengono prima esaurite dagli RNA mitocondriali utilizzando l'eonuclease dipendente dal terminator5-fosfatio (TEX), e le trascrizioni primarie rimaste vengono quindi mappate dall'analisi dell'estensione del primer5,6 . La terza strategia è quella di pre-cap le trascrizioni primarie utilizzando guanylyl transferase, e quindi la posizione di triposphated 5' è determinata dall'estensione primer insieme con ribonuclease o S1 nuclease protection analysis7,8 ,9. A differenza di quelli che dipendono dalla presenza/assenza di 7' triposfato, la quarta strategia combina la trascrizione in vitro, la mutagenesi diretta dal sito e l'analisi dell'estensione del primer per caratterizzare i promotori putativi e determinare la trascrizione siti di avvio8,10,11. Utilizzando queste strategie, molte trascrizioni primarie ed elaborate sono state determinate nei mitocondri delle piante.
Tuttavia, diversi studi hanno riferito che il trifosfato 5' delle trascrizioni primarie erano instabili, e sono stati facilmente convertiti in monofosfato per motivo sconosciuto2,4,12,13. Questo problema ostacola una chiara discriminazione dei due tipi di trascrizioni utilizzando tecniche a seconda della presenza/assenza di trifosfato 5' e precedenti sforzi per distinguere sistematicamente tra le trascrizioni primarie ed elaborate nell'impianto mitocondri fallito2,12.
In questo protocollo, combiniamo cRT-PCR, trattamento polifosforo RNA 5', RT-qPCR e macchia settentrionale per distinguere sistematicamente le trascrizioni primarie ed elaborate accumulate stabilmente nel mais (zea mays) mitocondrion (Figura 1). cRT-PCR consente la mappatura simultanea delle estremità 5' e 3' di una molecola di RNA, ed è di solito adattata alla trascrizione dei termini nelle piante2,12,14,15. Il polifosforasi di RNA 5' potrebbe rimuovere due fosfati dal termine di 1'trhafated 5', il che rende disponibili le trascrizioni primarie per l'auto-legatura da parte della ligase dell'RNA. Studi precedenti hanno dimostrato che il rRNA 26S maturo nel mais aveva elaborato il capolinea 5', ed era insensibile al polifosculo DI RNA 5 '1,16. Per ridurre al minimo l'influenza del triposfato instabile ai termini primari 5', i 5' RNA trattati con polifofofofosi e non trattati vengono normalizzati da rRNA 26S maturi, e le trascrizioni primarie ed elaborate vengono quindi differenziate confrontando cRT-PCR ottenuti dai due campioni di RNA. I risultati della mappatura cRT-PCR e della discriminazione sono verificati rispettivamente da Northern blot e RT-qPCR. Infine, i primer alternativi vengono utilizzati per amplificare le trascrizioni rilevate nella macchia settentrionale, ma non da cRT-PCR. Utilizzando questa strategia basata su cRT-PCR, la maggior parte delle trascrizioni a stato costante nel mitocondrio di mais sono state differenziate e mappate1.
Protocollo
1. Progettazione di Primer
- Progettare primer gene-specific per la trascrizione inversa (RT) utilizzando il software di progettazione del primer PCR (Table of Materials) in base alle regole generali di primer design17.
NOT: I primer RT sono altamente specifici per le trascrizioni di destinazione, e sono generalmente ancorati sulla parte 5' delle sequenze di codifica (mRNA maturi e RNA precursori), o 500-600 nt a valle della fine prevista 5' (18S e 26S rRNA). - Progettare coppie di primer divergenti per amplificare le trascrizioni circolari da cRT-PCR.
NOTA: i primer divergenti accoppiati fiancheggiano la giunzione 5'3' delle trascrizioni circolari e le loro posizioni variano tra le trascrizioni di destinazione analizzate (Figura S1). Alcune trascrizioni hanno UDR lunghe; ad esempio, nad2-1 5' UTR e rps4-1 3' UTR sono 1.985 e 1.826/1.834 nt, rispettivamente1. Se entrambi i primer sono ancorati all'area di codifica, sarà difficile amplificare le trascrizioni di destinazione. Al contrario, alcuni UTR sono brevi; ad esempio, i 3' UTR di nad2-1 e nad4-1 sono rispettivamente 34/35 e 29–31 nt, rispettivamente1. Se i primer accoppiati si trovano lontano dalle sequenze di codifica, la PCR avrà esito negativo. Generalmente, due coppie di primer divergenti sono progettati per ogni trascrizione di destinazione, mentre più coppie possono essere necessarie per mappare quelli i cui modelli di trascrizione sono complicati e/o gli UTR sono molto lunghi. - Progettare coppie di primer convergenti per preparare sonde di RNA per la macchia settentrionale.
NOT: I primer accoppiati si trovano sulla regione di codifica del gene bersaglio e la dimensione del prodotto PCR dovrebbe essere compresa tra 100 e 1.000 bp. Ogni primer in avanti deve contenere un sito enzimatico di restrizione per ridurre al minimo le sequenze vettoriali nelle sonde risultanti.
2. Preparazione del mitocondrio grezzo dai kernel in via di sviluppo del mais
- Sterilizza pestelli, mortai, imbuti di vetro, tubi e punte per autoclave e asciugali in forno.
- Eseguire tutte le procedure a 4 gradi centigradi o sul ghiaccio e pre-raffreddare tutte le soluzioni.
- Preparare 100 mL di buffer di estrazione (EB), composto da 0,3 M di saccarosio, 5 mM di tetrasodium pirofosfato, 10 mM KH2PO4, 2 mM EDTA, 1% [w/v] polyvinylpyrrolidone 40, 1% [w/v] bovine serum albumin, 5 mM L-cysteine e 20 mcorm asbica acida. Regolare a pH 7.3 con KOH e sterilizzare per filtrazione.
- Preparare 100 mL di buffer di lavaggio (WB) composto da 0,3 M di saccarosio, 1 mM EGTA e 10 mM MOPS (3-(N-morpholino)acido proposulfonico). Regolare a pH 7.2 con NaOH e sterilizzare per filtrazione.
NOT: Si consiglia di preparare EB e WB utilizzando DePC-trattato deionizzato H2O. - Raccogliere 20 g sviluppando kernel a 11-20 giorni dopo l'impollinazione (DAP) a un tubo da 50 mL posto sul ghiaccio, quindi trasferire i chicchi a mortai pre-raffreddati.
NOT: Utilizzare un rapporto di 100 mL di EB a 20 g di kernel di mais. - Aggiungere 10-20 mL di EB ghiacciato ad ogni mortaio e macinare completamente i chicchi.
- Aggiungere più EB, e filtrare i tessuti del terreno attraverso due strati di panno di filtro (Tabella dei materiali).
- Centrifugare il filtrato a 8.000 x g per 10 min e scartare il pellet.
- Trasferire il supernatante in un nuovo tubo e centrifugarlo a 20.000 x g per 10 min.
- Versare il supernatante, e risospendere il pellet in 6 mL WB.
- La sospensione è di cinque tubi da 1,5 mL senza RNase e centrifugarli a 14.000 x g per 5 min.
- Scartare il supernatante, congelare il pellet mitocondriale in azoto liquido e conservarlo a -80 gradi centigradi.
3. Estrazione di RNA mitocondriale
- Estrarre l'RNA mitocondriale con un reagente commerciale (Tabella dei materiali) secondo le istruzioni del produttore.
ATTENZIONE: Questo reagente contiene fenolo e guanidina isothiocyanate. Lavora con esso in un cappuccio a fume e indossa camice da laboratorio e guanti. - Sciogliere l'RNA mitocondriale isolato in DePC-trattato deionizzato H2O, e stimare la concentrazione di RNA e la purezza con uno spettrofotometro (Tabella dei materiali).
NOT: In genere, l'RNA mitocondriale di 250 g è ottenuto da 20 g di 15-DAP kernel di mais. - Preparare un gel di agarose composto da 1x tampone TAE (40 mM Tris, acido acetico da 20 mM, 1mM EDTA), 1,5% agarose (Tabella deimateriali) e 1x colorante di colorazione nucleare (Tabella dei materiali).
- Aggiungere un volume appropriato di buffer di carico 10x (0,5% blu bromofenolo, 0,5% xilene cyanol FF e 50% glicerolo), e caricare la miscela di buffer RNA mitocondriale/buffer di carico sul gel di agarose 1,5%.
- Eseguire il gel in 1x tampone TAE a 5–6 V/cm per 20-25 min e valutare l'integrità dell'RNA mitocondriale immaginando il gel con un sistema di documentazione del gel (Tabella dei materiali).
NOT: La presenza di due bande distinte (rispettivamente 3.510 e 1.970 NT di mais per 26S e 18S rRNA di mais) è uno standard accettabile per l'RNA mitocondriale intatto. Per escludere una possibile degradazione, l'integrità dell'RNA deve essere studiata durante le molteplici fasi della preparazione dell'RNA circolare (Figura2).
4. RNA 5' Trattamento polifosculo
- Impostare il trattamento RNA 5' polyphosphatase (Tabella e materiali) ( Tabella1) e incubare a 37 gradi centigradi per 30-60 min.
- Recuperare l'RNA trattato con polifosfosi da 5' con un kit di purificazione dell'RNA (Tabella dei materiali) secondo le istruzioni del produttore.
- Ripetere i passaggi da 3.3 a 3.5.
5. Circolarizzazione dell'RNA
- Preparare due reazioni circolarizzazioni utilizzando le stesse quantità di RNA mitocondriali trattati con polifofofofosi e non trattati da 5' e non trattati (Tabella2), e incubare entrambe le reazioni a 16 gradi centigradi per 12–16 h.
- Recuperare i due set di RNA auto-ligate utilizzando lo stesso kit del passaggio 4.2.
NOT: Va notato che solo una frazione di RNA mitocondriale sarà auto-migate, e l'RNA recuperato sarà una miscela di trascrizioni lineari e circolari. - Ripetere i passaggi da 3.3 a 3.5.
6. Trascrizione inversa
- Sintetizzare due serie di cDNA dalle stesse quantità di RNA trattati con polifosfasi da 5' e non trattati (200 ng).
- Preparare una miscela di primer aggiungendo un rapporto uguale di 26S-CRT e fino a 7 altri primer RT.
NOT: La concentrazione finale della miscela di primer dovrebbe essere di 1 M. - Preparare due pre-miscele combinando i reagenti elencati nella tabella 3, incubare a 65 gradi centigradi per 5 min, quindi raffreddare sul ghiaccio per 2 min.
- Assemblare due sistemidi reazione RT (tabella 4) e incubarli a 42 gradi centigradi per 50 min.
- Riscaldare entrambe le reazioni di RT a 70 gradi centigradi per 5 minuti, quindi raffreddarle sul ghiaccio.
7. Normalizzazione
- Preparare due reazioni PCR aggiungendo lo stesso volume di modello cDNA derivati da 5' RNA trattati con polifofofozosi o non trattati, primer divergenti che fiancheggiano la giunzione 5'-3' di rRNA circolare 26S (cioè 26S-CF1 e -CR1), ecc. (Tabella5).
- Eseguire le due reazioni in un ciclore termico (Tabella dei materiali) a condizioni descritte nella tabella 6.
- Preparare un gel di agarose composto da 1x tampone TAE, 1,0% agarose e 1x colorante di colorazione nucleare.
- Aggiungere 2 buffer di carico l-l 10x (0,5% di bromofenolo blu, 0,5% xilene cyanol FF e 50% glicerolo) a ciascuno dei due prodotti PCR e caricarli sul gel.
- Eseguire il gel in 1x tampone TAE a 5–6 V/cm per 30-40 min, e immagine utilizzando lo stesso sistema di documentazione gel come passo 3.5.
- Confrontare l'abbondanza dei due prodotti PCR utilizzando software (Tabella dei materiali, Figura 4A) e ottimizzare la normalizzazione modificando, se necessario, la quantità di cDNA del modello.
8. Amplificazione PCR
- Preparare coppie di reazioni PCR aggiungendo il volume appropriato dei cDNA normalizzati e una coppia di primer divergenti che fiancheggiano la giunzione 5'-3' delle trascrizioni bersaglio (Tabella 7).
NOT: La quantità di cDNA modello utilizzata per l'amplificazione PCR delle trascrizioni bersaglio è determinata dai risultati della normalizzazione di rRNA 26S. - Eseguire le reazioni PCR secondo il programma descritto nella tabella 8.
- Ripetere i passaggi da 7.3 a 7.5.
- Passare a primer divergenti nidificati e verificare i risultati PCR del primo round ripetendo i passaggi 8.1 e 8.2.
- Ripetere i passaggi da 7.3 a 7.5.
- Recuperare le bande prominenti che potrebbero essere ripetute in due cicli di amplificazione PCR utilizzando un kit di recupero del DNA gel (Tabella dei materiali).
9. Determinazione del Trascrizione Termini
- Clonare i prodotti PCR gordati in gel in un vettore smussato (Table of Materials) utilizzando tecniche standard.
- Eseguite la colonia PCR per selezionare cloni positivi contenenti gli inserti bersaglio e sequenziarli commercialmente.
NOT: I cloni positivi contenenti inserti di dimensioni variabili sono solitamente rilevati da una singola banda recuperata perché molte trascrizioni a stato costante in ambiente mitocondriale vegetale hanno dimensioni eterogenee 5' e/o 3' estremità1,12. - Allineare i dati di sequenziamento con il genoma mitocondriale del mais utilizzando lo strumento di ricerca di allineamento locale di base (BLAST) del centro nazionale per l'informazione biotecnologica (NCBI). Scegliere organismo "mais (taxid:4577)" e database di ricerca "Raccolta Nucleotide (nr/nt)".
- Trova la giunzione 5'3' della trascrizione circolare e determina le posizioni dei termini di trascrizione 5' e 3'.
- Calcolare la dimensione delle trascrizioni di destinazione.
10. Verifica dei risultati della mappatura cRT-PCR mediante l'ibridazione di RNA Gel Blot
NOT: L'ibridazione della macchia di gel di RNA viene eseguita utilizzando un kit commerciale (Tabella dei materiali), che contiene i reagenti per la trascrizione dell'etichettatura dell'RNA con digossigenina (DIG) e Polimerasi rna T7, ibridazione e rilevamento immunologico. Si prega di fare riferimento ai protocolli forniti in questo kit per maggiori dettagli. Assicurarsi che solo RNase attrezzature gratuite viene utilizzato per l'intera procedura.
- Amplificare il frammento di DNA utilizzato per preparare la sonda dell'RNA e clonarlo sullo stesso vettore del punto 9.1, che contiene un promotore T7 di 17 bp a monte del sito di inserimento.
- Linearizzare il costrutto utilizzando un enzima di restrizione adeguato, recuperare il plasmide linearizzato con un kit di purificazione del DNA (Tabella dei materiali) e dissolverlo in DePC-trattato deionizzato H2O.
- Etichettare le sonde RNA con DIG-11-UTP da un kit commerciale (Tabella dei materiali) secondo le istruzioni del produttore.
- Preparare 500 mL di buffer MOPS 10x (0,2 M MOPS, acetato di sodio 50 mM e 10 mM EDTA) utilizzando H2O. Regolare il pH 7.0 da NaOH e sterilizzare per filtrazione.
- Aggiungere 2-3 volumi di buffer di carico (50% formamide, 6,2% formaldeide, 1x MOPS, 10% glicerolo e 0,1% di blu bromofenolo) all'RNA mitocondriale preparato nei passaggi 3.1–3.3.
- Denaturare il campione di RNA/miscela di tampone di carico a 65 gradi centigradi per 10 min, quindi raffreddare sul ghiaccio per 1 min.
- Preparare un gel di agarose denaturato (2% formaldeide, 1,2% agarose e 1x MOPS) e caricare il campione di RNA/caricamento della miscela di buffer nel gel (1-2 g di RNA mitocondriale per pozzo).
- Eseguire il gel in 1x MOPS a 3-4 V/cm per 4 h.
- Preparare 2 L di 20x SSC (3 M NaCl, 0,3 M di citrato di sodio, pH 7.0). Trattarlo con 0,1% DEPC durante la notte e sterilizzare con autoclave.
- Sciacquare il gel due volte in 20x SSC (15 min ogni volta), e trasferire l'RNA gel a una membrana di nylon (Tabella dei materiali) con bonifico capillare con 20x SSC per 10–16 h.
- Fissare l'RNA a membrana cuocendo a 120 gradi centigradi per 30 min.
- Eseguire l'ibridazione dell'RNA e il rilevamento immunologico con un kit commerciale (Tabella dei materiali) secondo le istruzioni del produttore.
11. Discriminazione delle estremità primarie ed elaborate 5'
- Discriminare le estremità primarie ed elaborate 5' confrontando i prodotti cRT-PCR ottenuti dagli RNA normalizzati 5' trattati con polifofofosi e non trattati.
NOT: Per le trascrizioni primarie, l'abbondanza di prodotti PCR provenienti da 5' RNA trattato con polifosforasi è molto superiore a quella della controparte non trattata (Figura1, "Gene A" e Figura 4A). Tuttavia, per le trascrizioni trattate, un livello comparabile di prodotti PCR sarebbe amplificatodalle due serie di RNA mitocondriali (Figura 1, "Gene B"). - Progettare i primer RT-qPCR in base ai risultati del mapping cRT-PCR.
- Verificare i risultati della discriminazione cRT-PCR da parte di RT-qPCR.
NOT: I due campioni di cDNA derivati da 5' RNA trattati con polifosfozola e non trattati sono normalizzati dai prodotti RT-qPCR di rRNA maturo 26S.
Risultati
Stima dell'efficienza di circolarizzazione dell'RNA mitocondriale
In uno studio precedente, sia gli RNA totali che quelli mitocondriali sono stati utilizzati per la mappatura cRT-PCR della trascrizione mitocondriale termini in Arabidopsis (Arabidopsis thaliana) e i due tipi di RNA hanno dato risultati di mappatura simili12. Inizialmente, abbiamo anche usato RNA totali per la mappatura cRT-PCR della trascrizione mitocondriale termini nel mais. Dopo molte prove, abbiamo scoperto che le trascrizioni bersaglio erano difficili da rilevare. Come miglioramento, abbiamo arricchito l'RNA mitocondriale dai chicchi di mais sviluppando kernel, e ha reso possibile l'amplificazione delle trascrizioni circolari del bersaglio in un round di PCR.
Per spiegare la facile amplificazione delle trascrizioni bersaglio dopo l'arricchimento, abbiamo stimato l'efficienza della circolarizzazione dell'RNA eseguendo RT-PCR sul gene mitocondriale rappresentativo nad5 (Figura 3 e Figura S2). Il gene nad5 di mais contiene due introni trans- e due cis-splicing. Dopo l'autolegatura degli RNA totali e mitocondriali, i primi cDNA di filamento sono stati sintetizzati in modo indipendente utilizzando nad5-RT1 e -RT2 primer. I due primer potrebbero invertire trascrivere sia nad5 mRNA lineari che circolari (Figura S2). Quattro coppie di primer convergenti sono utilizzati per l'amplificazione PCR: sqF1&sqR1 e sqF2&sqR2 per RT-PCR (RT-sqPCR) semi-quantitativo e qF1&qR1 e qF2&qR2 per RT-qPCR. Per i cDNA trascritti da nad5-RT1, tutte e quattro le coppie di primer hanno rilevato mRNA maturi circolari e lineari; per la reazione di trascrizione inversa nad5-RT2, sqF2&sqR2 e qF2&qR2 hanno esaminato entrambe le forme di mRNA, mentre i prodotti PCR sqF1&sqR1 e qF1&qR1 sono stati derivati solo da prodotti rimando circolati. Sulla base di questa analisi, abbiamo utilizzato i prodotti PCR sqF2&sqR2 (per RT-sqPCR) e qF2&qR2 (per RT-qPCR) per normalizzare le reazioni di trascrizione inversa nad5-RT1 e -RT2 e abbiamo stimato il rapporto tra nad5 circolari sqF1&sqR1 (per RT-sqPCR) o qF1&qR1 (per RT-qPCR) PCR. Prima dell'autolegamento, gli RNA sono stati pre-trattati da RNA 5' polifosforasi per rendere tutte le trascrizioni disponibili per la circolarizzazione da parte della ligase dell'RNA. L'analisi RT-sqPCR ha mostrato una frazione molto piccola di nad5 mRNA è stato circolato quando è stato utilizzato l'RNA totale, ma il rapporto è stato drammaticamente aumentato quando è stato utilizzato l'RNA mitocondriale arricchito (Figura 3B). I risultati di RT-qPCR hanno mostrato che il rapporto tra nad5 mRNA auto-ligato è aumentato dal 3,7% al 32% dopo l'arricchimento (Figura 3C). L'analisi di nad1 ha dato risultati simili, e l'efficienza di circolarizzazione di nad1 mRNA è aumentata da 0,7% a 30% (Figura 3D-F).
In base a queste analisi, circa il 30% degli RNA mitocondriali del mais si sono autoprodotti dopo il miglioramento, e l'aumento dell'efficienza circolarizzazione spiega la facile individuazione delle trascrizioni bersaglio da parte del cRT-PCR.
Determinazione del mais cox2 mRNA utilizzando la strategia basata su cRT-PCR
Abbiamo usato il gene cox2 come esempio per introdurre la mappatura e la discriminazione delle trascrizioni mitocondriali del mais utilizzando la strategia basata su cRT-PCR (Figura 4).
All'inizio della mappatura del coXX2 mRNA, tre primer rivolte verso l'esterno CF1, CF2 e CR1 sono state progettate sulla regione di codifica genica (Figura 4D). CF1 e CF2 erano primer nidificati, e CF2 era 69 bp a valle di CF1. Utilizzando il modello cDNA sintetizzato nei passaggi 6.1–6.5 e normalizzato ai passaggi 7.1–7.6, la coppia primer CF1&CR1 ha amplificato due bande prominenti e i risultati dell'amplificazione sono stati ripetuti dai primer nidificati CF2&CR1 (Figura 4A). Inoltre, le due bande sono state fortemente amplificate dall'RNA trattato con polifofofala 5' poliphphatasi, mentre erano difficili da rilevare nella controparte non trattata, suggerendo che sono sensibili alla polifanfia dell'RNA 5' e hanno termini primari 5' .
Le due bande candidate sono state denominate cox2-1 e -2, e sono state recuperate indipendentemente dal gel di agarose e clonate in vettori. I risultati della Colonia PCR hanno mostrato che i cloni positivi contengono inserti con dimensioni variabili, implicando termini eterogenei 5' e/o 3' delle trascrizioni (Figura 5). I cloni positivi sono stati sequenziati commercialmente e i dati di sequenziamento sono stati allineati con il genoma mitocondriale del mais come descritto nel passaggio 9.3.
I risultati del sequenziamento e dell'allineamento hanno mostrato che le due trascrizioni erano identiche a 3'estremità, ma diverse per la lunghezza di 5' UTR (Figura 4D). Le estremità di cox2-1 e -2 sono state arricchite rispettivamente a 992-1.030 e 1.276-1.283 nt a monte dell'AUG, mentre la loro estremità era di 39 nt a valle del codone di arresto. Dedotte dai risultati cRT-PCR, le dimensioni calcolate di cox2-1 e -2 erano rispettivamente 1.804–1.832 e 2.088–2.095 nt.
Per verificare i risultati della mappatura cRT-PCR, l'ibridazione delle macchie di gel d'RNA è stata eseguita utilizzando la sonda derivata dalla regione di codifica genica cox2 e sono state rilevate due bande principali con dimensioni simili a cox2-1 e -2 (Figura 4C). Inoltre, due bande più grandi circa 2.500 e 2.800 nt sono state rilevate nella macchia settentrionale, ma non sono state amplificate da cRT-PCR. Le due bande più grandi sono state nominate rispettivamente come cox2-3 e -4. Per mapparli, sono stati progettati altri due primer rivolti verso l'esterno (ad esempio CR2 e CF3), ancorati alle UTR 5' e 3' di cox2-2, e le loro posizioni erano vicine alle estremità di trascrizione previste di cox2-3 e -4. Utilizzando la coppia di primer CF3&CR2, due bande principali sono state fortemente amplificate da cDNA derivati da 5' RNA trattato con polifofophatasi ma non dalla controparte non trattata (Figura 4A). I risultati del sequenziamento hanno mostrato che avevano termini identici da 5', mentre le estremità variabili 3'. Le dimensioni calcolate delle due trascrizioni erano rispettivamente 2.512/2.513 e 2.833–2.835 nt, ed erano di dimensioni vicine con le due bande più grandi rilevate nella macchia settentrionale. Inoltre, i risultati di cRT-PCR hanno suggerito che i cox2-3 e -4 hanno termini primari da 5'.
Per confermare i risultati della discriminazione, cinque primer rivolti verso l'esterno sono stati progettati in base ai risultati della mappatura cRT-PCR, ad esempio qCF1, qCF2, qCR1, qCR2 e qCR3 e qCF2&qCR3 per Analisi RT-qPCR di cox2-1, -2, -3 e -4, rispettivamente. L'abbondanza relativa di tutti e quattro i prodotti RT-qPCR nel campione trattato con poliphsphatasi da 5' era molto più elevata di quelli della controparte non trattata, il che conferma i risultati della discriminazione cRT-PCR.
In sintesi, il modello di trascrizione del gene cox2 di mais è complicato e la strategia basata su cRT-PCR discrimina efficacemente e mappa le isoforme multiple dell'mRNA cox2.

Figura 1: Panoramica della strategia basata su cRT-PCR. Illustrazione dei principali passi della strategia basata su cRT-PCR. I due campioni di RNA trattati e non trattati rispettivamente da RNA 5' polyphosphatae. I due campioni di cDNA derivati da 5' RNA trattati con polifosfofosi e non trattati sono normalizzati da rRNA maturi 26S nel mais, e le trascrizioni primarie ed elaborate sono discriminate confrontando i prodotti cRT-PCR. Per il gene A, l'RNA corrispondente ha una fine primaria di 5' perché l'abbondanza del prodotto PCR da 5' RNA trattato con polifosforasi è molto superiore a quello della controparte non trattata; per il gene B, i prodotti PCR provenienti dai due gruppi di RNA mitocondriali sono amplificati a un livello comparabile, il che implica un capolinea elaborato 5' dell'RNA corrispondente. L'area di codifica e le UTR sono rappresentate rispettivamente da linee grigie e in grassetto. CRT - primer per la trascrizione inversa; CF1, CF2, CR1 e CR2 - primer PCR che fiancheggiano la giunzione 5'-3' della trascrizione circolare; qCF e qCR - primer divergenti per RT-qPCR. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Valutazione dell'integrità dell'RNA mitocondriale mediante elettroforesi gel agarose. (A) RNA mitocondriali preparati da kernel di mais da 15 DAP. M - Marcatore molecolare del DNA. (B) Kernel RNA mitocondriale dopo il trattamento da 5' polifosfolasi e/o circolarizzazione da T4 RNA ligase. 5' - RNA mitocondriale dopo il trattamento da 5' polifosculo; T4&5' , RNA mitocondriale dopo il trattamento da 5' polifosforasi e circolarizzazione da parte della ligase t4 RNA; T4&5'- - RNA mitocondriale solo dopo la circolarizzazione. Fare clic qui per visualizzare una versione più grande di questa figura.
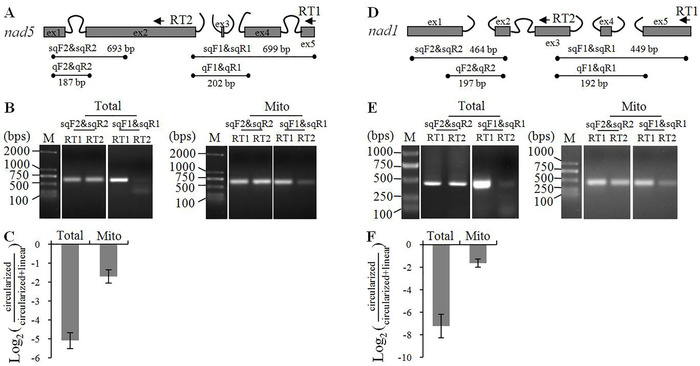
Figura 3: Efficienza di circolarizzazione di nad5 e nad1 mRNA stimati dagli RT-PCR. (A) e (D) Diagramma schematico dei geni nad5 e nad1, rispettivamente. Es. Gli esoni e gli introni vengono visualizzati rispettivamente come caselle grigie e linee curve. La posizione dei primer di trascrizione inversa RT1 e RT2 è indicata dalle frecce. I punti neri indicano la posizione dei primer PCR e viene visualizzata la dimensione prevista dei prodotti PCR. sqF1, sqF2, sqR1 e sqR2 sono primer per RT-sqPCR; qF1, qF2, qR1 e qR2 sono primer per RT-qPCR. (B) e (E) RT-sqPCR analisi dell'efficienza circolarizzazione dei nad5 e nad1 mRNA, rispettivamente. M - Marcatore molecolare del DNA. (C) e (F) RT-qPCR analisi dell'efficienza circolarizzazione dei nad5 e nad1 mRNA, rispettivamente. I due campioni cDNA invertiti dai primer RT1 e RT2 sono normalizzati da sqF2&sqR2 (per RT-sqPCR) o qF2&qR2 (per i prodotti PCR RT-qPCR). L'efficienza di circolarizzazione di nad5 e nad1 mRNA è stimata confrontando i prodotti PCR di sqF1&sqR1 (per RT-sqPCR) o qF1&qR1 (per RT-qPCR). circularized/(circolare-lineare) - qF1&qR1 su qF2&qR2 nella reazione RT2/ qF1&qR1 rispetto a qF2&qR2 nella reazione RT1. I valori sono mezzi e SD di tre repliche biologiche. Per escludere la potenziale influenza da 5 o più triposfati, gli RNA sono stati trattati da 5 ostiliasi prima dell'auto-legatura. Total e Mito: RNA totali e mitocondriali, rispettivamente. RT1 e RT2 - cDNA invertiti da primer RT1 e RT2, rispettivamente Fare clic qui per visualizzare una versione più grande di questa figura.
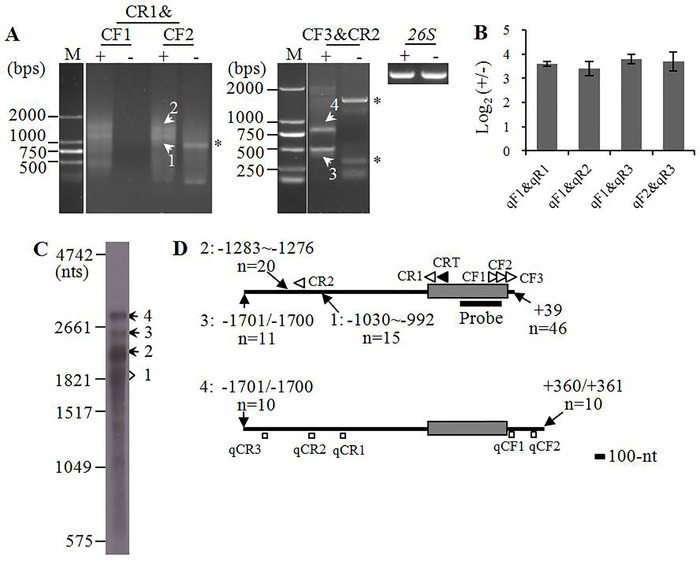
Figura 4: Mappatura del termine mRNA maturo cox2 utilizzando la strategia con maiuscole/minuscole cRT-PCR. (A) Separazione gel dei prodotti cox2 cRT-PCR. - - cDNA derivati da 5 o più RNA mitocondriali trattati con polifofofolosi e non trattati. I due campioni di cDNA sono stati normalizzati dall'amplificazione di 26S cDNA (26S). Per l'analisi cRT-PCR dell'mRNA cox2 circolare vengono utilizzati cinque primer e CF2 e CF2 e CF3 sono rispettivamente 69 e 138 bp a valle di CF1; CR1 e CR2 sono primer nidificati e CR2 è 1.288 bp a monte di CR1. Le bande indicate da '1' e '2' sono state amplificate sia dalla CF1&CR1 che dalla CF2&CR1, mentre le bande '3' e '4' sono state amplificate dalla CF3&CR2. Tutte e quattro le bande sono state sequenziate dalla clonazione. Le bande contrassegnate da asterischi non potevano essere ripetute, e sono state escluse dai risultati. M - Marcatore molecolare del DNA. (B) Analisi RT-qPCR della relativa abbondanza di mRNA cox2 circolari dopo il trattamento da 5' polifosfore. I due campioni di cDNA sono stati sintetizzati da una miscela di primer contenente 26S-CRT, cox2-CRT, nad5-CRT, nad6-CRT, nad7-CRT, nad9-CRT, cob-CRT e cox1-CRT a uguale rapporto, e il cDNA derivato da 26S maturo rRNA è stato utilizzato per la normalizzazione. cox2 su 26S nel campione trattato/cox2 su 26S nel campione non trattato. I valori rappresentano mezzi e SD di tre repliche biologiche. Sono indicati i primer utilizzati per RT-qPCR. (C) Analisi settentrionale delle trascrizioni di cox2. È stato caricato 2 RNA mitocondriale. Le bande corrispondenti a diverse isoforme di mRNA maturo cox2 sono contrassegnate. (D) Termini trascrizione di mRNA cox2 dedotti dai risultati cRT-PCR. Gli UTR e i riquadri di lettura aperti vengono visualizzati rispettivamente come linee in grassetto e caselle grigie. Vengono mostrate le posizioni dei termini 5' e 3' rispetto all'AUG (1) e all'UAA (-1) e i numeri di singoli cloni in sequenza in tali posizioni. Le posizioni dei primer di trascrizione inversa e di amplificazione PCR vengono visualizzate rispettivamente come teste di freccia chiuse e aperte. I primer rivolti verso l'esterno utilizzati per RT-qPCR vengono visualizzati come quadrati aperti e la posizione della sonda cox2 è indicata. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Selezione di cloni positivi contenenti inserti cox2-4 per colonia PCR. (A) Colonia PCR per vagliare singoli cloni contenenti inserti cox2-4. La dimensione dell'inserto cox2-4 è di circa 1.165 bp e quella delle sequenze vettoriali è 100 bp. La dimensione calcolata dei prodotti PCR colonia è di circa 1.265 bp, e quei cloni contenenti inserti di piccole dimensioni non sono stati sequenziati, cioè i numeri 11, 14, 22 e 23. (B) Sequenziamento dei risultati dei cloni positivi selezionati in (A). 5'/3' estremità : le estremità 5' e 3' relative rispettivamente all'AUG (-1) e all'UAA (-1). I risultati del sequenziamento dei numeri 12 e 20 sono stati esclusi perché erano molto più piccoli degli altri. Fare clic qui per visualizzare una versione più grande di questa figura.
| componente | Volume per una reazione di 50 l (L) | Concentrazione finale |
| 10 volte buffer polifosfore da 5 | 5 Del numero 3( | 1x (in modo non il |
| Inibitore di RNase (40 U/L) | 0,75 (in questo 0,05) | 0,6 U/L |
| RNA mitocondriale | X | 0,2 g/L |
| RNA 5' polifosforasi (20 U/ L) | 2.5 24,5 | 1 U/L |
| DePC trattato dionizzato H2O | fino a 50 l | - |
Tabella 1: Componenti di reazione del trattamento della polifosfato di RNA 5'
| componente | Volume per 20 reazione l (L) | Concentrazione finale |
| 10x T4 RNA ligase I tampone | 2 Il nome del sistema | 1x (in modo non il |
| dATP (1 mM) | 1 : il nome del | 50 M |
| PEG8000 (50%) | 6 È possibile: | 15% |
| Inibitore dell'RNA (40 U/L) | 0,5 0,5 | 1 U/L |
| 5' RNA mitocondriale trattato con polifosfofosi o non trattato | X | 0,1,0,2 g/ |
| T4 RNA ligase I (30 U/L) | 0,4 0 | 0,6 U/L |
| DePC trattati dizione H2O | a 20 l | - |
Tabella 2: Componenti di reazione della circolarizzazione dell'RNA mitocondriale.
| componente | Volume per 10 pre-miscela |
| Miscela primer (1 m totale) | 2 Il nome del sistema |
| miscela dNTP (10 mM ciascuna) | 1 : il nome del |
| RNA mitocondriale b trattato con polifosfolada 5' o non trattato | X |
| DePC trattato dionizzato H2O | a 10 l |
Tabella 3: Pre-miscela per la reazione di trascrizione inversa. un La miscela di primer contiene lo stesso rapporto di 26S-CRT e fino ad altri 7 primer RT, e la concentrazione finale è di 1 M. bDue pre-miscele sono preparate fianco a fianco, e contengono la stessa quantità (200 ng) di polifosfore circolari 5' o trattate con polifosfano o trattate con polifosfa RNA mitocondriale non trattato.
| componente | Volume per il sistema di reazione a 20 l | Concentrazione finale |
| Modello RNA/primer/dNTP | 10 del sistema | - |
| 5x buffer RTase | 4 DEL psu' | 1x (in modo non il |
| Inibitore di RNase (40 U/L) | 0,5 0,5 | 1 U/L |
| RTasi (200 U/L) | 1 : il nome del | 10 U/L |
| DePC trattati dizione H2O | a 20 l | - |
Tabella 4: Componenti di reazione della trascrizione inversa. - Il modello RNA/primer/dNTP pre-mixtures preparato al passo 6.3.
| componente | Volume per il sistema di reazione a 20 l | Concentrazione finale |
| Deionizzato H2O | 7 (in questo stato | - |
| Buffer di reazione 2x | 10 del sistema | 1x (in modo non il |
| miscela dNTP (10 mM ciascuna) | 0,4 0 | 0,2 mM ciascuno |
| 26S-CF1 (10 M) | 0,8 (in questo calibro) | 0,4 mM |
| 26S-CR1 (10 M) | 0,8 (in questo calibro) | 0,4 mM |
| Modelli cDNA derivati da RNA automatristatici trattati con polifosfato si o non trattati con polifosfato | 0,6 (in inglese) | - |
| Polimerasi del DNA (1 U/L) | 0,4 0 | 0,02 U/L |
Tabella 5: Componenti di reazione per l'amplificazione PCR di 26S cDNA. Inizialmente, per la normalizzazione viene utilizzato lo stesso volume dei modelli cDNA (0,6 ) . Se necessario, modificare la quantità di modello cDNAs per garantire la stessa abbondanza di 26S pcR prodotti tra le due reazioni PCR.
| passo | temperatura | ora | Numero ciclo |
| Denaturazione iniziale | 95 gradi centigradi | 3 min | 1 ciclo |
| Denaturazione | 95 gradi centigradi | 15 sec | 22–25 cicli |
| Primer annealing | 56 gradi centigradi | 15 sec | |
| interno | 72 gradi centigradi | 30 sec | |
| Estensione finale | 72 gradi centigradi | 5 min | 1 ciclo |
| tenere | 4 gradi centigradi | ∞ | - |
Tabella 6: condizioni PCR per amplificare 26S (in inglese) cDNA per la normalizzazione.
| componente | Volume per il sistema di reazione a 20 anni | Concentrazione finale |
| Deionizzato H2O | a 20 l | - |
| Buffer di reazione 2x | 10 del sistema | 1x (in modo non il |
| miscela dNTP (10 mM ciascuna) | 0,4 0 | 0,2 mM ciascuno |
| Primer avanti divergente (10 M) | 0,8 (in questo calibro) | 0,4 mM |
| Primer-reverse divergente (10 M) | 0,8 (in questo calibro) | 0,4 mM |
| Modello cDNA derivato dagli RNA circolari 5' trattati con polifosfosi o non trattati | X | - |
| Polimerasi del DNA (1 U/L) | 0,4 0 | 0,02 U/L |
Tabella 7: Componenti di reazione per l'amplificazione PCR delle trascrizioni circolari del bersaglio. Il volume dei cDNA del modello utilizzati in queste reazioni è determinato dai risultati della normalizzazione del rRNA 26S.
| passo | temperatura | ora | Numero ciclo |
| Denaturazione iniziale | 95 gradi centigradi | 3 min | 1 ciclo |
| Denaturazione | 95 gradi centigradi | 15 sec | 22-40 cicli c |
| Primer annealing un | 50-65 gradi centigradi | 15 sec | |
| Estensione b | 72 gradi centigradi | 0,5-1 min | |
| Estensione del finto | 72 gradi centigradi | 5 min | 1 ciclo |
| tenere | 4 gradi centigradi | ∞ | - |
Tabella 8: condizioni PCR per amplificare le trascrizioni di destinazione. un L'esatta temperata anneale dipende dalla temperatura di fusione dei primer PCR. b Il tempo di allungamento dipende dalla lunghezza del bersaglio da amplificare. Il tempo consigliato è 1 min per 1 kb del frammento PCR. c In generale, 30-35 cicli sono sufficienti per produrre una quantità adeguata di prodotto PCR. Per le trascrizioni abbondanti, aumentare il numero di cicli fino a 40 cicli.
Figura S1: Posizione dei primer per le trascrizioni mitocondriali di mais rappresentative. CRT - primer di trascrizione inversa; CF1, CF2, CR1 e CR2 - primer divergenti per amplificazione PCR; qCF e qCR - primer divergenti per RT-qPCR. La trascrizione del mais nad2-1, rps4-1 e nad4-1 è stata determinata in precedenza (et al., 20191). Vengono mostrate le posizioni delle estremità a 5'- e 3'rispetto all'AUG (n. 1) e all'ultimo nucleotide del codon di arresto (-1). Le aree di codifica e le UTR sono indicate rispettivamente come caselle grigie e linee in grassetto. Fare clic qui per scaricare questo file.
Figura S2: Principio per stimare l'efficienza circolarizzazione dell'mRNA nad5 nel mais. In una reazione di auto-legazione, solo una frazione degli RNA mitocondriali è circolarizzata. Per calcolare il rapporto tra mRNA nad5 circolare, vengono utilizzati due primer specifici del gene per sintetizzare i primi cDNA del filamento, cioè nad5-RT1 e -RT2. Nella reazione nad5-RT2 reverse transcription (RT), i prodotti PCR amplificati da sqF2&sqR2 (per RT-sqPCR) e qF2&qR2 (per RT-qPCR) sono derivati sia da nad5 lineari che circolari, mentre il sqF1&sqR1 (per RT-sqPCR) e qF1 I prodotti PCR &qR1 (per RT-qPCR) derivano solo da nad5 circolari; nella reazione nad5-RT1, tutte e quattro le coppie di primer PCR potrebbero amplificare entrambe le forme di nad5. Per calcolare il rapporto tra nad5 mRNA circolari, le due reazioni sono normalizzate da prodotti PCR sqF2&sqR2 o qF2&qR2. Confrontando l'abbondanza di prodotti PCR sqF1&sqR1 o qF1&qR1 tra le due reazioni RT, l'efficienza circolarizzazione dell'mRNA nad5 è approssimativamente stimata. ex - eson. Gli esoni e gli introni vengono visualizzati rispettivamente come caselle grigie e linee curve. Le posizioni dei numeri di rime nad5-RT1 e -RT2 sono indicate da frecce. I punti neri indicano le posizioni dei primer PCR e vengono visualizzate le dimensioni previste dei prodotti PCR. Fare clic qui per scaricare questo file.
Tabella S1: Informazioni primer. Fare clic qui per scaricare questo file.
Discussione
In uno studio precedente, gli RNA totali e mitocondriali della coltura delle sospensioni cellulari dell'Arabidopsis sono stati utilizzati per mappare i termini trascrizione mitocondriale da cRT-PCR, e risultati simili sono stati ottenuti12. Tuttavia, solo l'RNA mitocondriale arricchito è stato utilizzato per mappare la trascrizione mitocondriale termini in molti altri studi1,2,3,9. Abbiamo scoperto che l'arricchimento dell'RNA mitocondriale è un passo importante per la mappatura cRT-PCR della trascrizione mitocondriale termini nel mais. Dopo questo arricchimento, il rapporto tra l'RNA mitocondriale circolare è notevolmente aumentato dallo 0,3%–3,7% al 30% (Figura3 e Figura S2),il che rende possibile l'amplificazione delle trascrizioni di bersagli circolari in un round di PCR.
L'efficienza circolarizzazione dell'RNA mitocondriale è stata stimata dall'analisi RT-PCR sul mais nad1 e nad5, entrambi suddivisi in precursori indipendenti da introni cis-splicing. Poiché la potenziale influenza dalla presenza di RNA precursori e la variazione dell'efficienza della RT non potevano essere escluse, questa è solo una stima approssimativa dell'efficienza reale della circolazione dell'RNA mitocondriale.
Abbiamo alterato le condizioni di auto-ligazione alterando la concentrazione di RNA mitocondriale e PEG8000, così come allungando il tempo di incubazione, ma questi cambiamenti non sembravano influenzare l'efficienza dell'RNA circolare mitocondriale. Anche se non siamo sicuri se un'ulteriore purificazione del gradiente di Percoll possa aumentare l'efficienza della circolarizzazione, la qualità del mitocondrio grezzo preparato nella sezione 2 è sufficiente per la mappatura cRT-PCR della trascrizione mitocondriale nel mais. Poiché più cicli di PCR possono causare risultati falsi positivi, l'amplificazione delle trascrizioni di destinazione in un PCR round rende i risultati di mappatura più affidabili.
Il triposfato 5' delle trascrizioni primarie è instabile, e potrebbe essere convertito in monofato 5' per ragioni sconosciute. Questo problema ostacola una netta differenziazione tra le trascrizioni primaria ed elaborata utilizzando approcci a seconda della presenza/assenza di 7' triposfata1,2,4. La forma matura di mais 26S rRNA ha una fine monofosato 5' ed è insensibile al polifoszosi di RNA 5'. Per ridurre al minimo l'influenza del triphospahte instabile 5' viene utilizzato per normalizzare i due campioni di RNA, trattati e non trattati da 5' polifoszosi, ed è dimostrato essere un passo importante per differenziare i due tipi di trascrizioni nel mais Mitocondrio. Dopo la normalizzazione, le trascrizioni primarie ed elaborate potrebbero essere discriminate confrontando i risultati cRT-PCR e RT-qPCR ottenuti da 5 campioni di RNA trattati con polifofofolo e non trattati. Le trascrizioni primarie sono sensibili alla polifosfolasi da 5' e sono fortemente amplificate dal campione trattato con polifosfosi da 5' ma non (o a un livello molto basso a causa del trefosfato instabile 5') della controparte non trattata. Al contrario, le trascrizioni elaborate vengono rilevate a livelli comparabili tra i due campioni.
Nelle piante, i modelli trascrizioni di molti geni mitocondriali sono piuttosto complicati1,2. Ad esempio, i geni nad6 e atp6 di mais sono espressi sia come monocistron e dicistron, e nad6, atp6e atp6-na6 maturi mRNA hanno rispettivamente due, tre e due isoforme. Inoltre, la popolazione di trascrizione di un gene mitocondriale è in realtà una miscela di RNA precursori, maturi e degradanti. I PCR sono inclini ad amplificare molecole di piccole dimensioni, ed è difficile rilevare tutte le isoforme di trascrizione utilizzando una coppia di primer. Pertanto, l'ibridazione delle macchie di gel di RNA è necessaria per verificare i risultati della mappatura cRT-PCR e potrebbero essere necessari primer alternativi per amplificare tali trascrizioni rilevate nella macchia settentrionale, ma non per la prima volta cRT-PCR.
Questo protocollo include un passaggio RT-qPCR per verificare i risultati della discriminazione cRT-PCR. Poiché le isoforme multiple di alcuni mRNA maturi sono molto vicine nella dimensione1, è impossibile confermare tutti i risultati della discriminazione da parte di RT-qPCR e alcune trascrizioni a stato costante sono state determinate confrontando i risultati cRT-PCR tra i trattati solo RNA non trattati.
In questo protocollo, il termine 5' e 3' delle trascrizioni target è stato mappato mediante clonazione e sequenziamento dei prodotti cRT-PCR, e limita il numero di cloni singoli da sequenziare. Come metodo alternativo, i prodotti cRT-PCR potrebbero essere sequenziati dal sequenziamento di nuova generazione, che sequenzia migliaia di molecole per un insieme di trascrizioni a stato costante, e i risultati della mappatura sarebbero più accurati.
Oltre alla discriminazione del termine primario e lavorato 5' da cRT-PCR e RT-qPCR, il termine monofosfato 5' potrebbe essere determinato mediante l'identificazione delle strutture secondarie dell'RNA circostanti1,12. È risaputo che le strutture secondarie dell'RNA come il tRNA e l'elemento t potrebbero mediare la formazione finale della trascrizione mitocondriale dirigendo scissioni endonucleolitici di RNase P e/o RNase12,18,19, 20. Pertanto, i termini 5' adiacenti al tRNA o all'elemento t dovrebbero essere derivati dall'elaborazione post-trascrizione e contenere i monoffati.
Utilizzando questo protocollo, gran parte delle trascrizioni a stato costante sono state determinate nei mitocondri di mais1. Tuttavia, alcuni non sono stati differenziati e/o mappati per motivo sconosciuto1. Inoltre, riteniamo che la posizione del termine di trascrizione 5' sia meglio essere confermata dall'analisi dell'estensione del primer, anche se è inclusa in questo protocollo. A causa della mancanza di dati sperimentali, non siamo sicuri se questa strategia sia adatta ad altre specie vegetali, come il riso (Oryza sativa) e l'Arabidopsis . Oltre ai mitocondri vegetali, le trascrizioni primarie ed elaborate sono accumulate stabilmente in plastidi21, e non è certo se questa strategia potrebbe essere utilizzata per mappare e discriminare le trascrizioni plastidi.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dalla National Natural Science Foundation of China (n. 31600250, Y. , Science and Technology Projects of Guangzhou City (grant n. 201804020015, H.N.) e dal China Agricultural Research System (grant no. CARS-04-PS09, H.N.).
Materiali
| Name | Company | Catalog Number | Comments |
| Acetic acid | Aladdin, China | A112880 | To prepare 1x TAE buffer |
| Applied Biosystems 2720 Thermal Cycler | Thermo Fisher Scientific, USA | 4359659 | Thermal cycler for PCR amplification |
| Ascorbic acid | Sigma-aldrich, USA | V900134 | For preparation of extraction buffer |
| Biowest Agarose | Biowest, Spain | 9012-36-6 | To resolve PCR products and RNAs |
| Bovine serum albumin | Sigma-aldrich, USA | A1933 | For preparation of extraction buffer |
| Bromophenol blue | Sigma-aldrich, USA | B8026 | For preparation of loading buffer for agarose gel electrophoresis and Northern blot |
| DEPC | Sigma-aldrich, USA | V900882 | Deactivation of RNase |
| DIG Northern starter kit | Roche, USA | 12039672910 | For DIG-RNA labeling and Northern blot. This kit contains the reagents for transcription-labeling of RNA with DIG and T7 RNA polymerase, hybridization and chemiluminescent detection. |
| EDTA | Sigma-aldrich, USA | V900106 | For preparation of extraction buffer and 1x TAE buffer |
| EGTA | Sigma-aldrich, USA | E3889 | For preparation of wash buffer |
| Gel documentation system | Bio-Rad, USA | Gel Doc XR+ | To image the agarose gel |
| Glycerol | Sigma-aldrich, USA | G5516 | For preparation of loading buffer for agarose gel electrophoresis |
| GoldView II (5,000x) | Solarbio,. China | G8142 | DNA staining |
| Hybond-N+, Nylon membrane | Amersham Biosciences, USA | RPN119 | For Northern blot |
| Image Lab | Bio-Rad, USA | Image Lab 3.0 | Image gel, and compare the abundance of PCR products. |
| KH2PO4 | Sigma-aldrich, USA | V900041 | For preparation of extraction buffer |
| KOH | Aladdin, China | P112284 | For preparation of extraction buffer |
| L-cysteine | Sigma-aldrich, USA | V900399 | For preparation of extraction buffer |
| Millex | Millipore, USA | SLHP033RB | To sterile extraction and wash buffers by filtration |
| Miracloth | Calbiochem, USA | 475855-1R | To filter the ground kernel tissues |
| MOPS | Sigma-aldrich, USA | V900306 | For preparation of running buffer for Northern blot |
| NanoDrop | Thermo Fisher Scientific, USA | 2000C | For RNA concentration and purity assay |
| NaOH | Sigma-aldrich, USA | V900797 | For preparation of wash buffer |
| pEASY-Blunt simple cloning vector | TransGen Biotech, China | CB111 | Cloning of the gel-recovered band. It contains a T7 promoter several bps upstream of the insertion site. |
| Phanta max super-fidelity DNA polymerase | Vazyme, China | P505 | DNA polymerase for PCR amplification |
| Polyvinylpyrrolidone 40 | Sigma-aldrich, USA | V900008 | For preparation of extraction buffer |
| Primer Premier 6.24 | PREMIER Biosoft, USA | Primer Premier 6.24 | To design primers for reverse transcription and PCR amplification |
| PrimeScript II reverse transcriptase | Takara, Japan | 2690 | To synthesize the first strand cDNA |
| PureLink RNA Mini kit | Thermo Fisher Scientific, USA | 12183025 | For RNA purificaion |
| RNA 5' polyphosphatase | Epicentre, USA | RP8092H | To convert 5' triphosphate to monophosphate |
| RNase inhibitor | New England Biolabs, UK | M0314 | A component of RNA self-ligation and 5' polyphosphatase treatment reactions, and it is used to inhibite the activity of RNase. |
| Sodium acetate | Sigma-aldrich, USA | V900212 | For preparation of running buffer for Northern blot |
| Sodium chloride | Sigma-aldrich, USA | V900058 | To prepare 20x SSC |
| SsoFas evaGreen supermixes | Bio-Rad, USA | 1725202 | For RT-qPCR |
| T4 RNA Ligase 1 | New England Biolabs, UK | M0437 | For RNA circularization |
| Tetrasodium pyrophosphate | Sigma-aldrich, USA | 221368 | For preparation of extraction buffer |
| TIANgel midi purification kit | Tiangen Biotech, China | DP209 | To purify DNA fragments from agarose gel |
| Tris | Aladdin, China | T110601 | To prepare 1x TAE buffer |
| TRIzol reagent | Invitrogen, USA | 15596026 | To extract mitochondiral RNA. |
| Universal DNA purification kit | Tiangen Biotech, China | DP214 | To recover linearized plastmids from the restriction enzyme digestion reaction |
| Xylene cyanol FF | Sigma-aldrich, USA | X4126 | For preparation of loading buffer for agarose gel electrophoresis |
Riferimenti
- Zhang, Y., et al. Major contribution of transcription initiation to 5'-end formation of mitochondrial steady-state transcripts in maize. RNA Biology. 16 (1), 104-117 (2019).
- Choi, B. Y., Acero, M. M., Bonen, L. Mapping of wheat mitochondrial mRNA termini and comparison with breakpoints in DNA homology among plants. Plant Molecular Biology. 80 (4-5), 539-552 (2012).
- Calixte, S., Bonen, L. Developmentally-specific transcripts from the ccmFN-rps1 locus in wheat mitochondria. Molecular Genetics and Genomics. 280 (5), 419-426 (2008).
- Kuhn, K., Weihe, A., Borner, T. Multiple promoters are a common feature of mitochondrial genes in Arabidopsis. Nucleic Acids Research. 33 (1), 337-346 (2005).
- Jonietz, C., Forner, J., Holzle, A., Thuss, S., Binder, S. RNA PROCESSING FACTOR2 is required for 5' end processing of nad9 and cox3 mRNAs in mitochondria of Arabidopsis thaliana. ThePlant Cell. 22 (2), 443-453 (2010).
- Stoll, B., Stoll, K., Steinhilber, J., Jonietz, C., Binder, S. Mitochondrial transcript length polymorphisms are a widespread phenomenon in Arabidopsis thaliana. Plant Molecular Biology. 81 (3), 221-233 (2013).
- Mulligan, R. M., Lau, G. T., Walbot, V. Numerous transcription initiation sites exist for the maize mitochondrial genes for subunit 9 of the ATP synthase and subunit 3 of cytochrome oxidase. Proceedings of the National Academy of Sciences of the United States of America. 85 (21), 7998-8002 (1988).
- Lupold, D. S., Caoile, A. G., Stern, D. B. The maize mitochondrial cox2 gene has five promoters in two genomic regions, including a complex promoter consisting of seven overlapping units. Journal of Biological Chemistry. 274 (6), 3897-3903 (1999).
- Yan, B., Pring, D. R. Transcriptional initiation sites in sorghum mitochondrial DNA indicate conserved and variable features. Current Genetics. 32 (4), 287-295 (1997).
- Rapp, W. D., Lupold, D. S., Mack, S., Stern, D. B. Architecture of the maize mitochondrial atp1 promoter as determined by linker-scanning and point mutagenesis. Molecular and Cellular Biology. 13 (12), 7232-7238 (1993).
- Rapp, W. D., Stern, D. B. A conserved 11 nucleotide sequence contains an essential promoter element of the maize mitochondrial atp1 gene. The EMBO Journal. 11 (3), 1065-1073 (1992).
- Forner, J., Weber, B., Thuss, S., Wildum, S., Binder, S. Mapping of mitochondrial mRNA termini in Arabidopsis thaliana: t-elements contribute to 5' and 3' end formation. Nucleic Acids Research. 35 (11), 3676-3692 (2007).
- Binder, S., Stoll, K., Stoll, B. Maturation of 5' ends of plant mitochondrial RNAs. Physiologia Plantarum. 157 (3), 280-288 (2016).
- Hang, R. L., Liu, C. Y., Ahmad, A., Zhang, Y., Lu, F. L., Cao, X. F. Arabidopsis protein arginine methyltransferase 3 is required for ribosome biogenesis by affecting precursor ribosomal RNA processing. Proceedings of the National Academy of Sciences of the United States of America. 111 (45), 16190-16195 (2014).
- Wang, H. Q., et al. Maize Urb2 protein is required for kernel development and vegetative growth by affecting pre-ribosomal RNA processing. New Phytologist. 218 (3), 1233-1246 (2018).
- Maloney, A. P., et al. Identification in maize mitochondrial 26S rRNA of a short 5'-end sequence possibly involved in transcription initiation and processing. Current Genetics. 15 (3), 207-212 (1989).
- Green, R. M., Sambrook, J. Molecular Cloning: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor. New York. (2012).
- Canino, G., et al. Arabidopsis encodes four tRNase Z enzymes. Plant Physiology. 150 (3), 1494-1502 (2009).
- Gobert, A., et al. A single Arabidopsis organellar protein has RNase P activity. Nature Structural, Molecular Biology. 17, 740-744 (2010).
- Gutmann, B., Gobert, A., Giege, P. PRORP proteins support RNase P activity in both organelles and the nucleus in Arabidopsis. Genes & Development. 26 (10), 1022-1027 (2012).
- Stern, D. B., Goldschmidt-Clermont, M., Hanson, M. R. Chloroplast RNA metabolism. Annual Review of Plant Biology. 61, 125-155 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon