Method Article
Un metodo di estrazione multi-omics per l'analisi approfondita delle culture sincronizzate dell'Alga verde Chlamydomonas reinhardtii
In questo articolo
Riepilogo
L'analisi a livello di sistema di biomolecole multiple è fondamentale per ottenere informazioni funzionali e meccanicistiche sui processi biologici. In questo modo, viene descritto un ampio protocollo per l'estrazione ad alta produttività di lipidi, metaboliti, proteine e amido da un singolo campione raccolto dalla coltura sincronizzata di Chlamydomonas.
Abstract
Le microalghe sono state al centro della ricerca per le loro applicazioni nella produzione di composti di alto valore, cibo e carburante. Inoltre, sono preziosi modelli fotosintetici che facilitano la comprensione dei processi cellulari di base. Gli studi a livello di sistema consentono una comprensione completa e approfondita delle funzioni molecolari degli organismi. Tuttavia, più campioni e protocolli indipendenti sono necessari per studi di proteomica, lipidomica e metabolomica che introducono un errore e una variabilità più elevati. Un robusto metodo di estrazione ad alta produzione per l'estrazione simultanea di clorofilla, lipidi, metaboliti, proteine e amido da un singolo campione di alga verde Chlamydomonas reinhardtii è presentato qui. La configurazione sperimentale illustrata è per le colture Chlamydomonas sincronizzate utilizzando condizioni di luce/buio di 12 h/12 h. Sono stati raccolti campioni in un ciclo cellulare di 24 h per dimostrare che i metaboliti, i lipidi e i dati dell'amido ottenuti utilizzando varie piattaforme analitiche sono ben conformi. Inoltre, sono stati utilizzati campioni di proteine raccolti utilizzando lo stesso protocollo di estrazione per condurre un'analisi proteomica dettagliata per valutarne la qualità e la riproducibilità. Sulla base dei dati, si può dedurre che il metodo illustrato fornisce un approccio robusto e riproducibile per far progredire la comprensione dei vari percorsi biochimici e delle loro funzioni con maggiore fiducia sia per la ricerca di base che per quella applicata.
Introduzione
Le microalghe sono una ricca fonte di prodotti naturali (ad esempio, carburanti, nutrizione umana e animale, cosmetici e sostanze farmacologiche). Numerosi sforzi di ricerca sono effettuati per migliorare l'efficienza della produzione di prodotti di alto valore da microalghe1,2,3,4. La comprensione a livello di sistemi del metabolismo è un prerequisito per migliorare la qualità e la resa dei prodotti naturali5,6,7. Con l'avvento di tecniche genomiche funzionali e migliori metodi di spettrometria di massa, migliaia di geni, trascrizioni, proteine e metaboliti possono essere monitorati contemporaneamente. Tuttavia, più campioni sono necessari per studi di proteomica approfondita, lipidomica e metabolomica, che è spesso difficile da ottenere negli organismi unicellulari, soprattutto se devono essere eseguiti studi di corsi temporali. Inoltre, la raccolta e l'elaborazione di diversi campioni in combinazione con protocolli diversi per raccogliere i dati omici altamente complessi (ad esempio, proteomica, lipomica e metabolomica) introduce variabilità, rendendo così l'integrazione dei dati un compito impegnativo.
Chlamydomonas fornisce non solo un eccellente sistema microbico per lo studio dei processi cellulari, ma anche un modello conveniente per studiare la coordinazione del ciclo cellulare e metabolismo. Di conseguenza, è stata mostrata una forte coordinazione dell'espressione delle trascrizioni con il ciclo cellulare utilizzando la profilazione trascrittoma ad alta risoluzione della coltura sincronizzata di Chlamydomonas8. Circa l'80% delle trascrizioni analizzate ha mostrato una solida periodicità su un ciclo cellulare di 24 h8. Allo stesso modo, il peso secco, le proteine, la clorofilla, gli amminoacidi e gli acidi grassi di due diversi ceppi di Chlamydomonas hanno dimostrato di correlarsi con la divisione cellulare in uno studio in cui il campionamento veniva eseguito ogni 4 h9. Recentemente, è stato riferito che la dinamica dei metaboliti e dei lipidi dello spostamento cellulare sulla base di fasi specifiche del ciclo cellulare10. I sottili cambiamenti nelle diverse biomolecole sono stati possibili monitorare utilizzando un robusto tertmetile -butyl ether (MTBE): metanolo: metodo di estrazione a base d'acqua, che offre un punto di partenza ideale per l'analisi multiomica completa10 , 11.
Il protocollo presentato guida attraverso una strategia riproducibile ed efficiente10, per l'estrazione simultanea di lipidi, metaboliti, proteine e amido da un singolo campione di aliquota, per il tempo risolto studio metabolomico e lipidomico di in crescita sincrona culture Chlamydomonas. Oltre a illustrare i dati metabolici e lipidomici robusti e riproducibili10,qui è dimostrata anche la qualità dei campioni proteomici ottenuti dallo stesso pellet.
Protocollo
1. Pre-culture di Chlamydomonas reinhardtii
- Preparare le pre-colture di Chlamydomonas reinhardtii (ceppo selvaggio cc-1690 mt) trasferendo le cellule da piastre solide di media TAP a flaconetti Erlenmeyer con 200 mL di HSM medio12.
- Far crescere le precolture su uno shaker rotatoria a 24 gradi centigradi a 100 .m-2s-1 luce continua fino a quando la densità cellulare raggiunge i 2 x 106 celle/mL.
2. Sincronizzazione delle colture liquide Chlamydomonas nei fermentatori
NOTA: I parametri presentati in questo protocollo, come temperatura, luce e CO2, sono specifici per la sincronizzazione della deformazione CC-1690 mt. Per sviluppare una coltura sincronizzata utilizzando un altro ceppo, è necessario testare le condizioni ottimali.
- Trasferire la pre-coltura in un sistema di fermi (vedere figura supplementare 1 per il design del fermenter su misura), collegata a un dispositivo di raffreddamento ricircolante per mantenere la temperatura, con bubbling di CO2 filtrato (2%, v/v) e mescolato con un barra di agitazione magnetica nella parte inferiore del fermentatore con agitazione costante.
- Per avviare la sincronizzazione, impostare il fermenter a regime chiaro-scuro di 12:12 h, sotto 34 ,C e 200 -m-2s-1 luce e assicurarsi che contenga 500 mL di coltura nel mezzo HSM con una densità di celle di 1 x 106 celle/mL.
- Alla fine del ciclo di 24 h, ri-diluire la coltura a 1 x 106 cellule / mL con medio HSM fresco con perturbazione minima utilizzando un sistema di tubi in combinazione con pompe peristaliche, che permette prima di pompare una quantità distinta di coltura fuori e poi pompare in mezzo sterilizzato autoclave.
NOTA: Come alternativa alle pompe peristalitiche, le siringhe inocrative possono essere utilizzate per ritirare la coltura, mentre il volume richiesto di mezzo fresco può essere aggiunto direttamente nel fermenter in condizioni sterili. - Ripetere il passaggio 2.3 tre volte per ottenere le impostazioni cultura sincronizzate ilquarto giorno di inoculazione.
- Per convalidare la sincronizzazione delle cellule, raccogliere campioni a intervalli di ogni 2 h e misurare la densità delle celle e il volume delle celle utilizzando un contatore cellulare e un analizzatore di dimensioni (vedere Tabella dei materiali). A seconda della densità cellulare, diluire i campioni tra 1:10 e 1:100 e misurare in un intervallo di dimensioni di 30 – 1900 m3.
3. Raccogliere le cellule Chlamydomonas
- Etichettare 15 mL tubi centrifugati conici e preparare una dewar piena di azoto liquido.
- Raccogliere campioni utilizzando una siringa (50 cm3) attraverso il tubo di vetro interno del fermenter. Distribuire il campione in tubi di centrifuga conici che devono contenere 10-15 x 106 celle. Si noti il volume trasferito a ciascun tubo e misurare la densità delle celle e le dimensioni delle celle di ogni punto temporale utilizzando il contatore delle celle e l'analizzatore di dimensioni (vedere Tabella dei materiali)
- Pellet le cellule per centrifugazione per 5 min a 4000 x g. Quindi scartare il supernatante, congelare i pellet in azoto liquido e poi conservarli a -80 gradi centigradi.
4. Preparazione di buffer di estrazione e clorofilla di estrazione, lipidi e metaboliti
AVVISO: Il metanolo (MeOH) e MTBE sono infiammabili e possono causare irritazione delle vie respiratorie, degli occhi o della pelle a causa dell'esposizione prolungata e/o del contatto. Si prega di maneggiarli con cura solo in un cofano fume e utilizzare le procedure di sicurezza appropriate durante l'estrazione (lab coat, occhiali di sicurezza, guanti, ecc.).
-
Buffer di estrazione 1
- In un flaasso volumetrico da 100 ml, aggiungere 75 mL di MTBE, 25 mL di MeOH (grado UHPLC) e omogeneizzare (3:1, vol/vol).
- Aggiungere la seguente soluzione per gli standard interni: 50 -L di corticosterone (1 mg/mL in MeOH), 50 -L 1,2-diheptadecanoyl-SN-glycero-3-phos focolina (17:0 PC) (1 mg/mL in grado cloroformio HPLC), 25 - L di anch'io (1 mg/mL in acqua grado UHPLC) e 50 D-Sorbitol-1-13C (1 mg/mL in acqua di grado UHPLC).
- Trasferire il buffer di estrazione in una bottiglia di vetro pulito e pre-raffreddare la soluzione a -20 , 1 h prima dell'uso. Utilizzare una soluzione appena preparata per le estrazioni. Questa soluzione può essere conservata a 4 gradi centigradi per un massimo di 2 settimane.
-
Buffer di estrazione 2
- In un flacone volumetrico da 100 ml, aggiungere 75 mL di acqua di grado UHPLC e 25 mL di grado MeOH UHPLC e omogeneizzare.
- Trasferire il buffer di estrazione in una bottiglia di vetro pulita. Utilizzare una soluzione fresca per le estrazioni. Questa soluzione può essere conservata a temperatura ambiente per diverse settimane.
5. Estrazione di clorofilla, lipidi e metaboliti
- Disporre i tubi, con il pellet cellulare (contenente 10-15 x 106 cellule), in azoto liquido.
- Risospendere il pellet cellulare in ogni tubo con 1 mL di buffer di estrazione pre-raffreddato (-20 gradi centigradi) 1.
NOTA: Eseguire questo passaggio rapidamente per evitare l'evaporazione del buffer di estrazione della bassa viscosità. Dopo aver aggiunto il buffer di estrazione 1, mantenere i tubi a temperatura ambiente. - Vortice fino a quando le cellule sono ben omogeneizzati all'interno della miscela di estrazione e aliquota la miscela in 2 mL tubo di microcentrifuga.
- Sonicare le culture utilizzando il bagno di sonicazione (vedi Tabella dei materiali) in acqua ghiacciata raffreddata per 10 min.
- Incubare tutti i campioni su uno shaker orbitale a 1000 giri/mm per 60 min a 4 gradi centigradi.
- Per indurre la separazione di fase, aggiungere 650 l di Estrazione Buffer 2.
- Vortex seguì per breve tempo da centrifugazione a 20000 x g per 5 min a 4 gradi centigradi.
NOTA: Dopo questo passaggio, vi è una separazione delle fasi liquide e un pellet solido nella parte inferiore del tubo. Maneggiare i tubi con cura per evitare la miscelazione delle due fasi liquide. La fase MTBE superiore contiene lipidi e clorofilla, mentre la fase inferiore contiene i metaboliti polari e semipolari. Il pellet precipitato nella parte inferiore contiene proteine, amido e altre molecole insolubili.
6. Aliquota delle frazioni
- Trasferire 500 l- di fase MTBE superiore (lipidi) in un tubo etichettato da 1,5 mL. Asciugare i campioni utilizzando un concentratore a vuoto (vedere Tabella deimateriali) e conservarli a -80 gradi fino alla misurazione.
- Rimuovere la restante fase MTBE utilizzando una pipetta da 200.L.
- Trasferire 650 -L della fase inferiore (metaboliti polari e semipolari) in un nuovo tubo etichettato. Asciugare i campioni utilizzando un concentratore a vuoto (vedere Tabella deimateriali) e conservarli a -80 gradi fino alla misurazione.
- Rimuovere la fase inferiore rimanente rimuovendo il volume in eccesso.
- Congelare il pellet solido in azoto liquido e conservarlo a -80 gradi centigradi fino a un'ulteriore estrazione.
7. Determinazione dei metaboliti polari (metaboliti primari)
- Risospendere il pellet essiccato della fase polare (passaggio 6.3) nella soluzione methoxyamine-hydrochloride/pyridine per la methoxymization dei gruppi carbonili.
- Riscaldare i campioni a 37 gradi centigradi per 90 min.
- Derivare i campioni con N-metil-N-trimethylsilyltrifloracetamide (MSTFA) per 30 min a 37 gradi centigradi come descritto in precedenza13.
- Utilizzare la cromatografia a gas accoppiata alla spettrometria di massa a tempo di volo (GC-TOF-MS) per analizzare i metaboliti primari. I parametri di gradiente utilizzati erano in base al protocollo14descritto in precedenza.
8. Determinazione dei metaboliti non polari (Lipidi)
- Sospendere nuovamente il pellet essiccato della fase non polare (passaggio 6.1) in una miscela di acetonitrile:isopropanolo (7:3, v:v).
- Centrifuga a 20000 x g e separarla su una colonna C8 in fase inversa (100 mm x 1,1 mm e 1,7 m di particelle), utilizzando un sistema UPLC (vedere Tabella dei materiali). Le due fasi mobili erano acqua con acetato di ammonio di 1% 1 M, 0,1% acido acetico (Buffer A) e acetonitrile:isopropanolo (7:3) contenente 1% 1 M di acetato di ammonio, 0,1% acido acetico (Buffer B).
- Utilizzare i parametri della sfumatura in base al protocollo14descritto in precedenza.
9. Determinazione del contenuto di clorofilla
- Mescolare 100 l della fase MTBE (passaggio 6.1) con 900 l un'l di 90% di metanolo per un metodo vuoto e i campioni sperimentali.
- Misurare l'assorbimento utilizzando lo spettrofotometro a una lunghezza d'onda di 665 nm e 652 nm per distinguere tra clorofilla a e clorofilla b.
NOTA: l'assorbimento deve essere compreso tra 0,1 e 1 per un calcolo della concentrazione valido. Diluire i campioni, se necessario. - Calcolare il contenuto di clorofilla e b, e anche il contenuto totale di clorofilla secondo le seguenti formule.
Chla 16.82A665 – 9.28A652
Chlb - 36.92A652 – 16.54A665
Chla-b - 0,28A665 - 27,64A652
NOTA: le formule sono state descritte da Bar-Nun e Ohad15.
10. Estrazione e determinazione del contenuto proteico, digestione e analisi
NOTA: Per risospendere la proteina, è stato utilizzato il tampone pellet urea/thiourea (6 M urea, 2 M di titusi e astisia) con modifiche nel protocollo descritto in precedenza16. Tuttavia, qualsiasi buffer di scelta può essere utilizzato per la sospensione delle proteine.
- Preparare il tampone proteico utilizzando le seguenti concentrazioni: 6 M di urea e 2 M di tiourea.
- Sciogliere il pellet (passo 6.5) in 200 litri di tampone proteico (10,1).
- Incubare i campioni a temperatura ambiente per 30 min, seguita da centrifugazione a 20000 x g per 5 min.
- Trasferire il supernatante, che contiene le proteine, in un nuovo tubo.
- Determinare la concentrazione proteica dal saggio Bradford17.
- Digerire 50 g di proteine in soluzione con un protocollo di scelta. In questo caso, utilizzare il seguente protocollo.
- Riduci i 50 g di campioni di proteine usando 5 mM DTT per 30 min seguiti da un'alchilazione usando iodoacetamide di 10 m per 30 min a temperatura ambiente al buio.
- Aggiungere Trypsin/Lys-C Mix a un rapporto proteina:proteasi 25:1 (w/w), mescolare e incubare per 3 h a 37 .
- Diluire i campioni di sei pieghe utilizzando 50 mM TrisHCl (pH 8) e Incubare durante la notte a 37 .
- Terminare la digestione aggiungendo acido trifluoroacetico (TFA) ad una concentrazione finale dello 0,5-1%.
- Dopo la digestione, eseguire la dissalazione dei peptidi prima della spettrometria di massa utilizzando le punte dello stadio C18 ed eluire i peptidi digeriti18.
- Concentrare i campioni in prossimità della secchezza, lasciando 2-5 l di soluzione in un concentratore a vuoto (vedi Tabella dei materiali) senza riscaldamento.
- Risospendere i campioni nel buffer di carico (5% acetonitrile, 0,5% di acido formico) e analizzare le miscele di peptidi da LC-MS/MS utilizzando uno spettrometro di massa ad alta risoluzione (vedi Tabella dei materiali) collegato a un sistema nano-UPLC.
- Separare i peptidi utilizzando il sistema UPLC (vedere Tabella deimateriali) su una colonna ibrida di superficie carica in fase inversa di 20 cm (c'è la tabella deimateriali) con un diametro interno di 75 m e una dimensione delle particelle di 1,7 m.
- Caricare 4 l di campione e passare attraverso 90 min gradiente ad una portata di 300 nL/min.
- Impostare il gradiente. Utilizzare impostazioni parziali loop-offline con gradiente isocratico impostato al 3% del buffer B (99,9% acetonitrile - 0,1% di acido formica) tenuto per 14 min prima che il loop venga spostato in posizione online con la colonna, successivamente il gradiente viene aumentato linearmente per 50 min, fino al 20% viene raggiunto il buffer B.
- Entro i prossimi 15 min, aumentare la concentrazione di buffer B al 30%. Poi, nei seguenti 15 min, aumentare il buffer B al 40%, prima di raggiungere il 90% del Buffer B dopo un altro 4 min. Eseguire la fase di lavaggio, che è necessario per pulire la colonna, a 400 nL/min e tenere premuto per ulteriori 10 min (vedere tabella 1 per il gradiente dettagliato condizioni) e condizioni di vita
- Infine, riportare il sistema a una portata di 300 nL/min e una concentrazione del 3% tampone B entro 1 min. Equilibrate la colonna per 15 min prima che il campione successivo venga iniettato.
NOTA: Un elenco completo delle proteine identificate dopo l'analisi LCMS/MS è stato presentato nella tabella 2.
11. Estrazione e determinazione dei contenuti di amido
NOTA: Per la determinazione del contenuto totale di proteine e amido, il pellet solido è stato estratto in una procedura in due fasi come descritto in precedenza10.
- Aggiungere 500 lun dell'80% (v/v) di etanolo al pellet cellulare (passaggio 6,5) e incubare per 10 min a 80 gradi centigradi.
- Centrifuga a 4000 x g per 10 min a temperatura ambiente. Sciogliere quindi il pellet in 250 o L di acqua sterile seguita dall'aggiunta di 250 -L di acetato di sodio da 100 mM.
- Idrolizzare l'amido riscaldando per 3 h a 99 gradi centigradi. Digerire l'amido disciolto nei monomeri del glucosio durante la notte aggiungendo un mix enzimatico di anomilasi (4,2 unità per campione) e amiloglucosidasi (10 unità per campione). Incubare i tubi a 37 gradi durante la notte.
NOTA: Dopo l'incubazione, i campioni possono essere congelati a -20 gradi centigradi per alcuni giorni, o a -80 gradi centigradi per diversi mesi, prima delle misurazioni del glucosio. - Centrifugare l'estratto digerito a 20000 x g per 5 min e raccogliere il supernatante. Sciogliere il soldatore (con i monomeri di glucosio derivati dall'amido) in 100 mM di HEPES-buffer (pH 7) che contiene 5 mM MgCl2, 60 mg/mL ATP, 36 mg/mL NADP e glucose-6-fosfato-dehydrogenasi (1 unità per campione).
- Per analizzare il contenuto dell'amido, misurare innanzitutto l'assorbimento di base a 340 nm. Quindi aggiungere l'esaokinasi (1 unità per campione) per avviare la reazione. Infine, misurare l'aumento di NADPH-H (che indica il livello di amido digerito al glucosio) a 340 nm con un lettore di lastre di 96 pozze.
NOTA: La differenza del valore massimo a 340 nm (non deve superare l'intervallo lineare di 1) e la linea di base è equivalente alla concentrazione di glucosio dell'amido digerito. Una curva di glucosio dovrebbe essere fatta per determinare la concentrazione di glucosio in nmol di glucosio per mL.
Risultati
Chlamydomonas reinhardtii Sincronizzazione delle impostazioni cultura CC-1690
Per dimostrare i risultati rappresentativi per il protocollo specificato, presentiamo l'esempio di dati multiomici ottenuti dopo la raccolta e l'estrazione di campioni dalle culture Chlamydomonas reinhardtii sincronizzate10. Le colture sincronizzate di Chlamydomonas comprendono cellule appartenenti a una fase di crescita uniforme in un determinato momento. Le colture di Chlamydomonas sono state sincronizzate a un ciclo di luce/buio di 12 h/12h, 34 c con l'intensità della luce di 200 s-1 e la concentrazione di CO2 del 2%, v/v, descritta come concentrazione ottimale per lo sforzo CC-1690 mt . Queste condizioni erano state precedentemente ottimizzate e convalidate utilizzando vari parametri del ciclo cellulare10. Figura 1 mostra la distribuzione delle dimensioni delle celle misurata con Coulter Counter in punti temporali distinti di impostazioni cultura sincronizzate. Uno spostamento del volume cellulare può essere osservato man mano che le cellule crescono di dimensioni durante la fase di luce, seguite dal rilascio di cellule figlie a partire dalla fine della fase di luce da 10 h. Una volta rilasciate tutte le cellule figlie, è possibile osservare lo spostamento del volume cellulare man mano che le cellule figlie appena rilasciate vengono disposte per iniziare il ciclo successivo 10 (Figura 1).
Campionamento, -manipolazione ed estrazione
La raccolta rapida dei campioni viene effettuata utilizzando la centrifugazione e dopo aver scartato il supernatante, i pellet possono essere conservati a -80 gradi centigradi fino all'estrazione. Come descritto in precedenza (passaggio 5), l'estrazione di MTBE si traduce in tre fasi distinte: a) la fase organica è stata utilizzata per misurare i lipidi e i livelli di clorofilla (fattore di normalizzazione), b) fase polare è stata raccolta per misurare i metaboliti sul GCMS mentre, c) è stato utilizzato il pellet per misurare il contenuto di amido e le proteine. Una panoramica della distribuzione delle diverse fasi e della loro occupazione è illustrata nella figura 2.
Metaboliti polari e non polari
Sulla base dell'analisi GCMS della frazione polare, sono stati annotati 65 metaboliti, che coprono aminoacidi, acidi nucleici, intermedi di glicolisi, glucogenesi, ciclo di acido tricarboxylico, percorso del fosfato pentoso e poliamine (Figura 3A). L'analisi LCMS della fase neutra contenente lipidi ha portato all'identificazione di 204 specie di lipidi distinti che coprono varie classi di lipidi, vale a dire fosfatidilglyceroli, fosfatidylethanolamina, sulfoquinovosil diacylglycerols, monogalactosyldiacylglycerols, digalactosyldiacylglycerols, diacylglyceryltrimethylhomoserine, acidi grassi, diacylglycerides e triacylglycerides. Per visualizzare i cambiamenti globali nei metaboliti e nei lipidi nel ciclo cellulare, è stata utilizzata l'analisi dei componenti principali (PCA). L'IPA mostra una separazione delle fasi chiare e scure sia per la metabolomica che per i dati lipidomici. Inoltre, un cerchio semi-ciclico (parzialmente aperto) può essere notato per entrambi i dati (Figura3C, D). Il divario parziale nel modello circolare è attribuito al fatto che i campioni a 24 h del ciclo cellulare sono stati raccolti sotto il buio in contrasto con i campioni raccolti all'inizio del ciclo cellulare dopo 0,25 h di esposizione alla luce (Figura3C ,D).
Analisi delle proteine e dell'amido
Per esaminare la qualità del pellet proteico ottenuto a seguito dell'estrazione MTBE, sono stati utilizzati 6 campioni per l'analisi proteomica. La qualità dei dati proteomici ottenuti dissuadendo 50 proteine/campioni, è stata esaminata utilizzando uno strumento di controllo della qualità computazionale -Proteomics quality control (PTXQC)19, indicando dati riproducibili e di alta qualità dei dati proteomici ottenuti da tutte le repliche (Figura supplementare 1). La copertura funzionale molecolare delle proteine è stata esaminata utilizzando REVIGO20. Una panoramica dell'arricchimento funzionale delle 2463 proteine identificate (cfr. tabella 2), è presentata nella Figura 4A. Il pellet rimanente dopo l'estrazione delle proteine è stato utilizzato per la quantificazione riproducibile dell'amido, come indicato da una bassa deviazione standard tra varie repliche (Figura 4B).

Figura 1: Esempio illustrativo delle variazioni del volume cellulare nelle diverse fasi del ciclo cellulare in Chlamydomonas reinhardtii. Asse x che rappresenta il volume della cella mentre l'asse y rappresenta il numero di cella. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: flusso di lavoro illustrato per l'impiego di diverse fasi durante pellet cellulari di estrazione multi-omica. La figura è stata riutilizzata da Juppner, J.et al. 10. Fare clic qui per visualizzare una versione più grande di questa figura.
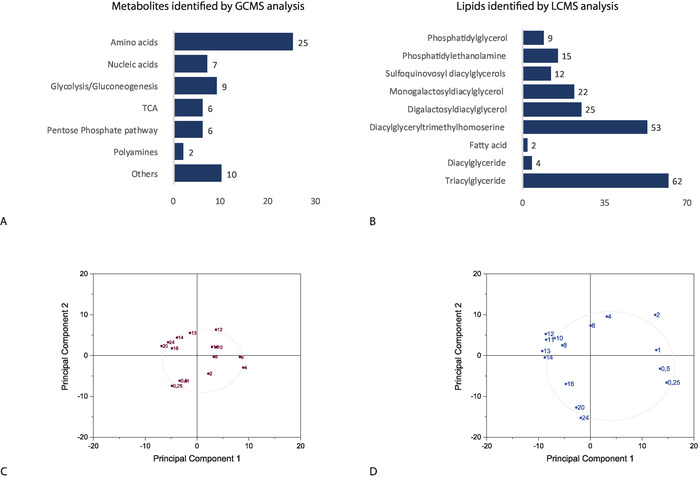
Figura 3: Esempio rappresentativo di metaboliti e lipidi identificati utilizzando il protocollo descritto. (A) Classi di metaboliti identificate dall'analisi GCMS. (B) Specie di lipidi appartenenti a diverse classi identificate dall'analisi LCMS. (C) Analisi dei componenti di principio dei livelli di metaboliti in un ciclo cellulare di 24 h. (D) Analisi del componente di principio dei lipidi su un ciclo cellulare di 24 h. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Esempio rappresentativo dei dati relativi alle proteine e all'amido. A) Arricchimento funzionale molecolare delle proteine identificate mediante l'analisi LCMS, mappa ad albero disegnata utilizzando dati di amido rappresentativi REVIGO 20 B che mostrano la riproducibilità del protocollo. Fare clic qui per visualizzare una versione più grande di questa figura.
Supplementare Figura 1: Progettazione personalizzata del sistema di fermenter per la temperatura e l'aerazione controllava la crescita sincrona delle culture Chlamydomonas. La figura è stata riutilizzata da Juppner, J.et al. 10. Fare clic qui per scaricare questo file.
Figura supplementare 2: Risultato rappresentativo per la qualità dei dati proteomica. Mappa di calore tracciata con lo strumento PTXQC computazionale19. Fare clic qui per scaricare questo file.
| Tempo (min) | % Buffer B al buffer A | |
| Da 0 a 15 min | Sfumatura lineare da 0 a 3% | Buffer A: 0,1% di acido formio nell'acqua di grado UPLC |
| Da 15 a 75 min | Sfumatura lineare dal 3% al 30% | Buffer B: 0,1% acido formico nel 60% acetonitrile di grado UPLC |
| Da 75 a 90 min | Sfumatura lineare dal 30% al 40% | Velocità di flusso 300 nL/min |
| Da 90 a 94 min | Sfumatura lineare dal 40% al 90% | Volume di iniezione 4 - L |
| Da 94 a104 min | colonna di lavaggio con 90% | |
| Da 105 a 120 min | Equilibrate la colonna per 15 min al 3% |
Tabella 1: cromatografia liquida di campioni di peptidi, parametri di gradiente.
Tabella 2: Elenco delle proteine identificate dopo l'analisi LCMS/MS. Fare clic qui per scaricare questo file.
Discussione
In questo articolo, abbiamo illustrato un protocollo di estrazione robusto e altamente applicabile per linfomiche complete, metabolomica, amido e analisi proteomica da un singolo pellet di 10-15 x 106 celle. Il metodo è stato implementato con successo in diversi studi per una vasta gamma di cellule e tessuti10,14,21,22,23,24,25 ,26,27,28,29,30,31,32,33,34 ,35,36. Qui, abbiamo presentato una pipeline graduale per l'analisi multiomica di diverse biomolecole da un singolo campione raccolto dalla coltura Chlamydomonas reinhardtii (CC-1690 mt.
Il protocollo fornisce un approccio robusto e riproducibile per elaborare più campioni contemporaneamente, per l'analisi di varie biomolecole. Tuttavia, una serie di misure critiche dovrebbe essere preso cura al fine di ridurre al minimo la variazione tecnica. In primo luogo, la raccolta delle cellule dovrebbe essere fatta il più rapidamente possibile, pur mantenendo le condizioni uniformi per tutti i campioni raccolti per preservare lo stato biologico delle cellule. Anche se abbiamo usato la centrifugazione per raccogliere le cellule, strategie di raccolta alternative possono essere utilizzate per la raccolta dei campioni. Tuttavia, è importante notare che diverse strategie di raccolta sono noti per influenzare lo stato metabolico delle cellule37 quindi, approccio di raccolta coerente deve essere utilizzato per tutti i campioni sperimentali. In secondo luogo, è importante evitare l'essiccazione della fase superiore non polare contenente clorofilla, poiché ciò può influenzare i livelli di clorofilla disciolta nel solvente che influisce sul fattore di normalizzazione per i campioni. Infine, occorre prestare attenzione durante la rimozione della fase polare rimanente per ottenere la proteina e il pellet di amido, per evitare di disturbare il pellet che può influenzare il contenuto di amido e proteine.
In questo modo presentato il protocollo di estrazione offre diversi vantaggi per l'analisi dei dati multi-omica. Oltre a ridurre al minimo il numero di campioni necessari, riduce anche la variazione tra i risultati analitici ottenuti per diverse biomolecole. Ciò consente il confronto diretto dei risultati ottenuti dai metaboliti primari, dai lipidi e dai dati proteomici. Analogamente, l'estrazione simultanea di più classi composte consente una strategia di normalizzazione coerente e uniforme dei diversi set di dati. Ciò è particolarmente applicabile se la normalizzazione è difficile da ottenere utilizzando peso secco o fresco38 o numero di cellulare.
Il protocollo può essere implementato per lo screening di routine di un campione biologico complesso. Questi set di dati metabolomici, lipimmici e proteomici olistici possono offrire informazioni complete sui cambiamenti sistematici nel metabolismo. Inoltre, i dati ottenuti dall'analisi proteomica, forniscono informazioni sui cambiamenti quantitativi (abbondanza) e qualitativi (modifiche) nelle proteine in relazione ai metaboliti. Di conseguenza, l'integrazione dei dati omici potrebbe rivelare informazioni approfondite sui cambiamenti indotti dalle perturbazioni genetiche o biotiche e/o abiotiche di un sistema biologico. Così, chiarire i cambiamenti molecolari di specifiche vie metaboliche o processi cellulari. Allo stesso modo, questi dati ad alto rendimento possono consentire l'identificazione di obiettivi per l'ingegneria metabolica e perfezionare o testare le previsioni da modelli metabolici su scala genoma37,39.
Divulgazioni
Gli autori non hanno divulgazioni.
Riconoscimenti
Siamo molto grati a Gudrun Wolter e a Nne Michaelis per un'eccellente assistenza tecnica. Ringraziamo tutti i membri del laboratorio Giavalisco per il loro aiuto. Siamo grati alla Max Planck Society per aver finanziato la ricerca e FAPESP per la borsa di studio di L A Giraldi
Materiali
| Name | Company | Catalog Number | Comments |
| Reagents and standards | |||
| 1,2-diheptadecanoyl-sn-glycero-3-phosphocholine (17:0 PC) | Avanti Polar Lipids | 850360P | Internal standard for lipids |
| 13C Sorbitol | Sigma Aldrich | 605514 | Internal standard for metabolites, ISOTEC® Stable Isotopes |
| Ampicillin | Sigma Aldrich | A9393-5G | Internal standard for metabolites |
| Corticosterone | Sigma Aldrich | 27840-500MG | Internal standard for metabolites, HPLC grade |
| Methanol (MeOH) | Biosolve Chemicals | 13684102 | ULC-MS grade |
| Methyl tert-butyl ether (MTBE) | Biosolve Chemicals | 13890602 | HPLC grade |
| Trypsin/Lys-C mix | Promega | V5072 | Enzymatic digestion of proteins |
| Water | Biosolve Chemicals | 23214102 | ULC-MS grade |
| Equipment | |||
| 1.5 ml Safe-lock microcentrifuge tubes | Eppendorf | 30120086 | Used for fractions |
| 2 ml Safe-lock microcentrifuge tubes | Eppendorf | 30120094 | Used for sample extarction |
| Balance | Sartorius Corporation | 14 557 572 | |
| Fermenter system | Glasbläserei Müller, Berlin, Germany | custom made fermenter of 800ml capacity | |
| Q-exactive HF Orbitrap Mass Spectrometer | Thermo Scientific | IQLAAEGAAPFALGMBFZ | |
| Refrigerated microcentrifuge | Eppendorf, model 5427R | 22620701 | |
| Reversed Phase (RP) Bridged Ethyl Hybrid (BEH) C8 column (100 mm×2.1 mm containing 1.7 μm diameter particles) | Waters, Machester, UK | 186002878 | Analysis of lipids |
| RP Charged Surface Hybrid (CSH) column (Waters) with an inner diameter of 75 μm and a particle size of 1.7 μm | Waters, Machester, UK | 186007477 | Analysis of proteins |
| RP High Strength Silica (HSS) T3 column (100 mm×2.1 mm containing 1.8 μm diameter particles) | Waters, Machester, UK | 186003539 | Analysis of metabolites |
| Shaker | Eppendorf Thermomixer 5436 | 2050-100-05 | |
| Sonicator | USC 300 TH | 142-0084 | standard preset sonication power at 45kHz |
| UPLC system | Waters Acquity UPLC system (Waters, Machester, UK) | ||
| Vacuum concentrator | Scan Speed Maxi Vac Alpha Evaporators | 7.008.500.002 | |
| Vortex mixer | Vortex-Genie 2, Model G560 | SI-0236 | |
| Z2 Coulter Particle Count and Size Analyzer | Beckman Coulter | 6605700 | particle (cells) volume and number analyser |
Riferimenti
- Spolaore, P., Joannis-Cassan, C., Duran, E., Isambert, A. Commercial applications of microalgae. Journal of Bioscience and Bioengineering. 101 (2), 87-96 (2006).
- Georgianna, D. R., Mayfield, S. P. Exploiting diversity and synthetic biology for the production of algal biofuels. Nature. 488 (7411), 329-335 (2012).
- Pulz, O., Gross, W. Valuable products from biotechnology of microalgae. Applied Microbiology and Biotechnology. 65 (6), 635-648 (2004).
- Perez-Garcia, O., Escalante, F. M., de-Bashan, L. E., Bashan, Y. Heterotrophic cultures of microalgae: metabolism and potential products. Water Research. 45 (1), 11-36 (2011).
- Mukhopadhyay, A., Redding, A. M., Rutherford, B. J., Keasling, J. D. Importance of systems biology in engineering microbes for biofuel production. Current Opinion in Biotechnology. 19 (3), 228-234 (2008).
- Chang, R. L., et al. Metabolic network reconstruction of Chlamydomonas offers insight into light-driven algal metabolism. Molecular Systems Biology. 7, 518 (2011).
- Dal'Molin, C. G., Quek, L. E., Palfreyman, R. W., Nielsen, L. K. AlgaGEM--a genome-scale metabolic reconstruction of algae based on the Chlamydomonas reinhardtii genome. Bmc Genomics. 12 Suppl 4, S5 (2011).
- Zones, J. M., Blaby, I. K., Merchant, S. S., Umen, J. G. High-Resolution Profiling of a Synchronized Diurnal Transcriptome from Chlamydomonas reinhardtii Reveals Continuous Cell and Metabolic Differentiation. Plant Cell. 27 (10), 2743-2769 (2015).
- Willamme, R., et al. Metabolomic analysis of the green microalga Chlamydomonas reinhardtii cultivated under day/night conditions. Journal of Biotechnology. 215, 20-26 (2015).
- Juppner, J., et al. Dynamics of lipids and metabolites during the cell cycle of Chlamydomonas reinhardtii. The Plant Journal. 92 (2), 331-343 (2017).
- Salem, M. A., Juppner, J., Bajdzienko, K., Giavalisco, P. Protocol: a fast, comprehensive and reproducible one-step extraction method for the rapid preparation of polar and semi-polar metabolites, lipids, proteins, starch and cell wall polymers from a single sample. Plant Methods. 12, 45 (2016).
- Harris, E. H., Harris, E. H. . The Chlamydomonas Sourcebook. 1, 248-249 (2009).
- Roessner, U., Wagner, C., Kopka, J., Trethewey, R. N., Willmitzer, L. Technical advance: simultaneous analysis of metabolites in potato tuber by gas chromatography-mass spectrometry. The Plant Journal. 23 (1), 131-142 (2000).
- Salem, M., Bernach, M., Bajdzienko, K., Giavalisco, P. A Simple Fractionated Extraction Method for the Comprehensive Analysis of Metabolites, Lipids, and Proteins from a Single Sample. Journal of Visualized Experiments. (124), (2017).
- Bar-Nun, S., Ohad, I. [34] Chloroplast membrane polypeptides. Methods in Enzymology. 69, 363-374 (1980).
- Giavalisco, P., et al. Proteome analysis of Arabidopsis thaliana by two-dimensional gel electrophoresis and matrix-assisted laser desorption/ionisation-time of flight mass spectrometry. Proteomics. 5 (7), 1902-1913 (2005).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Bittremieux, W., Valkenborg, D., Martens, L., Laukens, K. Computational quality control tools for mass spectrometry proteomics. Proteomics. 17 (3-4), 1600159 (2017).
- Supek, F., Bosnjak, M., Skunca, N., Smuc, T. REVIGO summarizes and visualizes long lists of gene ontology terms. PloS one. 6 (7), e21800 (2011).
- Hauck, O. K., et al. Uric acid accumulation in an Arabidopsis urate oxidase mutant impairs seedling establishment by blocking peroxisome maintenance. Plant Cell. 26 (7), 3090-3100 (2014).
- Caldana, C., et al. Systemic analysis of inducible target of rapamycin mutants reveal a general metabolic switch controlling growth in Arabidopsis thaliana. The Plant Journal. 73 (6), 897-909 (2013).
- Avin-Wittenberg, T., et al. Global analysis of the role of autophagy in cellular metabolism and energy homeostasis in Arabidopsis seedlings under carbon starvation. Plant Cell. 27 (2), 306-322 (2015).
- Hummel, J., et al. Ultra performance liquid chromatography and high resolution mass spectrometry for the analysis of plant lipids. Frontiers in Plant Science. 2, 54 (2011).
- Giavalisco, P., et al. Elemental formula annotation of polar and lipophilic metabolites using (13) C, (15) N and (34) S isotope labelling, in combination with high-resolution mass spectrometry. The Plant Journal. 68 (2), 364-376 (2011).
- Watanabe, M., et al. Comprehensive dissection of spatiotemporal metabolic shifts in primary, secondary, and lipid metabolism during developmental senescence in Arabidopsis. Plant Physiology. 162 (3), 1290-1310 (2013).
- Lambers, H., et al. Proteaceae from severely phosphorus-impoverished soils extensively replace phospholipids with galactolipids and sulfolipids during leaf development to achieve a high photosynthetic phosphorus-use-efficiency. New Phytologist. 196 (4), 1098-1108 (2012).
- Degenkolbe, T., et al. Differential remodeling of the lipidome during cold acclimation in natural accessions of Arabidopsis thaliana. The Plant Journal. 72 (6), 972-982 (2012).
- Bromke, M. A., et al. Liquid chromatography high-resolution mass spectrometry for fatty acid profiling. The Plant Journal. 81 (3), 529-536 (2015).
- Li, N., et al. FAX1, a novel membrane protein mediating plastid fatty acid export. PLOS Biology. 13 (2), e1002053 (2015).
- Hemme, D., et al. Systems-wide analysis of acclimation responses to long-term heat stress and recovery in the photosynthetic model organism Chlamydomonas reinhardtii. Plant Cell. 26 (11), 4270-4297 (2014).
- Sharma, D. K., et al. UPLC-MS analysis of Chlamydomonas reinhardtii and Scenedesmus obliquus lipid extracts and their possible metabolic roles. Journal of Applied Phycology. 27 (3), 1149-1159 (2015).
- Delgado, R., Munoz, Y., Pena-Cortes, H., Giavalisco, P., Bacigalupo, J. Diacylglycerol activates the light-dependent channel TRP in the photosensitive microvilli of Drosophila melanogaster photoreceptors. The Journal of Neuroscience. 34 (19), 6679-6686 (2014).
- Bozek, K., et al. Organization and evolution of brain lipidome revealed by large-scale analysis of human, chimpanzee, macaque, and mouse tissues. Neuron. 85 (4), 695-702 (2015).
- Khrameeva, E. E., et al. Neanderthal ancestry drives evolution of lipid catabolism in contemporary Europeans. Nature Communications. 5, 3584 (2014).
- Bozek, K., et al. Exceptional evolutionary divergence of human muscle and brain metabolomes parallels human cognitive and physical uniqueness. PLOS Biology. 12 (5), e1001871 (2014).
- Veyel, D., Erban, A., Fehrle, I., Kopka, J., Schroda, M. Rationales and approaches for studying metabolism in eukaryotic microalgae. Metabolites. 4 (2), 184-217 (2014).
- Allwood, J. W., et al. Inter-laboratory reproducibility of fast gas chromatography-electron impact-time of flight mass spectrometry (GC-EI-TOF/MS) based plant metabolomics. Metabolomics. 5 (4), 479-496 (2009).
- Feist, A. M., Herrgard, M. J., Thiele, I., Reed, J. L., Palsson, B. O. Reconstruction of biochemical networks in microorganisms. Nature Reviews Microbiology. 7 (2), 129-143 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Tutti i diritti riservati
Utilizziamo i cookies per migliorare la tua esperienza sul nostro sito web.
Continuando a utilizzare il nostro sito web o cliccando “Continua”, accetti l'utilizzo dei cookies.