Method Article
Analisi di basati su reazione a catena della polimerasi quantitativa del Microbiota intestinale murina dopo il trattamento antibiotico orale
In questo articolo
Riepilogo
Qui forniamo protocolli dettagliati per la somministrazione orale di antibiotici ai topi, raccolta di campioni di feci, estrazione del DNA e quantificazione di batteri fecali di qPCR.
Abstract
Il microbiota dell'intestino ha una centrale influenza sulla salute umana. Disbiosi microbico sono associato con immunopathologies molti comuni come malattia infiammatoria intestinale, asma e artrite. Così, la comprensione dei meccanismi alla base area interattiva microbiota-difese immunitarie è di importanza cruciale. Somministrazione di antibiotici, mentre aiutava la liquidazione dell'agente patogeno, inoltre induce cambiamenti drastici nella dimensione e nella composizione delle comunità batterica intestinale che possono avere un impatto sulla salute umana. Trattamento antibiotico nei topi ricapitola l'impatto e i cambiamenti a lungo termine nel microbiota umano da pazienti trattati con antibiotici e consente l'indagine dei link meccanicistico tra cambiamenti nelle comunità microbiche e funzione delle cellule immuni. Mentre sono stati descritti diversi metodi per il trattamento antibiotico di topi, alcuni di essi indurre grave disidratazione e perdita di peso che complica l'interpretazione dei dati. Qui, forniamo due protocolli per la somministrazione di antibiotici orale che può essere utilizzata per il trattamento a lungo termine dei topi senza indurre la perdita di peso maggiore. Questi protocolli fanno uso di una combinazione di antibiotici tale destinazione sia Gram-positivi e Gram-negativi batteri e possono essere forniti sia ad libitum nell'acqua potabile o mediante sonda gastrica orale. Inoltre, descriviamo un metodo per la quantificazione della densità microbica in campioni di feci di qPCR che può essere utilizzato per convalidare l'efficacia del trattamento antibiotico. La combinazione di questi approcci fornisce una metodologia affidabile per la manipolazione del microbiota intestinale e lo studio degli effetti del trattamento antibiotico in topi.
Introduzione
La mucosa del tratto gastrointestinale dei mammiferi è un ambiente unico, colonizzato da una miscela molto complessa di microrganismi che stabiliscono una relazione mutualistica con l'host. Il sistema di difesa della mucosa intestinale comprende uno strato epiteliale e una pletora di cellule immunitarie che limitano i commensali all'interno dell'intestino, preservando il loro numero e la diversità. Al contrario, organismi commensali sono necessari per lo sviluppo di un sistema immunitario pienamente funzionale. Mentre le interazioni tra ospite e batteri commensali sono normalmente benefiche, sta diventando sempre più chiaro che dysregulated sistema immunitario-microbiota diafonia può favorire lo sviluppo delle malattie infiammatorie croniche, tali asinflammatory dell'intestino malattia, l'artrite o asma1,2.
Il microbiota dell'intestino può essere alterato da vari fattori, ma forse i più drastici cambiamenti sono indotti dal trattamento antibiotico che altera gravemente la dimensione e la composizione della comunità batterica3,4. Mentre i benefici di antibiotici per trattare le infezioni sono indiscutibili, le modifiche di microbiota indotte tramite l'esposizione agli antibiotici in esseri umani inoltre possono modificare le difese immunitarie, che possono portare a effetti dannosi sulla salute. Per esempio, il trattamento antibiotico in esseri umani è stato collegato ad un rischio aumentato di Clostridium difficile-ha indotto diarrea, asma e alcuni tipi di cancro3. Trattamento antibiotico nei topi ricapitola l'impatto e le alterazioni a lungo termine delle Comunità di intestino di pazienti trattati con antibiotico e ha permesso l'indagine dei link meccanicistico tra cambiamenti nelle comunità microbiche e funzione delle cellule immuni. Tuttavia, parecchi rapporti hanno indicato che la somministrazione di antibiotici per l' acqua potabile ad libitum determina molto notevole perdita di peso come topi astengono dall'acqua potabile, presumibilmente a causa del suo cattivo gusto5,6. Così, in questi modelli la disidratazione severa concomitante alla somministrazione di antibiotici orale può complicare l'interpretazione degli esperimenti con l'obiettivo di identificare l'effetto del trattamento antibiotico in funzione delle cellule immuni.
Diversi approcci possono essere usati per esplorare la dimensione e la composizione delle comunità microbiche in vano intestinale7. Prossima generazione tecnologie di sequenziamento hanno fornito preziosi dati su questa questione8, tuttavia questi metodi sono relativamente costosi e richiedono analisi bioinformatiche esperto per l'interpretazione dei dati. D'altra parte, i metodi di coltura microbiologica tradizionale consentono il rilevamento delle specie batteriche, ma hanno bassa sensibilità e una grande frazione di batteri commensali (specialmente gli anaerobi) sono molto difficili o impossibili da coltivare con metodi attualmente disponibili8. Tecniche di reazione a catena (qPCR) della polimerasi quantitativa vengono utilizzati sempre più per quantificazione e identificazione di specie batteriche fecale, poichè forniscono una misura veloce e affidabile di coltura-indipendente della carica microbica totale. Di conseguenza, qPCR metodi si sono rivelati utili per studiare i cambiamenti nel microbiota associato all'età o alla progressione di parecchie malattie compreso la malattia infiammatoria intestinale9,10. In quest'ottica qPCR metodi forniscono un approccio veloce e conveniente per convalidare l'effetto dei vari trattamenti (compresi gli antibiotici) in carichi batteriche fecale e microbiota composizione10,11,12.
Qui, presentiamo un resoconto dettagliato passo-passo dei due protocolli distinti per somministrazione di antibiotici orale per topi, raccolta del campione fecale, estrazione del DNA, elaborazione di norme e la quantificazione dei batteri nei campioni fecali di qPCR. Questi protocolli forniscono un metodo affidabile per manipolare il microbiota intestinale nei topi e studio degli effetti del trattamento antibiotico in omeostasi intestinale e la malattia.
Protocollo
Esperimenti descritti qui sono stati eseguiti utilizzando 6-8 settimane vecchio selvaggio-tipo (C57BL6/J) topi mantenuti in una struttura (SPF) libera agente patogeno specifico. Tutti gli esperimenti sugli animali sono stati approvati da College Londra del re e l'Animal Welfare Institute di Francis Crick ed etici organo di ricorso e l'Home Office del Regno Unito. Prima di iniziare qualsiasi procedura di animale, assicurarsi che le autorizzazioni appropriate sono ottenute attraverso l'istituzione/organizzazione locale.
1. la somministrazione di antibiotici
Nota: Sono disponibili due metodi alternativi per il trattamento antibiotico: sonda gastrica orale (punto 1.1) e la somministrazione di antibiotici via acqua potabile (punto 1.2).
- Alimentazione mediante sonda gastrica orale
- Preparare scorte di singoli antibiotici da loro dissoluzione in acqua in autoclave alle seguenti concentrazioni: ampicillina 100 mg/ml, gentamicina 100 mg/ml, neomicina 100 mg/ml, metronidazolo a 10 mg/mL e vancomicina 100 mg/ml. Filtro sterilizzare usando un filtro da 0,45 µm. Aliquotare e conservare a-20 ° C.
- Preparare un cocktail di antibiotici con le scorte preparate in precedenza. Per un volume di 1 mL mix 50 µ l di ampicillina (5mg/mL finale), 50 µ l di gentamicina (5 mg/mL finale), 50 µ l di neomicina (5mg/mL finale), 500 µ l di metronidazolo (5mg/mL finale), 25 µ l di vancomicina (2,5 mg/mL finale) e 325 µ l di acqua. Preparare il cocktail fresco prima dell'uso.
- Caricare il mix antibiotico in una siringa sterile da 1 mL e difficoltà un'inezione di sonda gastrica appropriato (20 G per topi di 15 – 20 g), eliminando eventuali bolle.
- Topi di sonda gastrica con 200 µ l di miscela antibiotica.
- Afferrare saldamente la pelle sopra la spalla del mouse, allungare la testa e il collo per raddrizzare l'esofago. Dirigere la punta a sfera dell'alimentazione ago lungo il tetto della bocca e verso la parte posteriore della faringe. Poi, delicatamente passarla nell'esofago e iniettare la soluzione di 200 μL.
- Amministrare una volta cocktail antibiotica al giorno per tutta la durata dell'esperimento.
- Antibiotici nell'acqua potabile
Attenzione: Monitorare attentamente peso del mouse ogni giorno per le prime due settimane di somministrazione di antibiotici nell'acqua potabile.- Preparare un cocktail di antibiotici sciogliendo in 1 L d'acqua in autoclave i seguenti: 1 g di ampicillina (1 g/L finale), 1 g di neomicina (1 g/L finale), 1 g di metronidazolo (1 g/L finale), 0,5 g di vancomicina (0,5 g/L finale) e 8 bustine (0,75 g ciascuno) di artificiale dolcificante (60 g/L finale). Agitare fino a completa dissoluzione. Conservare a 4 ° C.
Nota: Dolcificante viene aggiunto per nascondere il sapore di antibiotici e prevenire la disidratazione di topi. Mentre parecchie marche di dolcificante possono funzionare, la concentrazione finale richiesta può essere diversa per ogni marca specifica. - Riempire il cocktail antibiotico in una bottiglia d'acqua (~ 100 mL/bottiglia) e collocare il flacone su una gabbia del mouse.
Nota: Utilizzare bottiglie marroni o coprire le bottiglie con un foglio per proteggere gli antibiotici dalla luce. - Sostituire il cocktail antibiotico con azione fresche due volte a settimana per tutta la durata dell'esperimento.
- Preparare un cocktail di antibiotici sciogliendo in 1 L d'acqua in autoclave i seguenti: 1 g di ampicillina (1 g/L finale), 1 g di neomicina (1 g/L finale), 1 g di metronidazolo (1 g/L finale), 0,5 g di vancomicina (0,5 g/L finale) e 8 bustine (0,75 g ciascuno) di artificiale dolcificante (60 g/L finale). Agitare fino a completa dissoluzione. Conservare a 4 ° C.
2. raccolta dei campioni di feci da sgabello, contenuto di ileo e parete dell'ileo
- Pesare ed etichettare 2 mL sterilizzato nell'autoclave provette per la raccolta del campione.
- Per la raccolta dei campioni di feci fresche, posizionare ogni mouse in un dispositivo di ritenuta e raccogliere fecale pellet direttamente dall'ano in un tubo di raccolta.
Nota: Possono anche ottenere campioni messa topi in una gabbia pulita in autoclave e raccogliere campioni di feci con il forcipe sterilizzato pulito. - Eutanasia i topi con CO2 asfissia seguita da dislocazione cervicale.
- Posare una carcassa di topo con l'addome completamente esposto e spruzzare la zona addominale con etanolo al 70%.
- Con delle forbici, pinze sterili, fare un'incisione trasversale nell'addome per esporre il peritoneo senza danneggiare qualsiasi tessuti interni. Sollevare il peritoneo e fare un'incisione per esporre gli intestini.
- Rimuovere gli intestini (dal colon per stomaco) con le pinze e le forbici e metterlo in una capsula di Petri sterile.
- Utilizzare con attenzione forcipe per stuzzicare l'intestino tenue (SI) dal arterie mesenteriche e grasso. Estendere l'intestino e collocarlo su un panno pulito lab.
- Con un righello, misurare e tagliare 4 cm dell'ileo distale del SI (parte più vicina al cieco). Tagliare ed eliminare il 1 cm dell'intestino prossimale al cieco. Ci sarà una parte di 3 cm dell'ileo che verrà utilizzato per raccogliere i batteri intestinali a sinistra.
- Tenere la parte di ileo (3 cm) sopra un tubo sterile mL 2. Raccogliere il contenuto intestinale estrudendo direttamente l'intestino e colleting il campione nel tubo. In questo esempio avrà batteri dal contenuto dell'ileo.
- Preparare una siringa da 20 mL con soluzione salina fredda tampone fosfato (PBS) e lavare la parte di ileo (scartare il flusso continuo).
- Posizionare la porzione di ileo su un tovagliolo di carta pulito e aperto longitudinalmente con le forbici.
- Raschiare l'interno della parete dell'ileo con un bisturi.
- Raccogliere tutti i batteri il bisturi lavando il bisturi con 1 mL di PBS sopra una provetta pulita. Rotazione a 8.000 × g per 5 min a pellet i batteri e scartare il surnatante. Questo esempio contiene batteri dalla parete dell'ileo.
Nota: I campioni fecali da sgabello, contenuto di ileo e parete possono essere congelati e conservati a-80 ° C fino all'utilizzo. - Pesare le provette contenenti i campioni dal contenuto sgabello e ileo e sottrarre il peso dai tubi a vuoti (dal punto 2.1) per ottenere il peso fecale in ciascun campione.
- Estrarre DNA batterico da sgabello, contenuto di ileo e ileo parete campioni usando i kit disponibili in commercio. Conservare i campioni di DNA a-20 ° C fino all'utilizzo.
3. quantificazione del Microbiota intestinale di qPCR
Nota: Questa procedura include la generazione di standard (punto 3.1) e il metodo per qPCR set-up per i campioni standard e fecali (punto 3.2)
- Generazione di uno standard per qPCR
- Reazione a catena della polimerasi (PCR) consente di amplificare il gene del rRNA 16S dal DNA genomico Estratto da una coltura batterica usando i reagenti13 e le condizioni PCR indicate nella tabella 1.
- Eseguire il prodotto PCR su un gel di agarosio 1.5% e purificare la band di DNA utilizzando un kit di estrazione del DNA commercialmente disponibile.
- Legare il purificato DNA frammento utilizzando una clonazione kit (vedere la Tabella materiali, utilizzare un vettore contenente marcatori di resistenza agli antibiotici e la fusione del gene della β-galattosidasi (LacZ) per la selezione di Colonia blu/bianco) e trasformare DH10 competente Escherichia coli seguendo le istruzioni del produttore. Piastra di trasformazione sul ampicillina (100 µ g/mL), piastre di agar Luria Bertani (LB) (20 µ g/mL) X-gal (10g/L triptone, 5g/L estratto di lievito, 10g/L NaCl, Agar 15 g/L). Incubare per una notte (O/N) a 37 ° C.
- Selezionare una colonia di singolo positivi (bianco) dalla piastra. Inoculare e in 5 mL di brodo LB contenente 100 µ g/mL ampicillina e O/N di incubare a 37 ° C, agitando a 250 giri/min.
- Dalla cultura O/N, isolare e purificare il plasmide utilizzando un kit commerciale miniprep secondo le istruzioni del produttore.
Nota: È importante sequenziare l'inserto del plasmide in questa fase, per garantire che il plasmide contiene solo una copia del gene del rRNA 16S e determinare la sua lunghezza in coppie di basi (bp). - Linearizzare il plasmide con un enzima di restrizione che taglia il plasmide una sola volta.
- Purificare il plasmide linearizzato utilizzando un kit disponibile in commercio.
- Determinare la concentrazione del plasmide misurando l'assorbanza a 260 nm utilizzando uno spettrofotometro.
- Calcolare il numero di copie di plasmide / µ l di campione utilizzando la seguente formula14:
numero di copie = (quantità * 6.022 × 1023) / (lunghezza * 1 × 109 * 650)
cui importo è la concentrazione di DNA ottenuta al passaggio 3.1.8 (in ng / µ l); e la lunghezza è la lunghezza totale del plasmide (con inserto) in bp. Il numero di copie saranno ottenuti come copie / µ l.
Nota: Ci sono strumenti online basati sulla formula di cui sopra che permettono il facile calcolo del numero di copie di plasmide. - Aliquotare e conservare lo standard come necessario a-20 ° C fino all'utilizzo.
- qPCR set-up per Standard e i campioni fecali
- Scongelare la qPCR standard (dal passaggio 3.1.10), DNA di campioni di feci (dal punto 2.15) e reagenti qPCR (da un kit disponibile in commercio) sul ghiaccio.
Nota: qPCR reagenti utilizzati in questo esempio sono il mix di reazione di SYBR Green qPCR e forward e reverse primer (tabella 2). - Diluire lo standard in acqua sterile privo di DNA in un range da 107 a 102 copie / µ l (per esempio, 1/5 diluizioni in serie da 107 a 6.4 x 102 copie / µ l). Diluire il campione fecale DNA a 1/2, 1/5, 1/10.
- Fare una reazione di mix master per il numero totale di reazioni più 1 (tabella 2).
- Mix 30 µ l di master mix e 5 µ l di modello (standard, campione o acqua per il controllo negativo)
- Aggiungere 10 µ l di questa miscela in ciascun pozzetto in una piastra 384 pozzetti ottico qPCR. Eseguire ogni reazione in triplice copia.
- Sigillare il posto di qPCR, centrifugare brevemente e caricare la piastra sulla macchina qPCR programmata con le seguenti condizioni in bicicletta: 95 ° C per 20 min, seguiti da 40 cicli di 95 ° C per 15 s e 60 ° C per 1 min.
- Ottenere i valori di CT per standard e campioni.
- Generare una curva standard tracciando i valori di CT per le norme contro il logaritmo del numero di copie del plasmide (come calcolato nel passaggio 3.1.9). Eseguire la regressione lineare della curva standard.
- Calcolare il numero di copie del rDNA 16S per campioni fecali interpolando i valori di CT (ottenuti al passaggio 3.2.2) nella curva standard.
Nota: numero di copie di rDNA 16S per ogni campione deve essere corretta considerando il fattore di diluizione del campione (come preparato al punto 3.2.2), gli acidi nucleici di volume finale che sono stati eluiti (al punto 2.15) e la quantità di campione fecale (calcolato nel passo 2.14) per ottenere copie del gene per grammo di feci.
- Scongelare la qPCR standard (dal passaggio 3.1.10), DNA di campioni di feci (dal punto 2.15) e reagenti qPCR (da un kit disponibile in commercio) sul ghiaccio.
Risultati
Qui forniamo due protocolli alternativi per il trattamento antibiotico orale dei topi. La figura 1 Mostra la percentuale di peso corporeo (relazionato al peso di linea di base originale per ogni animale) in topi trattati con antibiotici orali "gavage" (rosso) o nell'acqua potabile (blu) per 10 giorni consecutivi. Notevole perdita di peso non è trovata in topi che ricevono antibiotici mediante sonda gastrica orale. Tuttavia, quando i topi sono trattati con antibiotici ad libitum nell'acqua potabile, perdere peso (~ 10%) entro i primi giorni di somministrazione di antibiotici, ma recuperare successivamente aumento di peso normale (Figura 1). Comunque, circa 5-10% dei topi che ricevono antibiotici nell'acqua potabile può raggiungere > 20% perdita di peso entro la prima settimana di trattamento, nel qual caso essi sono euthanized.
La quantificazione dei batteri nei campioni fecali richiede l'uso di un'adeguata curva standard che si ottiene tracciando il log del numero di copie per lo standard (come calcolato al punto 3.2.2) contro i valori di CT ottenuti da qPCR (punto 3.2.7). La Figura 2A Mostra un esempio rappresentativo da una curva standard che soddisfa i criteri di prestazioni curva standard con un valore di2 R di 0.99827, un pendio di-3.09 e un'efficienza ((-1 + 10^(-1/slope))*100) del 110%. R2 valori di 0.99 ed efficienze PCR all'interno della gamma di 90-110% sono preferiti. All'interno della gamma lineare, l'equazione di regressione analisi consente la quantificazione dell'16S rDNA abbondanza all'interno i campioni fecali. Figura 2B indica il numero di copie di rDNA 16S nelle feci fecale, tenore di SI e parete di SI. In Figura 2B dati sono mostrati come 16S rDNA copie/g di campione fecale per sgabello fecale e tenore di SI. Per la parete SI, i dati sono presentati come numero totale di 16S rDNA copie ottenute da batteri recuperati da 3 cm del muro SI (come la quantità di materiale di partenza è troppo piccola per ottenere un peso preciso).
Per valutare l'effetto degli antibiotici sulla densità dei batteri nei campioni fecali, topi sono stati trattati con antibiotici mediante sonda gastrica orale al giorno per 10 giorni (giorni da 1 a 10) e campioni di feci sono stati raccolti prima (giorno 0), in diversi momenti durante il trattamento antibiotico ( giorni 5 e 10) e 7 giorni dopo l'arresto la somministrazione di antibiotici (giorno 17; Figura 3A). Come illustrato in Figura 3B, trattamento antibiotico induce una forte diminuzione del numero di 16S rDNA copie/g di feci rilevato ai giorni 5 e 10, mentre la densità dei batteri nelle feci recuperate ai livelli normali (paragonabili al pre-trattamento) 1 settimana dopo somministrazione di antibiotici è stata interrotta (giorno 17).

Figura 1: la somministrazione di antibiotici. I topi hanno ricevuto antibiotici orali "gavage" (rosso) o nell'acqua potabile (blu) per 10 giorni consecutivi. Trama indica pesi dei topi per tutta la durata degli esperimenti rispetto al peso originale prima della somministrazione di antibiotici (giorno 0). I dati vengono visualizzati come media ± SEM. Clicca qui per visualizzare una versione più grande di questa figura.
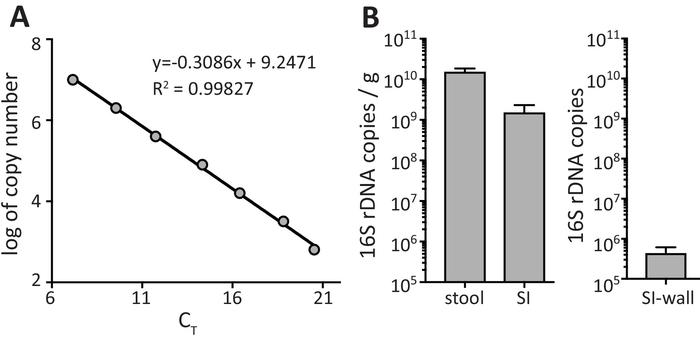
Figura 2: amplificazione del 16S rRNA gene qPCR di standard e i campioni fecali. (A) regressione lineare della curva standard con descrittori di curva standard. (B) calcolo delle abbondanze di gene da campioni fecali. I dati vengono visualizzati come media ± SEM. Clicca qui per visualizzare una versione più grande di questa figura.
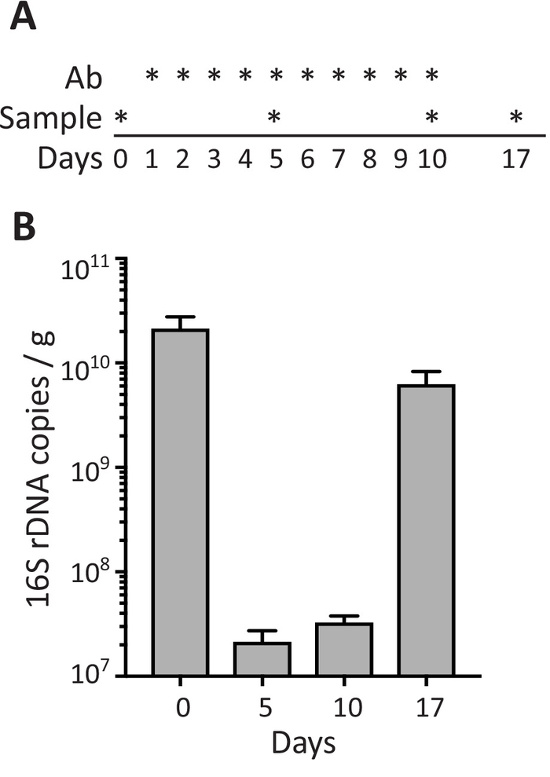
Figura 3: batteri fecali durante il trattamento antibiotico. (A) schema della pianificazione per la somministrazione di antibiotici orali "gavage" (Ab) e raccolta di campioni come raffigurato con *. (B), 16S rDNA copia per grammo di feci in campioni di feci da topi raccolti a giorni indicati. I dati vengono visualizzati come media ± SEM. Clicca qui per visualizzare una versione più grande di questa figura.
| Reagente | Volume | |
| DNA | 8 Μ l | |
| Buffer di 10 X | 2 Μ l | |
| dNTP (10mM) | 0,4 Μ l | |
| Eubatteri - primer F (10 mM) | 1 Μ l | |
| Eubatteri - primer R (10 mM) | 1 Μ l | |
| Taq polimerasi | 0.2 Μ l | |
| H2O | 7.4 Μ l | |
| Volume totale | 20 Μ l | |
| Sequenze dell'iniettore | ||
| Eubatteri - primer F | 5' ACTCCTACGGGAGGCAGCAGT 3' | |
| Eubatteri - primer R | 5' ATTACCGCGGCTGCTGGC 3' | |
| Condizioni di ciclismo | ||
| Temperatura | Tempo | Cicli |
| 94 ° C | 5 min | 1 x |
| 94 ° C | 30 s | 30 x |
| 60 ° C | 30 s | 30 x |
| 72 ° C | 1 min | 30 x |
| 72 ° C | 5 min | 1 x |
| 4 ° C | ∞ | 1 x |
Tabella 1: PCR reagenti e condizioni. Questa tabella fornisce i reagenti e il ciclismo di PCR per amplificare il gene del rRNA 16S da coltura batterica per la generazione di uno standard da usare nei saggi qPCR. Sequenze dell'iniettore sono stati originariamente pubblicati da Kruglov et al. 13.
| Reagente | Volume |
| Mix master SYBR Green (2x) | Μ l 17,5 |
| Eubatteri - primer F (10 mM) | 0,7 Μ l |
| Eubatteri - primer R (10 mM) | 0,7 Μ l |
| H2O | 11,1 Μ l |
Tabella 2: mix master qPCR. I volumi indicati (volume finale 35 µ l) sono per un singolo campione deve essere eseguito in triplice copia (da 10 µ l) su una piastra 384 pozzetti qPCR (contabilità per extra per pipettaggio errore 5 µ l). L'importo può essere scalato in base al numero di campioni da analizzare.
Discussione
Qui forniamo protocolli sperimentali per la somministrazione orale di antibiotici a topi e quantificazione dei batteri fecali di qPCR. La combinazione di antibiotici utilizzati in questo target di protocollo (contenente ampicillina, gentamicina, neomicina, metronidazole e vancomicina) batteri sia Gram-positivi e Gram-negativi, che offre attività battericida contro un ampio spettro di batteri. Sia mediante sonda gastrica orale e la somministrazione di antibiotici nell'acqua potabile notevolmente diminuire batterica fecale caricare5,6,12. Inoltre, entrambi i trattamenti hanno un profondo effetto sul fenotipo dei topi come si sviluppano diverse caratteristiche tipiche dei topi privo di germi tra cui dimensione ridotta della milza e allargata nell'intestino cieco. La selezione di un metodo particolare per somministrazione di antibiotici può forse dipende dalla durata dell'esperimento come richiede il metodo di alimentazione mediante sonda gastrica orale quotidiana somministrazione di antibiotici, essendo più alta intensità di lavoro e possibilmente causando più disagio per il animali nel lungo periodo.
Per la somministrazione di antibiotici nell'acqua potabile, deve usare cautela con l'aggiunta di dolcificante alla miscela antibiotica come questo è un fattore cruciale per mantenere i topi da disidratazione. Parecchi gruppi hanno dimostrato come la somministrazione di antibiotici in acqua potabile (senza aggiunta di dolcificante) conduce alla perdita di peso molto grave e rapido con tutti i topi perdere oltre il 20% del peso corporeo iniziale entro i primi giorni dell' esperimento5 , 6. nel nostro protocollo, l'uso di dolcificante saccarina-based sembrava essere sufficiente per mascherare il gusto antibiotico nell'acqua e topi perdita peso nei primi giorni dopo la somministrazione di antibiotici, ma ha recuperato la loro pesi rapidamente dopo che ( Figura 1). Ciò nonostante, nei nostri esperimenti 5-10% dei topi ancora raggiungere umano punto finale di > perdita del 20% del valore basale del peso corporeo e doveva essere euthanized. Abbiamo anche testato edulcoranti sucralosio-basato che completamente non è riuscito a prevenire la disidratazione di topi (100% dei topi ha perditi > 20% del peso) mentre altri autori hanno pubblicato simili fallimenti per dolcificanti a base di aspartame5,6. Aggiunto a questo, l'età, il background genetico e lo stato di salute generale dei topi utilizzati per gli esperimenti dovrebbe essere considerati, come essi possono influenzare la perdita di peso e benessere animale durante il trattamento antibiotico. Così, attento monitoraggio dei topi peso e stato di salute generale deve essere eseguita ogni giorno durante le prime due settimane di somministrazione di antibiotici orale.
qPCR metodi forniscono un approccio veloce e conveniente per la quantificazione del rRNA 16S in campioni di feci. Tuttavia, alcune limitazioni da considerare per quanto riguarda questa tecnica, tra cui: i) l'obbligo per uno standard di alta qualità affidabile; II) la progettazione e l'efficienza dei primer qPCR; III) il fatto che i microrganismi possono avere numeri di copia diversa del gene del rRNA 16S, così copie del gene non possono direttamente essere uguale cella conta15. Ciò nonostante, qPCR è un metodo sensibile e robusto che consente l'analisi rapida di campioni di feci. Questo metodo può essere particolarmente utile per verificare rapidamente l'effetto di vari trattamenti (compresi gli antibiotici) in carica batterica fecale come dettagliato qui. Inoltre, anche se forniamo un protocollo per la quantificazione di totale 16S rRNA, questo metodo può essere facilmente adattato (di progettazione primers specifici16) per consentire l'identificazione dei singoli taxa batterica, così fornendo sia quantitativa e informazioni qualitative sul microbioma dimensioni e composizione.
In sintesi, abbiamo fornito due protocolli per il trattamento antibiotico orale di topi e un metodo basato su qPCR di quantificare i cambiamenti più antibiotico-indotta di batteri fecali. Anche se questi protocolli possono essere ulteriormente ottimizzati e combinati con altri approcci secondo esigenze sperimentali, possono servire come strumenti rapidi, economici ed affidabili per manipolare il microbiota intestinale murino e studio degli effetti del trattamento antibiotico in omeostasi intestinale e la malattia.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato finanziato dalla UK Medical Research Council (grant a P.B. MR/L008157/1); R.J. è stata sostenuta da una borsa Marie Curie intraeuropee (H2020-MSCA-IF-2015-703639); P.M.B. è stata sostenuta da una borsa di studio da UK Medical Research Council e College Londra Consorzio del re di formazione dottorale in scienze biomediche (MR/N013700/1).
Materiali
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | Sigma-Aldrich (Merck) | A9518 | |
| Neomycnin trisulfate salt hydrate | Sigma-Aldrich (Merck) | N1876 | |
| Metronidazole | Sigma-Aldrich (Merck) | M3761 | |
| Vancomycin hydrochloride | Sigma-Aldrich (Merck) | V2002 | |
| Gentamicin sulfate salt | Sigma-Aldrich (Merck) | G3632 | |
| Tryptone | Sigma-Aldrich (Merck) | T7293 | |
| Yeast Extract | Sigma-Aldrich (Merck) | Y1625 | |
| NaCL | Sigma-Aldrich (Merck) | S7653 | |
| Sweetener Sweet'n Low | Sweet'N Low | Available in the UK from Amazon.co.uk | |
| X-Gal (5-brom-4-chloro-3-indoyl B-D-galactopyranoside) | Fisher scientific | 10234923 | |
| Phosphate Buffered Saline | Thermo Fisher Scientific (Gibco) | 10010023 | |
| Ultrapure Agarose | Thermo Fisher Scientific (Invitrogen) | 16500500 | |
| RT-PCR grade water | Thermo Fisher Scientific (Invitrogen) | AM9935 | |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530 | |
| Deoxynucleotide (dNTP) Solution Mix | New England BioLabs | N0447 | |
| iTaq Universal SYBR Green Supermix | Bio-Rad | 1725124 | with ROX |
| TOPO TA cloningTM for sequencing | Thermo Fisher Scientific (Invitrogen) | 450030 | |
| QIAamp fast DNA Stool mini kit | Qiagen | 51604 | |
| QIAprep spin Miniprep kit | Qiagen | 27106 | |
| QIAquick gel extraction kit | Qiagen | 28704 | |
| Syringe filter 0.45 µm | Fisher scientific | 10460031 | |
| Swann-MortonTM Carbon steel sterile scalpel blades | Fisher scientific | 11792724 | |
| Syringe (1 mL) | BD Plastipak | 303172 | |
| Syringe (20 mL) | BD Plastipak | 300613 | |
| 1.5 mL Crystal clear microcentriguge tube | StarLab | E1415-1500 | |
| 2 mL Ultra high recovery microcentrifuge tube | StarLab | I1420-2600 | |
| Oral dosing needles 20 G x 38 mm curved (pk/3) | Vet-Tech | DE008A | |
| Sterilin petri dish 50 mm | Scientific Laboratory Supplies | PET2020 | |
| Absolute qPCR plate seals | Thermo Fisher Scientific | AB1170 | |
| MicroAmpTM optical 384-well plate | Thermo Fisher Scientific (Applied Biosystems) | 4309849 | |
| ViiA7TM 7 real-time PCR system with 384-well block | Thermo Fisher Scientific (Applied Biosystems) | 4453536 | |
| Spectrophotometer (Nanodrop 1000) | Thermo Fisher Scientific | ND-1000 | |
| Labnet Prism microcentrifuge | Labnet | C2500 | |
| MultiGene Optimax Thermal cycler | Labnet | TC9610 |
Riferimenti
- Belkaid, Y., Hand, T. W. Role of the microbiota in immunity and inflammation. Cell. 157 (1), 121-141 (2014).
- Hooper, L. V., Littman, D. R., Macpherson, A. J. Interactions between the microbiota and the immune system. Science. 336 (6086), 1268-1273 (2012).
- Ubeda, C., Pamer, E. G. Antibiotics, microbiota, and immune defense. Trends in Immunology. 33 (9), 459-466 (2012).
- Dethlefsen, L., Huse, S., Sogin, M. L., Relman, D. A. The pervasive effects of an antibiotic on the human gut microbiota, as revealed by deep 16S rRNA sequencing. PLoS Biology. 6 (11), e280 (2008).
- Reikvam, D. H., et al. Depletion of murine intestinal microbiota: effects on gut mucosa and epithelial gene expression. PLoS One. 6 (3), e17996 (2011).
- Hill, D. A., et al. Metagenomic analyses reveal antibiotic-induced temporal and spatial changes in intestinal microbiota with associated alterations in immune cell homeostasis. Mucosal Immunology. 3 (2), 148-158 (2010).
- Fraher, M. H., O'Toole, P. W., Quigley, E. M. Techniques used to characterize the gut microbiota: a guide for the clinician. Nature Reviews Gastroenterology & Hepatology. 9 (6), 312-322 (2012).
- Eckburg, P. B., et al. Diversity of the human intestinal microbial flora. Science. 308 (5728), 1635-1638 (2005).
- Sokol, H., et al. Low counts of Fecalibacterium prausnitzii in colitis microbiota. Inflammatiry Bowel Diseases. 15 (8), 1183-1189 (2009).
- Bartosch, S., Fite, A., Macfarlane, G. T., McMurdo, M. E. Characterization of bacterial communities in feces from healthy elderly volunteers and hospitalized elderly patients by using real-time PCR and effects of antibiotic treatment on the fecal microbiota. Applied Environmental Microbiology. 70 (6), 3575-3581 (2004).
- Ubeda, C., et al. Vancomycin-resistant Enterococcus domination of intestinal microbiota is enabled by antibiotic treatment in mice and precedes bloodstream invasion in humans. Journal of Clinical Investigation. 120 (12), 4332-4341 (2010).
- Saez de Guinoa, J., et al. CD1d-mediated lipid presentation by CD11c(+) cells regulates intestinal homeostasis. EMBO Journal. 37 (5), (2018).
- Kruglov, A. A., et al. Nonredundant function of soluble LTalpha3 produced by innate lymphoid cells in intestinal homeostasis. Science. 342 (6163), 1243-1246 (2013).
- Wilhelm, J., Pingoud, A., Hahn, M. Real-time PCR-based method for the estimation of genome sizes. Nucleic Acids Research. 31 (10), e56 (2003).
- Kembel, S. W., Wu, M., Eisen, J. A., Green, J. L. Incorporating 16S gene copy number information improves estimates of microbial diversity and abundance. PLoS Computational Biology. 8 (10), e1002743 (2012).
- Yang, Y. W., et al. Use of 16S rRNA Gene-Targeted Group-Specific Primers for Real-Time PCR Analysis of Predominant Bacteria in Mouse Feces. Applied Environmental Microbiology. 81 (19), 6749-6756 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon