Method Article
Tempo di replicazione del DNA utilizzando Zebrafish come un sistema di modello In Vivo di profilatura
In questo articolo
Riepilogo
Zebrafish recentemente sono stati utilizzati come un sistema di modello in vivo per studiare il tempo di replicazione del DNA durante lo sviluppo. Qui è dettagliato i protocolli per l'utilizzo di embrioni di zebrafish per tempo di replicazione di profilo. Questo protocollo può essere facilmente adattato per studiare il tempo di replicazione in mutanti, tipi di cellule individuali, modelli di malattia e altre specie.
Abstract
Tempo di replicazione del DNA è una caratteristica importante di cellulare, che esibiscono le relazioni significative con i tassi di mutazione del DNA, trascrizione e struttura della cromatina. Cambiamenti nel tempo di replicazione si verificano durante lo sviluppo e nel cancro, ma il tempo di replicazione di ruolo svolge nello sviluppo e la malattia non è noto. Zebrafish recentemente sono stati stabiliti come un sistema di modello in vivo per studiare il tempo di replicazione. Qui è dettagliato i protocolli per l'utilizzo di zebrafish per determinare il tempo di replicazione del DNA. Dopo l'ordinamento delle cellule da embrioni e zebrafish adulto, ad alta definizione modelli di genoma del DNA replica temporizzazione possono essere costruiti determinando cambiamenti nel numero di copia di DNA attraverso l'analisi dei dati di sequenziamento di generazione successivi. Il sistema di modello di Danio rerio consente per la valutazione dei cambiamenti di temporizzazione di replica che si verificano in vivo durante lo sviluppo e può anche essere utilizzato per valutare i cambiamenti nei tipi di cellule individuali, modelli di malattia o linee mutanti. Questi metodi consentirà gli studi che studiano i meccanismi e i fattori determinanti di replica temporizzazione stabilimento e manutenzione durante lo sviluppo, il tempo di replicazione di ruolo gioca a mutazioni e tumorigenesi e gli effetti di perturbazione tempo di replicazione su sviluppo e malattia.
Introduzione
Per cellule a dividersi con successo, essi devono essere prima accuratamente e fedelmente replicate loro intero genoma. Duplicazione del genoma si verifica in un modello riproducibile, noto come il DNA replica temporizzazione programma1. Tempo di replicazione del DNA è correlata con l'organizzazione della cromatina, segni epigenetici e gene expression2,3. Cambiamenti nel tempo di replicazione si verificano nel corso dello sviluppo e sono significativamente correlati a trascrizionale programmi e alterazioni della cromatina marchi e organizzazione4,5. Inoltre, tempo di replicazione è correlato con frequenze mutazionale, e cambiamenti nella tempistica sono osservati in vari tipi di cancro6,7,8. Nonostante queste osservazioni, i meccanismi e i fattori determinanti di stabilimento di temporizzazione replica e regolamento sono ancora largamente sconosciuti e il ruolo che essa svolge nello sviluppo e la malattia è indeterminata. Inoltre, fino al recente la replicazione del genoma tempi di cambiamenti che si verificano nel corso dello sviluppo dei vertebrati aveva solo stato esaminato in modelli di coltura cellulare.
Zebrafish, Danio rerio, sono adatti a studiare replica temporizzazione in vivo durante lo sviluppo, come una singola coppia di accoppiamento può produrre centinaia di embrioni che si sviluppano rapidamente con molte somiglianze con lo sviluppo dei mammiferi9, 10. Inoltre, nel corso dello sviluppo di zebrafish, ci sono cambiamenti per il ciclo cellulare, organizzazione della cromatina e programmi trascrizionali che condividono relazioni con DNA replica temporizzazione11. Zebrafish sono anche un eccellente modello genetico, come essi sono particolarmente suscettibili alla manipolazione di transgenesi, mutagenesi e mutazioni mirate e schermi genetici hanno identificato molti geni richiesti per sviluppo vertebrato12. Di conseguenza, zebrafish può essere utilizzato per identificare i geni coinvolti nella manutenzione e creazione di temporizzazione replica e osservare gli effetti di deregolamentare il tempo di replicazione sullo sviluppo dei vertebrati. Linee transgeniche possono anche essere utilizzati per valutare il tempo di replicazione da tipi di cella individuale isolato timepoints inerente allo sviluppo differente o in condizioni di malattia. Importante, ci sono vari modelli di zebrafish della malattia umana che può essere utilizzato per studiare il ruolo del tempo di replicazione in malattia formazione e progressione9,13,14.
Recentemente, i profili di sincronizzazione replica primi sono stati generati dallo zebrafish, stabilendola come sistema modello per lo Studio in vivo15di sincronizzazione di replica. A tal fine, le cellule sono stati raccolti da embrioni di zebrafish in più fasi di sviluppo e in un tipo di cellule isolate da adulto zebrafish. Le cellule sono state quindi filtrate FACS (ordinamento di fluorescenza-attivato delle cellule) basato sul contenuto di DNA per isolare popolazioni di fase G1 e S. Utilizzando l'esempio di G1 come un controllo di numero di copia, variazioni del numeri di copie in popolazioni di fase S erano determinati ed utilizzati per dedurre replica relativa temporizzazione16. Cambiamenti nel tempo di replicazione possono essere direttamente confrontate tra diversi campioni inerente allo sviluppo e tipi di cellule e questo è stato utilizzato per determinare cambiamenti nel tempo di replicazione che si verificano in vivo nel corso dello sviluppo dei vertebrati. Questo metodo offre parecchi vantaggi sopra altri metodi di genomiche, soprattutto che non richiede etichettatura con gli analoghi della timidina o immunoprecipitazione di DNA4,6.
Qui è dettagliato i protocolli a tempo di replicazione del DNA di profilo genoma a ad alta risoluzione in zebrafish. Questi protocolli sono stati utilizzati per determinare le relazioni con le caratteristiche genomiche ed epigenetici nel genoma di zebrafish, nonché a modifiche all'analisi in queste relazioni che si verificano durante lo sviluppo. Questi protocolli sono anche facilmente adattati per studiare i cambiamenti nel tempo di replicazione in ceppi mutanti di zebrafish e nei modelli di malattia. Inoltre, questi metodi forniscono una base che può essere estesa, per studiare il tempo di replicazione in tipi cellulari specifici, dal primo procurandomi i tipi di cellule individuali da zebrafish. Zebrafish può servire come un eccellente in vivo sistema modello per studiare il tempo di replicazione e in ultima analisi rivelano le funzioni biologiche di questo importante tratto epigenetico.
Protocollo
Tutti gli animali erano gestiti in stretta conformità con protocolli approvati dal comitato di uso e Oklahoma Medical Research Foundation istituzionali cura degli animali.
1. configurazione di zebrafish adulto per l'allevamento
- Utilizzare un'ampia coorte di zebrafish adulto maschio e femmina di un singolo ceppo per l'allevamento. Ci sono piccole differenze nel corredo genetico di zebrafish di persino un singolo ceppo, utilizzare un'ampia coorte affinché i risultati siano rappresentativi della variabilità genetica della popolazione e non limitato a un piccolo sottoinsieme della popolazione.
- La notte prima che gli embrioni saranno raccolti, inserire decine di adulti di allevamento in vasche di allevamento, utilizzano un numero approssimativamente uguale di maschi e femmine. A seconda dell'esperimento, utilizzare uno delle seguenti strategie di allevamento.
- Allevamento di strategia 1: posto pochi zebrafish maschile e femminile nelle vasche di allevamento individuale, separare i maschi dalle femmine con un divisore. Se viene utilizzato questo approccio, installazione molte vasche di allevamento diversi e gli embrioni da ciascuno per garantire che la variabilità biologica è rappresentativo della popolazione della piscina.
- Allevamento di strategia 2: combinare decine di zebrafish maschile e femminile in un serbatoio di grande allevamento. Utilizzare questa strategia, purché ci siano un numero sufficientemente ampio di embrioni, è ragionevole supporre che sono venuti da una varietà di fondatori e sono pertanto geneticamente rappresentativo della popolazione.
2. gli accoppiamenti - raccolta, cernita e custodia embrioni di zebrafish per esperimenti a tempo
- Eseguire accoppiamenti temporizzati, inizio, non appena la luce cicli essendo la mattina. Eliminare eventuali embrioni generati durante la notte.
- Consentire pesce per riprodursi in acque poco profonde (3-5 cm) per un periodo di 10 min, con un doppio fondo per gli embrioni a fuggire attraverso.
- Dopo 10 min, raccogliere embrioni versando attraverso un colino e risciacquo in una piastra di 10 cm con una spruzzetta. Piscina tutti gli embrioni raccolti dallo stesso punto di tempo, li segnano con tempo di raccolta e posto immediatamente in un incubatore a 28,5 ° C.
- Centinaia di embrioni può essere previsto da una singola coppia di accoppiamento in un giorno. Consentono agli adulti di allevare in 10 min cicli fino a quando il numero di embrioni ottenuti è meno di venti.
- Circa 1-1,5 h dopo la raccolta, embrioni di sorta per rimuovere gli embrioni morti e non fertilizzati. Per ordinare gli embrioni, rimuovere dall'incubatore e osservare sotto un microscopio per dissezione.
- Embrioni di conteggio e posto ad una densità di 100 embrioni per piastra di 10 cm.
- Aggiungere mezzo fresco E3 (5 mM NaCl, 0,17 mM KCl, 0,33 mM CaCl2, 0,33 mM MgSO4) e restituire gli embrioni nell'incubatore di 28,5 ° C.
3. Dechorionate, deyolk e difficoltà di embrioni di zebrafish
Nota: Questa sezione del protocollo è progettata per gli embrioni prima della fecondazione post di 48h (hpf). Non c'è nessuna necessità di rimuovere la chorions di embrioni nelle fasi successive dello sviluppo (dopo 48 hpf), in quanto essi spesso naturalmente cadere. Non c'è nessun bisogno di deyolk o rimuovere il chorions di pesce più anziani i 5 giorni post fertilizzazione (dpf).
- Quando gli embrioni raggiungono età evolutiva desiderata, verificare adeguata fase di sviluppo morfologicamente sotto un microscopio chiaro secondo "Fasi di embrionale sviluppo di Zebrafish the"17.
- Trasferire gli embrioni in fasi fino a 1500 in una provetta conica da 15 mL e lavare più volte con E3.
- Aggiungere 1 mL di E3 e 20 µ l di soluzione di Pronase (30 mg/mL) per ogni 100 embrioni e agitare delicatamente. Fino a 1500 embrioni possono essere dechorionated in un singolo tubo con 15 mL di E3.
- Adagiare il tubo su un lato e organizzare gli embrioni in uno strato uniforme per garantire la massima superficie in rapporto al volume. Agitare delicatamente il tubo ogni 2-3 minuti fino a quando tutti i chorions sono caduti fuori gli embrioni. Il tempo di dechorionation totale varierà a seconda dell'attività di Pronase.
- Rimuovere il surnatante e sciacquare delicatamente gli embrioni 3 volte con 10 mL di E3. Posizionare gli embrioni in ghiaccio per il resto di questa sezione del protocollo.
- E3 di rimuovere e lavare gli embrioni con buffer di deyolking ghiacciata preparata al momento (0,5 x lattato Ginzburg pesce Ringer, senza calcio: 55 mM NaCl, 1,8 mM KCl, 1,25 mM NaHCO3).
- Aggiungere 1 mL di tampone deyolking ghiacciata per gli embrioni fino a 500.
- Utilizzando un P1000, delicatamente Pipettare embrioni su e giù 5 - 10 volte per interrompere il sacco del tuorlo.
- Trasferire 1 mL in una provetta da 1,5 mL microfuge e centrifugare a 4 ° C e 500 x g per 5 min.
- Rimuovere il surnatante e lavare la pallina con 1 mL di PBS 1X ghiacciata (soluzione salina tamponata con fosfato, pH 7.0) per rimuovere la soluzione di Ringer. Centrifuga a 4 ° C e 500 x g per 5 min.
- Con attenzione rimuovere il surnatante e risospendere le cellule in 1 mL di PBS 1X. Posizionare il tubo sul ghiaccio.
- Aggiungere 3 mL di gelida etanolo (EtOH) in una provetta conica 15 mL. Tubo a vortice di EtOH delicatamente a bassa definizione.
- Utilizzando un P1000, lentamente (1 goccia al secondo) del gocciolamento zebrafish embrione sospensione cellulare in EtOH mentre nel Vortex delicatamente.
- Aggiunta di un totale di 1 mL di sospensione di cellule di embrione, per una soluzione di fissaggio finale del 75% EtOH.
- Dopo 1 mL di cella sospensione è stato aggiunto, delicatamente ricciolo tubo per mescolare e mettere a-20 ° C per almeno 1 h. Si consiglia di posizionare i tubi al loro fianco per garantire la massima superficie in rapporto al volume e ridurre il numero di embrioni e cellule che si attaccano insieme. Conservare le cellule dell'embrione a-20 ° C per 2-3 settimane.
4. colorazione DNA e FACS ordinamento embrioni
Nota: Questa sezione del protocollo è progettata per gli embrioni a 1 dpf.
- Rimuovere le cellule di embrione di EtOH-fisso da-20 ° C e centrifugare a 4 ° C 1500 x g per 5 min.
- Posizionare il tubo sul ghiaccio e attentamente rimuovere surnatante. Utilizzando un P1000, delicatamente risospendere le cellule in 1 mL di preparati PBS freddo-BSA (1x PBS, 1% di sieroalbumina bovina).
- Centrifugare le cellule a 4 ° C e 1500 x g per 5 min e metterli su ghiaccio.
- Rimuovi surnatante e risospendere le cellule in ioduro di propidio (PI) soluzione (50 µ g/mL PI, 100 µ g/mL RNasi A, PBS, pH 7,0), di colorazione utilizzando 200 µ l per ogni 1000 gli embrioni.
- Consentire alle cellule di Incubare in soluzione di PI per 30 min in ghiaccio, mescolando delicatamente ogni 5 min.
- Posizionare un colino di cella in nylon 40 µm su un tubo conico da 50 mL su ghiaccio. Pipettare delicatamente le cellule per mescolare e filtrare attraverso le maglie.
Nota: Se più tubi di embrioni da timepoint stesso sono stati raccolti nella sezione 3 del protocollo, combinare questi embrioni a questo punto così che essi sono tutti ordinati insieme. - Utilizzo l'appartamento all'interno di fine di un stantuffo della siringa, delicatamente perturbare qualsiasi restanti grumi di cellule sulla mesh.
- Sciacquare le cellule attraverso il filtro con 500 µ l di PBS freddo-BSA per ogni 1000 embrioni.
- Posizionare una maglia 40-µm sopra un tubo conico di 15 mL sul ghiaccio e filtrare la sospensione cellulare dalla provetta conica da 50 mL una seconda volta nel tubo 15 mL.
- Utilizzando una cella appropriata strumento di ordinamento, impostare l'eccitazione laser per rilevamento 561 nm ed emissione a 610/20 per rilevare ioduro di propidio.
- Mix 15 tubo mL di cellule marcate brevemente da Vortex e posto in strumento a 4 ° C.
- Tasso di esecuzione celle a flusso lento, idealmente 1,0 mL/min, al fine di ottenere un profilo del ciclo cellulare pulito ed evitare di mescolare G1 o G2/M cellule con la S fase popolazione.
- In primo luogo, il cancello per FSC-A x SSC-A (zona di forward scatter di zona di dispersione laterale) per rimuovere i detriti (Figura 1A).
Nota: Cellule da embrioni possono essere diverse dimensioni a seconda del tipo di fase e delle cellule. Il filtro a densità neutra (ND) potrebbe essere necessario essere commutato tra 1.0 e 1.5 a seconda delle dimensioni delle cellule presenti. Una grande area per raccogliere la maggior parte delle cellule e rimuovere i detriti del cancello. - Secondo, cancello per SSC-area (SSC-A) da PI-area (PI-A), per rimuovere i detriti e cella doppietti (Figura 1B).
- In terzo luogo, il cancello per SSC-A di SSC-W (larghezza) per rimuovere qualsiasi residuo doppietti di cella (Figura 1).
- Visualizzare le popolazioni recintate su un istogramma con numero di cellulare (conteggio) sulla zona di asse y e propidio ioduro (PI-A) sull'asse x. Per 24 embrioni hpf, questo dovrebbe dare un profilo di ciclo cellulare stereotipata come in Figura 1.
- Per ordinare la popolazione di fase S, impostare il cancello dal lato destro del picco G1 al lato sinistro del picco G2, tra cui solo una minima quantità delle popolazioni G1 e G2. Per 24 embrioni hpf, ciò comporterà in genere circa 10-15% della popolazione con cancello (Vedi Figura 1).
- Per ordinare la popolazione di G1, impostare la porta sul lato sinistro del picco G1, di non passare la cima del picco. Regolare la larghezza del cancello G1 più stretta possibile (Vedi Figura 1).
- Nuovo cancello il G1 e popolazioni di fase S per garantire la corretta iniziale gating (Figura 1E).
- Ordinare le popolazioni di fase G1 e S in provette coniche da 15 mL con 3 mL di PBS-BSA. L'obiettivo dovrebbe essere quello di ottenere tra 500.000 e 1 milione o più celle ordinate per ogni frazione (fase G1 e S).
- Dopo l'ordinamento è completo, centrifugare le provette a 1500 x g e a 4 ° C per 10 min.
Nota: Il pellet cellulare piccolo sarà difficile da vedere ed è importante evitare di interrompere esso, quindi utilizzare preferibilmente un rotore oscillante sopra un rotore ad angolo fisso. - Con attenzione rimuovere il surnatante e risospendere il pellet con 1 mL di PBS 1X. Trasferire le cellule un 1,5 mL microfuge provetta e centrifugare a 6.000 x g e a 4 ° C per 5 min.
- Con attenzione rimuovere tutto il liquido dal tubo e congelare a secco pellet a-20 ° C. Conservare il pellet a-20 ° C per 2-3 settimane.
5. DNA isolamento, trattamento di RNAsi e purificazione del DNA
- Rimuovere il pellet cellulare da-20 ° C e posto sul ghiaccio.
- Aggiungere 400 µ l di tampone di SDS (50 mM Tris-HCl (pH 8.0), 10 mM EDTA, 1 M di NaCl, 0,5% SDS) a pellet e risospendere.
- Aggiungere proteinasi K a una concentrazione finale di 0,2 mg/mL e mescolare nel Vortex brevemente.
- Incubare i campioni a 56 ° C per 2 h, vortice ogni 15 min.
- Dopo 2 h, eseguire l'estrazione del fenolo-cloroformio aggiungendo 400 µ l di fenolo-cloroformio (fenolo: cloroformio: alcool isoamilico 25:24:1, saturo di 10 mM Tris, pH 8.0, 1 mM EDTA).
- Vortexare per 20 s e centrifugare il campione a 16.000 x g per 5 min separare le fasi.
- Rimuovere con cautela la fase acquosa superiore (400 µ l) e trasferirlo in una provetta da 1,5 mL.
- Eseguire EtOH precipitazione aggiungendo 40 µ l di acetato di sodio 3M (pH 5.2), 2 µ l di glicogeno e 1 mL di EtOH.
- Vortice per miscelare accuratamente e posizionare a-20 ° C durante la notte per precipitare il DNA. Campione può anche essere precipitato a-80 ° C o il ghiaccio secco per 1 h.
- Campione di centrifuga a 4 ° C e 16.000 x g per 30 min al DNA di pellet.
- Scartare la pallina surnatante e lavare con 1 mL 70% EtOH. Centrifuga a 4 ° C e 16.000 x g per 5 min.
- Scartare il pellet surnatante e asciugato all'aria a temperatura ambiente per 2-3 min.
- Sciogliere il pellet in 96 µ l di acqua in autoclave e aggiungere 4 µ l di RNasi A (2 mg/mL).
- Mescolare bene nel vortex e incubare a 37 ° C per 1 h.
- Eseguire l'estrazione del fenolo-cloroformio aggiungendo 100 µ l di fenolo-cloroformio e procedura riportata di seguito nei passaggi 5.2.1 - 5.2.2.
- Eseguire EtOH precipitazione aggiungendo 10 µ l di acetato di sodio 3M, 1 µ l glicogeno e 250 µ l di EtOH e seguendo la procedura in passaggi 5.3.1 - 5.3.5.
- Risospendere il pellet in 60 µ l di buffer di TE (10 mM Tris-HCl (pH 8.0), 10mm EDTA (pH8.0)) e calcolare la concentrazione di DNA mediante un metodo basato sulla fluorescenza quantitativo. Archivio del DNA a-20 ° C.
Nota: È importante quantificare DNA usando coloranti associazione DNA basata sulla fluorescenza o con altri mezzi più affidabili rispetto ai metodi basati su spettrometro UV.
6. preparazione di librerie di DNA e sequenziamento di nuova generazione
Nota: Un campione di riferimento numero G1 copia per ogni fonte biologica è necessario per ogni esecuzione di sequenziamento (cioè WT, linea mutante, transgenici, cellulare, ecc). Confrontare campioni di fase S tutti dalla stessa fonte biologica nel sequenziamento stesso eseguita allo stesso riferimento G1. Eseguire almeno due repliche biologiche di ogni campione per garantire la coerenza tra i campioni.
- Diluire 1 µ g di DNA ad un volume finale di 50 µ l e trasferirlo in una provetta specializzata per sonicazione.
- Taglio del DNA di genomic per un target di 250-300 bp in un ultrasonicatore concentrato, secondo le specifiche del produttore e protocollo.
- Verificare la qualità e le dimensioni del DNA genomico tranciato utilizzando una macchina per elettroforesi automatica, secondo le specifiche del produttore e protocollo. Garantire c'è un alto picco di ~ 250-300 bp per il DNA di genomic tranciato.
- Preparare le librerie di sequenziamento utilizzando un kit di preparazione libreria di DNA con i oligos multiplex per il sequenziamento su piattaforma Illumina, secondo il protocollo del produttore.
- Verificare la qualità della preparazione di libreria utilizzando l'elettroforesi automatica macchina, secondo il protocollo del produttore. Garantire c'è un alto picco di ~ 400-450 bp, e ci sono picchi minimi per dimeri dell'iniettore (80-85 bp) e adattatore dimeri (~ 120 bp).
- Quantificare le librerie utilizzando un kit di quantificazione di DNA libreria per sequenziamento di nuova generazione, secondo il protocollo del produttore.
- Diluire 15 µ l di libreria a una concentrazione di 5 nM e combinare multiplexed librerie come richiesto per raggiungere il desiderato in modo univoco mapping letto conteggi per campione.
Nota: Il numero di in modo univoco il mapping letture per campione determinerà la risoluzione. Uso da un minimo di 5 milioni mappato legge per valutare i tempi di replica per il genoma di zebrafish. Si raccomanda di iniziare con 10 milioni di letture. - Eseguire il sequenziamento del DNA di accoppiato-fine 100 bp intero genoma di campioni su una piattaforma di sequenziamento di nuova generazione, secondo le istruzioni del produttore.
7. analisi dei dati di sequenziamento
Nota: Le istruzioni riportate in questa sezione sono destinate come linea guida per l'analisi. Utilizzare metodi aggiuntivi per l'allineamento di sequenza, filtraggio, elaborazione, ecc. Questa sezione del protocollo si occuperà con il metodo preferito di analisi in questo lavoro. Se vengono utilizzati metodi aggiuntivi, regolare i parametri e le funzioni per soddisfare tali finalità. I seguenti comandi vengono immessi in Ubutnu o Mac terminal, con i pacchetti appropriati installati.
- Ottenere le versioni più recenti del genoma di zebrafish (danRer10, a partire da quando è stato scritto questo protocollo) dal browser del genoma UCSC utilizzando i seguenti comandi:
wget ftp://hgdownload.cse.ucsc.edu/goldenPath/danRer10/bigZips/danRer10.fa.gz-O danRer10.fa.gz- Decomprimere il file fa.gz con il seguente comando:
gunzip danRer10.fa.gz - Allineare i dati di sequenziamento genoma utilizzando BWA-mem utilizzando i seguenti comandi:
BWA mem -t 8 danRer10.fa read_1.fastq read_2.fastq > output.sam
- Decomprimere il file fa.gz con il seguente comando:
- Convertire il file. Sam .bam file tramite samtools con il seguente comando:
Mostra samtools -b input.sam > output.bam- Ordinare i file di sequenziamento allineati utilizzando samtools con il seguente comando:
samtools ordinare output.bam > output_sorted.bam - Indicizzare il file .bam con samtools con il seguente comando:
samtools Indice output.bam
- Ordinare i file di sequenziamento allineati utilizzando samtools con il seguente comando:
- Mark PCR Duplica con Picard utilizzando i seguenti comandi:
Java-Xmx2g-jar picard.jar MarkDuplicates \
INPUT=input.BAM
OUTPUT=output.BAM
REMOVE_DUPLICATES = false
METRICS_FILE=output_metrics.txt - Generare file di testo (. txt) di letture per ogni cromosoma utilizzando il seguente comando:
per i in {1,25}; fare CHROM = "chr" $i; Eco $CHROM; samtools Mostra input.bam $CHROM | cut -f 2,4,5,7,8,9 > readsChr. txt$ {i}; fatto
Nota: Le colonne nel file. txt risultante sono Flag, posizione, mapQ, coppia chr, coppia di posizione e dimensione del frammento, rispettivamente. - Leggere i file txt in Matlab (o qualsiasi altro software simile) utilizzando la funzione textscan.
- Filtrare la mappatura bassa qualità letture impostando una soglia di mapQ di 30.
- Rimuovere letture con le bandierine per allineamento non primario, duplicati PCR o mappatura di coppia ad un cromosoma diverso.
- Filtrare qualsiasi letture con loro coppia di là di una soglia di distanza di 2000 bp.
- Valutare le statistiche di letture risultante per determinare il numero di statistiche di lettura, copertura, dimensione dell'inserto e qualità (Figura 2).
- Determinare la copertura della lettura in 100 finestre bp fissata per ciascun campione contando il numero di letture in 100 intervalli di bp su tutta la lunghezza di ogni cromosoma.
- Calcolare che la mediana leggere totali per tutte le finestre.
- Filtrare le regioni di alta copertura rimuovendo windows maggiore di 5 deviazioni standard dalla mediana.
- Definire le aree per l'analisi nei campioni di riferimento G1 trovando segmenti lungo ciascun cromosoma con 200 letture utilizzando finestre scorrevoli.
- Determinare la profondità di lettura in ogni campione di fase S contando il numero di letture di fase S in windows 200-Leggi totali definiti per l'accompagnamento riferimento G1.
- Lisciare i dati non elaborati utilizzando la funzione csaps nel software, con un fattore di interpolazione (p) di 10-16.
- Normalizzare i dati levigati a z-score con media 0 e deviazione standard di 1.
Risultati
Utilizzando dati di intervallo di replica pubblicati, rappresentante replica temporizzazione profili e misure di controllo di qualità sono disponibili15. I passi iniziali dell'elaborazione coinvolgono allineando i dati di sequenziamento del genoma, calcolo lunghezza lettura e statistiche relative alla copertura del genoma e il filtraggio di bassa qualità, spaiati, e letture duplicate PCR. Statistiche di lettura per un esempio di sequenziamento di zebrafish tipici sono mostrate nella Figura 2. Dopo il filtraggio, leggi i conteggi sono determinati in finestre di dimensioni variabili e i dati sono levigata e normalizzato. Profili di temporizzazione replica tipica levigata/normalizzato di un cromosoma di zebrafish rappresentativo per repliche biologiche sono mostrati in Figura 3A. I profili per repliche biologiche e sperimentali dovrebbero essere visivamente molto simili (Vedi Figura 3A) e anche visualizzare alta correlazione lungo la lunghezza del cromosoma (Figura 3B). Essi dovrebbero anche visualizzare alta correlazione tra valori di intervallo genoma (Figura 3). Autocorrelazione, un metodo per valutare la continuità di disegno, deve essere utilizzato per valutare il grado di struttura nel set di dati (Figura 3D). L'autocorrelazione per programmi strutturati temporizzazione maturo dovrebbe essere elevato a brevi distanze e diminuire gradualmente man mano che aumenta la distanza. I campioni che mostrano un'elevata riproducibilità tra biologico e sperimentale replica e un segnale forte di autocorrelazione dovrebbe essere considerato come campioni di alta qualità e possa essere combinato per ottenere un campione di copertura superiore.
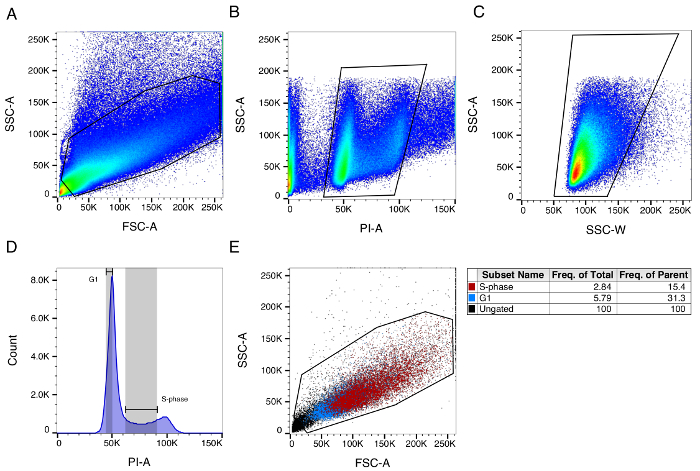
Figura 1: l'ordinamento di embrioni di zebrafish per analisi temporale replica. (A) Gated della popolazione di tutte le celle filtrate per zona di forward scatter (FSC-A) da zona di dispersione laterale (SSC-A). I colori rappresentano la densità di bidoni nella trama del puntino con alta-bassa densità rappresentati da colori che vanno dal rosso-verde-blu. (B) Gated popolazione delle cellule FSC x SSC gated ordinati per SSC-A per zona di ioduro di propidio (PI-A). (C) Gated popolazione del CSD x cellule PI gated ordinati per SSC-A di larghezza di dispersione laterale (SSC-W). Istogramma (D) di tutte le popolazioni gated visualizzati da un profilo di ciclo cellulare stereotipata. Cancelli di sorta per cellule in fase G1 e S vengono visualizzate da grigio sfumato boxed con linee nere. Popolazioni di fase (E) Backgated G1 e S mostrano che il gating iniziale catturato la maggior parte delle cellule. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: sequenziamento leggere le statistiche per un esempio di tipica zebrafish. (A) Reap mappatura qualità dalle statistiche dei valori mapQ. (B) istogramma della distribuzione delle dimensioni inserto per tutte le letture di sequenziamento. (C) le statistiche di lettura per totale letture mappate, legge contenente flag di bassa qualità, legge con coppie di mappatura ad un cromosoma diverso (coppia diff chr), legge con coppia oltre la soglia di distanza (distante coppia) e PCR duplicati. (D) numeri rappresentativi del totale mappato, letture non mappate e spaiate, copertura e leggere ad alta risoluzione per una corsa di sequenza tipica di un campione di zebrafish. Clicca qui per visualizzare una versione più grande di questa figura.
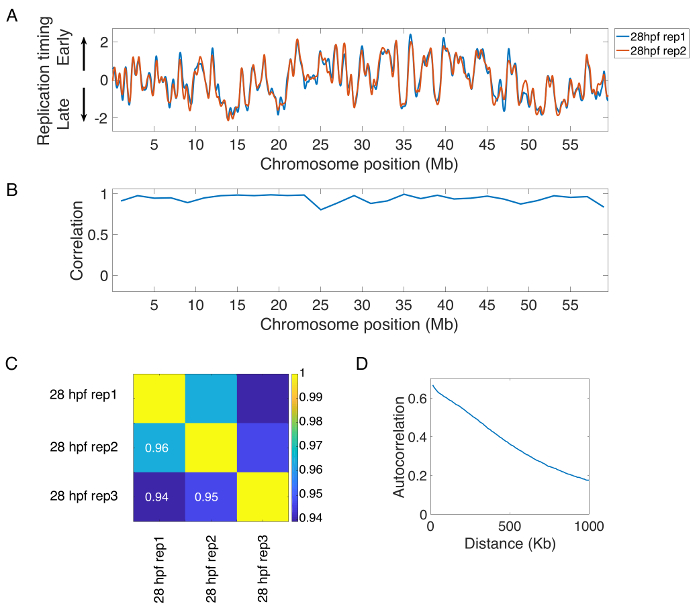
Figura 3: risultati di temporizzazione replica rappresentante zebrafish e misure di controllo di qualità. (A) replica dentate lungo la lunghezza di un rappresentante del cromosoma per due repliche biologiche di 28 embrioni hpf (Siefert, 2017). (B) correlazione tra valori di intervallo di replica a 2 Mb windows lungo la lunghezza di un cromosoma rappresentativo per il biologico replica di 28 embrioni hpf, mostrati in Figura 3A. (C) Genome-wide correlazione tra valori di intervallo di replica per il biologico (rep1 e rep2) e sperimentale (rep3) replicati di 28 embrioni hpf. La mappa dei colori rappresenta il coefficiente di correlazione di Pearson. Autocorrelazione (D) per un programma strutturato maturo temporizzazione Visualizza autocorrelazione alta a distanza ravvicinata che diminuisce gradualmente su distanze sempre più lunghi. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Zebrafish forniscono un sistema di modello nuovo e unico in vivo per studiare tempo di replicazione del DNA. Quando temporizzati accoppiamenti vengono eseguiti come dettagliato in questo protocollo sperimentale, migliaia di embrioni possa essere raccolti in un solo giorno per esperimenti. Questi embrioni si sviluppano in modo sincrono attraverso stadi di sviluppo precisamente cronometrati e distintamente caratterizzati. Zebrafish può essere facilmente ed accuratamente organizzato dalla morfologia utilizzando un microscopio stereoscopico, come embrioni di zebrafish sviluppano esternamente e sono otticamente chiari. Questo protocollo dettaglia l'uso di embrioni di zebrafish per studiare il tempo di replicazione durante lo sviluppo dei vertebrati.
Aree del protocollo che possono presentare problemi comprendono l'assicurazione lo sviluppo sincrono di embrioni di zebrafish, perdita di cellule durante la preparazione, l'ordinamento FACS e preparazione di biblioteca. I tempi di sviluppo di zebrafish è temperatura dipendente, pertanto uso zebrafish adattata ai cicli di luce/buio e ospitato a 28,5 ° C per gli esperimenti. Per garantire lo sviluppo sincrono, eseguire precisamente cronometrati accoppiamenti e raccogliere embrioni in molto tempo piccole-windows (10 min o meno). È fondamentale per mantenere gli embrioni a 28,5 ° C ed assicurare che spendono tempo minimo a temperatura ambiente. I tempi di sviluppo di zebrafish sono anche fortemente dipendenti dalla densità degli embrioni in una determinata area. È fondamentale per non embrioni di casa a densità maggiore di 100 embrioni per piastra di 10 cm, o non svilupperà correttamente. Il momento ottimale per rimuovere gli embrioni morti e non fecondati è quando essi hanno progredito alla fase cellulare 4-16 e può essere facilmente identificati come fertilizzato (uso "Fasi di embrionale sviluppo di Zebrafish the"17 come guida). Embrioni morti in genere sono visualizzati come palline nere e non fecondati embrioni come 1-cell. Lavorare più velocemente possibile per ridurre il tempo a temperatura ambiente.
Quando dechorionating embrioni, è importante garantire a tutte le chorions vengono rimossi da embrioni. Mantenere gli embrioni in pronase soluzione fino a quando la maggior parte dei chorions sono venuti fuori. Scartare eventuali embrioni rimanenti con chorions intatto o rimuovere loro chorions con le pinzette. Embrioni di pipetta con un movimento lento e uniforme. Non pipettare rapidamente come esso sarà soggetto le cellule per lo sforzo eccessivo e risultato nella perdita delle cellule. Inoltre è importante non utilizzare un P200, in quanto ciò può causare una maggiore sollecitazione di taglio che si traduce nella rottura delle cellule. Al contrario, una pipetta di plastica non può avere abbastanza un piccolo foro o fornire abbastanza shear stress per perturbare il sacco del tuorlo in maniera adeguata e disaggregare le cellule. Pipette alternativi potrebbero essere utilizzati fornito il foro e taglio sarebbe equivalente al P1000 ottimale.
Quando l'ordinamento 28 di FACS hpf, se una cella stereotypical ciclo profilo non è ottenuto, regolare la tensione sul laser PI affinché il picco G1 è centrato intorno a 50K. I guadagni per il FSC e SSC potrebbero anche bisogno di essere regolato in modo che le popolazioni di gating e cella appaiono simile a Figura 1. Se c'è ancora difficoltà, si deve attivare il filtro ND, e le tensioni FSC e SSC regolate per garantire la maggior parte delle cellule sono all'interno del cancello. Dovrebbe essere ovvio che ci sia PI che macchia, come ci sarà un intervallo che si approssima a doppio per il PI-A. L'istogramma dovrebbe mostrare due picchi chiari, un grande picco a 2N DNA per le cellule di G1 e un più piccolo picco alla doppia intensità per il DNA di 4N contenuto delle cellule in G2/M. Se questo ancora non si ottiene non ci può essere un numero basso di cellule intatte, problemi con la colorazione di PI o problemi con la fissazione e il protocollo dovrà essere ottimizzato di conseguenza.
Cellule da embrioni in anticipo (prima del 10 hpf) non darà profili tipico ciclo cellulare, come un'alta percentuale di queste cellule sono in fase S. Questi campioni possono essere trattati come campioni di fase S e rispetto a un riferimento di G1 da 24 embrioni hpf. Embrioni tra 10 hpf e 24 hpf possono dare profili intermedi ciclo cellulare e può essere ordinata se lo si desidera. La popolazione di S-fase può anche essere identificata e filtrata per incorporare gli analoghi della timidina come EDU (5-Etinil-2'-deoxyuridine) o BrdU (bromodeossiuridina) in brevi impulsi prima della fissazione, tuttavia, questo non è necessario per la determinazione accurata di tempo di replicazione.
Idealmente la preparazione libreria deve essere iniziata con 1 µ g di DNA. Teoricamente ~ 300.000 cellule ordinate produrrebbe circa 1 µ g di DNA (stima di ~3.3 pg/nucleo), tuttavia, c'è sempre perdita in tutto il protocollo. 500.000 a 1 milione di cellule ordinate dovrebbero dare più di 1 µ g di DNA. Un embrione di hpf 24 tipico avrà circa 18.000 cellule, 10-15% dei quali sarà in fase S. Gli embrioni nelle fasi precedenti dello sviluppo avranno considerevolmente meno cellule, ma avranno più elevate percentuali di cellule in fase S.
Esecuzione accurata purificazione e selezione dimensione usando i branelli magnetici è fondamentale per eliminare gli elementi indesiderati dalla preparazione libreria. Seguire attentamente le istruzioni del produttore e ottimizzazione supplementare può essere richiesto. Una piccola quantità di dimeri PCR non è in genere un problema importante, ma dimeri adattatore saranno sequenza molto efficiente quindi dovrebbe essere ridotto al minimo. Ciò può essere ottenuto regolando la scheda iniziale al rapporto DNA e esecuzione di dimensione accurata selezione durante la preparazione della biblioteca.
Sequenziamento di single-end può anche essere appropriato per le diverse esigenze sperimentali. Ad una estremità è più conveniente e fornisce la maggior parte delle informazioni richieste dal momento che questo approccio si basa sul conteggio delle letture; accoppiato-fine fornisce una superiore capacità di rimuovere duplicati ottici e PCR e di risolvere aree di sequenze ripetute e altre variazioni strutturali (che sono comuni nel genoma di zebrafish). La parte di analisi qui sotto si occuperà solo di sequenziamento accoppiato-fine letture. Più brevi letture di sequenziamento possono anche essere opportuna. Ciò fornirà un numero maggiore di letture ad un costo inferiore, ma la legge possono essere più difficile eseguire il mapping. La parte di analisi qui sotto è ottimizzata per 100 bp letture, e potrà essere necessario se viene utilizzata la lunghezza più corta di lettura.
Questo protocollo può essere anche facilmente adattato per ulteriori usi del modello zebrafish. E ' già stato utilizzato per l'analisi dei zebrafish tailfin fibroblasti (cellule ZTF), una cella primaria cultura riga isolata da tailfin di zebrafish adulto. Per studiare i tipi di cellule isolate da zebrafish, marcatori transgenici possono essere utilizzati per identificare e isolare i tipi di cellule individuali prima di macchiatura e ordinamento per contenuto di DNA. Una strategia potenziale sarebbe di utilizzare una linea transgenica esprimenti un marker fluorescente spinti da un promotore tessuto-specifica e di primo isolato cellule di FACS ordinamento, seguita la fissazione e l'ordinamento per il contenuto di DNA. Inoltre, l'uso di coloranti di DNA permeabili delle cellule può permettere per isolamento simultaneo di tipi cellulari individuali, come pure le popolazioni di fase G1 e S. Questi adattamenti di questo protocollo potrebbero anche consentire studi di temporizzazione replica in altri modelli in vivo .
Una possibilità emozionante per l'uso futuro del presente protocollo è quello di studiare il ruolo del tempo di replicazione in malattia. Ci sono diversi modelli di cancro in zebrafish, alcuni spontanea ed altri inducibile, che possono essere utilizzati per determinare quando si verificano modifiche nel tempo di replicazione durante oncogenesi. Questo permetterà di valutazione del tempo di replicazione di ruolo gioca nella progressione di malattia e di tumorigenesis. Inoltre, zebrafish sono un eccellente modello per lo screening di stupefacenti e può essere utilizzato per identificare farmaci che influiscono sulla regolazione tempi di replica, che hanno il potenziale per l'uso nel trattamento del cancro.
Zebrafish è un promettente nuovo modello per studiare il tempo di replicazione. Questo protocollo molti altri offrirà l'opportunità di utilizzare questo modello nello studio del ruolo del tempo di replicazione nello sviluppo e nella malattia. Questo protocollo può essere adattato per l'uso con altri in vivo sistemi di modello di sviluppo, come Drosophila melanogaster e Xenopus laevise sguardo in avanti al futuro i risultati da questi e da altri organismi.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato supportato dal National Institute of General Medical Sciences del National Institutes of Health attraverso concede 5P20GM103636-02 (tra cui il supporto core citometria a flusso) e 1R01GM121703, come pure premi dal centro di Oklahoma per cellule staminali adulte. Ricerca.
Materiali
| Name | Company | Catalog Number | Comments |
| NaCl | Fisher Scientific | BP358-10 | |
| KCl | Fisher Scientific | P217-500 | |
| CaCl2 | Fisher Scientific | C79-500 | |
| MgSO4 | EMD Millipore | MMX00701 | |
| NaHCO3 | Fisher Scientific | BP328-500 | |
| Pronase | Sigma | 10165921001 | protease solution |
| Phosphate buffered saline (PBS) | Sigma | D1408 | |
| Ethanol (EtOH) | KOPTEC | V1016 | |
| Bovine serum albumin (BSA) | Sigma | A9647-100G | |
| Propidium Iodide (PI) | Invitrogen | P3566 | |
| Tris-HCl | Fisher Scientific | BP153-500 | |
| EDTA | Sigma | E9844 | |
| SDS | Santa Cruz | sc-24950 | |
| Proteinase K | NEB | P8107S | |
| Phenol:Chloroform | Sigma | P3803-100ML | |
| Sodium acetate | J.T.Baker | 3470 | |
| Glycogen | Ambion | AM9510 | |
| RNase A | Thermo Scientific | EN0531 | |
| Quanit-iT | Invitrogen | Q33130 | Reagents for fluorescence-based DNA quantification |
| Covaris AFA microTUBE | Covaris | 520045 | specialized tube for sonication |
| Covaris E220 Sonicator | Covaris | E220 | focused ultrasonicator |
| Agilent 4200 Tapestation | Agilent | G2991AA | automated electrophoresis machine |
| D1000 ScreenTape | Agilent | 5067-5582 | Reagents for automated electrophoresis machine |
| NEBNext Ultra DNA Library Prep Kit for Illumina | NEB | Cat#E7370L | DNA library preparation kit |
| NEBNext Multiplex Oligos Kit for Illumina (Index Primers Set 1) | NEB | Cat#E7335S | multiplex oligos for DNA library preparation kit |
| NEBNext Multiplex Oligos Kit for Illumina (Index Primers Set 2) | NEB | Cat#E7500S | additional multiplex oligos for DNA library preparation kit |
| NEBNext Library Quant Kit for Illumina | NEB | E7630L | quantification kit for library preparation |
| Agencourt AMPure XP beads | Beckman Coulter | A63882 | magnetic beads |
| Illumina HiSeq 2500 | Illumina | SY–401–2501 | next generation DNA sequencing platform |
| 40 µm Falcon Nylon Cell Strainer | Fisher Scientific | 08-771-1 | |
| VWR Disposable Petri Dish 100 x 25 mm | VWR | 89107-632 | |
| 6.0 mL Syringe for Nichiryo Model 8100 | VWR | 89078-446 | |
| Posi-Click Tubes, 1.7 mL, Natural Color | Denville Scientific | C2170 (1001002) | Dnase/Rnase free |
| Vortex Genie 2 | Scientific Industries | SI-0236 | |
| Wash Bottles | VWR | 16650-022 | Low-Density Polyethylene, Wide Mouth |
| Strainer | VWR | 470092-440 | 6.9 cm, fine mesh |
| Corssing tank | Aquaneering | ZHCT100 | individual breeding tank |
| iSpawn | Techniplast | N/A | large breeding tank |
| FACSAria II | BD biosciences | N/A | cell sorting machine |
| Wild M5a steromicroscope | Wild Heerbrugg | N/A | dissecting microscope |
| Qubit 3 Fluorometer | Thermo Scientific | Q33216 | quantitative fluorescence-based method for determining DNA concentration |
| Matlab | Mathworks | version 2017a | |
| Matlab Statistics Toolbox | Mathworks | version 11.1 | |
| Matlab Curve Fitting Toolbox | Mathworks | version 3.5.5 |
Riferimenti
- Rhind, N., Gilbert, D. M. DNA replication timing. Cold Spring Harb Perspect Biol. 5 (8), a010132(2013).
- Pope, B. D., et al. Topologically associating domains are stable units of replication-timing regulation. Nature. 515 (7527), 402-405 (2014).
- Rivera-Mulia, J. C., et al. Dynamic changes in replication timing and gene expression during lineage specification of human pluripotent stem cells. Genome Res. 25 (8), 1091-1103 (2015).
- Hiratani, I., et al. Global reorganization of replication domains during embryonic stem cell differentiation. PLoS Biol. 6 (10), e245(2008).
- Hiratani, I., et al. Genome-wide dynamics of replication timing revealed by in vitro models of mouse embryogenesis. Genome Res. 20 (2), 155-169 (2010).
- Koren, A., et al. Differential relationship of DNA replication timing to different forms of human mutation and variation. Am J Hum Genet. 91 (6), 1033-1040 (2012).
- Ryba, T., et al. Abnormal developmental control of replication-timing domains in pediatric acute lymphoblastic leukemia. Genome Res. 22 (10), 1833-1844 (2012).
- Sima, J., Gilbert, D. M. Complex correlations: replication timing and mutational landscapes during cancer and genome evolution. Curr Opin Genet Dev. 25, 93-100 (2014).
- Veldman, M. B., Lin, S. Zebrafish as a developmental model organism for pediatric research. Pediatr Res. 64 (5), 470-476 (2008).
- Link, B. A., Megason, S. G. Zebrafish as a Model for Development. Sourcebook of Models for Biomedical Research. , 103-112 (2008).
- Siefert, J. C., Clowdus, E. A., Sansam, C. L. Cell cycle control in the early embryonic development of aquatic animal species. Comp Biochem Physiol C Toxicol Pharmacol. 178, 8-15 (2015).
- Hill, A. J., Teraoka, H., Heideman, W., Peterson, R. E. Zebrafish as a model vertebrate for investigating chemical toxicity. Toxicol Sci. 86 (1), 6-19 (2005).
- Dooley, K., Zon, L. I. Zebrafish: a model system for the study of human disease. Curr Opin Genet Dev. 10 (3), 252-256 (2000).
- Santoriello, C., Zon, L. I. Hooked! Modeling human disease in zebrafish. J Clin Invest. 122 (7), 2337-2343 (2012).
- Siefert, J. C., Georgescu, C., Wren, J. D., Koren, A., Sansam, C. L. DNA replication timing during development anticipates transcriptional programs and parallels enhancer activation. Genome Res. 27 (8), 1406-1416 (2017).
- Koren, A., et al. Genetic variation in human DNA replication timing. Cell. 159 (5), 1015-1026 (2014).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn. 203 (3), 253-310 (1995).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon