Method Article
Combinando la cristallografia a raggi x con piccolo angolo x-ray Scattering per modellare le regioni non strutturate di Nsa1 da S. Cerevisiae
In questo articolo
Riepilogo
Questo metodo descrive la clonazione, espressione e purificazione di Nsa1 ricombinante per determinazione strutturale di cristallografia a raggi x e small-angle x-ray scattering (SAXS) ed è applicabile per l'analisi strutturale di ibrido di altre proteine che contiene sia ordinato e disordinati domini.
Abstract
Determinazione della struttura Full-Length del fattore di assemblaggio del ribosoma Nsa1 da Saccharomyces cerevisiae (S. cerevisiae) è impegnativo a causa della disordinata e proteasi labile C-terminale della proteina. Questo manoscritto descrive i metodi per purificare ricombinante Nsa1 da S. cerevisiae per l'analisi strutturale di cristallografia a raggi x sia SAXS. Cristallografia a raggi x è stato utilizzato per risolvere la struttura del dominio N-terminale WD40 ben ordinato di Nsa1, e poi SAXS è stato utilizzato per risolvere la struttura del C-terminale di Nsa1 in soluzione. Dati di scattering di soluzione è stato raccolto da Full-Length Nsa1 in soluzione. Le ampiezze di scattering teorici sono state calcolate dalla struttura di cristallo ad alta risoluzione del dominio WD40, e poi una combinazione di corpo rigido e ab-initio modellazione ha rivelato il C-terminale di Nsa1. Attraverso questo approccio ibrido è stata ricostruita la struttura quaternaria della proteina intera. I metodi presentati qui dovrebbero essere generalmente applicabili per la determinazione strutturale di ibrido di altre proteine composta da un mix di domini strutturati e non strutturati.
Introduzione
I ribosomi sono macchine di grande ribonucleoproteina che svolgono il ruolo essenziale di tradurre mRNA in proteine in tutte le cellule viventi. I ribosomi sono composti da due subunità che sono prodotte in un processo complesso chiamato ribosoma biogenesi1,2,3,4. Assemblaggio del ribosoma eucariotico si basa sull'aiuto di centinaia di assemblaggio ribosomal essenziali fattori2,3,5. Nsa1 (Nop7 associato 1) è un fattore di assemblaggio ribosoma eucariotico che sia specificatamente richiesto per la produzione delle subunità ribosomali6, ed è noto come WD-ripetizione contenente 74 (WDR74) in più alti organismi7. WDR74 è stato indicato per essere richiesto per la formazione di blastocisti in topi8e il promotore di WDR74 è frequentemente mutato in cancro cellule9. Tuttavia, la funzione e i meccanismi precisi di Nsa1/WDR74 nell'assembly del ribosoma sono ancora in gran parte sconosciuti. Per iniziare a scoprire il ruolo di Nsa1/WDR74 durante la maturazione del ribosoma eucariotico, sono state effettuate analisi strutturali multiple, compreso cristallografia a raggi x e piccolo angolo x-ray scattering (SAXS)10.
Cristallografia a raggi x, spettroscopia di risonanza magnetica nucleare (NMR), microscopia elettronica e SAXS sono tutti importanti tecniche per studiare la struttura macromolecolare. Dimensioni, forma, disponibilità e stabilità delle influenze di macromolecole il metodo di biologia strutturale per cui una particolare macromolecola sarà meglio adatto, tuttavia combinando tecniche multiple attraverso un approccio di cosiddetti "ibridi" sta diventando un strumento sempre più utile di11. In particolare la cristallografia a raggi x e SAXS sono complementari e potenti metodi per la determinazione strutturale di macromolecole12.
Cristallografia fornisce strutture atomiche ad alta risoluzione che vanno da piccole molecole a grande macchinario cellulare come il ribosoma e ha portato a numerose scoperte nella comprensione delle funzioni biologiche delle proteine e altro macromolecole13. Inoltre, progettazione di farmaci basati su struttura sfrutta la potenza delle strutture cristalline per docking molecolare di metodi computazionali, aggiungendo una dimensione critica per droga scoperta e sviluppo14. Nonostante la sua ampia applicabilità, sistemi flessibili e disordinati sono difficili da valutare di cristallografia poiché impaccamento cristallino può essere ostacolato o mappe di densità elettronica potrebbero essere incomplete o di scarsa qualità. Al contrario, SAXS è un approccio strutturale a bassa risoluzione e basati su soluzioni in grado di descrivere sistemi flessibili che vanno dal loop disordinati e stazione termini a proteine intrinsecamente disordinati12,15,16. Considerando che è compatibile con una vasta gamma di particelle di dimensioni12, SAXS può lavorare sinergico con cristallografia per ampliare la gamma di domande biologiche che possono essere affrontate dagli studi strutturali.
Nsa1 è adatto per un approccio strutturale ibrida perché contiene un dominio ben strutturato di WD40 seguito da una C-terminale funzionale, ma flessibile che non è assoggettabile a metodi di cristallografia a raggi x. Di seguito è un protocollo per la clonazione, espressione e purificazione di S. cerevisiae Nsa1 per determinazione strutturale ibrida di cristallografia a raggi x e SAXS. Questo protocollo può essere adattato per studiare le strutture di altre proteine che sono costituiti da una combinazione di regioni ordinate e disordinate.
Protocollo
1. recombinant della proteina produzione e purificazione del Nsa 1
- Nsa1 Expression Design di plasmide e clonazione
- Ottenere o acquistare DNA genomic di S. cerevisiae .
- PCR amplificano le sequenze bersaglio di Nsa1 (Nsa1FL, residui 1-463) e C-terminale troncato Nsa1 (Nsa1ΔC, residui 1-434) con appositi primer usando il DNA genomic isolato da S. cerevisiae e una temperatura di fusione circa 60 ° C con un tempo di estensione di 1-2 min. Per amplificare Nsa1 sono stati utilizzati i seguenti primer:
SC_Nsa1_FLFw:CGCCAAAGGCCTATGAGGTTACTAGTCAGCTGTGTGGATAG
SC_Nsa1_FLRv:AATGCAGCGGCCGCTCAAATTTTGCTTTTCTTACTGGCTTTAGAAGCGC
SC_Nsa1ΔcFw:GGGCGCCATGGGATCCATGAGGTTACTAGTCAGCTGTGTGG
SC_Nsa1ΔcRv: GATTCGAAAGCGGCCGCTTAAACCTTCCTTTTTTGCTTCCC - Subclone Nsa1 nel pHMBP di vettore di espressione Escherichia coli (Escherichia coli) contenente un N-terminale 6 X-istidina tag seguita dal maltosio Binding Protein (MBP) e un sito di proteasi Tobacco Etch Virus (TEV) utilizzando tecniche di clonazione standard17 .
- Utilizzare sequenziamento del DNA per verificare che Nsa1 è stato clonato nel telaio con l'etichetta di sua MBP del N-terminale.
- Espressione della proteina Nsa1
- Trasformare il plasmid(s) di espressione in un adatto Escherichia coli ceppo di espressione con un sistema basato su promotore T7. Piastra trasformanti su piastre di agar LB contenente ampicillina di 100 µ g/mL e incubare la piastra invertita durante la notte a 37 ° C.
- Inoculare una cultura di 50 mL di LB con ampicillina 100 di µ g/mL dalla piastra di trasformazione e crescere in una notte con agitazione a 200 giri/min a 37 ° C.
- Inoculare 3 x 1000 mL di LB in Fernbach boccette con ampicillina µ g/mL 100 con 15 mL della coltura durante la notte e coltivarla con agitazione a 200 giri/min a 37 ° C.
Nota: Per soluzione di struttura proteica, incorporazione di selenomethionyl (SeMet) possa essere acquisiti coltivando cellule in medium minimo M9 che è completato con SeMet e una miscela dell'aminoacido di inibire la produzione di metionina prima dell'induzione, al contrario di LB media 18. - Inducono l'espressione di Nsa1 quando il OD600 raggiunge ~0.8 con aggiunta di isopropile β-D-1 thiogalactopyranoside (IPTG) ad una concentrazione finale di 1 mM, seguita da incubazione a 25 ° C durante la notte con agitazione a 200 giri/min.
- Raccogliere le cellule mediante centrifugazione a 4 ° C per 15 min a 5.050 x g.
Nota: Le cellule possono essere conservate a lungo termine a-80 ° C o utilizzate immediatamente per la purificazione della proteina.
- Purificazione della proteina Nsa1
- Risospendere le cellule in 25 mL di tampone di lisi (50 mM Tris-HCl, pH 7.5, 500 mM NaCl, 10% glicerolo, 10mm MgCl2) pre-refrigerati a 4 ° C, contenente una compressa di inibitore di proteasi privo di EDTA.
- Lisare cellule di sonicazione a 4 ° C (durata 7 min, 2 s il ciclo, 2 s fuori ciclo; ampiezza: 70%).
- Chiarire lisato mediante centrifugazione a 26.900 x g per 45 min a 4 ° C.
- Applicare chiarito lisato a una colonna di flusso di gravità caricata con 10 mL di resina di affinità immobilizzata cobalto, equilibrato con tampone di lisi.
- Consentire il supernatante di passare sopra la resina dalla gravità flusso a 4 ° C e lavare la resina due volte con 100 mL di tampone di lisi.
- Eluire Nsa1 con 20 mL di tampone di eluizione (50 mM Tris-HCl pH 7.5, 500 mM NaCl, 5 mM MgCl2, 5% glicerolo e imidazolo 200 mM).
- Prendere 15 μL dell'eluato ed eseguirlo su un gel di SDS-PAGE 4-15% per verificare che la proteina è eluita dalla resina di affinità (Figura 1A).
- Rimuovere il tag MBP di digestione della proteasi TEV. Aggiungere 1 mL di TEV proteasi19 (1 mg/mL di brodo) per l'eluizione di resina di affinità Nsa1 e mantenere in incubazione a 4 ° C durante la notte.
- Concentrato di MBP spaccati Nsa1 a ~ 5 mL con un filtro centrifugo con un peso molecolare di tagliare fuori di 10 kDa.
- Applicare Nsa1 MBP-spaccati a una colonna di gel-filtrazione, equilibrato nel buffer (20 mM Tris-HCl pH 7.5, 100 mM NaCl, 1 mM MgCl2, 5% glicerolo e 1mm β-mercaptoetanolo) (Figura 1B).
- Analizzare le frazioni di colonna da gel filtrazione per verificare che il MBP è spaccati e separate da Nsa1 da esecuzione 15 μL esempi su un gel di SDS-PAGE 4-15% (Figura 1B).
- Combinare le frazioni contenenti Nsa1 e concentrarsi a 8 mg/mL con un filtro centrifugo con un peso molecolare tagliato fuori di 10 kDa.
- Determinare la concentrazione di proteina misurando l'assorbanza a 280 nm in uno spettrofotometro utilizzando l'estinzione coefficiente 42530 M–1cm–1. Utilizzare proteine immediatamente per le prove di screening e cristallizzazione proteolitiche.
2. cristallizzazione e proteolitici Screening del Nsa 1
- Screening di cristallo di matrice sparsa di Nsa1
- Centrifugare a 500 μL dello stock di 8 mg/mL di Nsa1 a 16.000 x g a 4 ° C per 10 min.
- Installazione di prove di cristallizzazione utilizzando un robot di cristallizzazione e schermi a cristalli matrice sparsa. Riempire il serbatoio di ben 96 vassoi con 30 μl dei reagenti schermo singolo cristallo da schermi di cristallizzazione di matrice sparsa. Installazione di gocce di seduta con il robot mescolando 250 nL della soluzione bene con 250 nL della soluzione della proteina.
- Le piastre di cristallizzazione con nastro adesivo ed incubare li a 25 ° C.
- Ispezionare le piastre ogni due giorni per le prime 2-3 settimane con uno stereomicroscopio.
- Verificare che i potenziali hit di cristalli contengono proteine con un microscopio di UV.
Nota: Per Nsa1 due forme di cristallo differenti (cubi e ortorombico, Figura 2A) sono state ottenute entro 1 settimana dalla seguenti schermate: JCSG + condizione B11 (1,6 M sodio citrato tribasico disidratare, pH 6,5) e guidata precipitante sinergia schermo blocco 2 C11 di circostanza (20.1%(v/v) PEG 1500, 13.4%(v/v) PEG 400, 0.1 M Tris HEPES/idrossido di sodio, pH 7.5).
- Proteolitici Screening
Nota: Durante l'ottimizzazione della cristallizzazione, si scoprì che i cristalli ortorombico di Nsa1 ha presentato come conseguenza di clivaggio proteolitico e i cristalli non possono essere duplicati utilizzando la proteina full-length. Utilizzando una combinazione di proteolisi limitata accoppiata alla spettrometria di massa, è stato determinato che il C-terminale di Nsa1 era sensibile alla proteolisi e rimozione della coda C-terminale è stato richiesto per la successiva riproduzione del modulo (ortorombico Nsa1ΔC).- Preparare 1 mg/mL in soluzioni di riserva di proteasi del seguente proteasi α-chimotripsina, tripsina, elastasi, papaina, subtilisina ed endoproteinase Glu-C.
- Creare 01:10, 1: 100 e 1: 1000 diluizioni di ogni 1 mg/mL di brodo di proteasi con tampone di diluizione (10 mM HEPES, pH 7.5, 500 mM di cloruro di sodio).
- Pipettare 1 μL di stock di proteasi (01:10, 1: 100 e 1: 1000) in 9 aliquote del μL della proteina (1 mg/mL) per ogni proteasi essere proiettato.
- Incubare la soluzione a 37 ° C per 1 h.
- Fermare la reazione aggiungendo 10 μL di tampone del campione 2 x SDS-PAGE e calore la reazione a 95 ° C per 5 min.
- Analizzare il digest eseguendoli su un gel di SDS-PAGE 4-15% (Figura 2B).
- Identificare i domini proteasi resistente della proteina dall'analisi di spettrometria di massa in gel.
- Rimuovere le regioni proteasi-labile da proteina bersaglio mediante la creazione di costrutti di espressione troncato e seguendo il protocollo di clonazione, espressione e purificazione descritto in precedenza.
- Ottimizzazione di cristallizzazione
- Preparare o ottenere soluzioni dei seguenti reagenti iniziale cristallizzazione: 1,6 M sodio citrato tribasico disidratare pH 6.5, 100%(v/v) PEG 400, 50%(v/v) PEG 1500, 1m HEPES/idrossido di sodio, pH 7.5.
- Preparare una soluzione stock di Nsa1FL e Nsa1ΔC a 8 mg/mL come descritto sopra.
- Ottimizzare i cristalli cubici Nsa1 (Nsa1FL).
- Preparare una schermata di griglia 24 pozzetti con pozzetti contenenti 500 µ l con una pendenza di 1-1.6 M di citrato di sodio con pH 6.5 da una soluzione madre di 1,6 M sodio citrato tribasico con pH 6,5.
- Mescolare 1 µ l di proteina con 1 µ l di soluzione ben e posto il composto su un vetrino siliconato. Attentamente Inverti il vetrino di copertura sopra il pre-ingrassate bene e assicurarsi che sono ben sigillato, ma fate attenzione a non disturbare la goccia o rompere il vetrino di copertura. Ripetere questo processo fino a quando il vassoio è pieno e quindi conservare a 25 ° C.
Nota: Piccoli cristalli cubici dovrebbero apparire in 2-7 giorni. - Preparare un brodo di microseed dei cristalli cubici. Utilizzare un ciclo di nylon montato per trasferire ~ 10 piccoli cristalli cubici a una micro-centrifuga 1,5 mL contenente una perlina piccola e 50 µ l di 1,6 M sodio citrato tribasico wit a pH 6,5.
- Vortex la provetta di micro-centrifuga da 1,5 mL ad alta velocità (~ 3000 giri/min) per 1 min.
- Fare una serie di diluizioni seriali 10 volte delle riserve di semi con 1,6 M sodio citrato tribasico e vortice la miscela accuratamente per 5 s.
- Riempire ogni bene di una griglia di 24 pozzetti, schermo con 500 µ l di 1,6 M sodio citrato tribasico pH 6.5.
- Ottimizzare le condizioni di microseeding mediante l'istituzione di gocce con diversi rapporti di proteine con le soluzioni di ceppo (Figura 3A). Più grandi cristalli cubici di qualità elevata diffrazione dovrebbero apparire in 2-5 giorni (Figura 3).
- Ottimizzare i cristalli ortorombico (Nsa1ΔC)
- Preparare una schermata di griglia con 500 µ l in ogni pozzetto con 24 diverse condizioni (Figura 3B) dalle soluzioni di riserva di 50%(v/v) PEG 1500 e 100%(v/v) PEG 400. Oltre a gradienti di PEG 1500 e PEG 400, ciascun bene anche dovrebbe contenere 0,1 M HEPES/sodio idrossido a pH 7,5.
- Mescolare 1 µ l di soluzione proteica con 1 µ l di soluzione ben su un vetrino siliconato. Attentamente Inverti il vetrino di copertura sopra il pre-ingrassate bene e assicurarsi che sono ben sigillato. Ripetere questo processo finché non è stato riempito il vassoio intero. Conservare le piastre a 25 ° C.
Nota: Nsa1 ortorombico cristalli dovrebbero apparire in 2-7 giorni. - Ottimizzare ulteriormente i cristalli ortorombico di microseeding come descritto per i cristalli cubici (passaggi 2.3.3.3 per 2.3.3.7) utilizzando 500 µ l di 20.1%(v/v) PEG 1500, 13.4%(v/v) PEG 400 e 0,1 M HEPES/sodio idrossido pH 7.5 come la soluzione ben.
Nota: Nsa1 SeMet cristalli dovrebbero essere ottimizzati analoga ai cristalli nativi.
3. Nsa 1 struttura soluzione e raccolta di dati di diffrazione di raggi x
- Cryo-protezione di cristalli e raccolta di dati di diffrazione di raggi x
- Per i cristalli ortorombico (Nsa1ΔC), preparare una soluzione di crioprotettore 1 mL contenente 22.5%(v/v) PEG 1500, 15%(v/v) PEG 400 e 0,1 M HEPES/sodio idrossido pH 7,5.
- Riempire una schiuma Dewar con azoto liquido e pre-raffreddare un puck di cristallo. Usare cautela quando si lavora con azoto liquido e indossare occhiali e guanti protettivi.
- Attentamente e invertire il vetrino di copertura della cristallizzazione ben contenente cristalli sul palco di uno stereomicroscopio.
- Pipettare 2 µ l della soluzione crioprotettore su un vetrino di copertura nuovo.
- Allegare un ciclo di nylon montato di dimensioni adeguate per il cristallo di una bacchetta magnetica cryo.
- Con l'aiuto dello stereomicroscopio, trasferire rapidamente un cristallo alla soluzione crioprotettore con il ciclo di cryo montato.
- Almeno per 5 minuti nella soluzione di crioprotettore, portare il cristallo.
- Utilizzando l'aiuto dello stereomicroscopio, ciclo rapidamente il cristallo dalla soluzione crioprotettore e tuffo-congelamento in azoto liquido.
- Attendere che l'azoto liquido intorno all'anello per interrompere bollente e quindi rilasciare il ciclo dal tubo in una posizione specifica all'interno il puck di cristallo.
- Cubici Nsa1FL cristalli non devono essere prelevate e possono essere direttamente flash congelati (a seguito di passaggi 3.1.8-3.1.9 sopra).
- Sigillare il puck di cristallo utilizzando strumenti di cryo e trasferire in una canna di spedizione in un dewar pre-refrigerati. Conservare i cristalli in pucks/dewars fino alla raccolta dei dati.
- Se la raccolta di dati presso un sincrotrone, nave cristalli per il sincrotrone utilizzando un corriere a secco.
- Raccogliere i dati di diffrazione di raggi x seguendo tecniche standard20.
Nota: Per Nsa1 nativo e SAD (singola lunghezza d'onda dispersione anomala) set di dati sono stati raccolti a 100 K sulle linee di fascio di SER-CAT ID-22 e 22-BM della fonte del fotone avanzate all'Argonne National Laboratory (Chicago, IL). Il dataset di SAD Nsa1 è stato raccolto a λ = 0.97911 Å. Dati è stati registrati utilizzando un tempo di esposizione di s 1 e 0,5 ° oscillazioni. Il mosaicity di questi cristalli era tipicamente circa 0,3 °. - Elaborare e ridimensionare le immagini di diffrazione di raggi x per generare file di riflessione per ogni set di dati nel gruppo apposito spazio.
Nota: Nsa1 diffrazione DataSet sono stati elaborati con HKL200021. I cristalli cubici Nsa1 sono stati elaborati nel gruppo spazio P213 e i cristalli ortorombico sono stati elaborati in spazio gruppo P212121.
- Nsa1 struttura soluzione.
Nota: Ci sono diversi pacchetti di software di cristallografia che possono essere utilizzati per risolvere e perfezionare le strutture cristalline di cui Phenix e CCP422,23. Di seguito è riportato il protocollo per la soluzione della struttura Nsa1 utilizzando la suite di software Phenix22.- Analizzare il nativo e SAD scalati DataSet con phenix.xtriage22.
- Risolvere la struttura del Nsa1 con Phenix.autosol utilizzando il picco triste riflessione file22,24. Per eseguire il programma di AutoSol, immettere il numero di siti di selenometionina (siti = 9), il file di sequenza di fasta di Nsa1ΔC e la lunghezza d'onda utilizzata per la raccolta dati triste (λ = 0.97911 Å).
Nota: Dal dataset Nsa1 SAD, phenix.autosol dovrebbe essere in grado di determinare le fasi sperimentali e costruire la maggior parte del modello. - Aggiustamenti manuali al modello con Coot25, seguita da affinamento in phenix.refine22,26.
- Risolvere la struttura di cristallo ortorombico nativo ad alta risoluzione e il cristallo cubico da sostituzione molecolare usando Phaser27,28.
- Dopo soluzione struttura riuscita, controllare il modello e la densità dell'elettrone mappa folaga25.
- Costruire e perfezionare le strutture eseguendo cicli iterativi di raffinatezza in phenix.refine22,26 e modello di edificio in folaga25.
4. SAXS raccolta di dati, l'elaborazione e la modellazione
- Raccolta dati SAXS
Nota: Dati SAXS sono stati registrati per Full-Length S. cerevisiae Nsa1 verso la sorgente di luce avanzata, nella beamline di Sibille di alto-rendimento 12.3.1 Lawrence Berkeley National Laboratory, Berkeley, CA29.- Purificare Nsa1FL seguendo il protocollo descritto in precedenza. 24 h prima della spedizione di Nsa1FL per la beamline, eseguita proteina sopra una colonna di gel filtrazione pre-equilibrato in tampone A.
- Piscina le frazioni contenenti Nsa1 e determinare la concentrazione di proteine, come descritto in precedenza.
- Preparare 30 aliquote del μl di una serie di concentrazione di Nsa1 da 1 a 6,2 mg/mL Tampone desiderata.
- Trasferire una micropiastra ben chiaro completo gonna 96 insieme ai controlli da solo tampone A 20 μl di ogni serie di concentrazione di Nsa1.
Nota: Dati SAXS raccolti su soluzioni diluite con diverse concentrazioni di campione purificato per evitare l'aggregazione, repulsione fra le particelle e gli effetti di danni di radiazione. - Sigillare la micropiastra con un silicone mat e nave durante la notte a 4 ° C per la beamline di tenuta.
- Memorizzare la micropiastra a 4 ° C fino alla raccolta dei dati.
- Immediatamente prima di raccolta dei dati, girare la piastra a 3200 x g per 10 min a 4 ° C per rimuovere potenziali aggregati e bolle d'aria.
- Registrare dati SAXS.
Nota: Per Nsa1, SAXS dati sono stati registrati per il buffer prima e dopo ogni serie di concentrazioni di proteina. Trenta-tre analisi consecutive di 0,3 s sono stati raccolti per Nsa1FL sopra una serie di concentrazioni (1 a 6,2 mg/mL) a 10 ° C. - Eseguire la sottrazione di buffer per i dati SAXS.
Nota: Per Nsa1 sottrazione di buffer è stato fatto automaticamente presso la beamline ma sottrazione di buffer può essere eseguita anche utilizzando software di riduzione di dati come dispersione30 e la suite ATSAS31. Sottrazione di buffer è una parte critica di analisi dei dati SAXS e cura deve essere presa per garantire che il buffer utilizzato per sottrazione è identico al buffer del campione di proteina. - Media il 33 consecutivi scansioni per creare un file di *ave.dat per ciascuna concentrazione.
Nota: Sovrapporre i 33 fotogrammi consecutivi per confrontare le curve. Modifiche per le curve di dispersione nel corso del tempo è spesso indicativa di danno da radiazione. Escludere questi fotogrammi da una media. Facendo la media delle scansioni consecutive Nsa1 è stato effettuato automaticamente presso la beamline Sibille.
- Elaborazione dati SAXS e confronto di serie di concentrazioni
Nota: Ci sono diversi pacchetti di software che possono essere utilizzati per analizzare i dati SAXS. Per Nsa1 il raggio di girazione e funzioni di distribuzione pairwise distanza sono stati determinati utilizzando PRIMUS32 e Gnom33 da ATSAS 2.7.2 suite31 e confrontato attraverso tutte le concentrazioni di proteina per assicurare che non c'era nessuna radiazione concentrazione-dipendente inter-particella interazioni10o danni.- Aprire una shell di terminale e passare alla directory contenente i dati SAXS.
- Lanciare il PRIMUS32 GUI all'interno della shell di terminale.
- All'interno della GUI di PRIMUS, caricare le curve di dispersione (* ave.dat).
- Utilizzare AutoRg per determinare il raggio di girazione (R,g) e inoltrare l'intensità dello scattering I(0), per ogni curva di dispersione (* ave.dat).
- Generare Kratky trame per ogni curva di dispersione (* ave.dat), per valutare il grado di compattezza.
- Utilizzare AutoGNOM33 per calcolare la funzione di distribuzione pairwise (chiamato anche P(r)) per ogni curva di dispersione (* ave.dat). Immettere una partenza Dmax ≈ 3 * Rg e ottimizzare il valore dimax D per ottenere una curva uniforme p (r). Durante l'ottimizzazione del Dmax, assicurarsi che la funzione è coerente con la curva di dispersione sperimentale verificando il valore di2 χ segnalati e valutando visivamente generale adatta per la curva di dispersione p (r) calcolato.
- Calcolare il peso molecolare di Nsa1 dalle curve di dispersione utilizzando il volume di correlazione34.
- Confrontare i parametri strutturali per ciascuna concentrazione garantire che non esistono danni da radiazioni o effetti di concentrazione-dipendente.
Nota: Gli effetti di concentrazione possono manifestarsi come un crescente Rg e Dmax in relazione a una crescente concentrazione nella proteina. Dispersione in avanti I(0) diviso per la concentrazione del campione anche deve rimanere costante in tutta la serie di concentrazione di proteina.
- Modellazione di SAXS
Nota: Per determinare la posizione del C-terminale di Nsa1FL, Nsa1 cristallo struttura10 (PDB 5SUM) e le curve di dispersione di SAXS sono state utilizzate per realizzare una combinazione di corpo rigido e ab-initio di modellazione, utilizzando i programmi mazzo 35 ed Ensemble ottimizzazione metodo (EOM)36 dal ATSAS software suite31. Il dominio di WD40 di Nsa1 è stato trattato come un corpo rigido, mentre il C-terminale di Nsa1 insieme a diversi cicli disordinati dal dominio WD40 sono stati modellati per adattarle ai dati sperimentali di SAXS.- Generare il file PDB di ingresso. Il file PDB deve essere suddivisa ogni volta residui sono mancanti dalla catena principale, e il file PDB non può contenere molteplici conformazioni, leganti, o molecole d'acqua.
- Eseguire il programma Pre_BUNCH35 per preparare l'input file PDB per mazzo (pre_bunch.pdb). Inserire la sequenza della proteina dell'obiettivo (*.seq), il numero di domini/PDB generato al punto 4.3.1, e ciascuno dei singoli file PDB generato sopra.
- Calcolare le ampiezze di scattering per ogni singolo file PDB utilizzando il programma CRYSOL37. Per eseguire l'input CRYSOL il singolo file PDB e la curva di dispersione sperimentale SAXS (*. dat). Questo genererà un file di ampiezze (* .alm).
- Eseguire il programma mazzo35 per modellare il dominio di WD40 di Nsa1 contro i dati SAXS utilizzando un approccio combinato di corpo e ab-initio rigido. Input file PDB da Pre_BUNCH (pre_bunch.pdb), la curva di dispersione sperimentale SAXS (*. dat) e l'ampiezza individuo (* .alm) i file per ogni file PDB parziale generato dal CRYSOL.
- Confrontare il valore di2 χ da partenza PDB (5SUM) con quello dal modello ab-initio generato dal mazzo utilizzando i dati sperimentali di SAXS.
Nota: Un modello di gruppo di successo dovrebbe avere un valore di2 χ significativamente inferiore rispetto al modello di partenza. La dispersione teorica del modello mucchio dovrebbe anche descrivere bene i dati sperimentali come giudicato dall'ispezione visiva della sovrapposizione tra la dispersione teorica del modello ai dati SAXS mazzo e il relativo valore di2 χ (Figura 4, pipeline di centro). - Controllare manualmente i file di output da mazzo in Pymol38 per sovrapporre la struttura di cristallo con il modello generato dal mazzo e la busta SAXS (Figura 4, pipeline di centro).
- Eseguire nuovamente il mazzo 10 - 20 volte per generare modelli indipendenti per confermare che i modelli sono simili.
Nota: Per Nsa1 c'era una gamma di valori χ2 da ~ 1 a 3 da 20 sedute indipendenti. - Modellazione di ensemble
Nota: come un approccio facoltativo per mazzo eseguita EOM36 o minimo Ensemble ricerca39 (MES). EOM e MES utilizzare approcci ensemble, che ben si adattano per le proteine con domini flessibile/regioni che sono in molteplici conformazioni.- Eseguire il programma EOM36 utilizzando il server online ATSAS. Per eseguire EOM, inserire il file PDB da 4.3.1, la sequenza della proteina dell'obiettivo (*.seq) e i dati sperimentali di SAXS (*. dat).
- Confrontare i valori di2 χ da partenza PDB (5SUM) con quello dall'ensemble generato da EOM utilizzando i dati sperimentali di SAXS.
Nota: Un ensemble EOM successo dovrebbe avere un valore di2 χ significativamente inferiore rispetto al modello di partenza. La dispersione teorica dell'ensemble dovrebbe anche descrivere bene i dati sperimentali come giudicato dall'ispezione visiva della sovrapposizione tra la dispersione teorica dell'ensemble ai dati SAXS e il relativo valore di2 χ (Figura 4, il diritto Pipeline). Anche, bisogna confrontare i valori χ2 dal mazzo ed EOM per determinare quale modello meglio descrive i dati sperimentali di SAXS. - Controllare manualmente i conformisti EOM in Pymol38 per sovrapporre la struttura di cristallo con i conformeri generati da EOM (Figura 4). Nota il numero totale di conformeri e la frazione di occupazione per ogni conformero che contribuisce alla curva di dispersione. Per molecole più rigidi, come Nsa1, il numero di conformeri dovrebbe essere piccolo (1-5) (Figura 4, pipeline di destra)36.
- Eseguire nuovamente EOM parecchie volte per garantire risultati coerenti.
Nota: Per Nsa1 il numero di conformeri era in genere 3 o 4 con χ2 valori che vanno da 0,1 a 0,3.
Risultati
Nsa1 era PCR amplificati da DNA genomic di S. cerevisiae e subclonato in un vettore contenente un tag di affinità di 6 x-istidina N-terminale seguito da MBP e un sito di proteasi TEV. Nsa1 fu trasformato in cellule di e. coli BL21 (DE3) e alti rendimenti dell'espressione della proteina sono stati ottenuti dopo induzione con IPTG e crescita a 25 ° C durante la notte (Figura 1A). Nsa1 è stato purificato per affinità su resina di affinità immobilizzata cobalto, seguito dal clivaggio MBP con proteasi TEV e definitivamente risolta da cromatografia di esclusione di formato (Figura 1B). Frazioni da cromatografia di esclusione di formato contenente Nsa1 erano riunite, concentrate a 8 mg/mL e quindi utilizzate per le prove di cristallizzazione con un robot di cristallizzazione. Schermi a cristalli matrice sparsa iniziale ha reso due forme di cristallo differenti di Nsa1, cubi e ortorombica (Figura 2A).
Durante l'ottimizzazione dei cristalli cubici e ortorombico, si scoprì che i cristalli ortorombico abbia presentato come il risultato di clivaggio proteolitico del Nsa1. Proteolisi limitata e spettrometria di massa sono stati usati per determinare la regione di Nsa1 che era sensibile alla proteolisi, ed è stato osservato che Nsa1 era sensibile a un gradiente di concentrazione di elastasi la proteasi (Figura 2B). Analisi di spettrometria di massa successiva ha confermato che questo degrado è derivato da perdita del C-terminale di Nsa1. Una serie di troncamenti del C-terminale di Nsa1 sono stati generati, per rimuovere il C-terminus sensibile proteolitico (Figura 2). I cristalli ortorombico potrebbero essere ripetuti con il troncamento diΔC (residui 1-434) Nsa1, che in definitiva è stato usato per la determinazione della struttura triste. I cristalli ortorombico potrebbero essere ripetuti anche trattando Nsa1FL con elastasi per 1 ora a 4 ° C prima della creazione di vassoi di cristallo.
I cristalli cubici di Nsa1 sono stati ottimizzati utilizzando Nsa1FL attraverso una combinazione di sfumature di citrato di sodio, accoppiato con microseeding (Figura 3A). Questo ha reso grandi cristalli cubici riproducibile, con un limite di diffrazione di circa 2.8 Å risoluzione (Figura 3, a sinistra). I cristalli ortorombico potrebbero essere ottimizzati solo usando le varianti di troncamento del C-terminale del Nsa1, variando i gradienti di concentrazione di PEG 1500 e PEG 400, combinato con microseeding, che ha reso grandi cristalli con un limite di diffrazione di circa 1,25 Å risoluzione (Figura 3A-C). Fasi sperimentali di Nsa1 sono stati determinati mediante SeMet-SAD da un SeMet-derivato di Nsa1ΔC10.
Il dominio di WD40 β-elica a sette pale N-terminale di Nsa1 era ben risolto in entrambe le strutture di cristallo cubico e ortorombico, tuttavia entrambe le strutture mancavano la densità dell'elettrone per il C-terminus di Nsa1. SAXS è stato poi utilizzato per determinare la posizione del dominio C-terminale manca di Nsa1 in soluzione. Dopo l'ottimizzazione della concentrazione del campione per la raccolta dati, la struttura atomica parziale è stata utilizzata per eseguire la modellazione di corpi rigidi e generare una ricostruzione ab-initio i componenti mancanti. Il modello è stato valutato in termini di bontà dell'adattamento per le curve di dispersione calcolati i dati sperimentali (Figura 4, pipeline di centro). Il dominio di WD40 di Nsa1 da solo non è una buona misura dei dati sperimentali SAXS, come testimoniano la discrepanza tra la curva di dispersione sperimentale con la curva di dispersione teorica, che è stata generata dalla struttura di cristallo PDB ID 5SUM (Figura 4, pipeline di sinistra). Oltre alla modellazione di corpi rigidi, modellazione ensemble inoltre è stata fatta. Questo prodotto un insieme di 3 o 4 conformeri di Nsa1 e ha un valore più basso di2 χ (Figura 4, pipeline di destra). La riduzione di discrepanza di modello (χ2) utilizzando la modellazione ensemble ha rivelato il campionamento conformazionale della coda Nsa1 C-terminale in soluzione.

Figura 1. Espressione e purificazione di Nsa1 con un tag di fusione X-His-MBP 6. (A) analisi di SDS-PAGE di espressione della proteina in BL21 (DE3) cellule a 25 ° C durante la notte e la prima fase di purificazione cobalto affinità con la resina. (B) cromatogramma di esclusione di dimensione rappresentante dopo la scissione TEV. Le frazioni dalla cromatografia di esclusione di formato sono state analizzate da SDS-PAGE. Le frazioni da picco 1 contenente Nsa1 sono stati raccolti e utilizzati per l'analisi strutturale. Clicca qui per visualizzare una versione più grande di questa figura.
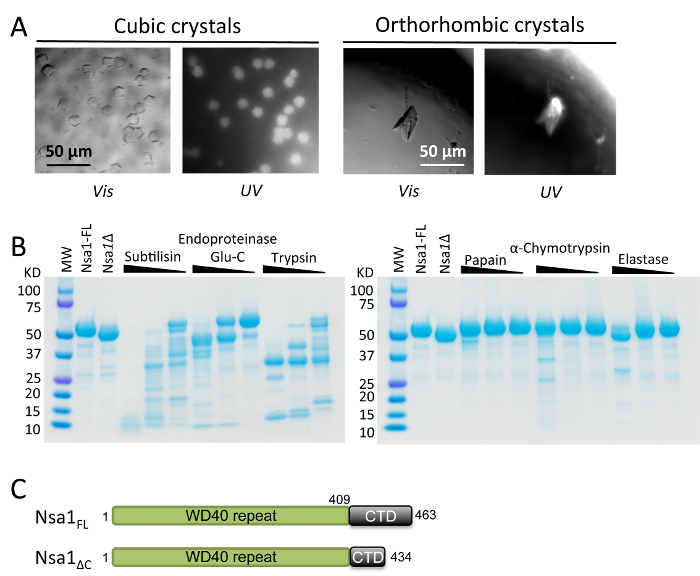
Figura 2. Il C-terminale di Nsa1 è sensibile alla proteolisi. (A) test di cristallizzazione iniziale di Nsa1 dato i due forme di cristalli diversi: ortorombico e cubi. Microscopio di UV è stato utilizzato per verificare che i cristalli contenevano proteico. Vis: Luce visibile, UV: microscopi UV. Barra della scala = 50 µm in ogni finestra. (B) proteolitici screening analizzati da SDS-PAGE. Tre diluizioni (01:10, 1: 100, 1: 1000) per ogni stock di proteasi (0.1, 0.01, 0,001 mg/mL) sono stati combinati con aliquote della proteina (1 mg/mL) ad essere proiettato. Resistente alle proteasi domini sono stati analizzati i da SDS-PAGE e spettrometria di massa dopo incubazione a 37 ° C per 60 min. Nsa1-FL: fresco purificato proteina, Nsa1Δ: purificato proteina conservata a 4 ° C per 3 settimane dopo che è stata osservata una forma degradata della proteina. Diagramma schematico (C) del Nsa1 Full-Length (superiore) e il troncamento del C-terminale costruire (inferiore). Clicca qui per visualizzare una versione più grande di questa figura.
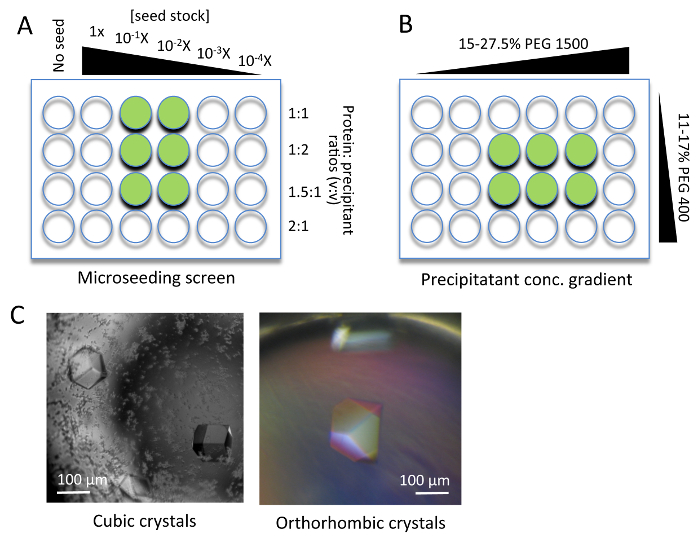
Figura 3. Ottimizzazione di cristallizzazione Nsa1. (A) un stock di seme è stato preparato da piccoli cristalli iniziale e usato per fare una serie di diluizioni (1 x ~ 1/104x) (microseeding). Mescolando 1 µ l di proteina con 1 µ l dello stock di seme diluito, i più grandi cristalli singoli è cresciuto entro una settimana. (B) gradiente di concentrazione precipitante per ottimizzazione ortorombico. (C) ottimizzato cristalli cubici e ortorombico, utilizzati per la raccolta dati. Cerchi verdi indicano la zona tipica dei vassoi di cristallo che ha prodotto raccolta dati cristalli di qualità. Clicca qui per visualizzare una versione più grande di questa figura.
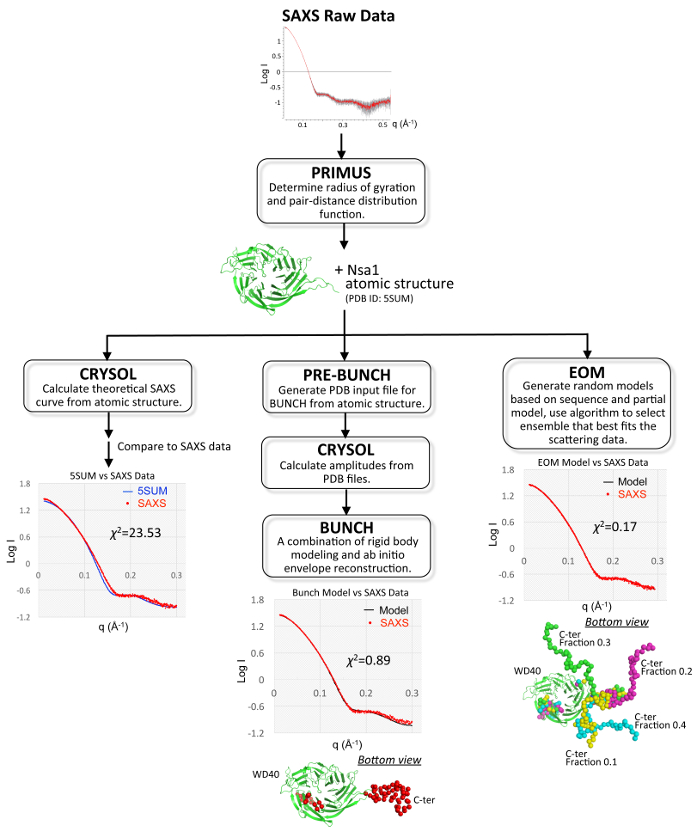
Figura 4. Schema di analisi SAXS Nsa1. Panoramica della pipeline utilizzata per elaborare dati SAXS e generare modelli di mazzo (centro) ed EOM (a destra). La pipeline di sinistra mostra la discrepanza tra la curva di dispersione sperimentale (cerchi rossi, concentrazione nella proteina: 6 mg/mL) con la curva di dispersione teorica (linea blu), che è stata generata dalla struttura di cristallo PDB ID 5SUM. I modelli sono stati valutati confrontando la curva di dispersione sperimentale SAXS (cerchi rossi, concentrazione nella proteina: 6 mg/mL) con la curva di dispersione derivata dal modello mucchio di Nsa1 (linea nera) o i conformisti EOM di Nsa1 (linea nera). In ogni modello, il dominio di WD40 è indicato in fumetto colorato in verde (PDB ID 5SUM), il C-terminale flessibile è mostrato in sfere colorate in rosso per mazzo e verde, magenta, ciano e giallo per i singoli conformeri EOM. La frazione di ogni conformero derivato da EOM è etichettata accanto al modello. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Usando questo protocollo, ricombinante Nsa1 da S. cerevisiae è stato generato per studi strutturali di cristallografia a raggi x sia SAXS. Nsa1 era ben educati in soluzione e cristallizzato in diverse forme cristalline. Durante l'ottimizzazione di questi cristalli, è stato scoperto che il C-terminale di Nsa1 era sensibile alla degradazione della proteasi. L'alta risoluzione, forma cristallina ortorombica solo potrebbe essere duplicato con varianti di troncamento di C-terminale di Nsa1, probabilmente perché il C-terminale flessibile di Nsa1 impedito impaccamento cristallino. La struttura del Nsa1 è stata risolta tramite cristallografia a raggi x ad alta risoluzione, ma il C-terminus non potrebbe essere costruito sia forma di cristallo, perché esso non è stato ordinato. La cristallografia è la prima tecnica per la determinazione di strutture di risoluzione atomica di macromolecole intorno al formato di Nsa1, tuttavia come con qualsiasi metodo, cristallografia hanno alcune limitazioni. Uno dei principali limiti della cristallografia è l'incapacità di risolvere disordinate regioni di proteine40,41.
Il C-terminale di Nsa1 è importante per la corretta localizzazione nucleolar della proteina, sottolineando la necessità di studiare la struttura10. Il C-terminale di Nsa1, è stato risolto da SAXS, una tecnica di biologia strutturale complementari per cristallografia a raggi x. Dati SAXS è stati registrati per Full-Length Nsa1 attraverso una serie di concentrazioni. Da questa serie di concentrazione, è stata determinata la concentrazione ottimale per la raccolta Nsa1 SAXS ed elaborazione. SAXS dati sono stati registrati per Nsa1 alle 6, 4.5 e 3,0 mg/mL. La regione Guinier, funzione p (r) e peso molecolare sono stati determinati in tutta la serie di concentrazione per garantire che il campione era ben educati e non aggregati nelle circostanze sperimentali testati. Per ricostruire la struttura Full-Length di Nsa1, l'ampiezza di scattering teorica è stata determinata dalla struttura di cristallo parziale e poi ab-initio metodi sono stati utilizzati per modellare il C-terminale flessibile. Da questo approccio ibrido, è stato determinato che il C-terminale flessibile di Nsa1 si estende verso l'esterno dal dominio WD40 ordinato.
Progressi nell'elaborazione di strumenti ha guidato la popolarità di SAXS per studi strutturali macromolecolari. SAXS misura il modello di dispersione dei raggi x da orientate casualmente proteine in soluzione per fornire informazioni strutturali a bassa risoluzione, tra cui massa molecolare e forma complessiva. Di conseguenza, SAXS è emerso come uno strumento di convalida potente struttura ortogonale per la cristallografia. Questo è in gran parte dovuto allo sviluppo di metodi di calcolo per calcolare la dispersione teorica delle strutture atomiche e confrontandole con sperimentale SAXS dati37. Utilizzando questo approccio, stato conformazionale, struttura quaternaria e assemblaggio di ordine più elevato osservato in un reticolo cristallino può essere paragonato alle caratteristiche strutturali della particella in soluzione. Inoltre, cicli disordinati e termini mancanti in strutture ad alta risoluzione determinate dalla cristallografia a raggi x possono essere modellati utilizzando dati di scattering di soluzione. Questo approccio strutturale ibrida utilizza la struttura di cristallo come un blocco di costruzione per la modellazione di SAXS-guida dei residui mancanti e ha dimostrato di essere efficace nella mappatura C-terminale di Nsa110, come pure altre macromolecole quali il virus di riossidazione A M1 matrice proteica42e morti-casella RNA chaperoni43. Software di modellazione avanzata basata su SAXS possa anche affrontare sistemi più complessi, quali proteine intrinsecamente disordinati, mappando il paesaggio conformazionale di questi sistemi utilizzando una serie di conformeri che insieme contribuiscono alla dispersione generale potenziale della particella in soluzione16,39. Presi insieme, recenti progressi nella strumenti di raccolta ed elaborazione dati SAXS contribuisce al successo di ibrido tra gli approcci di biologia strutturale per affrontare impegnativi sistemi biologici.
La combinazione di scattering di soluzione con strutture ad alta risoluzione è pronta a rispondere a domande importanti circa la flessibilità e la dinamica delle macromolecole44. Molte proteine, ad esempio Nsa1, hanno regioni dinamiche che sono importanti per la funzione biologica. In questo manoscritto è fornito un protocollo di modello che dettagli la combinazione di SAXS con determinazione della struttura ad alta risoluzione dalla cristallografia a raggi x. Oltre alla cristallografia a raggi x SAXS utilizzabile anche per complimentarmi con altre tecniche di biologia strutturale tra cui NMR, elettrone energia paramagnetica risonanza (EPR) e fluorescenza risonanza transfer (FRET), ulteriormente evidenziando l'importanza di SAXS come un complementare biologia strutturale tecnica45,46,47.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Dati di diffrazione sono stati raccolti a sud-est squadra regionale a accesso collaborativo (SER-CAT) ID-22 e 22-BM beamlines presso Advanced Photon fonte (APS), Argonne National Laboratory. I dati SAXS è stato raccolto sulle Sibille beamline presso Advance Light Source (ALS), Lawrence Berkeley National Laboratory. Vorremmo ringraziare il personale presso la beamline Sibille per il loro aiuto con elaborazione e raccolta dei dati remota. Siamo grati alla ricerca di spettrometria di massa National Institute di Environmental Health Sciences (NIEHS) e gruppo di sostegno per aiuto per determinare i limiti del dominio della proteina. Questo lavoro è stato supportato da noi programma nazionale di Istituto di salute intramurale ricerca; US National Institute of Environmental Health Sciences (NIEHS) (ZIA ES103247 a S. R. E.) e gli istituti canadesi di ricerca sanitaria (CIHR, 146626 a M.C.). Uso di APS è stata sostenuta dal dipartimento dell'energia statunitense, Office of Science, Office di base energia scienze sotto contratto n. W-31-109-ITA-38. Utilizzo di Advanced Light Source (ALS) è stata sostenuta dal direttore, Office of Science, ufficio di energia scienze di base, del US Department of Energy sotto contratto no. DE-AC02-05CH11231. Supporto aggiuntivo per la beamline Sibille SAXS deriva dal National Institute of Health project MINOS (R01GM105404) e un high-end strumentazione Grant S10OD018483. Vorremmo anche ringraziare Andrea Moon e Dr. Sara Andres per loro lettura critica di questo manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| Molecular Cloning of Nsa1 | |||
| pMBP2 parallel vector | Sheffield et al, Protein Expression and Purification 15, 34-39 (1999) | We used a modified version of pMBP2 which included an N-terminal His-tag (pHMBP) | |
| S. cerevisiae genomic DNA | ATCC | 204508D-5 | |
| Primers for cloning Nsa1 | |||
| SC_Nsa1_FLFw | IDT | CGC CAA AGG CCT ATGAGGTTACTAGTCAGCTGTGT GGATAG | |
| SC_Nsa1_FLRv | IDT | AATGCAGCGGCCGCTCAAATTTT GCTTTTCTTACTGGCTTTAGAAGC AGC | |
| SC_Nsa1_DeltaCFw | IDT | GGGCGCCATGGGATCCATGAGG TTACTAGTCAGCTGTGTGG | |
| SC_Nsa1_DeltaCRv | IDT | GATTCGAAAGCGGCCGCTTAAAC CTTCCTTTTTTGCTTCCC | |
| Recombinant Protein Production and Purification of Nsa1 | |||
| Escherichia coli BL21 (DE3) Star Cells | Invitrogen | C601003 | |
| pMBP- NSA1 and various truncations | Lo et al., 2017 | ||
| Selenomethionine | Molecular Dimensions | MD12-503B | |
| IPTG, Dioxane-Free | Promega | V3953 | |
| EDTA Free Protease Inhibitor Cocktail | Sigma-Aldrich | 4693159001 | |
| Sodium Chloride | Caledon Laboratory Chemicals | 7560-1-80 | |
| Magnesium Chloride hexahydrate | Sigma-Aldrich | M2670 | |
| Tris Buffer, 1 M pH7.5 | KD Medical | RGF-3340 | |
| Glycerol | Invitrogen | 15514-029 | |
| beta-mercaptoethanol | Sigma | M6250 | |
| 1M Imidazole, pH 8.0 | Teknova | I6980-06 | |
| Talon Affinity Resin | Clonetech | 635503 | |
| Amicon Ultra 15 mL Centrifugal Filter (MWCO 10K) | Millipore | UFC901024 | |
| HiLoad 16/600 Superdex 200 Prep Grade Gel Filtration Column | GE-Healthcare | 28989335 | |
| TEV Protease | Prepared by NIEHS Protein Expression Core | Expression plasmid provided by NCI (Tropea et al. Methods Mol Biology, 2009) | |
| 4-15% Mini-PROTEAN TGX Precast Protein Gels | BioRad | 456-8056 | |
| Crystallization, Proteolytic Screening | |||
| Crystal Screen | Hampton Research | HR2-110 | |
| Crystal Screen 2 | Hampton Research | HR2-112 | |
| Salt Rx | Hampton Research | HR2-136 | |
| Index Screen | Hampton Research | HR2-144 | |
| PEG/Ion Screen | Hampton Research | HR2-139 | |
| JCSG+ | Molecular Dimensions | MD1-37 | |
| Wizard Precipitant Synergy | Molecular Dimensions | MD15-PS-T | |
| Swissci 96-well 3-drop UVP sitting drop plates | TTP Labtech | 4150-05823 | |
| 3inch Wide Crystal Clear Sealing Tape | Hampton Research | HR4-506 | |
| Proti-Ace Kit | Hampton Research | HR2-429 | |
| PEG 1500 | Molecular Dimensions | MD2-100-6 | |
| PEG 400 | Molecular Dimensions | MD2-100-3 | |
| HEPES/sodium hydroxide pH 7.5 | Molecular Dimensions | MD2-011- | |
| Sodium Citrate tribasic | Molecular Dimensions | MD2-100-127 | |
| 22 mm x 0.22 mm Siliconized Coverslides | Hampton Research | HR3-231 | |
| 24 Well Plates with sealant (VDX Plate with Sealant) | Hampton Research | HR3-172 | |
| 18 mM Mounted Nylon Loops (0.05 mm to 0.5 mM) | Hampton Research | HR4-945, HR4-947, HR4-970, HR4-971 | |
| Seed Bead Kit | Hampton Research | HR2-320 | |
| Magnetic Crystal Caps | Hampton Research | HR4-779 | |
| Magnetic Cryo Wand | Hampton Research | HR4-729 | |
| Cryogenic Foam Dewar | Hampton Research | HR4-673 | |
| Crystal Puck System | MiTeGen | M-CP-111-021 | |
| Full Skirt 96 well Clear Plate | VWR | 10011-228 | |
| AxyMat Sealing Mat | VWR | 10011-130 | |
| Equipment | |||
| UVEX-m | JAN Scientific, Inc. | ||
| Nanodrop Lite Spectrophotometer | Thermo-Fisher | ||
| Mosquito Robot | TTP Labtech | ||
| Software/Websites | |||
| HKL2000 | Otwinoski and Minor, 1997 | ||
| Phenix | Adams et al., 2010 | ||
| Coot | Emsley et al., 2010 | ||
| ATSAS | Petoukhov et al., 2012 | https://www.embl-hamburg.de/biosaxs/atsas-online/ | |
| Scatter | Rambo and Tainer, 2013 | ||
| Pymol | The PyMOL Molecular Graphics System, Version 1.8 Schrödinger, LLC. | ||
| BUNCH | Petoukhov and Svergun, 2005 | ||
| CRYSOL | Svergun et al, 1995 | ||
| PRIMUS | Konarev et al, 2003 | ||
| EOM | Tria et al, 2015 |
Riferimenti
- Thomson, E., Ferreira-Cerca, S., Hurt, E. Eukaryotic ribosome biogenesis at a glance. J Cell Sci. 126 (Pt 21), 4815-4821 (2013).
- Woolford, J. L., Baserga, S. J. Ribosome biogenesis in the yeast Saccharomyces cerevisiae. Genetics. 195 (3), 643-681 (2013).
- Kressler, D., Hurt, E., Bassler, J. A Puzzle of Life: Crafting Ribosomal Subunits. Trends Biochem Sci. , (2017).
- Tomecki, R., Sikorski, P. J., Zakrzewska-Placzek, M. Comparison of preribosomal RNA processing pathways in yeast, plant and human cells - focus on coordinated action of endo- and exoribonucleases. FEBS Lett. , (2017).
- Kressler, D., Hurt, E., Bassler, J. Driving ribosome assembly. Biochim Biophys Acta. 1803 (6), 673-683 (2010).
- Kressler, D., Roser, D., Pertschy, B., Hurt, E. The AAA ATPase Rix7 powers progression of ribosome biogenesis by stripping Nsa1 from pre-60S particles. J Cell Biol. 181 (6), 935-944 (2008).
- Hiraishi, N., Ishida, Y., Nagahama, M. AAA-ATPase NVL2 acts on MTR4-exosome complex to dissociate the nucleolar protein WDR74. Biochem Biophy Res Co. 467 (3), 534-540 (2015).
- Maserati, M., et al. Wdr74 is required for blastocyst formation in the mouse. PLoS One. 6 (7), e22516 (2011).
- Weinhold, N., Jacobsen, A., Schultz, N., Sander, C., Lee, W. Genome-wide analysis of noncoding regulatory mutations in cancer. Nat Genet. 46 (11), 1160-1165 (2014).
- Lo, Y. H., Romes, E. M., Pillon, M. C., Sobhany, M., Stanley, R. E. Structural Analysis Reveals Features of Ribosome Assembly Factor Nsa1/WDR74 Important for Localization and Interaction with Rix7/NVL2. Structure. 25 (5), 762-772 (2017).
- Lander, G. C., Saibil, H. R., Nogales, E. Go hybrid: EM, crystallography, and beyond. Curr Opin Struc Biol. 22 (5), 627-635 (2012).
- Putnam, C. D., Hammel, M., Hura, G. L., Tainer, J. A. X-ray solution scattering (SAXS) combined with crystallography and computation: defining accurate macromolecular structures, conformations and assemblies in solution. Q Rev Biophys. 40 (3), 191-285 (2007).
- Jaskolski, M., Dauter, Z., Wlodawer, A. A brief history of macromolecular crystallography, illustrated by a family tree and its Nobel fruits. FEBS J. 281 (18), 3985-4009 (2014).
- Zheng, H., et al. X-ray crystallography over the past decade for novel drug discovery - where are we heading next?. Expert Opin Drug Dis. 10 (9), 975-989 (2015).
- Kikhney, A. G., Svergun, D. I. A practical guide to small angle X-ray scattering (SAXS) of flexible and intrinsically disordered proteins. FEBS Lett. 589 (19 Pt A), 2570-2577 (2015).
- Bernado, P., Mylonas, E., Petoukhov, M. V., Blackledge, M., Svergun, D. I. Structural characterization of flexible proteins using small-angle X-ray scattering. J Am Chem Soc. 129 (17), 5656-5664 (2007).
- Sheffield, P., Garrard, S., Derewenda, Z. Overcoming expression and purification problems of RhoGDI using a family of "parallel" expression vectors. Protein Expres Purif. 15 (1), 34-39 (1999).
- Doublie, S. Preparation of selenomethionyl proteins for phase determination. Methods Enzymol. 276, 523-530 (1997).
- Tropea, J. E., Cherry, S., Waugh, D. S. Expression and purification of soluble His(6)-tagged TEV protease. Methods Mol Biol. 498, 297-307 (2009).
- Wlodawer, A., Minor, W., Dauter, Z., Jaskolski, M. Protein crystallography for aspiring crystallographers or how to avoid pitfalls and traps in macromolecular structure determination. FEBS J. 280 (22), 5705-5736 (2013).
- Otwinowski, Z., Minor, W. Processing of X-ray diffraction data collected in oscillation mode. Macromolecular Crystallography, Pt A. 276, 307-326 (1997).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallogr D. 66 (Pt 2), 213-221 (2010).
- Winn, M. D., et al. Overview of the CCP4 suite and current developments. Acta Crystallogr D. 67 (Pt 4), 235-242 (2011).
- Terwilliger, T. C., et al. Decision-making in structure solution using Bayesian estimates of map quality: the PHENIX AutoSol wizard. Acta Crystallographica Section D. 65, 582-601 (2009).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Crystallogr D. 66 (Pt 4), 486-501 (2010).
- Afonine, P. V., et al. Towards automated crystallographic structure refinement with phenix.refine. Acta Crystallogr D. 68 (Pt 4), 352-367 (2012).
- McCoy, A. J. Solving structures of protein complexes by molecular replacement with Phaser. Acta Crystallogr D. 63 (Pt 1), 32-41 (2007).
- McCoy, A. J., et al. Phaser crystallographic software. J Appl Crystallogr. 40 (Pt 4), 658-674 (2007).
- Dyer, K. N., et al. High-throughput SAXS for the characterization of biomolecules in solution: a practical approach. Methods Mol Biol. 1091, 245-258 (2014).
- Forster, S., Apostol, L., Bras, W. Scatter: software for the analysis of nano- and mesoscale small-angle scattering. J Appl Crystallogr. 43, 639-646 (2010).
- Petoukhov, M. V., et al. New developments in the ATSAS program package for small-angle scattering data analysis. J Appl Crystallogr. 45, 342-350 (2012).
- Konarev, P. V., Volkov, V. V., Sokolova, A. V., Koch, M. H. J., Svergun, D. I. PRIMUS: a Windows PC-based system for small-angle scattering data analysis. J Appl Crystallogr. 36, 1277-1282 (2003).
- Svergun, D. I. Determination of the Regularization Parameter in Indirect-Transform Methods Using Perceptual Criteria. J Appl Crystallogr. 25, 495-503 (1992).
- Rambo, R. P., Tainer, J. A. Accurate assessment of mass, models and resolution by small-angle scattering. Nature. 496 (7446), 477-481 (2013).
- Petoukhov, M. V., Svergun, D. I. Global rigid body modeling of macromolecular complexes against small-angle scattering data. Biophys J. 89 (2), 1237-1250 (2005).
- Tria, G., Mertens, H. D., Kachala, M., Svergun, D. I. Advanced ensemble modelling of flexible macromolecules using X-ray solution scattering. IUCrJ. 2 (Pt 2), 207-217 (2015).
- Svergun, D., Barberato, C., Koch, M. H. J. CRYSOL - A program to evaluate x-ray solution scattering of biological macromolecules from atomic coordinates. J Appl Crystallogr. 28, 768-773 (1995).
- . The PyMOL Molecular Graphics System Version 1.8 Available from: https://pymol.org (2015)
- Pelikan, M., Hura, G. L., Hammel, M. Structure and flexibility within proteins as identified through small angle X-ray scattering. Gen Physiol Biophys. 28 (2), 174-189 (2009).
- Deller, M. C., Kong, L., Rupp, B. Protein stability: a crystallographer's perspective. Acta Crystallogr F. 72 (Pt 2), 72-95 (2016).
- Hinsen, K. Structural flexibility in proteins: impact of the crystal environment. Bioinformatics. 24 (4), 521-528 (2008).
- Shtykova, E. V., et al. Structural analysis of influenza A virus matrix protein M1 and its self-assemblies at low pH. PLoS One. 8 (12), e82431 (2013).
- Mallam, A. L., et al. Solution structures of DEAD-box RNA chaperones reveal conformational changes and nucleic acid tethering by a basic tail. Proc Natl Acad Sci U S A. 108 (30), 12254-12259 (2011).
- Papaleo, E., et al. The Role of Protein Loops and Linkers in Conformational Dynamics and Allostery. Chem Rev. 116 (11), 6391-6423 (2016).
- Rozycki, B., Boura, E. Large, dynamic, multi-protein complexes: a challenge for structural biology. J Phys Condens Matter. 26 (46), 463103 (2014).
- Schlundt, A., Tants, J. N., Sattler, M. Integrated structural biology to unravel molecular mechanisms of protein-RNA recognition. Methods. 118, 119-136 (2017).
- Thompson, M. K., Ehlinger, A. C., Chazin, W. J. Analysis of Functional Dynamics of Modular Multidomain Proteins by SAXS and NMR. Methods Enzymol. 592, 49-76 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon