Method Article
Scoperta di nuovi agenti patogeni intracellulari da amoebal co-coltura e approcci amoebal arricchimento
In questo articolo
Riepilogo
Coculture amoebal è un sistema di coltura cellulare usando amebe aderente a crescere selettivamente patogeni intracellulari in grado di resistere fagociti come amebe e macrofagi. Rappresenta quindi uno strumento chiave per scoprire nuovi agenti infettivi. Arricchimento amoebal permette la scoperta di nuove specie amoebal e dei loro specifici batteri intracellulari.
Abstract
Patogeni intracellulari come la legionella, micobatteri e organismi Chlamydia simili sono difficili da isolare perché spesso crescono poco o per niente su terreni selettivi che di solito vengono utilizzati per coltivare i batteri. Per questo motivo, molti di questi agenti patogeni sono state scoperte solo recentemente o seguente focolai importanti. Questi agenti patogeni sono spesso associati con amebe, che servono come cellula ospite e consentire la sopravvivenza e la crescita dei batteri. Intendiamo qui per fornire una dimostrazione di due tecniche che permettono l'isolamento e la caratterizzazione dei patogeni intracellulari presenti in campioni clinici e ambientali: la co-coltura amoebal e l'arricchimento amoebal. Coculture amoebal permette il recupero di batteri intracellulari inoculando campione indagato su un prato amoebal che può essere infettato e lisate dai batteri intracellulari presenti nel campione. Arricchimento amoebal permette il recupero di amebe presente in un campione clinico o ambientale. Thsi può portare alla scoperta di nuove specie amoebal ma anche di nuovi batteri intracellulari che crescono in particolare in queste amebe. Insieme, queste due tecniche aiutano a scoprire nuovi batteri intracellulari in grado di crescere in amebe. A causa della loro capacità di infettare amebe e resistere fagocitosi, questi batteri intracellulari potrebbero anche sfuggire fagocitosi da parte dei macrofagi e quindi, per essere patogeni eucarioti superiori.
Introduzione
Prima dell'avvento di diagnosi molecolare, microrganismi presenti in nicchie ambientali o in campioni clinici sono stati spesso individuati da loro coltivazione su diversi terreni selettivi, soprattutto su agar in piastre di Petri. Il fenotipo delle colonie batteriche e la loro attività metabolica quindi lasciata classificazione batterica a livello di specie. Brodo può essere anche usato per aumentare la sensibilità del rilevamento. Tuttavia, entrambe le tecniche non consentono il recupero di batteri che crescono lentamente o per niente su questi terreni. Questo è il motivo per cui gli approcci molecolari sono così ampiamente utilizzati al giorno d'oggi. Tuttavia, il rilevamento del DNA fornisce alcun indizio sulla vitalità dei batteri. Inoltre, contrariamente alla cultura, approcci molecolari non comportino un ceppo che può essere ulteriormente caratterizzato.
Studiare gli agenti patogeni che crescono male su terreni solidi o che le cellule hanno bisogno per crescere è complicato. La maggior parte di questi "difficile da coltivare" i batteri sono intr esigentibatteri acellulare, spesso scoperti e caratterizzati seguente grandi epidemie come era il caso di Legionella pneumophila. Questo batterio è stato caratterizzato in seguito ad un'epidemia che si è verificato nel corso di un convegno American Legion. Ben 182 persone sono state infettate e 29 morti a causa di una grave polmonite 1,2. E 'stato poi dimostrato che le amebe erano i padroni di casa naturali di questo batterio e che la loro presenza nelle reti albergo di sistema di aria condizionata e acqua è stata all'origine dello scoppio della malattia, la cosiddetta del legionario 3.
Amebe sono presenti in tutto il mondo e sono stati isolati dal suolo, l'aria, l'acqua e la mucosa nasale di volontari umani (recensiti 4). Queste amebe "vita libera" sono generalmente dividendo autonomamente nell'ambiente, ma può occasionalmente invadere padroni di casa permissive 5. Mangimi amebe su vari microrganismi attraverso la fagocitosi e la successiva digestione lisosomiale da hydrolases 6. Molti batteri intracellulari facoltativi o di obbligare sono in grado di resistere digestione e infettare e dividere in amebe come per esempio batteri o micobatteri Legionella, Chlamydia correlati (recensito in 7 e 8) così. Amebe a vita libera probabilmente rappresentano un importante bacino potenziale di batteri intracellulari che non sono ancora stati scoperti. Ciò ha portato il nostro gruppo ad attuare a Losanna due tecniche principali, chiamati co-coltura amoebal e arricchimento amoebal, che ha permesso di isolare i diversi gruppi diversi nuovi microrganismi intracellulari obbligati da vari campioni ambientali 9-15.
Poiché amebe sono fagociti professionali pascolo su batteri, un batterio in grado di resistere fagocitosi e di crescere all'interno di questi protisti potrebbe anche colonizzare fagociti umani ed essere patogeni verso gli esseri umani. Questo è stato parzialmente dimostrata per alcuni batteri Chlamydia-correlati, come Waddlia chondrophila. W. chondrophila può crescere non solo in amebe ma anche in diversi tipi cellulari quali cellule di mammifero epiteliali, macrofagi e linee cellulari di pesce 16-18. La co-coltura amoebal appare rilevante anche per la rilevazione di batteri intracellulari in campioni clinici 19,20, compresi sgabelli che sono fortemente contaminati da diverse specie batteriche 21.
Qui si descrivono le fasi principali di co-coltura amoebal e arricchimento amoebal, tra cui (a) il trattamento di campioni ambientali o cliniche; (b) la crescita di amebe su supporti axeniche e su un prato batterica di Escherichia coli e (c) la selezione e la caratterizzazione di batteri intracellulari.
Protocollo
1. Amoebal coculture
1.1 Preparazione del campione
- Campione ambientale
- I campioni di acqua
Filtrare il campione d'acqua (500 ml a 1 L) attraverso una membrana dimensione dei pori di 0,22 micron. Poi, scuotere la membrana in ameba saline medie PAS della pagina (120 mg di NaCl, 4 mg di MgSO4 • 7H 2 O, 4 mg di CaCl 2 • 2H 2 O, 142 mg di Na 2 HPO 4, e 136 mg di KH 2 PO 4 in 1 L di acqua distillata). - Campioni solidi
Risospendere campioni solidi come campioni di terreno o sabbia e campioni semi-solidi come fanghi attivi in acqua distillata o PBS e filtrare attraverso una membrana dimensione dei pori 0,22 micron. Poi, scuotere la membrana in PAS. - I campioni altamente contaminati da amebe endogena e protozoi
Prima sedimentare i campioni mediante centrifugazione a bassa velocità (180 xg) for 10 min o filtrare attraverso una membrana dimensione dei pori 5 micron. Ulteriori elaborare il surnatante (rispettivamente filtrato), come in 1.1.1.
Nota: Altre tecniche di decontaminazione potrebbero essere utilizzati per decontaminare ulteriori campioni ambientali: Riscaldare il campione a 50 ° C per 30 minuti o trattare con soluzioni acide o basiche 14.
- I campioni di acqua
- Campione clinico
Elaborare campioni clinici seconda delle loro proprietà chimico-fisiche. Filtrare o centrifugare liquidi per rimuovere grosse impurità. Risospendere campioni solidi. Per utilizzare tessuti, macinare, ad esempio utilizzando un homogeneiser Dounce o perline di vetro, e lisare le cellule per liberare i batteri intracellulari.
1.2 Amebe preparazione
- Preparato per brodo
Preparare i seguenti mezzi: un ricco supporto contenente peptone, estratto di lievito e glucosio (PYG, 100 g di peptone proteoso, 10 g di estratto di lievito, 4,9 g di MgSO4 • 7H 2O, 5 g di sodio citrato • 2H 2 O, 0,1 g di Fe (NH 4) 2 (SO 4) 2 • 6H 2 O, 1,7 g di KH 2 PO 4, 1,97 g di Na 2 HPO 4 • 7H 2 O , 45 g di glucosio, e 0,295 g di CaCl 2 a 5 L di acqua distillata) e un mezzo non nutritivo come PAS per specie Acanthamoeba. - Cultura amoebal
- Coltivare amebe (Acanthamoeba preferenzialmente castellanii ATCC 30010 o A. polyphaga Linc-Ap1) a 25 ° C in cella fiasche di coltura contenente 30 ml di PYG mezzo.
- Raccogliere il amebe forte agitazione del pallone e centrifugare la sospensione cellulare per 10 minuti a 1500 x g. Lavare il pellet due volte con mezzo di PAS. Contare le cellule in una diapositiva Kova e regolare il volume per ottenere una sospensione di 5 x 10 5 cellule per ml.
- Trasferire la sospensione amoebal in micropiastre. Utilizzare 1 ml per pozzetto per 12 - e 24-wpiastre ell, 500 microlitri per piastre 48 pozzetti e 300 microlitri per piastre da 96 pozzetti. Incubare la micropiastra per almeno 2 ore a 25 ° C. Questo permette sedimentazione e il fissaggio amebe al fondo di ciascun pozzetto.
1.3 coculture
- Esempio di inoculazione
- Seminare la piastra da 2.3.3 con diluizioni seriali del campione da 1.1 o 1.2, di solito con serie di diluizioni di 10 volte, partendo con 100 ml di campione non diluito.
- Centrifugare la micropiastra a 1.800 xg per 10 min a sedimenti sul prato amoebal microrganismi potenzialmente presenti nel campione. Questo aumenta il contatto e la fagocitosi dei microrganismi da amebe.
- Incubare le piastre per 45 minuti a 25 ° C e lavate tre volte con PAS sostituendo il materiale con PAS fresco. Aggiungere 1 ml per pozzetto di PAS con o senza aggiunta di antibiotici (streptomicina, penicillina, gentamicina e / o vancomicina), a seconda delle batteriologicheAL specie che sono cercato.
- Incubare la micropiastra a 32 ° C in atmosfera umidificata per evitare incistamento del amebe. Osservare ogni bene ogni giorno con un obiettivo 20X per rilevare la presenza di batteri invasori e lisi amebe.
- Nel caso di lisi, eseguire una subcultura su amebe fresco inoculando 100 pl di co-colture di un monostrato di circa 10 5 amebe / cm 2. Per isolare in particolare una determinata specie batteriche, inoculare anche agar specifico progettato per questi batteri (cioè BCYE agar per Legionella spp.).
- In assenza di lisi, prendere 100 ml di co-colture quattro a sette giorni dopo la prima inoculazione e inoculare una coltura fresca amoebal di 900 microlitri in una piastra da 24 pozzetti. Se rapida lisi delle amebe si osserva senza proliferazione di batteri, virus potrebbe essere presente. In questo caso, filtrare il supernatante a 0,22 micron e usare la sospensione filtrata per infettare amebe fresco.
1.4 isolamento batterica e la caratterizzazione
- Colorazione batterica
Eseguire co-colture direttamente su vetrini a 24 pozzetti. Rimuovere il supporto ed eseguire la colorazione o immunofluorescenza.- Modificato colorazione Romanowsky
- Lasciare asciugare il vetrino. Immergere il vetrino cinque volte nella soluzione fissativo (2 mg / L veloce verde in metanolo).
- Immergere il coprioggetto cinque volte nella soluzione colorante I (1,22 g / L Eosina G in tampone fosfato pH 6,6)
- Infine, immergere il vetrino 5 volte nella soluzione colorante II (1.1 g / L tiazina in tampone fosfato pH 6,6).
- Sciacquare il campione con acqua distillata. Lasciate asciugare e osservare al microscopio.
- Ziehl-Neelsen
- Lasciare asciugare il vetrino. Coprire il campione con Ziehl fucsina. Riscaldare il colorante con una fiamma fino all'apparizione vapori.
- Raffreddare a temperatura ambiente per almeno 5 mine risciacquare con acqua distillata. Coprire il campione con una soluzione di acido cloridrico al 3% in isopropanolo per 2 minuti e risciacquare con acqua distillata.
- Copertina per 30 sec con blu di metilene e risciacquare con acqua distillata. Lasciate asciugare e osservare al microscopio 22.
- Gimenez colorazione
- Preparare una soluzione stock fucsina base miscelando 100 ml di 10% fucsina basica (10 g di fucsina basica in 100 ml di etanolo al 95%), 250 ml di 4% fenolo acquoso e 650 ml di acqua distillata. Incubare a 37 ° C per 48 h prima dell'uso.
- Lasciate asciugare il vetrino e fissare mediante passaggio su fiamma. Coprire il campione con fucsina basica appena filtrata (4 ml di fucsina magazzino soluzione di base in 10 ml di 0,1 M tampone fosfato di sodio, pH 7,45) per 2 minuti.
- Risciacquare il campione con acqua e incubare in verde malachite (0,8% in acqua distillata) per 10 sec. Sciacquare nuovamente con acqua e ripetere la colorazione verde malachite. Sciacquare nuovamente con acqua. Lasciate che il vetrino asciutto, mount, e osservare al microscopio 23.
- Immunofluorescenza
- Fissare il vetrino incubando in metanolo per 5 min o con paraformaldeide al 4% per 10 min.
- Lavare tre volte con PBS e incubare per 2 ore in una soluzione bloccante (5% BSA, 0.1% saponina in PBS) a temperatura ambiente.
- Incubare il vetrino per 1 ora in una soluzione bloccante contenente anticorpi contro il microrganismo di interesse.
- Lavare nuovamente per tre volte in PBS e incubare per 1 ora con un anticorpo secondario diretto contro l'anticorpo primario e collegato a un fluoroforo. Lavare tre volte con PBS, montare il coprioggetto ed osservare al microscopio a fluorescenza.
- Modificato colorazione Romanowsky
- Rilevazione del DNA mediante PCR
Estrarre il DNA da 100 a 200 ml di co-coltura amoebal. Rileva microrganismi con primer universali rivolti al gene 16S rRNA o primer specifici per le specie di interesse come i micobatteri 24, Legionella 25 o membri del Chlamydiales 26.
2. Arricchimento amoebal
2.1. Preparazione del campione
Risospendere campioni solidi e semi-solidi in PAS vortex. Centrifugare la sospensione a bassa velocità (180 xg) per 10 min. Questo permette di arricchimento di amebe a vita libera nel pellet. Il supernatante può essere utilizzato per coculture amoebal e il pellet di arricchimento amoebal 21.
2.2. Preparazione medio
- Aggiungere 1,5 g di agar per 100 ml di PAS e sterilizzare in autoclave il mezzo 15 min a 121 ° C. Versare il terreno caldo in piastre di Petri e lasciare solidificare a temperatura ambiente.
- Grow Escherichia coli (ATCC 25922) in brodo LB o tioglicolato notte a 37 ° C. Lavare i batteri due volte con PBS e risospendere in mezzo PAS. Diluire 10x in PAS e diffondere 2-3 ml di questa diluizione su una piastra di PAS agar e lasciare asciugare.
2.3. Esempio di inoculazione
Aggiungere una goccia di campione (o un pezzo di filtro) su un lato della piastra e lasciare scorrere sulla piastra di Petri per formare una linea al centro del piatto.
2.4. La crescita amoebal e sottocultura amoebal
- Osservare la piastra di Petri quotidiano. Se viene rilevato un fronte di migrazione amoebal, tagliare un piccolo pezzo di agar al fronte di migrazione e inoculare un piatto fresco NNA Petri ricoperta da un prato di E. coli.
- Ripetere la reinoculazione più volte a seconda della purezza del campione, in modo da avere una coltura pura di un dato ceppo amoebal.
2.5. Amebe e batteri caratterizzazione
- Raschiare le cellule e risospendere in PAS.
- Estrarre il DNA e determinare l'identità di amebe e / o endosimbionti batterici mediante PCR e sequenziamento (16S rRNA di amplificazione per i batteri, risp. 18S rRNA per la amebe e sequenziamento, per esempio).
- Utilizzare queste cellule raschiate in PAS per inoculare amebe fresco ed eseguire una co-coltura amoebal per rilevare i batteri eventualmente presenti nel campione.
Risultati
Utilizzo di co-coltura amoebal e l'arricchimento amoebal, tutta una serie di batteri ambientali e / o patogeni sono stati scoperti (Tabella 1).
Coculture amoebal è stato utilizzato dal nostro gruppo ed altri per analizzare campioni ambientali, impianti di trattamento delle acque e sistemi di distribuzione dell'acqua. Una vasta gamma di microrganismi potrebbe essere isolato con questa tecnica. I più comuni batteri isolati da co-coltura amoebal sono membri del genere Mycobacterium che potrebbero essere recuperati dagli impianti di trattamento delle acque e delle reti idriche 13,14,24,27,28. Legionella e le specie α-Proteobacteria potrebbero anche essere isolati da impianti di trattamento delle acque e dalle reti idriche ospedaliere 14,24,28-31. Diverse specie Chlamydia correlati sono stati isolati da acqua di fiume e impianti di trattamento delle acque, come ad esempio Estrella lausannensis (Figure 2B-C 12,15,32,33.
Utilizzo di co-coltura amoebal, sono stati scoperti diversi virus giganti, come Mimivirus, Marseillevirus e Lausannevirus 34-36. Questi virus sono tutti in grado di infettare e moltiplicarsi all'interno di amebe e presentare una fase di eclisse tipico del loro stile di vita virale. I mimivirus è considerato come un delicato agente patogeno polmone umano da quando è stato implicato in una infezione accidentale di un tecnico di laboratorio che soffriva di polmonite 37. Potenziale patogenicità di altri virus giganti deve ancora essere indagato.
Arricchimento amoebal è stato spesso utilizzato in parallelo al amoebal co-coltura. Così, quando indaga su un sistema di depurazione delle acque e della rete idrica a valle, sono stati individuati 25 diversi ceppi amoebal, di cui 12 corrispondevano a nuove specie 14. Amebe erano presenti in ogni fase di purificazione e distribuzione dell'acqua, che indica una resistenza di questi protisti di ozonizzazione e clorazione. Intracellulare co batteriuld anche essere rilevata in questi amebe, mostrando l'importanza di amebe indigena nella trasmissione di batteri intracellulari 14. In un altro studio, amebe potrebbe essere isolato dal sistema di distribuzione dell'acqua di un ospedale 24. Una grande maggioranza dei ceppi isolati in questo studio corrisponde vermiformis Hartmannella, che è naturalmente in grado di sopravvivere a temperature relativamente elevate. Diversi batteri, come Legionella pneumophila potrebbe essere rilevato nel amebe indigena 24. Un altro esempio di batteri presenti in un'ameba specifico è Parachlamydia acantamoebae. Questo batterio Chlamydia legate stato isolato da mucosa nasale di volontari di sesso femminile da arricchimento amoebal (Figura 2A) 9 ed è un potenziale agente di polmonite 38. Questo mostra ancora l'importanza di amebe nella manutenzione e dispersione di agenti patogeni batterici che potrebbero essere particolarmente patogeni per immunocompromispazienti ED 39.
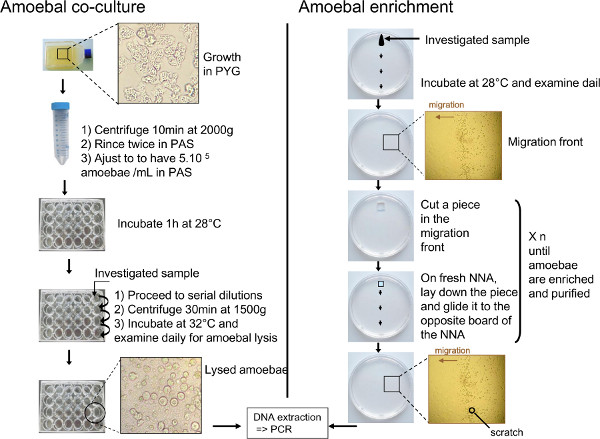
Figura 1. Schema di co-coltura amoebal e di arricchimento amoebal descrive i passi importanti di queste due tecniche. (Adattato da 40). Clicca qui per ingrandire la figura .

Figura 2. Esempi di batteri scoperti da co-coltura amoebal e la loro osservazione con differenti metodi di colorazione. A) La colorazione di cocco infettare amebe di Parachlamydia acanthamoebae Hall, con le modifiche metodo Romanowsky 24 ore dopo l'infezione, con un ingrandimento di 1.000 X. I batteri sono colorate in blu (arrows). aC) microscopia elettronica di Estrella lausannensis mostra la morfologia tipica stella di corpi elementari (frecce).
| Classe | Specie esempi | Riferimento |
| α-proteobatteri | Odyssella thelassonicensis | 10 |
| b-proteobatteri | Burkholderia cepacia | 36 |
| g-proteobatteri | Legionella drancourtii | 26 |
| Chlamydiae | Estrella lausannensis | 30 |
| Flavobacteriae | Amoebophilus asiaticus | 37 |
| Actinobacteria | Micobatteri spp. | 38 |
Tabella 1. Esempi di batteri provenienti da diverse classi scoperte da co-coltura amoebal.
Discussione
Coculture amoebal e l'arricchimento amoebal sono metodi efficaci che hanno permesso l'isolamento di molte nuove specie batteriche e amoebal. I risultati ottenuti con questi metodi confermano la presenza ubiquitaria di entrambi amebe e batteri-ameba resistere nell'ambiente, e la più interessante delle reti idriche artificiali che sono considerati essere controllato da trattamenti chimici come la clorazione e ozonizzazione. Coculture amoebal e l'arricchimento amoebal sono strumenti essenziali per isolare e coltivare questi microrganismi potenzialmente patogeni e di ottenere ceppi in coltura pura per poi approfondire la loro biologia e la patogenicità. Recentemente, questo metodo è stato adattato per high-throughput isolamento di virus giganti 41. Analogamente, coculture amoebal potrebbe essere automatizzato e usato come tecnica di routine per verificare la qualità microbiologica di campioni ambientali e dei sistemi idrici artificiali, come l'acqua potabile.
Coculture amoebal puòessere eseguito con diverse specie di amebe. Noi di solito preferiamo usare Acanthamoeba castellanii o A. polyphaga dal momento che sono meno inclini a incistamento di vermiformis Hartmannella e dal momento che presentano una relativamente grande spettro host. Altre specie possono essere utilizzati, ma il protocollo e il brodo devono essere adattati. È stato recentemente dimostrato che A. lenticulana (ATCC 30841) può essere usato nelle stesse condizioni come A. castellanii o A. polyphaga ma è diversa suscettibilità alle infezioni 42. Per evitare incistamento, è importante usare una temperatura di incubazione relativamente bassa (inferiore a 30 ° C) e per mantenere un'atmosfera umidificata.
Degno di nota, la replica continua di simbionti amoebal non comporta necessariamente lisi amoebal e quando specificamente alla ricerca di simbionti, lo screening mediante microscopia e / o PCR devono essere effettuati in modo sistematico. Per evitare lisi o distacco ce amoebal LLS a causa di proliferazione batterica, è utile effettuare l'inoculazione di campioni fortemente contaminati con diluizioni seriali di 10 volte.
Tuttavia, queste tecniche hanno limitazioni. Grazie all'uso di una singola specie amoebal, la co-coltura amoebal potrebbe non consentire l'isolamento di batteri che utilizzano come serbatoio un'altra specie amoebal. Inoltre, tali specifiche specie amoebal potrebbero non essere in grado di proliferare in E. prati coli e possono essere mancati da arricchimento amoebal. A seconda delle proprietà dei batteri o amebe indagati, potrebbe essere necessario testare diversi media, diverse specie amoebal e differenti specie batteriche per alimentare amebe (Enterobacter doacae e alcuni ceppi di Pseudomonas sono buone alternative). La contaminazione batterica di amebe oi supporti utilizzati per la co-coltura amoebal può verificarsi e può portare a risultati falsi positivi. Un controllo negativo è quindi obbligatorio per evitare tali risultati falsi positivi.
contenuto "> In conclusione, co-coltura amoebal e l'arricchimento amoebal sono due approcci complementari che rappresentano strumenti interessanti per specificare l'ecologia e la biodiversità delle amebe a vita libera e di batteri amebe resistere. Queste tecniche permettono la scoperta di molte nuove specie, comprese le amebe, batteri e virus giganti, che costituiscono la base per futuri studi per indagare la patogenicità di questi nuovi microrganismi.Divulgazioni
Gli autori dichiarano di non avere interessi finanziari in competizione.
Riconoscimenti
Ringraziamo Pr. Bernard La Scola per utili consigli tecnici e interessante discussione sulla co-coltura amoebal e di arricchimento amoebal. Ringraziamo anche il Dott. Vincent Thomas per il suo aiuto nella realizzazione tecnica nel nostro laboratorio.
Materiali
| Name | Company | Catalog Number | Comments |
| Glucose monohydrate | Merck, Darmstadt, Germany | 108342 | |
| 0.22 μm pore size membrane | Merck Millipore, Darmstadt, Germany | SCVPU11RE | |
| proteose peptone | Becton-Dickinson, Franklin Lakes, NJ | 211693 | |
| yeast extract | Becton-Dickinson, Franklin Lakes, NJ | 212750 | |
| Cell culture flasks | Becton-Dickinson, Franklin Lakes, NJ | 353135 | |
| Kova slide | Hycor, Indianapolis, IN | 87144 | |
| cell culture microplates | Corning Inc, Corning, NY | 3524 | |
| Diff-Quik staining kit | Siemens Healthcare diagn., Munich, Germany | 130832 | |
| Ziehl fuchsin | Fluka, St-Louis, MI | 21820 | |
| basic fuchsin | Sigma, St-Louis, MI | 857843 | |
| Phenol | Sigma, St-Louis, MI | P1037 | Corrosive and mutagenic |
| malachite green oxalate | Fluka, St-Louis, MI | 63160 | |
| Paraformaldehyde 16% solution | Electron Microscopy Sciences, Hatfield, PA | 15710 | |
| Saponin | Sigma, St-Louis, MI | 84510 |
Riferimenti
- Fraser, D. W., et al. Legionnaires' disease: description of an epidemic of pneumonia. New Engl. J. Med. 297, 1189-1197 (1977).
- McDade, J. E., et al. Legionnaires' disease: isolation of a bacterium and demonstration of its role in other respiratory disease. New Engl. J. Med. 297, 1197-1203 (1977).
- Rowbotham, T. J. Preliminary report on the pathogenicity of Legionella pneumophila for freshwater and soil amoebae. J. Clin. Pathol. 33, 1179-1183 (1980).
- Rodriguez-Zaragoza, S. Ecology of free-living amoebae. Crit. Rev. Microbiol. 20, 225-241 (1994).
- Booton, G. C., Visvesvara, G. S., Byers, T. J., Kelly, D. J., Fuerst, P. A. Identification and distribution of Acanthamoeba species genotypes associated with nonkeratitis infections. J Clin. Microbiol. 43, 1689-1693 (2005).
- Brussow, H. Bacteria between protists and phages: from antipredation strategies to the evolution of pathogenicity. Molecular microbiology. 65, 583-589 (2007).
- Greub, G., Raoult, D. Microorganisms resistant to free-living amoebae. Clin. Microbiol. Rev. 17, 413-433 (2004).
- Thomas, V., McDonnell, G., Denyer, S. P., Maillard, J. Y. Free-living amoebae and their intracellular pathogenic microorganisms: risks for water quality. FEMS Microbiol Rev. 34, 231-259 (2010).
- Birtles, R. J., Rowbotham, T. J., Storey, C., Marrie, T. J., Raoult, D. Chlamydia-like obligate parasite of free-living amoebae. Lancet. 349, 925-926 (1997).
- Amann, R., et al. Obligate intracellular bacterial parasites of acanthamoebae related to Chlamydia spp. Appl. Environ. Microbiol. 63, 115-121 (1997).
- Birtles, R. J., et al. Candidatus Odyssella thessalonicensis' gen. nov., sp. nov., an obligate intracellular parasite of Acanthamoeba species. Int. J. Syst. Evol. Microbiol. 50, 63-72 (2000).
- Thomas, V., Casson, N., Greub, G. Criblamydia sequanensis, a new intracellular Chlamydiales isolated from Seine river water using amoebal co-culture. Environ. Microbiol. 8, 2125-2135 (2006).
- Pagnier, I., Raoult, D., La Scola, B. Isolation and identification of amoeba-resisting bacteria from water in human environment by using an Acanthamoeba polyphaga co-culture procedure. Environ. Microbiol. 10, 1135-1144 (2008).
- Thomas, V., Loret, J. F., Jousset, M., Greub, G. Biodiversity of amoebae and amoebae-resisting bacteria in a drinking water treatment plant. Environ. Microbiol. 10, 2728-2745 (2008).
- Corsaro, D., et al. Novel Chlamydiales strains isolated from a water treatment plant. Environ. Microbiol. 11, 188-200 (2009).
- Goy, G., Croxatto, A., Greub, G. Waddlia chondrophila enters and multiplies within human macrophages. Microbes Infect. 10, 556-562 (2008).
- Kebbi-Beghdadi, C., Cisse, O., Greub, G. Permissivity of Vero cells, human pneumocytes and human endometrial cells to Waddlia chondrophila. Microbes Infect. 13, 566-574 (2011).
- Kebbi-Beghdadi, C., Batista, C., Greub, G. Permissivity of fish cell lines to three Chlamydia-related bacteria: Waddlia chondrophila, Estrella lausannensis and Parachlamydia acanthamoebae. FEMS Immunol. Med. Microbiol. 63, 339-345 (2011).
- Fry, N. K., Rowbotham, T. J., Saunders, N. A., Embley, T. M. Direct amplification and sequencing of the 16S ribosomal DNA of an intracellular Legionella species recovered by amoebal enrichment from the sputum of a patient with pneumonia. FEMS Microbiol. Lett. 67, 165-168 (1991).
- Rowbotham, T. J. Isolation of Legionella pneumophila serogroup 1 from human feces with use of amebic cocultures. Clin. Infect. Dis. 26, 502-503 (1998).
- Greub, G., La Scola, B., Raoult, D. Amoebae-resisting bacteria isolated from human nasal swabs by amoebal coculture. Emerging Infect. Dis. 10, 470-477 (2004).
- Isenberg, H. D. . Clinical microbiology procedures handbook. , (1992).
- Gimenez, D. F. Staining Rickettsiae in Yolk-Sac Cultures. Stain Technol. 39, 135-140 (1964).
- Thomas, V., Herrera-Rimann, K., Blanc, D. S., Greub, G. Biodiversity of amoebae and amoeba-resisting bacteria in a hospital water network. Appl. Environ. Microbiol. 72, 2428-2438 (2006).
- Miyamoto, H., et al. Development of a new seminested PCR method for detection of Legionella species and its application to surveillance of legionellae in hospital cooling tower water. Appl. Environ. Microbiol. 63, 2489-2494 (1997).
- Lienard, J., et al. Development of a new chlamydiales-specific real-time PCR and its application to respiratory clinical samples. J. Clin. Microbiol. 49, 2637-2642 (2011).
- Wang, Y., Ogawa, M., Fukuda, K., Miyamoto, H., Taniguchi, H. Isolation and identification of mycobacteria from soils at an illegal dumping site and landfills in Japan. Microbiol. Immunol. 50, 513-524 (2006).
- Corsaro, D., Pages, G. S., Catalan, V., Loret, J. F., Greub, G. Biodiversity of amoebae and amoeba-associated bacteria in water treatment plants. Int. J. Hygiene Environ. Health. 213, 158-166 (2010).
- La Scola, B., et al. Legionella drancourtii sp. nov., a strictly intracellular amoebal pathogen. Int. J. Syst. Evol. Microbiol. 54, 699-703 (2004).
- Thomas, V., Casson, N., Greub, G. New Afipia and Bosea strains isolated from various water sources by amoebal co-culture. Syst. Appl. Microbiol. 30, 572-579 (2007).
- La Scola, B., et al. Amoeba-resisting bacteria and ventilator-associated pneumonia. Emerging Infect. Dis. 9, 815-821 (2003).
- Collingro, A., et al. Recovery of an environmental Chlamydia strain from activated sludge by co-cultivation with Acanthamoeba sp. Microbiology. 151, 301-309 (2005).
- Lienard, J., Croxatto, A., Prod'hom, G., Greub, G. Estrella lausannensis, a new star in the Chlamydiales order. Microbes Infect. 13, 1232-1241 (2011).
- La Scola, B., et al. A giant virus in amoebae. Science. 299, 2033 (2003).
- Boyer, M., et al. Giant Marseillevirus highlights the role of amoebae as a melting pot in emergence of chimeric microorganisms. Proc. Natl. Acad. Sci. U.S.A. 106, 21848-21853 (2009).
- Thomas, V., et al. Lausannevirus, a giant amoebal virus encoding histone doublets. Environ. Microbiol. 13, 1454-1466 (2011).
- Raoult, D., Renesto, P., Brouqui, P. Laboratory infection of a technician by mimivirus. Ann. Internal Med. 144, 702-703 (2006).
- Greub, G. Parachlamydia acanthamoebae, an emerging agent of pneumonia. Clin. Microbiol. Infect. 15, 18-28 (2009).
- Lamoth, F., Greub, G. Amoebal pathogens as emerging causal agents of pneumonia. FEMS Microbiol. Rev. 34, 260-280 (2010).
- Lienard, J. G., Ashbolt, K., Sen, N. J. Ch. 6. Environmental microbiology, current technology and water applications. , 143-162 (2011).
- Boughalmi, M., et al. High-throughput isolation of giant viruses of the Mimiviridae and Marseilleviridae families in the Tunisian environment. Environ. Microbiol. , (2012).
- Ovrutsky, A. R., et al. Cooccurrence of Free-Living Amoebae and Nontuberculous Mycobacteria in Hospital Water Networks, and Preferential Growth of Mycobacterium avium in Acanthamoeba lenticulata. Appl. Environ. Microbiol. 79, 3185-3192 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon