Method Article
Protein Purification-libera Metodo di Binding Affinity Determinazione Thermophoresis microscala
In questo articolo
Riepilogo
Thermophoresis microscala (MST) può essere ampiamente usato per la determinazione di affinità di legame senza purificazione della proteina bersaglio da lisati cellulari. Il protocollo prevede sovraespressione della proteina GFP-fusa, lisi cellulare in condizioni non denaturanti, e la rilevazione di segnale MST in presenza di concentrazioni variabili di ligando.
Abstract
Caratterizzazione quantitativa delle interazioni della proteina è essenziale in praticamente ogni campo delle scienze della vita, in particolare di scoperta di nuovi farmaci. La maggior parte dei metodi attualmente disponibili di determinazione K D richiedono accesso purificata proteina di interesse, di generazione che può essere lunga e costosa. Abbiamo sviluppato un protocollo che consente di determinare l'affinità di legame per thermophoresis microscala (MST) senza purificazione della proteina bersaglio da lisati cellulari. Il metodo prevede sovraespressione della proteina GFP-fuso e lisi cellulare in condizioni non denaturanti. Applicazione del metodo di STAT3-GFP espresso transitoriamente in cellule HEK293 permesso di determinare per la prima volta l'affinità del fattore di trascrizione ben studiato per oligonucleotidi con sequenze differenti. Il protocollo è semplice e può avere una varietà di applicazione per studiare le interazioni delle proteine con piccole molecole, peptidi, DNA, RNA, e proteins.
Introduzione
Caratterizzazione quantitativa di affinità di interazioni intermolecolari è importante in molti settori della ricerca biomedica. Legame costante di dissociazione (K D) è essenziale non solo nella scoperta della droga, ma è anche un parametro importante caratterizzazione di ogni interazione binaria in qualsiasi sistema biologico. Metodi biochimici utilizzati per il rilevamento di interazioni proteina-proteina, come la immunoprecipitazione e lievito schermi a due ibridi, non ci informano su quanto stretto sono quelle interazioni, mentre affinità definisce se questo particolare complesso esiste in determinate condizioni in vivo. Nel processo di scoperta di nuovi farmaci, lo sviluppo del saggio di legame è uno dei più necessaria e spesso i passi più in termini di tempo. Metodi più utilizzati per la determinazione K D includono polarizzazione di fluorescenza, 1 tecnologia risonanza plasmonica di superficie (SPR), vincolante radioligando 2, 3 isotermica di titolazione calorimetria, 4 Equilibrium dialisi (ED), 5 ultrafiltrazione (UF), 5 e ultracentrifugazione (UC). 6 Tutte richiedono notevoli quantità di proteina purificata bersaglio. Thermophoresis microscala (MST) è un metodo rapido sviluppo che rileva la regia movimento delle molecole in un gradiente di temperatura microscopico. Eventuali modifiche del guscio di idratazione biomolecole comportano un cambiamento relativo di movimento lungo il gradiente di temperatura. 7 MST viene utilizzato per determinare affinità di legame ed è stato applicato per lo studio legame alle proteine fluorescente o ligandi fluorescenti ligando ad una proteina bersaglio. 8, 9 MST consente di misurare direttamente interazioni in soluzione, senza la necessità di immobilizzazione di una superficie (immobilizzazione tecnologia senza). Praticamente, qualsiasi associazione è accompagnato da un cambiamento nel segnale MST, anche se la dimensione del cambiamento differisce da sistema a sistema in modo significativo. Per la rilevazione di movimento molecola da MST, devono essere fluorescenzant. Questo importante limitazione del metodo può essere trasformato in un vantaggio. Se una proteina è espressa come una fusione GFP in qualsiasi sistema, sarà la sola molecola fluorescente e quindi può essere studiata senza separazione dal lisato cellulare o sistema di espressione acellulare. Generazione di lisati cellulari che permettono di condizioni vincolanti con manufatti minimali è la grande sfida. Qui si descrive un protocollo di cellule lisate e sperimentare MST che può essere utilizzato per molte proteine solubili e di membrana.
Proteine STAT sono latenti fattori di trascrizione attivati da citoplasmatici fosforilazione della tirosina in risposta a segnali extracellulari e sono coinvolti in molti processi biologici tra immunità, ematopoiesi, infiammazione e sviluppo. 10 Nei mammiferi, la famiglia STAT costituito STAT 1, 2, 3, 4 , 5A, 5B e 6. All Stats attivati sono noti per legarsi alla stessa sequenza di DNA, così chiamato motivi GAS, IFN-gamma attivato sequenza. Tuttavia, la transcrieffetti facoltativo, di diverse statistiche sono molto diversi. 11 Nonostante il coinvolgimento in molti processi patologici e ampi studi che producono oltre 17.000 pubblicazioni, la K D di interazioni STAT con diverse sequenze di DNA non sono stati determinati. Solo affinità relativa delle diverse statistiche varianti del motivo GAS è stato caratterizzato. 11 Difficoltà di espressione e purificazione di proteine sono i principali ostacoli nella caratterizzazione di STAT 'DNA selettività vincolante. Anche se la maggior parte degli studi si sono concentrati sul ruolo delle stats "attivato", che divenne sinonimo di fattore di trascrizione Tyr-fosforilata, il ruolo del non-fosforilata Stats (U-stats) nella regolazione della trascrizione sta emergendo rapidamente. 12 Tuttavia, questi meccanismi sono poco conosciute, e non era chiaro se U-stats in realtà si legano al DNA o di agire attraverso le interazioni con altri fattori di trascrizione. Abbiamo recentemente dimostrato che l'U-STAT3 può legarsi al DNA sequenzialeCES diverso da motivi a gas con ancora maggiore affinità. 13 La scoperta ha implicazioni significative per la nostra comprensione delle funzioni biologiche di questa importante proteina. Abbiamo applicato thermophoresis microscala per determinare affinità relative di STAT3 a GAS e AT-ricco oligonucleotide S 100 (Figura 4). Protocollo pressoché identico è stato utilizzato per la determinazione K D per il legame di un ligando diverso STAT3, un inibitore lipopeptide. 14 N. vincolante per un fattore di trascrizione correlato, GFP-STAT1 che è stato utilizzato come controllo negativo si poteva individuare confermando selettività di interazione. 14
Protocollo
1. Preparazione del lisato
Questo protocollo è destinato cellule aderenti esprimono alcuna proteina GFP-fusa. Numero di cellule necessarie possono variare da un minimo di 10 6 di ben 20 x 10 6 cellule, a seconda del livello di espressione della proteina. Per esempio, lisato di cellule HEK overexpressing GFP-STAT3 è stato preparato trattando cellule coltivate in fiasche T75 10 vicino al 70% di confluenza con 1 ml di tampone di lisi. Tuttavia, questo lisato stati diluiti 150 volte per fornire il livello ottimale di fluorescenza per esperimento MST. Protocollo di lisi cellulare dipende fortemente dalle proprietà e la localizzazione intracellulare della proteina in esame. Se si utilizza un detersivo è indesiderabile a causa dell'instabilità proteica, sonicazione descritta di seguito, potrebbe essere la scelta migliore. Diversi additivi possono essere aggiunti al tampone di lisi per prevenire reazioni di modificazione proteica: EDTA impedisce fosforilazione, vanadato sodico inibisce proteine fosfatasi tirosina, cosìdium fluoruro è un inibitore di Ser / Thr fosfatasi.
- Preparare tampone di lisi. Per estrarre a-easy-proteine citoplasmatiche la seguente composizione funziona bene: 25 mM Tris HCl, pH 8.0, e cocktail inibitore di proteasi diluita 100 volte (Sigma-Aldrich P2714, consistente in 2 mM AEBSF, 0.3 mM aprotinina, 130 pM Bestatina, 1 mM EDTA, 14 mM E-64, 1 mM leupeptina). In alternativa, per le proteine meno solubili, si può usare tampone RIPA commerciale con inibitori della proteasi cocktail e detergenti non denaturanti. Tenere il tampone sul ghiaccio.
- Lavare le cellule brevemente con PBS freddo utilizzando 10 ml di tampone per T75 pallone. Mantenere le cellule in ghiaccio per 5 minuti, o fino a quando le cellule iniziano staccandosi dal pallone. Raschiare le cellule con raschietto cella di staccare, se necessario.
- Risospendere le cellule in 10 ml di PBS freddo ghiaccio e trasferimento ad un prechilled 14 ml a fondo sferico da centrifuga.
- Cellule pellet per centrifugazione a 400-600 xga 4 ° C per 5 min. Combinare le cellule di diversi palloni, a questo punto if necessario.
- Rimuovere PBS surnatante e risospendere pellet in 200 ml di tampone di lisi ghiacciato, il trasferimento di sospensione per un pre freddo 1,5 ml provetta Eppendorf.
- Mantenere le cellule in ghiaccio per ridurre al minimo il surriscaldamento locale. Lyse cellule con 3x 10 sec impulsi di ultrasuoni al di ampiezza 30%, con un 2-3 mm punta pre-raffreddata. Tenere la punta sotto la superficie per ridurre al minimo la formazione di schiuma. Omettere questo passo quando si usano detergenti contenenti buffer e incubare in ghiaccio per 30 minuti, invece.
- Correggere la soluzione lisato di contenere la concentrazione salina fisiologica (NaCl 100 mM), se necessario, con 5 M NaCl.
- Raccogliere lisati mediante centrifugazione a circa 26.000 xga 4 ° C per 10 min.
- Determinare la diluizione lisato ottimale (come descritto nella sezione 3 di seguito) e la quantità di lisato necessaria per una titolazione (in genere circa 300 ml di lisato pre-diluito).
- Aliquotare il lisato e conservare a temperatura appropriata per la proteina in esame (-80 ° C, in molti casi).
- Poiché le interazioni proteina-ligando dipendono dalle condizioni di buffer, MST composizione del tampone viene scelta in base alle proprietà di un particolare sistema. E 'generalmente vantaggioso testare almeno due differenti tamponi.
- Preparare 2 5x buffer MST. Abbiamo avuto buone esperienze con queste due composizioni: HEPES (250 HEPES mM, pH 7.4, 25 mM MgCl2, 500 mM NaCl, 0.25% NP-40) e Tris HCl (250 mM Tris HCl, pH 7.4, 750 mM NaCl, 50 mM MgCl2; 0,05% Tween-20). L'aggiunta di BSA (5% nella miscela finale vincolante) può aiutare a prevenire l'adesione di proteine per tubi di plastica e di vetro capillari. NaN 3 (0,5 mM) può anche essere incluso per prevenire la crescita di microrganismi.
- Selezionare la sorgente di eccitazione LED con la lunghezza d'onda λ = 470 nm sullo strumento MST.
- Caricare capillari con il cell estratto diluito 2 e 10x con tampone MST.
- Eseguire l'operazione di "Trova Capillari" sul software di controllo dello strumento MST. Il range ottimale di fluorescenza in lisato diluito è da 400 a 1.500 unità di fluorescenza.
- La più alta concentrazione del ligando deve essere almeno 20 volte superiore alla costante di dissociazione atteso.
- La concentrazione minima ligando deve essere inferiore concentrazione molare della proteina fluorescente.
- Posizionare un rack tubo con un numero necessario (di solito 10-16) di 0,5 ml provette LoBind sul ghiaccio. Pipettare 25 ml di tampone MST al fondo di ogni provetta. Aggiungere 25 ml di soluzione madre ligando alla prima vascae (# 1, ligando più alta concentrazione) ed eseguire di serie di diluizioni doppie del ligando con il resto dei tubi. Tenere il rack con i campioni ligando su ghiaccio.
- Cella disgelo lisato lentamente sul ghiaccio.
- Diluire lisato cellulare con tampone MST per fornire il livello ottimale della proteina bersaglio fluorescente nelle reazioni di legame. Concentrazione proteica finale dovrebbe essere vicino al previsto K D o inferiore. Dovrebbe essere aggiustata per ottenere necessario numero di conteggi fluorescenza nella soluzione finale. Per la determinazione della concentrazione di GFP-STAT3 nel lisato, lo strumento è stato calibrato con un ausilio di fluoresceina. La concentrazione molare di GFP nel lisato è stato determinato usando rispettivamente il rapporto di fluoresceina e EGFP rese quantiche, 0,85 e 0,61. 15
- Selezionare la sorgente di eccitazione LED con λ = 470 nm sullo strumento MST.
- Luogo provette da 0,5 ml LoBind nel tube cremagliera di fronte tubi con ligando campioni di diluizione in serie. Aggiungere con cautela 15 ml di lisato cellulare al fondo di ogni tubo. Provare a non toccare pareti del tubo per evitare la perdita del campione.
- Aggiungere 15 microlitri di campione ligando con la più alta concentrazione (# 1) al tubo corrispondente # 1 con il lisato cellulare. Mescolare bene e cambiare una punta della pipetta. Ripetere questa fase con il resto dei tubi eccetto l'ultima, che dovrebbe contenere alcun ligando.
- Aggiungere 15 ml di tampone MST all'ultima provetta e mescolare bene.
- Riempire circa 2/3 della prima capillare con la miscela associazione dal tubo # 1, inclinarlo per spostare la soluzione verso il centro, e posizionare il capillare sul vassoio capillare nella posizione # 1 (la posizione più vicina all'apertura vassoio) . Ripetere questa operazione con il resto capillari. Estremità del capillare possono essere collegati con la cera per gli esperimenti più lunghi.
- Collocare il vassoio all'interno dello strumento MST e chiudere la porta dello strumento.
- Eseguire "Trova Capillari"comando per lasciare lo strumento trovare posizioni precise dei capillari e misurare la fluorescenza dei campioni.
- In base all'intensità del segnale di fluorescenza, regolare la potenza del LED (10-100%) per portarlo in 400-1,500 unità di intervallo.
- Fare clic sul pulsante "Start" per eseguire l'esperimento thermophoresis. Più di una potenza IR-laser può essere scelta per l'esperimento per trovare il gradiente di temperatura ottimale per il particolare sistema. Raccogliere dati 2-3 piste per lo stesso insieme di capillari per garantire la riproducibilità delle misurazioni. Ci vogliono 10-12 minuti per eseguire una serie di 16 capillari.
- Aprire il software di analisi.
- Caricare la cartella del progetto. Nel Informazioni Run Viewer apparso selezionare raccolti ad un laser di potenza specifiche curve thermophoretic IR. C'è una possibilità di aprire tutte le tracce thermophoretic raccolti in diverse condizioni quali la potenza del laser a infrarossi, LED power, temperatura, concentrazione, ecc. in una sola volta e poi la scelta di eventuali curve di analisi da loro accensione e spegnimento (clicca sul nome del dell'esperimento).
- Nella finestra del grafico i punti di valutazione, selezionare l'thermophoresis o thermophoresis con T-salto. Assicurarsi che le linee blu e rosse sono posizionati correttamente. Per ottenere i punti medi con deviazioni standard, selezionare "Utilizza media" o "Distinguere corre" per esecuzioni separate.
- Per tracciare una costante forma di dissociazione, selezionare "uso medio", inserire e fissare il valore di concentrazione molecola etichettato (controllare la piazza "concentrazione" in un menu a finestra in forma), e montare la curva. Il valore di K D con la sua deviazione standard compare in una informazione finestra pop-up separata. Per tracciare una misura con il metodo Hill, selezionare "Media", metodo di Hill, e poi misura la curva. Il valore di affinità EC 50 con la sua deviazione standard è mostrato in questo caso. La caratteristica di K D o Hill "confine" può essere utilizzato anche quando la saturazionein uno stato vincolato non viene raggiunto.
- Salvare i dati medi di adattarsi in un file di testo e trasferire in Excel.
- Plot F norma (fluorescenza normalizzata), Af norma (differenza di fluorescenza normalizzato se diversi esperimenti sono confrontati), o frazione legata (vedere la formula seguente) come funzione della concentrazione molecola non marcato (titolato).
2. MST Buffer selezione e preparazione
3. Determinazione ottimale lisato di diluizione
4. Determinazione del legante ottimale range di concentrazione
Fare riferimento alla Technologies strumento Finder Concentrazione NanoTemper per il ligando concentrazione gamma di stima.
5. Preparazione del lisato e diluizioni Ligand
6. Microscala Thermophoresis Studi di rilegatura
7. Microscala Thermophoresis Data Analysis
Frazione legata (frazione di molecole in un complesso) = (Therm (C)-non legato) / (bound-legato), dove Therm (C) è thermophoresis misurato per la concentrazione di C, non legato è thermophoresis per lo stato non legato (quando le molecole sono non in un complesso), e legato viene thermophoresis per lo stato completamente legato.
Risultati
Misurando l'affinità di non fosforilata STAT3 legame proteico oligonucleotidi.
Cellule HEK293 esprimenti STAT3-GFP sono stati utilizzati come fonte di fluorescente STAT3 per saggio di legame al DNA. I lisati cellulari sono stati preparati usando tampone RIPA (20x10 6 cellule / ml). Per studi di legame, i lisati sono stati diluiti con 150x MST tampone di legame al DNA per fornire il livello ottimale della proteina fluorescente nella reazione di legame (circa 20 nM). HEK293 cellule non transfettate sono state utilizzate per valutare fluorescenza di fondo, che si è rivelato essere non rilevabile anche nel lisato non diluito. Tuttavia, fluorescenza di fondo può essere più significativi in altri sistemi di espressione e deve quindi essere seguita. Titolazione serie composta da 11 miscele leganti e campione lisato senza il ligando è stato redatto. Ogni campione conteneva 15 microlitri di lisato cellulare diluito e 15 pl di oligonucleotidi soluzioni di varia concenioni. Composizione del buffer finale inclusa HEPES 25 mM, pH 7.2, 50 mM NaCl, 2,5 mM MgCl2, e 0,025% NP-40. I dati sono rilevati in capillari trattati serie su NT.115 strumento Monolith utilizzando il 50% di potenza IR-laser e sorgente di eccitazione a LED con λ = 470 nm a temperatura ambiente. Il legame di oligonucleotidi ad alta carica portato a cambiamenti significativi nel STAT3 mobilità nel gradiente di temperatura (Figura 2). Nella Figura 3, il segnale thermophoretic viene tracciata come funzione della concentrazione di oligonucleotidi. Ogni punto rappresenta la media di tre misurazioni. Software NanoTemper Analisi 1.2.20 è stato utilizzato per adattare i dati e per determinare i valori apparenti K D. Le apparenti costanti di dissociazione erano 37.9 ± 1.0 micron e 23,3 ± 0,6 micron per il gas e S 100, rispettivamente (Figura 4). Sostituzione di A-to-G comportato drammatica diminuzione di affinità S 100 mut # 1 (figura 4), mentre mutant S 100 mut # 2 non ha mostrato alcun rilevabile vincolante confermando così vincolante sequenza-selettivo di STAT3 a S 100. Sorprendentemente, S sequenze di 100 visualizzati leggermente più stretto legame di GAS in tre ripetizioni della misura.
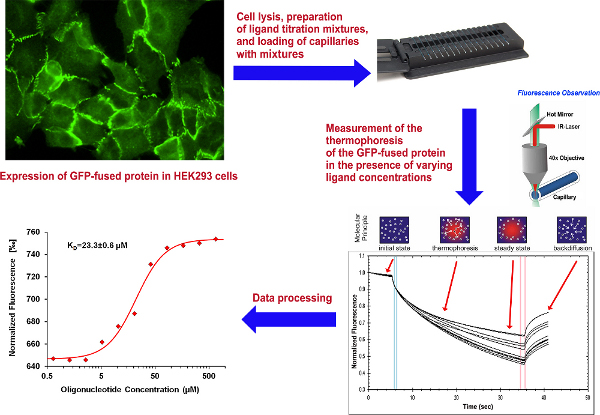
Figura 1. Schema generale di questo esperimento. Clicca qui per ingrandire la figura .

Figura 2. I dati non elaborati generati thermophoresis per l'interazione di GFP-STAT3 con AT-ricchi oligonucleotide. Diluito lisato containi ng 20 nM GFP-STAT3 è stato miscelato con quantità crescenti di oligonucleotide a doppio filamento (5'-AAAACAAAACGAAACAAACAAACTA) riportando le concentrazioni del ligando specificati. I dati sono stati raccolti a potenza laser 50% e il 100% a LED.
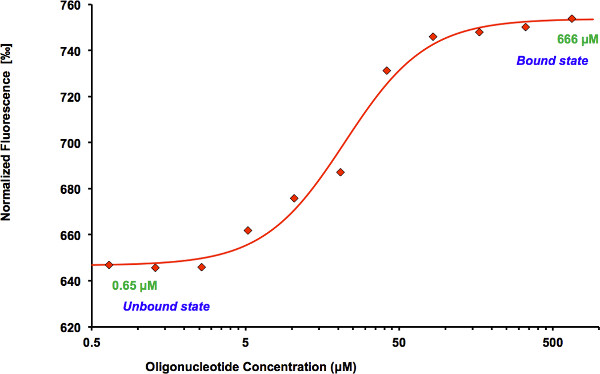
Figura 3. Curva vincolante generato dal software di analisi NanoTemper 1.2.231. Fluorescenza normalizzata (fluorescenza fluorescenza / iniziale caldo) è tracciata in funzione della concentrazione di oligonucleotidi. La proteina mostra un aumento della fluorescenza del bound rispetto allo stato non legato. I dati vengono montate con il metodo equazione Hill incorporato nel software di analisi NanoTemper.
0541fig4highres.jpg "src =" / files/ftp_upload/50541/50541fig4.jpg "/>
Figura 4. STAT3 vincolanti per gli oligonucleotidi con sequenze diverse. Microscala thermophoresis misure vincolanti di STAT3-GFP al gas (K D = 37.9 ± 1.0 mM), S 100 (K D = 23,3 ± 0,6 micron), S 100 mutante 1 (K D = 740 ± 21 micron), e S 100 mutante 2 (non vincolante). La concentrazione STAT3-GFP è stata mantenuta costante a circa 20 nM, e la concentrazione di oligonucleotidi varia 666-0,650 pM. La differenza di fluorescenza normalizzato [‰] viene tracciata come funzione della concentrazione oligonucleotide, e le curve vengono montate con il metodo Hill del software di analisi NanoTemper. Barre di errore rappresentano l'errore standard di tre misurazioni.
Discussione
Espressione e purificazione di proteine è un passo laboriosa e costosa, che è, tuttavia, necessario per la determinazione delle interazioni 'K D dal metodo più usato. Applicazione del MST permette di evitare la purificazione di proteine così in modo significativo semplificare ed accelerare la caratterizzazione quantitativa delle interazioni. Esso presenta vantaggi particolarmente significativo nel caso di-to-esprimere difficili e purificare proteine, quali le proteine di membrana e fattori di trascrizione.
La maggiore limitazione e esigenza di MST è la capacità di esprimere una proteina come una fusione con la proteina fluorescente verde. Tuttavia, i costrutti di espressione di maggioranza delle proteine umane e di topo GFP-fuse sono disponibili in commercio. Proteine GFP-fusi sono anche ampiamente utilizzati per il traffico e studi di degradazione e, quindi, può servire "doveri matrimoniali" in combinazione con studi MST.
Oltre alla mancanza di necessità di proteina purification, uso molto economico di tutti i reagenti e insensibilità a lievi variazioni nella composizione del buffer, MST offre altri vantaggi rispetto ai metodi tradizionali di K D determinazioni. A differenza di risonanza plasmonica di superficie, esperimenti MST-based in meno di 2 ore, consentono la determinazione di K D nella gamma più ampia e non soffrono di superficie artefatti immobilizzazione. Per esempio, non abbiamo potuto determinare K D per STAT3 vincolante per oligonucleotidi di SPR, anche se abbiamo investito notevole quantità di fondi per l'acquisto di purificata STAT3 proteine. K D dell'interazione era troppo alto, che è spesso il caso per fattori di trascrizione, e cadde fuori campo di concentramento di esperimento SPR.
Titolazione calorimetria funziona solo per le interazioni che sono accompagnati da cambiamenti di entalpia. Questa limitazione esclude molti casi. Nel frattempo, i cambiamenti nella mobilità thermophoretic, anche se differiscono molto in valori disistemi fferent, si verificano per la stragrande maggioranza delle interazioni. Uno dei maggiori vantaggi di MST è che funziona in una varietà di buffer e tollera la presenza di detergenti, micelle e bicelle nel sistema. Questa proprietà consente di applicarlo a proteine di membrana che sono praticamente impossibili da studiare con altri mezzi. Tuttavia, l'attenzione deve essere usata per evitare le variazioni di detersivo e contenuti micelle e la composizione durante la titolazione con il ligando.
Va tenuto inoltre conto quando si utilizzano estratti cellulari che la proteina sotto studio può esistere nella sua forma nativa, che è spesso complessi con altre proteine, acidi nucleici, e cofattori. Così, affinità di legame può essere differente da quello per una proteina isolata non coinvolto in formazioni complesse. Sovraespressione permette frequentemente titolando i partner di interazione lasciando la maggior parte delle molecole della proteina studiata in stato non legato. Questo è un altro motivo per cercare di ragve molto elevati livelli di espressione della proteina GFP-fusa sulle cellule trasfezione. L'altro parametro di difficile controllo è modificazioni post-traduzionali che può anche aggiungere una significativa eterogeneità al sistema. Limitazione aggiuntiva sta studiando legame alle proteine e piccole molecole che sono presenti nei lisati cellulari in grandi quantità o molecole con un'ampia specificità e la bassa affinità che possono interagire con più proteine presenti nel lisato. Wienken, CJ, et al. hanno trovato che K D determinata da MST in E. Estratto coli era più di un ordine di grandezza superiore in un buffer per l'interazione di interferone gamma con anticorpi 9. L'effetto può essere stato in parte dovuto alla proteolisi poiché l'estratto aveva inibitori della proteasi. Legame con l'albumina sierica piccola molecola è stata trovata anche a cambiare K D in studi di interazione con MST 9.
L'efficacia di estrazione può differire anche per differeproteine nt seconda localizzazione intracellulare e proprietà fisico-chimiche. Così, è utile provare diversi lisi cellulare e condizioni di estrazione della proteina. Per le proteine citoplasmatiche, senza usare detergenti, solo ultrasuoni per rottura delle cellule produce spesso ottimi rendimenti e dati. Nel frattempo, le proteine di membrana richiedono più approfondita delle condizioni di estrazione, con concentrazioni variabili e la natura dei detersivi, micelle lipidiche, o bicelle. Contenuti detergente deve essere tenuto al minimo per evitare di influenzare affinità di legame.
Nonostante le limitazioni elencate, abbiamo trovato studi MST di non-proteine purificate GFP-fusi per essere molto comoda per la quantificazione delle affinità di legame per molte proteine e tipi di ligando. Non vi è dubbio che l'estensione del metodo in molti laboratori consentirà di ampliare ulteriormente le applicazioni di MST-based studi di legame proteico.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari in competizione. Il contenuto di questa pubblicazione non riflettono necessariamente le opinioni o le politiche del Dipartimento di Salute e Servizi Umani, né menzione di nomi commerciali, prodotti commerciali, o organizzazioni implica l'approvazione da parte del Governo degli Stati Uniti.
Riconoscimenti
Questo lavoro è stato parzialmente supportato dal programma di ricerca intramurale del NIH, National Cancer Institute, Centro per la Ricerca sul Cancro; accordo di collaborazione di ricerca tra NSC e Calidris Therapeutics, American Cancer Society concessione IRG 97-152-17 di OT, e fondi federali dal National Cancer Institute, NIH, sotto HHSN26120080001E contratto.
Materiali
| Name | Company | Catalog Number | Comments |
| RIPA buffer | Millipore | 20-188 | Other manufacturer's buffers work as well |
| Protease inhibitors cocktail | Sigma-Aldrich | P2714 | |
| Monolith NT.115 | NanoTemper Technologies GmbH | G008 | |
| Monolith NT.115 Capillary Tray | NanoTemper Technologies GmbH | T001 | |
| Monolith NT.115 Standard Treated Capillaries | NanoTemper Technologies GmbH | K002 | |
| NT Control software | NanoTemper Technologies GmbH | 2.0.2.29 | |
| NT Analysis software | NanoTemper Technologies GmbH | 1.4.27 | |
| Table-top refrigerated centrifuge | Eppendorf | 5417R | Other microtube refrigerated centrifuges providing |
| Protein LoBind Tube 0.5 ml | Eppendorf | 22431064 |
Riferimenti
- Lea, W. A., Simeonov, A. Fluorescence polarization assays in small molecule screening. Expert. Opin. Drug Discov. 6, 17-32 (2011).
- Willander, M., Al-Hilli, S. Analysis of biomolecules using surface plasmons. Methods Mol. Biol. 544, 201-229 (2009).
- Hulme, E. C., Trevethick, M. A. Ligand binding assays at equilibrium: validation and interpretation. Br. J. Pharmacol. 161, 1219-1237 (2010).
- Ghai, R., Falconer, R. J., Collins, B. M. Applications of isothermal titration calorimetry in pure and applied research--survey of the literature from 2010. J. Mol. Recognit. 25, 32-52 (2012).
- Vuignier, K., Schappler, J., Veuthey, J. L., Carrupt, P. A., Martel, S. Drug-protein binding: a critical review of analytical tools. Analytical and Bioanalytical Chemistry. , 398-3953 (2010).
- Lebowitz, J., Lewis, M. S., Schuck, P. Modern analytical ultracentrifugation in protein science: a tutorial review. Protein Sci. 11, 2067-2079 (2002).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc. Natl. Acad. Sci. U.S.A. 103, 19678-19682 (2006).
- Zillner, K., Jerabek-Willemsen, M., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol. Biol. 815, 241-252 (2012).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat. Commun. 1, 100 (2010).
- Stark, G. R., Darnell, J. E. The JAK-STAT pathway at twenty. Immunity. 36, 503-514 (2012).
- Ehret, G. B., Reichenbach, P., et al. DNA binding specificity of different STAT proteins. Comparison of in vitro specificity with natural target sites. J. Biol. Chem. 276, 6675-6688 (2001).
- Yang, J., Stark, G. R. Roles of unphosphorylated STATs in signaling. Cell Res. 18, 443-451 (2008).
- Timofeeva, O. A., Chasovskikh, S., et al. Mechanisms of unphosphorylated STAT3 transcription factor binding to DNA. J. Biol. Chem. 287, 14192-14200 (2012).
- Timofeeva, O. A., Tarasova, N. I., et al. STAT3 suppresses transcription of proapoptotic genes in cancer cells with the involvement of its N-terminal domain. Proc. Natl. Acad. Sci. U.S.A. , (2013).
- Patterson, G., Day, R. N., Piston, D. Fluorescent protein spectra. J. Cell Sci. 114, 837-838 (2001).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon