Method Article
Analisi di espressione di mammifero Linker-istoni Sottotipi
In questo articolo
Riepilogo
Descriviamo una serie di saggi per analizzare i livelli di espressione di istoni linker H1. mRNA di geni H1 individuali sono quantitativamente misurato casuale trascrizione inversa primer a base seguita da PCR in tempo reale, mentre la quantificazione della proteina istoni H1 è ottenuta mediante analisi HPLC.
Abstract
Linker istone H1 si lega alla particella nucleo nucleosoma e il DNA linker, facilitando ripiegamento della cromatina nella struttura di ordine superiore. H1 è essenziale per lo sviluppo dei mammiferi 1 e regola l'espressione genica specifica in vivo 2-4. Tra le proteine altamente conservate istoniche, la famiglia degli istoni linker H1 è il gruppo più eterogeneo. Ci sono 11 sottotipi H1 in mammiferi che sono differenzialmente regolati durante lo sviluppo e in differenti tipi cellulari. Tali sottotipi H1 includono 5 somatica H1S (H1a-e), l'H1 sostituzione 0, 4 sottotipi H1 cellule germinali specifici e H1x 5. La presenza di sottotipi H1 multipli che differiscono nella affinità di legame al DNA e capacità compattazione cromatina 6-9 fornisce un ulteriore livello di modulazione della funzione cromatina. Così, l'analisi quantitativa di espressione dei sottotipi H1 individuali, sia di mRNA e di proteine, è necessario per una migliore comprensione del regolamento dell'istruzione superiorePer la struttura della cromatina e la funzione.
Qui si descrive una serie di saggi progettati per analizzare i livelli di espressione di singoli sottotipi H1 (Figura 1). espressione di mRNA di vari geni varianti H1 è misurata da un insieme di altamente sensibili e quantitativa PCR di trascrizione inversa (qRT-PCR) saggi, che sono più veloce, più accurata e richiedono campioni molto meno rispetto al metodo alternativo di analisi Northern blot. A differenza di molti altri messaggi mRNA cellulari, mRNA per geni più istone, compresa la maggior parte dei geni H1, non hanno una lunga coda polyA, ma contengono uno stem-loop struttura alla regione 3 'non tradotta (UTR) 10. Pertanto, cDNA sono preparati da RNA totale mediante trascrizione inversa usando primer casuali invece di oligo-dT primer. Tempo reale PCR con primer specifici per ciascuna sottotipi H1 (Tabella 1) vengono eseguite per ottenere una misurazione altamente quantitativa dei livelli di mRNA di sottotipo H1 individuales. Espressione dei geni costitutivi sono analizzati come controlli per la normalizzazione.
L'abbondanza relativa di proteine di ciascun sottotipo H1 e istoni nucleo è ottenuto mediante fase inversa la cromatografia liquida (RP-HPLC) su istoni totale estratto da cellule di mammifero 11-13. Il metodo HPLC eluizione e le condizioni qui descritte dare separazioni ottimali di sottotipi H1 mouse. Per quantificare il profilo HPLC, si calcola la percentuale relativa di sottotipi H1 individuali all'interno famiglia H1, oltre a determinare il rapporto di H1 nucleosoma nelle cellule.
Protocollo
1. Preparazione del campione ed estrazione RNA
- Prima di estrazione di RNA, tutte le superfici di lavoro e pipette strofinata con 70% etanolo e trattato con soluzione di decontaminazione RNasi, come RNasi Zap. Questa pratica riduce le possibilità di contaminazione da RNasi e la degradazione dell'RNA. Indossare guanti per tutte le procedure.
- Per estrarre l'RNA da tessuti di topo, sezionare l'organo di interesse da topo sacrificati, e lavare il tessuto in fosfato ghiacciato Soluzione salina tamponata (PBS: 0,13 M NaCl, 5 mM sodio fosfato bibasico eptaidrato, 5 mM sodio eptaidrato diidrogeno fosfato, pH 7,4) . Procedere immediatamente a RNA estrazione in fase 1.4. Se il tessuto fresco non deve essere trattati per l'estrazione di RNA, i campioni di tessuto dovrebbero essere scatto congelati immediatamente in azoto liquido e conservati a -80 ° C per un uso successivo.
- Se RNA deve essere estratto da colture di cellule aderenti, aspirare terreni di coltura, sciacquare con una quantità sufficiente di PBS e aggiungere reagente Trizol (Invitrogen) sulla piastra e passare al punto 1.4. Per le cellule coltivate in sospensione, raccogliere le cellule e le cellule pellet mediante centrifugazione. Eliminare i media, sciacquare il pellet brevemente con PBS, pellet e le celle con centrifugazione. Aggiungere il reagente Trizol e passare al punto 1.4.
- Reagente Trizol sufficiente è necessario per ottenere RNA di alta qualità. Usare 1 Reagente Trizol ml per estrarre RNA 50-100 mg di tessuto, 5 - 10 x 10 6 cellule (per colture in sospensione) o per 3,5 cm di piastra per le culture aderenti). Omogeneizzare il tessuto nel reagente Trizol con Polytron PT2100 omogeneizzatore (o equivalente). Procedere per estrarre l'RNA da campioni di tessuto o cellule in base al manuale del costruttore per il reagente Trizol (Invitrogen).
- Concentrazione di RNA viene misurata utilizzando NanoDrop 1000 (Thermo Scientific) e la qualità RNA viene analizzato mediante elettroforesi su gel. Le fasce di resa tipiche pg 1-10 di RNA per mg di tessuto o 5-15 pg di RNA per 1 x 10 6 cellule in coltura. Per eliminare l'contaminazione potenziale di RNA da quantità in tracce di DNA genomico, campioni di RNA sono trattati con DNasi RNase-free (Sigma AMP-D1) secondo le istruzioni del produttore. Ripetere la determinazione della concentrazione di RNA e elettroforesi su gel di garantire alcuna degradazione dell'RNA da questo trattamento. Conservare estratto RNA a -80 ° C.
* Nota: RNA possono anche essere estratti utilizzando RNAeasy kit (Qiagen) secondo il manuale kit, o da DNA / RNA kit (Qiagen) se sia il DNA e RNA sono desiderati.
2. Reverse Trascrizione PCR quantitativa (qRT-PCR)
- RNA totale è inversamente trascritto in cDNA usando Superscript III First-strand sistema di sintesi (Invitrogen). Poiché mRNA della maggior parte dei geni H1 mancano poli-A-code, è fondamentale utilizzare esameri casuali invece di oligo-dT come primer per la sintesi di cDNA. Tuttavia, se l'analisi di espressione di geni con messaggi poliadenilato a bassi livelli è inoltre desiderato, una miscela di esameri casuali e oligo-dT should essere utilizzato nella trascrizione inversa (RT) reazione per migliorare l'efficienza di trascrizione inversa di mRNA poliadenilato.
- Eseguire la reazione di RT in base al manuale del costruttore. Brevemente,
- In una provetta da 0,5 ml PCR, combinare 5 pg di RNA totale, 1 pl di 50 ng di esameri / pl casuali, 1 pl di miscela dNTP 10 mM, e aggiungere trattata con DEPC H 2 O per fare un volume totale di reazione di 10 pl. Mescolare bene e incubare per 5 minuti a 65 ° C, seguiti da 1 minuto incubazione su ghiaccio.
- Preparare 10 pl di miscela cDNA Synthesis: 2 microlitri di tampone 10xRT, 4 pl di 25 mM MgCl 2, 2 pl di 0,1 M DTT, 1 pl di RNaseOut (40 U / pl), 1 pl di SuperScript III RT (200 U / pl) e aggiungerlo al RNA / primer miscela.
- Incubare per 10 minuti a 25 ° C, seguita da 50 minuti a 50 ° C e terminare la reazione a 85 ° C per 5 minuti.
- Ogni reazione produce tipicamente 100-250 ng / pl del prodotto cDNA. Conservare i prodotti cDNA a -20° C o procedere immediatamente per real-time PCR quantitativa (qPCR).
- qPCR possibile quantificare con precisione le copie sequenza bersaglio ad alta efficienza e riproducibilità 14. Abbiamo scelto qPCR misurata dal colorante SYBR Green, che dà un segnale fluorescente solo quando si intercala con il DNA a doppia elica (dsDNA). Anche se non è specifica come Taqman dosaggio 14, questo metodo è più conveniente, più facile da adottare in laboratorio, ed offre una maggiore versatilità qPCR. Pertanto, è importante esaminare la trama di amplificazione (Figura 2A) e le curve di fusione derivati del prodotto qPCR (Figura 2B) per garantire l'efficienza di reazione e specificità.
| Sottotipi istone | Mouse nomenclatura istone | Umana nomenclatura istone | ||
| Gene il nome | Adesione n. | Gene nome | Adesione n. | |
| Histone H1a | Hist1h1a | NM_030609 | HIST1H1A (H1.1) | NM_005325 |
| H1b Histone | Hist1h1b | NM_020034 | HIST1H1B (H1.5) | NM_005322 |
| H1C Histone | Hist1h1c | NM_015786 | HIST1H1C (H1.2) | NM_005319 |
| H1D Histone | Hist1h1d | NM_145713 | HIST1H1D (H1.3) | NM_005320 |
| H1E Histone | Hist1h1e | NM_015787 | HIST1H1E (H1.4) | NM_005321 |
| H1 ° | H1f0 | NM_008197 | H1F0 | NM_005318 |
| Histone H1oo | H1foo | NM_183811 | H1FOO | NM_153833 |
| H1T Histone | Hist1h1t | NM_010377 | HIST1H1T | NM_005323 |
| H1t2 Histone | H1fnt | NM_027304 | H1FNT | NM_181788 |
| H1x Histone | H1fx | NM_198622 | H1FX | NM_006026 |
| Hils1 istone | Hils1 | NM_081792 | HILS1 | AY286318 |
Tabella 1. Histone nomenclatura sottotipo H1 nel topo e umane.
- Struttura avanti e indietro PCR primers specifici per ciascun gene H1 (Tabella 1). A causa della somiglianza di sequenza alta tra somatica H1S, in particolare nella regione corrispondente al dominio globulare centrale, è fondamentale garantire che i primer disegnati per un sottotipo H1 specifico non si allineano con oltre geni H1 o croce amplificare sottotipi H1 altri. È altresì importante notare che la maggior parte dei geni H1 non contiene introni. Così introne-inneschi che coprono tipicamente adottate per RT-PCR per evitare la contaminazione genomico non sono disponibili. Invece, campioni di RNA deve essere pre-trattati con DNasi (vedi 1.5) per eliminare qualsiasi quantità in tracce di contaminazioni genomico. Inoltre, RT (-)-qPCR deve essere eseguito in parallelo per convalidare la mancanza di contaminazione genomico nei campioni di cDNA.
- Anche primer design per i geni di riferimento interno, la cui espressione non vengono modificati tra i campioni. Spesso geni housekeeping, quali gliceraldeide-3-fosfato deidrogenasi (GAPDH) e beta-actina geni, sono scelti come geni di riferimento. qPCR segnali di geni housekeeping servono come controlli normalizzazione.
- Preparare ciascuna reazione PCR (volume totale 25 pl) come segue: 12,5 pl di 2x IQ SYBR Green Supermix (Bio-Rad) (dNTPs contenente, 50 U / ml DNA polimerasi iTaq, 6 mM MgCl 2, SYBR Green I e 20nM fluoresceina), 2 microlitri di 4 ng / ul cDNA, 1,25 microlitri di 10 nM avanti / mix primer reverse, e 9,25 microlitri di ddh 2 O, e mescolare bene in Microseal piastra a 96 pozzetti PCR. Utilizzare Microseal 'B' guarnizioni adesive (Bio-rad) per assicurare che la piastra di copertura è sigillato alla piastra. Toccare o brevemente vortex la piastra PCR, e far girare le miscele di reazione da una centrifugazione breve. Porre la piastra in unico colore MyIQ real-time PCR Detection System (Bio-rad) per qPCR.
- Si utilizzano le seguenti condizioni: qPCR 95 ° C per 3 minuti, seguiti da 40 cicli di 95 ° C per 10 secondi, 60 ° C per 20 secondi, 72 ° C per 30 secondi. Esaminare le curve di amplificazione (Figura 2A) per PCR efficienza e Ct (soglia di ciclo) valori. La linea di soglia può essere impostato automaticamente dal sistema ottico 2,0 IQ5 versione del software.
- L'efficienza primer e la concentrazione ottimale cDNA necessaria può essere testato da un saggio curva standard, in cui una diluizione seriale di DNA genomico Is usato per qPCR e valori Ct sono riportati in funzione della quantità log template DNA. Un saggio qPCR ottimizzato con primer di alta specificità ed efficienza darà una curva lineare standard, con il coefficiente di determinazione (R 2)> 0,98. Evitare primer con lunghezza amplicon più di 200 bp, che tendono ad avere scarsa efficienza di amplificazione.
- Poiché SYBR Green rileva qualsiasi dsDNA, è fondamentale per eseguire una corsa curva di fusione a seguito della qPCR per assicurare che l'amplicone desiderato, ma non dimeri di primer o contaminanti, vengono amplificati e rilevato. Per melt-analisi della curva, il programma strumento qPCR per riscaldare i campioni da 55 ° C a 95 ° C a 0,5 ° C con incrementi di raccolta dati. L'impostazione di default di melt-analisi della curva per MyIQ (Bio-rad) strumento è il seguente: 95 ° C per 1 minuto, 55 ° C per 1 minuto, seguita da 81 cicli di 10 secondi a 55 ° C setpoint, fondere curva, temperatura + 0,5 ° C (telecamera raccoglie i dati ad ogni ciclo).
- Dato che temperatura di fusione (Tm) di dsDNA dipende dalla lunghezza amplicone contenuto di GC, ampliconi differenti avranno differenti Tm (s). Considerare le curve di fusione dei prodotti derivati qPCR per confermare la temperatura specifica di fusione di ampliconi desiderate, nonché l'assenza di picchi di rumore ampliconi (Figura 2B).
- Preparare doppio o triplo, le reazioni per ogni dosaggio per l'analisi statistica. Include controlli negativi per qRT-PCR, come RT (-)-qPCR (qPCR con RNA come stampo senza trascrizione inversa) e qPCR senza cDNA, RNA o DNA sorgente aggiunti nella miscela PCR. RT (-)-qPCR può servire come controllo per il potenziale contaminazione di DNA genomico (RT (-) in Figura 2 e 3).
- Analizzare i dati con qPCR IQ5 Optical System Software versione 2.0 (Bio-rad). Normalizzare i valori di espressione dei geni H1 isoforma con l'espressione del gene housekeeping (GAPDH ad esempio, ß-actina, HPRT) per ottenere livelli di espressione di geni relativi H1.
* Tutte le procedure devono essere eseguite su ghiaccio oa 4 ° C.
- Staccare il tessuto del mouse e risciacquare con PBS ghiacciato. (Se non procede all'estrazione immediatamente scattare congelare e conservare i campioni come descritto al punto 1.2.) Tritare il tessuto in pezzi con lama di rasoio. Trasferimento carne macinata in un omogeneizzatore Dounce (B pestello). Aggiungere 10 ml tampone saccarosio (0,3 M saccarosio, 15 mM NaCl, 10 HEPES [pH 7,9], 2 mM EDTA, 0.5 mM PMSF, Complete Mini Tablet inibitore della proteasi Cocktail, aggiungere fresca) per grammo di tessuto. Omogeneizzare il tessuto con 10-15 colpi.
- Trasferire gli omogenati a un tubo 15 ml, centrifuga a 500 rpm per 30 secondi (modello centrifuga: Eppendorf 5810R); attentamente trasferire il surnatante in una nuova provetta (scartare il pellet-tessuto detriti), e centrifugare a 2000 rpm per 5 minuti, pellet di cellule. Passare al punto 3.4.
- Se istoni e cromatina vengono estratti da cellule cresciute in monostrato, risciacquocon PBS, aggiungere PBS al piatto cultura, e raccogliere le cellule con raschietto cellulare, e le celle a pellet per centrifugazione. Per cellule coltivate in sospensione, pellet cellule per centrifugazione.
- Risospendere il pellet di cellule in 10 ml di tampone saccarosio integrato con 0,5% NP-40 (per grammo importo di partenza tessuto o 10 (8) celle). Trasferire il campione ad un omogeneizzatore Dounce (B pestello) e Dounce 10 colpi in 20 minuti di incubazione. A questo punto, si ottengono nuclei. Esaminare la qualità nuclei sotto un microscopio. Pellet nuclei mediante centrifugazione a 2000 rpm per 5 minuti. Eliminare il surnatante.
- Risospendere il pellet nuclei in 3 ml di tampone Salt alta (0,35 M KCl, 10 mM Tris [pH 7,2], 5 mM MgCl 2, 0,5 mM PMSF - aggiungere fresco prima dell'uso) per 1 g di tessuto o 10 (8) cellule. Trasferire il campione ad un piccolo Dounce omogeneizzatore (B pestello) e omogeneizzare con colpi 5-10.
- Aliquotare la sospensione in 3 provette Eppendorf (1 ml ciascuna), incubare su ghiaccio per 20 minuti, seguiti dacentrifugazione a 14.000 rpm per 10 minuti a pellet cromatina. Eliminare il surnatante.
- Aggiungere 0,8 ml 0,2 NH 2 SO 4 su ciascuna pellet cromatina. Utilizzare un tubo eppendorf Dounce pestello per macinare il pellet bene fino a che il pellet è completamente dissociato. Incubare i campioni su una piattaforma rotante a 4 ° C per una notte. Totale istoni sono estratti con questo passaggio di trattamento con acido.
- Centrifugare a 14.000 rpm per 10 minuti. Trasferire il surnatante (estratti degli istoni) in due provette Eppendorf (400 pl / tubo). Eliminare il pellet. Aggiungere 2,5 volumi (1 ml) di etanolo ghiacciato a ciascuna provetta .. Conservare i campioni a -20 ° C per una notte.
- Centrifugare a 14.000 rpm per 10 minuti per gli istoni pellet totali, scartare il surnatante. Lavare i pellet tre volte con il 70% EtOH, lasciare in panchina per 20-30 minuti per asciugare all'aria. Conservare le proteine essiccati a -80 ° C o dissolvere in ddh 2 O e procedere all'analisi HPLC immediatamente. Proteine secchi possono essere memorizzatea -80 ° C per un massimo di 1 anno.
4. Analisi HPLC degli istoni linker
- Risospendere il istone pellet nel volume raccomandata di ddh 2 O a seconda della capacità della colonna a fase inversa e lo strumento HPLC. Usiamo colonna C18 a fase inversa 250 x 4,6 mm (Vydac) e Äktapurifier UPC 900 strumento (GE sanitaria) per l'analisi HPLC. Abbiamo tipicamente dissolvere 50-100μg di istoni totali in 100 pl di ddh 2 0 per l'analisi.
- Centrifugare a 14.000 rpm per 5 minuti per rimuovere residui insolubili. Saggio proteico Bradford viene utilizzato per determinare la quantità di proteina da iniettare nella colonna. Iniettare 50-100 microgrammi di proteine totali nella colonna a fase inversa sul sistema HPLC. Caricamento di una quantità eccessiva di proteine deve essere evitato per prevenire l'occlusione della colonna.
- Frazionare gli istoni linker e gli istoni core con un gradiente crescente di acetonitrile come indicato nella Tabella 2.
Tabella 2. Aumento gradiente di acetonitrile nel tempo.
- L'effluente viene monitorato a 214 nm, e il profili HPLC (Figure 4) sono stati registrati e analizzati utilizzando Äktapurifier UPC 900 (GE Healthcare) con 5,11 UNICORN software (GE Healthcare). Le frazioni proteiche possono essere raccolti anche con frazione di auto-collector (FRAC-920 - GE) per ulteriori analisi, ad esempio spettrometria di SDS-PAGE e di massa.
- A 214 I valori dei picchi di ciascun sottotipo H1 e H2B sono normalizzati per il numero di legami peptidici di ogni corrispondente proteina istone. La proporzione relativa dei singoli sottotipi H1 istone H1 all'interno della famiglia, così come il rapporto tra sottotipi H1 di particelle di nucleo nucleosoma possono essere calcolati da questi valori normalizzati A 214 (Figura 5).
5. Risultati rappresentativi
L'elenco dei sottotipi H1 mammiferi, diagramma di flusso complessivo ei risultati rappresentativi della analisi di espressione di geni singoli istone H1 sono mostrati nella Tabella 1, Figura 1 e Figura 2-5, rispettivamente.La figura 2a mostra curve di amplificazione tipici della H1A reazioni qPCR utilizzando cDNA preparato da fegato di topo e mESCs, mentre la figura 2B mostra le curve di fusione derivati gli ampliconi corrispondenti. La curva di fusione mostra un singolo picco caratteristico a temperatura di fusione (Tm) a 86 ° C per la PCR H1a amplicone, e manca di non-specifici picchi fondo, suggerendo elevata specificità del test H1a qPCR. Valutazione della trama di amplificazione (Figura 2A) mostra che le reazioni qPCR triplicato di ciascun campione dato segnali compatibili con quasi identici valori Ct, suggerendo alta riproducibilità. La mancanza di ampliconi build-up da RT (-)-qPCR reazioni indicano che la contaminazione da DNA genomico non era presente, o minima. Utilizzando i valori di Ct geni H1 e geni housekeeping, quali GAPDH, i relativi livelli di espressione di RNA di ciascun gene H1 sono stati calcolati. Esempi di risultati calcolati per H1 ° e geni H1A sono mostrati nella Figura 3. I livelli di espressione di mRNA relativi H1a sono più alti in mESCs rispetto al fegato di topo, mentre H1 ° espressione è molto più elevata nel fegato che nel mESCs.
La differenza di espressione di H1a o H1 ° in fegato di topo adulto rispetto MESC è evidente anche profili HPLC di istoni (Figura 4). H1 °, la H1 differenziazione specifica, viene accumulato ad una grande quantità nei tessuti maturi, pari al 27,2% del totale H1 nel fegato adulto (Figura 5A). Al contrario, H1 ° proteina è quasi assente in mESCs indifferenziate (Figura 4B). D'altra parte, H1a è altamente espresso, sia in trascritti di mRNA e proteine, in mESCs (Figura 3 e 4B). Attraverso quantificazione dei picchi H1 a profilo HPLC, la proporzione relativa di ciascun sottotipo H1 individuo all'interno della famiglia H1 è determinato (figura 5A). Inoltre, i valori di sottotipo H1 individuale (oH1 totale) per nucleosoma può essere calcolato il rapporto del valore normalizzato A 214 picco di sottotipi H1 corrispondenti (o somma totale di H1) alla metà della normalizzato A 214 valori per H2B (Figura 5B).
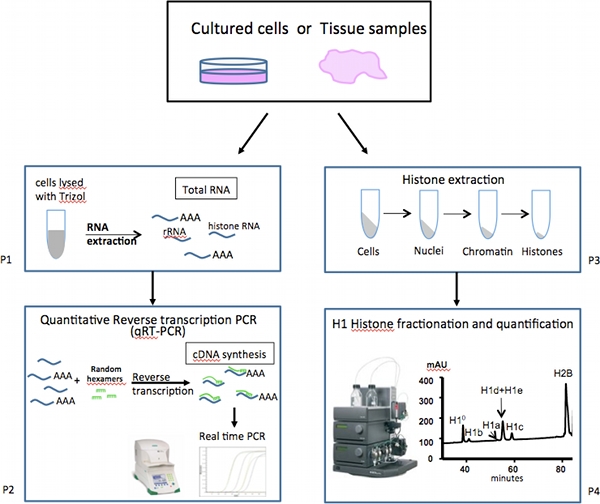
Figura 1. Schema generale di analisi di espressione di mammiferi linker-istoni sottotipi.
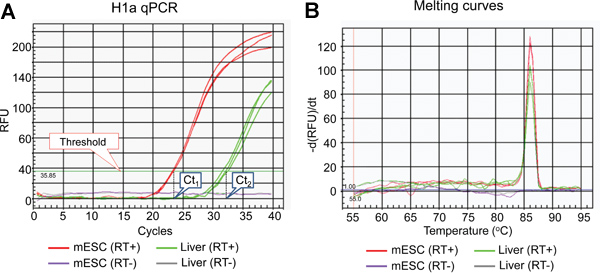
Figura 2. Risultati rappresentativi di dosaggio H1a qPCR. (A) plot di amplificazione del test H1a qPCR. La linea di soglia e valori Ct fissati dal Software IQ5 sistema ottico sono indicati. (B) derivati fusione curve di prodotti qPCR mostrato in (A).

Figura 3. QRT-PCR analisi dei livelli di mRNA e di H1a H1 ° in mESCs e adulti mOuse fegato. Asse Y rappresenta livelli di espressione relativi di geni H1 a quella del gene di riferimento GAPDH. qPCR con RT (-) campioni di RNA (senza trascrizione inversa) mostra segnali minimi o no.
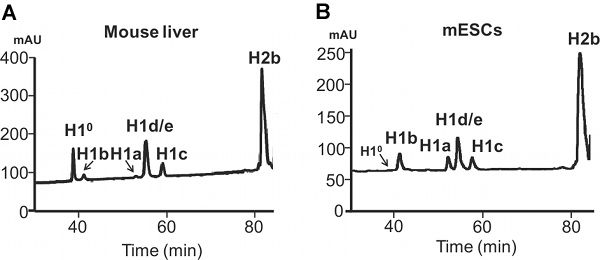
Figura 4. Analisi HPLC di istoni estratti da cellule di mammifero. Reverse-fase di analisi HPLC di 100 mcg istoni totale estratto dal fegato di topo adulto (A) e CES del mouse (B). Asse X: tempo di eluizione. Asse Y: mAU, milli-unità di assorbimento.

Figura 5. Composizione del sottotipo H1 e H1 per i rapporti nucleosoma nel fegato di topo adulto. A 214 I valori di area di picco per ciascuna isoforma H1 e H2B vengono calcolate usando software UNICORN 5,11 (GE Healthcare), e normalizzati per il numero di legami peptidici presenti nella proteina istone corrispondente. La somma di A normalizzato214 valori di tutti i sottotipi H1 è ottenuta come il valore totale per H1. La percentuale totale di H1 per ciascun sottotipo H1 (A), nonché il rapporto di H1 a nucleosomi (rappresentato da una metà del normalizzato A 214 valori di H2B) (B) nel fegato di topo adulto sono calcolati dal profilo HPLC mostrato in Figura 4A.
Discussione
L'insieme dei saggi qui presentati consentono un'analisi complessiva dei livelli di espressione di mammifero sottotipi istone linker. Adeguatamente progettati RT-PCR test forniscono misurazioni estremamente sensibili e accurate dei messaggi RNA da eventuali geni di mammifero H1. La parte critica della qRT-PCR per linker geni sottotipo istone è la preparazione di cDNA utilizzando primer casuale basato trascrizione inversa. mRNA di geni più istoni, incluse maggior parte dei geni H1, non contengono una lunga coda poli-A presentato in altre mRNA cellulari. Così il metodo tradizionale trascrizione inversa con oligo-dT primer non produrre in modo efficiente cDNA H1. Analisi espressione dei geni H1 pochi con trascritti di mRNA contenenti coda poli-A, come H1 0, è ugualmente efficace con esameri casuali in base qRT-PCR (Figura 3), probabilmente a causa della grande abbondanza di RNA H1. Tuttavia, una miscela di primer oligo-dT e le esameri casuali per la reazione RTzione possono essere adottate per migliorare l'efficienza RT di mRNA poliadenilato che sono di numero di copie basso, consentendo più ampia copertura dei geni analizzati da qRT-PCR. qRT-PCR dei geni di riferimento interni, quali housekeeping gene GAPDH, è incluso in modo che il relativo livello di espressione di geni specifici in H1 tessuti differenti o tipi cellulari possono essere confrontati (Figura 3). Combinando qRT-PCR con analisi curva standard, è anche possibile ottenere un numero di copie assoluti di cDNA H1 da vari campioni (dati non mostrati).
Qui si descrivono anche i protocolli per l'estrazione degli istoni e l'analisi HPLC delle proteine istoniche. Il vantaggio di questo metodo è che si può determinare le proporzioni relative di ciascun sottotipo H1 nel pool di proteine totali H1 e quantificare il rapporto di sottotipo H1 individuale (H1 e totale) per nucleosoma. Inoltre, rispetto ad altri metodi di analisi di proteine, come Western blotting, l'analisi HPLC pronisce misure più quantitative e riproducibili di tutti i sottotipi H1. I diversi livelli e la composizione dei sottotipi H1 nella cellula modulano la struttura della cromatina più alto ordine. Il rapporto di H1 a nucleosomi è in correlazione con compattazione cromatina ed è un fattore determinante per la lunghezza di ripetizione nucleosoma nella cromatina 2. Così, i metodi che abbiamo descritto qui dovrebbe avere ampie applicazioni nel campo della ricerca cromatina.
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Questo lavoro è supportato dal NIH concessione GM085261 e Georgia Cancer Coalition Distinguished Scholar Award (per YF).
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | |
| RNasi Zap | Applied Biosystems | AM9780 | |
| Reagente Trizol | Invitrogen | 15596-018 | |
| SuperScriptIII | Invitrogen | 18080-051 | |
| Etanolo assoluto | Fisher Scientific | BP2818-4 | |
| IQ SYBR Green | Bio-Rad | 170-8880 | |
| RNeasy Mini Kit | Qiagen | 74104 | |
| I deossiribonucleasi | Sigma | AMP-D1 | |
| Microseal piastra a 96 pozzetti PCR | Bio-Rad | MSP-9605 | |
| Microseal 'B' Sigilli adesivi | Bio-Rad | MSB-1001 | |
| Saccarosio | Agros Organics | AC40594 | |
| Sodio fosfato bibasico eptaidrato (Na 2 HPO 4 · 7H 2 O) | Fisher Scientific | BP332 | |
| Cloruro di sodio (NaCl) | Americano Bioanalytical | AB01915 | |
| Eptaidrato sodio fosfato monobasico (NaH 2 PO 4 · 7H2O) | Fisher Scientific | BP-330 | |
| HEPES | Fisher Scientific | BP310 | |
| Complete Mini tablet cocktail inibitore di proteasi | Roche Applied Science | 11836153001 | |
| EDTA | Sigma | E-5134 | |
| Phenylmethanesulfonyl fluoruro (PMSF) | Americano Bioanalytical | AB01620 | |
| Nonidet-40 (NP-40) | Americano Bioanalytical | AB01425 | |
| Cloruro di potassio (KCl) | Fisher Scientific | BP366 | |
| Tris [amminometano idrossimetil] | Americano Bioanalytical | AB02000 | |
| Magnesio cloruro (MgCl2) | Fisher Scientific | BP214 | |
| Acido solforico (H2SO4) | VWR | VW3648-3 | |
| Idrossido di ammonio (NH 4 OH) | Agros Organics | AC42330 | |
| Bradford Protein Assay | Bio-Rad | 500-0001 | |
| Acetonitrile | EMD | AX0145-1 | |
| Acido trifluoroacetico (TFA) | JTBaker | 9470-01 |
Riferimenti
- Fan, Y. H1 linker histones are essential for mouse development and affect nucleosome spacing in vivo. Mol. Cell Biol. 23, 4559-4572 (2003).
- Woodcock, C. L., Skoultchi, A. I., Fan, Y. Role of linker histone in chromatin structure and function: H1 stoichiometry and nucleosome repeat length. Chromosome Res. 14, 17-25 (2006).
- Shen, X., Gorovsky, M. A. Linker histone H1 regulates specific gene expression but not global transcription in vivo. Cell. 86, 475-483 (1996).
- Alami, R. Mammalian linker-histone subtypes differentially affect gene expression in vivo. Proc. Natl. Acad. Sci. U.S.A. 100, 5920-5925 (2003).
- Happel, N., Doenecke, D. Histone H1 and its isoforms: contribution to chromatin structure and function. Gene. 431, 1-12 (2009).
- Clausell, J., Happel, N., Hale, T. K., Doenecke, D., Beato, M. Histone H1 subtypes differentially modulate chromatin condensation without preventing ATP-dependent remodeling by SWI/SNF or NURF. PLoS One. 4, 0007243-0007243 (2009).
- Khadake, J. R., Rao, M. R. DNA- chromatin-condensing properties of rat testes H1a and H1t compared to those of rat liver H1bdec; H1t is a poor condenser of chromatin. Biochemistry. 34, 15792-15801 (1995).
- Orrego, M. Differential affinity of mammalian histone H1 somatic subtypes for DNA and chromatin. BMC Biol. 5, 22-22 (2007).
- Th'ng, J. P., Sung, R., Ye, M., Hendzel, M. J. H1 family histones in the nucleus. Control of binding and localization by the C-terminal domain. J. Biol. Chem. 280, 27809-27814 (2005).
- Wang, Z. F., Sirotkin, A. M., Buchold, G. M., Skoultchi, A. I., Marzluff, W. F. The mouse histone H1 genes: gene organization and differential regulation. J. Mol. Biol. 271, 124-138 (1997).
- Brown, D. T., Sittman, D. B. Identification through overexpression and tagging of the variant type of the mouse H1e and H1c genes. J. Biol. Chem. 268, 713-718 (1993).
- Fan, Y., Sirotkin, A., Russell, R. G., Ayala, J., Skoultchi, A. I. Individual somatic H1 subtypes are dispensable for mouse development even in mice lacking the H1(0) replacement subtype. Mol. Cell. Biol. 21, 7933-7943 (2001).
- Fan, Y., Skoultchi, A. I. Genetic analysis of H1 linker histone subtypes and their functions in mice. Methods Enzymol. 377, 85-107 (2004).
- Heid, C. A., Stevens, J., Livak, K. J., Williams, P. M. Real time quantitative PCR. Genome Res. 6, 986-994 (1996).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon