Method Article
Cambiamenti di equilibrio nella struttura di monitoraggio RNA da 'perossidativi' e 'ossidativo' Footprinting radicale ossidrile
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo descrive come quantificare il Mg (II)-dipendente formazione della struttura terziaria dell'RNA con due metodi di footprinting radicali idrossili.
Abstract
Molecole di RNA svolgono un ruolo essenziale in biologia. Oltre alla trasmissione di informazioni genetiche, l'RNA può piegare in unico strutture terziarie che compie un ruolo biologico specifico come regolatore, legante o catalizzatore. Informazioni sulla formazione terziaria contatto è essenziale per capire la funzione di molecole di RNA. I radicali idrossile (OH •) sono le sonde della struttura degli acidi nucleici a causa della loro elevata reattività e piccole dimensioni. 1 Quando utilizzato come sonda footprinting, i radicali idrossili mappare la superficie solvente accessibile della dorsale fosfodiestereo di DNA 1 e 2 con RNA sottile come singolo nucleotide risoluzione. Idrossile footprinting radicale può essere utilizzato per identificare i nucleotidi all'interno di una superficie di contatto intermolecolari, ad esempio nel DNA-proteina 1 e RNA-proteina complessi. Equilibrio 3 e 4 transizioni cinetica può essere determinato da condurre footprinting radicale ossidrile in funzione di una soluzione moltoil variabile o il tempo, rispettivamente. Una caratteristica fondamentale di footprinting è che l'esposizione limitata alla sonda (per esempio, 'single-hit cinetica') i risultati nel campionamento uniforme di ogni nucleotide del polimero 5.
In questo articolo il video, si usa il P4-P6 dominio del ribozima Tetrahymena per illustrare la preparazione dei campioni di RNA e la determinazione di un (II)-mediata isoterme Mg pieghevole. Descriviamo l'uso del noto protocollo di idrossile footprinting radicale che richiede H 2 O 2 (chiamiamo questo il protocollo 'perossidativi') e un valore, ma poco noto, alternativa che utilizza naturalmente disciolto O 2 (lo chiamiamo la ' ossidativo 'protocollo). Una panoramica della riduzione dei dati, la trasformazione e procedure di analisi è presentato.
Protocollo
1. Preparazione dei reagenti Footprinting
- Preparare un tampone di reazione 10x contenente 100 cacodilato sodio mM, 1 mM EDTA, e 1 M KCl. Regolare il pH a 7,4. Filtrare il tampone usando un dispositivo di acetato 0,2 mM filtro (Nalgene). Nota: non RNA pipettare direttamente nel buffer 10x.
- Preparare la miscela di reazione di titolazione per ogni reazione, come indicato nella tabella 1. Il volume del mix di titolazione (1x buffer e Mg (II) alla concentrazione desiderata) dovrebbe essere di 90 microlitri, prima di aggiungere 10μl di RNA nel buffer 1x.
- Preparare una digestione RNasi T1 tampone contenente urea 6.63M, citrato di sodio 20 mM, 1mM EDTA, 0,25 mg / tRNA microlitri, cyanol xilene 0,025%, e il blu bromofenolo 0,025%. Questo buffer può essere conservato a 4 ° C per un massimo di 6 mesi.
- La reazione di Fenton richiede preparati al momento le soluzioni acquose di 250 mm Fe (NH 4) 2 (SO 4) 2, 250 mM EDTA, sodio e 500 mM L-ascorbato. Le concentrazioni osservato nel capitolo 10,5 sono ottimali per esperimenti di equilibrio in un tampone come cacodilato di sodio che non eliminano i radicali e diverse concentrazioni di ioni bivalenti.
- Reagenti per la perossidazione (1.5.1) e di ossidazione (1.5.2) di reazione footprinting.
- La reazione di Fenton perossidativi richiede preparati al momento le soluzioni acquose di 100 mM Fe-EDTA soluzione di sodio e 50 mM di L-ascorbato e 1,5% di H 2 O 2. La Fe-EDTA soluzione viene preparata mescolando Fe (NH 4) 2 (SO 4) 2 con EDTA di avere 1,1 volte l'eccesso di EDTA su ferro.
- Poco prima di iniziare la reazione ossidativa scissione (3.4.2), creare il mix reazione di Fenton, combinando 2 l di Fe (NH 4) 2 (SO 4) 2 (250 mm), 2,2 ml di EDTA (250 mm), 62,5 microlitri L-ascorbato di sodio (500 mm), 22,5 reazione microlitri di buffer 10x e 135,5 ml di H 2 O per raggiungere le concentrazioni finali nella miscela di reazione footprinting (3.4.2) pari a 0.10 mM, 0,11 mm e 6,61 mm, rispettivamente.
2. Preparazione di RNA per Esperimento Footprinting
- L'RNA può essere prodotto da standard di trascrizione del DNA in vitro di modelli 6 o, se più corta di 50 nucleotidi, essere acquistato (ad es Integrated Technologies DNA, www.idtdna.com). Seguire le procedure di pulizia raccomandato di laboratorio per mantenere l'integrità del campione di RNA durante gli esperimenti footprinting 7.
- Defosforilazione 8 di RNA trascritto in vitro è necessario prima di 32 etichettatura P del 5 'di RNA. 10 pmol di RNA sono end-etichettati da kinasing. 9 La soluzione deve essere torbida dopo il successo inattivazione della fosfatasi.
- Purificare l'RNA radioattivo mediante elettroforesi su gel denaturante. L'RNA viene recuperato da due successive estrazioni gel utilizzando acetato di sodio 0,3 M (pH 5,5). RNA è precipitato mescolando 0,5 ml di soluzione acquosa con 1 ml di etanolo e successiveincubazione per 1 minuto in ghiaccio secco. Spin campione a 4 ° C, 12500 rpm per 0,5 ore. Eliminare il surnatante e lavare RNA a pellet con etanolo al 70%. Rimuovere il surnatante dopo centrifugazione ulteriore e secco di pellet nel vuoto.
- Sciogliere il 32 P-etichettati campioni di RNA in 330 ul di 1x Buffer.Pipet 30 microlitri della soluzione di RNA in un tubo di reazione, precipitato con etanolo e l'RNA secco nel vuoto; tenere questo tubo per generare la scala di riferimento (punto 3.5). Denaturare la soluzione tamponata di RNA da riscaldamento a 95 ° C per 2 min. Raffreddare i campioni a temperatura ambiente per 15 minuti e dare un giro rapido per abbattere i condensati sui coperchi dei tubi di riportare in soluzione.
3. Il Footprinting Experiment
- Il pettine elettroforesi determina il numero massimo di punti dati per gel analitici. Noi usiamo un 30 pettine bene. La scala di riferimento occupa 2 pozzi e quello di controllo uncleaved. Preparare 27 provette di reazione. La finale Mg (II) Concentration deve essere equamente distanziati su una scala logaritmica si estende ordini di grandezza attorno al punto medio di titolazione.
- Separatamente, incubare l'RNA e la (II) soluzioni Mg a 50 ° C per 5 min. Miscelare 10 ml di soluzione di RNA con 90 microlitri della corrispondente quantità di Mg (II) in 1x tampone per raggiungere un volume finale di 100 ul. Incubare le soluzioni per 30 minuti a 50 ° C.
- Lasciate che le soluzioni equilibrare a 25 ° C per 1 ora, mentre si piega.
- Perossidativi (3.4.1) o ossidativi (3.4.2) idrossile reazione footprinting radicale.
- Avviare la reazione di perossidazione mettendo gocce di 2 microlitri Fe-EDTA (100 mm), 2 microlitri di sodio L-ascorbato (50 mm) e 2 microlitri H 2 O 2 (1,5%), separati l'uno dall'altro nella parte superiore interna della reazione tubo contenente la soluzione di RNA e avviare la reazione di footprinting mescolando vigoroso. Arrestare la reazione dopo 15 secondi con l'aggiunta di 300 ml di etanolo assoluto freddo. Girare i tubi 3-5 volte. Precipitato, lavaggio easciugare il pellet come al punto 2.4.
- Avviare il ossidativo footprintingreaction radicale ossidrile aggiungendo 5 ml di preparati al momento della miscela di reazione Fenton (1,6). Incubare per 30 min a 25 ° C. Aggiungere 300 ml di etanolo assoluto freddo per placare la reazione e mescolare girando i tubi 3-5 volte. Precipitato, lavare e asciugare il pellet come indicato al punto 2.4.
- Generare un RNasi T1 campione di riferimento digerire secondo le procedure standard 10 utilizzando la soluzione preparata al punto 1.3.
- Sciogliere pellet di RNA a 8 microlitri gel colorante carico II (Ambion) e confermare l'RNA viene risospeso utilizzando il contatore Geiger.
- Preparare una denaturazione, 8% gel di sequenziamento poliacrilammide secondo protocolli standard. 11 campioni di carico tra cui due riferimenti e un controllo. Separare frammenti di RNA a 60 W - 75 W per 2,5 ore. Esporre gel essiccato ad uno schermo ai fosfori durante la notte. Scansione schermo al fosforo con un sistema di imaging per autoradiografia filmless, ad esempio,. Tempesta 865 (GE Healthcare) o Typhoon (GE Healthcare). Trasferimento di file immagine di gel sul tuo computer.
4. Analisi dei dati
- La reattività del radicale idrossile ogni nucleotide è determinato da quantificare l'intensità di ogni banda sul gel analitici. Scaricare, installare e aprire SAFA, un facile da usare software open source per il montaggio singola banda e la quantificazione. 12, 13, seguire le istruzioni per l'uso manuale. In breve, caricare la sequenza di RNA come file. Txt seguito dalla foto come gel. Gel file. Regolare intensità di banda, definire i vicoli, scegliere una corsia di ancoraggio, ed eseguire un allineamento gel. Assegnazione di bande di nucleotidi si verifica in riferimento alla scala RNasi T1 digestione. Quantificare gli integrali banda e utilizzare la normalizzazione / colorplot funzione per normalizzare e assegnare i residui potenzialmente invarianti. Salvare l'output come file. Txt.
- SAFA uscite un foglio di calcolo contenente le colonne che rappresentano corsie sul gel e le righe che rappresentano i integrated densità banda delle bande isolata corrispondente al frammento di RNA. In primo luogo, individuare i siti della protezione che mostrano un notevole cambiamento nella accessibilità al solvente confrontando il profilo di protezione deriva dalla non Mg (II) campione con il profilo del punto finale Mg (II) concentrazione (50 mM nel nostro esempio). Più basso è il valore, più protetto il nucleotide è contro l'attacco dei radicali idrossili e viceversa.
- Crea isoterme pieghevole dal integrali banda normalizzata di singoli o di gruppi di nucleotidi contro Mg (II) la concentrazione. Le isoterme sono individualmente scalata a saturazione frazionale di fi = L + (U - L) •
 dove f indica la densità integrata della band (s) oggetto di analisi, L e U rappresentano i limiti inferiore e superiore alla transizione e
dove f indica la densità integrata della band (s) oggetto di analisi, L e U rappresentano i limiti inferiore e superiore alla transizione e 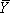 è la saturazione frazionale. 14 I dati sono idonei con una non-lineare lest piazza programma di analisi (si può usare o Origin (OriginLab) o GraphPad (GraphPad Software, Inc.).) all'equazione Hill (1),
è la saturazione frazionale. 14 I dati sono idonei con una non-lineare lest piazza programma di analisi (si può usare o Origin (OriginLab) o GraphPad (GraphPad Software, Inc.).) all'equazione Hill (1), 
dove K d è l'equilibrio costante di dissociazione, [M] corrisponde alla concentrazione della variabile che media la reazione pieghevole, Mg (II) in questo esempio, e n H è il coefficiente di Hill. Questo scala procedura la transizione alla saturazione frazionale, determina il suo punto medio e fornisce una prova fenomenologico di se una transizione è sigmoidale. Isoterme derivati dal sequenziamento gel (Figura 3A) che sono stati analizzati da SAFA (Figura 3B) e in forma, come descritto sopra sono mostrati in Figura 3C. Le isoterme di saturazione frazionale mostrato in Figura 3 sono stati generati dalla scala in un foglio i valori generati dalla SAFA migliori contro i limiti in forma superiore e inferiore (U e L), utilizzando la frazione equazione = valore / (U - L) - L / (U - L).
5. Rappresentante dei risultati:
La figura 3 mostra i risultati rappresentante P4-P6 RNA • esperimenti footprinting OH. L'immagine gel (a sinistra) indica che a) scissione di fondo è minima (corsia di destra), b) l'assegnazione singolo nucleotide è possibile grazie alle bande ben definite nella corsia T1 (RNasi T1 si unirà ad ogni G), ec) il radicale ossidrile frammentazione indotta di RNA è ben al di sopra di fondo. Il passaggio da bassa ad alta Mg (II) è affiliato con la diminuzione della densità integrata banda di singoli e gruppi di bande indicando formazione della struttura terziaria dell'RNA. Singoli o gruppi di nucleotidi contigui la cui scissione cambiamenti in concomitanza sono indicati come una 'protezione'. Il • Protezioni OH caratteristica del Mg (II) mediata ripiegamento della P4-P6 RNA strettamente corrispondere alle regioni solvente inaccessibili della molecola osservata nella sua struttura cristallina. 15
Molecole di RNA ripiegato c uno hanno estensioni molto diverse (ad esempio, quanto vicino al fondo del declino banda densità) di protezione dei contatti terziario che non sono stati correlati con evidente fenomeno strutturale e dinamico. Così, alcune molecole di RNA mostrerà ben definite transizioni strutturali, come mostrato in Fig. 3, e altri no. Intensità di banda sono quantificati e normalizzati per SAFA 12 analisi (Figura 3B). L'uscita è un complotto 'termico' visualizzando il relativo grado di protezione contro • OH. La transizione dei colori descrive il cambiamento di accessibilità su Mg (II) aggiunta. Bianco a spettacoli rosso o blu nucleotidi più accessibile o più protetti, rispettivamente. Ogni grado di ombreggiatura è affiliato con un valore numerico che può essere tracciata una curva di protezione (Figura 3C) e analizzati da un modello di associazione come ad esempio l'equazione di Hill (1). La costante di equilibrio di dissociazione affiliato Protezione 153-155 è all'incirca doppio rispetto a quello del corrispondente valore di Protezione 163-164.
CONTENUTO ">
Figura 1. Produzione di radicali ossidrile e P4-P6 pieghevole RNA. A) Fe (II) catalizza la produzione di radicali idrossili da perossido di idrogeno. Ascorbato riduce Fe (III) torna a ferro ferroso. B) In assenza di Mg (II), non P4-P6 struttura terziaria è formato, permettendo di penetrare radicali idrossili e unirà tutte le posizioni spina dorsale accessibile. Aggiunta di Mg (II) avvia pieghevole del P4-P6, consentendo solo la spina dorsale solvente accessibile ad essere spaccati dai radicali idrossili.

Figura 2. Schema di esperimento di footprinting radicali idrossile. A) Dephophorylation e 32 P 5'-end di etichettatura di RNA. B) Purificazione di 32 P-RNA marcato su un gel di poliacrilammide denaturante. C) Escissione di banda di RNA, dopo l'estrazione di RNA, e la precipitazione in etanolo. D) Prefolding epieghevole di RNA. E) L'aggiunta di preparati al momento miscela di reazione Fenton per generare i radicali idrossili. F) separazione frammento di RNA con elettroforesi su gel di poliacrilammide denaturante. G) La determinazione quantitativa di frammenti di RNA da un software SAFA.

Figura 3. Analisi dei frammenti di RNA dopo perossidativi reazione footprinting Fenton. (A) L'RNA è stato esposto ai radicali idrossile e prodotti di dissociazione sono stati risolti mediante elettroforesi su gel denaturante di poliacrilamide (PAGE). L'immagine mostra i prodotti di perossidazione scissione con crescente concentrazione di Mg (II). Corsie di riferimento e di controllo sono etichettati. Questo file immagine è l'ingresso per il programma SAFA che quantitates il volume di ciascuna banda. (B) La trama 'termica' generato da SAFA. (C) Un foglio di calcolo dei valori relativi densità integrata banda all'uscita numero nucleotide da SAFA è analizzato da un modello di associazione come la equati Hillsu (1). Aree di protezione sono stati scelti in base all'aumento della densità di banda integrale in funzione di Mg (II) la concentrazione. D K è la (II), Mg concentrazione alla quale è piegato metà del RNA nel sito monitorato. Il coefficiente di Hill, n H, è una misura della pendenza della curva che fornisce informazioni sulla cooperatività vincolante e fornisce una stima più bassa per il numero di ioni magnesio coinvolti nel ripiegamento di questo particolare sito.

Tabella 1. Volumi Rappresentante per la generazione di campioni di RNA contenenti differenti Mg (II) le concentrazioni.
Discussione
Footprinting radicale ossidrile è uno strumento prezioso per valutare l'area solvente superficie accessibile di acidi nucleici. Formazione qualitativa e quantitativa della struttura terziaria 14 può essere seguita in funzione di parametri quali il tipo e la concentrazione di ioni, pH, temperatura, proteine di legame o pieghevoli co-fattori. La combinazione vincente di un protocollo di dritto in avanti ed economico e l'accessibilità conseguente solvente e le informazioni su un pieghevole a singolo nucleotide livello rende questo metodo molto interessante. Tradizionalmente la reazione • footprinting OH viene eseguita utilizzando aggiunto H 2 O 2, un protocollo si fa riferimento a come 'perossidativi' (Figura 1A). L'alternativa ('ossidativo') protocollo 17 che utilizza la luce naturale sciolto O 2 per la produzione di piccole quantità di perossido di idrogeno tramite le reazioni 
Entrambi i metodi generate • OH attraverso l'ossidazione di Fe (II) a Fe (III) da H 2 O 2 (Figura 1A). Preferenza personale sembra determinare quale protocollo viene utilizzato dai membri del nostro laboratorio per la mappatura strutturale ed esperimenti di titolazione equilibrio. Le reazioni footprinting ossidativo e perossidazione circa 30 minuti e 1, rispettivamente. Questo svantaggio è compensato dal protocollo ossidativo è leggermente più semplice (un reagente meno è aggiunto) e la 'freschezza' sui profili ossidativo banda elettroforetica. Anche se questa osservazione suggerisce meno eterogeneità nella • prodotti di scissione OH, questa ipotesi non è stato testato. Si consiglia di utilizzare il protocollo ossidativo di analizzare un gran numero di campioni footprinting (> 15) o campioni che contengono H 2 O 2 componenti sensibili.
L'utilizzo della carica negativa EDTA complesso di Fe (II) (Figura 1A) impedisce l'associazione di Fe (II) con la spina dorsale del fosfato a carica negativa di nucleiciacido. 1 gratis Fe (II) possono legarsi al DNA e RNA e agire come un / o precipitante e possono catalizzare l'idrolisi della dorsale RNA.
Le concentrazioni presentato dei reagenti Fenton sono ideali per fendere P4-P6 RNA in singolo-hit condizioni cinetica necessaria per footprinting nel buffer indicato. Cinetica singolo di successo in modo che la molecola di RNA viene scissa in media solo una volta. 18 Se una quantità eccessiva di reagente Fenton è usato, prodotti RNA può derivare da più di un evento scissione. Questo bias di distribuzione frammento verso prodotti di scissione più breve. Il grado di scissione radicale deve essere regolata in modo che ~ 20% di fine marcata RNA è spaccati e ~ 80% di fine etichettati RNA rimane intatto. Dose-risposta esperimenti devono essere utilizzate per risalire al Fe (II) / H 2 O 2 per raggiungere concentrazioni di singolo-hit cinetica nelle condizioni soluzione di studio un investigatore di 19.
La relamappe di accessibilità ive solvente ottenuto dalla • Analisi footprinting OH può essere usato da solo per caratterizzare la struttura dell'RNA, le strutture pieghevoli e intermedie formate durante gli esperimenti pieghevole equilibrio. Questi dati possono essere combinati con i risultati di tecniche di affrontare cambiamenti di conformazione globale 7, 20 e / o studi di footprinting enzimatica e chimica 21 che sono stati ottenuti in condizioni identiche. A grana grossa modelli di strutture di RNA 22, possono essere sviluppate utilizzando radicali idrossile chimici e complementari e profili footprinting enzimatici come vincoli.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato sostenuto dalle concessioni dal National Institute of Health RO1-GM085130 e la National Science Foundation MCB0929394. Ringraziamo il Dott. Marion Schmidt per la sua ospitalità e per averci permesso di filmare nel suo laboratorio.
Materiali
| Name | Company | Catalog Number | Comments |
| Name | Company | Cat# | |
|---|---|---|---|
| Sodium Cacodylate (Caution! Toxic) | Sigma | C4945-25g | |
| EDTA (0.5 M) | Ambion | AM9260G | |
| DEPC treated water | Ambion | AM9915G | |
| Sodium Acetate (3 M) | Ambion | AM9740 | |
| MgCl2 (1 M) | Ambion | AM9530G | |
| Urea | Ambion | AM9902 | |
| Sodium Citrate | Sigma | W302600 | |
| tRNA | Sigma | R-7876 | |
| Sodium-L-ascorbate | Sigma | A7631-25g | |
| Fe(NH4)2(SO4)2 . 6 H2O | Sigma | F1543-500g | |
| RNase T1 | Fermentas | EN0541 | |
| Hydrogen Peroxide (30%) | Sigma | 349887 |
Riferimenti
- Tullius, T. D., Dombroski, B. A. Hydroxyl radical "footprinting": high-resolution information about DNA-protein contacts and application to lambda repressor and Cro protein. Proc. Natl. Acad. Sci. U.S.A. 83, 5469-5473 (1986).
- Celander, D. W., Cech, T. R. Iron(II)-ethylenediaminetetraacetic acid catalyzed cleavage of RNA and DNA oligonucleotides: similar reactivity toward single- and double-stranded forms. Biochemistry. 29, 1355-1361 (1990).
- Celander, D. W., Cech, T. R. Visualizing the higher order folding of a catalytic RNA molecule. Science. 251, 401-407 (1991).
- Sclavi, B., Sullivan, M., Chance, M. R., Brenowitz, M., Woodson, S. A. RNA folding at millisecond intervals by synchrotron hydroxyl radical footprinting. Science. 279, 1940-1943 (1998).
- Tullius, T. D., Dombroski, B. A., Churchill, M., Kam, L. Hydroxyl radical footprinting: a high-resolution method for mapping protein-DNA contacts. Methods. Enzym. 155, 537-558 (1987).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic. Acids. Res. 15, 8783-8798 (1987).
- Schlatterer, J. C., Brenowitz, M. Complementing global measures of RNA folding with local reports of backbone solvent accessibility by time resolved hydroxyl radical footprinting. Methods. 49, 142-147 (2009).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Zaug, A. J., Grosshans, C. A., Cech, T. R. Sequence-specific endoribonuclease activity of the Tetrahymena ribozyme: enhanced cleavage of certain oligonucleotide substrates that form mismatched ribozyme-substrate complexes. Biochemistry. 27, 8924-8931 (1988).
- Knapp, G. Enzymatic approaches to probing of RNA secondary and tertiary structure. Methods. Enzym. 180, 192-212 (1989).
- Slatko, B. E., Albright, L. M. Denaturing gel electrophoresis for sequencing. Curr. Protoc. Mol. Biol. Chapter 7, Unit 7.6-Unit 7.6 (2001).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. SAFA: semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. RNA. 11, 344-354 (2005).
- Simmons, K., Martin, J. S., Shcherbakova, I., Laederach, A. Rapid quantification and analysis of kinetic *OH radical footprinting data using SAFA. Methods. Enzym. 468, 47-66 (2009).
- Uchida, T., He, Q., Ralston, C. Y., Brenowitz, M., Chance, M. R. Linkage of monovalent and divalent ion binding in the folding of the P4-P6 domain of the Tetrahymena ribozyme. Biochemistry. 41, 5799-5806 (2002).
- Cate, J. H., Gooding, A. R., Podell, E., Zhou, K., Golden, B. L., Kundrot, C. E., Cech, T. R., Doudna, J. A. Crystal structure of a group I ribozyme domain: principles of RNA packing. Science. 273, 1678-1685 (1996).
- Petri, V., Brenowitz, M. Quantitative nucleic acids footprinting: thermodynamic and kinetic approaches. Curr. Opin. Biotechnol. 8, 36-44 (1997).
- Takamoto, K., Das, R., He, Q., Doniach, S., Brenowitz, M., Herschlag, D., Chance, M. R. Principles of RNA compaction: insights from the equilibrium folding pathway of the P4-P6 RNA domain in monovalent cations. J. Mol. Biol. 343, 1195-1206 (2004).
- Brenowitz, M., Senear, D. F., Shea, M. A., Ackers, G. K. Quantitative DNase footprint titration: a method for studying protein-DNA interactions. Methods. Enzym. 130, 132-181 (1986).
- Shcherbakova, I., Mitra, S. Hydroxyl-radical footprinting to probe equilibrium changes in RNA tertiary structure. Methods. Enzym. 468, 31-46 (2009).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for the study of protein association and assembly. Curr. Opinion. Chem. Biol. 10, 430-436 (2006).
- McGinnis, J. L., Duncan, C. D., Weeks, K. M. High-throughput SHAPE and hydroxyl radical analysis of RNA structure and ribonucleoprotein assembly. Methods. Enzym. 468, 67-89 (2009).
- Jonikas, M. A., Radmer, R. J., Laederach, A., Das, R., Pearlman, S., Herschlag, D., Altman, R. B. Coarse-grained modeling of large RNA molecules with knowledge-based potentials and structural filters. RNA. 15, 189-199 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon