Method Article
Analisi biofilm orale di espansori palatali da Fluorescence In Situ-ibridazione e microscopia confocale a scansione laser
In questo articolo
Riepilogo
Vi presentiamo un protocollo per l'analisi strutturale e compositiva delle risorse naturali biofilm orale da apparecchi ortodontici con In situ Ibridazione (FISH) e la microscopia confocale a scansione laser (CLSM). Campioni di biofilm orale sono stati raccolti da espansori palatali, raschiando acrilica sfalda la loro superficie e riferimento per l'elaborazione molecolare.
Abstract
Microscopia confocale a scansione laser (CLSM) di naturale biofilm eterogeneo è oggi facilitato da una gamma completa di tecniche di colorazione, uno dei quali è ibridazione in situ fluorescente (FISH). 1,2 Abbiamo eseguito uno studio pilota in cui campioni di biofilm orali raccolte da fisso apparecchi ortodontici (palatale expander) sono state macchiate dalla FISH, con l'obiettivo di valutare l'tridimensionale organizzazione del biofilm naturale e accumulo di placca. 3,4 PESCE crea l'opportunità di macchia cellule nel loro ambiente nativo biofilm con l'uso di 16S fluorescente rRNA-targeting sonde. 4-7,19 Rispetto alle tecniche alternative come l'etichettatura immunofluorescenza, questo è un poco costoso, tecnica di etichettatura preciso e semplice da indagare diversi gruppi di batteri nel biofilm consorzi misti. 18,20 sonde sono state usate Generale che si legano al Eubatteri ( EUB338 + + EUB338II EUB338III, di seguito EUBmix), 8-10 Firmicutes (LGC354 AC;. LGCmix di seguito), 9,10 e Bacteroidetes (Bac303) 11 Inoltre, le sonde specifiche vincolanti di Streptococcus mutans (MUT590) 12,13 e Porphyromonas gingivalis (conti correnti postali) 13,14 sono stati utilizzati . La durezza estrema dei materiali superficie interessata (in acciaio inox e resina acrilica) ci costrinse a trovare nuovi modi di preparare il biofilm. Poiché questi materiali di superficie non potrebbe essere facilmente tagliato con una cryotome, vari metodi di campionamento sono stati esplorati per ottenere intatto biofilm orale. La più realizzabile di questi approcci è presentata in questa comunicazione. Piccoli fiocchi del biofilm che trasporta resina acrilica sono stati raschiata con un bisturi sterile, facendo attenzione a non danneggiare la struttura del biofilm. Forcipe sono stati utilizzati per raccogliere biofilm dalle superfici in acciaio. Una volta raccolti, i campioni sono stati fissati e posti direttamente sul polysine vetrini rivestiti. FISH è stata eseguita direttamente su queste diapositive con le sonde mentioned sopra. PESCE protocolli diversi sono stati combinati e modificati per creare un nuovo protocollo che è stato facile da maneggiare. 5,10,14,15 Successivamente i campioni sono stati analizzati al microscopio confocale a scansione laser. Noto configurazioni 3,4,16,17 potrebbero essere visualizzati, tra cui funghi in stile formazioni e gruppi di batteri coccoidi pervaso da canali. Inoltre, la composizione batterica di queste strutture tipiche biofilm sono stati analizzati e immagini 2D e 3D creato.
Protocollo
1. Prelievo dei campioni
- Rimuovere fisso palatale espansori dopo 4 mesi di servizio intraorale. Utilizzare Sistema Campo Asciutto NOLA per isolare gli apparecchi prima di togliere loro (figure 1 e 2).
- Utilizzare una pinza sterilizzata, guanti e vassoi per rimuovere espansori senza l'aggiunta di contaminazione (figure 3 e 4).
- Oggetti Conservare in Sarstedt 120 fiale ml a -20 ° C. Prendere per laboratorio su ghiaccio e processo entro 24 ore.
2. Biofilm fissazione
- Raschiare fiocchi biofilm con un bisturi sterile, o raccogliere i pezzi con una pinza sterile.
- Aggiungi ghiacciata soluzione al 4% PFA fino campione è ben coperto.
- Incubare la miscela a +4 ° C (non congelare) per 3 a 12 ore. Tempi di fissazione più o temperature superiori fissazione può rendere le buste di cellule meno permeabile a sonde oligonucleotidiche gram-negativi.
- Rimuovere la soluzione PFA e lavare con ghiacciata PBS 1X. Ripetere questa operazione 2-3 volte al rTogliete residuo PFA.
- Risospendere il campione in 1 vol. ghiacciato 1X PBS e aggiungere 1 vol. ghiacciato del 96% (v / v) di etanolo.
- Conservare il campione a -20 ° C. Campioni fissati in base a questo protocollo può essere conservato per diversi mesi o anni.
3. Disidratazione dei campioni fisso
- Applicare 5-30 microlitri di PFA materiale fissato campione di un vetrino da microscopio.
- Secco a 46 ° C per circa 15 minuti oa temperatura ambiente per più tempo. Scongelare il lisozima e la formammide.
- Aggiungere 250 ml di lisozima (1mg/ml) a temperatura ambiente per 10 minuti, facilitando la penetrazione delle sonde nelle cellule dalla distruzione parziale delle pareti cellulari.
- Dip scivolare nel 50%, 80% e 96% (v / v) di etanolo per 3 minuti ciascuno. L'effetto disidratante della serie concentrazione di etanolo si disintegrerà delle membrane cellulari.
- Asciugare i vetrini a 46 ° C per 10 min.
4. Ibridazione in situ
- Preparare 1 ml diibridazione tampone fresco (vedi Tabella 1 per le concentrazioni). Concentrazioni formammide utilizzato in questo studio: 10% (EUBmix), 20% (Bac303, dei conti correnti postali, MUT590) o 45% (LGCmix).
- Scongelare le soluzioni oligonucleotide sonda. Sonde scongelati devono essere tenuti sul ghiaccio e al riparo dalla luce.
- Aggiungere 2 ml di ciascuna sonda a 200 l di tampone di ibridazione, mescolare bene e applicare la miscela al campione disidratato su un vetrino da microscopio.
- Mettere un pezzo di carta velina in un piatto piazza Petri e versare il buffer di ibridazione rimanente sulla carta velina.
- Collocare immediatamente la diapositiva orizzontalmente nel piatto e chiudere il piatto. Incubare in un forno a 46 ° C per 1-5 ore (90 min sarà sufficiente nella maggior parte dei casi). Le funzioni piatto come una camera di umidità impedendo l'evaporazione della soluzione di ibridazione dalla diapositiva. In particolare, l'evaporazione di formammide può causare non sonda specifica vincolante per non colpire le cellule.
- Preparare 50 ml di tampone di lavaggio (vedi Tabella 2 per concentrations). Preparare il tampone di lavaggio in un tubo da 50 ml e preriscaldare a 48 ° C a bagnomaria. La fase di lavaggio viene eseguita a 48 ° C.
- Rimuovere il piatto con la diapositiva dal forno di ibridazione. Immediatamente lavare il buffer di ibridazione con un piccolo volume di pre-riscaldato tampone di lavaggio, e trasferire la diapositiva nel buffer di lavaggio rimanenti.
- Posizionare il tubo contenente il tampone di lavaggio e lo scivolo di nuovo nel bagno d'acqua e incubare a 48 ° C per 10-15 minuti.
- Rimuove la pagina dal tubo e tuffo nella gelida DDH 2 O per 2-3 secondi per eliminare residui di tampone di lavaggio.
- Asciugare la diapositiva il più rapidamente possibile (l'uso di aria compressa è raccomandato). Essiccazione rapida riduce sonda dissociazione.
- Diapositive secchi possono essere conservati al buio a -20 ° C per diverse settimane senza significative perdite di sonda-conferito segnale di fluorescenza.
5. Microscopia
- Dopo la FISH e lavaggio, apply due gocce di antifadent vicino alle estremità destra e sinistra di una diapositiva (fette congelato dovrebbe essere portata a temperatura ambiente prima di questo passo).
- Mettere un coperchio microscopio scivolare sulla parte superiore e attendere che il antifadent si è diffuso oltre l'intera diapositiva. Si noti che una quantità eccessiva di antifadent può sfocare l'immagine microscopio.
- Osservare i campioni al microscopio confocale a scansione laser dotati di filtri adatti o laser. Abbiamo usato una Leica TCS unità (HCX PL APO/63x; NA 1.2). I dati possono essere analizzati con software come IMARIS o AMIRA.
- Diapositive integrati nel antifadent possono essere conservati a +4 ° C (non congelare) per un massimo di 7 giorni prima la sonda-conferito fluorescenza comincia a declinare. In alternativa, il antifadent può essere rimosso con DDH 2 O, e le diapositive secca può essere conservata a -20 ° C per un periodo prolungato di tempo.
6. Rappresentante dei risultati:
Raschiando biofilm off apparecchi ortodontici fissi (Figura 5) produce fiocchi adatto (Figura 6) che può essere ibridato direttamente su vetrini rivestiti per microscopia. In questo modo, i diversi gruppi di batteri orobiome possono essere identificati nel loro naturale ambiente tridimensionale da rRNA tagging batterica etichettati in modo diverso sonde specifiche (figure 7 e 8). Nella Figura 7, biofilm era macchiata di EUBmix (verde, tutti i batteri) e LGCmix (giallo, Firmicutes). Firmicutes visualizzati in verde, in quanto sono state colorate di giallo e blu. Nella Figura 8, biofilm era macchiata di EUBmix (rosso, tutti i batteri), Bac303 (blu, Bacteroidetes) e dei conti correnti postali (giallo, Porphyromonas gingivalis). Porphyromonas gingivalis è mostrata in bianco giallognolo, in quanto tutte e tre le sonde si legano al suo DNA e la sovrapposizione dei risultati di colori in un bianco segnale. Differenze morfologiche tra i gruppi di batteri possono essere identificati (Figure 9 e 10). Grandi gruppi di batteri coccoidi sono mostrati in figura 9, dove è stata eseguita con EUBmix colorazione (verde, tutti i bacteria). Diverse forme di batteri orali sono stati visualizzati in Figura 10, dove i batteri filamentosi coccoidi e possono essere distinte dalla colorazione con EUBmix (rosso). Inoltre, un tipico fungo-come la struttura del biofilm possono essere trattati tramite modellazione 3D dei dati CLSM (figure 11 e 12) Clicca qui per guardare un film della modellazione 3D.

Figura 1. Espansore palatale fisso in situ.
Figura 2. Sistema campo Nola a secco.

Figura 3. Pinze sterili, guanti e vassoio.

Figura 4. Rimosso espansione.

Figura 5. Biofilm raschiando con un bisturi sterile.

Fiocchi di resina Figura 6. Ibridato direttamente su lastre di vetro rivestito.

Figura 7 immagine CLSM:. Differenziazione di uno specifico gruppo di batteri. Sovrapposizione di dati 2D CLSM pila 3D. Biofilm colorati con EUBmix (verde, tutti i batteri) e LGCmix (giallo, Firmicutes).
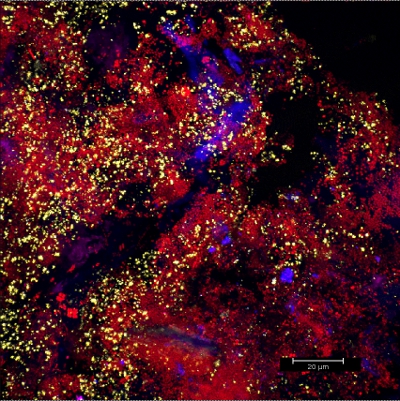
Figura 8 immagine CLSM:. Differenziazione di uno specifico gruppo di batteri. Sovrapposizione di dati 2D CLSM pila 3D. Macchiato biofilm con EUBmix (rosso, tutti i batteri), Bac303 (blu, Bacteroidetes) e dei conti correnti postali (giallo, Porphyromonas gingivalis).
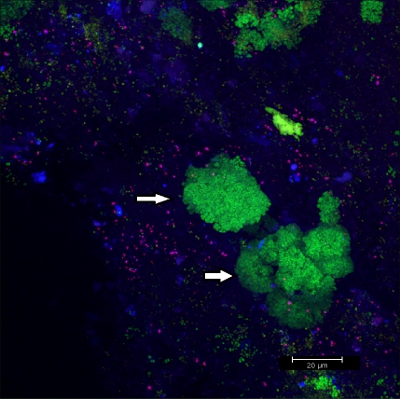
Figura 9 immagine CLSM:. Differenziazione delle morfologie specifiche. Sovrapposizione di dati 2D CLSM pila 3D. Biofilm colorati con EUBmix (verde, tutti i batteri). Grandi cluster di batteri coccoidi (frecce).
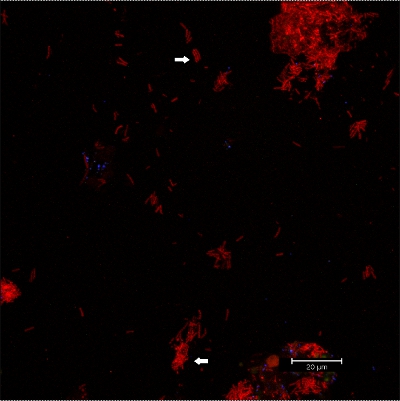
. Figura 10 immagini CLSM: differenziazione delle morfologie specifiche. Sovrapposizione di dati 2D CLSM pila 3D. Biofilm colorati con EUBmix (rosso, tutti i batteri). Coccoidi (freccia in basso) e batteri filamentosi (freccia in alto) si possono distinguere.
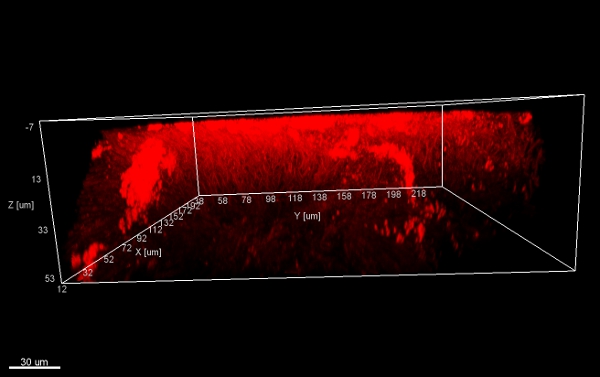
Figura 11. Struttura di funghi, viste 3D dal basso. Pile di fiocchi biofilm (raschiato la superficie di un apparecchio ortodontico), colorate con EUBmix e proceduressed con IMARIS (immagine CLSM).

Figura 12. Struttura di funghi, vista laterale 3D. Pile di fiocchi biofilm (raschiato la superficie di un apparecchio ortodontico) trattati con IMARIS (immagine CLSM).
| Ibridazione di buffer (200 mL) | |||
| Concentrazione Formammide | 10% | 20% | 45% |
| 5 M NaCl | 36 | 36 | 36 |
| 1 M Tris-HCl | 4 | 4 | 4 |
| 2% SDS | 1 | 1 | 1 |
| FA | 20 | 40 | 90 |
| DDH 2 O | 138 | 118 | 68 |
| Ogni pesce sonda | 2 | 2 | 2 |
Costituenti Tabella 1. Tampone di ibridazione (concentrazioni in microlitri).
| Tampone di lavaggio (50 ml) | |||
| Concentrazione Formammide | 10% | 20% | 45% |
| 5 M NaCl | 4500 | 2150 | 300 |
| 1 M Tris-HCl | 1000 | 1000 | 1000 |
| 0,5 M EDTA | 0 | 500 | 500 |
| DDH 2 O | 44 500 | 46 350 | 48 200 |
Costituenti Tabella 2. Tampone di lavaggio (concentrazioni in microlitri).
Movie 1. Clicca qui per guardare il film.
Discussione
Il presente protocollo descritto è un approccio altamente funzionale per biofilm colorazione raccolti dai materiali duri. Campionamento e ibridazione sono i passaggi più critici. Durante il campionamento, occorre fare attenzione che le fette di spessore adeguato sono raccolti per assicurare che la struttura del biofilm rimane intatto. Durante l'ibridazione, è essenziale per evitare eccessive fluttuazioni di temperatura, evitando legame non specifico, o la perdita di legame, delle sonde fluorescente. Questo protocollo FISH è un metodo elegante per macchiare normale biofilm eterogeneo direttamente su vetrini senza interrompere la sua composizione. Le slide possono essere immediatamente utilizzati per la microscopia, senza la necessità di spostare il campione ancora una volta. In questo modo, un miglioramento rispetto colorazione in tubi si ottiene una minore forza di taglio applicata al biofilm. Fette> 50 micron di spessore possono essere preparati e studiati in questo modo.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato sostenuto dal Fonds Igiene dell 'Università Medica di Graz. Tutti i soggetti hanno dato il loro consenso informato. Approvazione istituzionale del protocollo di studio è stato ottenuto presso l'Università Medica di Graz.
Riferimenti
- Sunde, P. T. Fluorescence in situ hybridization (FISH) for direct visualization of bacteria in periapical lesions of asymptomatic root-filled teeth. Microbiology. 149, 1095-1102 (2003).
- Sussman, M., Loya, Y., Fine, M., Rosenberg, E. The marine fireworm Hermodice carunculata is a winter reservoir and spring-summer vector for the coral-bleaching pathogen Vibrio shiloi. Environ. Microbiol. 5, 250-255 (2003).
- Kolenbrander, P. E. Communication among oral bacteria. Microbiol. Mol. Biol. Rev. 66, 486-505 (2002).
- Zijnge, V. Oral biofilm architecture on natural teeth. PLoS One. 5, e9321-e9321 (2010).
- Thurnheer, T., Gmur, R., Giertsen, E., Guggenheim, B. Automated fluorescent in situ hybridization for the specific detection and quantification of oral streptococci in dental plaque. J. Microbiol. Methods. 44, 39-47 (2001).
- Hojo, K., Nagaoka, S., Ohshima, T., Maeda, N. Bacterial interactions in dental biofilm development. J. Dent. Res. 88, 982-990 (2009).
- Jung, D. J. Visualization of initial bacterial colonization on dentine and enamel in situ. J. Microbiol. Methods. 81, 166-174 (2010).
- Al-Ahmad, A. Bacterial colonization of enamel in situ investigated using fluorescence in situ hybridization. J. Med. Microbiol. 58, 1359-1366 (2009).
- Bryers, J. D. Medical biofilms. Biotechnol. Bioeng. 100, 1-18 (2008).
- Cardinale, M., Vieira de Castro, J., Muller, H., Berg, G., Grube, M. In situ analysis of the bacterial community associated with the reindeer lichen Cladonia arbuscula reveals predominance of Alphaproteobacteria. FEMS Microbiol. Ecol. 66, 63-71 (2008).
- Aas, J. A., Paster, B. J., Stokes, L. N., Olsen, I., Dewhirst, F. E. Defining the normal bacterial flora of the oral cavity. J. Clin. Microbiol. 43, 5721-5732 (2005).
- Chin, M. Y., Busscher, H. J., Evans, R., Noar, J., Pratten, J. Early biofilm formation and the effects of antimicrobial agents on orthodontic bonding materials in a parallel plate flow chamber. Eur. J. Orthod. 28, 1-7 (2006).
- Loy, A., Maixner, F., Wagner, M., Horn, M. probeBase--an online resource for rRNA-targeted oligonucleotide probes: new features 2007. Nucleic. Acids. Res. 35, (2007).
- Diaz, P. I. Molecular characterization of subject-specific oral microflora during initial colonization of enamel. Appl. Environ. Microbiol. 72, 2837-2848 (2006).
- Dige, I., Nilsson, H., Kilian, M., Nyvad, B. In situ identification of streptococci and other bacteria in initial dental biofilm by confocal laser scanning microscopy and fluorescence in situ hybridization. Eur. J. Oral Sci. 115, 459-467 (2007).
- Kolenbrander, P. E. Bacterial interactions and successions during plaque development. Periodontology. 42, 47-79 (2000).
- Kolenbrander, P. E., Palmer, R. J., Periasamy, S., Jakubovics, N. S. Oral multispecies biofilm development and the key role of cell-cell distance. Nat. Rev. Microbiol. 8, 471-480 (2010).
- Chalmers, N. I. Use of quantum dot luminescent probes to achieve single-cell resolution of human oral bacteria in biofilms. Appl. Environ. Microbiol. 73, 630-636 (2007).
- Wecke, J. A novel technique for monitoring the development of bacterial biofilms in human periodontal pockets. FEMS Microbiol. Lett. 191, 95-101 (2000).
- Moter, A., Gobel, U. B. Fluorescence in situ hybridization (FISH) for direct visualization of microorganisms. J. Microbiol. Methods. 41, 85-112 (2000).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon