Method Article
Quantificazione di dsDNA con il F-7000 Hitachi Fluorescenza spettrofotometro e PicoGreen Dye
In questo articolo
Riepilogo
Dimostrazione di quantificazione del dsDNA utilizzando sonde molecolari PicoGreen colorante e Hitachi F-7000 Spettrofotometro a fluorescenza dotato di un accessorio lettore di micropiastre.
Abstract
Quantificazione del DNA, specialmente in piccole concentrazioni, è un compito importante, con una vasta gamma di applicazioni biologiche tra cui standard di test di biologia molecolare come la sintesi e la purificazione del DNA, applicazioni diagnostiche come la quantificazione dei prodotti di amplificazione del DNA, e la rilevazione di molecole di DNA in droga preparati. Durante questo video viene mostrata la capacità del spettrofotometro Hitachi F-7000 fluorescenza dotato di un accessorio Micro Reader piastra per eseguire quantificazione dsDNA utilizzando sonde molecolari Quant-it PicoGreen kit di reagenti coloranti.
Lo spettrofotometro a fluorescenza F-7000 offre alta sensibilità e le misure ad alta velocità. Si tratta di un sistema altamente flessibile in grado di misurare la fluorescenza, luminescenza e fosforescenza. Modalità di misurazione sono disponibili diversi, tra cui lunghezza d'onda di scansione, scansione del tempo, fotometria e 3-D di misura scansione. Lo spettrofotometro ha una sensibilità nel range di 50 picomoles di fluorescina quando si utilizza un volume di 300 microlitri del campione in micropiastra, ed è in grado di misurare la velocità di scansione di 60.000 nm / minuto. Dispone inoltre di un ampio range dinamico fino a 5 ordini di grandezza, che consente l'utilizzo di curve di calibrazione in un ampio range di concentrazioni. Il sistema ottico utilizza tutte le ottiche riflettenti per la massima energia e sensibilità. La gamma di lunghezze d'onda di serie è da 200 a 750 nm, e può essere estesa a 900 nm quando si usa uno dei fotomoltiplicatori opzionale vicino infrarosso. Il sistema permette il controllo della temperatura opzionale per il lettore di piastre 5-60 gradi Celsius usando un esterno opzionale circolatore controllato la temperatura del liquido. Il lettore di micropiastre consente l'utilizzo di 96 micropiastre bene, e la velocità di misura per 96 pozzetti è meno di 60 secondi quando si utilizza la modalità cinetica.
Controlli software per l'F-7000 e micropiastre Reader sono anche molto flessibili. I campioni possono essere impostati sia in formato colonna o riga, e qualsiasi combinazione di pozzi possono essere scelti per la misura del campione. Questo consente un utilizzo ottimale della micropiastra. Inoltre, il software consente l'importazione micro configurazioni di esempio piatto creato in Excel e salvato in valori separati da virgola, o in formato "csv". Micropiastre configurazioni di misura possono essere salvate e richiamate dal software per comodità e maggiore produttività. Risultati dei dati può essere emesso un rapporto standard, in Excel, o di un programma facoltativo Report Generator.
Protocollo
1. Strumento Setup
- Accendere il spettrofluorimetro e lasciare scaldare per 1 ora.
- Utilizzo di Excel crea un file con gli standard ed i dati del campione, come mostrato nella Figura 1 e salvarlo in valori separati da virgola formato "csv".
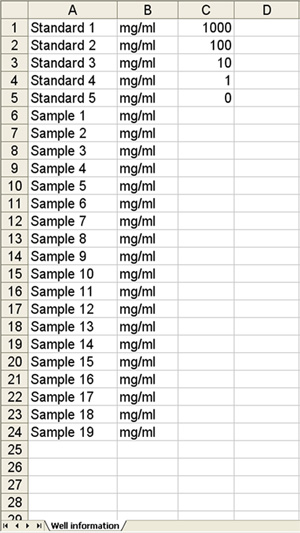
Figura 1. Standard e campioni di dati. - Istituito fluorimetro come segue:
- Clicca sul
 pulsante, si aprirà la finestra metodo di analisi, mostrata in Fig. 2.
pulsante, si aprirà la finestra metodo di analisi, mostrata in Fig. 2. 
Figura 2. Metodo di analisi finestra, nella scheda Generale.
Effettuare le seguenti selezioni:- Nel campo "Misura", selezionare Fotometria dal menu a discesa.
- Nel campo "Operatore", inserire il nome dell'operatore appropriato.
- Non è necessario inserire le informazioni nel campo "strumento". Questo viene selezionato automaticamente dal software.
- Il campo "Sampling" non è attivo quando si utilizza il lettore per micropiastre.
- Inserire eventuali commenti che è possibile includere nel rapporto nel campo "Commenti".
- Nel campo "accessori", inserire le informazioni relative agli accessori utilizzati per questo test.
- Nota i pulsanti sul lato destro della finestra.
- Il pulsante "Carica" permette di caricare Metodi di analisi precedentemente salvato.
- Il pulsante "Salva" consente di aggiornare una serie di parametri di prova o di metodo di analisi precedentemente salvato e caricato, utilizzando lo stesso nome.
- "Salva con nome" pulsante permette di salvare una nuova serie di parametri di prova o di metodo di analisi sotto il nome desiderato.

Figura 3. Metodo di analisi finestra, scheda quantificazione.- Nel "tipo di quantificazione", selezionare lunghezza d'onda. Questo indica che la lettura della fluorescenza sarà effettuata utilizzando la lunghezza d'onda selezionata.
- Nel "tipo di calibrazione" di campo, selezionare 1 ° ordine.
- Nel campo "Numero di lunghezze d'onda", selezionare 1.
- Nella "unità di concentrazione", immettere ng / ml.
- Lasciare le caselle non selezionate per "Calibrazione manuale" e "curva di Forza per lo zero".
- Nella "Digit dopo punto decimale", selezionare 2.
- Nel "basso limite di concentrazione", immettere 0.
- Nel "campo superiore limite di concentrazione, immettere 10000.
Ora cliccate sulla scheda "Strumenti" e effettuare le seguenti selezioni.
Figura 4. Metodo di analisi finestra, nella scheda strumento- Nella modalità "Dati:", selezionare fluorescenza.
- La "velocità Tagliere" campo non è attivo in questo momento, viene utilizzato solo per le misure fosforescenza.
- Nella modalità "Wavelength", selezionare Entrambi fisso WL.
- Nel campo "EX / WL1" entrare 480nm.
- Nella "EM / WL1" inserire 520nm.
- Tutti gli altri campi sotto EX e EM non sono attive in questo momento.
- Nella "EX fessura" campo 5.0nm selezionare dal menu a discesa.
- Nella "EM fessura" campo 5.0nm selezionare dal menu a discesa.
- Lasciate selezionata la casella di fronte al "Tensione PMT 1-1000V campo".
- Nella "PMT tensione", selezionare 700V dal menu a discesa.
- Nella "calc Auto statistica. Numero" campo selezionare 2.
- Nel campo "Replica", selezionare 1.
- Nel "tempo di integrazione" campo selezionare 0.1s.
- Nel campo "Ritardo" immettere o selezionare 0.
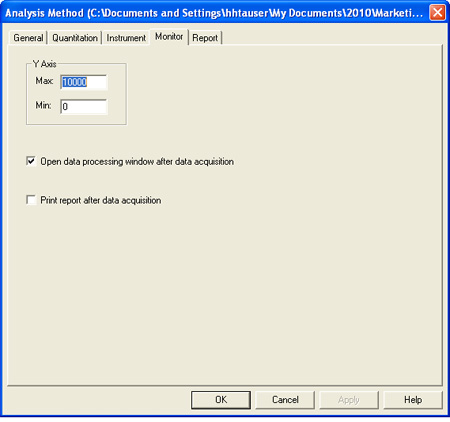
Figura 5. Metodo di analisi finestra, scheda Monitor.- Nella "asse Y", "Max:", selezionare 10000
- Nella "asse Y", "Min:", selezionare 0.
- Selezionare la casella a sinistra del "Aprire il trattamento dei dati dopo l'acquisizione dei dati".
- Lasciate selezionata la casella a sinistra del "rapporto di stampa dopo l'acquisizione dei dati", a meno che non si desidera un rapporto da stampare automaticamente dopo le letture sono state completate.
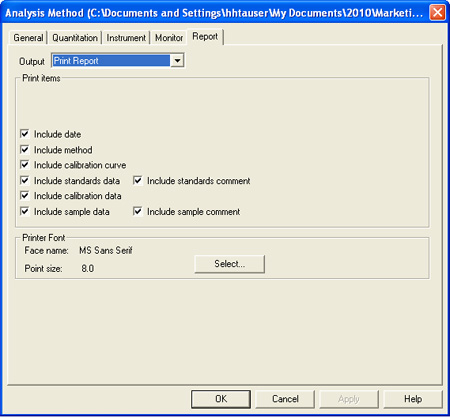
Figura 6. Metodo di analisi finestra, scheda Report.- Nel campo "Uscita", selezionare Stampa rapporto dal menu a discesa. Si può anche selezionare "Utilizza Microsoft Excel" se si desidera che i dati esportati in Excel, o "Stampa Foglio Usa Generator" se si utilizza il componente aggiuntivo opzionale Generator programma rapporto che permetterà generare report personalizzati fatto.
- Selezionare le caselle per gli elementi che si desidera includere nel rapporto.
- Per salvare tutti i parametri selezionati nelle diverse schede del "metodo di analisi" finestra, clicca di nuovo sulla scheda "Generale" e salvare con un nome di file e in una posizione in cui si riusciva a trovarli per un uso futuro, e fare clic sul " OK "per chiudere questa finestra.
- Questo completa l'impostazione dei parametri dello strumento di prova.
- Ora ci sarà istituito il lettore per micropiastre.
- Clicca sul
 pulsante, che porterà la finestra Setup MPR in cima.
pulsante, che porterà la finestra Setup MPR in cima.

Figura 7. MPR finestra di installazione, scheda Plate.- Clicca sulla posizione A1 e trascinare il mouse per E1, quindi fare clic sul
 pulsante. In questo modo selezionare le posizioni da utilizzare per gli standard. Quelle posizioni verranno evidenziati in colore verde.
pulsante. In questo modo selezionare le posizioni da utilizzare per gli standard. Quelle posizioni verranno evidenziati in colore verde. - Ora abbiamo bisogno di selezionare F1 posizioni H3 per i campioni. Fare clic e trascinare il mouse a partire F1 posizione, alla posizione H3 e quindi fare clic sul
 pulsante, questo metterà in evidenza le posizioni di colore giallo.
pulsante, questo metterà in evidenza le posizioni di colore giallo. - Quindi ripetere la stessa operazione per le posizioni A2 a E3, per selezionare quelle posizioni per le misure del campione.
- Ora, caricare il file di Comma Separated variabile (csv) preparati in anticipo, le prescrizioni e le informazioni del campione.

Figura 8. MPR finestra di installazione, scheda Informazioni Bene.- Quindi fare clic sul
 pulsante. Una finestra di Windows Explorer che si aprirà come mostrato nella Figura 9, che permette la localizzazione del "Bene information.csv" file e il caricamento.
pulsante. Una finestra di Windows Explorer che si aprirà come mostrato nella Figura 9, che permette la localizzazione del "Bene information.csv" file e il caricamento. 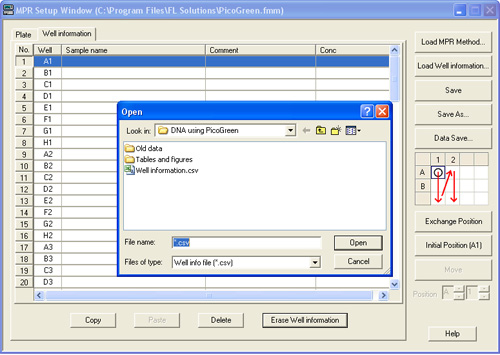
Figura 9. Caricamento ben campione.
Dopo aver caricato il "Bene informazione" del file, le "informazioni Well" sarà come mostrato nella Figura 10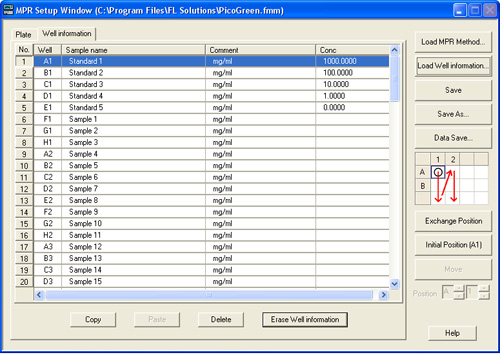
Figura 10. Loaded informazioni ben campione.
- Clicca sul
2. PicoGreen saggio Preparazione del campione
- Preparare 1X tampone TE (10 mM Tris-HCl, 1 mM EDTA) con l'aggiunta di 1 mL di 20 X TE Buffera 19 ml di dI H 2 O.
- Preparare la soluzione reagente PicoGreen aggiungendo 50 microlitri 200 X reagente PicoGreen a 9,95 ml di buffer di 1 X TE.
- Preparare gli standard (Lambda dsDNA) facendo diluizioni seriali di magazzino dsDNA (100 mg / mL). In primo luogo preparare una soluzione di 50 mg / mL dsDNA aggiungendo 25 l di magazzino dsDNA (100 mg / ml) e 25 microlitri 1 x tampone TE. Preparare quindi le soluzioni standard con l'aggiunta di volumi indicati di dsDNA e buffer come elencati nella tabella seguente:
Concentrazione di dsDNA Volume di dsDNA Volume di tampone 1 mg / mL 20 l di 50 mg / mL dsDNA 980 microlitri 1 X TE Buffer 0,1 mg / ml 100 l di 1 mg / mL dsDNA 900 microlitri 1 X TE Buffer 0,01 mg / ml 100 l di 0,1 mcg / mL dsDNA 900 microlitri 1 X TE Buffer 0,001 mg / ml 100 l di 0,01 mg / mL dsDNA 900 microlitri 1 X TE Buffer
3. Misura degli standard e fluorescenza del campione
- Pipettare 150 ml di ogni standard nei pozzetti A1-E1 di una piastra 96 pozzetti, per Figura 7, e secondo la seguente tabella:
Bene Standard A1 1 mg / mL B1 0,1 mg / ml C1 0,01 mg / ml D1 0,001 mg / ml E1 1 X TE Buffer - Pipettare 150 campioni microlitri sconosciuto in pozzi F1 per H3, mostrato in figura 7
- Versare la soluzione PicoGreen preparata al punto 1,2 in un trogolo progettato per multicanale uso pipetta. Utilizzare una pipetta multicanale per fornire 150 ul di soluzione PicoGreen ai pozzetti A1-H3. Mix di soluzioni e standard delicatamente con pipetta.
- Piastra in luogo buio e aspettare 2-5 minuti per la reazione di svilupparsi.
4. Rappresentante Risultati
Una buona curva di calibrazione viene valutata utilizzando i diversi parametri forniti dal F-7000 del software su misura degli standard e il calcolo della curva di calibrazione dal software.
Il coefficiente di determinazione indica la bontà di adattamento degli standard misurata e la curva di calibrazione calcolati. Quanto più questo valore è a "1", migliore è la misura del valore misurato e la curva di calibrazione.
Se il valore è ben lungi dall'essere "1", le norme devono essere ri-misurato, preparato ancora una volta, o l'idoneità della curva di calibrazione cambiato.
Figura 11 mostra un esempio di una curva di calibrazione con un coefficiente di determinazione calcolato = 0,99993, ottenuto per la quantificazione del DNA a doppia elica con PicoGreen colorante.
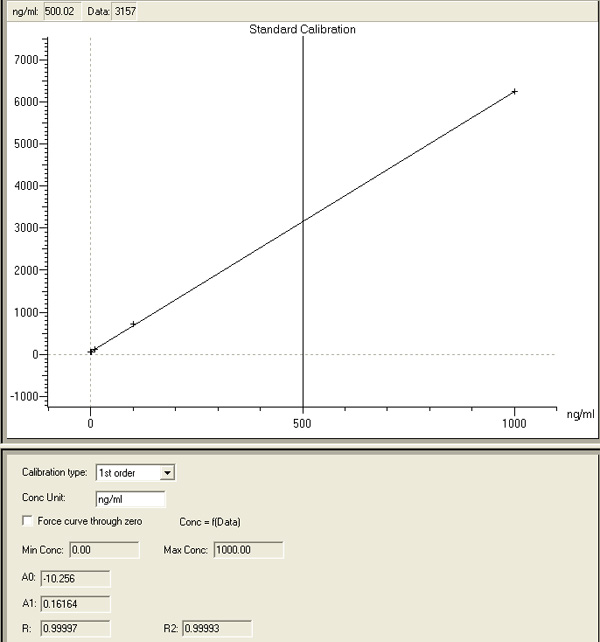
Figura 11. Esempio di curva di calibrazione dsDNA.
Discussione
Pipettaggio attenzione durante la preparazione dei campioni, così come usando uno spettrofotometro stabile e sensibile fluorescenza è essenziale per ottenere buoni risultati e riproducibili.
Nel caso dello spettrofotometro a fluorescenza utilizzata per questo test, si consiglia di utilizzare 300 microlitri volume del campione finale nel lettore per micropiastre. Abbassare il volume diminuisce la sensibilità, e un maggiore volume può causare la contaminazione incrociata tra pozzi.
Divulgazioni
Materiali
| Name | Company | Catalog Number | Comments | |
| Distilled H2O | Reagent | |||
| Quant-iT Picogreen dsDNA Assay Kit from Invitrogen | Reagent | P7589 | ||
| Nalge Nunc Fluorescence Microplates p/n 237108 | Supply | 237108 | ||
| 12 Channel Eppendorf Micropipette | Supply | 3516677 | ||
| 1000 μL Eppendorf Pipette | Supply | |||
| 200 μL Eppendorf Pipette | Supply | |||
| 10 mL serological pipet | Supply | |||
| Aluminum foil | Supply | |||
| Pipette tips | Supply | |||
| Hitachi F-7000 Fluorescence Spectrophotometer with FL Solutions 2.1 | Equipment | 5J1-0003 | ||
| Microplate Reader Accessory for F-7000 | Equipment | 5J0-0139 |
Riferimenti
- Singer, V. L., Jones, L. J., Yue, S. T., Haugland, R. P. Characterization of PicoGreen reagent and development of a fluorescence-based solution assay for double-stranded DNA quantitation. Analytical Biochemistry. 249 (2), 228-238 (1997).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon
Utilizziamo i cookies per migliorare la tua esperienza sul nostro sito web.
Continuando a utilizzare il nostro sito web o cliccando “Continua”, accetti l'utilizzo dei cookies.