זיהוי מיקרואורגניזמים סביבתיים עם תגובת שרשרת פולימראז ואלקטרופורזה ג'ל
Overview
מקור: מעבדות של ד"ר איאן פפר וד"ר צ'ארלס ג'רבה - אוניברסיטת אריזונה
מחבר מפגין: בראדלי שמיץ
תגובת שרשרת פולימראז (PCR) היא טכניקה המשמשת לזיהוי מיקרואורגניזמים הנמצאים בקרקע, במים ובסביבות אטמוספריות. על ידיגברת מקטעים ספציפיים של DNA, PCR יכול להקל על זיהוי וזיהוי של מיקרואורגניזמים היעד עד למין, זן, ורמת serovar / pathovar. ניתן להשתמש בטכניקה גם כדי לאפיין קהילות שלמות של מיקרואורגניזמים בדגימות.
פולחן המיקרואורגניזמים במעבדה באמצעות מדיית צמיחה מיוחדת הוא טכניקה ותיקה ונשאר בשימוש לגילוי מיקרואורגניזמים בדגימות סביבתיות. חיידקים רבים בסביבה הטבעית, בעודם בחיים, שומרים על רמות נמוכות של פעילות מטבולית ו/או זמני הכפלה ולכן מכונים אורגניזמים בני קיימא אך לא פולחניים (VBNC). השימוש בטכניקות מבוססות תרבות בלבד אינו יכול לזהות חיידקים אלה, ולכן אינו מספק הערכה יסודית של אוכלוסיות מיקרוביאליות בדגימות. השימוש ב- PCR מאפשר זיהוי של חיידקים הניתנים לתנור, אורגניזמים VBNC ואלה שאינם חיים או פעילים עוד, שכן הגברה של רצפים גנטיים אינה דורשת בדרך כלל העשרה מוקדמת של מיקרואורגניזמים הקיימים בדגימות סביבתיות. עם זאת, PCR אינו יכול להבדיל בין המצבים הנ"ל של כדאיות ופעילות. בשילוב עם טכניקה אחת או יותר המבוססת על תרבות, הכדאיות של תת-קבוצות מסוימות של מיקרואורגניזמים עדיין עשויה להיקבע.
Principles
הנחת היסוד של PCR היא להשתמש במחזורים חוזרים ונשנים של שינויי טמפרטורה רציפים כדי להשיג הגברה מעריכית של DNA. סינתזת הדנ"א מתבצעת על ידי אנזימי פולימראז DNA המתקבלים מחיידקים החיים במעיינות חמים, כגון תרמיוס אקווטיקה (Taq). פולימראזים אלה הם חום יציב, המאפשר להם לעמוד בטמפרטורות הגבוהות המשמשות במהלך PCR.
רצף היעד, המכונה אמפליסון, מוגבר מתבנית ה- DNA באמצעות שתי רצועות קצרות של נוקלאוטידים המכונים "פריימרים". בגלל הספציפיות הגבוהה של כריכת חומצת גרעין משלימה, הפריימרים מאפשרים הגברה ממוקדת של רצפים ספציפיים מאוד של עניין. על ידי עיצוב פריימרים שרק יגבירו רצף ייחודי (או שילוב ייחודי של רצפים) מאורגניזם מעניין, ניתן להשתמש ב- PCR כדי לזהות באופן דיפרנציאלי את נוכחות הדנ"א של האורגניזם בין כל החומרים הגנטיים הקיימים במדגם סביבתי מורכב.
כדי לבצע PCR, מכונה המכונה thermocycler משמש מחזור אוטומטי דרך הטמפרטורות השונות הנדרשות עבור התגובה. כל מחזור מחולק לשלושה שלבים. הראשון, המכונה "denaturation", מוגדר בדרך כלל מעל 92 °C (70 °F) ונמשך כ -30 מעלות צלזיוס. דנטורציה משמשת לפירוק מולקולות DNA לגדילים בודדים, כדי לאפשר לתגובת ההגברה להמשיך.
השלב השני, "חישול", מוגדר 2-3 °C (5 °F) מתחת לטמפרטורת ההיתוך של שני פריימרים, בדרך כלל בין 50-65 °C (50 °F), וגם נמשך כ -30 מעלות צלזיוס. טמפרטורת ההיתוך היא הטמפרטורה שבה 50% מהדנ"א הכפול נפרדו לגדילים בודדים, ולכן שלב ההמסה מאפשר לפריימרים להיקשר לאתרי היעד שלהם בתבנית הדנ"א.
השלב השלישי של מחזור PCR הוא "התארכות" או "הרחבה", כאשר פולימראז ה- DNA נקשר לדופלקס תבנית פריימר ומזרז סינתזה של המוצר. מוגדר על 72 °C עבור פולימראז טאק, משך שלב זה תלוי באורך של אמפליסון, בדרך כלל 30 s / 500 bp. לאחר כל מחזור, ה- DNA המוגבר שוב מושמצת ומשמש כתבנית חדשה, מה שמוביל לעלייה מעריכית בכמות מוצרי ה- PCR.
לאחר השלמת התגובה, ניתן לפתור את מוצרי ה- PCR לפי גודל על "ג'ל" העשוי בדרך כלל אגרוז פולימר, תהליך המכונה אלקטרופורזה. שדה חשמלי מוחל על פני הג'ל, והמטענים השליליים בעמוד השדרה של מולקולות ה- DNA גורמים להם לנדוד לעבר הקצה החיובי של השדה. באופן כללי, מולקולות DNA ליניאריות גדולות יותר ייקח יותר זמן לנסוע דרך מטריצת הג'ל.
Procedure
1. איסוף דוגמאות
- לאסוף אדמה באמצעות אוגר או את חפירה עד לעומק נחוש. אם אוספים אדמה מהקרניזון (האזור הצר של האדמה המקיפה ומושפעת משורשי הצמח והמיקרואורגניזמים הקשורים אליהן), אוספים ישירות מסביב לשורשים הצמחיים רק על ידי פגיעה בקרקע לחבית איסוף.
- לאסוף דגימת מים על ידי טבילת בקבוק פלסטיק סטרילי לתוך המים תוך החזקת הקצה של מקל הטבילה.
2. מיצוי והכנה של חומצות גרעין
- לאסוף אורגניזמים ווירוסים מהדגימה, ולחלץ דנ"א ורנ"א מהם. לפרטים, אנא עיינו בסרטון החינוך המדעי JoVE על מיצוי חומצת גרעין קהילתית.
- אחסן את הדנ"א שחולץ בצינורות מיקרופוגה מסומנים. אם ה- DNA צריך להיות מאוחסן בלילה או לתקופות זמן ארוכות יותר, להקפיא אותו ב -20 °C (70 °F), ולהפשיר את הצינורות בטמפרטורת החדר כאשר מוכן לשימוש.
- אם החומר הגנטי שיש להתבצע הוא RNA (בין אם זה הגנום של וירוסי RNA, או ה- RNA המתומלל של אורגניזמים תאיים), בצע שעתוק הפוך (RT) על המדגם כדי ליצור DNA משלים (cDNA) לפני שתמשיך ל- PCR. לפרטים, אנא עיינו בסרטון החינוך המדעי של JoVE ב- RT-PCR.
3. תגובת שרשרת פולימראז
- מניחים את האנזים PCR(למשל, טאק פולימראז) על קרח ומפשירים את ריאגנטים אחרים (חיץ PCR, dNTPs, פריימרים) בתוך מכסה המנוע "נקי" המיועד בטמפרטורת החדר. האנזים מאוחסן ב -20 מעלות צלזיוס אך לעולם אינו קופא. הוא רגיש לטמפרטורה, ולכן יש לשמור על קור רוח ועל חשיפתו לטמפרטורת הסביבה ממוזערת
- חשב את הנפח של כל ריאגנט הדרוש כדי ליצור "תערובת ראשית" של כל הריאגנטים הקבועים בין כל התגובות (טבלה 1). הקפד להסביר פקדים חיוביים(לדוגמה,תבנית הידועה כמכילה את אזור היעד) ושלילי(לדוגמה,ללא תבנית) בחישובים. הוסף 10% נוספים לאמצעי האחסון הסופי כדי להסביר שגיאת צנרת. כרכים פריימר תלויים בדיקות עבור אורגניזמים ספציפיים; עיין בספרות שפורסמה עבור הערכים המתאימים.
- באמצעות צינור מיקרו-קשירה נמוך, הממזער את ההידבקות של מולקולות ריאגנט לקירות הפלסטיק, מוסיפים את הנפחים המחושבים של כל ריאגנט כדי להרכיב את תערובת האב. מערבולת בעדינות וצנטריפוגות כל ריאגנט לפני הוספת. לאחר תערובת המאסטר מוכן, מערבולת לערבב ולאסוף על ידי צנטריפוגה
- הכן רצועות PCR 8-tube. ייעד צינור לכל דגימה, כולל פקדים חיוביים ושליליים
- מחלקים את הנפח המתאים של תערובת PCR לכל שפופרת של הרצועה
- הוסף את הנפח המתאים של תבנית DNA מן הדגימות, כמו גם 5 μL של התבנית החיובית ו 5 μL של מים כיתה מולקולרית כמו שליטה שלילית, לתוך צינורות PCR בהתאמה.
- מניחים את המכסה בבטחה על רצועת 8 הצינורות וצנטריפוגה למשך מספר שניות באמצעות מיני צנטריפוגה
- מניחים את רצועת 8 הצינורות בתרמוסקלר
- הגדר את תוכנית ה- PCR המתאימה לפעול על התרמו-מחזור. תוכנית טיפוסית מורכבת מהפעולות הבאות
- דנטורציה ב 94 °C (55 °F) במשך 3 דקות.
- 30-40 מחזורים של הגברה: denaturation ב 94 °C (30 °F) עבור 30 s, חישול בטמפרטורה ספציפית פריימר (בדרך כלל בין 50-60 °C (50 °F) עבור 30 °C (30 °F), והרחבה ב 72 °C (72 °F) עבור 30 ° C / 500 bp.
- הארכה סופית ב 72 °C (7-10 דקות).
4. הכנת ג'ל אגרוז
- בהתבסס על נפח הג'ל הרצוי וריכוז הג'ל (טבלה 2), לשקול את הכמות המתאימה של אבקת אגרוז לתוך בקבוק ארלנמאייר 125 מ"ל.
- מוסיפים את הנפח המתאים של מאגר ג'ל לבקבוקון, ומערבבים את הבקבוק ביד.
- מחממים את תערובת האגרוז במיקרוגל בעוצמה גבוהה במשך דקה אחת.
- מוציאים את הבקבוקון מהמיקרוגל ומערבבים ביד כדי לוודא שכל האגרוז נמס. אם האגרוז לא נמס לחלוטין, מיקרוגל חוזר במרווחים של 30-s.
- לאחר אבטחה הדוקה של המכסה על הבקבוקון, לקרר אותו ל 50 °C (50 °F) על ידי סיבוב תחת מים קרים זורמים.
- הוסף 1 μL של אתידיום ברומיד (EtBr) לתערובת אגרוז באמצעות מיקרופיפט המיועד EtBr. EtBr הוא צבע הקושר חומצות גרעין כפולות נטושות פלואורסים כתומים כאשר הוא מואר באור UV. שים לב כי EtBr הוא פוטנציאל מסרטן, כך ציוד מגן אישי (משקפי מגן, חלוק מעבדה, כפפות עמידות EtBr) חייב להיות משוחק.
- יוצקים את הג'ל המותך לתוך מגש יציקה של ג'ל אלקטרופורזה. ודא כי אין בועות לכודות בתוך agarose. מניחים מסרק לתוך הג'ל ומהדקים בבטחה. המתן כ 20-30 דקות עד שהג'ל יתמצה.
- לאחר הג'ל התגבש, להסיר את המסרק בזהירות מבלי לגרום לדמעות בג'ל. המסרק יוצר בארות בג'ל לטעינת הדגימות.
5. אלקטרופורזה ג'ל
- מניחים את ג'ל אגרוז מוצק לתוך תא אלקטרופורזה.
- מוסיפים את מאגר הריצה המתאים לתא עד שהג'ל בקושי שקוע.
- על חתיכת Parafilm, כתמי פיפטה של תרכיז צבע טעינה. כלול גם סולם DNA של מגוון מתאים לגדלים הצפויים של מוצרי PCR. לחלופין, השתמש קבוצה חדשה של צינורות microfuge, אחד עבור כל מדגם.
- לאחר ה- PCR, יש לאחזר את רצועת 8 הצינורות מהתרמוסקלר, ובקצרה צנטריפוגה כדי לאסוף עיבויים. הוסיפו נפח מתאים של מוצר DNA לצבע הטעינה והפפטה למעלה ולמטה כדי לערבב. לדוגמה, 2 μL של צבע טעינה 10x מעורבב עם 8 μL של מדגם כדי לתת נפח סופי של 10 μL, עם הצבע ב 1x.
- פיפטה כל תערובת דגימת צבע לתוך בארות המיועדות בג'ל אגרוז, נזהר לא לנקב את בארות.
- חבר את האלקטרודות לתא האלקטרופורזה. הדנ"א טעון לרעה, כך שהוא "רץ" לעבר האלקטרודה החיובית. לכן, לחבר את האלקטרודה החיובית לצד הנגדי של התא, שם בארות היו טעונות. הגדר את ספק הכוח למתח המתאים למערכת החיץ ולגודל תא הג'ל, והגדר אותו לפעול. בועות קטנות צריכות להיות גלויות נעות במעלה צידי התא אם האלקטרופורזה מתקדמת כראוי.
- ברגע שחזית הצבע התקדמה מספיק במורד הג'ל, כבה את אספקת החשמל. בזהירות להעביר את הג'ל לתוך transilluminator או הדמיה חזותית ולהדליק את אור UV כדי לדמיין את רצועות ה- DNA על הג'ל.
- לנתח את גודל הרצועה ואת המיקום על הג'ל. השווה את מיקומי הרצועה של הדגימות לשליטה החיובית כדי לקבוע אם DNA מהאורגניזם מעניין נמצא במדגם (איור 1).
| רכיב | נפח לכל צינור (μL) | נפח עבור 5 צינורות (μL) | ריכוז סופי |
| 10x חוצץ אקס טאק | 5.0 | 25 | 1x |
| 2.5 מ"מ של dNTPs | 4.0 | 20 | 0.2 מ"מ |
| פריימר קדמי* | 2.0 | 10 | 400 ננומטר |
| פריימר הפוך* | 2.0 | 10 | 400 ננומטר |
| מולקולרית H2O | 31.75 | 158.75 | - |
| אקס טאק | 0.25 | 1.25 | 2.5 U |
| תערובת PCR | 45 | 225 |
טבלה 1. אמצעי אחסון ריאגנט עבור תערובת מאסטר PCR. *נפחי הפריימר משתנים בהתאם לאי-ההסתעפות של האורגניזם. התאם את נפח המים המולקולריים כדי להפוך את הנפח הסופי 45 μL. אמצעי אחסון של רכיבים אחרים אינם אמורים להשתנות.
| % מומלץ של אגרוז | רזולוציה אופטימלית עבור שברי DNA ליניאריים (זוגות בסיס) |
| 0.5 | 1,000-30,000 |
| 0.7 | 800-12,000 |
| 1.0 | 500-10,000 |
| 1.2 | 400-7,000 |
| 1.5 | 200-3,000 |
| 2.0 | 50-2,00 |
טבלה 2. גודל שבר DNA טווחים נפתרו בצורה אופטימלית על ידי אחוזי ג'ל אגרוז שונים.
Results
באיור 1, סולם הדנ"א (נתיב 1) מספק התייחסות לגודל ולריכוז המשוער של רצועות של מוצרי ה-PCR. השליטה השלילית (נתיב 2) אינה מכילה כל חומר גנטי, בעוד הפקד החיובי (נתיב 3) מוגבר מתבניות הידועות כמכילות את ה- DNA המשמש כיעד כדי לציין את הגודל והמיקום של רצועות היעד. דגימות 4, 6, 8 ו-9 מציגות דפוס פס דומה לזה של השליטה החיובית, ולכן מצביעות על כך שדגימות אלה מכילות את החומר הגנטי של היעד. ניתן להסיק כי האורגניזם קיים בסביבות שמהן הושגו דגימות אלה.
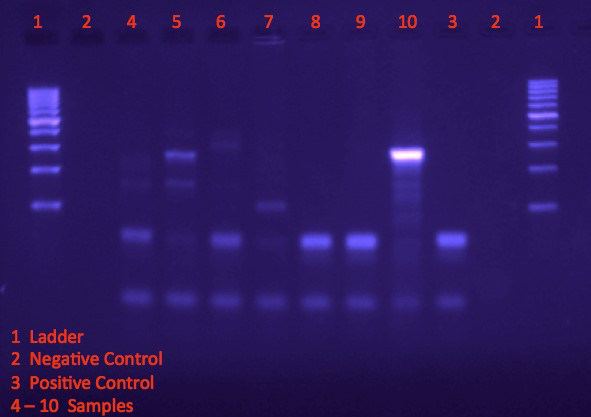
איור 1. לדמיין להקות על ג'ל אגרוז בעקבות אלקטרופורזה.
Application and Summary
PCR יכול להיות מועסק כדי לקבוע במהירות את נוכחות או היעדר פתוגנים בסביבה. לדוגמה, פריימרים ספציפיים אמבה אוכל המוח, Naegleria fowleri, יגבירו את ה- DNA וייצרו רצועות חזקות על ג'ל אם האורגניזם נמצא במדגם. אם אורגניזם יחיד אינו העניין העיקרי, אלא גנים הקשורים לייצור רעלים ממגוון אורגניזמים, PCR יכול לשמש גם כדי לקבוע את נוכחותם או היעדרם של חומרים גנטיים ספציפיים אלה.
PCR יכול לשמש גם כהליך אישור בעת ניתוח חיידקים סביבתיים במעבדה. אם שיטת תרבית אינה יכולה להבדיל בין אורגניזמים מסוימים הנמצאים במדגם סביבתי, אז PCR אולי משמש להבחין באופן ספציפי בין חיידקים המועמדים.
PCR קונבנציונאלי ניתן לשנות במספר דרכים למטרות ניסיוניות מסוימות. ניתן להשתמש ב- PCR כדי לנתח תבניות RNA חד-גדיליות על-ידי צימוד לשלב שעתוק הפוך (RT-PCR). מעבר לקביעת נוכחות לעומת היעדרות, PCR כמותי (qPCR) יכול למדוד את הריכוז עבור DNA ספציפי של עניין.
Tags
Skip to...
Videos from this collection:

Now Playing
זיהוי מיקרואורגניזמים סביבתיים עם תגובת שרשרת פולימראז ואלקטרופורזה ג'ל
Environmental Microbiology
44.6K Views

קביעת תכולת הלחות בקרקע
Environmental Microbiology
359.3K Views

טכניקה אספטית במדעי הסביבה
Environmental Microbiology
126.4K Views

כתמי גרם של חיידקים ממקורות סביבתיים
Environmental Microbiology
100.2K Views

הדמיית מיקרואורגניזמים קרקע באמצעות מצג שקופיות מגע ומיקרוסקופיה
Environmental Microbiology
42.2K Views

פטריות נלאמנטיות
Environmental Microbiology
57.3K Views

הפקת דנ"א קהילתי ממושבות חיידקים
Environmental Microbiology
28.8K Views

ניתוח RNA של דגימות סביבתיות באמצעות RT-PCR
Environmental Microbiology
40.4K Views

כימות מיקרואורגניזמים סביבתיים ווירוסים באמצעות qPCR
Environmental Microbiology
47.8K Views

ניתוח איכות מים באמצעות אורגניזמים מחוון
Environmental Microbiology
29.5K Views

בידוד חיידקים צואתיים מדגימות מים על ידי סינון
Environmental Microbiology
39.3K Views

זיהוי בקטריופאג'ים בדגימות סביבתיות
Environmental Microbiology
40.7K Views

פולחן ופירום חיידקים מדגימות קרקע
Environmental Microbiology
184.3K Views

ניתוח עקומת גדילה חיידקית והיישומים הסביבתיים שלה
Environmental Microbiology
295.9K Views

1 200 אצות באמצעות מתודולוגיה של Culturable
Environmental Microbiology
13.8K Views
Copyright © 2025 MyJoVE Corporation. All rights reserved