Method Article
ניתוחים מבניים של משתנה שבר שרשרת יחיד ספציפי לקולטן גורם גדילה אפידרמלי באמצעות גישת In Silico
In This Article
Summary
פרוטוקול חיזוי זה של מידול הומולוגיה של נוגדנים מלווה בעגינה של Pyrx לקולטן נוגדנים וסימולציה דינמית מולקולרית. שלוש השיטות העיקריות הללו משמשות להמחשת אזורי הקישור המדויקים של קולטני נוגדנים ויציבות הקישור של המבנה הסופי.
Abstract
נוגדנים משתנים של שרשרת אחת (scFv) נבנו בעבר משרשראות קלות וכבדות משתנות שאליהן הצטרף מקשר (Gly4-Ser) 3. המקשר נוצר באמצעות תוכנת מידול מולקולרי כמבנה לולאה. כאן, אנו מציגים פרוטוקול לניתוחבסיליקו של נוגדן scFv שלם המקיים אינטראקציה עם קולטן גורם הגדילה האפידרמלי (EGFR). המודלים ההומולוגיה, עם Pyrx של עגינת חלבון-חלבון וסימולציה דינמית מולקולרית של נוגדן scFv האינטראקציה ו-EGFR ראשית, המחברים השתמשו בתוכנית מידול מבנה חלבון וב-Python למידול הומולוגיה, ומבנה הנוגדן scFv עוצב להומולוגיה. החוקרים הורידו את תוכנת Pyrx כפלטפורמה במחקר העגינה. הסימולציה הדינמית המולקולרית הופעלה באמצעות תוכנת מידול. התוצאות מראות שכאשר סימולציית MD הייתה נתונה למזעור אנרגיה, למודל החלבון הייתה אנרגיית הקישור הנמוכה ביותר (-5.4 קק"ל/מ'). בנוסף, סימולציית MD במחקר זה הראתה כי נוגדן EGFR-scFv המעוגן היה יציב במשך 20-75 ns כאשר תנועת המבנה גדלה בחדות ל-7.2 Å. לסיכום, בוצע ניתוח בסיליקו, וסימולציות העגינה המולקולרית והדינמיקה המולקולרית של נוגדן scFv הוכיחו את יעילותה של התרופה הטיפולית-חיסונית המתוכננת scFv כטיפול תרופתי ספציפי ל-EGFR.
Introduction
שינויים קונפורמטיביים בחלבון (ליגנד וקולטן) מתרחשים תמיד על סמך פונקציות מבוססות מבנה. חקר חריצי הקישור האפשריים של החלבון וחיזוי אינטראקציית הקישור היציבה היא שיטה מתקדמת להכנת תרופות לשימוש טוב יותר בגוף האדם. מידול הומולוגיה ואחריו עגינה וסימולציה דינמית מולקולרית היא שיטה פשוטה לחיזוי מדויק של אינטראקציות יציבות של קשירה בין שאריות של קולטנים ונוגדנים בנויים המשמשים כרפואה מותאמת אישית ספציפית 1,2. מבנה המודל החזוי יכול להראות שינויים קונפורמטיביים וסידורים מחדש באתרי קשירת ליגנד-קולטן, במיוחד בממשק הנוגדן-קולטן. ישנן סיבות רבות לשינויים אלה, כגון סיבוב של שרשראות צד, טרנספורמציה מבנית גלובלית או שינויים מורכבים יותר. הסיבה העיקרית למידול הומולוגיה היא להבחין בין המבנה השלישוני של חלבון לבין המבנה הראשוני שלו 2,3.
קולטן טירוזין קינאז הנקרא קולטן גורם גדילה אפידרמלי (EGFR) ממלא תפקידים ביולוגיים רבים בתאי סרטן, כולל אפופטוזיס 4,5, התמיינות 6,7, התקדמות מחזור התא 8,9, התפתחות 9,10 ושעתוק11. EGFR הוא אחד היעדים הטיפוליים הידועים לסרטן השד12. ביטוי יתר של פעילות קינאז רגילה כגון EGFR מוביל בדרך כלל להתקדמות תאים סרטניים, שיכולה להיות מדוכאת על ידי סוגים רבים של מעכבי סרטן13. קולטן גורם הגדילה האפידרמלי (EGFR) שימש כקולטן למשתנה שבר השרשרת היחיד שנבנה במיוחד כדי לעבוד נגד קולטן זה. המבנה החזוי שלו שימש לבדיקת פעילות קשירת הנוגדנים.
במאמר זה, מבנה הנוגדנים scFv עוצב באמצעות תוכנת מידול עם סקריפט Python ושיטת מידול הומולוגיה14,15. ניתן לבנות מודל הומולוגיה מרצפי החלבונים וחומצות האמינו של קולטנים וליגנדים16,17. בנוסף, נעשה שימוש בטכנולוגיות ביואינפורמטיקה מתקדמות כגון עגינה מולקולרית כדי לחזות כיצד ליגנדים של מולקולות קטנות ייקשרו לאתר קשירת המטרה הנכון. העגינה תאזן את הפיתוח של תרופות חדשות המכוונות למחלות מרובות. התנהגות הקישור נלקחת בחשבון 5,18.
יתר על כן, עגינה מולקולרית היא טכניקה קריטית כדי להקל ולהאיץ את התפתחות קשירת קולטני הליגנד. עגינה מולקולרית מאפשרת למדענים לסנן באופן וירטואלי ספריית ליגנדים כנגד חלבון מטרה ולחזות את קונפורמציות הקישור והזיקה של הליגנדים לחלבון קולטן המטרה. סימולציה דינמית מולקולרית (MNS) מדגימה כיצד השאריות נעות בחלל, מדמה את תנועות הנוגדנים לעבר הקולטנים שלהם, ולבסוף מודיעה על מאמצי תכנון הנוגדנים. מחקר זה הוא חיזוי חדשני של מידות קופסאות הרשת שהחליטו כיצד נוגדן scFv נקשר ל-EGFR וזיהוי האנרגיה והזמן של קשירה זו בסימולציית MD.
Protocol
1. חיזוי מבנה משני של חלבון משתנה שבר שרשרת בודד (scFv)
- בנה את המבנה התלת-ממדי של משתנה השבר היחיד (scFv) עם בנק נתוני חלבון BLAST (PDB), מספור KABAT ותוכנת המידול. ה-scFv מורכב מקישור (Gly4-Ser) המחבר שרשרת כבדה משתנה (VH) ושרשרת קלה משתנה (VL).
- השתמש בתוכנת המידול המולקולרי כדי לבנות את המקשר כמבנה לולאה, ובצע את כל השיטות הללו כמתואר במחקרים קודמים 2,19,20.
2. בחירת תבניות וחיזוי מבנה תלת מימד scFv ו-EGFR ומידול הומולוגיה
- בחר תבנית 1ivo עבור מבני EGFR (בהתבסס על הרזולוציה הגבוהה שלה). הורד את 1ivo.pdb file מ-pdb webאתר, כפי שמוצג באיור 1B.
- הכן את הקלט 1ivo.pdb file כמתואר להלן.
- בקובץ 1ivo.pdb, הסר את כל הליגנדים החיצוניים על ידי פתיחת אתר pdb.org ובחירת 1ivo. מבנה, ומחפש את שם הליגנדים תחת כותרת המולקולה הקטנה בדף מבנה 1ivo באתר pdb.
- מצא את שם הליגנד NAG. פתח את הקובץ 1ivo.pdb שהורד מאתר pdb ומצא את שאריות הסיום (TER).
- מחק את שאריות הליגנדים החיצוניים במבנה 1ivo, החל מהשאריות לאחר TER ולפני שהשאריות מסתיימות. שמור את 1ivo.pdb file במערכת.
- הכן את הקובץ 1ivo.pdb שנשמר כמתואר להלן.
- הורד את תוכנת העגינה של Autodock (autodock.scripps.edu) מאזור בחירת החלון. לחץ על הקובץ Open 1ivo.pdb.
- השתמשו בפקודה Edit כדי לבחור Add Hydrogen > Add, לאחר מכן בחרו Polar Only ולאחר מכן לחצו על Ok.
- השתמש בפקודה Edit כדי להוסיף חיובי Kollman (איור משלים 1). השתמשו בפקודה 'עריכה' למחיקת מים. שמור את הקובץ 1ivo.pdb במחשב.
- צמצם את האנרגיה של מבנה 1ivo.pdb כמתואר להלן.
- הורד את SPDBV. תוכנה מבית http://spdbv.vital-it.ch/disclaim.html. פתח את הקובץ 1ivo.pdb.
- בחר הכל. בחר את הפקודה ביצוע > מזעור אנרגיה > אישור (איור משלים 2). שמור את קובץ 1ivo במחשב.
- הכן את המודל המלא scFv באמצעות מודלים הומולוגיה כמתואר להלן.
- הורד את תוכנת המידול17 ואת מעטפת סקריפט Python 3.7.9 מהחלון- 64. שמור את קבצי התוכנה שהורדת בכונן D.
- הכן את קבצי הקלט כמתואר להלן.
- טען את קובץ scFv Pdb בפורמט fasta מאתר NCBI ושנה את שם הקובץ TARGET.ali. כמתואר בקובץ קידוד משלים 1. בחר את התבנית באמצעות המקטע Blast ב-NCBI, הדבק את הקובץ ברצף, בחר בפורמט pdb 7det.pdb כמתואר בקובץ קידוד משלים 2 ולאחר מכן שלח. לאחר מכן, השתמש באתר pdb.org כדי להשיג את קובץ התבנית.
- הכן את קובץ הקלט השלישי כ-align2d.py (Python) כמתואר בקובץ קידוד משלים 3, שנפתח כפי שמוצג באיור משלים 3A. לחץ על הצג עוד אפשרות ולאחר מכן עבור אל ערוך עם IDLE > ערוך עם EDLE (64 סיביות). הפעל באמצעות הפקודה Run module 5 align2d.py כדי לקבל שני קבצי פלט: Tar- 7det.ali ו- Tar- 7det.pap.
- השלם את שלושת השלבים הקודמים כדי להשתמש בפקודה בקובץ הקלט האחרון.
- הוסף קובץ קלט חדש model-single.py (קובץ פייתון פקודה) כפי שמוצג בקובץ קידוד משלים 4 וכמתואר להלן.
- לחץ על האפשרות הצג עוד ולאחר מכן עבור אל ערוך עם IDLE > ערוך עם EDLE3.7 (64 סיביות). הפעל באמצעות הפקודה (הפעל מודול 5) כפי שמוצג באיור משלים 3B.
הערה: קבצי הפלט המתקבלים הם ששת הקבצים של המודלים ההומולוגיה המוצגים באיור משלים 3C.
- לחץ על האפשרות הצג עוד ולאחר מכן עבור אל ערוך עם IDLE > ערוך עם EDLE3.7 (64 סיביות). הפעל באמצעות הפקודה (הפעל מודול 5) כפי שמוצג באיור משלים 3B.
3. חיזוי והערכה של מבנה משני של הקולטן
- זהה את התיקון והדיוק של מודלים הומולוגיה כמתואר להלן.
- צור את עלילת Ramachandran עבור דגמי scFv ודגמי EGFR על ידי הורדת כלי ההדמיה מ-https://discover.3ds.com/discovery-studio-visualizer-download.
- פתח את file ולאחר מכן לחץ באמצעות לחצן העכבר הימני ובחר את רצף התצוגה (איור משלים 4). העתק את הרצף והדבק אותו במסד הנתונים הציורי של מבנים תלת-ממדיים (pdbsum) www.ebi.ac.uk/thornton-srv/databases/pdbsum/.
- בחר חיפוש לפי רצף, הדבק את העותק של הרצף ולאחר מכן שלח אותו. צור את העלילה כפי שמוצג באיור 1B,D.
4. עגינה של חלבון-חלבון
- הורד את תוכנת כלי הסינון הווירטואלי.
- עבור לקובץ> קרא מולקולות > טען 1ivo.pdb. לחץ לחיצה ימנית על החלבון בלוח העגינה האוטומטית כדי ליצור מקרומולקולה. לחץ שוב באמצעות לחצן העכבר הימני כדי ליצור ליגנד (איור משלים 5).
- לחץ בחלונית העגינה האוטומטית כדי לבחור חלבון ולאחר מכן בחר ליגנד.
- פתח את רשימת החלבונים. לאחר מכן, מהרשימה, בחר חלבון scFv.
- עבור אל החלפת מצב כדורי בחירה. התאם את תיבת הרשת למרכז הקולטן. לחץ על Forward כאשר הכפתור הוורוד העגול מופיע.
- כדי להכין את קבצי ה-pdbqt הן עבור מבני נוגדן scFv והן עבור מבני EGFR (1ivo), השתמש בשלבים הבאים.
- עבור אל C Drive > Program Files (86) > משתמשים, ולאחר מכן בחר את קובץ ה-pyrx המכיל מקרומולקולות וקבצי פלט חלבון שנשמרו כקובץ pdbqt.
- לאחר מכן, שמור את קובץ הנוגדן pdbqt של משתנה שבר שרשרת אחת (scFv).
- הורד את תוכנת PyMOL ב-PyMOL | pymol.org. השתמש בתוכנת PyMOL כדי להציג את תצורות ה-EGFR של קולטני נוגדנים scFv.
- עבור לקובץ ופתח את C:\Users\ilham\.mgltools\PyRx\Macromolecules\protein. הכן את תצורות העגינה של scFvantibody המקיים אינטראקציה עם הקולטן באיור 2A כמתואר להלן.
- השתמש באפשרות התצוגה כדי להציג את הקולטן (1ivo) file כשאריות רצף עם רקע לבן המוצג באיור משלים 6.
- הצג את תצורת העגינה file ברזולוציה גבוהה יותר כדי לראות את צבע הליגנד בצבעי שאריות ירוק ואדום. הצג את המשטח הקשיח של הקולטן (1ivo) בצהוב.
- עבור לקובץ ופתח את C:\Users\ilham\.mgltools\PyRx\Macromolecules\protein. הכן את תצורות העגינה של scFvantibody המקיים אינטראקציה עם הקולטן באיור 2A כמתואר להלן.
- הכן את תצורות העגינה של scFvantibody המקיים אינטראקציה עם הקולטן באיור 2B, כמתואר להלן.
- הורד את תוכנת העגינה מאזור בחירת החלון. השתמש ב-Autodock כדי להציג את התצורות והקונפורמציות של EGFR של קולטני נוגדנים scFv.
- ב-Autodock, בחר/י באפשרות ״נתח״ ולאחר מכן פתח/י את התוצאה של Autodock Vina. עבור אל קובץ ופתח את C:\Users\ilham\.mgltools\PyRx\Macromolecules\protein.
- בחר את קובץ ה-pdb של קולטן החלבון, ולאחר מכן בחר את האזור של תצורת הליגנד (מבנה נוגדני scFv). חבר את המשטח הנוקשה של הקולטן עם תצורת המבנה המעוגן והסתר את שאר הקולטן. הסתר את השאריות הרחוקות של הקולטן מהשאריות המחוברות לליגנד, כפי שמוצג באיור 2B.
- קומפלקס החלבון-חלבון נחשב אז מוכן לביצוע סימולציית MD.
- הורד את תוכנת העגינה מאזור בחירת החלון. השתמש ב-Autodock כדי להציג את התצורות והקונפורמציות של EGFR של קולטני נוגדנים scFv.
5. סימולציה דינמית מולקולרית (MD Simulation) של קומפלקס עגינת הנוגדנים EGFR-scFv
- הורד את תוכנת הסימולציה MD והשתמש בה באופן הבא.
- הכן את קובץ ה-pdb של EGFR (1ivo) באמצעות אשף התיקון כמו באיור משלים 7A. הפעל את קטע העיבוד המקדים כדי לחדד את הקובץ. שלח את הקובץ המעודכן להגדרה בבונה המערכות.
- טען את תוכנת הדמיית הדינמיקה המולקולרית מספריית העבודה. הוסף את היונים ושדרג את המעודן file כדי להגיע ל-20 Å כדי להגיש את העבודה (טבלה 1), מוצגת גם באיור משלים 7B.
- טען את ה-EGFR (1ivo) pdb מהקובץ המיובא, ולאחר מכן בחר צעדי זמן של 100 ns כדי להפעיל אותו (איור משלים 7C).
- התחל את ניתוח הסימולציה לאחר השלמת סימולציית MD כמתואר להלן.
- צור תיקיית עבודה ושמור אותה בקובץ cms . טען את קובץ ה-cms כדי לבצע שלב זה בהדמיית MD.
- צור ספריית עבודה לתיקיות הפרויקט ודווח על ערכי האנרגיה. השתמש S.I.D. pdf כדי לדווח על הסימולציה, כפי שמוצג באיור 3A, ועל דיאגרמת האינטראקציה וקשר H, כפי שמוצג באיור 3B.
- טען את קובץ ה-pdf של cms על ידי גלישה מהתיקייה והשתמש ב-TIP3P כמודל למזעור נפח הקבצים.
- צור את קובץ המסה כדי לבצע שלב זה המוצג באיור משלים 7D. שמור קובץ pdf דרך התוכנה, ונתח את הנתונים, וכתוצאה מכך איור 4, איור 5 ואיור 6.
- צור הגדרת סיום MDsimulation על ידי יצירת קובץ הפתרון. מצא את התוצאות בתיבת הגבול, כפי שמוצג באיור 7.
תוצאות
באמצעות טכנולוגיית תצוגת פאג'ים, הגן scFv anti-EGFR נוצר מקו ההיברידומה של תאי B של העכבר C3A820,21. דגמי המבנה המשתנה של שבר שרשרת בודד (scFv) של מבני VH ו-VL נבנו בנפרד, על פי Chua et al.22. לאחר מכן, הדגמים נראו כסרטים שיוצרו באמצעות RasMol. לאחר מכן, באמצעות תוכנת מידול מולקולרי, נעשה שימוש בפפטיד סינתטי [Gly4Ser)3 כדי לחבר את מבני ה-VH וה-VL שמודלו בנפרד. אינטראקציית הליגנד עם הקולטן מופעלת כמבנה מקומי המשנה את אתרי הקישור הנקראים קצוות משתנים. האזור הקובע את ההשלמה (CDR) שנמצא במבני חלבון נוגדנים) יוצר קשירת חריצים טבעית לקשירת אפיטופ 3,23. קשירה אנטיגנית גורמת לשינוי קונפורמציה של CDR כדי להתאים את שאריות 183 לצורה האידיאלית ולהקל על התכונות הפיזיקוכימיות הנכונות של השאריות. במקום להשוות את המבנים הגבישיים של המולקולות הקשורות לנוגדנים והמולקולות הלא קשורות לנוגדנים, בחינה דינמית של תהליך הקישור נחשבת לדרך הטובה ביותר להבין את המנגנון התלת-ממדי האלוסטריהבסיסי 23.
כפי שמוצג באיור 1A-D, העלילה משמשת כדי לקבוע אם הקונפורמציה של עמוד השדרה נחזה נכון. כפי שמוצג באיור 1A,C, רוב הפפטידים נמצאים במרכז הנכון של העלילה, מה שמדגים את הדיוק והנכונות של המבנים באיורים 1B,D. המודלים מוערכים לאחר מכן באמצעות עלילות רמאצ'נדרן כדי לקבוע את הדיוק של מודלים אלה24. תהליך הבנייה וההערכה המלא של מודל scFv הוכן כך שפרופיל האנרגיה הממוצע היה מתחת לאפס. שתי חלקות Ramachandran הראו את אישור עמוד השדרה הנכון לתבנית הקולטן של 3njp ותבנית נוגדנים scFv של 1osq. רוב הפפטידים מרוכזים β יריעות24, ואילו השאר נמצאים בסליל α של חלקה25. עלילת רמאצ'נדרן היא השיטה המהירה והקלה ביותר לאיתור תיקון ודיוק פפטידים מבניים24,26.
איור 2A מראה את המבנה הגבישי של אינטראקציית הקישור בין הקולטן לליגנד במבנה פני השטח. איור 2B מציג את פרופיל האינטראקציה של אישור והכריכה הנמוכה ביותר. עגינת חלבון-חלבון בוצעה באמצעות תוכנת PyRx, והליגנד נשמר גמיש בעוד מבנה החלבון נשמר נוקשה. המקרומולקולה והליגנד הוכנו בעזרת תוכנת הכלים MGL 1.5.7. הליגנד סונתז על ידי סיבוב כדי להפוך אותו לגמיש, והמקרומולקולה נוצרה על ידי הסרת מולקולות מים והוספת אטומי מימן. 186 מבני גביש קשורים לנוגדנים ובלתי קשורים לנוגדנים שימשו לאחסון האנרגיה הפוטנציאלית הנובעת מהאינטראקציה בין כל אטום בליגנד הגמיש13. בתחום הפונקציונלי של מולקולת החלבון שנבחרה, הליגנד עוגן סביב האזור השמורשהתגלה 16. האינטראקציה המועדפת נבחרה על סמך קונפורמציית הקישור החיובית בין המקרומולקולה לליגנד בעלת אנרגיית הקישור הנמוכה ביותר (BE), כפי שנקבע על ידי תוכנית העגינה25,26. גודל תיבת הרשת היה 40 Å x 40 Å x 40 Å x 40 Å לאורך צירי X, Y ו-Z וממורכז ב-90.653, 56.2181 ו-50.1986, בעוד שגודל תיבת רשת העגינה הממוקדת הוגדר ל-40 Å x 40 Å x 40 Å וממורכז בקואורדינטות X, Y ו-Z של 49.086, 25.0 ו-25.0, בהתאמה. גודל תיבת הרשת נוצר באופן ידני.
איור 3A מציג את האינטראקציה בין שאריות ליגנד-קולטן (אינטראקציה עם קולטן). כפי שמוצג, השאריות הירוקות הכהות מראות את השאריות שהיו להן קשירה חזקה יותר בין נוגדן scFv-EGFR (1ivo) עם קשרי H. בניגוד לשאריות האדומות עם סוגי מליטה שונים, כפי שמוצג באיור3A, המספר הכולל של שאריות הוא 1117, המוגדרות כ-A=513, B=510, C=7 ו-D =447. A הוא הקולטן, ישנם 8685 אטומים כבדים, ומטעני האינטראקציה הם +55. יתר על כן, שמונה אינטראקציות של קשרי מימן מוצגות באיור 3B: אינטראקציה של קשר 1H של DNA. חלבון עם asn:32 של קולטן חלבון, אינטראקציה של קשר H אחד של DNA. חלבון עם ASN:33 של קולטן חלבון, 4 קשרי H עם 3tpo, 1 קשר H עם tp 3p45 וקשר 1H עם t3p5627. לכן, אינטראקציות אלה מציגות יציבות וחוזק מורכבים מעוגנים טובים מאוד.
כפי שמוצג באיור 3B, קשרי המימן ליציבות העגינה (אינטראקציה של ליגנד-קולטן) ומספר קשרי המימן שזוהו קובעים תמיד את חוזק יציבות העגינה. קיפול חלבון נעשה סביר יותר על ידי קשרי מימן, ולשרשרת יש גישה לכל דרגות החופש כדי להשיג את המבנה האידיאלי שלה. עם זאת, עבור קשירה נוקשה, מולקולות החלבון כבר מקופלות 27,28,29 , מה שמשאיר את השרשראות עם שש דרגות חופש בלבד בתרגום וסיבוב כדי לסדר את עצמן לתצורה המוגבלת המועילה ביותר.
נצפו סימולציות דינמיקה מולקולרית (MDS) של קשירת קולטן EGF עם חלבון scFv. בטבלה 1, ניתן להשתמש במידע נגד יון/מלח, החלת שיטת NaCl על קשירת קולטן EGF עם אינטראקציית קשירת חלבון scFv, וסטיית שורש-ממוצע-ריבוע (RMSD) כדי לזהות אזורים של מצבים מטה-יציבים בנוסף למצב היציב. המחברים הבחינו כי המבנים החזויים אינם יציבים, וקיימת אפשרות שהמסלול המתקבל על ידי סימולציה אינו אמין. לכן, סימולציית MD יכולה להראות את דפוס התנועה העקבי של המסלול המתקבל מהעגינה של אינטראקציית הקולטן-נוגדן, וכתוצאה מכך כוח היגוי המייצר ערכים יציבים במבנים החזויים.
כפי שמוצג בטבלה 1, החישובים מתבצעים עם ריכוזי NaCl הנעים בין C = 0.5 מ' ל- C = 0.75 מ'. ניתוחים מבוצעים על האופן שבו יונים משפיעים על רשת המים המקושרת למימן 258 ועל המבנה הטטרהדרלי30. בגלל תצורה לא טטרה-דיהדרלית, הניתוח מדגים כי הגדלת תכולת המלח מורידה את הסידור הטטרהדרלי של המים31. אנו מזהים את המקור והתכונות הזוויתית של מבנה לא טטרהדרלי זה בשיטת השכן הקרוב ביותר. יתר על כן, המבנה הטטרהדרלי של המים מושפע באופן משמעותי יותר מיוני ההשוואה Na+ ו-Cl- מאשר מיונים אחרים30,31. גישת RMSD היא השיטה הסטנדרטית. ב-RMSD, החלבון ממוקם על ציר ה-Y. מסגרות החלבון ומסגרת הייחוס של עמוד השדרה המבני מסתדרות לעתים קרובות. כאשר קשרי מימן מוערכים בסמיכות למבנה היציב ביותר של כלוב ה-Trp, ה-RMSD מחושב 32,33.
כפי שמוצג באיור 4, חושבה הסטייה הריבועית הממוצעת של השורש (RMSD); החלבון RMSD עלה בהדרגה מ-3.2 Å ל-6 Å עבור צעד הזמן הראשון של 0 עד 20 ns. לאחר מכן, מ-20 ל-75 ns, החלבון RMSD נשמר בטווח של 4 Å עד 5.6 Å ולאחר מכן השתנה מ-75 ns ל-90 ns. לבסוף, תנועות החלבון RMSD עלו בחדות עד 7.2 Å וירדו עד 6.4 Å עד לשלב הזמן האחרון. זה מצביע על התנועה החופשית של החלבונים. זה מראה את חוסר היציבות של המבנה המעוגן כאשר צעד הזמן גדל, והיציבות של המבנה המעוגן של הקולטן-ליגנד מסופקת כאשר צעד הזמן של RMSD הוא בטווח של 20 עד 75 ns. התרשים לעיל מראה כי ה-RMSD מעריך את הקונפורמציה המבנית לאורך כל הסימולציה. ניתוח RMSD הצביע על כך שהסימולציה הייתה מאוזנת ויציבה.
תנודת שורש ממוצעת ריבועית (RMSF), מוצגת באיור 5. ב-RMSF, תזוזה של אטום מסוים הושוותה למבנה ייחוס, ונמדד מספר האטומים הממוצע. הוא משמש לעתים קרובות כדי להבחין אם מבנה יציב בסולם הזמן של הסימולציות או לא. יתר על כן, באיור 5, החלבון מראה RMSF בטווח של 2.4 Å עבור שאריות בין 1-100. לאחר מכן, הוא יורד ל-2a עבור 200-300 שאריות; יתר על כן, הוא משתנה ל-5.6 Å או 500 שאריות ואז יורד בחדות ל-2.4 Å עבור שאריות עד 1000 ns. לבסוף, יש שיא חד אחד במיקום 1000 עם ירידה עד 2.4 Å בטרמינל. תוצאות RMSF עוזרות לאפיין שינויים במיקומי אטומי הליגנד במהלך עגינה25. תוצאות אלו מייצגות את תהליך הקשירה, ובדרך כלל, המודל הסופי, המודל הקרוב ביותר, מייצג את מבנה הגביש במונחים של RMF, וניתן להראות אינטראקציות קשירה בבירור בתמונות סופיות, כפי שהודגם במחקר הקודם על ידי Huang et al.2.
כפי שנצפהב-34, המבנה המעוגן של נוגדן EGFR-scFv היה יציב מעל 1000 ns של סימולציית MD. לכן, אלמנטים של מבנה משני של חלבון (SSEs), כגון סלילי אלפא וגדילי בטא, נוטרו לאורך כל הסימולציה כדי לצפות בשינויים במדד השאריות ביצירת חלבון SSEs35,36. הקצאה לאורך זמן מוצגת באיור 6. אלמנטים של מבנה משני של חלבון (SSEs), כגון סלילי α (איור 6 באדום) ו-β גדילים (איור 6 בכחול), מנוטרים לאורך כל הסימולציה. התרשים לעיל מדווח על התפלגות SSE לפי אינדקס שאריות בכל מבנה החלבון. כפי שמוצג באיור 7, המודל המצויר מציג מקטעי חלבון, ליפידים ומולקולות מים המסודרים בקווים ישרים ואת אטומי הזרחן של השומנים בחרוזים.

איור 1: תרשים לבחירת תבנית. העלילה משמשת כדי לקבוע אם קונפורמציה של עמוד השדרה נחזה נכון. כפי שמוצג ב-(A) ו-(B), רוב הפפטידים נמצאים במרכז הנכון של העלילה, מה שמדגים את הדיוק והנכונות של המבנים ב-(C) ו-(D). אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
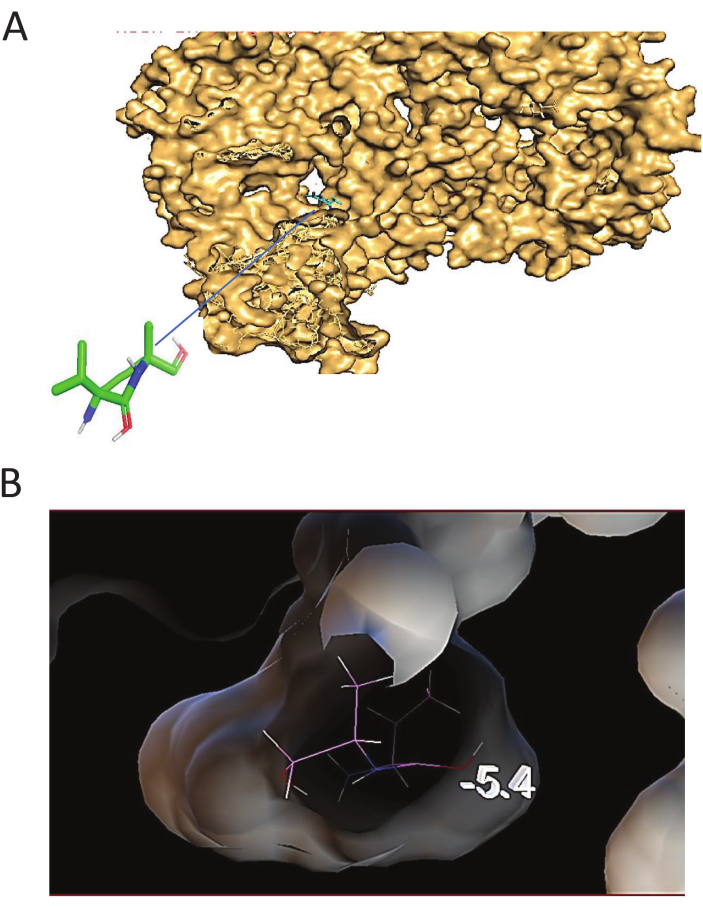
איור 2: מבנה ותצורת העגינה. (A) מבנה העגינה של נוגדן הקולטן-scFv מראה את המבנה הגבישי של אינטראקציית הקישור בין הקולטן לליגנד במבנה פני השטח. (ב) תצורת עגינה. תצורת עגינה פרופיל אינטראקציה של אישור והכריכה הנמוכה ביותר שהוצגה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 3: אינטראקציה בין שאריות ליגנד-קולטן. (A) אינטראקציה של שאריות של מבנה קולטן ליגנד מראה שהמספר הכולל של מספרי שאריות הוא 1117 שאריות המוגדרות ל-A = 513, B = 510, C = 7, D = 447. A הוא הקולטן. מספר האטומים הכבדים הוא 8685, בעוד שמטעני האינטראקציה הם +55. היישור הירוק הכהה הוא האינטראקציה החזקה ביותר עם קשרי H. יישור שאריות קולטני ליגנד מראה שהירוק הוא הקשר החזק ביותר עם קשר H, הירוק הבהיר הוא קשר קל יותר, והאדום הוא הקשר החלש יותר. (B) אטומי הליגנד מקיימים אינטראקציה עם שאריות החלבון. האינטראקציות בין הליגנד לחלבון הקולטן מתרחשות ביותר מ-30.0% מזמן הסימולציה. שאריות הקולטן מחוברות לקשר H של אטומי הליגנד. שאריות הקולטנים בעיגולים ירוקים כהים מייצגות את האינטראקציה החזקה ביותר של קשר H בין קולטן EGFR לנוגדני scFv. ישנם שמונה קשרי H, הנחשבים לאינטראקציה חזקה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
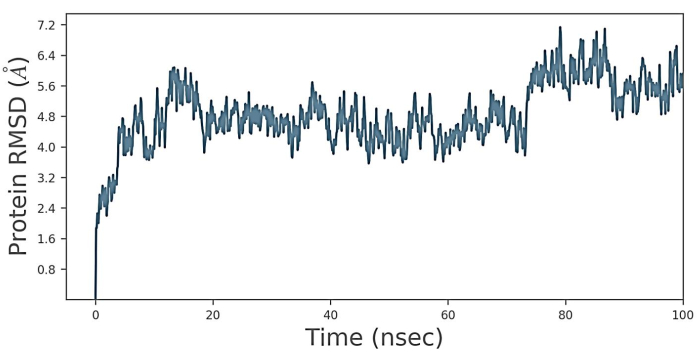
איור 4: סטייה ריבועית ממוצעת שורש (RMSD). תרשים RMSD של קומפלקס קולטן-ליגנד לזמן סימולציה של 20 ns. נצפה כי חלבון יוצר 3.2 Å עד 6 Å עבור צעד הזמן הראשון של 0 עד 20 ns. בנוסף, החלבון, מ-20 ns ל-75 ns צעד זמן. התנועה נשמרת מ-4 Å עד 5.6 Å ו-75 עד 90 ns. התנועה עולה בחדות ל-7.2 Å ואז יורדת ל-6.4 A עד לשלב הזמן האחרון. תוצאות אלה הוכיחו כי המבנה המעוגן של נוגדן EGFR-scFv היה יציב לאורך 1000 ns של סימולציית MD. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
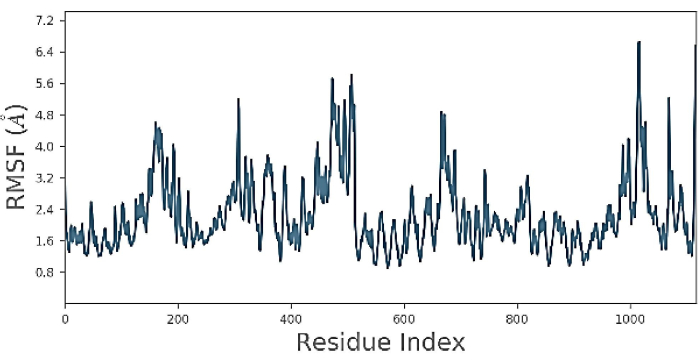
איור 5: תנודות שורש ממוצע ריבועי (RMSF). האיור מראה שהחלבון RMSF מועבר 2.4 Å משאריות בין 1-100. ואז התנועה יורדת ל -2a עבור 200-300 שאריות. יתר על כן, החלבון משתנה ל-5.6 Å עבור 500 שאריות, ואז יורד בחדות ל-2.4 Å עבור שאריות עד 1000. יש שיא חד אחד במיקום 1000 עם ירידה ל-2.4 Å בטרמינל. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 6: יסודות מבנה משני של חלבון (SSE) ואינדקס השאריות שלו. התפלגות יסודות המבנה המשני (SSE) לפי אינדקס שאריות בכל מבנה החלבון מוצגת בתרשים למעלה. התרשים התחתון עוקב אחר הקצאת ה-SSE של כל שאריות לאורך זמן, ואילו התרשים שלהלן מסכם את הרכב ה-SSE עבור כל מסגרת מסלול לאורך הסימולציה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
איור 7: סימולציות של דו-שכבות ליפידים תוך שימוש בשדה הכוח ENERGY. סימולציות של שכבות דו-שכבתיות של שומנים באמצעות שדה הכוח ENERGY. הדמיית MD משמשת לחקירת תכונות גיאומטריות על ידי הצגת מצב המצויר. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
תרשים משלים 1: הוספת אישומים של קולמן. אנא לחץ כאן להורדת קובץ זה.
תרשים משלים 2: מזעור אנרגיית מבנה pdb. אנא לחץ כאן להורדת קובץ זה.
תרשים משלים 3: הכנת קבצי קלט ב-Python. אנא לחץ כאן להורדת קובץ זה.
איור משלים 4: שלבים ליצירת חלקת רמאצ'נדרן. אנא לחץ כאן להורדת קובץ זה.
תרשים משלים 5: יצירת ליגנד. אנא לחץ כאן להורדת קובץ זה.
תרשים משלים 6: הכנת תצורת עגינה. אנא לחץ כאן להורדת קובץ זה.
תרשים משלים 7: סימולציה דינמית מולקולרית ליצירת מתחם העגינה. אנא לחץ כאן להורדת קובץ זה.
קובץ קידוד משלים 1. אנא לחץ כאן להורדת קובץ זה.
קובץ קידוד משלים 2. אנא לחץ כאן להורדת קובץ זה.
קובץ קידוד משלים 3. אנא לחץ כאן להורדת קובץ זה.
קובץ קידוד משלים 4. אנא לחץ כאן להורדת קובץ זה.
| סוג | סה"כ מספר. | ריכוז [מ"מ] | תשלום |
| קלרנית | 166 | 75.746 | -166 |
| נה | 111 | 50.65 | 111 |
טבלה 1: מידע נגד יון/מלח.
Discussion
EGFR הוא קולטן המטרה העיקרי של סרטן השד. ביטוי יתר של EGFR מגדיל את מקרי סרטן השד ברחבי העולם. בינתיים, נוגדנים ספציפיים כגון משתני שבר שרשרת בודד הם נוגדנים הנעים בקלות במחזור הדם ובעלי קצב פינוי מהיר בגוף. נוגדנים הם פתרון נבון ותרופה אימונותרפית יעילה37. לכן, תכנון תרופות מבוסס מבנה חייב לזהות תרופות מעכבות, כגון נוגדנים scFv, הפועלות באופן ספציפי נגד קולטן מטרה (EGFR). בהתבסס על תכנון תרופות בסיליקו , החוקר במחקר זה עבד על ניסויים ביציבות של נוגדן scFv-EGFR או מבנה מעוגן חלבון-חלבון שטופל כעיצוב תרופה מבוסס מבנה. שיטות סימולציה של MD שימשו במחקר זה כדי לחקור את היציבות והדיוק של המבנה המעוגן כתרופה מעכבת סרטן השד. ניתוח ניסיוני של ביולוגיה מולקולרית שהוזכר במחקרים קודמים21, הראה את יכולתם של נוגדנים scFv להיקשר עם זיקה וסגוליות רבה ל-EGFR.
מתודולוגיה זו נחשבת לגישה מוצלחת להוכחת יציבות האינטראקציה המחייבת בין הקולטן למבנה העגינה של הנוגדנים, שהוזכרה במחקרים רבים38,39. כפי שמוצג באיור 1A, D, בתוכנת מודל ההומולוגיה של המבנה, כתיבת השם המדויק של הקבצים בגיליון הפקודות היא השלב הקריטי ביותר בשיטה זו. בנוסף, הכנת קובץ 1ivo.pdb נחוצה באופן סביר כדי לשמש כקולטן ללא ליגנדים חיצוניים המזהמים את תהליך העגינה. בנוסף, איור 1A,C מציג עלילות Ramachandran לתיקון כלים וזיהוי דיוק של מבנה מודל ההומולוגיה של מודלים של מבנה הקולטן ומבנה הנוגדנים, והעלילות מציגות את מספרי השאריות בסלילי α ושאריות β יריעות בחלקת Ramachandran של עמוד השדרה החלבוני של מבנים אלה. ככל שקיימות יותר שאריות באזור סלילי α של שאריות β היריעות, כך הדיוק של המבנה גדול יותר. ישנן דרכים רבות ליצור עלילת רמאצ'נדרן. שימוש ב-www.ebi.ac.uk/thornton-srv/databases/pdbsum/ הוא אתר שמכין את העלילה ומציג את שמות התבניות כדי להציג את מיקומי השאריות.
עגינה מולקולרית היא אחת הטכניקות הנפוצות ביותר בתכנון תרופות מבוססות מבנה. אינטראקציות חלבון-חלבון דורשות סידורים מחדש קונפורמטיביים, במיוחד בממשק נוגדן-אנטיגן. שינויים כאלה עשויים להיות פשוטים כמו סיבוב שרשראות צד או עשויים לכלול שינויים מסובכים יותר, כגון ארגון מחדש מוחלט של המבנה2. לכן, המגבלות הטכניות הספציפיות של גישה זו טופלו, שכן העגינה פועלת בנקודה עיוורת של מידות תיבת הרשת, וחיזוי סוג ועוצמת האות המופק בתהליך האינטראקציה הוא תמיד קריטי. עגינה יכולה לחזות את קונפורמציה הקישור של הליגנדים כממצא חדשני. תוכנת העגינה המשמשת כאן גמישה, ומאפשרת לליגנדים וקולטנים לקיים אינטראקציה עם אפשרויות רבות בעת הכנת קופסאות רשת. כפי שמוצג באיורים 2A,B, גישת הכריכה הנמוכה ביותר נספרה, והשאריות המחוברות מוצגות באיור 2A.
בנוסף, נרשמו שמונה קשרי מימן, כפי שמוצג באיור 3B, בטווח של 70 Å. ה-RMSD שוכלל, וזיקת הקישור הגבוהה ביותר יוצרת קשר מימן חזק עם Asn70, הנחשב חיוני לפעילות ועוסק באינטראקציות נוספות משמעותיות עם שאריות קשירת קולטנים אחרות40, כפי שמוצג באיור 3A,B. גישת הניתוק של ורלט סיימה את האינטראקציות הבלתי מוגבלות, אינטראקציות קולומב (פוטנציאל אלקטרוסטטי) ולנארד ג'ונס (אטרקציות ואן דר ואלס) ב-10 Å. מכיוון שקשרי H רבים נמצאים בחיבור חלבון-ליגנד, קונפורמציות חלבון טובות יותר לחוזק האינטראקציה, כפי שהוזכר במחקרים קודמים29.
במחקר דומה 1,19, החוקרים גילו כי למרות שכל המבנים הם באיכות סבירה ובעלי שונות מבנית קלה, רוב הגישות עובדות על מבני גביש מסוימים באותה משפחת חלבונים ונכשלות באחרים. מחקר זה צפה עקביות טובה יותר על פני קונפורמציות חלבון שונות של אותו רצף. מעניין לציין כי מדדי איכות כגון רזולוציה, מדד דיוק עקיפה של Cruickshank או שאריות לא פתורות אינם יכולים לחזות במדויק את ההצלחה או הכישלון של מבנה ספציפי. בנוסף, אתרים קריפטיים נבדקו ברבים מהמחקרים הללו. למרות שהחוקר חיפש את אתר הכריכה שהורד מאתר בנק נתוני החלבון (pdb) כגישה סבירה להתייחס להשערת הפעילות האנטי-COVID-19 לכאורה, pdb הוא המקור העיקרי לאתרי הקישור שהוזכרו במחקרים רבים 1,29.
במחקר זה, סימולציית MD הסבירה את ניתוח האנרגיה הפוטנציאלית, ניתוח תנודות מבניות וניתוח יציבות קואורדינטות. התנועות המחושבות עבור המונומר של החלבון שחווה את השינוי הקונפורמטיבי הגדול ביותר בצורתו הבלתי קשורה (החופשית) תואמות את השינויים המבניים שנראו אמפירית כמתרחשים בעת קשירה41. סימולציות MD מחושבות על ידי פתרון משוואות התנועה הקלאסיות. ה-RMSD משתמש בדינמיקה מולקולרית (MD) כדי לדמות כיצד אטומים ומולקולות מתקשרים במערכת לאורך תקופה מסוימת. ה-RMSD מחושב בדרך כלל עבור המצב ההתחלתי של המבנה. הסטייה הראתה את החלקים הניידים ביותר של המבנה42. בנוסף, ה-RMSD מנתח עד כמה מבנה שונה מהתייחסות לאורך זמן. סימולציה זו זיהתה בדרך כלל את יציבות המבנה המעוגן. ה-RMSD שימש לקביעת הקונפורמציה של חוזק החלבון וערכי הסטייה שלו שהופקו במהלך הסימולציה 2,5,40,43. במספרים המתקבלים, ככל שהסטיות קטנות יותר, כך נוצר מבנה החלבון יציב יותר. כפי שמוצג באיור 4, ערך ה-RMSD עבור המבנה המעוגן נרשם כ-20 ns-75 ns כאשר תנועת המבנה הגיעה ל-7.5 Å, מה שהוכיח את יציבות המבנה44.
עם זאת, RMSF מחשב את ממוצע ה-RMSD על סמך הזמן של המבנה המדומה45. ניידות גבוהה מסומנת על ידי אזור במבנה עם ערכי RMSF גבוהים החורגים לעתים קרובות ממבנה הייחוס, כגון הקולטן לפני הגירוי. כפי שמוצג באיור 5, חלבונים לעתים קרובות נתונים רק לעמוד השדרה או לאטום הפחמן α ב-RMSF. לכן, ניתוח RMSF מציע שינויים קונפורמטיביים ומראה שרשראות צד גמישות יותר.
לבסוף, נעשה שימוש בסימולציית MD כדי לחקור תכונות גיאומטריות. על ידי הצגת מצב הקריקטורה באיור 7, הממברנה של טופולוגיית חלבון EGFR ותנועתה מנותחים באמצעות RMSD מכיוון שהיא שונה מ-20 ns-75 ns. האיור מציג את רצף החלבונים, עם מולקולות מים ושומנים מסודרים ואטומי הזרחן של השומנים בחרוזים15,46.
למרות שאינטראקציות חלבון-חלבון קשורות לתהליכים ומסלולים פיזיולוגיים חשובים, הן מהוות מטרות פוטנציאליות לגילוי תרופות. תרופות מולקולות קטנות, לעומת זאת, לא עמדו בהבטחה זו. משטחי אינטראקציה רבים של חלבון-חלבון אינם יכולים להכיל קשירה של מולקולות קטנות דמויות תרופות כתוצאה מכך. בנוסף, מחקר זה ביצע מודלים הומולוגיה, עגינה וסימולציות דינמיות מולקולריות. התוצאות מראות כי המודל המוקרן של קומפלקס קולטני נוגדנים scFv מציג איכות מעולה וחסר קונפליקטים סטריים משמעותיים. כתוצאה מכך, מודל זה עשוי לפתוח את הדלת למחקר מבני, פונקציונלי וטיפולי נוסף המתמקד במיוחד במחקרי סיליקו . בנוסף, מחקר זה יכול להוביל למחקרים עתידיים ממוקדי תרופות לקביעת ההליכים של שאריות חומצות אמינו שונות ולחזות כיצד אינטראקציות קולטן-נוגדן וקולטני התנועה שלהם משנים את התנועה שלהם, ובכך להשפיע על תכנון מבנה התרופה. לפיכך, התאמה גיאומטרית מובטחת רק לאחר הארגון מחדש המבני של החלבונים שנגרם על ידי אינטראקציית הזיהוי שלהם.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
ללא.
Materials
| Name | Company | Catalog Number | Comments |
| Autodock software | Center for Computational structural Biology | AutoDock (scripps.edu) | |
| Desmond Maestro 19.4 software | Schrodinger | www.schrodinger.com | |
| Download Discovery Studio 2021 | Dassault Systems | https://discover.3ds.com/discovery-studio-visualizer-download. | |
| Modeler Version 9.24[17] | University of California | https://salilab.org/modeller/9.24/release.html | |
| Pictorial database of 3D structures (pdbsum) | EMBL-EBI | www.ebi.ac.uk/thornton-srv/databases/pdbsum/ | |
| PyMOL software | Schrodinger | PyMOL | pymol.org | |
| Pyrx software | Sourceforge | Download PyRx - Virtual Screening Tool (sourceforge.net) | |
| Python script 3.7.9 shell from the window (64) | Python | Python Release Python 3.7.9 | Python.org | |
| SPDBV software | Expasy | http://spdbv.vital-it.ch/disclaim.html |
References
- Clark, J. J., Orban, Z. J., Carlson, H. A. Predicting binding sites from unbound versus bound protein structures. Sci Rep. 10 (1), 15856(2020).
- Huang, Y., et al. A stepwise docking molecular dynamics approach for simulating antibody recognition with substantial conformational changes. Comput Struct Biotechnol J. 20, 710-720 (2022).
- Mahgoub, I. O., Ali, A. M., Hamid, M., Alitheen, N. M. Single chain fragment variable (scFv) secondary structure prediction and evaluation. FASEB J. 25, (2011).
- Kim, H., et al. Titanium dioxide nanoparticles induce apoptosis by interfering with EGFR signaling in human breast cancer cells. Environ Res. 175, 117-123 (2019).
- Mahgoub, E. O., Abdella, G. M. Improved exosome isolation methods from non-small lung cancer cells (NC1975) and their characterization using morphological and surface protein biomarker methods. J Cancer Res Clin Oncol. 149 (10), 7505-7514 (2023).
- Shaurova, T., Zhang, L., Goodrich, D. W., Hershberger, P. A. Understanding lineage plasticity as a path to targeted therapy failure in EGFR-mutant non-small cell lung cancer. Front Genet. 11, 281(2020).
- Mahgoub, E. O., Bolad, A. K. Construction, expression and characterisation of a single chain variable fragment in the Escherichia coli periplasmic that recognise MCF-7 breast cancer cell line. J Cancer Res Ther. 10 (2), 265-273 (2014).
- Mahgoub, I. O. Design, expression and characterization of a single chain fragment variable anti-mcf-7 antibody; A humanized antibody derived from monoclonal antibody. Ann Res Conf Proc. 2014 (1), (2014).
- Sun, X. L., et al. Dimeric (-)-epigallocatechin-3-gallate inhibits the proliferation of lung cancer cells by inhibiting the EGFR signaling pathway. Chem Biol Interact. 365, 110084(2022).
- Mahgoub, E. O., Haik, Y., Qadri, S. Comparison study of exosomes molecules driven from (NCI1975) NSCLC cell culture supernatant isolation and characterization techniques. FASEB J. 33, 647(2019).
- Madeddu, C., et al. EGFR-mutated non-small cell lung cancer and resistance to immunotherapy: Role of the tumor microenvironment. Int J Mol Sci. 23 (12), 6489(2022).
- Mahgoub, I., Bolad, A. K., Mergani, M. and immune-characterization of single chain fragment variable (scFv) antibody recognize breast cancer cells line (MCF-7). J. Immunother Cancer. 2, 6(2014).
- Ladner, R. C., Sato, A. K., Gorzelany, J., de Souza, M. Phage display-derived peptides as therapeutic alternatives to antibodies. Drug Discov Today. 9 (12), 525-529 (2004).
- Khan, M. H., Manoj, K., Pramod, S. Reproductive disorders in dairy cattle under semi-intensive system of rearing in North-Eastern India. Vet World. 9 (5), 512-518 (2016).
- Al-Refaei, M. A., Makki, R. M., Ali, H. M. Structure prediction of transferrin receptor protein 1 (TfR1) by homology modelling, docking, and molecular dynamics simulation studies. Heliyon. 6 (1), 03221(2020).
- Anbuselvam, M., et al. Structure-based virtual screening, pharmacokinetic prediction, molecular dynamics studies for the identification of novel EGFR inhibitors in breast cancer. J Biomol Struct Dyn. 39 (12), 4462-4471 (2021).
- Oduselu, G. O., Ajani, O. O., Ajamma, Y. U., Brors, B., Adebiyi, E. Homology modelling and molecular docking studies of selected substituted benzo d imidazol-1-yl) methyl) benzimidamide scaffolds on Plasmodium falciparum adenylosuccinate lyase receptor. Bioinform Biol Insights. 13, 1177932219865533(2019).
- Mahgoub, E. O., et al. et al. of exosome isolation techniques in lung cancer. Mol Biol Rep. 47 (9), 7229-7251 (2020).
- Shree, P. Targeting COVID-19 (SARS-CoV-2) main protease through active phytochemicals of ayurvedic medicinal plants -Withania somnifera (Ashwagandha), Tinospora cordifolia (Giloy) and Ocimum sanctum (Tulsi) - a molecular docking study. J Biomol Struct Dyn. 40 (1), 190-203 (2022).
- Mahgoub, I. O. Expression and characterization of a functional single-chain variable fragment (scFv) protein recognizing MCF7 breast cancer cells in E. coli cytoplasm. Biochem Genet. 50 (7-8), 625-641 (2012).
- Mahgoub, E. O. Single chain fragment variables antibody binding to EGF receptor in the surface of MCF7 breast cancer cell line: Application and production review. Open J Genet. 7 (2), 84-103 (2017).
- Heng, C. K., Othman, R. Y. Bioinformatics in molecular immunology laboratories demonstrated: Modeling an anti-CMV scFv antibody. Bioinformation. 1 (4), 118-120 (2006).
- Mahgoub, E. O., Ahmed, B. Correctness and accuracy of template-based modeled single chain fragment variable (scFv) protein anti- breast cancer cell line (MCF-7). Open J Genet. 3 (3), 183-194 (2013).
- Khare, N., et al. Homology modelling, molecular docking and molecular dynamics simulation studies of CALMH1 against secondary metabolites of Bauhinia variegata to treat Alzheimer's disease. Brain Sci. 12 (6), 770(2022).
- Hu, J., Rao, L., Fan, X., Zhang, G. Identification of ligand-binding residues using protein sequence profile alignment and query-specific support vector machine model. Anal Biochem. 604, 113799(2020).
- Santos, L. H. S., Ferreira, R. S., Caffarena, E. R. Integrating molecular docking and molecular dynamics simulations. Methods Mol Biol. 2053, 13-34 (2019).
- Xu, D., Tsai, C. J., Nussinov, R. Hydrogen bonds and salt bridges across protein-protein interfaces. Protein Eng. 10 (9), 999-1012 (1997).
- Das, S., Chakrabarti, S. Classification and prediction of protein-protein interaction interface using machine learning algorithm. Sci Rep. 11 (1), 1761(2021).
- Mahmoud, S. S. A., Elkaeed, E. B., Alsfouk, A. A., Abdelhafez, E. M. N. Molecular docking and dynamic simulation revealed the potential inhibitory activity of opioid compounds targeting the main protease of SARS-CoV-2. Biomed Res Int. 2022, 1672031(2022).
- Villada, C., Ding, W., Bonk, A., Bauer, T. Simulation-assisted determination of the minimum melting temperature composition of MgCl2-KCl-NaCl salt mixture for next-generation molten salt thermal energy storage. Front. Energy Res. 10, 809663(2022).
- Hog, S. E., Rjiba, A., Jelassi, J., Dorbez-Sridi, R. NaCl salt effect on water structure: a Monte Carlo simulation study. Phys Chem Liq. 60 (5), 767-777 (2022).
- Maruyama, Y., Igarashi, R., Ushiku, Y., Mitsutake, A. Analysis of protein folding simulation with moving root mean square deviation. J Chem Inf Model. 63 (5), 1529-1541 (2023).
- Winarski, K. L., et al. Vaccine-elicited antibody that neutralizes H5N1 influenza and variants binds the receptor site and polymorphic sites. Proc Natl Acad Sci U S A. 112 (30), 9346-9351 (2015).
- Awal, M. A., et al. Structural-guided identification of small molecule inhibitor of UHRF1 methyltransferase activity. Front Genet. 13, 928884(2022).
- Sharma, P., Hu-Lieskovan, S., Wargo, J. A., Ribas, A. adaptive, and acquired resistance to cancer immunotherapy. Cell. 168 (4), 707-723 (2017).
- Geerds, C., Wohlmann, J., Haas, A., Niemann, H. H. Structure of Rhodococcus equi virulence-associated protein B (VapB) reveals an eight-stranded antiparallel β-barrel consisting of two Greek-key motifs. Acta Crystallogr F Struct Biol Commun. 70 (7), 866-871 (2014).
- Mahgoub, E., et al. The therapeutic effects of tumor treating fields on cancer and noncancerous cells. Arabian J Chem. 14 (10), 103386(2021).
- Hospital, A., Goni, J. R., Orozco, M., Gelpi, J. L. Molecular dynamics simulations: advances and applications. Adv Appl Bioinform Chem. 8, 37-47 (2015).
- Kaur, J., Tiwari, R., Kumar, A., Singh, N. Bioinformatic analysis of Leishmania donovani long-chain fatty acid-CoA ligase as a novel drug target. Mol Biol Int. 2011, 278051(2011).
- Páll, S., Hess, B. A flexible algorithm for calculating pair interactions on SIMD architectures. Comp Phy Comm. 184 (12), 2641-2650 (2013).
- Tobi, D., Bahar, I. Structural changes involved in protein binding correlate with intrinsic motions of proteins in the unbound state. Proc Natl Acad Sci U S A. 102 (52), 18908-18913 (2005).
- Sinha, S., Wang, S. M. of VUS and unclassified variants in BRCA1 BRCT repeats by molecular dynamics simulation. Comput Struct Biotechnol J. 18, 723-736 (2020).
- Patel, D., Athar, M., Jha, P. C. Exploring Ruthenium-based organometallic inhibitors against Plasmodium falciparum calcium dependent kinase 2 (PfCDPK2): A combined ensemble docking, QM/MM and molecular dynamics study. Chem Select. 6 (32), 8189-8199 (2021).
- Khan, A., et al. Higher infectivity of the SARS-CoV-2 new variants is associated with K417N/T, E484K, and N501Y mutants: an insight from structural data. J Cell Physiol. 236 (10), 7047-7057 (2021).
- Zhang, Q., Shao, d, Xu, P., Jiang, Z. Effects of an electric field on the conformational transition of the protein: Pulsed and oscillating electric fields with different frequencies. Polymers. 14 (1), 123(2021).
- Mahtarin, R., et al. et al. and dynamics of membrane protein in SARS-CoV-2. Biomol Struct Dyn. 40 (10), 4725-4738 (2022).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ISSN 2578-6326
Copyright © 2025 MyJoVE Corporation. All rights reserved
We use cookies to enhance your experience on our website.
By continuing to use our website or clicking “Continue”, you are agreeing to accept our cookies.