Method Article
בידוד ועיבוד של אדיפוציטים לבנים של עכברים לניתוח טרנסקריפטום ואפיגנום
In This Article
Summary
הפרוטוקול הנוכחי מסכם שיטה אוניברסלית לבידוד, טיהור ועיבוד במעלה הזרם של אדיפוציטים לבנים של עכברים המותאמים לריצוף RNA כולל במורד הזרם, חילוץ גרעינים על ידי SONication (NEXSON) ו-ChIP-seq.
Abstract
השמנת יתר היא מחלה מורכבת המושפעת מגנטיקה, אפיגנטיקה, הסביבה והאינטראקציות ביניהם. אדיפוציטים בוגרים מייצגים את סוג התא העיקרי ברקמת השומן הלבנה. הבנת האופן שבו אדיפוציטים מתפקדים ומגיבים לאותות (אפי) גנטיים וסביבתיים חיונית לזיהוי הגורמים להשמנה. RNA וכרומטין בודדו בעבר מאדיפוציטים באמצעות עיכול אנזימטי. בנוסף, פותחו פרוטוקולים לבידוד גרעיני, שבו הטיהור מושג על ידי מיון תאים המופעל פלואורסצנטי (FACS) של מדווחים טרנסגניים ספציפיים לאדיפוציטים. אחד האתגרים הגדולים ביותר להשגת תפוקה ואיכות גבוהים במהלך פרוטוקולים כאלה הוא הכמות המשמעותית של שומנים הכלולה ברקמת השומן. הפרוטוקול הנוכחי מתאר הליך אופטימלי לבידוד אדיפוציטים בוגרים הממנף את הפטן כדי להפריד שומנים ממטרות העניין (RNA/כרומטין). ה-RNA המתקבל הוא בעל שלמות גבוהה ומייצר תוצאות RNA-seq באיכות גבוהה. כמו כן, ההליך משפר את קצב תפוקת הגרעינים ומייצר תוצאות ChIP-seq הניתנות לשחזור על פני דגימות. לכן, המחקר הנוכחי מספק פרוטוקול בידוד אדיפוציטים עכבריים אמין ואוניברסלי המתאים למחקרי טרנסקריפטום ואפיגנום של גנום שלם.
Introduction
השמנת יתר מובנת בדרך כלל כמחלה של הצטברות עודף שומן התורמת לסיכון מוגבר לסוכרת מסוג 2, מחלות לב וכלי דם ומספר צורות של סרטן 1,2,3. בעוד שההבנה הנוכחית של השמנת יתר מושרשת מאוד בגנטיקה (הן ממחקרים בבני אדם והן ממחקרים במכרסמים), כ-30%-70% מהנטייה למחלות מטבוליות אינה גנטית במקורה 4,5,6,7,8 ונותרה לא מוגדרת כהלכה.
רקמת השומן ממלאת תפקיד קריטי בהשמנת יתר ובמחלות מטבוליות אחרות 9,10. רקמת השומן מורכבת מאדיפוציטים בוגרים ומחלק כלי הדם הסטרומלי, כולל פרה-אדיפוציטים, תאי אנדותל ותאי חיסון. עדיין לא ברור כיצד כל סוג תא תורם להשמנת יתר וכיצד חוסר ויסות אדיפוציטים תורם להשמנה. פרוטוקולי בידוד וטיהור הניתנים לשחזור ויעילים למחקרי אפיגנום אדיפוציטים בוגרים מעניינים את התחום.
אדיפוציטים בוגרים בודדו זה מכבר לניתוחי ביטוי גנים11 ומחקרי אפיגנום12,13. ישנן שתי אסטרטגיות עיקריות לבידוד אדיפוציטים. הראשון הוא להשתמש בעיכול אנזימטי כדי להפריד את השומנים הבוגרים משאר סוגי התאים במקטע כלי הדם הסטרומלי11,14. השני הוא להוציא את רקמת השומן כדי לשחרר גרעינים שלמים ולאחר מכן לשחזר את הגרעינים על סמך מדווח פלואורסצנטי על ידי מיון תאים מופעל פלואורסצנטי (FACS)12,13, הדורש מודלים מיוחדים של דיווח טרנסגני. האתגר הטכני בכל מקרה הוא שאדיפוציטים בוגרים מכילים ריכוזים גבוהים של ליפידים (איור 1), מה שמפחית את האיכות ו/או התפוקה של סך RNA15,16 וגרעינים17. כאן, מתואר הליך עיכול אנזימטי אופטימלי לבידוד אדיפוציטים בוגרים, שבו היתרון של הפטן הוא להמיס ולהסיר שומנים במהירות וביעילות18 לפני מיצוי RNA או שלבי בידוד הגרעינים על ידי מיצוי גרעינים על ידי SONication (NEXSON)19. הפרוטוקול מבטיח התאוששות ואיכות מצוינת של ה-RNA הכולל למחקרים ברחבי הגנום ומשפר משמעותית את התפוקה של גרעינים שלמים עבור ChIP-seq הניתן לשחזור.
Protocol
כל הניסויים בבעלי חיים אושרו על ידי הוועדה המוסדית לטיפול ושימוש בבעלי חיים (IACUC), מספר פרוטוקול: 18-10-028. עכברי C57BL/6J זכרים בני 12 שבועות הומתו עם CO2 ונותחו כדי לאסוף את כריות השומן מרקמת השומן הלבנה האפידידימלית (eWAT).
1. בידוד אדיפוציטים
- מכינים את מאגר העיכול (טבלה 1) ומחממים ל-37 מעלות צלזיוס באמבט מים.
- שים את כרית השומן מרקמת השומן הלבנה האפידידימלית (eWAT) בצלחת פטרי. חותכים את הרקמה לחתיכות קטנות (5 מ"מ על 5 מ"מ) בעזרת מלקחיים ואזמל ב -5 מ"ל DMEM.
הערה: עדיף צלחת פטרי שאינה מכילה ציפוי הידבקות תאים. חשוב גם להחזיק את כרית השומן בעדינות כדי למנוע נזק לתאים. ככל שמטפלים יותר ברקמת השומן, כך התשואה נפגעת. שיטת אזמל/מלקחיים עדיפה על חיתוך רפידות השומן במספריים או במלקחיים בלבד מכיוון שהם עלולים לפגוע באדיפוציטים. - מסננים את עודפי ה-DMEM מבלי להפריע לפסולת הרקמות.
הערה: אין לאסוף את פסולת הרקמות מפני השטח של צלחת הפטרי. - מוסיפים 5 מ"ל של חיץ עיכול לצלחת הפטרי, מערבבים כדי לשחרר שברי רקמות מהצלחת, ויוצקים את כל התוכן לתוך צינור צנטריפוגה של 50 מ"ל. חזור על התהליך עד שכל שברי הרקמה מועברים לצינור הצנטריפוגה.
- הוסף את מאגר העיכול הנותר לצינור הצנטריפוגה כך שהנפח הסופי יהיה ~20 מ"ל.
- דגרו את שברי הרקמה באמבט מים רועד של 37 מעלות צלזיוס ב-100 סל"ד למשך 20-30 דקות, או עד ששברי הרקמה קטנים מ-1 מ"מ על 1 מ"מ.
- הוסף 0.4 מ"ל של 0.5 מ' EDTA ו-0.2 מ"ל של 0.5 מ' EGTA לצינור הצנטריפוגה, והמשיך לרעוד ב-37 מעלות צלזיוס למשך 10 דקות.
- העבירו את תרחיף התאים עם פיפטה של 10 מ"ל דרך מסנן מטריצה עדין (420 מיקרומטר, ראה טבלת חומרים) על גבי צינור צנטריפוגה נוסף של 50 מ"ל כדי להסיר את הרקמה הלא מעוכלת.
- צנטריפוגה את צינור הצנטריפוגה של 50 מ"ל ב-200 x g למשך 5 דקות בטמפרטורת החדר כדי להפריד אדיפוציטים טהורים (צפים בחלק העליון) משבר כלי הדם הסטרומלי (גלולה בתחתית) (איור 2A).
- יוצקים את שכבת השומן הצפה לצינור חדש של 50 מ"ל, נזהרים לא לשבש את הגלולה.
הערה: הימנע משימוש בקצות פיפטה להעברת אדיפוציטים צפים מכיוון שהם יכולים להיצמד לדופן קצה הפיפטה (ומכאן, להפחית את התפוקה). חיץ עיכול כלשהו יעבור לתוך הצינור החדש; השתמש במזרק ובמחט כדי להסיר את שאריות העיכול מתחת לשכבת השומן. - השתמש באדיפוציטים המטוהרים למיצוי RNA מלא (שלב 2.) ו/או ChIP-seq (שלב 3. ושלב 4.) באופן מיידי.
הערה: המשתמש יכול להחליט להשתמש בכל תמצית השומן לשלב 2. או שלב 3., בהתאם למטרת המחקר. המשתמש יכול גם לפצל את כל תמצית השומן לשתי מנות ולהמשיך בשלב 2. ושלב 3. במקביל לקבלת תוצאות ניתוח מהדגימות הזהות מבחינה ביולוגית.
2. מיצוי RNA כולל
הערה: בצע שלב זה במכסה מנוע כימי.
- הוסף מגיב בידוד RNA (1 מ"ל) (ראה טבלת חומרים) לאדיפוציטים המטוהרים בצינור הצנטריפוגה של 50 מ"ל (שלב 1.11.).
- פיפטה למעלה ולמטה באמצעות קצה פיפטה של 1 מ"ל כדי להמיס היטב את האדיפוציטים במגיב בידוד ה-RNA ולהעביר לצינור מיקרו-צנטריפוגה של 2 מ"ל.
- דגרו את הצינור למשך 15 דקות בטמפרטורת החדר.
- הוסף 0.5 מ"ל של הפטן לתוך צינור הצנטריפוגה והמערבולת במלוא המהירות למשך 30 שניות.
הערה: השומנים יתמוססו בהפטן (איור 2B). - צנטריפוגה את הצינור ב-1,000 x גרם למשך 10 דקות ב-4 מעלות צלזיוס.
- רכשו את השכבה התחתונה של ריאגנט בידוד RNA עם מזרק ומחט 30 G, והעבירו לצינור חדש של 1.5 מ"ל.
- הוסף 0.1 מ"ל של 1bromo-3chloropropane (ראה טבלת חומרים) לתמצית ריאגנט בידוד RNA ומערבולת במהירות מלאה למשך 30 שניות. דגרו את הצינור בטמפרטורת החדר למשך 15 דקות.
- צנטריפוגה את הצינור ב-12,000 x גרם למשך 15 דקות ב-4 מעלות צלזיוס. השתמש בפיפטה כדי לאסוף את השלב המימי העליון ולהעביר אותו לצינור חדש.
- מוסיפים 0.5 מ"ל איזופרופנול ודוגרים למשך 10 דקות בטמפרטורה של 4 מעלות צלזיוס. צנטריפוגה את הצינור ב-12,000 x גרם למשך 10 דקות ב-4 מעלות צלזיוס.
הערה: ה-RNA ישקע ככדור בתחתית הצינור. - השתמש בפיפטה כדי להסיר את הנוזל העליון, היזהר להימנע מלגעת בגלולה.
- הוסף 1 מ"ל של 75% EtOH ומערבולת במהירות מלאה לזמן קצר.
- צנטריפוגה את הצינור ב-7,500 x גרם למשך 5 דקות ב-4 מעלות צלזיוס. הסר בזהירות את הסופרנטנט בעזרת קצה פיפטה. אל תפריע לגלולה.
- יבש את הגלולה באוויר בטמפרטורת החדר למשך 10 דקות או עד שהגלולה הופכת צלולה/שקופה.
- ממיסים את הגלולה ב-50 מיקרוליטר של מים נטולי נוקלאז.
3. קיבוע אדיפוציטים עבור ChIP-seq
הערה: בצע את שלב 3. במכסה מנוע כימי.
- הוסף 0.3 מ"ל של 0.5 מ"ל EDTA, 0.2 מ"ל של 0.5 מ"ל EGTA, 14.8 מ"ל של DMEM ו-10 מ"ל של הפטן לתוך צינור 50 מ"ל המכיל אדיפוציטים מבודדים (שלב 1.11).
- הוסף 0.7 מ"ל של 16% פורמלדהיד (0.7% סופי) וסובב על מסובב צינור (ראה טבלת חומרים) ב-10 סל"ד למשך 5 דקות בטמפרטורת החדר.
הערה: הגדר טיימר וספור במשך 5 דקות. אם מעבדים מספר דגימות, הוסף פורמלדהיד ברצף במרווחים של 30 שניות כדי לאפשר תזמון מדויק ובר השוואה של קיבוע בכל הדגימות. - הוסף 1.78 מ"ל של 1.25 מ' גליצין (0.125 מ' סופי) וערבב על המסובב ב-10 סל"ד למשך לא יותר מ-5 דקות בטמפרטורת החדר.
הערה: פורמלדהיד נשאר פעיל גם לאחר הוספת גליצין. לכן, חשוב להפריד את השכבות על ידי צנטריפוגה (שלב 3.4.) מיד לאחר שלב הדגירה של 5 דקות. אם יש יותר מדגימה אחת, הוסף גליצין לכל דגימה כדי לעצור את התגובה ברצף במרווחי זמן של 30 שניות. - לאחר מכן, צנטריפוגה ב -200 x גרם למשך 5 דקות בטמפרטורת החדר.
הערה: חייבות להיות שלוש שכבות נוזל ברורות ובדידות (איור 2C). השכבה הלבנה האמצעית מכילה את האדיפוציטים הקבועים. - השתמש בקצה פיפטה של 1 מ"ל כדי להעביר את שכבת השומן (לבן) לצינור צנטריפוגה טרי של 15 מ"ל, תוך מזעור כמות הקיבוע וההפטן המועברים לצינור החדש.
- מלאו את הצינור ב-10 מ"ל (טמפרטורת החדר) DMEM בתוספת טבלית קוקטייל מעכב פרוטאז (PIC, ראה טבלת חומרים). מערבבים היטב על ידי היפוך.
- צנטריפוגה את הצינור בטמפרטורה של ≤200 x גרם למשך 5 דקות בטמפרטורת החדר. שמור על כוח הצנטריפוגה על 100 x g כדי למזער את התפוצצות התאים.
- חזור על שלבים 3.5.-3.7. 1x לפני שעוברים לשלב 3.9.
- השתמש בקצה פיפטה של 1 מ"ל כדי להעביר את שכבת השומן (לבן) לצינור מיקרו-צנטריפוגה של 1.5 מ"ל או 2 מ"ל.
- צנטריפוגה את הצינור ב-100-200 x גרם למשך 5 דקות בטמפרטורת החדר, והשתמש בקצה פיפטה של 1 מ"ל כדי להסיר את הפטן (השכבה העליונה) ואת ה-DMEM (השכבה התחתונה).
- אחסן את השומנים הקבועים בטמפרטורה של -80 מעלות צלזיוס עד 6 חודשים עד לשימוש.
4. מיצוי גרעינים וגזירת כרומטין
הערה: הליך זה מותאם מ-Arrigoni et al.19.
- הפעל את הסוניקטור (ראה טבלת חומרים) והגדר את שיא ההספק ל-75 וואט, מקדם החובה ל-2%, ו-200 מחזורים/פרץ, כאשר מצנן אמבט המים מוגדר ל-20 מעלות צלזיוס.
- הוסף קוקטייל מעכב פרוטאז (PIC) למאגר מעבדת פרנהאם ולמאגר הגזירה (טבלה 1).
הערה: אנא בדוק עוד כמה סוניקטורים ופרמטרים מוצעים ב- Arrigoni et al.19. - השעו מחדש את האדיפוציטים הקבועים (משלב 3.) עם 750-800 מיקרוליטר של מאגר מעבדת פרנהאם קר כקרח בצינור סוניקציה של 1 מ"ל (ראה טבלת חומרים).
הערה: מומלץ לא למלא את הצינור במלוא קיבולתו. אחרת, האדיפוציטים יצופו לחלק העליון ביותר של הצינור, מה שמפחית את יעילות הסוניקציה. - סוניקציה של הדגימה למשך 2.5 דקות.
הערה: בדוק את מיצוי הגרעינים במיקרוסקופ ניגודיות פאזה כדי לקבוע אם יש צורך בסוניקציה נוספת. הגרעינים חייבים להיות עגולים ושלמים, כפי שמוצג באיור 2D. - לפני שתמשיך הלאה, הגדר את הספק השיא של הסוניקטור ל-140 וואט, מקדם החובה ל-5%, ו-200 מחזורים/פרץ, כאשר מצנן אמבט המים מוגדר ל-4 מעלות צלזיוס.
- השתמש בפיפטה כדי להעביר את הסופרנטנט לצינור מיקרו-צנטריפוגה של 1.5 מ"ל. צנטריפוגה את הצינור ב-1,000 x גרם למשך 5 דקות ב-4 מעלות צלזיוס.
- הסר בזהירות את הסופרנטנט מהגלולה (גרעינים) ושמור את הגלולה.
- שטפו את הגלולה עם 1 מ"ל של מאגר מעבדת פרנהאם על קרח.
- צנטריפוגה את הצינור ב-1,000 x גרם למשך 5 דקות ב-4 מעלות צלזיוס, השתמש בפיפטה כדי להסיר את הסופרנטנט ושמור את הגלולה.
- חזור על שלבים 4.8.-4.9. פי 1 לפני המעבר לשלב 4.11.
הערה: ניתן לאחסן את הגרעינים המבודדים והשטופים בטמפרטורה של -80 מעלות צלזיוס למשך 3 חודשים עד לשימוש נוסף. - השעו מחדש את הגרעינים המבודדים ב-1 מ"ל של מאגר גזירה, והעבירו אותם לצינור סוניקציה חדש של 1 מ"ל. ודא שאין בועות בצינור.
- סוניקציה של הגרעינים בסוניקטור בהספק שיא של 140 וואט, מקדם חובה של 5%, ו-200 מחזורים/פרץ למשך 12 דקות כדי לגזור את הכרומטין ב-4 מעלות צלזיוס.
- העבירו את הליזאט לצינור מיקרופוגה של 1.5 מ"ל.
הערה: הכרומטין הגזוז משתחרר מהגרעינים וכלול במאגר (ליזאט). - צנטריפוגה את הצינור ב-10,000 x גרם למשך 5 דקות ב-4 מעלות צלזיוס כדי לגלול פסולת בלתי מסיסה. העבירו את הסופרנטנט (כרומטין) לצינור מיקרו-צנטריפוגה חדש של 1.5 מ"ל.
- ניתן לאחסן את הכרומטין הגזוז בטמפרטורה של 4 מעלות צלזיוס למשך 10 ימים או בטמפרטורה של -80 מעלות צלזיוס למשך 3 חודשים עד לשימוש נוסף.
הערה: בדיקת איכות נוספת: יש לנקוט 25 מיקרוליטר של הכרומטין הגזוז כדי לנתק ולהסיר את ה-RNA עם RNase נטול DNase בעקבות הדוח שפורסם בעבר19. במחקר הנוכחי, ה-DNA שהתקבל טוהר עם ערכת טיהור DNA בהתאם להוראות היצרן, ה-DNA כומת עם ערכת כימות DNA במכשיר כימות ה-DNA, וגודל השבר הוערך על ידי מכשיר האלקטרופורזה האוטומטי (ראה טבלת חומרים). גודל הכרומטין הגזוז צריך להיות בטווח של 100-800 bp. שאר כרומטין הגזירה יכול להמשיך ליישום ChIP-Seq במורד הזרם.
תוצאות
אדיפוציטים בודדו משש כריות שומן, מיצוי RNA מבוסס הפטן בוצע (שלב 2.), וה-RNA שהתקבל נותח במכשיר האלקטרופורזה האוטומטי. מספר שלמות ה-RNA המחושב (RIN) עבור כל הדגימות היה >8 (איור 3A), מה שמצביע על הכנת RNA איכותית וניתנת לשחזור. ערכת הכנת ה-RNA הכוללת שימשה לאחר מכן להכנת ספריות RNA, וכל דגימה רצפה על הרצף של הדור הבא כדי להגיע לעומק קריאה של ≥40 מיליון קריאות. ציון ה-Phred עבור כל הדגימות (איור 3B) ולכל רצף (איור 3C) היה ≥30, מה שמעיד על רצפי DNA באיכות גבוהה20. לפיכך, הסרת הפטן תומכת בבידוד של RNA איכותי בעל תפוקה גבוהה המתאים לריצוף RNA.
בשלב קיבוע הפורמלדהיד בוצעו גם שלושה תכשירי בידוד גרעיני המכילים הפטן ותכשיר בקרה אחד ללא הפטן. לאחר מכן נותח הכרומטין הגזוז במכשיר האלקטרופורזה האוטומטי. בשני המקרים, הכרומטין נחתך לטווח גודל של 100-800 bp (איור 4A), אידיאלי להליכי ChIP-seq במורד הזרם19. חשוב לציין, ~פי 5 יותר כרומטין התקבל מהדגימות שטופלו בהפטן (11.5 ננוגרם/מיקרוליטר, 9.42 ננוגרם/מיקרוליטר ו-9.6 ננוגרם/מיקרוליטר) ביחס לדגימות הלא מטופלות (2.2 ננוגרם/מיקרוליטר). H3K4me3 ו-H3K27me3 ChIP-seq בוצעו בעקבות Arrigoni et al.19. כפי שמוצג באיור 4B, מסלולי האותות משלוש דגימות עצמאיות שטופלו בהפטן היו דומים למסלול של הדגימה הלא מטופלת בהפתן עבור שני סימני ההיסטון. הטיפול בהפטן אינו מפריע לאיכות ה-ChIP-seq אך משפר משמעותית את תפוקת הגרעינים (והכרומטין הגזוז).

איור 1: אדיפוציטים מבודדים. אדיפוציטים מבודדים נצבעו ב-DAPI (כחול) כדי להכתים את הגרעין וב-BODIPY (ירוק) עבור השומנים. טיפות השומן הציטוזוליות מהוות עד 95% מנפח האדיפוציטים ומהוות אתגר טכני להשגת תפוקות גבוהות במהלך מיצוי RNA וכרומטין סרגל קנה מידה = 100 מיקרומטר. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
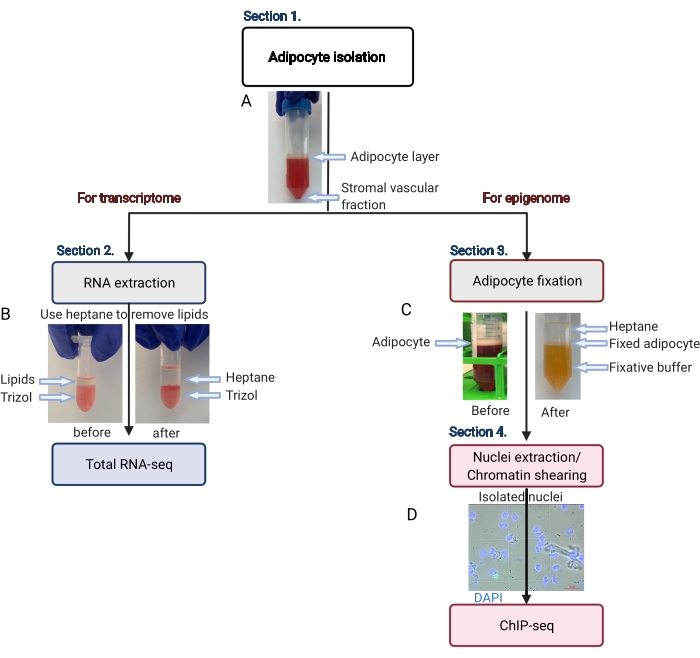
איור 2: זרימה סכמטית של בידוד השומן לניתוח טרנסקריפטום ואפיגנום. כל זרימת העבודה מתוארת, מבידוד אדיפוציטים ועד יישום טרנסקריפטום או אפיגנום. השלבים העיקריים והתוצאות המייצגות מוצגים. (A) האדיפוציטים המבודדים צפים על השכבה העליונה, ונפרדים מחלק כלי הדם הסטרומלי ככדור בתחתית הצינור. (B) השימוש בהפטן להסרת שומנים לפני מיצוי RNA עם ריאגנט בידוד RNA. (C) השימוש בהפטן להסרת שומנים במהלך קיבוע אדיפוציטים. (D) התמונה המייצגת של גרעיני שומן מבודדים חייבת להיות שלמה ועגולה. סרגל קנה מידה = 20 מיקרומטר. התוכנית נוצרה עם BioRender.com. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
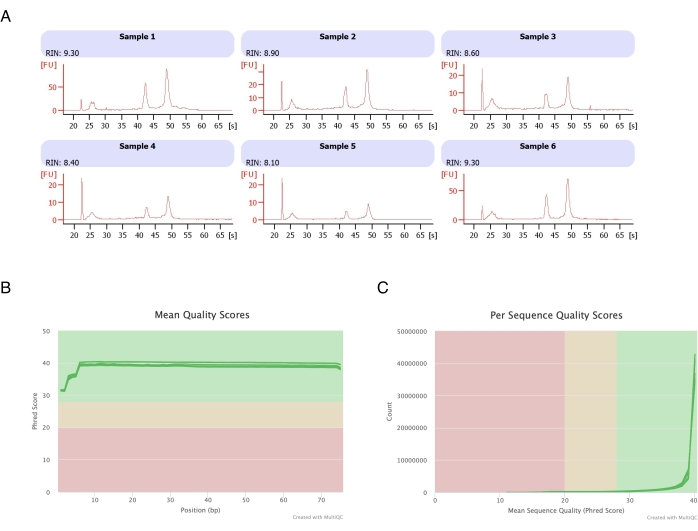
איור 3: אלקטרופרוגרמה מייצגת של שלמות RNA וציוני איכות לאחר רצף RNA. (A) דגימות 1-6 מייצגות שישה שכפולים של אדיפוציטים שטופלו בהפטן, וניתוח שלמות ה-RNA שלהם הופעל על מכשיר האלקטרופורזה האוטומטי. מספר שלמות ה-RNA (RIN) התבסס על יחסי RNA ריבוזומליים 18S ו-28S ומייצג את איכות ה-RNA. קנה מידה מ-1 (מושפל בהרבה) ל-10 (הכי פחות מושפל). (ב,ג) ציון איכות קריאת הרצף נקבע על ידי ניתוח multiQC (B) עבור ציון האיכות הממוצע בבסיסים (C) ועבור ציון האיכות לכל רצף. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
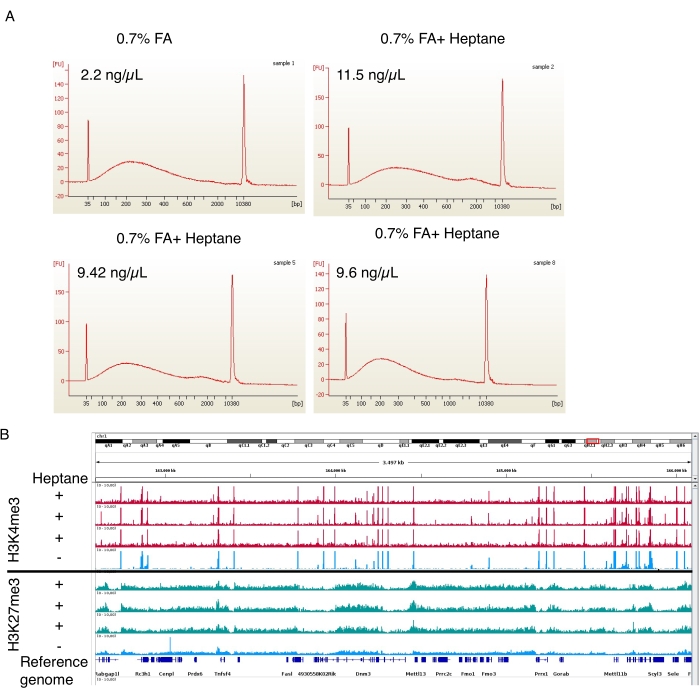
איור 4: אלקטרופרוגרמה מייצגת של כרומטין גזוז ופסגות העשרה מ-ChIP-seq. (A) פרופילים מייצגים ממכשיר האלקטרופורזה האוטומטי המציגים את התפלגות גודל הכרומטין של הבקרה (מדגם 1, משמאל למעלה) ותכשירי כרומטין אדיפוציטים שטופלו בהפתן (דגימות 2, 5 ו-8, מימין למעלה ושני תחתונים). ריכוזי הכרומטין בתכשירים הסופיים מצוינים בפינה השמאלית העליונה של כל תמונה. (B) צילום מסך של דפדפן הגנום שנעשה באמצעות Integrative Genomics Viewer (IGV). החלק העליון של התרשים מציג פרופילי H3K4me3 ChIP-seq עבור שלוש דגימות (אדומות) שטופלו בהפטן ובקרה אחת (כחולה). הפאנל התחתון מראה את אותו הדבר עבור ChIP-seq של H3K27me3. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
| מאגר | הרכב |
| מאגר עיכול (עבור 3000 מ"ג או פחות של כרית שומן / 20 מ"ל) | 20 מ"ל של מדיום הנשר המותאם (DMEM) של Dulbecco |
| 0.3 גרם BSA ללא חומצות שומן | |
| 0.1 גרם קולגנאז סוג 2 | |
| מאגר מעבדת פרנהאם | צינורות 5 מ"מ (pH 8) |
| 85 מ"מ KCl | |
| 0.5% איג'פל | |
| מאגר גזירה | 10 מ"מ Tris-HCl pH 8 |
| 0.1% SDS | |
| 1 מ"מ EDTA |
טבלה 1: הרכב המאגרים השונים ששימשו במחקר הנוכחי.
Discussion
פרוטוקול בידוד השומן המוצג כאן מבוסס על שיטות עיכול אנזימטיות מקובלות11,14 להפרדת אדיפוציטים בוגרים (צפים) מחלק כלי הדם הסטרומלי הנותר של רקמת השומן הלבנה. הוא מספק גישה פשוטה ואוניברסלית לטיהור אדיפוציטים בוגרים בכל דגם עכבר. כפי שהודגם לעיל, הפרוטוקול מתאים לשימוש במורד הזרם עבור ניתוחי טרנסקריפטום שלם ו-ChIP-seq. הוא מספק תפוקה מספקת ליצירת פרופילים אפיגנומיים מרובים (למשל, מספר שינויים בהיסטון בתוספת RNA-seq) מאותה כרית שומן בודדת.
כדי להבטיח את התשואות הגבוהות המאפשרות הכנת נתוני אפיגנום תואמים כאלה, שלב קריטי הוא שימוש בהפטן כדי להמיס שומנים הרחק מאדיפוציטים שלמים ומאדיפוציטים שמתמזגים במהלך התהליך. מניסיוננו, שלב זה מגדיל באופן משמעותי את יכולת השחזור והתפוקה על ידי מניעת אובדן RNA וגרעינים לשכבת השומנים. סיבוב רציף של הצינורות המכילים אדיפוציטים העוברים קיבוע חשוב לא פחות מכיוון שהוא משפר את ההומוגניות שבה מוציאים את השומנים מהאדיפוציטים. במקום המאגר המקבע של פורמלדהיד 1% שדווח בחלק גדול מהספרות של ChIP-seq 21,22, נמצא כי הפחתת הריכוז ל-0.7% פורמלדהיד נחוצה כדי למנוע קיבוע יתר. זמן גזירת הכרומטין עבר אופטימיזציה גם ל-12 דקות כדי להשיג טווח גודל כרומטין אופטימלי (100-800 bp) עבור ChIP-seq.
הפרוטוקול עבר אופטימיזציה למחקרי טרנסקריפטום ואפיגנום בתפזורת. לפרוטוקול הנוכחי עדיין יש מגבלות משלו עבור יישומי טרנסקריפטום ואפיגנום של תא בודד. בהתחשב בהטרוגניות התאים המדווחת בשדה השומן23,24, ניתן לפתח שיטת בידוד זו כדי להתאים אותה לבדיקות של תא בודד. בהקשרים כאלה, סמנים מוגבלים לאדיפוציטים כגון בורון-דיפירומתן (BODIPY)25 ופריליפין-1 (PLIN1)26, ASC-127, או סמני גרעינים ספציפיים לאדיפוציטים יכולים להיות בעלי ערך במתן אפשרות לכימות של פרמטרים תאיים נוספים, פונקציונליים ורלוונטיים28. מלבד היישום למחקרים גנומיים, פרוטוקול זה עשוי גם לשפר את המחקרים הפרוטאומיים של אדיפוציטים, שיש להם מכשול נוסף בשל תכולת השומנים הגבוהה באדיפוציטים29.
ניתן להשתמש בפרוטוקול זה גם כדי לחלץ בהצלחה גרעיני אדיפוציטים אנושיים (נתונים לא מוצגים), ולהרחיב את יישומו למחקר על השמנת יתר אנושית.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
אנו אסירי תודה להדמיה האופטית של MPI-IE, לריצוף, ליבה ביואינפורמטית וכוח אדם. עבודה זו נתמכה על ידי מימון מ-MPG, מכון המחקר ואן אנדל, מחקר Horizon 2020 של האיחוד האירופי, NIH (R01HG012444 ו-R21HG011964), הסכם מענק מארי סקלודובסקה-קירי מס' 675610, והמשרד הפדרלי לחינוך ומחקר במסגרת פרויקט מספר 01KU1216 (Deutsches Epigenom Programm, DEEP).
Materials
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde Solution (w/v). Methanol-free | ThermoFisher SCIENTIFIC | 28908 | |
| 1-bromo-3-chloropropane | SIGMA | B9673 | |
| 5 M NaCl | invitrogen | AM9759 | |
| Automated electrophoresis instrument (2100 Bioanalyzer Instrument) | Agilent Technologies | G2939BA | |
| BODIPY | ThermoFisher SCIENTIFIC | D3922 | |
| Collagenase Type 2 | Worthington Biochemical Corp. | 43D14184B | |
| Complete, EDTA-free, Protease inhibitor cocktail (PIC) | Roche | 4693159001 | EDTA free |
| DAPI | ThermoFisher SCIENTIFIC | D1306 | |
| DMEM (+ GlutaMAX) | life technologies | 61965-026 | |
| DNA purification kit (PCR purification kit) | Qiagen | 28104 | |
| DNA quantification instrument (Qubit 4 Fluorometer) | Fisher SCIENTIFIC | Q33238 | |
| DNA quantification kit (DNA high sensitivity assay) | Agilent Technologies | 5067-4626 | |
| EDTA | Fisher chimical | E478-500 | |
| EGTA | Fisher chimical | O2783-100 | |
| EtOH | PHARMCO | 111000200 | |
| Fatty acid-free BSA | SERVA | 11932.01 | bovine albumin fraction V, fatty acid-free lyophilized |
| Glycine | SIGMA | G7126-100G | |
| Heptane | SIGMA | H2198-1L | |
| High Sensitivity RNA Analysis | Agilent Technologies | 5067-1513 | |
| Igepal | SIGMA | 18896-50ML | |
| KCl | SIGMA | P3911-25G | |
| Matrix filter (420um) | Tisch Scientific | ME17238 | |
| Next-Generation Sequencer (HiSeq 3000 Sequencer) | Illumina | ||
| Nuclease-free water | Invitrogen | 4387936 | |
| PIPES | ACROS organics | 5625-37-6 | |
| Proteinase K | ThermoFisher SCIENTIFIC | EO0491 | |
| RNA isolation reagent (Trizol) | ThermoFisher SCIENTIFIC | 15596026 | |
| RNase A, DNase-free | ThermoFisher SCIENTIFIC | EN0531 | |
| Rotator (Thermo Scientific Tube Revolver Rotator) | ThermoFisher SCIENTIFIC | 88881001 | |
| SDS, Sodium dodecyl sulfate | SIGMA | 8170341000 | |
| Sonication tube (1 mL milliTUBE with AFA Fiber) | Covaris | 520135 | |
| Sonicator (Evolution Focused-ultra-sonicator (S220 or E220)) | Covaris | 500217 or 500239 | |
| Total RNA Prep kit ( Illumina Stranded Total RNA Prep kit) | Illumina | 20040525 | |
| Tris-HCl | Fisher bioreagents | BP153-500 |
References
- Albuquerque, D., Stice, E., Rodríguez-López, R., Manco, L., Nóbrega, C. Current review of genetics of human obesity: From molecular mechanisms to an evolutionary perspective. Molecular Genetics and Genomics. 290 (4), 1191-1221 (2015).
- Weihrauch-Blüher, S., Schwarz, P., Klusmann, J. H. Childhood obesity: Increased risk for cardiometabolic disease and cancer in adulthood. Metabolism. 92, 147-152 (2019).
- Weihrauch-Blüher, S., Wiegand, S. Risk factors and implications of childhood obesity. Current Obesity Reports. 7 (4), 254-259 (2018).
- Gluckman, P. D. Epigenetics, the life-course and metabolic disease. Nature Reviews Endocrinology. 8 (2), 74-76 (2012).
- Loh, M., Zhou, L., Ng, H. K., Chambers, J. C. Epigenetic disturbances in obesity and diabetes: Epidemiological and functional insights. Molecular Metabolism. 27, 33-41 (2019).
- Sayols-Baixeras, S., et al. DNA methylation and obesity traits: An epigenome-wide association study. The REGICOR study. Epigenetics. 12 (10), 909-916 (2017).
- Xia, Q., Grant, S. F. A. The genetics of human obesity. Annals of the New York Academy of Sciences. 1281 (1), 178-190 (2013).
- Yokoi, N. Epigenetic dysregulation in pancreatic islets and pathogenesis of type 2 diabetes. Journal of Diabetes Investigation. 9 (3), 475-477 (2018).
- Frayn, K. N. Obesity and metabolic disease: Is adipose tissue the culprit. Proceedings of the Nutrition Society. 64 (1), 7-13 (2005).
- Kawai, T., Autieri, M. V., Scalia, R. Adipose tissue inflammation and metabolic dysfunction in obesity. American Journal of Physiology-Cell Physiology. 320 (3), 375-391 (2021).
- Cat, A. N. D., Briones, A. M., Touyz, R. M., Schiffrin, E. L. . Hypertension: Methods and Protocols. , 283-295 (2017).
- Ambati, S., et al. Adipocyte nuclei captured from VAT and SAT. BMC Obesity. 3, 35 (2016).
- Roh, H. C., et al. Simultaneous transcriptional and epigenomic profiling from specific cell types within heterogeneous tissues in vivo. Cell Reports. 18 (4), 1048-1061 (2017).
- Jacobsen, M. J., et al. Epigenetic and transcriptomic characterization of pure adipocyte fractions from obese pigs identifies candidate pathways controlling metabolism. Frontiers in Genetics. 10, 1268 (2019).
- Janke, J., Engeli, S., Gorzelniak, K., Sharma, A. M. Extraction of total RNA from adipocytes. Hormone and Metabolic Research. 33 (4), 213-215 (2001).
- Cirera, S. Highly efficient method for isolation of total RNA from adipose tissue. BMC Research Notes. 6 (1), 472 (2013).
- Rajbhandari, P., et al. Single cell analysis reveals immune cell-adipocyte crosstalk regulating the transcription of thermogenic adipocytes. eLife. 8, 49501 (2019).
- Buddrick, O., Jones, O. A. H., Morrison, P. D., Small, D. M. Heptane as a less toxic option than hexane for the separation of vitamin E from food products using normal phase HPLC. RSC Advances. 3 (46), 24063-24068 (2013).
- Arrigoni, L., et al. Standardizing chromatin research: A simple and universal method for ChIP-seq. Nucleic Acids Research. 44 (7), 67 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Schmidt, D., et al. ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods. 48 (3), 240-248 (2009).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: A tool for the study of chromatin complexes. Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
- Rondini, E. A., Granneman, J. G. Single cell approaches to address adipose tissue stromal cell heterogeneity. Biochemical Journal. 477 (3), 583-600 (2020).
- Corvera, S. Cellular heterogeneity in adipose tissues. Annu Review of Physiology. 83, 257-278 (2021).
- Katz, L. S., Geras-Raaka, E., Gershengorn, M. C. Heritability of fat accumulation in white adipocytes. American Journal of Physiology-Endocrinology and Metabolism. 307 (3), 335-344 (2014).
- Hansen, J. S., de Maré, S., Jones, H. A., Göransson, O., Lindkvist-Petersson, K. Visualization of lipid directed dynamics of perilipin 1 in human primary adipocytes. Scientific Reports. 7 (1), 15011 (2017).
- Ussar, S., et al. ASC-1, PAT2, and P2RX5 are cell surface markers for white, beige, and brown adipocytes. Science Translational Medicine. 6 (247), (2014).
- Onogi, Y., Khalil, A. E. M. M., Ussar, S. Identification and characterization of adipose surface epitopes. Biochemical Journal. 477 (13), 2509-2541 (2020).
- Kim, E. Y., et al. Recent advances in proteomic studies of adipose tissues and adipocytes. International Journal of Molecular Sciences. 16 (3), 4581-4599 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ISSN 2578-6326
Copyright © 2025 MyJoVE Corporation. All rights reserved
We use cookies to enhance your experience on our website.
By continuing to use our website or clicking “Continue”, you are agreeing to accept our cookies.
