Method Article
יעיל SARS-CoV-2 כמותי הפוך Transcriptase PCR רוק אסטרטגיה אבחון באמצעות רובוטים צנרת קוד פתוח
* These authors contributed equally
In This Article
Summary
הפרוטוקול מתאר שיטת אבחון SARS-CoV-2 המשתמשת באוטומציה בקוד פתוח כדי לבצע בדיקות מולקולריות RT-qPCR של דגימות רוק. גישה מדרגית זו יכולה להיות מיושמת מעקב קליני בריאות הציבור, כמו גם כדי להגדיל את הקיבולת של מעבדות אוניברסיטאיות קטנות יותר.
Abstract
הופעתו של משבר הבריאות העולמי SARS-CoV-2 לאחרונה הציבה אתגרים מרכזיים למחקר אפידמיולוגי ובדיקות קליניות. מגפת COVID-19, המאופיינת בשיעור הדבקה גבוה ובתמותה נמוכה, דרשה בדיקות אבחון מדויקות ויעילות, במיוחד באוכלוסיות סגורות כגון אוניברסיטאות מגורים. הזמינות הראשונית של בדיקות חומצת גרעין, כמו דגימות האף, הוגבלה עקב לחץ שרשרת האספקה שגם עיכב את הדיווח על תוצאות הבדיקה. בדיקת תגובת שרשרת פולימראז כמותית (RT-qPCR) המבוססת על רוק, הוכיחה כי היא דומה ברגישות ובספציפיות לשיטות בדיקה אחרות, ואיסוף הרוק פחות פולשני פיזית למשתתפים. כתוצאה מכך, פיתחנו בדיקת אבחון מולטיפלקס RT-qPCR למעקב אוכלוסין של אוניברסיטת קלמסון והקהילה שמסביב. הבדיקה השתמשה ברובוטים ובתרמוציקלים לטיפול בנוזל בקוד פתוח במקום במערכות אוטומציה קלינית מורכבות כדי לייעל את זרימת העבודה ואת גמישות המערכת. אוטומציה של RT-qPCR מבוסס רוק מאפשרת זיהוי מהיר ומדויק של מגוון רחב של ריכוזי RNA נגיפיים הן עבור דרישות בדיקה בקנה מידה גדול והן עבור דרישות בדיקה בקנה מידה קטן. התפנית הממוצעת עבור המערכת האוטומטית הייתה < 9 שעות עבור 95% מהדגימות < 24 שעות עבור 99% מהדגימות. העלות עבור בדיקה אחת הייתה $2.80 כאשר כל הריאגנטים נרכשו בכמויות גדולות.
Introduction
נגיף נשימה חריף חמור הקשור לקורונה-2 (SARS-CoV-2), נגיף קורונה חדש, הופיע בסוף 2019 והתפשט במהירות באוכלוסיות העולם1. זיהום SARS-CoV-2 גורם למחלת קורונה 2019 (COVID-19), מחלה מדבקת מאוד עם תסמינים נשימתיים ודלקתיים חמורים. יכולת קבילות גבוהה בשילוב עם תמותה נמוכה הצביעו על כך שהנגיף יתפשט במהירות דרך אוכלוסיות וידרוש בדיקות אבחון מוגברות 2,3. המלצות בריאות הציבור עודדו סינון רחב היקף של אוכלוסיות כדי לבודד מקרים ולאחר מכן להפחית את שיעורי ההדבקה4,5,6. יתר על כן, מודלים של מעקב אוכלוסין גילו כי הגדלת תדירות הבדיקות והפחתת זמן הדיווח השפיעו יותר על הפחתת השידור מאשר הגברת הרגישות לבדיקה7. הסיבה לכך היא ככל הנראה כי אנשים נגועים יכולים להיות בהסגר מוקדם יותר, ובכך לשבור את שרשראות ההדבקה.
תקן ההגברה המקורי של חומצת הגרעין (NAAT) היה מטליות האף (NP) שעובדו על ידי RT-qPCR8. עם זאת, סיבוכים מתעוררים עם צורה זו של בדיקות עבור אוכלוסיות גדולות מאוד, כגון עלות יחסית מוגברת ולחץ שרשרת אספקה מוגברת9,10. יתר על כן, הן איסוף ועיבוד דגימות של שיטות NAAT נפוצות (כולל מטליות NP, דגימות oropharyngeal, מטליות טורבינות באמצע, ודגימות האף) מסתמכים על ציוד מיוחד, ריאגנטים, ואנשי צוות רפואי9,10.
תחליף הולם לבדיקות NP RT-qPCR ספוגיות הוא בדיקות מבוססות רוק, המהווה כלי אבחון מדויק לזיהוי SARS-CoV-211,12,13,14. ביצוע ישיר של RT-qPCR על דגימות רוק מניב רגישות וספציפיות דומות כמו מטליות NP15. אחד היתרונות העיקריים של בדיקות רוק יש על בדיקות ספוגית NP הוא שזה מאפשר איסוף עצמי של דגימות16. זה מצמצם את הצורך בצוותים רפואיים וממקסם את הקלות של איסוף מדגם עבור חולים על ידי היותו פחות פולשני מאשר דגימות NP. בנוסף, מאז דגימות רוק אינם דורשים חוצצים כדי להסיר את המדגם מספוגית (כמו במקרה של דגימות NP), בדיקות מבוססות רוק יכול לנצל חומצה ריבונוקלאית מבוססת חום (RNA) מיצוי ישירות, אשר מקטין את עלויות הבדיקה על ידי הסרת הצורך במאגרים נוספים, מדיה הובלה, ו / או ריאגנטים חילוץ RNA14,17.
המעבדה למחקר וחינוך באוניברסיטת קלמסון לאבחון מחלות והתערבות (REDDI) הוקמה כדי לענות על צרכי האוניברסיטה לבדיקות ומעקב של COVID-19. באוכלוסיות סגורות, כולל אוניברסיטאות, בדיקות מעקב תכופות בשילוב עם ריחוק חברתי הניבו את התוצאות החיוביות ביותר במודלים האפידמיולוגיים של שכיחות המחלה18. הפרוטוקולים המאוחדים של CDC 2019-nCOV RT-qPCR19 ו- RockivaDirect14 הותאמו, ואוטומציה נוצלה בזרימת העבודה הקלינית כדי להפחית את העלות ולשפר את זמן התפנית. קבוצות קודמות השתמשו ברובוטים לטיפול בנוזל בקוד פתוח עבור שלבי חילוץ SARS-CoV-2 RNA20,21, אך הגדלנו את השימוש ברובוטים להכנת לוחות בדיקה ודגימות עומס22. כאן אנו מראים כי הפרוטוקול המותאם והשימוש במערכות טיפול בנוזל בקוד פתוח (איור 1) מאפשרים RT-qPCR מהיר ומדויק המבוסס על רוק ומהווה אסטרטגיה יעילה למעקב נרחב אחר בריאות הציבור.
Protocol
כל המחקרים בוצעו בהתאם לאוניברסיטת קלמסון ולוועדות הביקורת המוסדית לבריאות פריזמה (Prisma Health IRB # Pro00099491, 1 ביולי 2020).
1. התקנה של רובוט טיפול בנוזל בקוד פתוח
- התקן מודולי סינון אוויר חלקיקי (HEPA) בעלי יעילות גבוהה (ראה טבלת חומרים) בחלק העליון של כל רובוט שמטפל בנוזלים בהתאם להוראות היצרן.
- צרף פיפטה P20 8 ערוצים להרכבה השמאלית של רובוטי הכנת צלחת המיקס הראשית בהתאם להוראות היצרן.
- חברו פיפטה P20 להרכבה הימנית של רובוטי הטעינה לדוגמה בהתאם להוראות היצרן.
- הורד את סקריפטים מותאמים אישית Python (קובץ משלים 1 וקובץ משלים 2) במחשבים המתאימים.
- פתח את TigerSaliva Full 384 Loading.py ביישום שולחן העבודה בדגימת טעינת מחשבים רובוטיים. לחץ על כיול והגדר הן את הפיפטה והן את התוכנית לפי הוראות התוכנה.
- פתח את 12 Plates.py מלא ביישום שולחן העבודה במחשב הרובוט הראשי Mix. לחץ על כיול והגדר הן את הפיפטה והן את התוכנית לפי הוראות התוכנה.
- הדפס ארונות תקשורת לדוגמה מותאמים אישית עם מדפסת תלת-ממדית תלת-ממדית (3D) מותכת עם קובץ עיצוב (CAD) בעזרת מחשב (https://www.myminifactory.com/object/3d-print-141363). הדפס 16 ארונות תקשורת לכל רובוט טעינה לדוגמה, עבור שני סטים של שמונה מתלים.
2. הכנה של 20x מולטיפלקס N1 + P1 בדיקה / תערובת פריימר
- הכן אצווה של 20x מולטיפלקס N1 + P1 בדיקה / פריימר לערבב (נפח כולל של 20 מ"ל, טבלה 1) בצינור חרוטי 50 מ"ל בסביבה סטרילית הרחק SARS-CoV-2 RNA סינתטי או דגימות המטופל.
- Aliquot 1.6 מ"ל של התערובת באמצעות פיפטה סרולוגית לתוך צינורות צנטריפוגה סטריליים 2.0 מ"ל תווית כראוי.
- יש לאחסן את האליקוטים במקפיא בטמפרטורה של 20 מעלות צלזיוס עד לקבלת אפשרות מוכנה לשימוש.
3. הכנת תערובת שליטה חיובית
הערה: אין ליצור תערובת שליטה חיובית באותה סביבה סטרילית כמו תערובת בדיקה/פריימר או רכיבי תערובת ראשיים אחרים. יש להשתמש במיכל נפרד של מים ללא נוקלאז.
- לדלל SARS-CoV-2 RNA סינתטי (N1) מ 1,000,000 עותקים גנים / μL (cpμ) ל 10,000 cpμ על ידי הוספת 10 μL של פתרון מלאי 990 μL של מים ללא נוקלאז. Aliquot 25 μL של 10,000 cpμ לתוך צינורות סטריליים 0.2 מ"ל תווית כראוי. יש לאחסן את ה-aliquots שאינם בשימוש בטמפרטורה של -80°C.
הערה: יש לאחסן RNA סינתטי בטמפרטורה של -80°C כדי למנוע השפלה. - לדלל Hs_RPP30 DNA סינתטי (P1) מ 200,000 cpμ ל 10,000 cpμ על ידי הוספת 50 μL של פתרון מלאי למים 950 μL ללא נוקלאז. Aliquot 25 μL של 10,000 cpμ לתוך צינורות סטריליים 0.2 מ"ל תווית כראוי. יש לאחסן את ה-aliquots שאינם בשימוש בטמפרטורה של -80°C.
- לדלל כל רכיב לריכוז סופי של 200 cpμ על ידי הוספת 20 μL של SARS-CoV-2 ו- Hs_RPP30 10,000 מניות cpμ ל 960 μL של מים ללא נוקלאז, עבור סך של 1000 μL. Aliquot 20 μL של מעורב 200 cpμ שליטה חיובית לתוך 0.2 mL צינורות תווית כראוי. יש לאחסן aliquots בטמפרטורה של -80°C עד לקבלת מוכנות לשימוש.
4. הכנת צלחות תערובת מאסטר
הערה: צרו תערובת ראשית בסביבה סטרילית הרחק מרנ"א סינתטי של SARS-CoV-2 או מדגימות מטופלים. כל הרכיבים חייבים להיות מופשרים לחלוטין לפני הוספת לתערובת; ללא הפשרה מתאימה, הריכוזים עשויים להיות שגויים. הפשרה לקויה מסומנת על ידי נוכחות של קרח או צבע לא אחיד של ריאגנטים. יש לאחסן על בלוק מקפיא בעת הכנת התערובת.
- הכן אצווה של תערובת מאסטר מולטיפלקס (נפח כולל של 48 מ"ל, טבלה 2) בצינור חרוטי 50 מ"ל.
- הומוגניזציה התערובת על ידי הפיכת הצינור מעל 3x. אין לערבב על ידי צנרת למעלה ולמטה או מערבולת כמו זה יפגע באנזים.
- מלאו עמודים 1-4 של מאגר באר סטרילי בעומק 96-באר על ידי העברת 1.48 מ"ל של תערובת מאסטר לכל באר. חרוטי אחד 50 מ"ל ממלא ארבעה עמודים, מספיק עבור 12 צלחות.
- מכסים את המאגר העמוק עם אטם רדיד אלומיניום ומניחים אותו ברובוט הטיפול בנוזלים של תערובת מאסטר ייעודית. מניחים את מאגר הבאר העמוקה המלאה על סיפון 10, מניחים שש צלחות ריקות של 384 בארות על סיפונים 1-6, ואת תיבת הקצה P20 על סיפון 11. לחשוף את צלחת הבאר העמוקה ותיבות קצה ולסגור את הרובוט.
- אתחל את פרוטוקול ההפעלה המותאם אישית של Python על-ידי לחיצה על התחל הפעלה ביישום שולחן העבודה של הרובוט.
- לאחר 40 דקות, הריצה תושהה. כסו את 384 לוחות הבאר המלאים עם אטמי נייר כסף בזמן שהם נשארים ברובוט ולחצו עם הרולר כדי להבטיח דבקות. סמן את הקצה של כל צלחת באמצעות מזהה אצווה.
- מקם שש לוחות ריקים חדשים של 384 בארות על סיפונים 1-6 וחדש את פרוטוקול ההפעלה על-ידי לחיצה על המשך הפעלה. לאחר השלמת הריצה, לכסות את הסט הסופי של 384 לוחות היטב עם חותמות רדיד.
- Pipette 2 μL של תערובת המאסטר הנותרת לעמודים 1-3 ו 22-24 של צלחת ריקה 384-well לבקרת איכות אצווה. לאטום עם חותם ברור אופטי ולהפעיל על התרמוציקלר (סעיף 10.1-10.2). אם לבארות כלשהן יש ערכי מחזור סף N1 (Ct) או אם ליותר מ- 10 בארות יש ערכי P1 Ct, האצווה מזוהמת ולא ניתן להשתמש בה.
- יש לאחסן את צלחות התערובת המוכנות בטמפרטורה של 4 מעלות צלזיוס ולהשתמש בהן תוך 7 ימים ממועד ההכנה.
5. איסוף מדגם, צריכה וטיפול בחום
- הנחו את המשתתפים להימנע מאכילה, שתייה, עישון או היגיינת שיניים 30 דקות לפני איסוף הרוק. להנחות את המשתתפים לאסוף לפחות 1 מ"ל של רוק כי באופן טבעי בריכות בפה ולהפקיד אותו לתוך צינור חרוטי סטרילי 50 מ"ל ללא חומרים משמרים, ולאחר מכן לכסות את הצינור (קובץ משלים 3).
- לטהר את החלק החיצוני של צינורות איסוף רוק עם 70% אתנול או חיטוי מגבונים ולהעביר אותם למעבדה לבדיקה.
- רשום הגעה לדוגמה על-ידי סריקת כל ברקוד לדוגמה בגיליון האלקטרוני של הצריכה היומית (קובץ משלים 4).
- יש לטפל בחום בדגימות הסרוקות במשך 30 דקות בתנור בטמפרטורה של 95 מעלות צלזיוס. יש להסיר דגימות בעת לבישת כפפות עמידות בחום.
הערה: דגימות לא מטופלות יציבות בטמפרטורת החדר (23 °C (60 °F) עבור עד 72 שעות. לאחר טיפול בחום, דגימות חייב להיות מאוחסן ב 4 °C (60 °F), אם לא מעובד באופן מיידי.
6. הקצאה לדוגמה
- פתח את הגיליונות האלקטרוניים היומיים של טעינת מדגם עבור כל רובוט טעינה לדוגמה (קובץ משלים 5) במחשב בתחנת הקצאת הדגימה.
- הקצה 188 דגימות לכל צלחת 384-well כדלקמן: דגימות 1-48 נחשבות לרבעון 1, דגימות 49-96 נחשבות לרבע 2, דגימות 97-144 נחשבות לרבע 3, ודגימות 145-188 נחשבות לרבע 4. המדגם מנותח בכפילות.
- מגשי תוויות עם שם צלחת, תאריך ומספר רבעון. סרוק את הדגימות לפי הסדר לתוך גיליון העבודה של טעינת הדגימה לדוגמה.
הערה: ייתכן שיהיה צורך להפעיל באופן ידני דוגמאות מלוחות קודמים, כפי שהוגדר באיור 3. עיין בסעיפים 8.1-8.3 לקבלת הוראות הקצאה וטעינה ידניות לדוגמה.
7. הפעלת רובוטים לטעינת מדגם
- בתחנת הטעינה לדוגמה, בשורה שני סטים מלאים של שמונה מתלים מודפסים 3D המתאים למיקום הסיפון ברובוט.
- בטלו את התקלה בצינורות רבע 1 והניחו אותם במתלים מודפסים בתלת-ממד, החל ממיקום A1 בארון תקשורת 1. מלאו כל ארון תקשורת משמאל לימין ומלמעלה למטה. המשיכו בתבנית טעינה זו בארון תקשורת 2, ולאחר מכן המשיכו למתלים 4 ו-5 (עיינו באיור 2C).
הערה: ארונות תקשורת אינם ממוספרים ברציפות עקב הפרמטרים של תוכנית הרובוטים. - מקם מתלים טעונים רבע 1 מדגם על סיפונים 1, 2, 4, 5, 7, 8, 10, 11. מניחים טיפים P20 על סיפונים 3 ו -9. כדי לפשט את תהליך ההגדרה, טען חומרים מהחלק האחורי לחזית לתוך הרובוט.
- קח צלחת תערובת מאסטר מוכנה מראש מן 4 °C (60 °F), לתייג אותו עם שם צלחת ולהשתמש להב חד לחתוך קו בנייר כסף סביב בארות הבקרה (N23/24, O23/24, ו P23/24).
- מניחים את צלחת התערובת הראשית על סיפון 6 ומקלפים את כיסוי נייר הכסף, ומשאירים מאחור את המלבן הקטן המכסה את בארות הבקרה. לחשוף את תיבות הקצה ולסגור את הרובוט.
- אתחל את פרוטוקול ההפעלה המותאם אישית של Python על-ידי לחיצה על התחל הפעלה דרך יישום שולחן העבודה הרובוטי. כל רבעון לוקח 24.5 דקות כדי לטעון על הצלחת; הגדר שעון עצר כתזכורת.
- בזמן שהרובוט פועל, שחררו וטענו את צינורות הדגימה של רבע 2 לקבוצה השנייה של מתלים מודפסים בתלת-ממד כמתואר בסעיף 7.2.
- כאשר הרובוט עוצר, הסר את המתלים של רבע 1 והחלף אותם במתלים רבע 2. לחץ על חדש הפעלה ביישום שולחן העבודה.
- סכום רבע 1 צינורות מדגם ולאחסן אותם במקרר 4 °C (4 °F) בזמן ההמתנה לתוצאות.
חזור על תהליך טעינה זה עבור רבעים 3 ו- 4. - מעבירים את הצלחת הטעונה לארון בטיחות ביולוגית. כדי למזער את הזיהום, יש לשמור על צלחת מכוסה במהלך ההעברה.
8. טעינה ידנית מדגם
הערה: בצע הפעלה ידנית אחת בדגימות חוזרות (N1 שידור חוזר או הפעלה חוזרת, ראה איור 3) במקרה של טעינה רובוטית לקויה.
- אסוף דגימות חוזרות והקצה אותן כדוגמאות האחרונות ברבעון 4 (ראה סעיף 6.2). מספר דוגמאות, סרוק את הברקודים והזן את המיקום לדוגמה המקורי והתוצאה היא גיליון העבודה לטעינת הדגימה לדוגמה.
- העבר דגימות חוזרות לארון הבטיחות הביולוגית. אין לטעון צינורות מדגם חוזרים למתלי הטעינה של הרובוטים.
- Pipette 2 μL של כל מדגם חוזר לבארות הנכונות בעקבות דיאגרמת פריסת הלוח (קובץ משלים 6). השתמש בפיפטה ייעודית להוספת דגימות מטופלים. שמור על בארות בקרה מכוסות בנייר כסף תוך הוספת דגימות כדי למזער את הזיהום.
9. הוספת פקדים ללוחות בדיקה
- לקלף את כיסוי נייר הכסף מעל בארות בקרה באמצעות מלקחיים.
- Pipette 2 μL של מים ללא נוקלאז (ללא בקרת תבנית) לבארות N23-N24 ו 2 μL של 200 cpμ שליטה חיובית מעורבת (עיין בסעיף 3) לבארות O23-O24. פיפטה 2 μL של שליטה מדגם חולה חיובי מאושר לתוך בארות M23-M24 כמו בקרה נוספת. השאר בארות P23-P24 ריק כדי לפקח על איכות אצוות תערובת מאסטר.
- לכסות את הצלחת עם חותם ברור אופטית ולהשתמש רולר אפליקטור כדי לדבוק חותם לכל הבארות. מערבולת את הצלחת ב 2500 סל"ד במשך 30 שניות לערבב ביסודיות. צנטריפוגה הצלחת ב 500 x g במשך 1 דקה.
10. ביצוע RT-qPCR
- צור תוכנית פרוטוקול בתוכנת התרמו-מחזור בהתאם לתנאים המתוארים (טבלה 3). שמור את הפרוטוקול עבור לוחות עתידיים. מניחים את הצלחת האטומה בתרמוציקלר ומפעילים את הפרוטוקול.
- יצא ערכי Ct כקובץ .xslx והעתק את הערכים לגיליון האלקטרוני של הטעינה לדוגמה (קובץ משלים 5).
הערה: גליונות אלה תוכננו בהתאמה אישית עבור קבצי פלט Ct מתוכנות היצרן וייתכן שיהיה צורך בשינוי כדי לקבל תבניות אחרות.
11. קביעת תוקף הלוח
- אמת הן את הפקד החיובי והן את הדגימות החיוביות הידועות ו/או את הפקד השלילי כדי לשקול את תוצאות הלוחות כחוקות. להעריך את בארות הבקרה עם הקריטריונים הבאים.
- לשליטה חיובית, בדוק אם לפחות בקרה חיובית אחת (O23/O24) מייצרת ערכי Ct של בין 22-28 עבור בדיקות P1 ו- N1. לחלופין, בארות המדגם החיוביות הידועות (M23/M24) מייצרות ערכי P1 ו- N1 Ct <33 בבדיקות P1 ו- N1.
- לשליטה שלילית, ודא שאין ערכי N1 או P1 Ct באחת משתי הבקרות השליליות היטב (N23/N24). ודא שערכי Ct כוללים עקומות הגברה חוקיות לפני ביטול תוקף הלוח.
12. פירוש תוצאות מדגם
- קבעו את תוצאת המטופל בעקבות הדיאגרמה (איור 3) ודיווחו על דגימות שנפתרו.
- הערך את תוצאת P1 כתקפה או כלא חוקית. אם P1 מפיק תוצאה של Ct <33, שקול את הבאר תקפה והמשיך לתוצאה מ- N1. אם P1 מפיק תוצאה של ערך Ct >=33 או ללא Ct, שקול את הבאר לא חוקית.
- הערך את תוצאת N1 ככן, לא או לא*. אם N1 מייצר תוצאה של Ct <33, הבאר היא כן. אם N1 אינו מפיק ערך Ct, הבאר היא לא. אם N1 מייצר > Ct =33, הבאר היא NO *. ודא שכל ערכי N1 Ct משויכים לעקומת הגברה אמיתית. אם לערך Ct עבור N1 אין עקומת הגברה, הבאר היא לא.
- זהה דוגמאות חוזרות (N1 הפעל מחדש או הפעל מחדש), תייג אותן במספר מדגם פנימי ובסוג מדגם והחזר אותן לזרימת העבודה של הטעינה (סעיף 8.1-8.3).
- הערך את תוצאת P1 כתקפה או כלא חוקית. אם P1 מפיק תוצאה של Ct <33, שקול את הבאר תקפה והמשיך לתוצאה מ- N1. אם P1 מפיק תוצאה של ערך Ct >=33 או ללא Ct, שקול את הבאר לא חוקית.
13. ניקוי מעבדה
- רובוטים לטיפול בנוזלים
- נקו את כל הצדדים עם חומר חיטוי ברמת הביניים. אין להשתמש באתנול מכיוון שהוא ישפיל את הפלסטיק.
- יש לנגב בעדינות קצה פיפטה ופח אשפה עם אלכוהול (70% אתנול או 100% איזופרופנול) לנגב. נגב את המקלדת והעכבר.
- ארון בטיחות ביולוגית
- נקו את כל המשטחים עם חומר חיטוי ברמת הביניים. הפעל את אור UV במשך 15 דקות.
תוצאות
קבענו את טווח הזיהוי של בדיקות RT-qPCR ופריימרים לתכולת חומצת גרעין סינתטית הן עבור SARS-CoV-2 (N1) והן עבור Hs_RPP30 (P1). דילול סדרתי של פי 10 של ריכוזים ידועים של רנ"א SARS-CoV-2 סינתטי משולב ודנ"א Hs_RPP30 סינטטי במים נעשה. הנוסחה הבאה שימשה להמרת משקל מולקולרי למספר עותק גנים
מספר עותק גנים = (ng * 6.0221 x 1023)/((אורך בזוגות בסיס*660 גרם/מול) *1 x 109 ng/g)
ו- RT-qPCR בוצע. לאחר ביצוע RT-qPCR, עקומות ליניאריות לזיהוי N1 (איור 4A) וזיהוי P1 (איור 4B) הראו מקדמי מתאם טובים במגוון רחב של ריכוזי העתקת גנים (R2= 0.9975 ו- R2= 0.9884, בהתאמה). תוצאה זו מציינת כי השילוב של פריימר וערכות בדיקה אינו מעכב והוא יכול לזהות במדויק SARS-CoV-2 RNA בהעתקת גן אחת / μL (Cq = 33). עותק גן אחד שווה בערך לעותק ויראלי אחד; עם זאת, לא קבענו מספרי העתקה ויראליים כמותיים ברוק בשל האופי הכמותי למחצה של RT-qPCR. ניסינו לדמות דגימות רוק חיוביות על ידי העלאת RNA SARS-CoV-2 סינתטי של ריכוזים ידועים לרוק ללא וירוסים (הן מטופלים בחום והן ללא חום מטופלים) אך לא הצלחנו לייצר הגברה N1 בריכוזים נמוכים של RNA (נתונים לא הוצגו). ייתכן שהסיבה לכך היא השפלת RNase או גורמים מבלבלים אחרים.
כמו כן, נבדקה השונות הבין-מדומה בין שיטות טעינת מדגם אוטומטיות וידניות. כדי להעריך את השונות הבין-אסאית, 20 דגימות חיוביות ייחודיות נטענו באמצעות המדריך (המתואר בסעיף 8.1-8.3) ואוטומטיות (המתוארות בשיטות סעיף 7.1-7.11). ערכי N1 Ct הושוו כדי לקבוע אם רובוטים לטיפול בנוזל וטעינת מדגם ידנית הניבו תוצאות שוות ערך (איור 5A). הקשר הליניארי בין שיטות ידניות ואוטומטיות יצר מקדם מתאם גבוה (R2= 0.9088), המציין כי שתי השיטות שוות ערך מבחינה פונקציונלית. ככל שערכי N1 Ct גדלו, השונות של ערכי Ct גדלה גם היא. מגמה זו נובעת ככל הנראה מההתפלגות ההטרוגנית של חלקיקים ויראליים בתוך הרוק, אשר בולט יותר כאשר פחות חלקיקים נמצאים. כדי להעריך את השונות הפנימית, נעשתה השוואה בין ערכי N1 Ct מבארות משוכפלות של דגימות רוק ייחודיות באמצעות שתי שיטות טעינת הדגימה (איור 5B). הקשר הליניארי בין שכפולים של טעינת מדגם אוטומטית (R2= 0.9622) יצר מקדם מתאם מעט גבוה יותר מזה של טעינה ידנית (R2= 0.9589), המציין רבייה גבוהה של זיהוי SARS-CoV-2 עבור שתי שיטות הטעינה.
לבסוף, נעשתה הערכה של הפחתת צמיגות הרוק ביחס לשיטות הטיפול בחום (איור 6). הרוק הושג ממקור אחד כדי למנוע שונות מדגם. שונות רבה יותר בערכי P1 Ct בתוך שיטת טיפול בחום אחת עשויה להצביע על צמיגות מדגם גבוהה יותר כמו רוק צמיג לא ניתן לשאוף ולחלק בדיוק. שתי שיטות הטיפול בחום של 30 דקות ו-60 דקות יצרו ירידה משמעותית בשונות המדגם בהשוואה ללא בקרת טיפול (p = 0.0006 ו- p = 0.0429, בהתאמה). לא היה הבדל משמעותי בין טיפולים של 30 דקות ל -60 דקות (p = 0.2245); לכן, שיטת הטיפול בחום של 30 דקות יושמה כדי לקצר את זמן העיבוד.

איור 1: זרימת עבודה במעבדה המשתמשת במערכת האבחון מבוססת הרוק RT-qPCR. (A) דגימות נאספות ומטופלות בחום ב- 95 °C למשך 30 דקות. דגימות שטופלו ממוינות ומעקב עם מידע על מטופלים באמצעות מערכת גיליון אלקטרוני פנימית. רובוט שמטפל בנוזלים טוען דגימות לבארות כפולות של לוחות תערובת ראשיים מוכנים. טכנאי טוען ידנית את הפקדים, אוטם את הצלחת ומניח את הצלחת בתרמוציקלר לעיבוד. התוצאות מנותחות באמצעות מערכת מחשב אוטומטית ומאומתות על ידי טכנאי. (ב) טכנאי מכין ריאגנטים לתערובת הראשית המתווספים למאגר באר עמוקה בארון בטיחות ביולוגי סטרילי. מאגרי באר עמוקה מלאים נטענים לרובוט ייעודי לטיפול בנוזלים. לוחות שהושלמו חתומים בנייר כסף, מסומנים בתווית ומאוחסנים בטמפרטורה של 4 מעלות צלזיוס. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.

איור 2: פריסות המשמשות לרובוט המטפל בנוזלים. (A) פריסת סיפון לרובוטים להכנת לוחות מיקס ראשיים. עם פיפטה בת שמונה ערוצים, הרובוט מתוכנת לאסוף טיפים של פיפטה, לשאוף תערובת מאסטר ממאגר באר עמוק של 96 בארות, לחלק תערובת ראשית ללוחות ריקים של 384 בארות, ולהוציא את קצות הפיפטה לפח אשפה. זה חוזר על עצמו במשך שש צלחות לכל ריצה. (ב) הגדרת סיפון עבור רובוטי טעינה לדוגמה. עם פיפטה חד-ערוצית, הרובוט מתוכנת להרים קצה פיפטה, לשאוף דגימת רוק, לחלק דגימת רוק לתוך בארות כפולות של צלחת תערובת ראשית של 384 בארות, ולהוציא את קצה הפיפטה לפח אשפה. זה חוזר על עצמו עבור 48 דגימות לכל ריצה. (ג) הזמנת טעינת צינור לדוגמה עבור ארונות תקשורת מודפסים בתלת-ממד. חצים אדומים מציינים סדר טעינה בתוך ארון תקשורת, והמספרים בקופסאות הלבנות מציינים את סדר הטעינה של כל קבוצת המתלים. ההתקנה כולה תטען 188 דגימות כפולות ללוחית של 384 בארות. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 3: תרשים זרימה לדוגמה שנוצר. דגימות עם P1 תקף N1 חיובי נקבעו להיות דגימות רוק אנושי חיובי עבור SARS-CoV-2. תוצאות מדגם חוקיות וחיוביות/שליליות נחשבו חד משמעיות. דגימות שלא הניבו תוצאות חד משמעיות בהפעלה הראשונה סווגו כהפעלה חוזרת (RR מסומן) או N1 Rerun (מסומן N1 RR). דגימות הפעלה חוזרת לא היו הגברה P1 תקף, ודגימות N1 Rerun היה הגברה N1 חיובית בשכפול יחיד. אם לא ניתן היה לייצר הגברה P1 חוקית על-ידי הפעלה ידנית עוקבת, או שלשני העותקים המשוכפלים היו ערכי N1 Ct מעל הסף החיובי (Ct >33), תוצאות המדגם נחשבו לא חד משמעיות. למטרות קליניות, דגימות מטופלים שלא הגיעו למעבדה, היה כמות מספקת של רוק לפיפטה או ניזוקו נחשבו לא חוקיים. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 4: זיהוי RT-QPCR של N1 (SARS-CoV-2) RNA סינתטי ו-P1 (Hs_RPP 30) DNA סינתטי. עקומות סטנדרטיות שורטטו עם סטיות תקן כדי לקבוע את טווח הזיהוי המדויק באמצעות שילוב בדיקה /פריימר זה. (A) ערכי ה- Ct הממוצעים (n =4) שהושגו בדילולים המתאימים שורטטו כנגד הכמות המשוערת של RNA סינתטי (1x100 עד 1x104 עותקי RNA ב- 10 μL של תגובת RT-qPCR). (B) ערכי ה- Ct הממוצעים (n =3) שהושגו בדילולים המתאימים שורטטו כנגד הכמות המשוערת של DNA סינתטי (1 x 100 ל 1 x 104 עותקים גנטיים ב 10 μL של תגובת RT-qPCR). לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
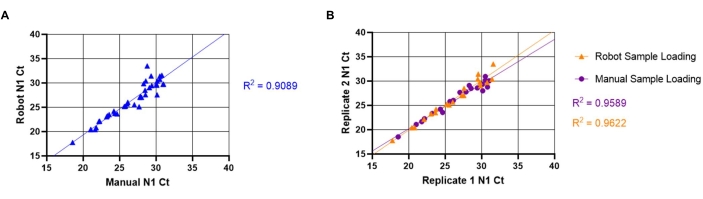
איור 5: השוואה בין ערכי Ct ידניים ואוטומטיים להעברת רוק SARS-CoV-2 (N1). דגימות הרוק החיוביות הידועות SARS-CoV-2 (n =20) נטענו כפולות לצלחת תערובת ראשית של RT-qPCR על ידי רובוט שמטפל בנוזלים. לדגימות יש ערך Ct הנע בין 18-32 עבור N1. אותן דגימות נטענו באופן ידני לבארות כפולות במיקום לוח אחר. (A) ערכי N1 Ct שהתקבלו מדגימות ייחודיות באמצעות טעינת הרובוט והדגימה הידנית הוחלפו כדי לקבוע שונות בין-בדיקה בין טעינה ידנית לרובוט. (ב) שונות תוך-בדיקה נקבעה גם באמצעות שכפול מוחלף של ערכי N1 Ct שהתקבלו הן מטעינת רובוט והן טעינת מדגם ידנית. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 6: הערכת שיטות טיפול בחום להפחתת צמיגות ברוק. SARS-CoV-2 רוק שלילי נאסף ממקור אחד ו aliquots טופלו בחום במשך 0 דקות, 30 דקות, או 60 דקות ב 95 °C (95 °F). ערכי P1 Ct משכפולים טכניים (n = 12) של כל תנאי שורטטו כדי לקבוע את השונות בין שיטות הטיפול. השוואות זוגיות בין קבוצות הוערכו באמצעות בדיקת t לא מזווגת (*** מציינת p <0.001, * מציין p <0.05). לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
איור משלים 1: השוואה של N1 Ct בדגימות רוק P1 Ct נמוכות. הדגימות החיוביות עם P1 Ct נמוך נבחרו בהשוואה ל- N1 Ct (n = 106). ערכי N1 Ct נעו בין 14-33, המציין כי הבדיקה יש טווח דינמי בדגימות רוק הדומה לעקומה הסטנדרטית. נא לחץ כאן כדי להוריד קובץ זה.
| רכיב | רצף (5'→3') | ריכוז מלאי | נפח | ||
| 2019-nCoV-N1 בדיקה | /5FAM/ACCCCGCAT/ZEN/TACGTGGTGGACC/3IABKFQ | 50 מיקרומטר | 500 μL | ||
| 2019-nCoV-N1-For | GACCCCAAAATCAGCGAAAT | 100 מיקרומטר | 2000 μL | ||
| 2019-nCoV-N1-Rev | TCTGGTTACTGCCAGTTGAATCTG | 100 מיקרומטר | 2000 μL | ||
| Hs RPP30 Cy5 בדיקה | /5Cy5/TTCTGACCT/ZEN/GAAGGCTCTGCG/3IABkFQ | 50 מיקרומטר | 500 μL | ||
| HS-RPP30-For | AGATTTGGACCTGCG | 100 מיקרומטר | 2000 μL | ||
| Hs-RPP30-Rev | GAGCGGCTGTCTCCACAAGT | 100 מיקרומטר | 2000 μL | ||
| מים | - | - | 11000 μL | ||
טבלה 1: רכיבים של N1 + P1 בדיקה / תערובת פריימר.
| רכיב | ריכוז מלאי | עוצמת קול לכל תגובה | ריכוז סופי | אמצעי אחסון של אצווה | ||
| לונה חמהתחילת תערובת אנזימים RT | פי 20 | 0.5 μL | פי 1 | 3 מ"ל | ||
| תערובת תגובת חוצץ לונה | פי 2 | 5.0 μL | פי 1 | 30 מ"ל | ||
| N1 + P1 פריימר / תערובת בדיקה | nCoV N1 F: 10 מיקרומטר | 0.5 μL | 500 ננומטר | 3 מ"ל | ||
| nCoV N1 R: 10 מיקרומטר | 500 ננומטר | |||||
| בדיקה nCoV N1: 2.5 מיקרומטר | 125 ננומטר | |||||
| RPP_30 P1 F: 10 מיקרומטר | 500 ננומטר | |||||
| RPP_30 P1 R: 10 מיקרומטר | 500 ננומטר | |||||
| בדיקה RPP_30 P1: 2.5 מיקרומטר | 125 ננומטר | |||||
| מים ללא נוקלאז | --- | 2 μL | --- | 12 מ"ל | ||
| סכום ביניים | --- | 8 μL | --- | 48 מ"ל | ||
| תבנית | 2 μL | |||||
טבלה 2: רכיבים של תערובת מאסטר מולטיפלקס SARS-CoV-2.
| במה | טמפרטורה (°C) | משך | מספר מחזורים |
| תמלול הפוך | 55 | 10 דקות | 1 |
| דנטורציה ראשונית | 95 | 1 דקות | 1 |
| טאצ'דאון | 95 | 10 שניות | 3 |
| 72 | 30 שניות | ||
| 95 | 10 שניות | 3 | |
| 69 | 30 שניות | ||
| 95 | 10 שניות | 3 | |
| 66 | 30 שניות | ||
| הגברה ראשית | 95 | 10 שניות | 40 |
| 65 | 30 שניות |
טבלה 3: טאצ'דאון פרוטוקול RT-qPCR. תנאי התרמו-רכיבה על אופניים לבדיקת אבחון RT-qPCR SARS-CoV-2 חד-שלבית.
| שלב טאצ'דאון | אין צעד טאצ'דאון | |||
| ממוצע N1 Ct | ממוצע P1 Ct | ממוצע N1 Ct | ממוצע P1 Ct | |
| דוגמה 1 | 19.65 | 22.7 | 27.8 | 28.3 |
| דוגמה 2 | 22.24 | 24.9 | 28.77 | 30.5 |
| דוגמה 3 | 18.85 | 19.2 | 24.65 | 25.9 |
| דוגמה 4 | 25.56 | 22.8 | 31.93 | 29.2 |
| דוגמה 5 | 22.34 | 24.8 | 38.48 | 40.0 (זיהוי שנכשל) |
טבלה 4: השוואה של ערכי Ct טאצ'דאון עבור חמש דגימות חיוביות מול ערכי Ct ללא טאצ'דאון.
| לדוגמה | טייגרסליבה | בדיקת SARS-CoV-2, המבוססת על רוק, זמינה מסחרית | ||
| N1 Ct | P1 Ct | ערך Covid-19 | ערך RNasep | |
| D11 | 16.4 | 18.1 | 20.86 | 23.4 |
| E11 | 18.9 | 19.1 | 25.6 | 21.2 |
| F11 | 19.5 | 18.4 | 22.8 | 22.2 |
| G11 | 22.2 | 19.1 | 23.7 | 22.9 |
| H11 | 26.4 | 21.3 | 32.2 | 26.7 |
| A12 | 14.8 | 16.5 | 29.15 | 19 |
| B12 | 24 | 19.6 | 31.05 | 21.35 |
| C12 | 14.9 | 17.5 | 20.84 | 18.9 |
טבלה 5: השוואה בין תוצאות TigerSaliva Ct ותוצאות בדיקת SARS-CoV-2 המבוססות על רוק זמינות מסחרית. שתי הבדיקות בוצעו על אותן דגימות רוק (n =8).
קובץ משלים 1: סקריפט מותאם אישית ליצירת צלחת מיקס מאסטר רובוט. אנא לחץ כאן כדי להוריד קובץ זה.
קובץ משלים 2: סקריפט מותאם אישית לעיבוד רוק על רובוטים טעינת מדגם. אנא לחץ כאן כדי להוריד קובץ זה.
קובץ משלים 3: הוראות לאיסוף עצמי של דגימות רוק באיכות גבוהה מהמשתתפים. פרטים נוספים ניתן למצוא בתיאור הווידאו הקצר של תהליך הבדיקה הזמין https://www.clemson.edu/centers-institutes/reddilab/index.html. נא לחץ כאן כדי להוריד קובץ זה.
קובץ משלים 4: גיליון אלקטרוני של צריכת דגימה. לחץ כאן כדי להוריד קובץ זה.
קובץ משלים 5: גיליון אלקטרוני של טעינה לדוגמה. לחץ כאן כדי להוריד קובץ זה.
קובץ משלים 6: דיאגרמת פריסת לוח 384-well לדוגמה. לחץ כאן כדי להוריד קובץ זה.
Discussion
הבדיקה המתוארת בפרוטוקול הוערכה על ידי מחקר אימות עצמאי. נמצא כי הבדיקה הייתה 98.9% ספציפיות (1.1% חיובי כוזב) ו 90.0% רגישות (10.0% שלילי כוזב) כאשר מוערך נגד דגימות nasopharyngeal מזווג נלקח באותו זמן (n = 837; 817 שלילי, 20 חיובי). חשוב לציין, שלושה משתתפים שנבדקו חיוביים עם TigerSaliva ושלילי עם ספוגית האף נבדקו מחדש עם מטליות 48 שעות מאוחר יותר והחזירו תוצאות חיוביות, מה שמצביע על כך ש- TigerSaliva עשוי להיות מסוגל לזהות זיהומי SARS-CoV-2 מוקדם יותר במהלך המחלה.
דימינו דגימות רוק חיוביות על ידי ספייק רוק ללא וירוסים (הן מטופלים בחום והן לא מטופלים בחום) עם ריכוזים ידועים של SARS-CoV-2 RNA סינתטי וביצע דילול של פי 10 כדי לקבוע את מגבלת ה- Ct ברוק. הגן N1 לא היה ניתן לזיהוי מתחת 10,000 עותקים גנטיים (כ Ct = 28) בדגימות חיוביות מדומות. אנו חושדים שזה בגלל השפלת RNase או גורמים מבלבלים אחרים. עם זאת, האינטראקציה של RNases רוק עם RNA סינתטי עירום הוא כנראה שונה מן האינטראקציה עם חלקיקים ויראליים, גם לאחר שהם כבר denatured על ידי חום. דגימות רוק חיוביות זוהו עם Ct >30 ומעבדות חיצוניות השיגו נתוני רצף גנטי SARS-CoV-2 מדגימות אלה. אנו משערים כי החלבונים הנגיפיים מספקים הגנה מפני השפלת RNA בדגימות רוק החולה.
השלב הקריטי ביותר בפרוטוקול הוא יישום אוטומציה להכנת תערובת ראשית ועיבוד מדגם רוק (סעיפים 4 ו -7 בהתאמה). הדבר מאפשר תהליכי משימה חופפים, מה שמקצר באופן דרסטי את זמן התפנית. צעד קריטי נוסף הוא פרשנות תוצאות קליניות (סעיפים 11 ו-12). יצירת קטגוריות תוצאות ביניים (שידור חוזר והפעלה חוזרת של N1) מזערה גם את התרחשותן של תוצאות בדיקה לא חד משמעיות.
הדגמנו שהשונות בין שיטות טעינה ידניות ואוטומטיות של דגימת רוק היא זניחה (איור 5A) ושאוטומציה עשויה לשפר את יכולת השכפול של זיהוי SARS-CoV-2 (איור 5B). אוטומציה צריכה להיות מועדפת כדי להקל על הבדיקות בעת תכנון והרחבה של מעבדות קליניות25. זרימת העבודה במעבדה משופרת עם יישום משימות אוטומטיות רובוט26. יכולות הקוד הפתוח של הרובוטים המטפלים בנוזל מאפשרות יישום של סקריפטים מותאמים אישית לעיצוב פרוטוקול. זה הופך רובוטים לטיפול בנוזלים למערכת זולה וניתנת לשינוי בהשוואה לשיטות אוטומציה קלינית מסורתיות. זוהי גם אסטרטגיה אידיאלית לביצוע משימות מעבדה שחוזרות על עצמן. הרמה הגבוהה של יכולת התאמה אישית של המערכת מתורגמת לחופש לשנות תוכנות מעבדה (למשל, צינורות איסוף, טיפים פיפטה, או לוחות 384-well) במקרה של מחסור. לכן, אוטומציה באמצעות רובוטים לטיפול בנוזלים היא בת קיימא הן עבור מעקב בקנה מידה גדול בקנה מידה קטן ומחקר.
היתרון העיקרי של אסטרטגיית בדיקה זו הוא זמן תפנית קצר בהרבה ביחס למעבדות קליניות אחרות. השימוש ברובוטים אוטומטיים לטיפול בנוזלים ממלא תפקיד מרכזי בהפחתת זמן התפנית, אך שימוש בו-זמני ברובוטים ותרמוציקלים הוא גם אינסטרומנטלי למקסם את יעילות הבדיקה. רובוט אחד ותרמוסיקר צריך להיות מופעל כזוג, שבו שתי המכונות משמשות יחד לטעינת מדגם ללא הפרעה וניתוח תוצאות מדגם. לאחר שנוצרת זרימה קבועה של דגימות שהוקצו, ניתן להפעיל את כל זוגות המכונות בו-זמנית. שימוש מתמיד בו-זמני ברובוטים ובתרמו-מחזורים מגדיל באופן דרסטי את יכולת הבדיקה ואת היעילות, וזה חיוני כדי להתאים לנפח הבדיקות הגבוה.
בניגוד לפרוטוקולים מבוססים אחרים של SARS-CoV-2 RT-qPCR, כללנו שלב טאצ'דאון בפרוטוקול התרמוציקלר כדי לשפר את חישול הגשושית וערכות הפריימר לגנים היעד27, ובכך להפחית את הסיכון להגברה כושלת. התוצאות הראו כי הטאצ'דאון שיפר את הזיהוי של דגימות חיוביות מבלי להסתכן באובדן של איגוד פריימר ספציפי (טבלה 4). קבענו שניתן לזהות בו-זמנית מגוון רחב של עותקי RNA של SARS-CoV-2 (איור 4A) ועותקי דנ"א Hs_RPP30 (איור 4B) על-ידי בדיקת RT-qPCR.
מגבלה אחת של רובוטים לטיפול בנוזלים היא האפשרות של זיהום צולב מדגימות חיוביות במהלך העברת רוק. רוק הוא נוזל ויסקולסטי28 ועשוי להיגרר על פני בארות סמוכות לאחר שהופץ מקצה הפיפטה. יתר על כן, ההטרוגניות של הרוק29 עלולה לגרום להפצה לא אחידה של חלקיקים ויראליים בכל המדגם. זה מגדיל את האפשרות של תוצאות חיוביות שגויות ושליליות, המחייבות את ייעודן של דגימות N1 Rerun ו- Rerun. עם זאת, 14.1% מהדגימות שהוגדרו בתחילה כ- N1 Rerun נפתרו כחיוביות עבור SARS-CoV-2 והיו בסיכון גבוה פי 30 מאשר דגימות שידור חוזר כדי לפתור כחיוביות לאחר בדיקה חוזרת. כתוצאה מכך, הבחנה בין שידור חוזר ל-N1 Rerun (איור 3) אפשרה הפרדה מדויקת יותר של דגימות שעשויות להיות חיוביות, והגבירה את הרגישות והספציפיות של בדיקת האבחון שלנו. פרמטרים אחרים שהתקבלו לבדיקות רוק אבחנתיות לא עשו הבחנה זו12,14,24,30,31.
דגימות רוק יכול להיות קשה פיפטה בשל הטרוגניות וצמיגות32. טיפול בחום מפחית כראוי חלבונים בביומטריקס הרוק, מפחית את הצמיגות ומבטל את הצורך בריאגנטים להפקת RNA9, שהיו נדירים בשלבים המוקדמים של המגפה10. טיפול בחום ממושך גם מנטרל וירוסים קיימים33 המאפשר עיבוד מעבדה ברמות בטיחות ביולוגית נמוכות יותר. כתוצאה מכך, מיצוי RNA מבוסס חום (המתואר בסעיף 5.4) יושם כדי להפחית את הצמיגות באמצעות denaturation חלבון (איור 6). בהתבסס על התוצאות, אנו מניחים כי טיפול בחום עשוי גם הומוגניזציה דגימות רוק בנוסף denaturing ביומטריקס החלבון. קבוצות אחרות שילבו טיפול בחום וטיפול בחלבון K כדי להגביר את ההומוגניות9,14,34. בחרנו שלא ליישם את הצעד הזה מכיוון שהוא עלול לפגוע בחלבוני וריון בקצב שמשאיר את הרנ"א הנגיפי חשוף לירידה בחום35. יתר על כן, דילול מדגם עם proteinase K עשוי להסוות דגימות חיוביות המכילות פחות חלקיקים ויראליים ובכך, הפחתת הרגישות. בנוסף, תוצאות הבדיקה הושוו לבדיקת SARS-CoV-2 מבוססת רוק זמינה מסחרית (Logix Smart COVID-19) המשתמשת בהפקת RNA חרוז מגנטי (טבלה 5). נמצא כי הבדיקה הנוכחית התאימה יותר לאיתור דגימות חיוביות חלשות בהשוואה לבדיקה הזמינה מסחרית.
קשה לכמת מספר העתקת וירוס ברוק באמצעות RT-qPCR בלבד, כי qPCR הוא חצי כמותי. קיימת שונות אינהרנטית בין ערכי Ct שמקורם במגבלות טכניות. ניתן לקבוע את מספר עותק הגן מערכי Ct (איור 4) והוא שווה בערך למספר עותק נגיפי. פתרון אפשרי אחד כדי לקבוע את מספר העותק הנגיפי בדגימות רוק הוא ddPCR, המספק כימות קשה של עותקי גנים בתגובה. עם זאת, אנו מאמינים כי זה מספיק כדי לספק תוצאות איכותיות לרופאים ותוכן ויראלי יחסית ניתן להשוות על פני דגימות מעובדות עם השיטות שלנו.
למרות כמה מגבלות המתעוררות בעת שימוש ברוק, בדיקת SARS-CoV-2 על ידי RT-qPCR מבוסס רוק מוכיחה להיות שיטה יעילה לזיהוי RNA ויראלי מהיר ואמין בכל קנה מידה של בדיקות. הדבר נכון במיוחד כאשר יחד עם ניצול של מערכות טיפול בנוזל קוד פתוח. ניתן לשנות גישה זו לבדיקה כדי לזהות רצפי חומצות גרעין אחרים הרלוונטיים לאבחון, כגון סוכני מחלות זיהומיות, סמני מחלות או וירוסים אחרים. זה הופך את הבדיקה ישימה הן למאמצי אבחון קליניים והן למאמצי אבחון מחקריים.
Disclosures
למחברים אין מה לחשוף. הבדיקה המתוארת בפרוטוקול נופלת תחת רוקDirect EUA שהוגש על ידי בית הספר לבריאות הציבור של ייל.
Acknowledgements
המחברים מודים להנהלתו של קלמסון, לצוות הרפואי ולעובדי המעבדה הקלינית במעבדת REDDI שעזרו ליישם ולנהל את בדיקות SARS-CoV-2. אנו מודים לד"ר פיליפ באקהולטס ולד"ר קרולין בניסטר מאוניברסיטת דרום קרוליינה על ייעוץ ראשוני לפרויקטים וקשרים בתעשייה לרכישת ציוד. אנו מודים לסטודנטים, פרופסורים ואנשי צוות רבים על עזרתם באיסוף דגימות. תודה לתלמידי חקירה יצירתית על איסוף נתוני עקומה סטנדרטית. המימון למחקר זה התקבל ממענק המכונים הלאומיים לבריאות P20GM121342 (הוענק ל- DD ו- LGP), מחלקת האתלטיקה של קלמסון, סגן נשיא אוניברסיטת קלמסון למחקר, ומושל דרום קרוליינה & ועדת ביקורת אג"ח משותפת.
Materials
| Name | Company | Catalog Number | Comments |
| 100% EtOH | Fisher scientific | 22-032-601 | |
| 20 uL Filtered Pipette Tips | Opentrons | 20uL tips | |
| 2mL Microcentrifuge Tubes | Fisher Scientific | 14-666-313 | Alternate product may be used |
| Armadillo PCR Plate, 384-well, clear, white wells | Thermo Scientific | AB3384 | Alternate product may be used |
| Celltreat 2mL 96 Deep Well Plates | Fisher Scientific | 50-828-743 | For mastermix preparation |
| Clear PCR Sealing Sheets | Thermo Scientific | AB0558 | Alternate product may be used |
| DPEC Treated Water | Ambion (Thermo Scientific) | AM9916 | |
| Flip Cap 50 mL Conical Tubes | VWR | 75845-210 | For sample collection |
| Foil PCR Sealing Sheets | Thermo Scientific | AB0626 | For storage of mastermix plates, Alternate product may be used |
| HS_RPP30 Synthetic DNA | Integrated DNA Technologies | 299788131 | P1 positive control |
| Luna Buffer Probe One-Step Reaction | New England Biolabs | M3006B | |
| Luna WarmStart RT Enzyme Mix | New England Biolabs | M3002B | |
| nCOV_N1 Forward Primer, 100 nmol | Integrated DNA Technologies | 10006830 | |
| nCOV_N1 Probe Aliquot, 50 nmol | Integrated DNA Technologies | 10006832 | Probe can be synthesized by other vendors with SYBR or FAM fluophores |
| nCOV_N1 Reverse Primer, 100 nmol | Integrated DNA Technologies | 10006831 | |
| Opentron HEPA Filter Module | Opentrons | N/A | Not required, but useful to reduce contamination |
| Opentron Multichannel Attachment, P20 | Opentrons | 999-00005 | For mastermix preparation |
| Opentron OT-2 Liquid Handling Robot | Opentrons | OT-2 | |
| Opentron Pipette Attachment, P20 | Opentrons | 999-0000215 | For sample loading |
| Oven | Memmert | UF450 PLUS 208V-3PH | |
| PCR Tubes (rnase, dnase free) | Fisher Scientific | 14-230-225 | For aliquots of positive and neg controls |
| PolarSafe Aluminum Cooling Block, 15-Well (1.5/2.0 mL Tubes) | VWR | 10808-952 | |
| PolarSaf Aluminum Cooling Block, 24-Well (0.5mL tubes) | VWR | 10808-956 | |
| RNAse P (ATTO 647) Probe, 50 nmol | Integrated DNA Technologies | 10007062 | Probe can be synthesized by other vendors with Cy5 fluorophore |
| RNAse P Forward Primer, 100nmol | Integrated DNA Technologies | 10006836 | |
| RNAse P Reverse Primer, 100 nmol | Integrated DNA Technologies | 10006837 | |
| Sars-CoV-2 Synthetic RNA Control 2 | Twist Biosciences | 102024 / 103907 / 103909 | N1 Positive Control |
| Scanners | Code | CR1500 | Only required when scaling up |
| Small HEPA Filtered Hood | Erlab | Captair Bio 321 | For mastermix preparation |
| Thermocycler CFX384 Touch | Biorad | CFX384 Touch | Alternate models can be used, e.g. CFX384 Opus |
| X-acto Knife Set | Staples | N/A | To cut foil for keeping control wells covered |
References
- Zhu, N., et al. A novel coronavirus from patients with pneumonia in China, 2019. New England Journal Medicine. 382 (8), 727-733 (2020).
- Chen, J. Pathogenicity and transmissibility of 2019-nCoV-A quick overview and comparison with other emerging viruses. Microbes and Infection. 22 (2), 69-71 (2020).
- Petersen, E., et al. Comparing SARS-CoV-2 with SARS-CoV and influenza pandemics. The Lancet. Infectious Diseases. 20 (9), 238-244 (2020).
- Honein, M. A., et al. Summary of Guidance for Public Health Strategies to Address High Levels of Community Transmission of SARS-CoV-2 and Related Deaths, December 2020. MMWR. Morbidity and Mortality Weekly Report. 69 (49), 1860-1867 (2020).
- World Health Organization. Surveillance strategies for COVID-19 human infection: interim guidance. World Health Organization. , Available from: https://apps.who.int/iris/handle/10665/332051 (2020).
- Screening testing for early detection of SARS-CoV-2 infection. Division of viral diseases. Centers for Disease Control. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/hcp/tsting-overview.html#PublicHealthSurveillance (2020).
- Larremore, D. B., et al. Test sensitivity is secondary to frequency and turnaround time for COVID-19 screening. Science Advances. 7 (1), 5393(2021).
- Sethuraman, N., Jeremiah, S. S., Ryo, A. Interpreting Diagnostic Tests for SARS-CoV-2. JAMA. 323 (22), 2249-2251 (2020).
- Chu, A. W., et al. Evaluation of simple nucleic acid extraction methods for the detection of SARS-CoV-2 in nasopharyngeal and saliva specimens during global shortage of extraction kits. Journal of clinical virology: the official publication of the Pan American Society for Clinical Virology. 129, 104519(2020).
- Guan, D., et al. Global supply-chain effects of COVID-19 control measures. Nature Human Behaviour. 4 (6), 577-587 (2020).
- Bastos, M. L., Perlman-Arrow, S., Menzies, D., Campbell, J. R. The Sensitivity and Costs of Testing for SARS-CoV-2 Infection With Saliva Versus Nasopharyngeal Swabs: A Systematic Review and Meta-analysis. Annals of Internal Medicine. 174 (4), 501-510 (2021).
- Pasomsub, E., et al. Saliva sample as a non-invasive specimen for the diagnosis of coronavirus disease 2019: a cross-sectional study. Clinical Microbiology and Infection: The Official Publication of The European Society of Clinical Microbiology and Infectious Diseases. 27 (2), (2021).
- To, K. K., et al. Consistent Detection of 2019 Novel Coronavirus in Saliva. Clinical Infectious Diseases: An Official Publication of The Infectious Diseases Society of America. 71 (15), 841-843 (2020).
- Vogels, C. B., et al. SalivaDirect: A simplified and flexible platform to enhance SARS-CoV-2 testing capacity. Med (New York, N.Y.). 2 (3), 263-280 (2021).
- Griesemer, S. B., et al. Evaluation of Specimen Types and Saliva Stabilization Solutions for SARS-CoV-2 Testing. Journal of Clinical Microbiology. 59 (5), 1418-1420 (2021).
- Wehrhahn, M. C., et al. Self-collection: An appropriate alternative during the SARS-CoV-2 pandemic. Journal of Clinical Virology: The Official Publication of The Pan American Society for Clinical Virology. 128, 104417(2020).
- Barza, R., Patel, P., Sabatini, L., Singh, K. Use of a simplified sample processing step without RNA extraction for direct SARS-CoV-2 RT-PCR detection. Journal of Clinical Virology: The Official Publication of the Pan American Society for Clinical Virology. 132, 104587(2020).
- Paltiel, A. D., Zheng, A., Walensky, R. P. Assessment of SARS-CoV-2 Screening Strategies to Permit the Safe Reopening of College Campuses in the United States. JAMA Network Open. 3 (7), 2016818(2020).
- Centers for Disease Control. 2019-Novel Coronavirus (2019-nCoV) Real-Time RT-PCR Primers and Probes. U.S. Department of Health and Human Services. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/lab/rt-pcr-panel-primer-probes.html (2020).
- Cresswell, K., Ramalingam, S., Sheikh, A. Can Robots Improve Testing Capacity for SARS-CoV-2. Journal of Medical Internet Research. 22 (8), 20169(2020).
- Villanueva-Cañas, J. L., et al. Implementation of an open-source robotic platform for SARS-CoV-2 testing by real-time RT-PCR. PLoS One. 16 (7), 0252509(2021).
- Robot Boosts COVID-19 Testing Efficiency. University of South Carolina College of Pharmacy. , Available from: https://sc.edu/study/colleges_schools/pharmacy/about/news/2020/robot-boosts-testing-effort.php (2020).
- Matic, N., et al. Practical challenges to the clinical implementation of saliva for SARS-CoV-2 detection. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of The European Society of Clinical Microbiology. 40 (2), 447-450 (2021).
- Sahajpal, N. S., et al. SalivaSTAT: Direct-PCR and Pooling of Saliva Samples Collected in Healthcare and Community Setting for SARS-CoV-2 Mass Surveillance. Diagnostics. 11 (5), Basel, Switzerland. 904(2021).
- Genzen, J. R., et al. Challenges and Opportunities in Implementing Total Laboratory Automation. Clinical Chemistry. 64 (2), 259-264 (2018).
- Archetti, C., Montanelli, A., Finazzi, D., Caimi, L., Garrafa, E. Clinical Laboratory Automation: A Case Study. Journal of Public Health Research. 6 (1), 881(2017).
- Hecker, K. H., Roux, K. H. High and low annealing temperatures increase both specificity and yield in touchdown and stepdown PCR. BioTechniques. 20 (3), 478-485 (1996).
- Bhat, P. P., et al. Formation of beads-on-a-string structures during break-up of viscoelastic filaments. Nature Physics. 6, 625-631 (2010).
- Miller, C. S., et al. Current developments in salivary diagnostics. Biomarkers in Medicine. 4 (1), 171-189 (2010).
- Moreno-Contreras, J., et al. Saliva Sampling and Its Direct Lysis, an Excellent Option To Increase the Number of SARS-CoV-2 Diagnostic Tests in Settings with Supply Shortages. Journal of Clinical Microbiology. 58 (10), 01659(2020).
- Wang, W., et al. Detection of SARS-CoV-2 in Different Types of Clinical Specimens. JAMA. 323 (18), 1843-1844 (2020).
- Landry, M. L., Criscuolo, J., Peaper, D. R. Challenges in use of saliva for detection of SARS CoV-2 RNA in symptomatic outpatients. Journal of Clinical Virology: The Official Publication of The Pan American Society for Clinical Virology. 130, 104567(2020).
- Lista, M. J., et al. Resilient SARS-CoV-2 diagnostics workflows including viral heat inactivation. PLoS One. 16 (9), 0256813(2021).
- Brotons, P., et al. Validation and implementation of a direct RT-qPCR method for rapid screening of SARS-CoV-2 infection by using non-invasive saliva samples. International Journal of Infectious Diseases: IJID: Official Publication of The International Society for Infectious Diseases. 110, 363-370 (2021).
- Batéjat, C., Grassin, Q., Manuguerra, J. C., Leclercr, I. Heat inactivation of the severe acute respiratory syndrome coronavirus 2. Journal of Biosafety and Biosecurity. 3 (1), 1-3 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved