Method Article
Analyse semi-quantitative du peptidoglycane par chromatographie liquide Spectrométrie de masse et bioinformatique
Dans cet article
Résumé
Ce protocole couvre une analyse détaillée de la composition du peptidoglycane à l’aide de la spectrométrie de masse par chromatographie liquide couplée à un logiciel avancé d’extraction de caractéristiques et d’analyse bioinformatique.
Résumé
Le peptidoglycane est un composant important des parois cellulaires bactériennes et une cible cellulaire commune pour les antimicrobiens. Bien que certains aspects de la structure du peptidoglycane soient assez conservés dans toutes les bactéries, il existe également des variations considérables entre les Gram positifs/négatifs et entre les espèces. En outre, il existe de nombreuses variations, modifications ou adaptations connues du peptidoglycane qui peuvent se produire au sein d’une espèce bactérienne en réponse à une phase de croissance et / ou à des stimuli environnementaux. Ces variations produisent une structure très dynamique qui est connue pour participer à de nombreuses fonctions cellulaires, y compris la croissance / division, la résistance aux antibiotiques et l’évitement de la défense de l’hôte. Pour comprendre la variation au sein du peptidoglycane, la structure globale doit être décomposée en ses parties constitutives (appelées muropeptides) et évaluée pour la composition cellulaire globale. La peptidoglycomique utilise la spectrométrie de masse avancée combinée à une analyse de données bioinformatiques de haute puissance pour examiner la composition du peptidoglycane dans les moindres détails. Le protocole suivant décrit la purification du peptidoglycane à partir de cultures bactériennes, l’acquisition de données d’intensité de muropeptides par chromatographe liquide – spectromètre de masse et l’analyse différentielle de la composition du peptidoglycane à l’aide de la bioinformatique.
Introduction
Le peptidoglycane (PG) est une caractéristique déterminante des bactéries qui sert à maintenir la morphologie cellulaire, tout en fournissant un soutien structurel aux protéines et autres composants cellulaires 1,2. L’épine dorsale du PG est composée d’acide muramique N-acétyl lié au β-1,4-1,4 (MurNAc) et de N-acétyl glucosamine (GlcNAc)1,2. Chaque MurNAc possède un peptide court lié au résidu ᴅ-lactyle qui peut être réticulé avec des peptides adjacents liés au disaccharide (Figure 1A,B). Cette réticulation produit une structure en forme de maille qui englobe toute la cellule et est souvent appelée sacculus (Figure 1C). Au cours de la synthèse du PG, des précurseurs sont générés dans le cytoplasme et transportés à travers la membrane cytoplasmique par des flippases. Les précurseurs sont ensuite incorporés dans le PG mature par les enzymes transglycosylase et transpeptidase, qui produisent respectivementles liaisons glycosidique et peptidique3. Cependant, une fois assemblées, il existe de nombreuses enzymes produites par les bactéries qui modifient et / ou dégradent le PG pour effectuer un certain nombre de processus cellulaires, y compris la croissance et la division. En outre, il a été démontré que diverses modifications du PG confèrent des adaptations spécifiques à la souche, aux conditions de croissance et au stress environnemental, qui ont été impliquées dans la signalisation cellulaire, la résistance aux antimicrobiens et l’évasion immunitaire de l’hôte4. À titre d’exemple, une modification courante est l’ajout d’un groupe acétyle C6 sur le MurNAc qui confère une résistance en limitant l’accès aux liaisons glycane β-1,4 aux enzymes lysozymes produites par l’hôte qui dégradent PG 4,5,6. Chez les entérocoques, la substitution du ᴅ-Ala terminal de la chaîne latérale peptidique par ᴅ-Lac confère une plus grande résistance à l’antimicrobien, la vancomycine 7,8.
La procédure générale d’isolement et de purification des PG est restée relativement inchangée depuis qu’elle a été décrite dans les années 19609. Les membranes bactériennes sont dissoutes par traitement thermique avec SDS, suivi de l’élimination enzymatique des protéines liées, des glycolipides et de l’ADN restant. Le sacculus intact purifié peut ensuite être digéré en composants individuels par hydrolyse de la liaison β-1,4 entre GlcNAc et MurNAc. Cette digestion produit des disaccharides GlcNAc-MurNAc avec toutes les modifications structurelles et / ou réticulations intactes et sont appelés muropeptides (Figure 1B).
L’analyse de la composition du PG a d’abord été réalisée par séparation chromatographique liquide à haute pression (CLHP) pour purifier chaque muropeptide, suivie d’une identification manuelle des muropeptides10,11. Cela a depuis été remplacé par la spectrométrie de masse en tandem par chromatographie liquide (LC-MS), qui augmente la sensibilité de détection et diminue la charge de travail manuelle de purification de chaque muropeptide individuel. Cependant, la nature longue et complexe de l’identification manuelle des muropeptides est restée un facteur limitant, réduisant le nombre d’études menées. Ces dernières années, avec l’émergence des technologies « omiques », l’extraction automatisée de caractéristiques LC-MS est devenue un outil puissant, permettant la détection et l’identification rapides de composés individuels dans des échantillons complexes à partir de très grands ensembles de données. Une fois les caractéristiques identifiées, le logiciel bioinformatique compare statistiquement la variation entre les échantillons à l’aide d’une analyse différentielle isolant même les différences minimes entre les ensembles de données complexes et les affichant graphiquement à l’utilisateur. L’application de logiciels d’extraction de caractéristiques pour l’analyse de la composition PG vient tout juste de commencer à être explorée 12,13,14 et couplée à l’analyse bioinformatique 12. Contrairement à l’analyse protéomique qui bénéficie des bases de données de protéines facilement disponibles qui prédisent la fragmentation peptidique permettant une identification entièrement automatisée, il n’existe actuellement aucune bibliothèque de fragmentation pour la peptidoglycomie. Cependant, l’extraction de caractéristiques peut être couplée à des bases de données structurelles connues et prévues pour prédire l’identification des muropeptides12. Nous présentons ici un protocole détaillé pour l’utilisation de l’extraction de caractéristiques à base de LC-MS combinée à une bibliothèque de muropeptides pour l’identification automatisée et l’analyse différentielle bioinformatique de la composition PG (Figure 2).
Protocole
1. Préparation d’échantillons de peptidoglycane
- Croissance des cultures bactériennes
REMARQUE : La croissance des cultures bactériennes varie en fonction de l’espèce bactérienne et des conditions de croissance examinées. Les paramètres expérimentaux à tester définiront les conditions de croissance.- Cultiver des cultures bactériennes dans les conditions de croissance requises pour la souche bactérienne et la conception expérimentale. Cultiver des bactéries sous forme de cultures triples (répliques biologiques), c’est-à-dire trois colonies distinctes par souche ou condition de croissance.
NOTE: Les conditions de croissance et la phase de croissance sont connues pour avoir des effets significatifs sur la composition du PG 1,2,10. Il faut prendre grand soin de maintenir la cohérence entre les cultures et les répétitions pour s’assurer que les changements de composition sont dus aux paramètres expérimentaux et non à une erreur expérimentale. - Refroidir rapidement la culture à 4 °C, recueillir par centrifugation (11 000 x g, 10 min, 4 °C) et congeler la pastille cellulaire à -20 °C. Laver la pastille de la cellule avec du phosphate de sodium prérefroidi à 4 °C, 20 mM pH 6,5 avant la congélation. La production de la pastille de cellules congelées doit être effectuée aussi rapidement et de manière aussi cohérente que possible entre les échantillons afin de limiter l’activité des enzymes qui pourraient modifier et/ou dégrader le PG pendant le processus de collecte. Les échantillons peuvent être traités directement par le processus d’extraction (section 1.2) sans congélation; Cependant, assurez-vous que tous les échantillons sont traités de la même manière.
REMARQUE : Pour assurer une quantité suffisante de produit pour les étapes ultérieures, une pastille à cellules humides de taille importante est utilisée. Cela produit une pastille de sacculi suffisamment grande, qui est facilement visualisée et entretenue pendant les étapes de lavage répétitives (sections 1.2.5 et 1.2.14) sans perte significative de produit. Selon la bactérie et les conditions de croissance, ce rendement variera probablement. Pour la bactérie à Gram négatif Pseudomonas aeruginosa, PAO1, 4 L d’une culture de 0,5 OD600 ont produit une pastille cellulaire de 3–4 g et étaient suffisantes pour produire ~10 mg de sacculi purifiés (section 1.2.17)12. Il s’agit d’un excès de sacculi important par rapport à ce qui est requis pour la LC-MS (section 2); Cependant, il contribuera à la précision de la mesure (section 1.2.17) et à la normalisation (section 1.3).
- Cultiver des cultures bactériennes dans les conditions de croissance requises pour la souche bactérienne et la conception expérimentale. Cultiver des bactéries sous forme de cultures triples (répliques biologiques), c’est-à-dire trois colonies distinctes par souche ou condition de croissance.
- Extraction de sacculi peptidoglycanes
NOTE: Le protocole d’extraction de PG est adapté de la réf.9,11,15. Ce protocole extraira le PG des cellules bactériennes individuelles comme un sacculi entier, exempt d’autres composants cellulaires. Le protocole peut être utilisé avec des bactéries à Gram négatif ou à Gram positif. Cependant, pour les cellules à Gram positif, des ajustements peuvent être nécessaires pour isoler la structure PG plus épaisse et éliminer les polymères associés à la paroi cellulaire; tels que les acides teichoïques.- Resuspendre les pastilles cellulaires congelées à environ 1:10 du volume de culture initial de 20 mM de phosphate de sodium pH 6,5. Effectuer cette étape à 4 °C (peut représenter 1 à 8 mg de poids de pastilles à cellules humides par mL de tampon11,12).
NOTE: Pour maintenir l’état d’acétylation du PG, un pH de 6,5 ou moins est nécessaire15,16. - Ajouter goutte à goutte la suspension cellulaire au phosphate de sodium de dodécylsulfate (SDS) 20 mM pH 6,5 pour un volume final de 1:1 (c.-à-d. que la concentration finale est de 4 % de SDS), en agitant doucement dans une fiole à fond rond munie d’un condenseur refroidi à l’eau. (Figure 2, étape 2)
- Maintenir une ébullition douce pendant 30 min à 3 h sous agitation pour assurer une dissociation complète de la membrane. Assurez-vous que le mélange résultant est complètement clair, sans amas cellulaires ou viscosité restants. Une ébullition plus longue est préférable pour garantir une dissociation complète.
- Laissez-le refroidir à température ambiante. L’échantillon peut être laissé à température ambiante pendant la nuit.
REMARQUE: Lorsque des FDS sont présentes, conserver les échantillons à température ambiante pour conserver les FDS dans la solution. - Recueillir le sacculi sous forme de pastille par ultracentrifugation à 70 000 x g pendant 40 min (ou le temps nécessaire pour sédimenter complètement les sacculi) à 25 °C.
- Lavage répétitif des sacculi par ultracentrifugation successive (section 1.2.5) et suspension dans ~50 mL de phosphate de sodium à température ambiante 20 mM pH 6,5 jusqu’à ce que le tampon de lavage ait une concentration SDS ~0,001%. En règle générale, 5 à 7 lavages suffisent.
REMARQUE: Pour tester la concentration de la FDS restante dans le tampon de lavage, utilisez le colorant colorimétrique, Stains-all17. - Resuspendre les sacculi dans 5–10 mL de phosphate de sodium à température ambiante 20 mM pH 6,5.
- Sonifier brièvement l’échantillon (~40 %, 50 W, 20 kHz, 20 s) à température ambiante pour disperser les touffes.
REMARQUE: Une sonication prolongée provoquera mécaniquement le cisaillement de la structure PG18. - Compléter l’échantillon avec 50 μg/mL d’amylase, de DNase et de RNase, 10 mM de sulfate de magnésium, et digérer à 37 °C pendant 1 h avec agitation ou nutation.
REMARQUE: La digestion de l’amylase élimine tout glycogène restant piégé dans le sacculi11. - Ajouter 100 μg/mL de pronase et digérer à 37 °C pendant une nuit avec agitation ou nutation et ~0,02 % d’azoture de sodium.
NOTE: La digestion de la pronase élimine les enzymes ajoutées (de la rubrique 1.2.9) et élimine les lipoprotéines liées de manière covalente au PG.
ATTENTION : L’azoture de sodium est hautement toxique et nécessite des méthodes d’utilisation et d’élimination appropriées. - Ultracentrifugeuse à 70 000 x g pendant 40 min (ou le temps nécessaire pour sédimenter complètement les sacculi) à 25 °C pour éliminer l’azoture de sodium.
- Remettez la pastille en suspension dans 25 mL de 2% SDS 20 mM phosphate de sodium pH 6,5.
- Faire bouillir pendant 1 h dans un cuiseur à vapeur ou dans la fiole à fond rond munie d’un réfrigérant refroidi à l’eau (point 1.2.2).
REMARQUE: La deuxième étape d’ébullition SDS élimine toutes les protéines et contaminants restants du sacculi. - Répéter le lavage des sacculi (section 1.2.6) avec ~50 mL d’eau distillée double à température ambiante (ddH2O) jusqu’à ce que la concentration de FDS soit de ~0,001 %.
- Remettez en suspension la pastille dans une quantité suffisante deddH2Opour suspendre les sacculi, lavez le récipient (p. ex. 25 mL) et congelez toute la nuit à -80 °C. Le volume peut varier car l’échantillon sera lyophilisé à l’étape suivante, bien que les petits volumes nécessitent moins de temps pour se lyophiliser.
- Le lendemain, lyophilisez la suspension et conservez à température ambiante.
- Mesurer la quantité de sacculi lyophilisés obtenue sur une balance analytique.
- Diluer les sacculi dans le ddH2O à 10 mg/mL et soniquer brièvement pour briser les amas avant une analyse plus approfondie.
- Resuspendre les pastilles cellulaires congelées à environ 1:10 du volume de culture initial de 20 mM de phosphate de sodium pH 6,5. Effectuer cette étape à 4 °C (peut représenter 1 à 8 mg de poids de pastilles à cellules humides par mL de tampon11,12).
- Quantification du peptidoglycane purifié
- Quantifier la quantité de sacculi purifiés de la section 1.2.18 pour s’assurer que les données sur les spécifications massiques sont égalisées pendant l’analyse différentielle (section 3.2). Suivez la méthodologie détaillée décrite dans la référence15.
NOTE: Les sacculi purifiés (section 1.2.18) sont décomposés en composants individuels de sucre et d’acides aminés par hydrolyse acide. Les composants individuels sont séparés et quantifiés par chromatographie par échange d’anions à l’aide de la détection ampérométrique pulsée. Compte tenu de la structure de PG (Figure 1), les muropeptides individuels sont composés d’un seul résidu MurNAc et d’un seul résidu GlcNAc. Par conséquent, la quantification de la concentration de l’un ou l’autre résidu représente la quantité de muropeptides 1:1 dans l’échantillon. MurNAc est préféré en raison de la séparation nette des pics des autres composants PG pendant la chromatographie16.
- Quantifier la quantité de sacculi purifiés de la section 1.2.18 pour s’assurer que les données sur les spécifications massiques sont égalisées pendant l’analyse différentielle (section 3.2). Suivez la méthodologie détaillée décrite dans la référence15.
2. Acquisition de données de spectrométrie de masse
- Préparation de muropeptides pour la spectrométrie de masse
- Compléter 800 μg de sacculi purifiés avec 100 μg/mL de mutanolysine, 100 mM d’acétate d’ammonium pH 5,5 et 50 mM de chlorure de magnésium dans une réaction de 100 μL.
- Digestion à 37 °C pendant la nuit.
- Ajouter 1:1 volume 0,5 M tampon de borate pH 9,0 et compléter avec ~10 mg/mL de borohydrure de sodium (NaBH4).
NOTE: La mutarotation des sucres cycliques entre les formes α et β anomères provoquera de multiples formations de pics lors de la séparation par chromatographie liquide des muropeptides. Le traitement avec NaBH4 élimine l’interconversion entre les deux formes en réduisant MurNAc en muramitol11. Le traitement ne réduit pas les résidus de sucre 1,6-anhydro MurNAc ou 1,4-liés.
ATTENTION : La réaction duNaBH4 produit de petites quantités d’hydrogène gazeux. La réaction NaBH4 créera des bulles et les tubes de microfuge doivent être maintenus ouverts pour permettre au gaz de s’échapper. - Incuber l’échantillon à température ambiante pendant ~20–30 min.
- Centrifuger brièvement pour déposer l’échantillon dans le tube de microfuge et éliminer les bulles.
- Ajuster le pH à <4,0 en utilisant de l’acide phosphorique 1:5, ajouté par incréments de 5 μL. Testez le pH à l’aide de bandelettes de test de pH décisif.
- Centrifuger à ~17 000 x g pendant 1 min pour sédimenter toute matière insoluble restante.
- Filtre à l’aide de filtres microcentrifuges de 0,2 μm.
- Les échantillons sont centrifugés pendant 10 minutes à 30 000 x g avant l’injection dans la LC-MS pour s’assurer que les particules ne sont pas injectées dans la SEP.
- Configuration de LC-MS
- Fixer une colonne de particules superficiellement poreuses C18 (100 mm x 2,1 mm, taille des pores <3 μm) à un spectromètre de masse QTF, quadripôle, avec une précision de détection m/z minimale à quatre décimales.
- Effectuer la séparation par chromatographie liquide des muropeptides
NOTA: Chaque triple biologique (section 1.1.1) doit être passé trois fois par la LC-MS (sections 2.2.2 à 2.2.3) (triplat technique). Par conséquent, chaque condition testée aura un total de neuf fichiers de données LC-MS. L’acquisition des données a été effectuée à l’aide de logiciels disponibles dans le commerce (voir le tableau des matières). Cependant, le logiciel d’acquisition doit être choisi en fonction des machines EM. Ce qui suit représente un guide pour la configuration du MS avec des paramètres spécifiques pour l’exécution de ce protocole. Pour une description détaillée, veuillez vous référer au manuel du fabricant.- Pour la séparation chromatographique, préparer les solvants suivants à 0,1 % d’acide formique (A) et d’acétonitrile avec 0,1 % d’acide formique (B).
- Mettre en place une méthode de séparation chromatographique en utilisant les paramètres suivants.
- Réglez le débit sur 0,4 mL/min.
- Conditionnez la colonne pendant 10 min à 2 % B (~24 volumes de colonne).
- À l’aide d’un échantillonneur automatique, injecter 10 μL d’échantillon de la section 2.1.9.
REMARQUE : Exécutez un échantillon initial via le protocole LC-MS avant de commencer la collecte de données. Cette exécution n’est pas utilisée pour les données, mais pour augmenter la reproductibilité du temps de rétention pour toutes les exécutions suivantes. La reproductibilité du temps de rétention est requise pendant le traitement spectral (section 3.1) pour l’identification et le regroupement précis des pics du rapport masse/charge (m/z). - Séparer les muropeptides en utilisant 2% B pendant 5 min (~12 volumes de colonne), puis l’augmenter à 15% B sur 13 min (~30 volumes de colonne), l’augmenter à 50% B sur 10 min (~24 volumes de colonne), et enfin l’augmenter à 98% B sur 2 min (~5 volumes de colonne).
REMARQUE: Jeter (envoyer aux déchets) les 2 premières minutes et les 5 dernières minutes de la pente. - Terminez par un lavage de colonne à 98 % B pendant un rééquilibrage de 6 min (~14 volumes de colonne) et de 20 min (~47 volumes de colonne).
- Effectuer la détection par spectrométrie de masse des muropeptides
- Étalonner l’axe de masse en mode positif à l’aide d’un mélange de réglage dans de l’acétonitrile contenant des étalons de masse de référence LC-MS en suivant les instructions du fabricant MS.
NOTE: Le MS est réglé avant le début des cycles chromatographiques (section 2.2.2.1). - Configurez une méthode d’acquisition de données MS à l’aide des paramètres suivants.
NOTE: Les données MS et MS/MS sont collectées (sections 2.2.3.2 à 2.2.3.6) simultanément à la séparation chromatographique des muropeptides (sections 2.2.2.4 et 2.2.2.5). Les paramètres chromatographiques et MS (sections 2.2.2.2 et 2.2.3.2) sont une méthode unique qui est ajoutée lors de la configuration d’une liste de travail pour exécuter plusieurs échantillons en séquence. - Réglez la tension capillaire de l’électropulvérisation à 4,0 kV et la température du gaz de séchage à 350 ° C avec un débit de 13 L / min.
NOTA : L’azote (pureté >99 %) doit être utilisé comme gaz de nébulisation, de séchage et de collision pendant toute collecte de données par spectrométrie de masse. - Réglez la pression du nébuliseur à 40 psi et réglez le fragmenteur sur 150 V.
- Réglez les tensions RF de la buse, de l’écumeur et de l’octapôle sur 1000 V, 65 V et 750 V, respectivement.
- Réglez la plage de balayage sur 300–2 000 m/z en mode ion positif 4 GHz (plage dynamique étendue).
- Définissez la collecte de données en utilisant l’acquisition MS/MS dépendante des données avec une fréquence de balayage MS et MS/MS de 1 spectre/seconde. Sélectionnez cinq masses de précurseurs par cycle, dans l’ordre de charge simple, double et triple.
- Réglez les énergies de collision de fragmentation MS/MS à 15, 20 et 30 eV.
- Étalonner l’axe de masse en mode positif à l’aide d’un mélange de réglage dans de l’acétonitrile contenant des étalons de masse de référence LC-MS en suivant les instructions du fabricant MS.
3. Analyse différentielle de l’abondance des muropeptides
- Traitement spectral du chromatogramme LC-MS
REMARQUE : L’extraction récursive des caractéristiques a été effectuée à l’aide d’un logiciel disponible dans le commerce (voir le tableau des matières). D’autres logiciels d’extraction de fonctionnalités peuvent être utilisés. Cependant, d’autres logiciels peuvent nécessiter un traitement manuel supplémentaire des données pour effectuer l’extraction récursive très robuste. Divers logiciels utilisent la fonction terminologique, l’entité et le composé de manière presque interchangeable. Pour l’analyse PG, tous se réfèrent à l’identification du chromatogramme ionique LC-MS représentatif d’un muropeptide individuel (p. ex., figure 3). Au cours du traitement spectral (section 3.1), une caractéristique représente les multiples pics m/z qui englobent les multiples espèces ioniques possibles d’un seul muropeptide qui sont regroupées sous un seul marqueur composé.- Sous Fichier, démarrez un nouveau projet.
- Ajoutez les fichiers de données LC-MS QTOF et attribuez des fichiers de données individuels à une condition / groupe expérimental, par exemple, deux conditions de croissance différentes.
- Exécutez l’assistant de traitement des données Extraction de caractéristiques récursives par lots (petites molécules/peptides) et définissez les filtres de traitement des données pour qu’ils correspondent aux paramètres des conditions LC-MS et à l’instrumentation pour identifier, regrouper et vérifier avec précision les pics m/z représentant des muropeptides individuels. À partir du chromatogramme, examiner la dérive du temps de rétention et la variation de m/z de pics similaires connus pour définir les paramètres de filtre initiaux.
REMARQUE : L’extraction de caractéristiques récursives utilise un algorithme initial d’extraction de caractéristiques moléculaires non ciblées pour identifier et aligner les caractéristiques du chromatogramme (pics m/z ) dans tous les fichiers de données. Une fois créées, ces entités sont utilisées pour réévaluer les fichiers de données originaux (récursifs) avec un algorithme d’extraction de caractéristiques moléculaires ciblées afin d’améliorer la fiabilité et la précision des caractéristiques identifiées. Il est préférable de définir l’extraction initiale non ciblée avec une fenêtre de détection étroite et d’utiliser des paramètres de détection plus larges pour l’extraction récursive afin d’identifier les pics dans tous les fichiers de données qui peuvent manquer lors de l’extraction initiale. L’exécution de l’extraction récursive des fonctionnalités peut prendre des heures en fonction du nombre d’échantillons, de la complexité des données et du matériel informatique présent - Passez en revue les résultats de l’extraction des fonctionnalités. Si un nombre important d’entités n’ont pas réussi à s’aligner dans un groupe, ajustez les paramètres de filtrage récursifs pour élargir/restreindre la fenêtre de détection selon vos besoins. Pour ce faire, inspectez le chromatogramme et le profil isotopique de chaque caractéristique extraite pour vous assurer que la détection des caractéristiques a été effectuée de la même manière dans tous les fichiers de données. En outre, pour chaque entité identifiée dans un fichier de données, examinez le score, les avertissements éventuels et si la fonctionnalité a réussi les paramètres de filtre choisis.
Remarque : Il faut veiller à garder les fenêtres de détection suffisamment étroites pour que les fonctionnalités distinctes ne soient pas regroupées par erreur. Ceci est souvent noté par des profils isotopiques disparates entre les fichiers de données, ou des variations significatives m/z / temps de rétention. Inversement, s’il existe plusieurs caractéristiques avec des temps de rétention m/z et similaires, il est possible que les paramètres de filtre aient été trop stricts, ce qui a entraîné la division d’un pic MS en deux caractéristiques. Par conséquent, exécutez à nouveau l’extraction des entités (section 3.1.3) avec des paramètres de filtre de temps de rétention ajustés pour permettre un meilleur regroupement de ces entités. La visualisation des pics d’abondance les plus faibles indiquera si les filtres utilisés (section 3.1.3) ont identifié avec précision les pics au-dessus du bruit de fond. Si les pics d’abondance les plus faibles semblent similaires à l’arrière-plan, réexécutez l’extraction d’entités avec de nouveaux paramètres de filtre d’arrière-plan. - Exportez les données sous la forme d’un fichier d’échange composé (.cef) (un format compatible avec le logiciel statistique) ou d’un fichier séparé par colonne (.csv) contenant la masse, le temps de rétention et l’abondance de chaque caractéristique pour chaque échantillon.
- Analyse différentielle des caractéristiques spectrales
REMARQUE : L’analyse différentielle a été effectuée à l’aide d’un logiciel disponible dans le commerce (voir le tableau des matières). D’autres logiciels bioinformatiques peuvent être utilisés.- Ouvrez le programme et, lorsque vous y êtes invité, démarrez un nouveau projet.
- Suivez les instructions pour l’importation et l’analyse des données. Lors de l’importation des données, téléchargez les fichiers extraits de la fonctionnalité (section 3.1). Au cours de l’analyse des données, choisissez la signification et le changement de pli pour l’analyse différentielle et sélectionnez les données de référence à l’intensité médiane dans tous les fichiers de données. Ne définissez pas de filtres de données (si cela a été fait lors de l’étape précédente de traitement des spectres (section 3.1)) car l’application de filtres annulerait l’extraction robuste des caractéristiques récursives. Cependant, comme pour l’extraction de caractéristiques, l’analyse différentielle doit être alignée en fonction de la masse (m/z) et du temps de rétention en raison de la dérive dans la collecte de données LC-MS. Utilisez les paramètres déterminés dans l’extraction de caractéristiques (section 3.1).
REMARQUE : Normaliser entre les fichiers de données à l’aide de la quantification des muropeptides (section 1.3) pour s’assurer que les variations sont dues à des paramètres expérimentaux et non à des variations dans la purification des sacculi (section 1.2). Utilisez l’option de mesureur externe pour ajuster chaque fichier de données en fonction des différences de concentration de MurNAc dans l’échantillon. - Une fois l’analyse terminée, examiner les analyses graphiques et statistiques résultantes pour identifier les muropeptides qui démontrent un changement d’abondance significatif entre les conditions expérimentales testées.
- Sous le navigateur de projet, cliquez avec le bouton droit sur les différentes analyses et choisissez une option d’exportation pour enregistrer les détails de l’entité sous la forme d’un fichier de données séparé par colonne (.csv) qui contient le m/z, le temps de rétention, les valeurs d’intensité brutes et normalisées, la valeur p, le FDR et les changements de pli pour chaque entité. Plusieurs analyses doivent être sauvegardées pour obtenir toutes les données pertinentes.
- Exportez un deuxième fichier .csv contenant uniquement les muropeptides qui ont dépassé les analyses statistiques, y compris la valeur p <0,05 et le changement de pli >2.
- Annoter l’identité du muropeptide aux caractéristiques spectrales
NOTE: Chaque caractéristique identifiée doit se voir attribuer une structure muropeptidique prédite basée sur le m / z et cette annotation confirmée par l’examen de la fragmentation MS / MS. Après confirmation des annotations, il peut être nécessaire d’effectuer et d’affiner l’analyse différentielle (section 3.2).- Dans le logiciel d’analyse différentielle, sous Interprétation des résultats, sélectionnez ID browser. Ajouter une bibliothèque de structures muropeptidiques attendues et sélectionner des paramètres similaires à ceux utilisés précédemment (section 3.1.3). Cela produira une annotation muropeptide prédite pour chaque caractéristique identifiée. Une bibliothèque de structures muropeptidiques peut être produite à l’aide du m/z pour les structures muropeptidiques prévues et du logiciel de base de données MS (voir le tableau des matériaux). Cependant, une bibliothèque des m /z de >6 000 muropeptides possibles peut être trouvée dans la référence12.
- Sélectionnez l’annotation du muropeptide prédit en fonction du score correspondant et de la pertinence biologique du muropeptide prédit, c’est-à-dire choisissez le muropeptide le plus susceptible d’être présent dans l’échantillon biologique.
- Confirmer manuellement l’annotation prédite du muropeptide en comparant les pics m /z du chromatogramme MS/MS au m/z prédit de toutes les fragmentations possibles d’une structure muropeptidique connue (p. ex., figure 4).
- Visualisez les données MS et MS/MS à l’aide d’un programme de visualisation par chromatogramme (voir le tableau des matériaux, figure 4).
- Dessinez la structure prédite du muropeptide à l’aide d’un éditeur moléculaire (programme de dessin de structure chimique) (voir le tableau des matériaux, figure 4, encart gris). Utilisez l’outil de fragmentation de masse pour afficher le m/z des fragments MS lorsque chaque liaison est rompue individuellement ou en combinaison.
REMARQUE: Selon l’énergie de fragmentation utilisée pour la SEP / SEP, la fragmentation peut se produire à n’importe quelle liaison dans la structure du muropeptide. Cependant, certaines liaisons sont plus facilement / fréquemment fragmentées à des niveaux d’énergie plus faibles. Par exemple, la fragmentation dans la chaîne latérale peptidique se produit le plus souvent au niveau de la liaison amide entre les acides aminés. Lors de l’évaluation de la fragmentation, il est important de noter que les résidus de GlcNAc sont très facilement fragmentés à partir du muropeptide. Par conséquent, la fragmentation de la structure muropeptidique connue doit être évaluée avec et sans GlcNAc. En raison de la fragmentation du GlcNAc à la source, plusieurs caractéristiques extraites dans le traitement spectral (section 3.1) peuvent représenter une structure muropeptidique unique. Si elles sont trouvées, ces caractéristiques doivent être fusionnées et l’analyse différentielle réévaluée. - Comparer toutes les fragmentations possibles de la structure du muropeptide qui a été déterminée (section 3.3.3.2) au chromatogramme MS/MS (section 3.3.3.1). Pour confirmer l’annotation du muropeptide, les pics m /z de fragments multiples doivent être trouvés dans le chromatogramme MS/MS avec une fenêtre d’alignement m/z très minimale (Figure 4).
- Afin d’élucider l’identité des muropeptides en cas de fragmentation ambiguë de la SM/SM, répéter les sections 2.2.2 à 2.2.3 avec des échantillons (section 2.1.9) pour l’acquisition de données MS/MS supplémentaires incorporant une liste précurseur préférée de m/z et un temps de rétention avec des énergies de collision de fragmentation MS/MS supplémentaires.
- Pour les entités co-éluantes qui ont été annotées comme le même muropeptide, exécutez à nouveau l’analyse différentielle (section 3.2.2) et fusionnez les caractéristiques extraites.
- Évaluation des changements globaux dans les modifications des muropeptides
- Modifier le fichier .csv (section 3.2.4) des muropeptides à variation de pli élevé statistiquement significatif afin d’inclure une seule colonne pour chaque modification de muropeptide. Remplissez cette colonne avec une désignation pour chaque muropeptide annoté (section 3.3) (p. ex., GlcNAc ou MurNAc acétylé versus dé-N-acétylé).
- Téléchargez le fichier .csv modifié dans Perseus22,23. Importez les valeurs d’intensité normalisées dans la zone Importation principale et importez la désignation de modification dans la zone Importation catégorielle.
- Sous Annoter les lignes, cliquez sur Annoter catégoriquement les lignes et ajoutez des fichiers de données à chaque paramètre expérimental.
- Sous test, cliquez sur deux exemples de tests pour effectuer le test t d’un élève (valeur p < 0,05, FDR < 0,05, s0 = 1).
- Cliquez sur 1D pour effectuer l’annotation 1D22,23. Une annotation FDR 1D < 0,05 indique un changement d’abondance significatif pour la modification du muropeptide entre les paramètres expérimentaux testés. La définition de la valeur de seuil (s0) = 1 affichera les scores FDR d’annotation 1D pour tous les muropeptides.
- Dans un logiciel graphique (voir le tableau des matériaux), produire une carte thermique du changement de pli d’abondance pour chaque modification du muropeptide et montrer le score d’annotation 1D pour démontrer la signification (Figure 5B). Le changement de pli de chaque modification de muropeptide peut être produit dans Microsoft Excel en utilisant les intensités brutes de tous les muropeptides individuels qui contiennent la modification.
Résultats
La sensibilité accrue de détection des machines MS associée à un logiciel de reconnaissance de pics très puissant a amélioré la capacité d’isoler, de surveiller et d’analyser les compositions de substances d’échantillons complexes dans les moindres détails. Grâce à ces avancées technologiques, des études récentes sur la composition du peptidoglycane ont commencé à utiliser des techniques automatisées d’extraction de caractéristiques LC-MS 12,13,14,24 par rapport à l’ancienne méthodologie basée sur HPLC 11,25,26,27,28,29,30,31 . Bien qu’il existe de nombreux progiciels génériques d’extraction de caractéristiques disponibles, les logiciels commerciaux utilisant l’extraction de caractéristiques récursives sont rapides et très robustes en identifiant et en combinant automatiquement toutes les charges, les isotopes et les versions adduits de chaque muropeptide trouvé dans l’ensemble de données LC-MS (Figure 3). En outre, les temps de rétention initiaux, les modèles m/z et isotopiques des entités extraites sont utilisés pour réévaluer (récursivement) l’ensemble de données afin d’assurer une identification précise de chaque entité dans tous les fichiers de données. Par conséquent, l’algorithme récursif aide à valider et à augmenter la confiance dans l’identification des pics. La plupart des programmes génériques d’extraction de caractéristiques ne regroupent pas les charges/isotopes, etc. et nécessitera cela comme une étape manuelle supplémentaire. En outre, les programmes génériques seront moins robustes car les entités sont extraites séparément dans chaque fichier de données et non comme un jeu de données entier, qui fait partie de l’algorithme récursif.
Le protocole peptidoglycomique présenté ici a récemment été utilisé pour examiner les changements de composition de PG entre deux conditions de croissance physiologique, à savoir le biofilm planctonique en nage libre et le biofilm communstationnaire 12. En utilisant un QTOF MS très sensible couplé à l’extraction de caractéristiques récursives, 160 muropeptides distincts ont été reconnus et suivis. Cela représentait huit fois le nombre de muropeptides identifiés dans cet organisme précédemment 29,32, et plus du double des muropeptides identifiés à l’aide d’autres méthodologies dans d’autres organismes10,14,24.
L’association de chaque pic m/z extrait des données MS à un muropeptide particulier est facilitée par le croisement avec une base de données de structures muropeptidiques connues et prévues. Le chromatogramme MS/MS de fragmentation (Figure 4) pour chaque caractéristique extraite est comparé au profil de fragmentation (Figure 4, encart gris) du muropeptide proposé à l’aide de la base de données.
Les données peptidoglycomiques peuvent être visualisées de différentes manières en fonction de la configuration expérimentale et des questions posées. Une telle analyse graphique peut inclure l’analyse en composantes principales (ACP), les nuages de points, les diagrammes de volcans, les cartes thermiques et l’analyse hiérarchique de clustering. Par exemple, les diagrammes de volcans mettent en évidence des muropeptides qui démontrent une ampleur statistiquement significative de changement d’abondance entre les conditions testées (Figure 5A). Ces muropeptides sélectionnés qui représentent des changements d’abondance significatifs entre les conditions testées peuvent être examinés plus avant pour les modifications des muropeptides. Ces modifications peuvent inclure la présence de substitutions d’acides aminés, de changements d’acétylation ou de présence d’activité amidase. Lorsqu’ils sont examinés ensemble, plusieurs muropeptides possédant la même modification peuvent être examinés pour une tendance vers une condition expérimentale (Figure 5A - points surlignés en vert) et l’ensemble du groupe évalué pour la signification (Figure 5B). Le suivi d’une modification muropeptidique de cette manière peut indiquer une activité enzymatique particulière affectée par le paramètre expérimental. De plus, les valeurs aberrantes de cette tendance peuvent indiquer une activité enzymatique ayant une spécificité ou une fonction biologique particulière (figure 5A – points angulaires surlignés).

Figure 1 : Exemple d’une structure typique de peptidoglycane à Gram négatif. (A) Chez les bactéries à Gram négatif, le peptidoglycane est situé dans le périplasme entre les membranes interne et externe. (B) Un muropeptide unique est constitué d’une N-acétyl glucosamine liée à la β-1,4 (GlcNAc) (bleu) et d’un acide muramique N-acétyle (MurNAc) (violet) avec une chaîne latérale peptidique annexée (orange). La chaîne latérale peptidique peut être réticulée à la chaîne latérale du muropeptide adjacent produisant le peptidoglycane mature de type maille (A). La purification implique l’isolement du peptidoglycane de la cellule entière sous forme de sacculus où tout autre matériel cellulaire a été enlevé. (C) Micrographie électronique à transmission d’un sacculi peptidoglycane. En comparaison, le PG à Gram positif peut consister en un plus grand éventail de variations de structure et fait partie de la classification taxonomique à Gram positif33. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Flux de travail de la peptidoglycomique. Préparation des échantillons. Étape 1, cultiver et granuler des cellules bactériennes (section 1.1). Étape 2, purifier les sacculi peptidoglycanes par ébullition SDS à 4% (rubrique 1.2). Acquisition de données. Étape 3, digestion enzymatique des sacculi pour produire des muropeptides par rupture de la liaison β-1,4 entre la N-acétylglucosamine (GlcNAc) et l’acide N-acétylmuramique (MurNAc) du squelette peptidoglycane (rubrique 2.1). Étape 4, analyse de l’intensité des muropeptides par LC-MS/MS (rubrique 2.2). Analyse des données. Étape 5, l’extraction récursive des caractéristiques identifie et recueille toutes les charges, adduits et isotopes associés à un seul muropeptide (rubrique 3.1). Étape 6, identification des muropeptides en comparant la fragmentation prévue avec les chromatogrammes MS/MS (rubrique 3.3). Étape 7, analyse différentielle bioinformatique (section 3.2) comparant les changements de composition du peptidoglycane entre différents paramètres expérimentaux. Étape 8, examiner le changement global des modifications des muropeptides dans les différents paramètres expérimentaux à l’aide de l’annotation 1D (section 3.4). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Exemple d’extraction d’entités récursives. Pour un muropeptide représentant une chaîne latérale peptidique de l’alanine (A), de l’iso-ᴅ-glutamate (E), de l’acide méso-diaminopimélique (m), de l’alanine (A) réticulée à l’AE m A de la chaîne latérale muropeptidique adjacente (1864,8 m/z). La caractéristique extraite pour 1864,8 m/z comprend des charges (+1, +2 et +3), des adduits (p. ex., sodium et potassium), une perte de GlcNAc (1 ou 2 GlcNAc) et plusieurs pics isotopiques pour chaque variation (p. ex., encart zoomé). Veuillez cliquer ici pour voir une version agrandie de cette figure.
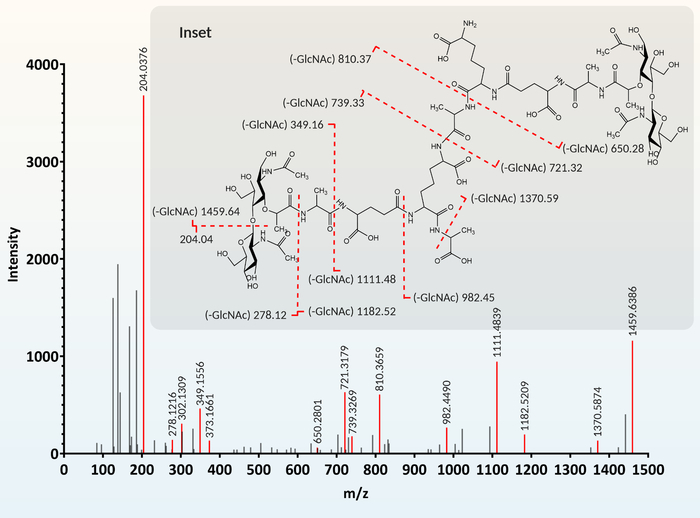
Figure 4 : Fragmentation et identification des muropeptides. Pour l’annotation, chaque pic m/z (caractéristique) extrait du chromatogramme MS reçoit une structure muropeptidique proposée basée sur la similitude avec une bibliothèque de muropeptides. Pour confirmer cette structure proposée, les fragments MS/MS prévus sont générés à l’aide d’un programme de dessin chimique (encart gris). Cette fragmentation prédite est comparée au chromatogramme MS/MS. Lorsque les fragments prédits (encart gris) correspondent au chromatogramme MS/MS, la structure du muropeptide proposée est confirmée. La figure a été modifiée à partir de la référence12. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Analyse différentielle de la composition du peptidoglycane. (A) Tracé du volcan du changement de pli et de la signification statistique des changements d’intensité du muropeptide entre peptidoglycane purifié à partir de P. aeruginosa cultivé en culture planctonique ou stationnaire en biofilm. Tous les muropeptides qui ont une modification qui représentait un changement dans l’arrangement typique des acides aminés dans la chaîne latérale peptidique sont mis en évidence. Les muropeptides substitués par des acides aminés qui ont montré une tendance à la diminution de l’abondance du peptidoglycane dérivé du biofilm sont mis en évidence en vert. Les muropeptides substitués par des acides aminés qui étaient des valeurs aberrantes à cette tendance et ont montré une abondance accrue dans le peptidoglycane dérivé du biofilm sont mis en évidence en orange. (B) Carte thermique du changement global du pli de l’abondance de tous les muropeptides substitués par des acides aminés avec une abondance accrue (orange) et une abondance diminuée (vert) dans les biofilms. Ces muropeptides ont été regroupés et évalués pour déterminer si la substitution d’acides aminés s’était produite sur des monomères, des dimères réticulés, ou si le quatrième (AE m +), le cinquième (AE m A +) ou les deux acides aminés (AEm ++) ont été substitués. La signification de chaque groupe de muropeptides a été évaluée par annotation 1D avec FDR < 0,05 pour la signification et le score 1D associé est affiché. L’annotation 1D ne peut être effectuée que sur plus de 2 muropeptides (par exemple, la substitution AEm++ n’a été trouvée que sur deux muropeptides). Par conséquent, dans ce cas, la signification doit être examinée pour les muropeptides individuels et non sur le groupe. La figure a été modifiée à partir de la référence12. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Ce protocole décrit une méthode pour purifier le peptidoglycane à partir de cultures bactériennes, un procédé de détection LC-MS et une analyse de composition à l’aide de techniques bioinformatiques. Ici, nous nous concentrons sur les bactéries à Gram négatif et quelques légères modifications seront nécessaires pour permettre l’analyse des bactéries à Gram positif.
La préparation des muropeptides est restée pratiquement la même depuis sa première production dans les années 1960 9,11,15. Une fois purifiés, les sacculi (rubrique 1.2.18) sont digérés en muropeptides individuels à l’aide de l’enzyme muramidase mutanolysine de Streptomyces globisporus. La mutanolysine digère la structure PG en brisant la liaison β-1,4-glycosidique libérant des muropeptides individuels constitués d’un disaccharide GlcNAc-MurNAc avec une chaîne latérale peptidique ajoutée et comprend toute modification ou réticulation (Figure 1).
Une limite de la méthodologie précédente utilisée pour étudier la composition du PG a été l’identification manuelle fastidieuse des muropeptides. En raison de la complexité et de la difficulté, les adduits, les charges et/ou les isotopes peuvent ou non avoir été inclus dans l’analyse. En outre, la plupart des études ont limité l’analyse aux muropeptides les plus abondants, donc les plus faciles à purifier. Par conséquent, en raison de la nature complexe de la méthodologie, relativement peu d’analyses détaillées de haut niveau de la composition du GE ont été effectuées. Les analyses de type « omique » ont utilisé des améliorations technologiques récentes pour la production et l’analyse statistique d’ensembles de données LC-MS relativement volumineux et complexes pour la vue d’ensemble de haut niveau des systèmes biologiques. L’application de peptidoglycomique permettra d’analyser la composition PG dans les moindres détails.
Dans la peptidoglycomie, l’extraction récursive des caractéristiques réduit la charge de travail manuelle et augmente la précision en examinant tous les fichiers de données à la fois. Un algorithme d’extraction de caractéristiques récursives est utilisé pour identifier, aligner et regrouper des caractéristiques spectrales uniques (pics m/ z) dans plusieurs fichiers de données chromatographiques LC-MS, ce qui automatise l’identification des pics muropeptides m/z . Cet algorithme utilise l’appariement de motifs isotopiques qui prend les nombreux isotopes potentiels, adduits ioniques et états de charge et condense les multiples pics m / z en son composé unique représentatif (ou caractéristique), qui dans ce cas représenterait un seul muropeptide (Figure 3). La vérification du groupe d’entités spectrales est effectuée en comparant le temps de rétention, m/z et l’appariement isotopique dans chaque fichier de données chromatographiques afin d’assurer une extraction robuste de la caractéristique dans l’ensemble de données. Les algorithmes génériques de recherche de caractéristiques peuvent ne pas inclure la correspondance isotopique ou l’alignement, le regroupement ou la vérification des pics m/z sur plusieurs échantillons et nécessiteront un traitement manuel supplémentaire des données pour effectuer cette extraction de caractéristiques.
Une fois les caractéristiques identifiées, les algorithmes d’analyse différentielle bioinformatique traitent le très grand ensemble de données dans son ensemble, permettant ainsi des comparaisons et des interprétations utiles à partir des données complexes. L’utilisation de ces analyses graphiques bioinformatiques est un moyen puissant de visualiser et d’interpréter de grands ensembles de données afin d’examiner les tendances qui peuvent indiquer des processus biologiques. Ce n’est que récemment que ces analyses graphiques de haute puissance ont été utilisées pour examiner le peptidoglycane dans les moindres détails12. L’analyse différentielle (section 3.2) évalue les changements d’abondance des muropeptides individuels entre différentes conditions expérimentales. Cependant, dans le contexte de cellules bactériennes entières, l’activité des enzymes modificateurs PG pourrait entraîner de multiples structures muropeptidiques distinctes en fonction de la spécificité de l’activité catalytique (c.-à-d. l’ajout d’un groupe acétyle sur le disaccharide pourrait se faire avec ou sans modification de la chaîne latérale peptidique). Par conséquent, l’évaluation des changements d’abondance globale d’une modification particulière sur tous les muropeptides annotés individuels donnera un aperçu de l’activité enzymatique agissant sur le PG (Figure 5) Par conséquent, l’analyse différentielle est utilisée pour étudier les changements d’abondance des muropeptides individuels; tandis que l’annotation 1D examine les changements d’abondance d’une modification PG particulière. L’analyse différentielle de couplage avec annotation 1D permet d’évaluer la composition PG à la fois au niveau du muropeptide individuel et également en tant qu’indicateur de l’activité enzymatique globale du PG.
Lors de l’analyse différentielle, il est important de noter que PG est composé de quelques muropeptides très abondants et de nombreux muropeptides de faible abondance12. Par conséquent, le niveau de référence est très important afin d’éliminer tout biais des muropeptides à haute abondance au cours des étapes ultérieures de l’analyse. De plus, en raison des multiples tests t effectués, une correction statistique pour réduire les faux positifs doit être appliquée. La valeur par défaut est souvent le taux de fausses découvertes (FDR)19 de Benjamini-Hochberg. D’autres corrections telles que le taux d’erreur familial Bonferroni (FWER)20,21 plus conservateur sont possibles.
Dans le logiciel bioinformatique, le pic m / z identifié dans l’extraction de caractéristiques se voit également attribuer une structure prédite. D’autres analyses de type « omique » (p. ex. protéomique) bénéficient de la disponibilité de grandes bases de données sur les composés, qui permettent l’identification des composés grâce à l’appariement prédictif des spectres de fragmentation. Actuellement, il n’existe pas de bibliothèque de fragmentation prédite par muropeptide et la confirmation de l’identification des muropeptides reste une étape manuelle. Cependant, à mesure que les bases de données de fragmentation peptidoglycomique se développeront et deviendront accessibles au public, cette étape d’identification manuelle deviendra plus automatisée et accessible en éliminant ou en réduisant fortement les sections 3.3.3 et 3.3.4.
Chez Escherichia coli , PG est constitué de ~3,5 x 106 muropeptides par cellule34. Dans les limites de détection du QTOF MS, même les muropeptides les plus abondants les plus faibles peuvent encore représenter des centaines de copies d’un seul muropeptide dans une cellule12. Par conséquent, la compréhension des changements apportés même aux muropeptides les plus abondants les plus bas peut fournir des informations utiles sur l’activité biologique des enzymes ciblant les PG dans la cellule.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Les auteurs aimeraient remercier la Dre Jennifer Geddes-McAlister et le Dr Anthony Clarke pour leur contribution au perfectionnement de ce protocole. Ces travaux ont été appuyés par des subventions de fonctionnement des IRSC accordées au C.M.K (PJT 156111) et une BESC Alexander Graham Bell du CRSNG décernée à l’EMA. Des figures ont été créées le BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| C18 reverse phase column - AdvanceBio Peptide column (100 mm x 2.1 mm 2.7 µm) | Agilent | LC-MS data acquisition | |
| Heating mantle controller, Optichem | Fisher | 50-401-788 | for 4% SDS boil |
| Heating Mantle, 1000mL Hemispherical | Fisher | CG1000008 | for 4% SDS boil |
| Incubator, 37°C | for sacculi purification and MS sample prep | ||
| Leibig condenser, 300MM 24/40, | Fisher | CG121805 | for 4% SDS boil |
| Lyophilizer | Labconco | for lyophilization of sacculi | |
| Magentic stirrer | Fisher | 90-691-18 | for 4% SDS boil |
| mass spectrometer Q-Tof model UHD 6530 | Aglient | LC-MS data acquisition | |
| microcentrifuge filters, Nanosep MF 0.2 µm | Fisher | 50-197-9573 | cleanup of sample before MS injection |
| Retort stand | Fisher | 12-000-102 | for 4% SDS boil |
| Retort clamp | Fisher | S02629 | for 4% SDS boil |
| round bottom flask - 1 liter pyrex | Fisher | 07-250-084 | for 4% SDS boil |
| Sonicator model 120 | Fisher | FB120 | for sacculi purification |
| Sonicator - micro tip | Fisher | FB4422 | for sacculi purification |
| Ultracentrifuge | Beckman | sacculi wash steps | |
| Ultracentrifuge bottles, Ti45 | Fisher | NC9691797 | sacculi wash steps |
| Water supply | City | for water cooled condenser | |
| Software | |||
| Chemdraw | Cambridgesoft | molecular editor for muropeptide fragmentation prediction | |
| Excel | Microsoft | viewing lists of annotated muropeptides, abundance, isotopic patterns, etc. | |
| MassHunter Acquisition | Aglient | running QTOF instrument | |
| MassHunter Mass Profiler Professional | Aglient | bioinformatic differential analysis | |
| MassHunter Personal Compound Database and Library Manager | Aglient | muropeptide m/z MS database | |
| MassHunter Profinder | Aglient | recursive feature extraction | |
| MassHunter Qualitative analysis | Aglient | viewing MS and MS/MS chromatograms | |
| Prism | Graphpad | Graphing software | |
| Perseus | Max Plank Institute of Biochemistry | 1D annotation | |
| Material | |||
| Acetonitrile | Fisher | A998-4 | |
| Ammonium acetate | Fisher | A637 | |
| Amylase | Sigma-Aldrich | A6380 | |
| Boric acid | Fisher | BP168-1 | |
| DNase | Fisher | EN0521 | |
| Formic acid | Sigma-Aldrich | 27001-500ML-R | |
| LC-MS tuning mix - HP0321 | Agilent | G1969-85000 | |
| Magnesium chloride | Sigma-Aldrich | M8266 | |
| Magnesium sulfate | Sigma-Aldrich | M7506 | |
| Mutanolysin from Streptomyces globisporus ATCC 21553 | Sigma-Aldrich | M9901 | |
| Nitrogen gas (>99% purity) | Praxair | NI 5.0UH-T | |
| Phosphoric acid | Fisher | A242 | |
| Pronase E from Streptomyces griseus | Sigma-Aldrich | P5147 | |
| RNase | Fisher | EN0531 | |
| Sodium azide | Fisher | S0489 | |
| Sodium borohydride | Sigma-Aldrich | 452890 | |
| Sodium dodecyl sulfate (SDS) | Fisher | BP166 | |
| Sodium hydroxide | Fisher | S318 | |
| Sodium Phosphate (dibasic) | Fisher | S373 | |
| Sodium Phosphate (monobasic) | Fisher | S369 | |
| Stains-all | Sigma-Aldrich | E9379 |
Références
- Vollmer, W., Blanot, D., de Pedro, M. A. Peptidoglycan structure and architecture. FEMS Microbiology Reviews. 32, 149-167 (2007).
- Pazos, M., Peters, K. Peptidoglycan. Sub-cellular Biochemistry. 92, 127-168 (2019).
- Typas, A., Banzhaf, M., Gross, C. A., Vollmer, W. From the regulation of peptidoglycan synthesis to bacterial growth and morphology. Nature Reviews. Microbiology. 10 (2), 123-136 (2011).
- Yadav, A. K., Espaillat, A., Cava, F. Bacterial strategies to preserve cell wall integrity against environmental threats. Frontiers in Microbiology. 9, 2064 (2018).
- Bera, A., Herbert, S., Jakob, A., Vollmer, W., Götz, F. Why are pathogenic staphylococci so lysozyme resistant? The peptidoglycan O-acetyltransferase OatA is the major determinant for lysozyme resistance of Staphylococcus aureus. Molecular Microbiology. 55 (3), 778-787 (2005).
- Brott, A. S., Clarke, A. J. Peptidoglycan O-acetylation as a virulence factor: its effect on lysozyme in the innate immune system. Antibiotics. 8 (3), 94 (2019).
- Putty, S., Vemula, H., Bobba, S., Gutheil, W. G. A liquid chromatography-tandem mass spectrometry assay for D-Ala-D-Lac: A key intermediate for vancomycin resistance in vancomycin-resistant Enterococci. Analytical Biochemistry. 442 (2), 166-171 (2013).
- Arthur, M., et al. Evidence for in vivo incorporation of D-lactate into peptidoglycan precursors of vancomycin-resistant Enterococci. Antimicrobial Agents and Chemotherapy. 36 (4), 867-869 (1992).
- Mardarowicz, C. Isolierung und Charakterisierung des Murein-Sacculus von Brucella. Z. Naturforsdig. 21, 1006-1007 (1966).
- Glauner, B., Höltje, J. V., Schwarz, U. The composition of the murein of Escherichia coli. The Journal of Biological Chemistry. 263 (21), 10088-10095 (1988).
- Glauner, B. Separation and quantification of muropeptides with high-performance liquid chromatography. Analytical Biochemistry. 172, 451-464 (1988).
- Anderson, E. M., et al. Peptidoglycomics reveals compositional changes in peptidoglycan between biofilm- and planktonic-derived Pseudomonas aeruginosa. The Journal of Biological Chemistry. 295 (2), 504-516 (2020).
- van der Aart, L. T., et al. High-resolution analysis of the peptidoglycan composition in Streptomyces coelicolor. Journal of Bacteriology. 200 (20), 00290 (2018).
- Bern, M., Beniston, R., Mesnage, S. Towards an automated analysis of bacterial peptidoglycan structure. Analytical and Bioanalytical Chemistry. 409, 551-560 (2017).
- Brott, A. S., Sychantha, D., Clarke, A. J. Assays for the enzymes catalyzing the O-acetylation of bacterial cell wall polysaccharides. Methods in Molecular Biology. 1954, 115-136 (2019).
- Clarke, A. J. Compositional analysis of peptidoglycan by high-performance anion-exchange chromatography. Analytical Biochemistry. 212 (2), 344-350 (1993).
- Rusconi, F., Douard Valton, &. #. 2. 0. 1. ;., Nguyen, R., Dufourc, E. Quantification of sodium dodecyl sulfate in microliter-volume biochemical samples by visible light spectroscopy. Analytical Biochemistry. 295, 31-37 (2001).
- Verwer, R. W. H., Beachey, E. H., Keck, W., Stoub, A. M., Poldermans, J. E. Oriented fragmentation of Escherichia coli sacculi by sonication. Journal of Bacteriology. 141 (1), 327-332 (1980).
- Benjamini, Y., Hochberg, Y. Controlling the false discovery rate: A practical and powerful approach to multiple testing. Journal of the Royal Statistical Society: Series B (Methodological). 57 (1), 289-300 (1995).
- Article, R., Alonso, A., Marsal, S., Julià, A., James Carroll, A. Analytical methods in untargeted metabolomics: state of the art in 2015. Frontiers in Bioengineering and Biotechnology. 3, 23 (2015).
- Xi, B., Gu, H., Baniasadi, H., Raftery, D. Statistical analysis and modeling of mass spectrometry-based metabolomics data. Methods in Molecular Biology. 1198, 333-353 (2014).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Cox, J., Mann, M. 1D and 2D annotation enrichment: a statistical method integrating quantitative proteomics with complementary high-throughput data. BMC Bioinformatics. 13, 12 (2012).
- Chang, J. D., Foster, E. E., Thadani, A. N., Ramirez, A. J., Kim, S. J. Inhibition of Staphylococcus aureus cell wall biosynthesis by desleucyl-oritavancin: A quantitative peptidoglycan composition analysis by mass spectrometry. Journal of Bacteriology. 199 (15), 00278 (2017).
- Glauner, B., Höltje, J. V. Growth pattern of the murein sacculus of Escherichia coli. The Journal of Biological Chemistry. 265 (31), 18988-18996 (1990).
- Espaillat, A., et al. Chemometric analysis of bacterial peptidoglycan reveals atypical modifications that empower the cell wall against predatory enzymes and fly innate immunity. Journal of the American Chemical Society. 138 (29), 9193-9204 (2016).
- Quintela, J. C., Caparros, M., de Pedro, M. A. Variability of peptidoglycan structural parameters in Gram-negative bacteria. FEMS Microbiology Letters. 125, 95-100 (1995).
- Quintela, J. C., Zollner, P., Portillo, F. G., Allmaier, G., de Pedro, M. A. Cell wall structural divergence among Thermus spp. FEMS Microbiology Letters. 172, 223-229 (1999).
- Torrens, G., et al. Comparative analysis of peptidoglycans from Pseudomonas aeruginosa isolates recovered from chronic and acute infections. Frontiers in Microbiology. 10, 0868 (2019).
- Antignac, A., et al. Detailed structural analysis of the peptidoglycan of the human pathogen Neisseria meningitidis. The Journal of Biological Chemistry. 278 (34), 31521-31528 (2003).
- De Jonges, B. L. M., Changi, Y. S., Gage, D., Tomaszs, A. Peptidoglycan composition of a highly methicillin-resistant Staphylococcus aureus strain. The role of penicillin binding protein 2A. The Journal of Biological Chemistry. 267 (16), 11248-11264 (1992).
- Lee, M., et al. Muropeptides in Pseudomonas aeruginosa and their role as elicitors of β-lactam-antibiotic resistance. Angewandte Chemie - International Edition. 55, 6882-6886 (2016).
- Schumann, P. Peptidoglycan Structure. Methods in Microbiology. 38, 101-129 (2011).
- Wientjes, F. B., Woldringh, C. L., Nanninga, N. Amount of peptidoglycan in cell walls of Gram-negative bacteria. Journal of Bacteriology. 173 (23), 7684-7691 (1991).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon