Method Article
Combiner la microdissection de capture laser et le QPCR microfluidique pour analyser les profils transcriptionnels des cellules uniques : une approche de biologie des systèmes à la dépendance aux opioïdes
Dans cet article
Résumé
Ce protocole explique comment collecter des neurones uniques, des microglies et des astrocytes du noyau central de l’amygdale avec une grande précision et une spécificité anatomique à l’aide de la microdissection de capture laser. En outre, nous expliquons notre utilisation de microfluidique RT-qPCR pour mesurer un sous-ensemble du transcriptome de ces cellules.
Résumé
L’hétérogénéité transcriptionnelle profonde dans les cellules simples anatomiquement adjacentes suggère que la fonctionnalité robuste de tissu puisse être réalisée par la diversité cellulaire de phénotype. Les expériences à cellule unique qui étudient la dynamique du réseau des systèmes biologiques démontrent des réponses cellulaires et tissulaires à diverses conditions à une résolution biologiquement significative. Ici, nous expliquons nos méthodes pour recueillir des cellules uniques à partir d’endroits anatomiquement spécifiques et mesurer avec précision un sous-ensemble de leurs profils d’expression génique. Nous combinons la microdissection de capture laser (LCM) avec des réactions microfluidiques de la chaîne quantitative de polymérase de transcription inverse (RT-qPCR). Nous utilisons également cette plate-forme microfluidique RT-qPCR pour mesurer l’abondance microbienne du contenu intestinal.
Introduction
La mesure des profils d’expression génique des cellules simples a démontré une hétérogénéité phénotypique étendue dans un tissu. Cette complexité a compliqué notre compréhension des réseaux biologiques qui régissent la fonction tissulaire. Notre groupe et d’autres ont exploré ce phénomène dans de nombreux tissus et conditions1,2,3,4,5,6. Ces expériences suggèrent non seulement que la régulation des réseaux d’expression génique sous-tend une telle hétérogénéité, mais aussi que la résolution à cellule unique révèle une complexité dans la fonction tissulaire que la résolution au niveau tissulaire ne parvient pas à apprécier. En effet, une petite minorité de cellules peut répondre à une condition ou un défi spécifique, mais l’impact de ces cellules sur la physiologie globale peut être considérable. En outre, une approche de biologie du système qui applique des méthodes multivariées à des jeux de données de haute dimension provenant de plusieurs types de cellules et tissus peut élucider les effets de traitement à l’échelle du système.
Nous combinons LCM et microfluidique RT-qPCR pour obtenir de tels ensembles de données. Nous adoptons cette approche ici en contraste avec la collecte de cellules individuelles via le tri cellulaire activé par fluorescence (FACS) et en utilisant le séquençage de l’ARN (ARN-seq) pour mesurer leur transcriptome. L’avantage de LCM par rapport à FACS est que la spécificité anatomique exacte des cellules simples peut être documentée avec LCM, relativement et absolument. En outre, alors que l’ARN-seq peut mesurer plus de fonctionnalités que RT-qPCR, microfluidique RT-qPCR est moins cher et a une sensibilité plus élevée et la spécificité7.
Dans cette expérience représentative, nous avons étudié les effets de la dépendance aux opioïdes et le sevrage opioïde naltrexone-précipité sur le rat neuronal, microglia, et l’expression de gène d’astrocyte dans le noyau central de l’amygdale (CeA) et l’abondance de microflora intestinale4. Quatre groupes de traitement ont été analysés : 1) Placebo, 2) Morphine, 3) Naltrexone, et 4) Retrait(figure 1). Nous avons constaté que la dépendance aux opioïdes n’altait pas considérablement l’expression des gènes, mais que le sevrage des opioïdes incitait l’expression de gènes inflammatoires, Tnf en particulier. Les astrocytes étaient le type de cellule le plus affecté. Le microbiome intestinal a été profondément affecté par le sevrage des opioïdes comme indiqué par une diminution du rapport Firmicutes à Bacteroides, qui est un marqueur établi de dysbiose intestinale8,9.
Protocole
Cette étude a été réalisée conformément aux recommandations du Comité de soins et d’utilisation des animaux (IACUC) de l’Université Thomas Jefferson et du Drexel University College of Medicine. Le protocole a été approuvé par l’Université Thomas Jefferson et le Drexel University College of Medicine IACUC.
1. Modèle animal
- Insérez deux granules de sulfate de morphine à libération lente de 75 mg ou deux granules placebo sous-cutanée chez les rats sprague-Dawley mâles adultes.
- Utilisez une robe et des gants de façon appropriée pour une chirurgie stérile mineure. Raser le rat dorsum avec des tondeuses si nécessaire.
- Appliquer la pommade vétérinaire sur les yeux de l’animal. Anesthésiez le rat avec environ 20 s d’inhalation d’isoflurane. L’anesthésie est confirmée par la perte de conscience.
- Faire une incision de ligne médiane dans le dorsum de rat avec des ciseaux émoussés perlés-stérilisés et séparer le derme de la paroi du corps avec une sonde perlée-stérilisée. Insérez les granulés sous le derme avec des forceps stérilisés par perle. Suture l’incision fermée avec une aiguille stérile.
REMARQUE : L’ensemble de la procédure prend environ 5 min par rat. Des gants stériles frais sont utilisés pour chaque rat. - Placez le rat dans une cage d’isolement pour la récupération posturgique. Vérifiez s’il y a un rythme cardiaque et un rythme respiratoire régulier. Observez le rat jusqu’à ce que la conscience soit retrouvée. Évaluer la douleur posturgique.
- Évaluer les rats 8 h posturgery et tous les 12 h après pour la récupération et l’infection. Placez les rats dans une cage avec le reste de la cohorte lorsqu’ils sont complètement récupérés après une chirurgie, environ 24 h de postsurgery.
- Donnez une injection intraperitonéal de naltrexone (75 mg/kg) aux cohortes G et de retrait après 6 jours d’exposition à la morphine.
REMARQUE : Il y avait quatre cohortes de rats dans cette expérience représentative (voir la figure 1).
2. Récolte d’échantillons
- Récoltez le cerveau 6 jours après l’insertion de granulés ou 24 h après l’injection de naltrexone.
- Placez l’animal dans une chambre isoflurane pendant environ 30 s ou jusqu’à ce que la perte de conscience se produise, indiquée par un manque de mouvement et une diminution de la fréquence respiratoire.
- Utilisez une guillotine pointue pour décapiter rapidement l’animal.
- Disséquez le cerveau du crâne de l’animal.
- Utilisez un rasoir portatif pointu pour faire les incisions brutes suivantes au cerveau enlevé : Tout d’abord, trancher le cervelet et jeter. Deuxièmement, séparer le tronc cérébral du cerveau avec une incision transversale. Troisièmement, hémisect le cerveau avant et / ou tronc cérébral avec une incision sagittale de ligne médiane.
- Placer le cerveau avant et le tronc cérébral dans un moule en plastique encastrement des tissus avec 3-4 cm de composé optimal de température de coupe (OCT) dans le fond du moule. Couvrez le reste de l’échantillon avec OCT.
- Placez immédiatement le moule en tissu plastique avec l’échantillon recouvert d’OCT dans un bain contenant de la glace sèche et du méthanol. Ne laissez pas le méthanol se déverser dans le moule à ensenchir les tissus. Conservez le moule d’intégration avec l’échantillon de cerveau dans le bain de méthanol-glace jusqu’à ce que la collecte des tissus soit terminée (maximum de 10-15 min).
- Placer l’échantillon de cerveau dans un congélateur à -80 oC dès que possible.
- Récoltez les échantillons d’intestin en même temps.
- Après décapitation rapide, faire une incision de ligne médiane dans l’abdomen de l’animal avec un scalpel.
- Trouvez le cecum et coupez sa connexion au côlon ascendant.
- Presser le contenu cécal dans un tube conique de 15 ml.
- Placez immédiatement le tube conique sur de la glace sèche et mettez-le dans un congélateur à -80 oC dès que possible.
REMARQUE : Le contenu de l’intestin grêle peut également être recueilli en utilisant les mêmes méthodes qu’un contrôle négatif.
3. Trancher
- Trancher le cerveau pré-cerveau hémisé à l’aide d’un cryostat.
- Retirer le moule d’intégration en plastique avec le cerveau avant du congélateur de -80 oC et les placer dans un cryostat de -20 oC.
- Retirer l’échantillon de cerveau pré-cerveau hémisected intégré octogénaire du moule d’intégration. Utilisez un rasoir pour trancher les coins du moule d’intégration en plastique verticalement si nécessaire. Montez le cerveau avant pour rostral à coronale caulique en coupant sur un mandrin cryostat utilisant OCT.
REMARQUE : Les repères anatomiques pour identifier le CeA comprennent le tractus optique et la stria terminalis(figure 2B). Le tract optique s’en branches du chiasme optique et suit dorsal-latéral comme le cerveau est tranché rostral à caudal. Lorsque le tractus optique a une morphologie similaire à ce qui est vu dans un bregma atlas du cerveau de rat -2,12 mm10, les tranches d’essai peuvent être vues sous un microscope. La morphologie du tractus optique et de la stria terminalis peut être vérifiée dans un atlas du cerveau de rat10 pour identifier la bregma et si le CeA entoure les terminaux de stria. - Trancher 10 m d’épaisseur sections coronales du rostral hémisected de cerveau avant à caudal jusqu’à ce que des sections contenant le CeA soient atteintes.
REMARQUE : La largeur et la hauteur des sections sont d’environ 200 mm. - Recueillir 10 sections de m contenant le CeA, ou la région cérébrale préférée, en dégelant 10 sections de m sur des toboggans en verre ordinaire. Placez immédiatement les toboggans en verre sur une casserole en métal reposant sur de la glace sèche. Placez les diapositives avec des sections cérébrales dans un congélateur de -80 oC dès que possible.
REMARQUE : Plusieurs tranches peuvent être placées sur la même lame. Si vous utilisez une tache de type cellulaire différente pour les tranches sur la même lame, laissez environ 100 mm entre les tranches afin qu’un stylo hydrophobe puisse être utilisé pour séparer les solutions d’anticorps spécifiques de type cellulaire sur la glissière. Laisser environ 20 mm du bord de la glissière de chaque côté de la tranche.
4. Coloration d’immunofluorescence
- Tacher les sections de cerveau pour la cellule cérébrale de choix (par exemple, neurone, microglie, astrocyte, etc.) en utilisant l’immunofluorescence.
- Retirer une ou plusieurs diapositives avec des sections de 10 m du CeA du congélateur de -80 oC.
- Fixer les diapositives avec 75% d’éthanol pour 30 s. Retirer l’excédent de liquide.
- Bloquer les tranches pour 30 s avec 2% d’antigène de sérum bovin (BSA) dans 1x phosphate tampon salin (PBS). Laver 1x avec PBS.
- Ajouter une solution d’anticorps primaire à la glissière pendant 2 min. Laver 1x avec une solution BSA de 2%.
REMARQUE : La solution principale d’anticorps est composée de 2 % d’anticorps primaires, de 1 % de RNase Out et de 96 % de la même solution BSA PBS pour l’étape de blocage au-dessus (étape 4.1.3). L’expérience représentative a utilisé un anticorps anti-NeuN, un anticorps anti-Cd11MD et un anticorps anti-GFAP dans les quantités suivantes : 3 l de l’inhibiteur primaire, 1,88 l d’inhibiteur de l’ARN et 145,12 ll de solution BSA. - Ajouter la solution d’anticorps secondaires à la glissière pendant 3 min. Laver 1x avec PBS.
REMARQUE : La solution secondaire d’anticorps est composée de 1 l d’étiquette fluorescente anti-souris de chèvre 488 nm (1:500), de 2,5 l d’inhibiteur de l’ARN, de 1,3 L de DAPI (1:10 000) et de 196,5 L de 2 % de BSA.
5. Série de déshydratation de l’éthanol et du xylène
- Trempez les diapositives dans 75% d’éthanol pour 30 s. Immédiatement après, trempez les diapositives dans 95% d’éthanol pour 30 s. Immédiatement après, tremper les diapositives dans 100% d’éthanol pour 30 s. Immédiatement après, tremper les diapositives dans un deuxième récipient contenant 100% d’éthanol pour 30 s.
- Après la série de déshydratation de l’éthanol, tremper les diapositives dans le xylène fraîchement versé pendant 1 min. Immédiatement après, tremper les diapositives dans un deuxième récipient de xylène pendant 4 min.
- Retirer les lames du bain de xylène et laisser sécher l’air dans l’obscurité pendant 5 minutes.
- Placer les toboggans dans un desiccateur pendant 5 minutes pour sécher davantage.
6. Microdissection de capture au laser
- Si taché, placez la glissière dans le microscope et trouvez la région d’intérêt (CeA) à l’aide de repères anatomiques (c.-à-d. le chiasme optique et la stria terminalis).
- Utilisez la fluorescence pour identifier le type de cellule tachée et son noyau dans la région d’intérêt. Choisissez une cellule ou plusieurs cellules si vous effectuez des échantillons à cellules individuelles. Marquez les cellules d’intérêt à l’aide du logiciel LCM.
- Placez le bouchon LCM sur le dessus de la tranche sur la région d’intérêt.
- Utilisez des plans d’essai d’un laser infrarouge (IR) pour ajuster la force, la taille et la durée du laser IR afin que l’adhésif de bouchon de LCM ne fonde que sur la zone de la cellule unique sélectionnée. Cela garantit qu’aucune autre cellule ne sera collectée autre que les cellules sélectionnées.
REMARQUE : Dans cette expérience représentative, 10 pools cellulaires du même type de cellule ont été utilisés comme un seul échantillon pour limiter la variabilité de cellule à cellule dans l’expression des gènes entre les échantillons avec le même traitement. Cependant, cette méthode peut être utilisée pour de véritables expériences cellulairessimples 1,3. - Sélectionnez les cellules individuelles à collecter pour analyse à l’aide des outils logiciels LCM(figure 2C). Les cellules sélectionnées doivent se trouver dans la zone anatomique du CeA (ou de la région cérébrale de choix) en fonction de l’atlas du cerveau des rats et de la bregma10. Les cellules doivent être à au moins 3 m des noyaux tachés adjacents.
- Tirez le laser IR pour recueillir les cellules individuelles identifiées.
- Placez le bouchon dans la station de contrôle de la qualité (QC) et visualyez-le pour s’assurer que seules les cellules désirées ont été sélectionnées. Si d’autres cellules ont été sélectionnées par erreur, un laser ultraviolet peut être utilisé pour détruire les cellules indésirables pendant que le bouchon reste dans la station QC.
- Prenez une photo de la section tissulaire d’où la cellule a été recueillie pour documenter sa spécificité anatomique. Enregistrez la distance de la tranche de la bregma si nécessaire à l’aide d’un atlas du cerveau de rat comme référence10.
- Retirez le bouchon LCM de la station QC, fixez le dispositif d’extraction de l’échantillon et la pipette de 5,5 l de tampon de lyse sur l’échantillon.
REMARQUE : La solution tampon de lyse se compose de 0,5 l d’améliorateur de lyse et de tampon de résuspension de 5 l. - Placez l’appareil ExtracSure sur un tube de microcentrifuge de 0,5 mL et placez-le sur une plaque chauffante à 75 oC pendant 15 minutes.
- Tourner l’échantillon et le tampon de lyse pour 30 s à basse vitesse (0,01-0,02 x g) et placer l’échantillon recueilli dans un congélateur de -80 oC.
7. Microfluidique à cellule unique RT-qPCR
- Préamplification de l’ARNm à cellule unique pour 96,96 Dynamic Array Chip
- Combinez les amorces de gènes qPCR avant et inverse de l’ARNm pour tous les gènes analysés dans un pool d’amorce pour la préamplification (concentration de 500 nM par amorce). Par exemple, 1 L de 100 amorces de M dans 80 l plus 120 l de tampon de suspension d’ADN.
REMARQUE : Les séquences d’amorce utilisées pour l’expérience représentative peuvent être trouvées dans O’Sullivan et coll.4. - Dans une nouvelle plaque PCR de 96 x 96, ajoutez 1 L de 5x VILO à chaque puits.
- Retirez les échantillons de LCM à cellule unique des échantillons stockés à -80 oC, laissez décongeler brièvement, centrifugeuse brièvement à basse vitesse (0,01-0,02 x g),et ajoutez 5,5 l de l’échantillon de monocellulaire lysed à la plaque PCR. Chaque échantillon est ajouté à son propre puits.
- Placer la plaque PCR avec les échantillons et VILO dans le thermocycleur et chauffer à 65 oC pendant 1,5 min. Tourner la plaque pendant 1 min à 1 300 x g à 4 oC et placer la plaque sur la glace.
- Ajoutez 0,15 l de 10x cDNA synthèse maître mélange, 0,12 T4 Gène 32 protéine, et 0,73 L de tampon de suspension d’ADN à chaque puits.
- Placez la plaque PCR dans le thermocycleur et exécutez le protocole suivant : 25 oC pour 5 minutes, 50 oC pendant 30 min, 55 oC pendant 25 minutes, 60 oC pour 5 minutes, 70 oC pendant 10 min, 4 oC à la fin.
- Ajouter 7,5 L de mélange de maître en polymérase Taq à chaque puits.
- Ajouter 1,5 L de la piscine d’apprêt (voir ci-dessus) à chaque puits.
- Placez la plaque PCR dans le thermocycleur et exécutez le protocole de préamplification suivant : 95 oC pendant 10 min, suivi de 22 cycles de 96 oC pendant 5 secondes, 60 oC pour 4 min.
- Ajoutez 0,6 L d’exonuclease I zone tampon de réaction 10x, 1,2 L exonuclease I, et 4,2 l de tampon de suspension d’ADN à chaque puits.
- Placez la plaque PCR dans le thermocycler et exécutez le protocole suivant : 37 oC pendant 30 min, 80 oC pendant 15 min.
- Ajouter 54 L de tampon TE à chaque puits. Tourner la plaque PCR à 1 300 x g à 5 min. Magasinez à 4 oC si vous continuez immédiatement à l’étape suivante. Conserver à -20 oC en attendant plus de 12 h pour la prochaine étape.
- Combinez les amorces de gènes qPCR avant et inverse de l’ARNm pour tous les gènes analysés dans un pool d’amorce pour la préamplification (concentration de 500 nM par amorce). Par exemple, 1 L de 100 amorces de M dans 80 l plus 120 l de tampon de suspension d’ADN.
- Préparer la plaque d’échantillon pour la puce Dynamic Array 96.96.
- Dans une nouvelle plaque DE 96 puits PCR, ajouter 0,45 L de colorant de liaison d’ADN 20x et 4,55 l de mîtalisme ROX faible à chaque puits.
- Ajouter 3 L d’échantillon préamplifié à chaque puits, faire tourner la plaque PCR à 1 300 x g,puis mettre la plaque sur la glace.
- Préparez la plaque d’essai pour la puce Dynamic Array 96.96.
- Dans une nouvelle plaque PCR de 96 puits, ajoutez 3,75 L de réactif de chargement d’essai GE de 2x et 1,25 l de tampon de suspension d’ADN à chaque puits.
- Ajouter 2,5 L de l’amorce qPCR de M à chaque puits correspondant. Tourner la plaque PCR à 1 300 x g pendant 5 min.
- Chargez et exécutez la puce Dynamic Array 96.96.
- Émiz la puce avec du liquide de la ligne de commande.
- Placez la puce dans un contrôleur IFC HX et exécutez le script Prime (136X).
- Ajoutez 6 L de l’échantillon de la plaque d’échantillon PCR dans les puits d’échantillon correspondants dans la puce dynamic Array 96.96.
- Ajoutez 6 L de l’échantillon de la plaque d’essai PCR dans les puits d’essai correspondants dans la puce Dynamic Array 96.96.
- Utilisez des aiguilles pour faire éclater les bulles d’air dans les puits de la puce Dynamic Array 96.96.
- Placez la puce Dynamic Array 96.96 dans le contrôleur IFC HX et exécutez le script Load Mix (136x).
- Retirez la puce du contrôleur IFC HX, décollez l’autocollant de protection et placez la puce Dynamic Array 96.96 dans une plate-forme microfluidique RT-qPCR. Exécuter le protocole GE Fast 96 x 96 PCR (30 cycles).
REMARQUE : La qualité et la validité de l’ARN des résultats sont évaluées par plusieurs méthodes, y compris la validation d’essai par électrophoresèse de gel, les courbes de température fondante, l’échantillon et les répliques d’essai, et les parcelles standard de série de dilution. En outre, les résultats transcriptionnels peuvent être validés par des méthodes indépendantes sur l’hémisection de cerveau comprenant les essais occidentaux de tache et d’immunofluorescence.
8. Mesurer l’abondance bactérienne avec le RT-qPCR microfluidique
- Extraire l’ADN bactérien en suivant les instructions du kit d’extraction de l’ADN des selles.
- Estimer la concentration d’ADN bactérien à l’aide de qPCR.
- Ajouter l’ADN bactérien extrait à une nouvelle plaque PCR. Ajouter 1 l d’ADN bactérien extrait et 9 l de tampon de suspension d’ADN.
- Préparer la plaque d’essai pour la puce Dynamic Array 48.48 (voir les étapes 7.2.1-7.2.2)
- Préparer la plaque d’échantillon pour la puce Dynamic Array 48.48 (voir les étapes 7.3.1-7.3.2)
- Dans une nouvelle plaque DE 96 puits PCR, ajoutez 0,45 L de colorant de liaison d’ADN 20x et 4,55 l de mîtalisme ROX bas à 48 puits.
- Ajouter 3 L de l’échantillon de la plaque PCR contenant l’ADN bactérien aux 48 puits et faire tourner la plaque PCR à 1 300 x g pendant 5 min. Conserver à 4 oC.
- Chargez et exécutez la puce Dynamic Array 48.48 (voir étapes 7.4.1-7.4.7).
Résultats
La sélection des cellules simples a été validée visuellement et moléculairement. Visuellement, la morphologie cellulaire a été vue avant la collecte de cellules. Les cellules recueillies ont ensuite été vues à la station DE QC et la tache cellulaire des noyaux (DAPI) chevauchait la fluorescence du marqueur de sélection de cellules uniques. La figure 2A montre des images représentatives d’une diapositive avec le cerveau pré-cerveau hémisé contenant le CeA. Les images ultérieures(figure 2B-D) montrent la sélection des cellules individuelles et leur élimination du tissu pour l’analyse transcriptomique. Moléculairement, les marqueurs spécifiques au type cellulaire ont démontré une expression accrue dans ce type de cellule(figure 1C). Nous avons examiné les neurones, les microglies et les astrocytes et mesuré l’expression de NeuN, Maf, et Gfap, respectivement. Les chiffres ont été publiés à l’origine dans O’Sullivan et al.4.
En outre, les contrôles peuvent également être exécutés dans la plate-forme microfluidique pour valider les résultats d’expression (par exemple, l’analyse d’autres secteurs du même tissu pour démontrer la spécificité de noyau). Un tissu séparé pourrait également être comparé à l’échantillon désiré pour démontrer la spécificité d’apprêt dans le tissu d’intérêt. Des gènes de contrôle positifs et négatifs peuvent également être inclus (p. ex., les gènes connus pour être absents du tissu sélectionné ou exprimés haut et fort). Trois ou quatre gènes d’entretien ménager devraient également être inclus non seulement à des fins de normalisation des données, mais aussi comme une mesure de la qualité expérimentale. Ces gènes devraient démontrer la plus faible variance d’expression dans tous les échantillons et traitements. Dans cette expérience représentative, aucune région autre de cerveau n’a été analysée, mais les gènes d’entretien ménager Ldha et Actb ont été employés pour la normalisation. Gapdh a été utilisé comme un contrôle interne.
La figure 3 affiche certaines des méthodes multivariate utilisées par notre groupe pour analyser nos données. Nous avons constaté que les astrocytes dans le groupe de retrait étaient le type de cellule le plus affecté. Sur la base de ces données dans le cadre d’autres études, nous spéculons que les astrocytes jouent un rôle clé dans l’inflammation dans le CeA pendant le sevrage des opioïdes et que cela contribue aux symptômes physiques et émotionnels qui conduisent la recherche de drogues par le renforcement négatif. Nous montrons également les données de microflore intestinale(figure 4).
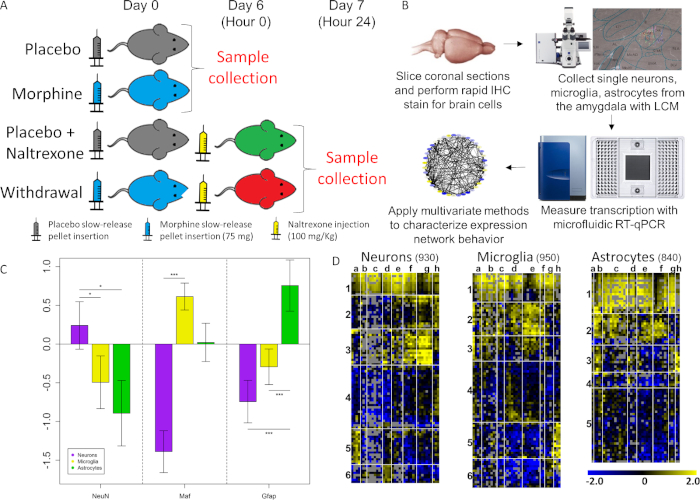
Figure 1 : Flux de travail à cellule unique RT-qPCR et hétérogénéité transcriptionnelle. (A) Protocole expérimental (n 4 pour chaque condition) (B) Mesure de transcriptome d’échantillon de dix cellules. (C) L’intrigue de barre affiche les valeurs médianes d’expression de -'' Neurones et violet; microglies et jaune; astrocytes et vert. Les barres d’erreur montrent une erreur standard. 0,05, 0,05, 0,0003; Le test de signification honnête de Tukey. (D) La carte thermique montre l’expression de tous les échantillons sur 40 gènes analysés. Les rangées sont des échantillons mis en commun à 10 cellules (930 échantillons neuronaux, 950 échantillons microgliaux, 840 échantillons d’astrocytes tels qu’ils sont dénotés); les nombres indiquent les amas d’échantillons et les colonnes sont les gènes. Le chiffre est modifié à partir d’O’Sullivan et al.4. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
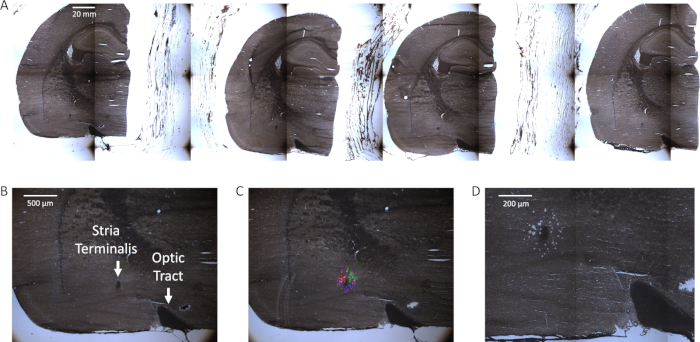
Figure 2 : Capture laser des images de microdissection. (A) Quatre tranches de pré-cerveau de rat hémisected déshydraté contenant le CeA sur une glissière. Des tranches ont été placées sur la glissière exactement à 10 m de la tranche précédente. La gauche est antérieure et la droite est postérieure. La distance de la bregma peut être estimée à l’aide d’un atlas du cerveau de rat et des repères, y compris le tractus optique et la stria terminalis. (B-D) Séquence d’images montrant la sélection de cellules individuelles dans le CeA (C) et leur élimination du tissu (D). Plusieurs bouchons LCM ont été utilisés pour sélectionner ces cellules. Un bouchon est utilisé pour choisir 10 cellules d’un type cellulaire. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
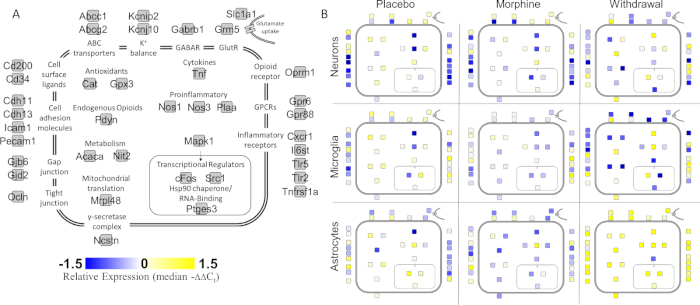
Figure 3 : Résultats représentatifs 1. (A) Un schéma de dessin animé d’une cellule affichant les gènes analysés et leur emplacement. Les symboles génétiques étiquetés ici sont une référence pour le panneau B. (B) Les carrés colorés représentent l’expression relative de gène (médiane ----- Valeur de cœurs) pour les gènes représentés dans le panneau A. L’emplacement des carrés représente la localisation cellulaire ou la fonction de la protéine correspondante. Les panneaux affichent l’expression relative du gène représentée par la couleur entre les traitements et les types de cellules. Jaune et expression élevée; bleu et faible expression; blanc - expression neutre. Le chiffre est modifié à partir d’O’Sullivan et al.4. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 4 : Résultats représentatifs 2. (A) Réseaux de corrélation des gènes. La corrélation Pearson a été effectuée sur les valeurs de -Ct dans un type de traitement et de cellule. Les nœuds dénotent les gènes et leur couleur signifie les niveaux d’expression relatifs (la valeur médiane de -Ct pour chaque gène). Les bords dénotent les corrélations d’expression et l’épaisseur signifie la force de la corrélation d’expression . Des corrélations avec une valeur q et 0,001 sont affichées. Bords noirs et corrélations positives; bords verts et corrélations négatives. (B) Parcelles à barres de gènes sélectionnés démontrant l’expression différentielle significative des gènes. Les statistiques ont été calculées à l’aide d’ANOVA imbriqués #p de lt; 0,1, p 'lt; 0,05, 'p 'lt; 0,01, 'p 'lt; 0.0001 (n '4 animaux pour tous les traitements). Le chiffre est modifié à partir d’O’Sullivan et al.4. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
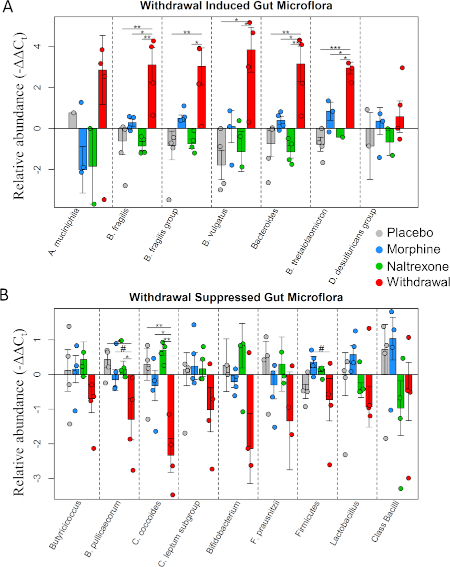
Figure 5 : Abondance relative de microflore intestinale. Les barplots affichent l’abondance relative des espèces bactériennes (valeurs de la maladie). #p’a lt; 0,1, p 'lt; 0.05, 'p 'lt; 0.008, 'p '0.0009; ANOVA bidirectionnel; n 4 animaux pour chaque traitement. Le chiffre est modifié à partir d’O’Sullivan et al.4. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Discussion
La biologie unicellulaire a démontré l’hétérogénéité des phénotypes cellulaires et la robustesse de la fonction tissulaire. Ces résultats ont permis de mieux comprendre l’organisation des systèmes biologiques à la fois à des échelles macro et micro. Ici, nous décrivons la combinaison de deux méthodes, LCM et qPCR microfluidique, pour obtenir des mesures de transcriptome à cellule unique qui fournissent la spécificité anatomique et l’exactitude transcriptionnelle à un coût relativement faible(figure 1). Notre groupe adopte une approche de biologie des systèmes et mesure souvent plusieurs tissus chez le même animal. Nous trouvons ces méthodes à la fois flexibles et fructueuses pour déterminer comment les systèmes biologiques répondent à divers défis au niveau transcriptionnel. En outre, nous utilisons ces méthodes dans la cartographie anatomique des phénotypes cellulaires dans des conditions de base.
Nous fournissons des données et des chiffres modifiés d’une publication récente explorant comment le CeA réagit à la dépendance et au retrait des opioïdes4. Dans cet exemple, nous avons utilisé la même plate-forme microfluidique qPCR pour mesurer l’abondance relative de la microflore intestinale. Les méthodes et le flux de travail sont résumés à la figure 1 et ont été initialement publiés dans O’Sullivan et coll.4. Les principaux résultats de RT-qPCR microfluidique à haut débit peuvent être validés par la suite par des mesures protéiques telles que la tache occidentale ou l’immunofluorescence4.
L’un des principaux défis de cette approche de biologie des systèmes est de déterminer des mécanismes biologiques causals spécifiques. La logique floue est une solution validée que nous avons employée avec succès pour déduire des agents dans le comportement de réseau de régulation desgènes 2. La manipulation des modèles animaux peut également être utilisée pour donner un aperçu des mécanismes systémiques. Par exemple, le même protocole fourni dans les présentes avec l’ajout d’une cohorte de rats avec une vagotomie gastrique donnera des données qui fournissent un aperçu de la circulation de l’information via le nerf vague.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont pas d’intérêts financiers concurrents.
Remerciements
Les travaux présentés ici ont été financés par NIH HLB U01 HL133360 attribué à JS et RV, NIDA R21 DA036372 décerné à JS et EVB, et T32 AA-007463 attribué à Jan Hoek à l’appui de SJO’S.
matériels
| Name | Company | Catalog Number | Comments |
| 20X DNA Binding Dye | Fluidigm | 100-7609 | NA |
| 2x GE Assay Loading Reagent | Fluidigm | 85000802-R | NA |
| 48.48 Dynamic Array IFC for Gene Expression | Fluidigm | BMK-M-48.48 | NA |
| 96.96 Dynamic Array IFC for Gene Expression | Fluidigm | BMK-M-96.96 | NA |
| Anti-Cd11β Antibody | Genway Biotech | CCEC48 | Microglia Stain |
| Anti-NeuN Antibody, clone A60 | EMD Millipore | MAB377 | Neuronal Stain |
| ArcturusXT Laser Capture Microdissection System | Arcturus | NA | NA |
| Biomark HD | Fluidigm | NA | RT-qPCR platform |
| Bovine Serum Antigen | Sigma-Aldrich | B4287 | |
| CapSure Macro LCM Caps | ThermoFisher Scientific | LCM0211 | NA |
| CellDirect One-Step qRT-PCR Kit | ThermoFisher Scientific | 11753500 | Lysis buffer solution components |
| DAPI | ThermoFisher Scientific | 62248 | Nucleus Stain |
| DNA Suspension Buffer | TEKnova | T0221 | |
| Exonuclease I | New Englnad BioLabs, Inc. | M0293S | NA |
| ExtracSure Sample Extraction Device | ThermoFisher Scientific | LCM0208 | NA |
| Fisherbrand Superfrost Plus Microscope Slides | ThermoFisher Scientific | 22-037-246 | Plain glass slides |
| GeneAmp Thin-Walled Reaction Tube | ThermoFisher Scientific | N8010611 | |
| GFAP Monoclonal Antibody | ThermoFisher Scientific | A-21294 | Astrocyte Stain |
| Goat anti-Mouse IgG (H+L), Superclonal™ Recombinant Secondary Antibody, Alexa Fluor 488 | ThermoFisher Scientific | A28175 | Seconadry Antibody |
| IFC Controller | Fluidigm | NA | NA |
| RNaseOut | ThermoFisher Scientific | 10777019 | |
| SsoFast EvaGreen Supermix with Low Rox | Bio-Rad | PN 172-5211 | Rox master mix |
| SuperScript VILO cDNA Synthesis Kit | ThermoFisher Scientific | 11754250 | Contains VILO and SuperScript |
| T4 Gene 32 Protein | New Englnad BioLabs, Inc. | M0300S | NA |
| TaqMan PreAmp Master Mix | ThermoFisher Scientific | 4391128 | NA |
| TE Buffer | TEKnova | T0225 | NA |
Références
- Park, J., et al. Inputs drive cell phenotype variability. Genome Research. 24, 930-941 (2014).
- Park, J., Ogunnaike, B., Schwaber, J., Vadigepalli, R. Identifying functional gene regulatory network phenotypes underlying single cell transcriptional variability. Progress in Biophysics and Molecular Biology. 117, 87-98 (2015).
- Park, J., et al. Single-Cell Transcriptional Analysis Reveals Novel Neuronal Phenotypes and Interaction Networks Involved in the Central Circadian Clock. Frontiers in Neuroscience. 10, 481(2016).
- O'Sullivan, S. J., et al. Single-Cell Glia and Neuron Gene Expression in the Central Amygdala in Opioid Withdrawal Suggests Inflammation With Correlated Gut Dysbiosis. Frontiers in Neuroscience. 13, 665(2019).
- Buettner, F., et al. Computational analysis of cell-to-cell heterogeneity in single cell RNA-sequencing data reveals hidden subpopulations of cells. Nature Biotechnology. 33, 155-160 (2015).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Reviews Immunolology. 18, 35-45 (2018).
- SEQC/MAQC-III Consortium. A comprehensive assessment of RNA-seq accuracy, reproducibility and information content by the Sequencing Quality Control Consortium. Nature Biotechnology. 32, 903-914 (2014).
- Rowin, J., Xia, Y., Jung, B., Sun, J. Gut inflammation and dysbiosis in human motor neuron disease. Physiological Reports. 5, (2017).
- Tamboli, C. P., Neut, C., Desreumaux, P., Colombel, J. F. Dysbiosis in inflammatory bowel disease. Gut. 53, 1-4 (2004).
- Paxinos, G., Watson, C. The Rat Brain in Stereotaxic Coordinates: Hard Cover Edition. , (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon