Method Article
Visualisation des Variants génétiques, quelques cibles et des Mutations ponctuelles dans le cadre de tissu morphologique avec un ARN essai In Situ hybridation avec
Dans cet article
Résumé
Nous décrivons ici une analyse de l’hybridation in situ qui permet la détection sensible et spécifique des séquences aussi courtes que 50 nucléotides avec résolution de simple-nucléotide au niveau unicellulaire. L’analyse, qui peut être réalisé manuellement ou automatiquement, pouvez activer la visualisation des variants d’épissage, courtes séquences et des mutations dans le cadre de tissu.
Résumé
Parce que la médecine de précision dépend fortement de la détection précise des biomarqueurs, il y a un besoin croissant de technologies normalisées et robustes qui mesurent RNA biomarqueurs in situ dans les échantillons cliniques. Tandis qu’essais de mouture-et-bind comme RNAseq et RT-PCR quantitative permettent des mesures d’expression de gène très sensibles, ils exigent également l’extraction de l’ARN et prévenir ainsi les analyse de l’expression précieuse dans le cadre de tissu morphologiques. L’essai in situ hybridation (ISH) avec décrite ici permet de détecter des séquences de l’ARN cible aussi courte que 50 nucléotides à une résolution de simple-nucléotide et au niveau de single-cell. Ce test est complémentaire à l’essai commercial développé précédemment et permet la sensible et spécifique le in situ détection de variants d’épissage, quelques cibles et des mutations ponctuelles dans les tissus. Dans ce protocole, sondes ont été conçus pour cibler les jonctions exon unique pour deux variants d’épissage cliniquement importants, EGFRvIII et METΔ14. La détection de séquences cibles courte a été démontrée par la détection spécifique des séquences CDR3 de T-cellule récepteurs α et β dans la ligne de cellules T Jurkat. Également montré est l’utilité de ce test ISH pour la distinction de séquences d’ARN cible à résolution mononucléotidiques (mutations ponctuelles) par le biais de la visualisation des variations mononucléotidiques EGFR L858R et KRAS G12A lignées cellulaires à l’aide de coloration automatisé plates-formes. En résumé, le protocole montre un essai d’ARN ISH spécialisé qui permet la détection de variants d’épissage, de courtes séquences et mutations in situ pour une performance manuelle et sur des automates.
Introduction
Technologies haut débit transcriptomique tels que les puces à ADN et le séquençage de RNA de génération (RNAseq) améliorent exponentiellement la découverte de biomarqueurs RNA avec valeur clinique diagnostique, pronostique et prédictif de maladies diverses, y compris le cancer1,2. Pour faire progresser l’utilisation de ces biomarqueurs dans la médecine de précision, il y a une grande nécessité de technologies normalisées et robustes qui peut mesurer des biomarqueurs RNA dans le contexte de tissu d’échantillons cliniques. Bien que largement établie grind-lier et dosages comme RNAseq et RT-PCR quantitative activez Mensurations d’expression de gène hautement sensible, l’homogénéisation des tissus requis et ARN impliquent la perte de in vivo la spécificité cellulaire de type et l’information morphologique3. Méthodologies de détection conventionnels en situ ARN n’ont pas la sensibilité et la spécificité nécessaire pour mesurer de manière fiable rares ou peu exprimant des biomarqueurs RNA dans le contexte de tissu4.
Un commercial essai in situ hybridation (ISH) avec (e.g., le test de RNAscope) est une technologie qui a relevé ces défis et permet également la visualisation très sensible et spécifique des molécules d’ARN unique supérieures à 300 nucléotides dans le contexte morphologique de tissu. Ce type de test utilise un oligonucléotide unique sonde d’environ 6 à 20 paires de double-Z sonde combinée avec une amplification de signal avancé basé sur l’hybridation5.
Cette étude décrit une analyse de RNA ISH spécialisée, BaseScope, complémentaires à la technologie commerciale précédemment conçue pouvant détecter des séquences de l’ARN cible aussi courte que 50 nucléotides à une résolution de nucléotide. Ce test aborde la complexité complexe de la transcriptomique et est applicable pour la détection précise des jonctions exon, séquences cibles courte et des mutations ponctuelles dans le cadre de tissu (tableau 1), en utilisant aussi peu qu’une paire de sonde double-Z. Ce rapport montre le protocole d’essai complet et son utilisation dans la détection de variants d’épissage, CDR3 séquences des clones de lymphocytes de récepteurs, et des mutations mononucléotidiques FFIP cell lines et tissus tumoraux.
Protocole
Les échantillons de tumeur humaine utilisés dans cette étude ont été dépersonnalisées et acquis auprès de sources commerciales conformément aux directives éthiques pour la recherche locales.
1. l’échantillon, l’équipement et préparation du réactif
-
Préparation des échantillons FFPE
- Tissus
- Immédiatement après dissection, fixer le tissu (divisée en blocs de 3 à 4 mm d’épaisseur) dans 10 % de formol tamponné neutre (FBN) pour 16 – 32 h à température ambiante (RT).
NOTE : Temps de Fixation variera selon la taille et le type de tissu. - Laver l’échantillon avec 1 x solution saline tamponnée au phosphate (PBS) et mettre en attente à l’aide d’une série standard éthanol (EtOH) (70 % EtOH durant 30 à 60 min, 80 % EtOH durant 30 à 60 min, 90 % EtOH durant 30 à 60 min, 95 % EtOH durant 30 à 60 min, 3 x 100 % pour EtOH 30 – 60 min) suivie de xylène.
- Incorporer l’échantillon dans la paraffine à l’aide de procédures standard6 et découper les blocs de paraffine comme nécessaires pour éliminer les excès paraffine.
Remarque : Taille de bloc variera selon la taille de l’échantillon de tissu, mais une taille typique est de 0,75 x 0,75 pouces2 ou plus petit.
- Immédiatement après dissection, fixer le tissu (divisée en blocs de 3 à 4 mm d’épaisseur) dans 10 % de formol tamponné neutre (FBN) pour 16 – 32 h à température ambiante (RT).
- Lignées cellulaires
- Recueillir et granules des cellules selon les méthodes recommandées pour la lignée cellulaire spécifique.
- Fixer les cellules dans 10 % de formaldéhyde à ta pendant 24 h sur un rotateur.
- Préparer les cellules comme une pastille en gel traitement préchauffée (e.g., Histogel). Permettre le culot à se solidifier en plaçant le gel-culot sur un morceau de parafilm sur la glace et laisser reposer pendant 2 à 3 min. Submerge le culot de gel électrolyte dans du PBS 1 x.
- Déshydrater et incorporer les boulettes de cellule comme indiqué au point 1.1.1.
- Section Préparation
- Couper les tissus/cellules embarqués en 5 sections de ± 1 μm à l’aide d’un microtome, monter les sections sur lames de verre adhésive électrostatiquement et les sécher toute la nuit à température ambiante.
Remarque : Les diapositives peuvent être stockés à RT sous la dessiccation pendant 3 mois. - Déposer les lames dans une grille coulissante et cuire les lames de tissu monté dans un four à circulation d’air à 60 ° C pendant 1 h avant d’effectuer le test.
Remarque : Utilisez les diapositives immédiatement ou stockez-les à RT avec gel de silice pendant 1 semaine. Dégradation de l’ARN peut entraîner un stockage prolongé.
- Couper les tissus/cellules embarqués en 5 sections de ± 1 μm à l’aide d’un microtome, monter les sections sur lames de verre adhésive électrostatiquement et les sécher toute la nuit à température ambiante.
- Tissus
-
Préparation de matériel
- Régler le four de l’hybridation à 40 ° C. Bien mouiller le papier humidification supprimer toute résiduelle dH2O. Insérez le papier dans le bac de contrôle d’humidité et insérez le plateau dans le four de l’hybridation à Préchauffer pendant au moins 30 min avant utilisation.
-
Préparation du réactif
- Remplir deux compensation agent plats avec 200 mL de xylène et de remplir les deux plats de coloration avec 200 mL de 100 % EtOH, qui sera utilisé pour le déparaffinage des sections.
- Préparer, 200 mL de réactif de récupération disponibles dans le commerce 1 x cible en ajoutant 180 mL de dH2O à 20 mL de réactif de récupération 10 x cible. Placer les deux titulaires de glisser dans un cuit-vapeur. Remplir un porte-lames avec 200 mL de 1 x réactif de récupération cible et remplir les autres porte-lames avec 200 mL de dH2O. chaleur les deux solutions à ébullition avec le cuit-vapeur.
- Préchauffé de chaud 50 x tampon de lavage à 40 ° C pendant 10 à 20 min. préparer 3 L 1 x tampon de lavage par dilution de 60 mL de tampon de lavage 50 x avec L 2,94 de dH2O.
- Sous une hotte, préparer hématoxyline de 50 % Gill contre-coloration solution en ajoutant 100 mL de l’hématoxyline Gill dans 100 mL de dH2O. Sous la hotte, préparer le réactif de bleuissement (0,02 % (p/v) d’eau ammoniaque) en ajoutant 1,43 mL d’hydroxyde d’ammonium 28 – 30 % à 250 mL de dH2O.
- Réchauffer au préalable les sondes de la cible à 40 ° C pendant 10 min avant l’hybridation de la sonde et apporter les réactifs d’amplification (0 – 6 AMP) RT.
2. ARN essai In Situ hybridation avec
-
Déparaffinage et déshydratation
- Après la cuisson comme indiqué au point 1.1.3.2 (point d’arrêt facultatif 1), Déparaffiner les sections dans le xylène pendant 5 min avec agitation. Déparaffiner à nouveau dans le xylène frais pendant 5 min Dehydrate dans 100 % EtOH pendant 2 min avec agitation et répéter encore une fois au frais 100 % EtOH pendant 2 min.
- Air sec les diapositives pendant 5 min à 60 ° C dans un four à circulation d’air ou à ta jusqu'à ce qu’ils soient complètement secs (point d’arrêt facultatif 2).
-
Prétraitement de l’échantillon
- Incuber les sections avec ~ 4 gouttes de prêt-à-l’emploi de peroxyde d’hydrogène pendant 10 min à RT pour étancher l’activité peroxydase endogène. Décanter la solution de la glisse et les rincer deux fois avec dH2O.
- Incuber les sections avec 200 mL de réactif récupération cible pour 15 à 30 min à 100 ° C dans un cuit-vapeur.
Remarque : Le temps d’Incubation peut varier selon le type de tissu. Dans ce protocole, récupération de cible a été effectuée pendant 15 min pour les échantillons de tumeur et boulettes de cellule. - Décanter la solution de la glisse, rincer deux fois avec dH2O, trempez-les dans 100 % EtOH pendant 3 min et sécher les lames à 60 ° C dans un four à circulation d’air ou à la droite jusqu'à ce que complètement à sec.
- Dessiner une barrière hydrophobe autour de la section à l’aide d’un stylo hydrophobe, environ 0,75 x 0,75 pouces2.
Remarque : Il n’est pas recommandé pour dessiner une petite barrière. Pour les plus grandes sections, une plus grande barrière devront être tirées. - Laissez la barrière sécher complètement pendant 1 min, ou laisser une nuit au RT (point d’arrêt en option 3).
- Déposer les lames dans un support Dia et placez le support de diapositive dans la barre de contrôle d’humidité. Ajouter 4 gouttes de protéase III à chaque diapositive et les incuber pendant 15 à 30 min à 40 ° C dans le four d’hybridation pour la digestion des protéines.
Remarque : La période d’Incubation peut varier selon le type de tissu. Dans ce protocole, digestion protéasique a été réalisée pour 30 min pour des échantillons de tumeur et 15 min à pellets de la cellule. - Décanter la solution de la glisse et les rincer deux fois avec dH2O.
-
Hybridation de la sonde
- Ajouter 4 gouttes de la solution de sonde appropriée de prêt-à-utiliser pour couvrir la totalité de la section. Si vous utilisez de plus grandes sections, ajouter environ 5 à 6 gouttes.
- Hybrider les sondes pendant 2 h à 40 ° C dans le four de l’hybridation. Décanter la solution de la glisse et laver les diapositives dans 200 mL de 1 x tampon de lavage pendant 2 min à ta avec agitation occasionnelle. Répéter la procédure de lavage dans cette étape.
-
Amplification du signal
- Incuber les sections avec ~ 4 gouttes d’AMP 0 par lame à 40 ° C au four pendant 30 min. décanter la solution hybridation et laver les diapositives dans 200 mL de tampon de lavage 1 x pendant 2 min à ta avec agitation occasionnelle. Répéter la procédure de lavage dans cette étape.
- Incuber les sections avec ~ 4 gouttes de AMP 1 par lame à 40 ° C au four pendant 15 min. décanter la solution hybridation et laver les diapositives dans 200 mL de 1 x tampon de lavage pendant 2 min à ta avec agitation occasionnelle. Répétez la procédure de nettoyage.
- Incuber les sections avec ~ 4 gouttes de AMP 2 par lame à 40 ° C au four pendant 30 min. décanter la solution hybridation et laver les diapositives dans 200 mL de 1 x tampon de lavage pendant 2 min à ta avec agitation occasionnelle. Répétez la procédure de nettoyage.
- Incuber les sections avec ~ 4 gouttes d’huile de 3 AMP par lame à 40 ° C au four pendant 30 min. décanter la solution hybridation et laver les diapositives dans 200 mL de 1 x tampon de lavage pendant 2 min à ta avec agitation occasionnelle. Répétez la procédure de nettoyage.
- Incuber les sections avec ~ 4 gouttes de 4 AMP par lame à 40 ° C au four pendant 15 min. décanter la solution hybridation et laver les diapositives dans 200 mL de 1 x tampon de lavage pendant 2 min à ta avec agitation occasionnelle. Répétez la procédure de nettoyage.
- Incuber les sections avec ~ 4 gouttes d’huile de 5 AMP par lame à ta pendant 30 min. décanter la solution et laver les diapositives dans 200 mL de 1 x tampon de lavage pendant 2 min à ta avec agitation occasionnelle. Répétez la procédure de nettoyage.
- Incuber les sections avec ~ 4 gouttes de 6 AMP par lame à ta pendant 15 min. décanter la solution et laver les diapositives dans 200 mL de 1 x tampon de lavage pendant 2 min à ta avec agitation occasionnelle. Répétez la procédure de nettoyage.
-
Détection de signal
- Préparer le colorant Fast Red, solution de travail. Pour une diapositive avec une barrière de2 0,75 x 0,75 pouces, ajouter 2 μL de la teinture rouge Fast-B à 120 μL de Fast Red-A dans un tube et bien mélanger.
NOTE : Selon la taille de la barrière hydrophobe et le nombre de diapositives, volumes de la solution de travail Fast Red changeront. - Décanter le liquide en excès de la glisse et ajouter le colorant rouge rapide solution aux diapositives de travail. Incuber les lames pendant 10 min à la droite dans la barre d’État avec un couvercle pour éviter l’exposition à la lumière.
Remarque : Utilisez le Fast Red solution moins de 5 min de préparation de travail et ne l’exposez pas aux rayons directs du soleil ou lumière UV. - Décanter la solution Fast Red et rincer les lames deux fois avec l’eau du robinet. Replacez les diapositives dans une grille coulissante.
- Préparer le colorant Fast Red, solution de travail. Pour une diapositive avec une barrière de2 0,75 x 0,75 pouces, ajouter 2 μL de la teinture rouge Fast-B à 120 μL de Fast Red-A dans un tube et bien mélanger.
-
Contre-coloration
- Contre-coloration les coupes de tissus avec une solution de 50 % Gill hématoxyline pendant 2 min à RT. Wash les lames avec l’eau du robinet et répétez cette opération plusieurs fois jusqu'à ce que les lames sont claires, alors que les sections restent pourpres. Tremper les lames dans l’eau ammoniaque 0,02 % pour bleuissement (tremper 2 à 3 fois). Remplacer l’eau de l’ammoniac avec l’eau du robinet et de laver les diapositives 3 à 5 fois.
-
Faites glisser le montage
- Sèche les diapositives dans un four à circulation d’air à 60 ° C pendant 15 min ou à la droite jusqu'à ce que complètement à sec. Placez 1 à 2 gouttes de réactif montage sur chaque diapositive et lieu lamelles couvre-objet sur chaque section. Éviter tout piégeage de bulles d’air. Sécher à l’air les diapositives pendant au moins 5 min.
Remarque : Le substrat Fast Red est sensible à l’alcool. Ne pas déshydrater les diapositives de l’alcool.
- Sèche les diapositives dans un four à circulation d’air à 60 ° C pendant 15 min ou à la droite jusqu'à ce que complètement à sec. Placez 1 à 2 gouttes de réactif montage sur chaque diapositive et lieu lamelles couvre-objet sur chaque section. Éviter tout piégeage de bulles d’air. Sécher à l’air les diapositives pendant au moins 5 min.
-
Visualisation
- Observer les diapositives sous un microscope à fond clair standard.
Résultats
In situ hybridation dosage workflow :
Le flux de travail est représenté dans la Figure 1 et se compose de quatre parties : perméabilisation de cellules ou tissus avec récupération de la cible et protéase solutions, hybridation des sondes d’ARN, de la cible du signal amplification et visualisation du signal. Le signal peut aussi être quantifié à l’aide de systèmes de logiciels d’imagerie numérique ou d’une manière semiquantitative basée sur le nombre de points par cellule. La procédure manuelle décrite à la Figure 2 a également été entièrement automatisée dans les systèmes commerciaux de coloration à l’auto.
Représentant de coloration pour la détection de jonction exon (variante d’épissure de EGFRvIII) : EGFRvIII est une variante du récepteur de facteur de croissance épidermique, qui résulte d’une suppression de génomique dans le cadre des exons 2 à 7, conduisant à la signalisation d’oncogènes constitutivement actif7 . Le test servait à identifier le statut de EGFRvIII dans des échantillons de tumeur FFIP glioblastome (GBM). Seul double-Z sondes ont été conçus pour enjamber les jonctions exon pour détecter soit WT, mutant, ou les deux transcriptions (Figure 3 a). Les sondes WT EGFR couvrent les jonctions des exons 1 et 2 (E1/E2) ou des exons 7 et 8 (E7/E8), alors que les sondes de EGFRvIII spécifiques couvrent la jonction des exons 1 et 8 (E1/E8). Une sonde commune qui s’étend sur la jonction des exons 8 et 9 (E8/E9) était également utilisée pour détecter EGFR total (les deux versions WT et EGFRvIII transcriptions). Toutes les sondes servent ensuite à déterminer le statut EGFR dans les échantillons FFPE GBM. Les deux exemples représentatifs, illustrés à la Figure 3 b ont été tirées d’une étude plus vaste. EGF a été confirmée par une méthode indépendante, RT-PCR. Les deux sondes WT détecté le signal dans les deux échantillons, ce qui indique que les deux échantillons exprimant l’EGFR WT (Figure 3 b). Cependant, la sonde mutante ne montrait de détection de signal dans l’EGFRvIII + échantillon, confirmant que cet échantillon est en effet positif pour la variante de EGFRvIII (Figure 3 b, panneaux de gauche). À l’inverse, la sonde mutante ne détecte pas de signal dans l’EGFRvIII-l’échantillon (Figure 3 b, panneaux de droite). Globalement, ces résultats démontrent que le dosage de jonction exon peut identifier EGFRvIII statut dans des échantillons de tumeur GBM FFPE.
Représentant une coloration pour quelques cibles :
Le CDR3, ou la région déterminante complémentaire 3, est un domaine très variable dans les récepteurs des lymphocytes T. En règle générale, la séquence CDR3 sont assez courts ; par exemple, les séquences de CDR3 α et β de la lignée de cellules T Jurkat sont respectivement de 51 et 48 nucléotides de long (Figure 4 a). Pour identifier les séquences spécifiques de la CDR exprimés dans les cellules Jurkat, sondes antisens pour CDR3 α et β qui sont exprimées dans la lignée de cellules T Jurkat ont été générés, en plus des sondes de sens pour CDR3 α et β pour servir de témoin négatif des sondes. Toutes les sondes ont ensuite été testées dans les cellules Jurkat FFIP préparés avec le test. Une coloration robuste a été observée avec des sondes pour les deux CDR3 anti-sens α et β dans les cellules Jurkat, tandis que le sens des sondes ne détectées peu à aucun signal (Figure 4 b). Ces résultats démontrent la capacité du test objectif court de discerner entre les séquences de CDR très variables, mais courts pour les clones de T-cell receptor.
Représentant une coloration pour point mutation EGFR L858R :
Mutation ponctuelle sondes ont été développées pour détecter les variations de nucléotide et petites insertions ou délétions (INDELs) dans le cadre de la tumeur. Figure 5 a démontre des aptitudes pour la détection in situ de la mutation ponctuelle EGFR L858R (2573T > G). Deux sondes ont été conçus : un pour détecter les L858R muté séquence EGFR et un autre pour détecter la séquence de l’EGFR L858 WT. Les deux sondes ont été testés dans deux lignées cellulaires préparés FFIP : H2229, qui exprime seulement EGFR L858 WT ; et H1975, qui est hétérozygote pour la mutation de l’EGFR L858R. La sonde de mutant L858R détecté le signal que dans la lignée de cellules H1975, mais pas dans la lignée de cellules H2229. Cependant, la sonde WT détecté le signal dans les deux lignées cellulaires. De même, Figure 5 b visualise in situ la détection de la mutation ponctuelle KRAS G12A (35 G > C). Deux sondes ont été conçus pour détecter les séquences KRAS G12A MT et KRAS G12 WT et puis testés sur la lignée cellulaire de HuT78 (qui seulement se déclare KRAS G12 WT) et la lignée de cellules de SW116 (qui est hétérozygote pour la mutation KRAS G12A). Alors que la sonde de KRAS G12 WT détecté le signal dans les deux lignées cellulaires, la sonde KRAS G12A détecté seulement le signal dans la lignée de cellules SW116. Pris ensemble, ces résultats démontrent la capacité technique de l’essai de mutation ponctuelle dans la détection de polymorphismes dans le contexte de cellules et de tissus.
Représentant de coloration pour le test automatisé in situ :
Tests automatisés permettent un plus grand nombre d’échantillons à exécuter de façon plus fiable, minimisant la variabilité inter-utilisateur et temps pratique et générer des résultats reproductibles constamment. Par conséquent, une version automatisée de l’essai a été développée. Pour illustrer la coloration automatisé avec ce dosage, détection de la variante d’épissure METΔ14 a été étudiée. Cette variante est le résultat de 14 d’exon du gène MET étant ignorées pendant le pré-ARNm épissage, qui conduit à l’activation constitutive et transformation oncogénique du récepteur MET8,9. Pour détecter spécifiquement la variante METΔ14, visaient deux sondes de jonction exon : une qui s’étend sur la jonction des exons 13 et 15 (E13/E15) pour détecter la transcription variante de METΔ14 et l’autre qui s’étend sur la jonction des exons 14 et 15 (E14/E15) pour détecter le WT TME transcription (Figure 6 a). Les deux sondes ont ensuite été testées en 2 lignées de cellules FFIP préparés : H596, qui exprime la variante METΔ14, et A549, qui exprime le gène WT a rencontré. Les deux sondes ont montré des modèles d’expressions mutuellement exclusives, avec la sonde E13/E15 seulement détecter de signal dans les cellules de la H596 et la sonde E14/E15 seulement détecter de signal dans les cellules A549 (Figures 6 b et 6C). Enfin, la sonde pour dapB a montré aucun signal n’indiquant aucun signal de fond (Figures 6 b et 6C). Dans l’ensemble, ces données montrent une détection spécifique du MET variant METΔ14 in situ utilisant le test automatisé de BaseScope.
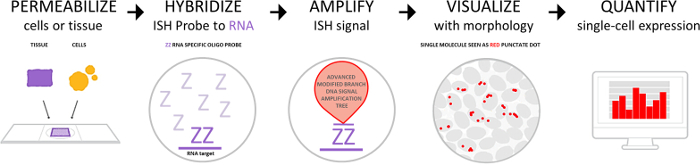
Figure 1 : Le flux de travail test. Le workflow se compose de 4 grandes étapes : prétraitement pour permeabilize des cellules ou tissus, hybridation de la sonde à l’ARN, cible l’amplification du signal et détection de signal par visualisation sous microscope fond clair ou fluorescent. Les points individuels peuvent être quantifiées à l’aide d’une plateforme d’analyse image numérique. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : Illustration du protocole de dosage manuel. Le test complet peut être complété en 9 h. prétraitement, la durée peut varier selon le type de tissu, il est donc conseillé de consulter annexe A du manuel de recommandations de prétraitement de tissu au sujet de la période d’incubation. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 3 : Des images représentatives de détection de jonction exon. (A) montré ici sont l’organisation exon transcriptions EGFR WT et EGFRvIII et un schéma illustrant les sondes de jonction exon double-Z qui chevauchent la jonction pour détecter EGFR WT ou EGFRvIII. (B) l’exon jonction test a été réalisé sur deux échantillons de glioblastome FFIP utilisant les sondes énumérés au point A, ainsi que d’un dapB sonde de contrôle, POLR2A et la sonde de contrôle négatif, positif. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 4 : Détection de séquences images représentatives de cible court. (A) montré ici sont des séquences de CDR3 dans les cellules Jurkat. La séquence en noir est une séquence commune flanquante et la séquence en rouge est unique à CDR3α ou à CDR3β. Sondes de séquence cible court unique double-Z ont été conçus contre ces séquences. (B) le test court cible a été réalisée sur les cellules Jurkat préparés comme un culot cellulaire FFPE en utilisant des sondes anti-sens ou sens ciblant les séquences en (A), et dapB a été utilisé comme contrôle négatif. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
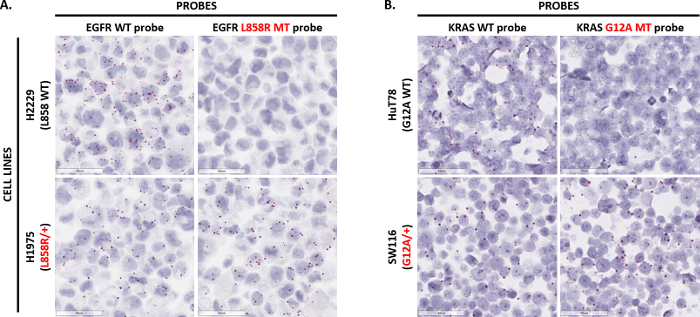
Figure 5 : Des images représentatives de détection de mutation ponctuelle. (A) l’analyse a été réalisée sur H2229 cellules (homozygotes pour L858 EGFR) et H1975 (hétérozygote pour la mutation de l’EGFR L858R) préparé comme un culot à l’aide de sondes de mutation ponctuelle double-Z ciblant la séquence de l’EGFR L858 WT ou EGFR L858R FFIP séquence mutée. (B), l’essai de mutation ponctuelle a été réalisée sur les cellules HuT78 (homozygotes pour KRAS G12A) et les cellules SW116 (hétérozygotes pour la mutation KRAS G12A) préparés comme un culot cellulaire FFIP à l’aide de sondes de mutation ponctuelle double-Z ciblant la séquence KRAS G12 WT ou Séquence G12A KRAS muté. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 6 : Des images représentatives de coloration automatisé avec le test de jonction exon. (A) il est décrit ici est l’organisation exon transcriptions rencontré WT et METΔ14 et un schéma illustrant les sondes de jonction unique double-Z exon qui chevauchent la jonction pour détecter rencontré WT ou METΔ14. (B) et (C), le dosage de jonction exon automatisé a été réalisée sur les cellules de H596 (exprimant METΔ14) et les cellules A549 (exprimant rencontré WT) préparés comme un culot cellulaire FFIP utilisant les sondes énumérés au point A, ainsi que de la dapB de sonde de contrôle négatif. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Jonction exon | Courte séquence | Mutation ponctuelle |
| Variante/isoforme d’épissure | Séquences entre 50 et 300 nt | Mutation ponctuelle |
| RNA circulaire (circRNA) | Séquences très homologues | Indel court |
| Fusion de gènes | CDR3 séquence des clones TCR | Gène d’édition |
| Knockout (KO) de la gène | Pré-miARN | |
| Petits ARN nucléolaire (ARNpno) | ||
| Gène d’édition |
Tableau 1 : demandes de la in situ dosage. Il existe 3 grandes catégories pour les applications de ce test : jonction exon, séquence cible court et mutation ponctuelle. Chaque colonne sont quelques exemples d’application spécifiques pour chaque catégorie.
Discussion
Dans ce rapport, le nouveau protocole d’essai ISH et ses demandes ont été examinées en détail. Le dosage permet la visualisation directe des jonctions exon, séquences courtes-cible et hautement homologues et des mutations ponctuelles dans le cadre de tissu. Le test est basé sur la technologie de RNAscope5 et est donc capable de détection de molécules simples. Toutefois, en raison d’un système d’amplification avancé, le signal peut être détecté avec des sondes contenant aussi peu qu’une paire de double-Z ou d’une longueur de modèle cible de seulement 50 nucléotides. Parce que les sondes peuvent être aussi courts qu’une seule double-Z dans la longueur, cela permet la détection des jonctions exon, séquences courtes-cible et hautement homologues et mutations ponctuelles (tableau 1).
Pour les bons résultats de l’essai, il y a plusieurs recommandations techniques. Tout d’abord, les tissus doivent être fixés dans du formol tamponné neutre en 10 % frais (NBF) à température ambiante pendant 16 – 32 h10. Underfixation (< 16 h) ou overfixation (> 32 h) empêchera la performance du test et peut nécessiter une optimisation supplémentaire. En second lieu, afin d’assurer un contrôle optimal de la température et l’humidité, qui sont nécessaires pour l’amplification de l’hybridation et signal sonde robuste, la diapositive du système et l’hybridation four de traitement doit être utilisée pour obtenir la procédure protocole 2,3 à 5 (sonde de prétraitement, protéase l’hybridation, amplification du signal et détection de signal). Troisième, l’excès de tampons résiduelles doivent être convenablement décanté avant chaque étape tout au long du protocole (mais pas tellement que les coupes de tissus dessèchent). Si les diapositives s’assèchent, signal non spécifique important se développe. Quatrièmement, selon le type de tissu, optimisation de prétraitement peut être nécessaire. À l’aide de la protéase tort ou d’effectuer une durée sous-optimale peut entraîner en sous - ou over - digestion et affectera négativement le signal. Enfin, il est important de toujours exécuter les contrôles positifs et négatifs avec les sondes de test. Sondes de contrôle négatif s’assurer qu’il n’y a aucun signal de fond, et des sondes de contrôle positif, s’assurer que le test a été fait correctement et que la qualité du RNA dans l’échantillon est optimale pour l’interprétation des résultats de la sonde. S’il n’a pas de signal avec la sonde de contrôle positif, alors la qualité RNA dans l’échantillon est probable sous-optimal et un signal ne peut pas s’afficher avec la sonde de test.
En plus de l’analyse manuelle, la possibilité d’effectuer le test sur des automates a été également démontrée (Figure 6). Ce dosage automatisé de ISH donne un ratio signal-bruit élevé et est applicable pour les mêmes applications, comme le montre le tableau 1; Toutefois, les avantages d’un test automatisé incluent la normalisation des conditions expérimentales, minimisation de la variabilité inter-utilisateur et de temps de pratique et provision pour le criblage à haut débit d’échantillons de tissus de manière fiable.
Alors que l’immunohistochimie (IHC) et qRT-PCR permettent pour la détection de variants d’épissage (particulièrement EGFRvIII), dans les échantillons cliniques FFIP, ces techniques peuvent manquer la spécificité nécessaire et n’offrent pas d’aperçu de la résolution spatiale de l’épissure expression de type Variant, respectivement11,12. Un avantage majeur du test dans le présent protocole est sa visualisation très sensible et spécifique des jonctions d’épissure tout en préservant le cadre tissu morphologiques. Ici, capacité de l’essai de cibler précisément les jonctions exon unique pour plusieurs variants d’épissage, y compris l’EGFRvIII et METΔ14, a été démontrée (Figures 3 et 6). En outre, le test s’est avéré de détecter la variante d’épissure AR-V7 dans le cancer de la prostate, plusieurs isoformes de ErbB4 dans le cerveau et la confirmation de la débouchure de Cdr1as d’ARN circulaire dans les souris cerveau3,13,14.
Détection de la cible courte séquence de ce dosage ISH permet pour la visualisation de séquences d’ARN aussi courtes que 50 nucléotides de long, comme en témoigne la détection de séquences CDR3 dérivées de cellules Jurkat (Figure 3). Le test permet aussi de détecter des séquences de gènes qui sont hautement homologues aux autres membres de la famille ou des espèces, comme indiqué par Revêchon et al., qui ont utilisé le test court cible pour détecter les progerin humain exprimé dans la souris blanche du tissu adipeux sous-cutané15. En outre, petits ARN nucléolaire (ARNpno), la modification de gènes CRISPR et précurseur-micro-ARN peuvent être détecté in situ avec le test court cible. Plus récemment, Fu al associé ce dosage ISH IHC pour identifier les cellules précises dans la rétine exprimant le pré-miARN mir125b16.
Mutation de profilage dans les tumeurs est essentielle pour l’étude de la progression des tumeurs et pour le développement de thérapies ciblées. Alors que la mutation de profilage peut être atteint par l’intermédiaire de séquençage haut-débit, cette technologie ne peuvent pas résoudre totalement intratumorale des altérations génétiques hétérogénéité ou un lien avec la morphologie cellulaire17,18. La détection des mutations ponctuelles à l’aide de ce test ISH permet la distinction de séquences d’ARN cible à une résolution de base unique, tel qu’il est validé par la détection des variations de nucléotide de EGFR L858R et G12A KRAS dans les lignées cellulaires (Figure 5). En outre, Baker et al utilisé l’essai de mutation ponctuelle pour cibler plusieurs mutations dans les oncogènes BRAF, KRAS et PIK3CA dans le cancer colorectal,18. Ils ont pu identifier et dans l’espace carte rares sous-clones mutants des cellules tumorales, en fin de compte montrant la manière dont ils contribuent à l’hétérogénéité intra-tumeur.
En résumé, un dosage de RNA ISH spécialisé a été développé. Cette méthodologie permet une détection de variants d’épissage, de courtes séquences et mutations in situ. Il est sensible, précis, quantifiables et adaptables à la performance par des méthodes manuelles et sur des automates.
Déclarations de divulgation
Tous les auteurs sont employés par Advanced Cell Diagnostics, Inc.
matériels
| Name | Company | Catalog Number | Comments |
| HybEZ Oven (110 or 220 VAC) or HybEZ II Oven (110 or 220V) | ACD | 310010 or 310013 (HybEZ™), 321710 or 321720 (HybEZ™ II) | |
| HybEZ Humidity Control Tray (with lid) | ACD | 310012 | |
| ACD EZ-Batch Slide Rack (20 slide capacity) 1 rack 310017 | ACD | 310017 | |
| HybEZ Humidifying Paper | ACD | 310015 | |
| ImmEdge Hydrophobic Barrier Pen (required) | Vector Laboratory | H-4000 | |
| SuperFrost Plus Slides (required) | Fisher Scientific | 12-550-15 | |
| 10% neutral-buffered formalin (NBF) | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Paraffin wax | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Microtome | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Gill’s Hematoxylin I | American Master Tech Scientific/MLS | HXGHE1LT | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Xylene | Fisher Scientific/MLS | X3P-1GAL | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tissue-Tek Vertical 24 Slide Rack | American Master Tech Scientific/MLS | LWSRA24 | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tissue-Tek Staining Dishes | American Master Tech Scientific/MLS | LWT4457EA | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tissue-Tek Clearing Agent Dishes, xylene resistant | American Master Tech Scientific/MLS | LWT4456EA | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| 100% alcohol (EtOH) | American Master Tech Scientific/MLS | ALREACS | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| VectaMount Permanent Mounting Medium (required) | Vector Labs | H-5000 | |
| Cover Glass, 24 mm x 50 mm | Fisher Scientific/MLS | 12-545-F | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Ammonium hydroxide, 28–30% | Sigma-Aldrich/MLS | 320145-500mL | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Carboy (>3L) | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Oster Steamer Model 5712, Black and Decker Steamer HS3000, or the Braun Multiquick FS 20 Steamer | / | / | |
| Digital thermometer | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Water bath or incubator, capable of holding temperature at 40 +/– 1 °C | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Pipettors and tips, 1–1,000 μL | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Distilled water | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tubes (various sizes) | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Fume hood | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Graduated cylinder | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Parafilm | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Paper towel or absorbent paper | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Microcentrifuge | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Microscope and accessories | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Drying oven, capable of holding temperature at 60 +/– 1 °C | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Formaldehyde | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Histogel | Fisher Scientific/MLS | 22-110-678 | |
| BaseScope Reagent Kit - RED | Advanced Cell Diagnostics | 322900 | |
| BaseScope Hs-EGFR-E1E2 | Advanced Cell Diagnostics | 701701 | |
| BaseScope Hs-EGFR-E1E8 | Advanced Cell Diagnostics | 701711 | |
| BaseScope Hs-EGFR-E7E8 | Advanced Cell Diagnostics | 701721 | |
| BaseScope Hs-EGFR-E8E9 | Advanced Cell Diagnostics | 701731 | |
| BaseScope Hs-MET-E14E15 | Advanced Cell Diagnostics | 701811 | |
| BaseScope Hs-MET-E13E15 | Advanced Cell Diagnostics | 701801 | |
| BaseScope Hs-KRAS-G12A | Advanced Cell Diagnostics | 705491 | |
| BaseScope Hs-KRAS-G12-nt35WT | Advanced Cell Diagnostics | 705531 | |
| BaseScope Hs-EGFR-L858R | Advanced Cell Diagnostics | 705451 | |
| BaseScope Hs-EGFR-L858WT | Advanced Cell Diagnostics | 705461 | |
| BaseScope Control Probe Pack Human | Advanced Cell Diagnostics | 322975 |
Références
- Bolha, L., Ravnik-Glavač, M., Glavač, D. Circular RNAs: Biogenesis, Function, and a Role as Possible Cancer Biomarkers. International Journal of Genomics. 2017, 6218353 (2017).
- Yamada, A., Yu, P., Lin, W., Okugawa, Y., Boland, C. R., Goel, A. A RNA-Sequencing approach for the identification of novel long non-coding RNA biomarkers in colorectal cancer. Scientific Reports. 8 (1), 575 (2018).
- Erben, L., He, M. X., Laeremans, A., Park, E., Buonanno, A. A Novel Ultrasensitive In Situ Hybridization Approach to Detect Short Sequences and Splice Variants with Cellular Resolution. Molecular Neurobiology. , (2017).
- Mahmood, R., Mason, I. In-situ hybridization of radioactive riboprobes to RNA in tissue sections. Methods in Molecular Biology. 461, 675-686 (2008).
- Wang, F., et al. RNAscope: a novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Paraffin processing of tissue. Protocols Online Available from: https://www.protocolsonline.com/histology/sample-preparation/paraffin-processing-of-tissue/ (2016)
- An, Z., Aksoy, O., Zheng, T., Fan, Q. W., Weiss, W. A. Epidermal growth factor receptor and EGFRvIII in glioblastoma: signaling pathways and targeted therapies. Oncogene. , (2018).
- Frampton, G. M., et al. Activation of MET via diverse exon 14 splicing alterations occurs in multiple tumor types and confers clinical sensitivity to MET inhibitors. Cancer Discovery. 5 (8), 850-859 (2015).
- Awad, M. M., et al. MET Exon 14 Mutations in Non-Small-Cell Lung Cancer Are Associated With Advanced Age and Stage-Dependent MET Genomic Amplification and c-Met Overexpression. Journal of Clinical Oncology. 34 (7), 721-730 (2016).
- Hammond, M. E., et al. American Society of Clinical Oncology/College of American Pathologists guideline recommendations for immunohistochemical testing of estrogen and progesterone receptors in breast cancer. Archives of Pathology and Laboratory Medicine. 134 (6), 907-922 (2010).
- Gan, H. K., Cvrljevic, A. N., Johns, T. G. The epidermal growth factor receptor variant III (EGFRvIII): where wild things are altered. Federation of European Biochemical Societies Journal. 280 (21), 5350-5370 (2013).
- Wheeler, S. E., Egloff, A. M., Wang, L., James, C. D., Hammerman, P. S., Grandis, J. R. Challenges in EGFRvIII detection in head and neck squamous cell carcinoma. Public Library of Science One. 10 (2), e0117781 (2015).
- Zhu, Y., et al. Novel Junction-specific and Quantifiable In Situ Detection of AR-V7 and its Clinical Correlates in Metastatic Castration-resistant Prostate Cancer. European Urology. , (2017).
- Piwecka, M., et al. Loss of a mammalian circular RNA locus causes miRNA deregulation and affects brain function. Science. , (2017).
- Revêchon, G., et al. Rare progerin-expressing preadipocytes and adipocytes contribute to tissue depletion over time. Scientific Reports. 7 (1), 4405 (2017).
- Fu, Y., et al. Functional ectopic neuritogenesis by retinal rod bipolar cells is regulated by miR-125b-5p during retinal remodeling in RCS rats. Scientific Reports. 7 (1), 1011 (2017).
- Yates, L. R., et al. Subclonal diversification of primary breast cancer revealed by multiregion sequencing. Nature Medicine. 21 (7), 751-759 (2015).
- Baker, A. M., et al. Robust RNA-based in situ mutation detection delineates colorectal cancer subclonal evolution. Nature Communications. 8 (1), 1998 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon