Method Article
Pyrosequencing pour l'identification microbienne et Caractérisation
Dans cet article
Résumé
Pyroséquençage est une technique polyvalent qui facilite le séquençage du génome microbien qui peut être utilisé pour identifier des espèces bactériennes, de discriminer les souches bactériennes, et de détecter des mutations génétiques qui confèrent une résistance aux agents anti-microbiens. Dans cette vidéo, la procédure pour la production microbienne de l'amplicon, amplicon pyrosequencing et analyse de la séquence d'ADN sera démontrée.
Résumé
Pyroséquençage est une technique polyvalent qui facilite le séquençage du génome microbien qui peut être utilisé pour identifier des espèces bactériennes, de discriminer les souches bactériennes et de détecter des mutations génétiques qui confèrent une résistance aux agents anti-microbiens. Les avantages de pyroséquençage pour des applications microbiologiques comprennent criblage à haut débit rapide et fiable et une identification précise des microbes et des mutations du génome microbien. Pyroséquençage comprend le séquençage de l'ADN en synthétisant le brin complémentaire d'une base unique à la fois, tandis que la détermination de l'être nucléotidique spécifique incorporé au cours de la réaction de synthèse. La réaction se produit sur la matrice ADN simple brin immobilisé, où les quatre désoxyribonucléotides (dNTP) sont ajoutés de façon séquentielle et les dNTP non incorporées sont dégradés par voie enzymatique avant l'addition de la prochaine dNTP à la réaction de synthèse. Détection de la base spécifique incorporé dans la matrice est contrôlée par la génération d'chemilumsignaux inescent. L'ordre des dNTP qui produisent des signaux chimiluminescents détermine la séquence d'ADN de la matrice. La capacité de séquençage en temps réel de la technologie de pyroséquençage permet l'identification microbienne rapide dans un seul essai. En outre, l'instrument pyroséquençage, permet d'analyser la diversité génétique complète de la résistance aux médicaments anti-microbienne, y compris le typage des SNP, mutations ponctuelles, insertions et suppressions, ainsi que la quantification de multiples copies du gène qui peuvent survenir dans une certaine résistance anti-microbienne motifs.
Introduction
Pyroséquençage est une méthode rapide et précise aux acides nucléiques de séquence qui est basé sur le principe de la "séquençage par synthèse". "Le séquençage par synthèse" implique l'utilisation d'une seule matrice d'ADN de brin pour synthétiser le brin complémentaire d'une base en même temps, et la détection de la base constituée (A, T, G ou C), à chaque étape de détection d'un signal chimiluminescent. La réaction pyroséquençage comprend matrice d'ADN, dNTP (dATP, dGTP, dTTP, dCTP), l'ADN polymérase, ATP sulfurylase, la luciférine, la luciférase, et apyrase. La réaction de synthèse est initiée par l'addition de l'un des quatre dNTP et de l'ADN polymérase de l'ADN matrice simple brin biotinylé. ADN polymérase incorpore le dNTP complémentaire sur le modèle, avec la libération subséquente de pyrophosphate (figure 1). ATP sulfurylase convertit proportionnellement le pyrophosphate à l'ATP. ATP agit comme un catalyseur pour la conversion de l'intermédiaire de luciférase de la luciférine à oxyluciférine, quigénère de la lumière (Figure 2). L'intensité de la lumière est proportionnelle au nombre de nucléotides incorporés et détermine s'il ya une ou plusieurs dNTP spécifique (dATP, dTTP, dGTP, dCTP ou) est présent sur le brin matrice de façon séquentielle (figure 3). Toute dNTP non incorporée est dégradée par l'apyrase avant l'addition de la prochaine dNTP pour la poursuite de la réaction de synthèse. Cette "séquençage par synthèse" réaction est répété avec addition de chacune des quatre dNTP jusqu'à ce que la séquence d'ADN de la matrice d'ADN monocaténaire est déterminée.
Pour visualiser une animation de la réaction pyroséquençage Cliquez ici pour voir le film .
Protocole
Introduction: Une colonie bactérienne unique devrait être utilisée pour inoculer un bouillon de culture approprié qui est incubé pendant une nuit. Un culot bactérien est ensuite traitée pour purifier l'ADN génomique en utilisant un kit d'isolation de génomique disponible dans le commerce. L'ADN génomique purifié doit avoir un rapport 260/280 (nm)> 1,8 à assurer que l'échantillon est exempt de contamination par des protéines. La quantité d'ADN génomique nécessaire pour la génération de la matrice est de 10 à 20 ng pyroséquençage.
A. amplification de la matrice
(Manuel PCR PyroMark; www.qiagen.com / manuels 1)
Mise en place de la réaction PCR
Note: Une amorce doit être biotinylé à son extrémité 5 ', et il est recommandé qu'elle soit purifiée par HPLC pour la préparation du modèle. Nous recommandons l'PyroMark Assay conception Software 2.0 pour la PCR et seconception d'amorce de séquence qui sont optimisés pour le séquençage de courte lecture, cependant, Primer-BLAST ( http://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome ) de NCBI pouvez également être utilisé pour la conception d'amorces.
- Décongeler les solutions requises (listées dans le tableau 1) et mélanger soigneusement chaque. Mettre en place la réaction selon le tableau 1. Tout le travail peut être fait à la température ambiante.
Note: Le PCR Master Mix contient MgCl2 pour une concentration finale de 1,5 mm, ce qui donne des résultats satisfaisants dans la plupart des cas. Si un Mg 2 + concentration plus élevée est requise, jusqu'à 3,5 ul de 25 mM MgCl 2 peut être ajouté à une réaction.
- Bien mélanger le mélange réactionnel par un léger pipetage de haut en bas, puis distribuer des volumes appropriés dans des tubes PCR.
- Ajouter template ADN dans chaque tube PCR. Nous recommandons 10 ng d'ADN génomique.
- Programmer le thermocycleur selon le tableau 2. Une prise finale à 4 ° C est recommandé pour plus de commodité. Si vous utilisez un cycleur thermique sans couvercle chauffant, les réactions de superposition avec huile minérale 100 pi.
- Placer les tubes PCR dans le thermocycleur et démarrer le programme de cyclisme.
B. vide Protocole Workstation
Immobiliser les produits de PCR biotinylés
- Faites un mélange-maître selon la figure 4.
Remarque: Avant de pipetage, secouez doucement la bouteille de billes de Sepharose revêtues de streptavidine pour assurer une suspension homogène.
- Selon le volume de produit de PCR utilisé, dispenser 60-75 ul mélange maître dans chaque puits d'une plaque PCR pour obtenir un volume total de 80 pi par puits.
- Ajouter 5-20 ul produit PCR biotinylé à chaque puits.
- Sceller le puits avec bande caps et agiter la plaque PCR à 1400 rpm pendant 5-10 min à température ambiante (15-25 ° C) en utilisant un agitateur orbital.
La dénaturation de l'ADN et l'addition d'amorce de séquençage
- Diluer les amorces de séquençage à 0,3 um avec un tampon d'hybridation et de distribuer 25 ul dans chaque puits d'une plaque de réaction. Placer la plaque sur le poste de travail.
- Remplir les auges de poste de travail selon le schéma de la figure 5.
- Tournez sur la pompe à vide, et assurez-vous que l'interrupteur de l'outil à vide est activée (Figure 6). Rincer les sondes filtre à l'eau de haute pureté (Milli-Q 18,2 MW x cm ou équivalent) en creux 5 (figure 5). Remplir le creux avec de l'eau de haute pureté frais pour l'utilisation dans l'étape 12.
- En travaillant rapidement, positionner la plaque PCR avec des perles sur le poste de travail. S'assurer que les deux plaques sont dans la même orientation que quand les échantillons ont été chargés.
- Avec le passage à vide en marche, abaissez le videoutil dans les puits de la plaque PCR pendant 20 secondes ou jusqu'à ce que le volume a été aspiré. Les billes seront capturés par l'outil à vide (figure 7).
- Avec le vide toujours ON, rincer l'outil avec 70% d'éthanol (bac 1) pendant 20 secondes ou jusqu'à ce que le volume a été aspiré.
- Avec vide ON, rincer l'outil avec une solution de dénaturation (bac 2) pendant 20 secondes ou jusqu'à ce que le volume a été aspiré.
Note: La Solution de dénaturation contient de l'hydroxyde de sodium, qui est un irritant oculaire et cutanée. Toujours porter une blouse, des gants et des lunettes de protection.
- Avec ON vide, rincer avec l'outil tampon de lavage (bac 3) pendant 20 secondes ou jusqu'à ce que le volume a été aspiré.
- Avec vide ON, soulever l'outil à vide pour au-delà de 90 ° à la verticale pendant 5 secondes, pour permettre à tout le liquide à s'écouler hors de l'outil à vide. Arrêtez la pompe et aligner l'outil à vide avec la plaque de réaction. Abaisser l'outil à vide dans l'puits et agiter doucement de gauche à droite pour libérer les billes dans le contenant amorce de séquençage puits.
- Afin de nettoyer l'outil à vide, agiter les sondes filtre à l'eau de haute pureté (bac 4) pendant 10 sec.
- Passez l'aspirateur en marche et rincer l'outil avec de l'eau de haute pureté (creux 5) pendant 20 secondes ou jusqu'à ce que le volume a été aspiré.
- Soulevez l'outil à vide au-delà de 90 ° C vertical pendant 5 secondes, puis passer l'aspirateur hors tension et stocker dans la position "parking".
Recuit amorce de séquençage de brins d'ADN
- Placer la plaque de réaction dans un support de plaque de pré-chauffé.
- Chauffer la plaque sur un bloc de chauffage à 80 ° C pendant 2 min.
- Retirer la plaque du support et le déposer sur le comptoir pour refroidir à la température ambiante pendant 5 min.
PyroMark Q24 opération
Note: Allumer l'appareil en utilisant l'interrupteur d'alimentation situé au-dessus du cordon d'alimentation. Étoiletup peut prendre jusqu'à 1 min.
- Remplir une cartouche en suivant les instructions fournies dans le PyroMark Or Q24 Réactifs Manuel 2. Pour déterminer les volumes nécessaires, mettre en place un fichier courir dans le logiciel de l'instrument, et sous le menu Outils, sélectionnez Information préalable Run
- Si l'instrument est déjà allumé, veiller à ce qu'il ne remplit pas une course, puis ouvrez le couvercle. Un signal d'avertissement sonore est émise lorsque le couvercle est ouvert quand il est dangereux de faire.
- Ouvrez la porte cartouche et insérez la cartouche avec l'étiquette vers l'avant (figure 8). Assurez-vous que la cartouche est insérée correctement (une ligne doit être visible à l'avant) et fermer la porte.
- Ouvrez le cadre plaque de rétention et positionner la plaque avec des échantillons à l'intérieur de l'instrument.
- Fermer le châssis de plaque de maintien et le couvercle de l'appareil (figure 9).
Démarrage d'une course
- Insérez la clé USB avec le fichier d'exécutions créé avec le logiciel de l'appareil dans le port USB à l'avant de l'instrument.
- Sélectionnez "Exécuter" dans le menu principal et appuyez sur "OK".
- Utilisez les flèches de défilement pour sélectionner le fichier d'exécution désiré et appuyez sur "Select".
Note: L'instrument commence la distribution de réactifs lorsque tous les niveaux de pression et de température prédéfini est atteint (cela peut prendre plusieurs minutes). Au cours d'une course, l'écran de l'appareil affiche le pyrogramme du puits sélectionnés en temps réel. Utilisez les flèches de défilement pour afficher le pyrogramme d'autres puits.
Remplir une course
- Lorsque l'instrument confirme que la course est terminée et le fichier d'exécution a été enregistré sur la clé USB, appuyez sur "Fermer".
- Retirez la clé USB. Si elle a été retirée avant la course terminée, insérez la clé USB. Sélectionnez "Administration" puis "Copier Runs non enregistrées».
- Le fichier d'exécution peut être ouvert et analysées en utilisant le logiciel de l'appareil et comparéà une bibliothèque de microbes connus en utilisant le logiciel Identifire.
Résultats
Les résultats d'une course de pyrosequencing typique utilisant le logiciel montre l'emplacement des amorces sens et antisens utilisées pour la production d'amplicon, et l'amorce de séquençage utilisée pour la réaction de pyroséquençage dans le 16S séquence ribosomique conservé (Figure 10). L'séquence hypervariable entre le primaire des sites permet d'identification bactérienne d'un grand nombre d'espèces bactériennes à l'aide de l'amorce de séquençage conservée (5'-TACATGCAAGTCGA). Comme un contrôle interne de la qualité, la mise entre parenthèses de la séquence d'ADN de la région variable peut être contrôlé pour assurer que la région d'ADN correcte a été analysé . Le logiciel permet de comparer pyroséquençage et l'alignement de la séquence générée à une base de données interne de séquences ribosomiques bactériens d'identification bactérienne. En outre, une séquence peut être analysé afin de déterminer les mutations qui confèrent une résistance aux antibiotiques. Par exemple, l'analyse des mutations dans les gènes ribosomique 23S de Helicobpylori tère montre deux profils de mutation (GAA ou AGA) qui confèrent une résistance aux antibiotiques (Figure 11). L'analyse de plusieurs isolats du même patient peut être en même temps dosé pour suivre l'émergence de la résistance aux médicaments au fil du temps pour les enquêtes épidémiologique des foyers de résistance aux médicaments microbiennes.
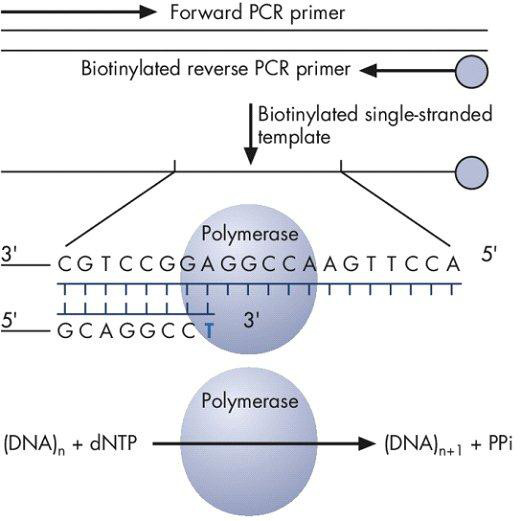
Figure 1. Produit PCR biotinylé est utilisé comme modèle pour incorporer dNTP par l'ADN polymérase, conduisant à la génération de pyrophosphate (PPi).
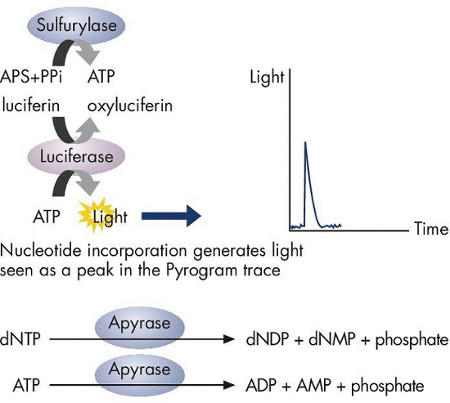
Figure 2. ATP sulfurylase convertit proportionnellement pyrophosphate de l'ATP. ATP agit comme un catalyseur pour la conversion de l'intermédiaire de luciférase de la luciférine à oxyluciférine, qui génère de la lumière qui est proproportionnel à la quantité d'ATP. La lumière est enregistrée en pointe sur la trace de pyrogramme et indique incorporation de nucléotides. Les dNTPs non constituées en société sont dégradées par apyrase avant la prochaine dNTP est ajouté pour la suite de la synthèse.

Figure 3. L'intensité de la lumière générée indique si a été constituée de une ou plusieurs dNTP spécifique (dATP, dTTP, dGTP, dCTP ou) sur le brin matrice de manière séquentielle.
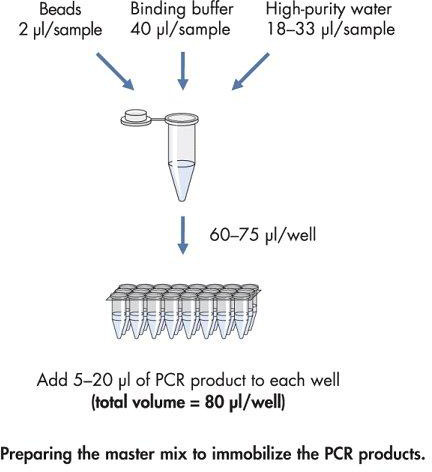
Figure 4. Diagramme pour la préparation du master mix pour immobiliser le produit PCR biotinylé.

GE = "always"> Figure 5. PyroMark poste de travail, avec plaque PCR, plaque PyroMark et lieux de dépression.

Figure 6. Emplacements de l'interrupteur à vide positions ON et OFF.

Figure 7. outil à vide. d'une manipulation correcte de l'outil à vide.

Figure 8. Porte cartouche ouverte avec cartouche en place.

Figure 9. Cartouche insérée correctement avec la porte fermée.

Figure 10. Pyroséquençage résultats de l'identification de bactéries à base de. Amorces de PCR sont conçus pour les régions conservées de la matrice d'ADN et l'amorce de séquençage est positionné immédiatement en amont d'une identification de la séquence d'ADN hypervariable bien caractérisé dans le amplicon (en bleu).

Figure 11. La détection de la résistance aux antibiotiques dans Helicobacter pylori en utilisant pyrosequencing. Analyse des mutations dans les gènes 23S qui confèrent une résistance antibactérienne à Helicobacter pylori contenant la GAA ou séquences AGA. Cliquez ici pour agrandir la figure .
| Composant | Volume par réaction | Concentration finale |
| Pyro PCR Master Mix, 2x | 12,5 pi | 1x |
| Concentré CoralLoad, 10x | 2,5 pl | 1x |
| MgCl2 25 mM (facultatif) | Variable | ≥ 1,5 mM |
| Q-Solution, 5x (facultatif) | 5 pl | 1x |
| Primer A / B Primer | Variable / Variable | 0,2 μM/0.2 iM |
| Eau sans RNase | Variable | - |
| Volume total (après avoir ajouté l'ADN matrice) | 25 pl |
Tableau 1. PCR mélange réactionnel.
| & Nbsp; | Autres commentaires | ||
| Étape d'activation initiale PCR | 15 min | 95 ° C | HotStartTaq ADN polymérase est activé |
| 3 étape cyclisme: Dénaturation | 30 sec | 94 ° C | |
| Recuit | 30 sec | 60 ° C 56 ° C | Pour l'ADN génomique Pour l'ADN converti au bisulfite |
| Prolongement | 30 sec | 72 ° C | |
| Nombre de cycles | 45 | ||
| Extension finale | 10 min | 72 ° C |
Tableau 2. PCR caractéristiques du cycle.
Discussion
Plusieurs nouvelles méthodes de séquençage de nouvelle génération ont été commercialisés. Trois des méthodes les plus couramment utilisés sont le séquençage ion des semi-conducteurs, le séquençage en temps réel de la molécule unique, et le séquençage par synthèse (SBS). 3-11 Chacune de ces méthodes dépend de séquençage par synthèse, mais emploient de nouvelles plates-formes pour la détection des nucléotides incorporés. Dans ion semiconducteur séquençage, un simple brin d'ADN servant de matrice pour le brin de séquençage. Polymérase et 12 nucléotides sont ajoutés séquentiellement comme dans pyroséquençage. Quand un nucléotide est ajouté au brin d'ADN en croissance, un ion d'hydrogène est libéré. L'ion d'hydrogène est détecté par un capteur à effet à base de transistors de champ.
Molécule unité séquençage en temps réel dépend du guide d'ondes en mode zéro (ZMW). 13 Le ZMW est une petite structure où une seule molécule de la polymérase est fixé au fond du puits. Il est éclairé de telle manière qu'une floreMolécule d'odeur peut être détectée. Chaque nucléotide est étiqueté avec une molécule fluorescente. Comme un nucléotide marqué par fluorescence est incorporé, un détecteur identifie le nucléotide. Lorsque le nucléotide suivant est ajouté, la molécule fluorescente est clivé. 14
Le séquençage par synthèse (SBS) utilise une méthode unique pour amplifier l'ADN cible de sorte que des groupes de séquences uniques sont générés. 15 gabarits simples brin sont générées, puis le brin complémentaire est synthétisé. Chaque nucléotide est marqué avec une molécule fluorescente et après chaque addition de base de la fluorescence de la base ajoutée est enregistrée.
Déclarations de divulgation
Production et le libre accès à cet article est parrainé par Qiagen.
Auteurs Ahmed, Durocher, Jessen et Vardi sont des employés de Qiagen Inc. qui produit des réactifs et des instruments utilisés dans cet article.
Remerciements
Ce projet a appuyé en partie par l'Université Johns Hopkins, Bureau du prévôt par le Science Initiative passerelle et Qiagen Inc.
matériels
| Name | Company | Catalog Number | Comments |
| PyroMark PCR Master Mix, 2x | Qiagen | 978703 | |
| CoralLoad Concentrate, 10x | Qiagen | 978703, 978705 | |
| Q-Solution, 5x | Qiagen | 978703, 978705 | |
| MgCl2, 25 mM | Qiagen | 978703, 978705 | |
| RNase-Free Water | Qiagen | 129112 | |
| Primers | Qiagen | 978776 or 978777 | Primers should be purchased from an established oligonucleotide manufacturer (i.e. QIAGEN Pyromark Custom Assays, IDT, etc). Lyophilized primers should be dissolved in TE to provide a stock solution of 100 μM. |
| Pyromark Gold Q24 Reagents | Qiagen | 970802 | |
| High-purity water (Milli-Q 18.2 MΩ x cm or equivalent) | |||
| Streptavidin Sepharose High Performance Beads | GE Healthcare | 17-5113-07 | |
| PyroMark Annealing Buffer | Qiagen | 979009 | |
| PyroMark Denaturation Solution | Qiagen | 979007 | |
| PyroMark Wash Buffer | Qiagen | 979008 | |
| PyroMark Q24 | Qiagen | 9001514 | |
| PyroMark Q24 Cartridge | Qiagen | 979202 | |
| PyroMark Q96 HS Tip Holder Box | Qiagen | 979105 | |
| PyroMark Q24 Vacuum Workstation | Qiagen | 9001516 | |
| PyroMark Q24 Plate Holder | Qiagen | 9022273 | |
| Orbital shaker | Fisher | 11-675-297 | |
| Heat block | Fisher | 11-718Q |
Références
- . . PyroMark PCR Handbook. , (2009).
- . . PyroMark Q24 User Manual. , (2012).
- Elahi, E., Ronaghi, M. Pyrosequencing: a tool for DNA sequencing analysis. Methods Mol. Bio. 255, 211-219 (2004).
- Fakhrai-Rad, H., Pourmand, N., Ronaghi, M. Pyrosequencing: and accurate detection platform for single nucleotide polymorphisms. Hum. Mutat. 19 (5), 479-485 (2002).
- Langaee, T., Ronaghi, M. Genetic variation analyses by pyrosequencing. Mutat. Res. 573 (1-2), 96-102 (2005).
- Liu, Z., Lozupone, C., Hamady, M., Bushman, F. D., Knight, R. Short pyrosequencing reads suffice for accurate microbial community analysis. Nucleic Acids Res. 35 (18), e120 (2007).
- Novais, R. C., Thorstenson, Y. R. The evolution of Pyrosequencing for microbiology: From genes to genomes. J. Microbiol. Methods. 86 (1), 1-7 (2011).
- J, Metagenomic pyrosequencing and microbial identification. Clin. Chem. 55 (5), 856-866 (2009).
- Quince, C., Lanzen, A., Curtis, T. P., Davenport, R. J., Hall, N., Head, I. M., Read, L. F., Sloan, W. T. Accurate determination of microbial diversity from 454 pyrosequencing data. Nat. Methods. (9), 639-6341 (2009).
- Ronaghi, M., Karamohamed, S., Pettersson, B., Uhlen, M., Nyren, P. Real-time DNA sequencing using detection of pyrophosphate release. Anal. Biochem. 242, 84-89 (1996).
- King, C., Scott-Horton, T. Pyrosequencing: A simple method for accurate genotyping. J. Vis. Exp. (11), e630 (2008).
- Rothberg, J. W., Heinz, , et al. An integrated semi-conductor device that enabling non-optical genome sequencing. Nature. 475, 348-352 (2011).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-Mode Waveguides for Single-Molecule Analysis at high concentrations. Science. 299, 682-686 (2003).
- Eid, J., Fehr, A. Real-Time DNA Sequencing from Single Polymerase Molecules. Science. 323, 133-138 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon