Method Article
Análisis de estabilidad de plásmidos con microfluídica de gotas de código abierto
En este artículo
Resumen
Se presenta un flujo de trabajo microfluídico accesible y de código abierto para el análisis paralelizado de la retención de plásmidos en bacterias. Al emplear la microscopía de fluorescencia para cuantificar la presencia de plásmidos en microcolonias unicelulares dentro de microgotas de gel, este método proporciona una alternativa precisa, accesible y escalable al recuento tradicional de placas.
Resumen
Los plásmidos desempeñan un papel vital en la biología sintética al permitir la introducción y expresión de genes extraños en varios organismos, lo que facilita la construcción de circuitos y vías biológicas dentro y entre las poblaciones celulares. Para muchas aplicaciones, es fundamental mantener plásmidos funcionales sin selección de antibióticos. Este estudio presenta un flujo de trabajo microfluídico basado en hardware abierto para analizar la retención de plásmidos mediante el cultivo de células individuales en microgotas de gel y la cuantificación de microcolonias mediante microscopía de fluorescencia. Este enfoque permite el análisis paralelo de numerosas gotas y microcolonias, lo que proporciona una mayor potencia estadística en comparación con el recuento tradicional de placas y permite la integración del ensayo en otros flujos de trabajo de microfluídica de gotas. Mediante el uso de plásmidos que expresan proteínas fluorescentes junto con una tinción de ADN fluorescente no específica, se pueden identificar y diferenciar colonias individuales en función de la pérdida de plásmidos o la expresión de marcadores fluorescentes. En particular, este flujo de trabajo avanzado, implementado con hardware de código abierto, ofrece un control preciso del flujo y una gestión de la temperatura tanto de la muestra como del chip microfluídico. Estas características mejoran la facilidad de uso, la solidez y la accesibilidad del flujo de trabajo. Si bien el estudio se centra en Escherichia coli como modelo experimental, el verdadero potencial del método radica en su versatilidad. Se puede adaptar para diversos estudios que requieran la cuantificación de señales de fluorescencia a partir de plásmidos o tinciones, así como para otras aplicaciones. La adopción de hardware de código abierto amplía el potencial para realizar bioanálisis de alto rendimiento utilizando tecnología accesible en diversos entornos de investigación.
Introducción
Los plásmidos son elementos genéticos esenciales en las células procariotas, contribuyendo significativamente a la evolución microbiana a través de la transferencia lateral de ADN y la rápida adaptación a los cambios ambientales 1,2. Estas moléculas de ADN extracromosómicas portan genes que proporcionan rasgos ventajosos, como resistencia a los antibióticos, funciones metabólicas y factores de virulencia, lo que las hace valiosas para la investigación en microbiología, biología sintética y estudios de evolución2. Sin embargo, el mantenimiento de plásmidos en poblaciones celulares es un desafío debido a la carga metabólica de replicación y segregación, lo que a menudo resulta en la pérdida de plásmidos sin presión de selección3. Además, la herencia estable requiere mecanismos como toxina-antitoxina y sistemas de partición, lo que añade complejidad al mantenimiento de los plásmidos. La evaluación de la estabilidad de los plásmidos en condiciones variables es crucial tanto para la investigación fundamental como para las aplicaciones prácticas que utilizan plásmidos como elemento primario de investigación 4,5. La mayoría de los métodos actuales para evaluar la estabilidad de los plásmidos tienen limitaciones significativas: los métodos basados en citometría de flujo proporcionan datos indirectos a nivel de población, requieren equipos costosos y carecen de visualización directa de las colonias6. Los métodos de transcriptómica y proteómica a granel son costosos, proporcionan solo respuestas celulares promedio y no pueden cuantificar directamente la retención de plásmidos en colonias individuales6. Los métodos tradicionales, como la dilución en serie y el passaging, son sencillos pero requieren mucho tiempo y carecen de precisión y representabilidad7. En general, inferir o proyectar cuantitativamente el número de colonias que conservan un plásmido funcional específico a lo largo del tiempo o las presiones selectivas sigue siendo un desafío.
Para abordar estos desafíos, se presenta un nuevo flujo de trabajo microfluídico que utiliza instrumentos de investigación de hardware abierto para cuantificar señales fluorescentes en múltiples colonias aisladas de bacterias, utilizando Escherichia coli como modelo. Este método permite un análisis preciso y de alto rendimiento de la retención de plásmidos en diversas condiciones o presiones selectivas. El análisis de resolución de una sola célula proporciona un método preciso para manipular colonias aisladas, lo que proporciona datos confidenciales sobre la cuantificación de plásmidos que pueden ayudar a evaluar las tasas de retención y pérdida4.
La microfluídica, en particular la microfluídica de gotas, se ha convertido en una poderosa herramienta para encapsular y manipular células individuales en entornos controlados8. Específicamente, las gotas de microgel pueden encapsular células individuales para un análisis preciso y de alto rendimiento sin la necesidad de mantener gotas suspendidas en aceite9, lo que permite un estudio controlado de la dinámica de plásmidos en un microambiente definido. Después de la encapsulación de suspensiones celulares directamente desde una punta de pipeta10, se pueden utilizar técnicas de fluorescencia para monitorear el crecimiento de microcolonias dentro de las gotas, lo que permite un análisis detallado de la retención y segregación de plásmidos bajo diferentes presiones de selección3.
Las ventajas de este método sobre las técnicas tradicionales de cultivo a granel incluyen una mayor precisión, una menor variabilidad y la capacidad de realizar análisis de alto rendimiento. La tecnología microfluídica de código abierto supera las limitaciones de los costosos sistemas propietarios, como los problemas de accesibilidad, adaptabilidad y mantenimiento, que a menudo obstaculizan el progreso de la investigación 11,12,13. Al demostrar cómo aplicar el flujo de trabajo experimental avanzado del análisis de retención de plásmidos en microgeles con instrumentación de código abierto, se proporciona un método accesible y confiable para la investigación en biología de plásmidos, aplicaciones de biología sintética y técnicas de análisis de gotas microfluídicas.
En resumen, este artículo presenta un método accesible para evaluar cuantitativamente la retención de plásmidos en E. coli con alto poder estadístico. Las capacidades de este método lo convierten en una herramienta valiosa para avanzar en la comprensión de la biología de plásmidos y mejorar las aplicaciones de la biología sintética.
Protocolo
La Figura 1proporciona una descripción general esquemática para evaluar la estabilidad de plásmidos en E. coli. Los detalles de los reactivos y el equipo utilizado se enumeran en la Tabla de Materiales. Los datos sin procesar y los scripts de visualización están disponibles en https://doi-org.remotexs.ntu.edu.sg/10.17605/OSF.IO/6YWJK.
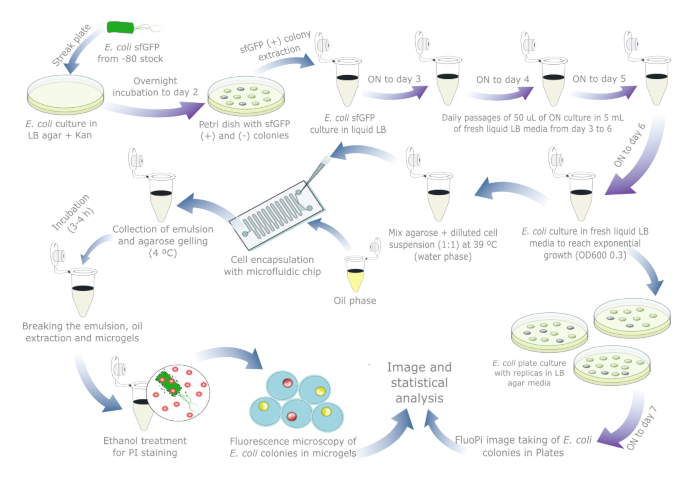
Figura 1: Protocolo día a día para evaluar la estabilidad de plásmidos en E. coli. Las flechas azules indican los pasos durante el día y las flechas moradas indican la incubación durante la noche. Cada incubación de líquido y agar se realizó a 37 °C junto con un tubo/placa de control negativo separado. Tenga en cuenta que la preparación y los pasajes del cultivo celular no son necesarios para muestras del mundo real en las que ya se puede haber producido una pérdida de plásmidos, por lo que el protocolo debe reducirse a uno o dos días si se incluye un cultivo de referencia en placa. Haga clic aquí para ver una versión más grande de esta figura.
1. Preparación de chips microfluídicos
NOTA: En este protocolo, se pueden utilizar diferentes diseños de chips comerciales o personalizados para la encapsulación de celdas que pueden generar gotas de agua en aceite de menos de 100 μm de diámetro en un régimen de goteo. Para este estudio, el chip se diseñó y fabricó (ver datos del artículo https://doi-org.remotexs.ntu.edu.sg/10.17605/OSF.IO/6YWJK) siguiendo el mismo método de diseño y fabricación que se informó en un informe publicado anteriormente14.
- Obtenga o prepare un chip microfluídico PDMS sobre vidrio utilizando un molde maestro diseñado para la generación de microgotas de gel.
- Inyecte una solución repelente al agua (fluoroalquilsilano) en el chip para hacer que los microcanales internos sean hidrofóbicos. Infunda la solución en las entradas y asegúrese de que todos los canales estén llenos de líquido. Deje reposar los canales llenos durante unos 30-60 s.
- Retire la solución del dispositivo expulsando aire hacia los microcanales internos. Use una jeringa de aire vacía para enjuagar el aire y una toallita absorbente sobre los otros puertos para evitar salpicaduras.
- Hornee el dispositivo tratado a 65 °C durante 15 minutos en una placa calefactora para evaporar el exceso de solución. Alternativamente, guarde el dispositivo en un refrigerador (4 °C) durante la noche.
NOTA: El chip microfluídico está listo para usar. El protocolo se puede pausar aquí.
2. Preparación de la muestra
- Recolección de células
NOTA: El cultivo celular en gotitas puede proporcionar datos relevantes sobre la dinámica de los plásmidos. Como modelo experimental de bacterias, se utiliza una cepa TOP10 de E. coli con el plásmido pCA_Odd1 (ver datos depositados https://doi-org.remotexs.ntu.edu.sg/10.17605/OSF.IO/6YWJK) que codifica una proteína fluorescente verde supercarpeta (sfGFP) y resistencia a la kanamicina15. Las bacterias, el plásmido y los medios de crecimiento pueden variar según el sistema experimental.- Prepare Luria Bertoni (LB)-Agar disolviendo 25 g de LB premezclado y 12 g de agar en 800 mL de agua destilada (dH2O). Además, prepare los medios LB líquidos disolviendo 25 g de LB premezclado en 800 mL dedH2O. Esterilize las soluciones en clave y deje que se enfríen a unos 60 °C. Muévase a un entorno estéril, como una campana de flujo, para los siguientes pasos.
NOTA: El polvo de LB premezclado contiene 10 g de triptona, 5 g de extracto de levadura y 10 g de NaCl. También se pueden utilizar medios de agar LB automezclados. - Añadir 50 μL de kanamicina (preparada a 100 mg/mL) a 50 mL de LB-Agar líquido (concentración final: 100 μg/mL). Mezcle la solución invirtiendo el tubo unas cuantas veces.
- Vierta alrededor de 15 mL de agar LB líquido por placa de Petri (90 mm x 15 mm). Prepare dos placas: una para el control negativo (monitoreo de contaminación) y otra para el cultivo del experimento. Deje que la solución de agar LB se enfríe y solidifique hasta que el color cambie de oscuro a claro en ambas placas.
- Use un asa estéril para esparcir la cepa de E. coli en la placa de cultivo utilizando la técnica de placa de rayas. Vuelva a colocar el caldo de glicerol E. coli a –80 °C inmediatamente después de su uso. Cierre ambas placas e incube durante la noche a 37 °C (día 1).
- El día 2, compruebe la placa de control negativa para detectar cualquier colonia como señal de contaminación (si es así, repita los pasos de 2.1.4.). Identifique colonias fluorescentes individuales en la placa de E. coli utilizando un transiluminador de luz azul para preparar un stock de cultivo líquido.
- En condiciones estériles, abra la placa y utilice un asa estéril o una punta de pipeta de 200 μL para recoger la colonia seleccionada y transferirla a un tubo de cultivo con 5 mL de medio LB líquido fresco y 5 μL de kanamicina (preparado a 100 mg/mL). Prepare un tubo de control negativo sin inoculación para el monitoreo de la contaminación. Incubar los tubos de cultivo a 37 °C durante la noche mientras se agitan a 220 rpm.
- Pase la muestra durante 3 días adicionales en medios sin antibióticos para simular las condiciones en las que puede ocurrir la pérdida de plásmidos. En los días 3, 4 y 5, transfiera 50 μL del cultivo nocturno a un nuevo tubo de cultivo que contenga 5 mL de LB líquido sin antibióticos. Incubar a 37 °C durante la noche con agitación a 220 rpm. Repita este paso hasta el día 6, obteniendo un cultivo final de 5 mL después de cuatro pasadas totales.
- Transfiera 50 μL del cultivo final durante la noche a un nuevo tubo de cultivo que contenga 2 mL de LB líquido. Permita que el cultivo alcance una densidad óptica (OD600) de 0,3 utilizando un espectrofotómetro (3-4 h aproximadamente).
NOTA: El cultivo fresco debe usarse antes de que aumente el OD600 para garantizar que las bacterias estén en la fase de crecimiento logarítmico. Si no se logra la concentración deseada, repita desde el paso 2.1.8.
- Prepare Luria Bertoni (LB)-Agar disolviendo 25 g de LB premezclado y 12 g de agar en 800 mL de agua destilada (dH2O). Además, prepare los medios LB líquidos disolviendo 25 g de LB premezclado en 800 mL dedH2O. Esterilize las soluciones en clave y deje que se enfríen a unos 60 °C. Muévase a un entorno estéril, como una campana de flujo, para los siguientes pasos.
- Mezcla de célula y agarosa para encapsulación
NOTA: El control de concentración es esencial para garantizar la encapsulación de gotas de una sola célula. La concentración de células requerida se puede calcular para una tasa de encapsulación objetivo y un volumen de gota específico, como se muestra en el siguiente ejemplo:
Volumen de gota (V drop): Si se generan gotas de 50 μm

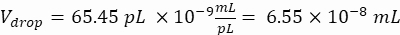
Células deseadas por gota (Cpd): Una célula por cada cinco gotas en promedio (0,2 células/gota)

Factor de dilución: La concentración celular inicial se obtiene en el paso 2.1.8

Aquí, se generan gotas de 50 μm o 100 μm (sin división de gotas) (65-520 pL) donde se encapsula aproximadamente una célula por cada cinco gotas, o 1,6 células por gota sin división. Para E. coli, utilice el factor de conversión de 1 unidad OD600 ≈ 7,8 x 108 células/mL16. Multiplique el valor de OD600 del paso 2.1.8 por el factor de conversión para obtener la concentración inicial (células/mL) del cultivo.- Volver a suspender E . coli del cultivo preparado en LB líquido sin kanamicina a una concentración de 6,2e+6 células/ml (concentración objetivo para gotas de 50 μm de diámetro). Mantenga la suspensión de bacterias a temperatura ambiente hasta que se mezcle con la agarosa.
NOTA: Los antibióticos suprimirán la formación de microcolonias negativas, por lo que es esencial excluirlas de los medios para los experimentos de pérdida de plásmidos. - Prepare la agarosa a temperatura de gelificación ultrabaja calentándola a 90 °C en LB líquido a una concentración del 2% (p/v). Agite la mezcla durante 10 minutos en una coctelera con temperatura controlada.
- Reduzca la temperatura del termoagitador a 39 °C para enfriar la solución de agarosa. Paralelamente, coloque el tubo de suspensión de bacterias en el agitador térmico durante 4 minutos para calentarlo a 39 °C.
- Mezclar las bacterias y las suspensiones de agarosa en una proporción 1:1 para obtener una concentración de agarosa del 1% (p/v) con suspensión celular a 3,1e+6 células/mL. Prepare la solución de control negativo (monitoreo de contaminación) con la misma concentración de agarosa usando LB líquido en lugar de la suspensión de bacterias.
NOTA: La suspensión de células de agarosa debe usarse rápidamente para evitar cambios de concentración debido al crecimiento de bacterias. Mantenga el agitador térmico a 39 °C para mantener el líquido de agarosa hasta que se cargue en el calentador de punta para la generación de gotas.
- Volver a suspender E . coli del cultivo preparado en LB líquido sin kanamicina a una concentración de 6,2e+6 células/ml (concentración objetivo para gotas de 50 μm de diámetro). Mantenga la suspensión de bacterias a temperatura ambiente hasta que se mezcle con la agarosa.
3. Cultivo de una sola colonia de alto rendimiento
- Configuración experimental
- Construya u obtenga la plataforma de flujo de código abierto completa (consulte https://doi-org.remotexs.ntu.edu.sg/10.17605/OSF.IO/6YWJK), incluidos los controladores de presión de gas y los sensores de flujo. Como alternativa, cree la etapa de microscopía mejorada con luz estroboscópica de hardware de código abierto más sencilla (consulte las instrucciones de compilación https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ y el repositorio de proyectos https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage).
NOTA: También se puede utilizar una configuración de microfluídica tradicional con un microscopio comercial, una cámara de alta velocidad y capacidades de calentamiento de muestras. - Integre el sistema controlador de presión y caudal de código abierto basado en Raspberry Pi como se documenta en el repositorio designado (https://github.com/wenzel-lab/modular-microfluidics-workstation-controller).
NOTA: El control del módulo se ilustra y se realiza una copia de seguridad en el repositorio de datos (https://doi-org.remotexs.ntu.edu.sg/10.17605/OSF.IO/6YWJK. Como alternativa, se pueden emplear controladores de presión tradicionales o bombas de jeringa de alto par. - Incluya un portaobjetos de vidrio y calentadores de punta de pipeta (https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring) en la configuración para controlar la temperatura de la muestra de celda de agarosa a medida que ingresa al chip.
NOTA: Estos calentadores, ilustrados en el repositorio de datos (https://github.com/wenzel-lab/flow-microscopy-platform y https://doi-org.remotexs.ntu.edu.sg/10.17605/OSF.IO/6YWJK), son una característica importante del sistema de controlador de código abierto que permite el trabajo con agarosa y pueden no estar disponibles en otros sistemas comerciales.
- Construya u obtenga la plataforma de flujo de código abierto completa (consulte https://doi-org.remotexs.ntu.edu.sg/10.17605/OSF.IO/6YWJK), incluidos los controladores de presión de gas y los sensores de flujo. Como alternativa, cree la etapa de microscopía mejorada con luz estroboscópica de hardware de código abierto más sencilla (consulte las instrucciones de compilación https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ y el repositorio de proyectos https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage).
- Encapsulación de una sola célula
- Coloque el chip microfluídico en la platina de microscopía estroboscópica, asegurándose de que la unión de generación de gotas (intersección de las fases acuosa y oleosa) sea visible.
- Ajuste el calentador de punta de pipeta y el calentador de portaobjetos de vidrio a 40 °C mediante la interfaz del software de control.
- Utilice una jeringa con tubo y tapón de PDMS para cargar la mezcla de agarosa al 1 % con suspensión celular en una punta de pipeta de 200 μL. Inserte la punta en el calentador de puntas y colóquela en la entrada de la fase acuosa en el chip microfluídico. Cambie el sello PDMS de la punta por uno conectado a la tubería del sistema de control de flujo e inicie la infusión de la suspensión celular.
- Inserte el extremo del tubo de salida en un tubo de desagüe y establezca caudales o presiones de dos fases en la interfaz de usuario para suministrar fluido lentamente al canal microfluídico. Utilice 200 μL/h (180 mbar) para la fase acuosa y 1700 μL/h (320 mbar) para la fase oleosa. Espere 1 minuto para la estabilización de la generación de gotas.
NOTA: Los valores de presión dependen de los tamaños de canal del diseño del chip, y es posible que los valores de flujo deban ajustarse para diferentes diseños de uniones de generación de gotas. - Una vez que la generación de gotas sea estable, transfiera los desechos y el tubo de recolección al tubo de recolección. Continuar recolectando gotas hasta que el depósito de muestra esté vacío. Repita los pasos 3.2.3 a 3.2.5 para encapsular la solución de control negativo del paso 2.2.4.
NOTA: La recolección de muestras debe completarse dentro de los 15 minutos. - Almacene los tubos de recolección en hielo durante la generación de gotas o colóquelos a 4 °C después del experimento durante 1 h para permitir que la agarosa se gelifique dentro de las gotas.
NOTA: El chip microfluídico se puede reutilizar si los microcanales permanecen desatascados y se carga la misma suspensión. Deseche la punta de pipeta después de completar la generación de la emulsión (pasos 3.2.3–3.2.5).
- Crecimiento de colonias y liberación de la emulsión
- Transfiera las microgotas de gel que contienen bacterias y el control negativo en gotas a una cámara de incubación ajustada a 37 °C.
- Incube las microgotas durante al menos 4 h, o toda la noche, para permitir un crecimiento suficiente de la colonia. Confirme que el control negativo no muestra signos de contaminación a través de la microscopía de campo claro.
- Para liberar las colonias de la emulsión, retire la mayor cantidad de aceite posible de debajo de la emulsión de microgotas de gel con una pipeta o una jeringa con una aguja (en este caso, se utilizó una aguja de tamaño 21 G).
- Transfiera 50 μL de las microgotas de gel a un nuevo microtubo y guárdelo a 4 °C para su posterior análisis de gotas. A la emulsión restante, agregue una mezcla 1:1 de aceite fluorado con 1H, 1H, 2H, 2H-perfluoro-1-octanol (PFO) en un volumen igual a la emulsión.
- Agregue aproximadamente 200 μL de tampón salino tamponado con fosfato (PBS), o 0,9% p/v de tampón NaCl, encima de la emulsión. Agite la mezcla y gírela brevemente en una centrífuga de velocidad fija.
- Retire con cuidado la fase oleosa de la parte inferior de la interfaz líquida y deseche 100 μL de PBS de la parte superior. Repita los pasos 3.3.4–3.3.5 para obtener microgeles lavados en tampón PBS con un residuo de aceite mínimo o nulo.
NOTA: Los microgeles se depositan en la interfaz líquida; Evite eliminarlos junto con la fase oleosa.
4. Análisis de una sola colonia
- Tinción celular
NOTA: Muchas combinaciones de tinte diferentes funcionan para este protocolo. En esencia, se debe elegir una tinción de ADN o de pared celular que tenga un color distinto al de la proteína fluorescente codificada por el plásmido y que pueda analizarse utilizando las combinaciones de filtros disponibles en el microscopio de fluorescencia. Aquí, el ADN de las células se tiñó con yoduro de propidio (PI) para distinguir su fluorescencia de la proteína fluorescente verde codificada en los plásmidos, pero se pueden usar muchas otras tinciones de ADN.
PRECAUCIÓN: El PI es un carcinógeno potencial y debe manipularse con el equipo de protección personal adecuado. Deseche el tinte de manera segura y de acuerdo con las regulaciones locales.- Centrifugar los microgeles lavados a aproximadamente 80 x g durante 5 min a temperatura ambiente. Deseche el sobrenadante con una pipeta.
- Transfiera 50 uL de los microgeles a un nuevo microtubo para que sirva como control negativo para el tratamiento con etanol.
NOTA: La tinción PI solo ingresa a las células con membranas comprometidas, como las afectadas por el tratamiento con etanol. - Agregue un volumen igual de etanol al 70% a los microgeles restantes y mezcle brevemente con un vórtice. Incubar a temperatura ambiente durante 15 min para permeabilizar las membranas bacterianas para la tinción de PI. Repita el paso 4.1.1.
- Agregue un volumen igual de 0.9% p/v de NaCl a los microgeles y brevemente en vórtice. Repita el paso 4.1.1.
- Añadir 2 μL de PI (1 mg/mL) a ambas muestras de microtubos. Mezclar bien e incubar en la oscuridad a temperatura ambiente durante 15 min.
NOTA: Si el testigo negativo presenta alguna señal fluorescente roja a través de microscopía de fluorescencia, la integridad de las colonias puede haberse visto comprometida durante su exposición a otras soluciones en los pasos anteriores.
- Microscopia
NOTA: Las gotas y los microgeles se visualizan con un microscopio de epifluorescencia invertida para obtener la distribución del tamaño de las gotas y la fluorescencia de las colonias bacterianas en los microgeles (Figura 2). En este caso, se utiliza una plataforma de microscopía invertida (https://github.com/wenzel-lab/SQUID-bioimaging-platform)17 de código abierto con un objetivo de 10x 0,3NA, una matriz de LED blancos para la iluminación de campo claro y un LED de 470 nm para la excitación. Los microscopios de epifluorescencia comerciales se pueden utilizar para obtener imágenes de gotas y microgeles. La calibración es necesaria porque la iluminación y los filtros varían según el modelo, la marca y las proteínas fluorescentes utilizadas.- Transfiera 2 μL de microgotas de gel a un portaobjetos de la cámara de imágenes y agregue 5 μL de aceite fluorado para ayudar a formar una monocapa de gotas para obtener imágenes óptimas.
NOTA: Las cámaras de recuento de células o las cámaras microfluídicas simples pueden ayudar a esparcir la emulsión finamente y ralentizar el proceso de secado. - En el microscopio, active la iluminación blanca de matriz LED desde la parte superior para obtener imágenes de campo claro. Monte el portaobjetos preparado, concéntrese en la muestra y localice una monocapa de gotas. Captura una imagen de campo claro.
- Sin mover la muestra, capture una imagen de fluorescencia de las colonias cambiando al LED de 470 nm para la excitación. Ajuste la rueda de filtros para alinearla con el filtro de longitud de onda verde para imágenes sfGFP. Escanee todas las áreas que contengan monocapas de gotas y repita los pasos 4.2.2–4.2.3 para garantizar la solidez estadística en el análisis de gotas.
- Transfiera 2 μL de los microgeles teñidos a un chip de cámara de imagen y 5 μL de NaCl al 0,9% p/v para ayudar a formar una monocapa de microgeles. Selle la entrada y la salida del chip para evitar la evaporación durante la toma de imágenes.
NOTA: La evaporación puede afectar a la colocalización de las etiquetas fluorescentes. Las cámaras microfluídicas se recomiendan para la obtención de imágenes prolongadas. - Repita los pasos 4.2.2–4.2.3. Ajuste el filtro de intervalo de longitud de onda roja para imágenes PI y capture las imágenes respectivas.
- Después de completar la obtención de imágenes en un lugar, encuentre la siguiente área adecuada en el portaobjetos con una monocapa de microgeles y repita el proceso de obtención de imágenes para garantizar la solidez estadística y el análisis completo de la muestra.
- Transfiera 2 μL de microgotas de gel a un portaobjetos de la cámara de imágenes y agregue 5 μL de aceite fluorado para ayudar a formar una monocapa de gotas para obtener imágenes óptimas.

Figura 2: Imágenes de microscopía y su análisis. Imágenes de fluorescencia y análisis de colonias en microgeles. (De la A a la C) Canales de imagen adquiridos mediante microscopía de campo claro y fluorescencia con el microscopio invertido. La imagen compuesta (D) muestra la presencia de una colonia negativa (solo fluorescencia roja) dentro de los microgeles. (E-H) Resultados del flujo de trabajo de análisis de imágenes. Al generar ROI, las colonias se pueden identificar en los canales rojo y verde, y las señales se pueden cuantificar para definir la presencia de colonias negativas. Barras de escala: 50 μm. Haga clic aquí para ver una versión más grande de esta figura.
- Análisis de imágenes
NOTA: Para analizar las señales de fluorescencia de las colonias encapsuladas e identificar eventos raros, las imágenes de campo claro y de fluorescencia se pueden procesar con Fiji/ImageJ (Figura 3). Estos pasos se pueden implementar en un script de macros y los valores de los parámetros pueden variar en función de la configuración óptica.- Abra las imágenes de los canales verde y rojo. Defina una región de interés rectangular (ROI) a partir de las coordenadas (500, 500) con un ancho y un alto de 2000 píxeles cada uno.
NOTA: Estos parámetros se aplican a imágenes de 3000 x 3000 píxeles y el ROI define el área con mejor iluminación. - Recorte la imagen al ROI rectangular definido. Reste un valor constante de 10 de la intensidad de cada píxel y aplique un filtro de suavizado a la imagen para reducir el ruido de fondo y hacer que los objetos de interés sean más distintos.
NOTA: Solo se utilizará esta región para un análisis más detallado. - Duplique la imagen del canal rojo y conviértala en una máscara binaria para identificar los eventos raros relacionados con el plásmido. Establezca los valores de umbral entre 10 y 255. Los píxeles dentro de este rango se considerarán en primer plano (objetos de interés), mientras que otros se tratarán como fondo.
- Realizar operaciones morfológicas para cerrar pequeños huecos y rellenar los huecos dentro de los objetos de interés. Aplique el algoritmo de cuenca hidrográfica para separar objetos superpuestos dentro de la máscara binaria.
- Analice las partículas de la máscara binaria. Considere solo partículas con un tamaño superior a 30 píxeles y una circularidad entre 0,50 y 1,00. Los resultados se resumen y se agregan a la tabla de resultados. Guarde el conjunto de ROI detectados por el análisis de partículas para su posterior visualización.
- Muestra los ROI guardados en las imágenes de los canales verde y rojo. Mida la intensidad u otras propiedades de los ROI dentro de estas imágenes. Registre las mediciones en la tabla de resultados y guarde los resultados en archivos CSV separados para su posterior análisis estadístico.
- Abra las imágenes de los canales verde y rojo. Defina una región de interés rectangular (ROI) a partir de las coordenadas (500, 500) con un ancho y un alto de 2000 píxeles cada uno.
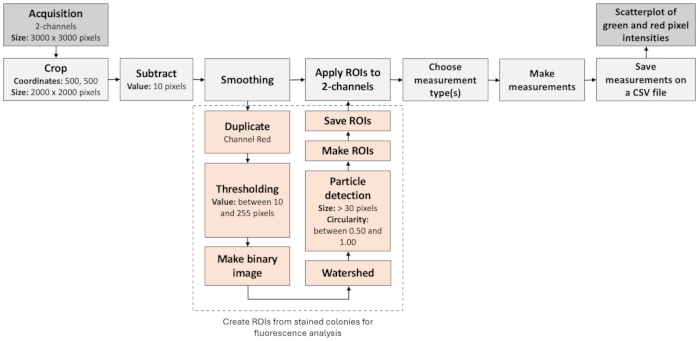
Figura 3: Flujo de trabajo de análisis de imágenes para identificar colonias negativas. La figura ilustra un flujo de trabajo paso a paso para procesar y evaluar automáticamente las imágenes de fluorescencia. El flujo de trabajo se basa en la colocalización de etiquetas de fluorescencia, así como en el análisis de partículas. Haga clic aquí para ver una versión más grande de esta figura.
5. Ensayo de comparación de placas de agar
NOTA: Para comparar el método de las gotas con un ensayo tradicional en placa, se obtuvo la cuantificación de colonias fluorescentes de la misma cepa de E. coli en el paso 2.1.8. se realizó utilizando placas de Petri. Esto sirvió como un método de control analógico para medir la estabilidad del plásmido sfGFP. Consulte también la ilustración del método en la Figura 1.
- Prepare LB-agar (sin antibiótico) como se describe en el paso 2.1.1 de la sección Recolección de células. Enfriar el agar por debajo de 60 °C y homogeneizar.
- En un ambiente estéril, vierta 15 ml del medio de agar en cada placa de Petri. Deje que los platos se solidifiquen con las tapas parcialmente abiertas hasta que estén listos para usar.
- Inocular tres placas con 10 μL cada una de un cultivo de E. coli en medio LB con un OD600 de 0,007 ± 0,002. Extienda uniformemente con un esparcidor en forma de L. Incubar las placas a 37 °C, cerradas e invertidas. Prepare una placa de control sin bacterias en las mismas condiciones.
- Después de 24 h, capture imágenes de fluorescencia de las placas utilizando el FluoPi, un sistema de imágenes de fluorescencia (https://github.com/RudgeLab/FluoPi) de código abierto15.
NOTA: La FluoPi consta de una cámara Raspberry Pi con una luz de excitación azul a una longitud de onda centrada en 470 nm y filtros acrílicos de excitación y emisión. - Cuente manualmente las colonias fluorescentes y no fluorescentes en cada placa utilizando las imágenes capturadas.
Resultados
Validación de encapsulación celular y formación de microcolonias
La encapsulación celular se puede confirmar visualmente mediante la realización de microscopía de campo claro en las microgotas de gel antes de romper la emulsión y lavar los microgeles. En la Figura 4 se muestra un resultado representativo de la emulsión en este paso.
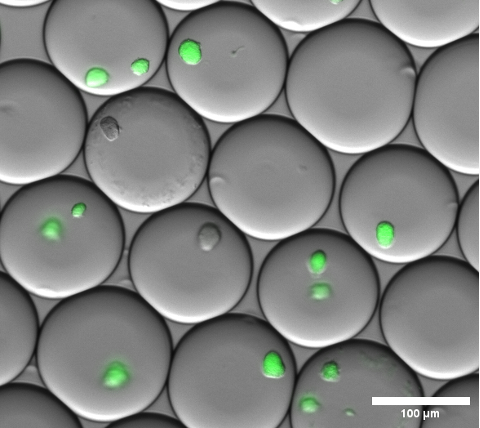
Figura 4: Sección de una imagen de superposición de microscopía de fluorescencia. Después de la incubación durante la noche, microcolonias representativas de sfGFP expresan colonias de E. coli dentro de microgotas de gel. Se utilizó un objetivo de microscopio con aumento de 10x y 0,30 NA. Barra de escala: 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
Resultados del análisis de imágenes
Una vez que los microgeles se tiñen y se obtienen el campo claro, así como los canales de fluorescencia adquiridos en varias posiciones, se pueden visualizar las colonias identificadas como negativas en las imágenes originales (ver Figura 5A). Los datos extraídos de todas las imágenes de un experimento se pueden trazar para mostrar la relación de fluorescencia de varias colonias, destacando aquellas que perdieron la fluorescencia codificada por plásmidos (ver Figura 5B). Los resultados indican que 100 colonias perdieron su funcionalidad plásmida o plásmido de un total de 2785 microcolonias analizadas, lo que corresponde al 3,6%.
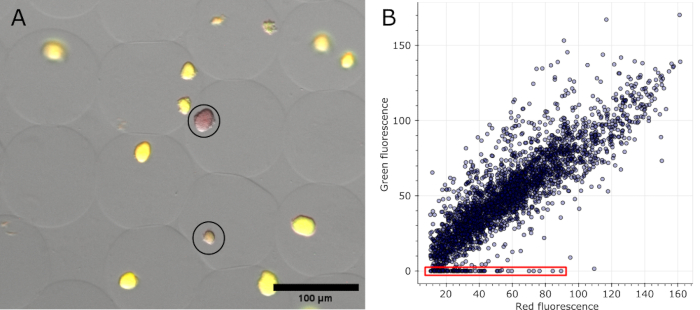
Figura 5: Cuantificación de microcolonias negativas. (A) Sección de una imagen de superposición de microscopía de fluorescencia. Después de la eliminación del aceite y la tinción, las colonias representativas en los microgeles que expresaban sfGFP y dos colonias negativas mostraron la fluorescencia roja de la tinción de ADN (con un círculo negro). Barra de escala: 100 μm. (B) Diagrama de dispersión de los valores de fluorescencia de microcolonias individuales extraídos de 16 imágenes de microscopía multicanal. Las colonias sin fluorescencia verde se contaron como negativas, como se indica en rojo en el gráfico. Haga clic aquí para ver una versión más grande de esta figura.
Cuantificación en placa de agar
Las imágenes de las placas triplicadas se muestran en la Figura 6, con colonias no fluorescentes indicadas por flechas blancas. La primera placa (Figura 6A) mostró un total de 213 colonias, de las cuales 1 no era fluorescente. La segunda placa (Figura 6B) tenía un total de 49 colonias, sin colonias no fluorescentes. La tercera placa (Figura 6C) mostró un total de 252 colonias, 6 de las cuales no eran fluorescentes. Estos resultados corresponden a una tasa media de pérdida de plásmidos de colonias del 2,3%, con una gran desviación estándar de 3,2.

Figura 6: Identificación de colonias negativas en placas. (De la A a la C) Colonias de E. coli fluorescentes y no fluorescentes en placas de agar LB (diámetro: 90 mm, altura: 15 mm). El inóculo, derivado de E. coli con sfGFP de -80 °C, se estregó el día 1, se cultivó con antibiótico el día 2 y se diluyó 1:100 diariamente desde el día 3 hasta el día 6 para permitir la pérdida de plásmidos. Las colonias se incubaron a 37 °C durante 24 h y se tomaron imágenes en una cámara FluoPi. Las colonias no fluorescentes se mejoraron con GIMP y se indicaron con flechas blancas. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Se ha demostrado que un método basado en microgotas en gel identifica y cuantifica eficazmente colonias con y sin expresión genética codificada por plásmidos de proteínas fluorescentes, como sfGFP. Las colonias que no expresan suficientemente el producto plásmido se identifican utilizando una tinción de ADN fluorescente (en este caso, yoduro de propidio) que tiñe todas las colonias y presenta una longitud de onda de emisión diferente. Esta integración de la microfluídica de gotas, la gelificación y la microscopía de fluorescencia, utilizando tecnología de código abierto, permite ejecutar un flujo de trabajo avanzado en muchos entornos de investigación11,13. La exitosa generación de microgotas de gel permite flujos de trabajo avanzados de biología molecular de una sola célula, incluida la lisis celular, la amplificación de un solo genoma, las pantallas de interacción celular metabólica, el intercambio de medios, etc. Estas ventajas se utilizan en este protocolo para cultivar, teñir y analizar microcolonias de una manera más escalable que en los ensayos tradicionales basados en placas.
Pasos críticos
El proceso de encapsulación es una parte crítica y delicada del protocolo. Se requiere un control preciso de las concentraciones de ingredientes, los caudales y las presiones para generar microgeles uniformes dentro de un rango de tamaño específico y controlar el número promedio de células por gota. Además, el mantenimiento de la concentración y la temperatura de la mezcla de células y agarosa evita la formación de grumos o la gelificación prematura. El control de temperatura de la suspensión líquida de celdas de agarosa en una punta de pipeta es una implementación particularmente ventajosa de nuestra estación de trabajo de microfluídica de hardware de código abierto que proporciona una generación de microgel mucho más fácil y robusta en comparación con los esfuerzos para controlar la temperatura de las bombas de jeringa y los tubos. Dado que las células se mezclan con el medio de crecimiento de agarosa antes de la encapsulación y el cultivo, los microgeles de agarosa deben generarse rápidamente para evitar cambios importantes en la concentración celular. Para ello, se optimizó un diseño de chip microfluídico de división de gotas inspirado en Abate et al.18.
Modificaciones y solución de problemas
Fueron necesarias varias calibraciones y modificaciones para refinar el protocolo original. La encapsulación de la agarosa es mucho más desafiante que las gotas normales de agua en aceite, lo que requiere el diseño de un sistema para mantener la agarosa en estado líquido y garantizar que el flujo de la fase acuosa alcance un rango de tamaño de partícula homogéneo. Los cambios en la viscosidad de la agarosa debido a la gelificación afectan el caudal, lo que lleva a tamaños de partícula más grandes. La microscopía requiere una cuidadosa selección de filtros y fuentes de luz para garantizar señales de excitación y emisión que no se superpongan para una diferenciación clara. Inicialmente, se eligió DAPI para teñir bacterias, pero su señal de emisión se superpuso con sfGFP, lo que provocó que sfGFP se detectara en el canal de detección azul. Cambiamos a PI porque su emisión está bien separada de sfGFP en longitudes de onda largas (luz roja).
Si bien la pérdida de plásmidos se cuantificó utilizando el método propuesto, el plásmido sfGFP utilizado fue inesperadamente estable, mostrando apenas casos de pérdida de plásmidos en la primera generación de células cultivadas sin antibióticos, incluso en condiciones de estrés como medios de pH 9 e incubación a 40 °C. Esta observación es consistente con los hallazgos de otros grupos de investigación 1,19. La estabilidad del plásmido limitó la demostración de las capacidades de cuantificación completas del método para las generaciones iniciales de cultivos celulares, pero demostró que el método es lo suficientemente sensible como para detectar incluso pequeñas diferencias en la retención de plásmidos. La observación de la estabilidad de plásmidos altos en generaciones tempranas tiene una implicación importante para los cribados microfluídicos de gotas que utilizan un ensayo de selección negativa, como la inhibición de bacterias objetivo. Esto significa que la pérdida de plásmidos de los objetivos de selección es una fuente baja de resultados de selección falsos positivos. Dado que las cribas microfluídicas de gotas suelen superar a otras cribas de alto rendimiento, como los flujos de trabajo de los robots de pipeteo, en órdenes de magnitud de rendimiento, es necesario evaluar y tener en cuenta estos eventos poco frecuentes.
Limitaciones
A pesar de sus ventajas, existen limitaciones en el método presentado. La fabricación de dispositivos microfluídicos requiere experiencia y una atención meticulosa a los detalles, así como un estricto control experimental de los caudales para garantizar la eficiencia de la encapsulación determinista. Estos aspectos pueden requerir optimización para diferentes configuraciones experimentales. Si bien este método se basa en la microscopía de fluorescencia para la detección de señales, lo que requiere acceso a equipos de imagen adecuados, este equipo se puede fabricar utilizando hardware de código abierto, lo que lo hace más accesible. Además, los microgeles se pueden procesar en citometría de flujo comercial con boquillas grandes, lo que mejora aún más la accesibilidad y el rendimiento experimental. Los clasificadores de gotas también se pueden utilizar para este análisis citométrico.
Además, si bien el método está diseñado para detectar señales fluorescentes de plásmidos, tinciones u otros marcadores, se limita a las células que se pueden marcar con fluorescencia, lo que puede no aplicarse a todas las cepas bacterianas o condiciones experimentales. Sin embargo, el método se puede adaptar para incorporar otros tipos de microscopías, como la microscopía de contraste de fase o la microscopía de campo claro, lo que permite aplicaciones de fenotipado más allá de la fluorescencia. Además, se puede combinar con técnicas espectroscópicas como FTIR o espectroscopía Raman, ampliando sus capacidades para analizar composiciones químicas e información estructural de las células encapsuladas. Estas adaptaciones amplían el rango de su aplicabilidad, convirtiéndolo en una herramienta versátil para diversos entornos de investigación.
Importancia y aplicaciones
Los ensayos tradicionales para la pérdida de plásmidos19 no permiten una buena cuantificación de la proporción de células que perdieron su expresión, información que puede ser muy importante en el diseño de métodos experimentales y diversas aplicaciones biológicas. Por lo general, los tipos de colonias se enumeran en ensayos en placa de agar, donde se pueden obtener colonias aisladas bien definidas, como se muestra en la Figura 4. Sin embargo, es difícil identificar con confianza las colonias superpuestas; En nuestras manos, no siempre obtenemos una densidad de colonias óptima, y se necesitan muchas placas para obtener buenas estadísticas de eventos de pérdida de plásmidos de baja frecuencia. El método propuesto ofrece un enfoque más robusto para cuantificar con precisión las señales fluorescentes procedentes de colonias aisladas con un mayor número de colonias que los métodos análogos de placa de agar porque, en las microgotas, las colonias se desarrollan por separado, son más pequeñas y son fáciles de cargar en las cámaras de imagen, lo que permite la cuantificación basada en microscopía o citometría de flujo de grandes números de colonias. Esto puede mejorar significativamente la representación estadística del método y permitir la integración en otros flujos de trabajo de gel-microgotas.
El uso de hardware de código abierto 11,20 permite a los investigadores personalizar el diseño de la estación de trabajo microfluídica y ajustar con precisión la velocidad de flujo; Por lo tanto, el tamaño de partícula admite varios tipos de células y condiciones experimentales. Esta flexibilidad se extiende a la posible incorporación de otros tipos de microscopía, como el contraste de fase o la espectroscopia, ampliando la aplicabilidad del método. La capacidad del método para evaluar la estabilidad del plásmido en diversas condiciones es crucial para aplicaciones que requieren retención de plásmidos sin selección de antibióticos, en condiciones particulares de estrés o en varias generaciones de cultivo. La versatilidad y adaptabilidad del método presentado lo hacen valioso para diversas aplicaciones de investigación en campos como la biología sintética, el monitoreo ambiental y el diagnóstico clínico2.
Divulgaciones
Los autores declaran que ningún interés financiero o relación personal contrapuestos podría haber influido en el trabajo reportado en este artículo.
Agradecimientos
Este trabajo forma parte de los proyectos financiados a T.W. por ANID FONDECYT 1241621 Regular y el proyecto de la Iniciativa Chang Zuckerberg 'Latin American Hub for Bioimaging Through Open Hardware'. T.W. también está agradecido por la financiación de CIFAR, como Azrieli Global Scholar en el programa CIFAR MacMillan Multiscale Human.
Materiales
| Name | Company | Catalog Number | Comments |
| 1H,1H,2H,2H-Perfluoro-1-octanol | Sigma-Aldrich | 370533-25G | For breaking emulsion |
| 70% ethanol | For cell permeabilization | ||
| Agar-Agar | Winkler | 9002-18-0 | |
| Biopsy Punch | 0.75 mm and 1.8 mm | ||
| Blue LED transilluminator | IO Rodeo | ||
| Culture tube | 15 mL | ||
| Desiccator | With vacuum pump | ||
| Disposable cup | For mixing PDMS | ||
| Disposable fork | For mixing PDMS | ||
| E. coli TOP10 strain | |||
| FluoPi microscope | https://github.com/wenzel-lab/FluoPi | Green fluorescence imaging system for analyzing plates | |
| Fluorinated Oil | 3M | Novec 7500 | |
| Glass slide heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of microfluidic chip | |
| Glass Slides | |||
| Hotplate | Mechanic | For evaporating Aquapel | |
| Image analysis software | Fiji/ImageJ | 2.14.0/1.54f | |
| Incubator | Mundo Lab | MLAB Scientific / For incubation of plates and microgels | |
| Isopropanol | For cleaning glass slides | ||
| Kanamycin | 100 ug/mL concentration | ||
| L-shaped spreader | For spreading bacteria on agar plates | ||
| Master mold | Chip design on silicone or glass wafer | ||
| Microtubes | 2 mL | ||
| NaCl solution | Sodium chloride 0.9% w/v | ||
| Open-source hardware strobe-enhanced microscopy stage | https://github.com/wenzel-lab/flow-microscopy-platform | For bright-field microscopy | |
| Petri dish | Citotest | 2303-1090 | 90 x 15 mm |
| Pipette tip heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of pipette tip | |
| Plasma Cleaner | Diener Electronic | 117056 | For bonding PDMS with a glass slide |
| Plasmid pCA_Odd1 | Encodes sfGFP and kanamycin resistance | ||
| Polytetrafluoroethylene (PTFE) tubing | Adtech Polymer Engineering Ltd | ||
| Pre-mixed Luria Bertoni medium | US Biological Life Science | L1520 | |
| Propidium iodide (PI) | For staining | ||
| Raspberry Pi-based pressure and flow controller system | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller | For controlling pressure and flow rates | |
| Silicone elastomer base | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Silicone elastomer curing agent | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Spectrophotometer | For measuring absorbance | ||
| SQUID microscope | https://github.com/wenzel-lab/SQUID-bioimaging-platform | Multi-fluorescence imaging system for analyzing stained cells | |
| Sterile loop | For picking a colony and streaking plating | ||
| Surfactant | Sphere Fluidics | Pico-Surf | |
| Syringes | NIPRO | With filters and tubing | |
| Temperature-controlled shaker | Mundo Lab | DLAB HCM100-Pro | |
| Tweezer | |||
| Ultra-low gelling temperature agarose | Sigma-Aldrich | A2576-5G | For generating hydrogel beads |
| Water repelent solution (fluoroalkyl silane) | Aquapel | For treating microchannels of PDMS device |
Referencias
- Wein, T., Hülter, N. F., Mizrahi, I., Dagan, T. Emergence of plasmid stability under non-selective conditions maintains antibiotic resistance. Nat Commun. 10 (1), 2595 (2019).
- Rodríguez-Beltrán, J., DelaFuente, J., León-Sampedro, R., MacLean, R. C., Millán, &. #. 1. 9. 3. ;. S. Beyond horizontal gene transfer: The role of plasmids in bacterial evolution. Nat Rev Microbiol. 19 (6), 347 (2021).
- Wein, T., Dagan, T. Plasmid evolution. Curr Biol. 30 (19), R1158-R1163 (2020).
- Chen, S., Larsson, M., Robinson, R. C., Chen, S. L. Direct and convenient measurement of plasmid stability in lab and clinical isolates of E. coli. Sci Rep. 7 (1), 4788 (2017).
- Rouches, M. V., Xu, Y., Cortes, L. B. G., Lambert, G. A plasmid system with tunable copy number. Nat Commun. 13 (1), 3908 (2022).
- Silva, F., Queiroz, J. A., Domingues, F. C. Evaluating metabolic stress and plasmid stability in plasmid DNA production by Escherichia coli. Biotechnol Adv. 30 (3), 691-708 (2012).
- Wang, R., et al. Construction of novel pJRD215-derived plasmids using chloramphenicol acetyltransferase (cat) gene as a selection marker for Acidithiobacillus caldus. PLoS ONE. 12 (8), e0183307 (2017).
- Moragues, T., et al. Droplet-based microfluidics. Nat Rev Methods Primers. 3 (1), 32 (2023).
- Vitalis, C., Wenzel, T. Leveraging interactions in microfluidic droplets for enhanced biotechnology screens. Current Opinion in Biotechnology. 82, 102966 (2023).
- Sinha, N., Subedi, N., Wimmers, F., Soennichsen, M., Tel, J. A pipette-tip based method for seeding cells to droplet microfluidic platforms. J Vis Exp. (144), e57848 (2019).
- Wenzel, T. Open hardware: From DIY trend to global transformation in access to laboratory equipment. PLOS Biol. 21 (1), e3001931 (2023).
- Murillo, L. F. R., Wenzel, T. Welcome to the journal of open hardware. J Open Hardware. 1 (1), (2017).
- Shin, J. H., Choi, S. Open-source and do-it-yourself microfluidics. Sens Actuators B Chem. 347, 130624 (2021).
- Pryszlak, A., et al. Enrichment of gut microbiome strains for cultivation-free genome sequencing using droplet microfluidics. Cell Rep Methods. 2 (1), 100137 (2021).
- Pollak, B., et al. Universal loop assembly: open, efficient and cross-kingdom DNA fabrication. Biology. 5 (1), (2020).
- Volkmer, B., Heinemann, M. Condition-dependent cell volume and concentration of Escherichia coli to facilitate data conversion for systems biology modeling. PLoS ONE. 6 (7), e23126 (2011).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , (2020).
- Abate, A. R., Weitz, D. A. Faster multiple emulsification with drop splitting. Lab Chip. 11 (11), 1911-1915 (2011).
- Lau, B. T. C., Malkus, P., Paulsson, J. New quantitative methods for measuring plasmid loss rates reveal unexpected stability. Plasmid. 70 (3), 353-361 (2013).
- Oellermann, M., et al. Open hardware in science: The benefits of open electronics. Integr Comp Biol. 62 (4), 1061-1075 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados