Method Article
Reconstrucción y análisis en 3D de estructuras neuronales subcelulares delgadas mediante datos de microscopía electrónica de barrido de haz de iones enfocados
En este artículo
Resumen
Un canal metodológico flexible para identificar, visualizar y cuantificar procesos neuronales subcelulares delgados dentro de volúmenes de imágenes de microscopía electrónica de barrido con haz de iones enfocados utilizando paquetes de software de código abierto fáciles de usar.
Resumen
Los avances recientes en las tecnologías de microscopio electrónico de barrido permiten ahora el análisis tridimensional rápido (3D) de procesos subcelulares ultrafinos. Aquí, se presenta una línea metodológica para identificar, visualizar y analizar procesos neuronales delgados, como los que se proyectan en los botones presinápticos de otras neuronas (denominados 'espínulas'). Utilizando paquetes de software disponibles gratuitamente, este protocolo demuestra cómo utilizar un árbol de decisión para identificar estructuras subcelulares neuronales comunes utilizando criterios morfológicos dentro de volúmenes de imágenes de microscopía electrónica de barrido con haz de iones focalizado (FIB-SEM), con especial atención a la identificación de una diversidad de espínulas que se proyectan en botones presinápticos. En particular, este protocolo describe cómo rastrear las espínulas dentro de las sinapsis neuronales para producir reconstrucciones en 3D de estas proyecciones subcelulares delgadas, sus neuritas progenitoras y socios postsinápticos. Además, el protocolo incluye una lista de programas de software de código abierto disponibles gratuitamente para analizar datos FIB-SEM y ofrece consejos (por ejemplo, suavizado, iluminación) para mejorar las reconstrucciones 3D para su visualización y publicación. Este protocolo adaptable ofrece un punto de entrada al análisis rápido a nanoescala de estructuras subcelulares dentro de volúmenes de imágenes FIB-SEM.
Introducción
Las investigaciones sobre las relaciones estructura-función de los componentes subcelulares nanométricos a menudo se benefician de la visualización y el análisis en 3D1. Sin embargo, los estudios de microscopía electrónica de transmisión de sección en serie se han visto limitados temporal y espacialmente por la necesidad de utilizar un cuchillo de diamante para cortar y alinear cientos o miles de secciones ultrafinas en serie de ≥40 nm. Estas limitaciones han limitado la capacidad de muestrear y analizar eficazmente estructuras subcelulares delgadas (<40 nm de diámetro), y la necesidad de dominar el corte en serie ultrafino ha obstaculizado la aplicación de análisis estructurales 3D 2,3. Sin embargo, los avances recientes en la microscopía electrónica de barrido por haz de iones focalizados (FIB-SEM) han revolucionado la velocidad y la resolución de los volúmenes de imagen obtenibles y ahora permiten el análisis cuantitativo de estructuras subcelulares delgadas como la retícula endoplásmica lisa 4,5, las sinapsis neuronales 3,6 y las vesículas sinápticas 7,8 a escala. Además, el uso más amplio de los volúmenes de imágenes FIB-SEM ha acelerado el desarrollo de repositorios de volúmenes de imágenes FIB-SEM9 y software de análisis 3D de libre acceso (por ejemplo, Espina10, IMOD11, Neuromorph12, Reconstruct13, TrakEM14) que amplían el alcance de esta tecnología y ahora permiten investigaciones sobre la estructura y función de las estructuras subcelulares finas.
Una de estas características subcelulares a nanoescala es la "espínula" sináptica neuronal. Las espínulas son proyecciones delgadas (~0,06-0,15 μm de ancho, 0,1-1 μm de largo), en forma de dedo, que emanan de una neurona y son encapsuladas por la neurita (por ejemplo, el botón presináptico) de otra neurona15,16. Las espínulas incrustadas dentro de los procesos neuronales han sido reportadas en la literatura de microscopía electrónica durante casi 60 años17, y las protuberancias en forma de espínulas son una característica conservada 18,19,20 y ubicua 21,22 de las sinapsis excitatorias. Sin embargo, a pesar de la omnipresencia de las espínulas sinápticas, sus funciones siguen siendo oscuras y hay una escasez de datos necesarios para explicar su abundancia y conservación estructural. Esta falta de caracterización experimental de las espínulas se ha debido principalmente a la dificultad de analizar cuantitativamente la prevalencia y el tamaño de las espínulas. Sus pequeñas dimensiones son las más adecuadas para los análisis que utilizan una resolución z (profundidad) previamente inalcanzable (es decir, ≤15 nm).
Aquí, se presenta una línea de análisis FIB-SEM para identificar, visualizar y analizar estructuras subcelulares delgadas (con secciones transversales de ≤40 nm de ancho) que pueden servir como punto de entrada tanto para los recién llegados como para los expertos de FIB-SEM. Este protocolo sirve como un manual para identificar estructuras neuronales subcelulares dentro de un volumen de imágenes FIB-SEM en 3D, enfatizando cómo usar criterios específicos para reconocer y clasificar subtipos de espínulas y sinapsis. Además, el protocolo demuestra cómo importar volúmenes de imágenes a una plataforma de software de análisis 3D gratuita (Reconstruct), utilizar este software para rastrear espínulas dentro de las sinapsis neuronales excitadoras y producir reconstrucciones 3D de estas proyecciones subcelulares, sus neuritas parentales y botones presinápticos encapsulados. Por último, el protocolo muestra cómo utilizar un software de corte 3D gratuito y de código abierto (Blender) para suavizar la "piel" de las reconstrucciones 3D para su visualización y posible publicación, detallando las ventajas y los posibles inconvenientes de esta técnica.
Protocolo
1. Datos de volumen de imagen y tamaño del objeto subcelular: consideraciones y registro
- Obtener un volumen de imagen FIB-SEM de calidad del área de interés que contiene los objetos subcelulares de interés.
NOTA: Este protocolo utiliza segmentos de un volumen de imagen FIB-SEM de la corteza visual primaria de un hurón adolescente tardío (24,2 x 16,2 x 2,4 μm, vóxeles isotrópicos de 4 nm), y un volumen FIB-SEM de libre acceso del hipocampo CA1 de rata adulta (10,2 x 7,7 x 5,3 μm; vóxeles isotrópicos de 5 nm) puesto a disposición por el laboratorio Knott (https://www.epfl.ch/labs/cvlab/data/data-em/). Es fundamental preparar los bloques de tejido para FIB-SEM utilizando un protocolo que preserve los detalles ultraestructurales y produzca un alto contraste en condiciones de imagen FIB-SEM23,24. Es importante destacar que el tejido debe ser visualizado de tal manera que la resolución del vóxel de la imagen FIB-SEM resultante sea de <0,5 × del tamaño del diámetro de la estructura subcelular más pequeña de interés. Por ejemplo, debido a que los sitios de protrusión de la espínula en los botones presinápticos pueden ser tan pequeños como 30 nm de diámetro, un tamaño de vóxel de <15 nm sería apropiado para muestrear estas estructuras por encima del criterio de Nyquist. Además, es útil obtener imágenes de tejido con una resolución isotrópica (igual en todas las dimensiones) (por ejemplo, vóxeles de 5 x 5 x 5 nm) para permitir la visualización y reconstrucción de estructuras finas que tienen dimensiones iguales en todos los planos x/y/z. - Descargue e instale la distribución de Fiji de ImageJ25 (https://imagej.net/software/fiji/downloads).
- Alinee el volumen de la imagen.
NOTA: Si bien los sistemas FIB-SEM modernos producen volúmenes de imagen aproximadamente alineados, el registro de imagen a imagen aún puede sufrir de deriva de subpíxeles.- En Fiyi, vaya a Plugins | Regístrese y elija Register Virtual Stack Slices. Consulte el Manual del usuario para obtener ayuda (https://imagej.net/Register_Virtual_Stack_Slices). Comience con un registro rígido y, a continuación, utilice una técnica afín si es necesario.
- Escala el volumen de la imagen.
- Abra el volumen de imagen alineado en Fiji haciendo clic en Archivo | Importación | Secuencia de imágenes y navegando a la carpeta con las imágenes alineadas.
- En la ventana de diálogo, haga clic en Ordenar nombres numéricamente si las imágenes se nombran en orden numérico. Haga clic en Usar pila virtual para un rendimiento más rápido de Fiji.
- Una vez abierta la imagen, haga clic en Imagen | Propiedades e inserte el tamaño físico de los píxeles del volumen de la imagen en micrómetros en los cuadros de altura, anchura y profundidad de píxeles. Escriba mmm OR μm en cada uno de los cuadros situados más a la derecha. Guarde este volumen de imagen alineado como un archivo .tiff haciendo clic en Archivo | Guardar como | Tiff.
NOTA: Si la imagen se escaló, la parte superior de la imagen ahora debe indicar el tamaño de la imagen en micrómetros (por ejemplo, 9,82 x 8,65 μm).
2. Identificación de neuritas y espínulas sinápticas
- Mientras se desplaza por la pila (usando las teclas de flecha o la rueda del mouse), consulte el árbol de decisión de identificación de neuritas (Figura 1) y ejemplos de neuritas seccionadas (Figura 2) para ubicar e identificar los objetos de interés.
- Identificar las espínulas sinápticas como protuberancias unidas a la membrana de una neurita que se envuelve completamente dentro de una neurita presináptica o postsináptica.
NOTA: La identificación positiva de la espínula sináptica requiere evidencia visual de la membrana del objeto que proyecta la espínula invaginando en la membrana invertida de una estructura presináptica o postsináptica y la presencia de la espínula unida a la membrana completamente encapsulada por la membrana de la estructura presináptica o postsináptica en al menos una imagen.
- Identificar las espínulas sinápticas como protuberancias unidas a la membrana de una neurita que se envuelve completamente dentro de una neurita presináptica o postsináptica.
- Creación de regiones de interés (ROI) en Fiyi
- Haga clic en una herramienta apropiada en la barra de herramientas de Fiji (por ejemplo, herramienta ovalada) y luego haga clic y arrastre en la imagen para formar un ROI. Registre este ROI en el Administrador de ROI escribiendo la letra "T".
NOTA: El Administrador de ROI ahora debería aparecer con las coordenadas de la imagen como su nombre predeterminado. - Cambie el nombre del ROI como desee haciendo clic en el botón Cambiar nombre en el Administrador de ROI y escribiendo el nombre deseado en el cuadro de texto que aparece. Cambie el nombre de las ROI en función de criterios cuantitativos (por ejemplo, el12º botón presináptico que se analiza y tiene dos sinapsis con espinas postsinápticas podría denominarse B12_s,s).
NOTA: Para asociar cada ROI con un "corte" de imagen (en lugar de tenerlo presente en toda la pila de imágenes) y para hacer que los nombres de ROI sean visibles en imágenes individuales, haga clic en estas opciones en el Administrador de ROI | Más | Opciones y, a continuación, haga clic en las casillas de verificación Mostrar todo y Etiquetas en la parte inferior del Administrador de ROI. Tenga en cuenta que este paso es crucial cuando se guardan los ROI dentro de un volumen de imagen, ya que asociar los ROI con todo el volumen de la imagen en lugar de con imágenes individuales puede provocar un doble recuento de objetos dentro del volumen. - Para guardar todos los ROI que se encuentran actualmente en el Administrador de ROI como un archivo, primero, haga clic en el botón Deseleccionar en el Administrador de ROI, haga clic en el botón Más y desplácese hacia abajo hasta Guardar.
NOTA: Este archivo de .zip guardado ahora se puede abrir en Fiji para volver a cargar todos estos ROI en el Administrador de ROI.
- Haga clic en una herramienta apropiada en la barra de herramientas de Fiji (por ejemplo, herramienta ovalada) y luego haga clic y arrastre en la imagen para formar un ROI. Registre este ROI en el Administrador de ROI escribiendo la letra "T".
3. Determine el área de interés y transfiera el volumen de la imagen al software de análisis 3D
- Determine si es deseable transferir todo el volumen de la imagen al software de reconstrucción y análisis 3D, o si se debe crear una subpila más pequeña (ver 3.3.2) para analizar un número menor de imágenes y/o región de interés de la pila original.
- Realice un pequeño estudio "piloto" en un área relativamente pequeña que contenga los objetos de interés para estimar el tamaño del efecto y el número de objetos de interés necesarios para obtener la potencia deseada para las comparaciones estadísticas. Utilice una estimación conservadora del tamaño del volumen de imagen necesario para muestrear los objetos de interés si estos objetos subcelulares se distribuyen de manera no uniforme dentro del volumen de la imagen.
- Para utilizar una parte del volumen de la imagen para el análisis 3D, primero duplique el volumen de la imagen haciendo clic en Imagen | Duplicado.
- Para utilizar una parte recortada de la imagen para el análisis, elija la herramienta rectángulo de la barra de herramientas Fiji y dibuje la región de interés deseada; Si no lo desea, continúe con el siguiente paso.
- Ir a la imagen | Pilas | Herramientas | Crear Substack. Escriba el rango de imágenes que se incluirán en la subpila (por ejemplo, incluya las imágenes 1 a 500 escribiendo 1 - 500) en el cuadro de texto.
- Consulte las consideraciones de muestreo anteriores (nota después de 1.1) para determinar la resolución z requerida en función del tamaño de los objetos de interés. Por ejemplo, si el volumen de la imagen tiene una profundidad de vóxel de 5 nm, es posible muestrear el objeto de interés a intervalos de 10 nm en la dimensión z para que Fiji conserve todas las demás imágenes de la subpila. Para hacer esto, escriba el número de la enésima imagen después del rango (por ejemplo, para incluir solo todas las demás imágenes en un rango de imágenes de 1 a 500, escriba 1 - 500 - 2).
NOTA: Cada paquete de software de análisis 3D (consulte la Tabla 1) tendrá sus propios requisitos de importación de archivos. Este protocolo se centra en la importación de volúmenes de imagen guardados como archivos .tiff en el software de volumen de imagen, Reconstruct.
4. 3D reconstrucciones y análisis de estructuras subcelulares delgadas en Reconstruct
NOTA: Es muy ventajoso usar un mouse equipado con una rueda mientras se usa Reconstruct. Además, si la mayoría o todos los trazos se realizarán manualmente, el uso de un lápiz óptico para dibujar contornos en una computadora con pantalla táctil puede aumentar drásticamente la eficiencia del rastreo.
- Descargue e instale Reconstruct (https://synapseweb.clm.utexas.edu/software-0) para usarlo en un equipo que ejecute Microsoft Windows en un entorno nativo o virtual.
- Inicie una nueva reconstrucción creando una nueva carpeta para los archivos de reconstrucción, haciendo clic en Series | Nuevo, y guardando esta reconstrucción (archivo .ser) con el nombre de archivo deseado en la carpeta que se acaba de crear.
- Importe el volumen de imagen .tiff guardado en Reconstruir.
- Ir a Series | Importación | Imágenes, haga clic en Seleccionar en la ventana Importar imágenes y localice el archivo de volumen de imagen. En la ventana Importar imágenes , busque los archivos de volumen de imagen que aparecen como una línea separada para cada imagen del volumen.
- Importante: Escriba el tamaño de píxel correcto en el cuadro de texto Tamaño de píxel para que corresponda al tamaño de píxel x/y (en μm) del volumen de la imagen. Una vez hecho esto, haga clic en el botón Importar en la ventana Importar imágenes .
NOTA: Una vez que se hayan importado las imágenes, debe haber una imagen en el medio de la ventana Reconstruir y la lista de Importar imágenes debe estar vacía. - Si hay una imagen en el centro de la ventana Reconstruir y la lista de Importar imágenes está vacía, haga clic en Salir en la ventana Importar imágenes .
- Establezca el grosor de la sección (es decir, la altura del vóxel o la dimensión z) haciendo clic en Sección | Secciones de lista, resaltando todas las secciones enumeradas y, a continuación, haciendo clic en Modificar | Espesor en el menú Lista de secciones y escribiendo el grosor de la sección (en μm) en el cuadro de texto (por ejemplo, 0,005 para una sección de 5 nm de grosor).
NOTA: Recuerde que si se trabaja con una subpila de un volumen de imagen original más grande que solo incluye cada enésima sección (consulte el paso 3.3.3), es necesario tener en cuenta estas secciones "faltantes" en el grosor total de la sección. Por ejemplo, si el grosor de la sección del volumen de la imagen original (también conocido como altura del vóxel) era de 0,005 μm y la subpila solo incluye una de cada tres secciones (por ejemplo, 1-500-3), el grosor de la sección de esta subpila es de 0,015 μm (es decir, 0,005 μm x 3 secciones).
- Haz rastros de los objetos de interés.
- Ir a la serie | Opciones | Nombres/Colores para dar un nombre al nuevo objeto. Elija un color para el contorno y el relleno interior de los trazos del objeto y, a continuación, haga clic en Aceptar.
NOTA: Reconstruct intentará conectar todos los seguimientos con el mismo nombre que un objeto. Consulte el manual de reconstrucción para conocer las convenciones de nomenclatura de seguimiento (por ejemplo, no incluya espacios en los nombres). Los colores de traza son simplemente para visualizar trazas; el color de la reconstrucción 3D de este objeto se puede alterar fácilmente más tarde. - En la barra de herramientas, seleccione la herramienta Dibujar a mano alzada que se parece a un lápiz. Mantenga pulsado el botón izquierdo del ratón y arrastre el ratón para trazar la membrana del objeto; Suelte el botón izquierdo del ratón para autocompletar el seguimiento. Haga clic en Re Pág / Av Pág o utilice la rueda del ratón para avanzar a la siguiente sección óptica en el volumen de la imagen y hacer trazos progresivamente hasta completar la estructura.
- Ir a la serie | Opciones | Nombres/Colores para dar un nombre al nuevo objeto. Elija un color para el contorno y el relleno interior de los trazos del objeto y, a continuación, haga clic en Aceptar.
- Rastreo de espínulas sinápticas
NOTA: Las espínulas incrustadas dentro de los botones presinápticos aparecen como estructuras transparentes unidas a la membrana, ocasionalmente con material opaco a los electrones similar a la actina y/o pliegues similares a la membrana en su interior.- Avance a través del volumen de la imagen y localice el sitio de invaginación de la espínula en el botón presináptico, teniendo cuidado de diferenciar las espínulas de la retícula endoplásmica, vacuolas, endosomas, autofagosomas y otros orgánulos subcelulares.
NOTA: A diferencia de los orgánulos unidos a la membrana, asegúrese de que las espínulas sinápticas siempre se proyecten desde (es decir, la membrana evertida) una neurita/glía y sean recibidas por (es decir, membrana invertida) una estructura pre o postsináptica. Esta evidencia visual de la invaginación de la espílnula debe ocurrir en ≥2 imágenes, y una espínula completamente encapsulada unida a la membrana debe estar presente dentro de la estructura pre o postsináptica en la imagen ≥1. Las espínulas pequeñas pueden estar recubiertas de clatrina. Para diferenciar los eventos de invaginación de la espina de la endocitosis recubierta de clatrina a la neurita pre o postsináptica, asegúrese de que la identificación positiva de las espinulas recubiertas de clatrina requiera la confirmación visual de la membrana everstora coincidente del objeto padre de la espinula que se proyecta en la membrana inversora de una estructura pre o postsináptica dentro de la misma imagen. - Avance a través del volumen de la imagen para localizar el sitio de proyección/origen de la espínula de su neurita/glía madre; consulte la Figura 1 y la Figura 2 para ayudar a identificar estas estructuras principales.
- Comienza el trazo de la espínula en el sitio de protuberancia de la membrana en la estructura receptora (por ejemplo, botón presináptico) y continúa dibujando la espinula durante el tiempo que aparezca como una estructura unida a la membrana dentro de la neurita presináptica o postsináptica encapsulante.
- Continúe manteniendo presionado el botón izquierdo del mouse (o siga dibujando a lo largo de la membrana de la espícula con un lápiz óptico si usa una computadora con pantalla táctil) mientras traza la membrana de la espílnea con el puntero del mouse.
- Cuando se alcance el punto donde se inició el seguimiento, suelte el botón izquierdo del ratón.
NOTA: Si se suelta el botón izquierdo del ratón demasiado pronto, se completará automáticamente el seguimiento, a menudo con resultados no deseados. Si esto sucede, simplemente presione eliminar mientras este seguimiento está resaltado en la Lista de seguimiento. - Repita los pasos 4.5.2-4.5.5 para cada sección en la que aparezca la espínula.
NOTA: Recuerde trazar la espícula como una estructura encapsulada unida a una membrana dentro del botón cuando sea apropiado. Debido a que Reconstruct no permite la sustracción automática de estructuras subcelulares encapsuladas (por ejemplo, espínulas, orgánulos subcelulares) del volumen de la estructura encapsulante, esto se puede realizar post-hoc (por ejemplo, simplemente restando el volumen de la espílnela del volumen total del botón presináptico).
- Avance a través del volumen de la imagen y localice el sitio de invaginación de la espínula en el botón presináptico, teniendo cuidado de diferenciar las espínulas de la retícula endoplásmica, vacuolas, endosomas, autofagosomas y otros orgánulos subcelulares.
- Reconstrucción de objetos en 3D en Reconstruct
- Ir a Series | Opciones | 3D y asegúrese de que se haya hecho clic en el botón de opción Trazos .
- Abra la lista de objetos haciendo clic en Objetos | Objetos de lista.
- Una vez que se carguen los nombres de los objetos trazados en la ventana, haga doble clic en el nombre de los objetos en la Lista de objetos para verlos en 3D. Espere a que aparezca la ventana Escena 3D .
- Asegúrese de que el inicio, el final y los recuentos de los seguimientos sean correctos en la lista de objetos de cada objeto, de modo que el recuento = fin - inicio. Si el recuento ≠ fin - Inicio de un objeto, es posible que se hayan omitido por error uno o varios seguimientos de este objeto.
- Gire los trazos 3D de los objetos manteniendo pulsado el botón izquierdo del ratón y moviendo el ratón y/o manteniendo pulsado el botón derecho del ratón y moviendo el ratón hacia delante y hacia atrás para acercar y alejar. Asegúrese de que los seguimientos del objeto estén correctamente alineados y de que parezca haber una transición suave entre cada seguimiento sucesivo.
- Si es necesario, vuelva a trazar trazas individuales para eliminar los errores que provoquen bordes afilados y/o transiciones entre trazas.
- Coloque una piel de malla tetraédrica en el objeto navegando a Serie | Objeto | 3D y haciendo clic en el botón de radio de la superficie de Boissonnat .
- En la ventana Escena 3D , haga clic en Escena | Desactive y, a continuación, haga doble clic en el nombre del objeto deseado en la lista de objetos.
- Gire los objetos para garantizar transiciones suaves entre trazos y vuelva a dibujar trazos si es necesario.
- Para cambiar los colores y la transparencia del objeto, haga clic en el nombre del objeto en la Lista de objetos y, a continuación, haga clic en Escena | Color/transparencia... en el menú Lista de objetos . Haga doble clic en el nombre del objeto en Lista de objetos para ver este cambio en la ventana Escena 3D .
NOTA: El color, la transparencia y el suavizado de los objetos se pueden modificar con un mayor grado de control y especificidad utilizando Blender (ver 5.4-5.6).
- Análisis cuantitativo de objetos 3D
- Ir a Series | Opciones | Listas y haga clic en todas las mediciones deseadas en las secciones Lista de objetos y Lista de trazas de esta ventana.
- Exporte las medidas del objeto abriendo la Lista de objetos (Objeto | Objetos de lista...) y luego haga clic en Lista | Guardar.
- Guarde las medidas del objeto tal y como se muestran en la Lista de objetos como un archivo delimitado por comas (.csv).
- Importe el archivo .csv en el software de análisis de datos de su elección. Para importar a una hoja de cálculo (por ejemplo, Microsoft Excel), simplemente haga doble clic en el archivo o abra un nuevo libro en blanco, haga clic en Archivo | Importación | CSV y siga las instrucciones.
- Para medir las distancias entre objetos en 3D (por ejemplo, para medir la distancia de proyección de una espílnela sináptica dentro de un botón presináptico), utilice la función Z-trace en Reconstruir.
- En primer lugar, asigne un nuevo nombre al seguimiento navegando a la serie | Opciones | Nombres/Colores y escribir un nombre para el trazado Z.
- A continuación, haga clic en la herramienta Z-trace en la barra de herramientas.
- Para dibujar la traza Z de la distancia de proyección de una espínula sináptica, haga clic en el inicio de la espínula sináptica haciendo clic con el botón izquierdo del ratón y, a continuación, pulse los botones Re Pág o Av Pág para avanzar por el volumen de la imagen mientras hace clic en puntos sucesivos a lo largo de la membrana de la espínula sináptica dentro de cada sección.
- Complete este trazo Z haciendo clic con el botón derecho del ratón.
- Exporte las mediciones de Traza Z haciendo clic en Trazar | Lista de seguimientos... para abrir la Lista de rastreo y luego haga clic en Lista | Guardar para guardar las mediciones de la lista de rastreo como un archivo .csv.
- Adición de un cubo de escala
- Navegue a una de las primeras o últimas imágenes de la pila
- Ir a la serie | Opciones | Nombres/Colores y escriba un nombre para el cubo de escala.
- En la barra de herramientas, elige la herramienta Dibujar rectángulo y dibuja un cuadrado pequeño (sin importancia) adyacente a los objetos, pero sin oscurecerlos. Ir a la serie | Opciones | 3D, haga clic en la opción del botón de opción Caja en el área Generar y especifique el tamaño del cubo de escala (en μm) escribiendo sus dimensiones en las cajas A:, B:, C: en el área Tamaño (por ejemplo, escriba 0,5 para A, B y C para generar un cubo de escala de 0,5 μm/lado).
NOTA: Antes de intentar reconstruir el cubo de escala en 3D, asegúrese de que los objetos 3D deseados ya se hayan reconstruido en la escena 3D utilizando la opción de superficie Trazos o Boissonnat . Recuerde volver a cambiar la opción Generar a la superficie de Boissonnat y los tamaños de A, B y C a -1 después de reconstruir el cubo de escala (es decir, después del paso 4.8.5). - Abra la lista de objetos y haga doble clic en el nombre del cubo de escala; espere a que aparezca el cubo de escala en la ventana de escena 3D .
- Para mover el cubo de escala en relación con los otros objetos, vaya a la sección Serie | Opciones | 3D y escriba el número de μm que desee mover el cubo de escala a los cuadros X, Y y/o Z . Haga clic en Aceptar para cerrar la ventana y, a continuación, haga doble clic en el nombre del cubo de escala en la lista de objetos.
NOTA: Alternativamente, esto se puede hacer usando Blender (ver más abajo).
5. Importar, cambiar el color, suavizar y agregar transparencia a las reconstrucciones 3D en Blender
NOTA: Para obtener asistencia detallada en el uso de Blender, consulte el manual de Blender y / o la miríada de videos de "cómo hacerlo" sobre el uso de cada función de Blender (simplemente haga una búsqueda en la web de Blender Y Función Deseada). Lo que sigue es una breve introducción sobre cómo volver a colorear, suavizar y agregar transparencia a las reconstrucciones en 3D.
- Descargue e instale la última versión de Blender, el paquete de software 3D gratuito y de código abierto (www.blender.org).
- En Reconstruir, guarde las reconstrucciones 3D abriendo los objetos deseados en la ventana de escena 3D y, a continuación, haga clic en Escena | Exportar como | VRML 2.0.
- En Blender, vaya a Archivo | Importación | X3D Extensible 3D (.x3d/.wrl). Haga clic en el archivo VRML 2.0 que se guardó de Reconstruct y luego haga clic en el botón Importar X3D/VRML2 .
- Vuelve a colorear las reconstrucciones.
- En la ventana Colección de escenas , en la esquina superior derecha, haga clic para anular la selección o desmarcar el icono de ojo para el objeto Cubo predeterminado y haga clic en los nombres de los objetos individuales para resaltar cada objeto de la escena. Cambie el nombre de estos objetos según corresponda.
- Haga clic en el nombre del primer objeto para volver a colorear en la ventana Colección de escenas .
- Haga clic en el contexto circular. Material en la parte inferior de la tira derecha de iconos y, a continuación, haga clic en Agregar material. A continuación debería aparecer una lista de opciones. A continuación, haga clic en la barra junto a la opción Color base y elija la luminancia y el tono deseados para el objeto.
- Para modificar el aspecto metálico, el brillo y las cualidades reflectantes del objeto, ajuste las opciones Metálico, Especular, Rugosidad y Brillo para producir el efecto deseado.
NOTA: Para que aparezcan ciertas opciones en la escena (por ejemplo, transparencia, brillo), puede ser necesario estar en la configuración Sombreado de ventanilla: Aspecto Dev o Sombreado de ventana gráfica: Renderizado . Para cambiar a cualquiera de las configuraciones de la ventana gráfica, coloque el cursor sobre los íconos del globo terráqueo en la parte superior derecha y haga clic en la configuración de la ventana gráfica deseada. - Para cambiar el ángulo de iluminación, haga clic en el objeto Luz en la ventana Colección de escenas , haga clic en el icono Mostrar artilugio en la parte superior derecha y luego arrastre la luz alrededor de la escena para lograr el efecto de iluminación deseado. Cambie la intensidad de la luz en la ventana Datos del objeto que aparece debajo de la ventana Colección de escenas cuando se selecciona el objeto Luz .
- Para rotar objetos en los ejes seleccionados por separado, haga clic en los nombres de los objetos en la ventana Colección de escenas , presione la tecla R , luego haga clic en la letra del eje de rotación (tecla x, y o z ) y mueva el mouse para producir la rotación deseada. Para agarrar y mover objetos, selecciónelos en la ventana Colección de escenas , presione la tecla G y, a continuación, use el mouse para mover objetos por la escena.
- Volver a mallar (es decir, suavizar) las reconstrucciones.
- Haga clic en el nombre del objeto de interés en la ventana Colección de escenas y, a continuación, utilice la bola de gimball y la rueda del ratón para centrar y acercar el objeto.
- Haga clic en el icono de llave inglesa Context.Modifiers en la tira de iconos de la derecha, haga clic en Agregar modificador y, a continuación, en Remallar (bajo el encabezado Generar ). Use el menú desplegable para seleccionar Suavizar, haga clic en el botón Sombreado suave y cambie la profundidad y la escala del árbol para lograr la suavidad deseada.
NOTA: Es útil estar en la ventana Sombreado de la ventana Viewport Shading: Wireframe (opción de icono de globo terráqueo en la parte superior derecha) mientras se vuelve a enmallar para ver los resultados directamente. El remallado creará mallas tetraédricas de diferentes tamaños en el objeto. Al suavizar el objeto para eliminar los artefactos de sección o trazado, tenga cuidado de conservar su forma y contornos originales. Comience con una Profundidad de Octree = 7 y una Escala = 0.95, y luego disminuya estos números lentamente si es necesario. Preste especial atención a los objetos pequeños y delgados, como las espínulas y las densidades postsinápticas, ya que el pequeño volumen de estos objetos disminuye el rango de suavizado que se puede hacer sin alterar su forma original.
- Añada transparencia a los objetos.
NOTA: Hacer que los objetos sean transparentes es importante cuando se desea mostrar la relación 3D de un objeto (por ejemplo, espínulas, vesículas presinápticas, mitocondrias, endosomas) que reside dentro de otro objeto (por ejemplo, un botón presináptico).- Haga clic en el nombre del objeto deseado en la ventana Colección de escenas . Desplácese hasta la parte inferior del contexto. Material (icono de la parte inferior derecha en la lista de la derecha), abra la sección Configuración y use el menú desplegable para cambiar el Modo de fusión a Mezcla alfa. A continuación, abra la sección Superficie , desplácese hacia arriba hasta Alfa y cambie el número a un valor inferior a uno (1 = totalmente opaco, 0 = totalmente transparente).
- Exporte instantáneas de las reconstrucciones 3D.
- Una vez que se haya obtenido el color, el suavizado y la iluminación óptimos para las reconstrucciones 3D, guarde la imagen dentro de la escena haciendo clic en el menú Ventana y seleccionando Guardar captura de pantalla.
- En las opciones Guardar captura de pantalla en la parte inferior izquierda, elija el tipo de archivo, el formato de color y la compresión deseada.
- Escriba el nombre de la nueva captura de pantalla en el cuadro de texto del nombre de archivo anterior y, a continuación, guarde la captura de pantalla en la ubicación deseada.
Resultados
Cuantificación del porcentaje de espínulas sinápticas dentro de la población de botones presinápticos excitatorios en la corteza visual primaria de hurones
Aunque se han observado protuberancias en forma de espínulas de las neuritas en los botones presinápticos excitatorios durante décadas19,26, su importancia potencial para la función sináptica ha permanecido oscura. Estos experimentos se diseñaron para determinar la proporción de botones presinápticos excitatorios que contienen espínulas a lo largo del desarrollo postnatal en la corteza visual primaria (V1) del hurón para determinar la importancia potencial de las espínulas para la función sináptica en relación con los hitos del desarrollo. En consecuencia, se adquirieron volúmenes de imágenes FIB-SEM isotrópicas alineadas de 4 nm/vóxeles de un hurón (p)21 (15,1 x 14,1 x 2,8 μm) del día postnatal, p46 (9,7 x 8,4 x 2,7 μm), p60 (24,2 x 16,2 x 2,4 μm) y >p90 (24,2 x 16,2 x 2,4 μm) V1, fotografiado con un FEI Helios 660 DualBeam FIB-SEM con una inclinación de 52°, Distancia de trabajo de 4,2 mm, tensión de aceleración de 3 kV y corriente de 400 pA en modo de retrodispersión. Aunque las pilas se alinearon aproximadamente con el software FEI en la adquisición, todas las pilas pasaron por la alineación de subpíxeles utilizando Fiji (consulte 1.4.3). Estas edades corresponden a antes del inicio de la experiencia visual correlacionada (es decir, apertura de ojos; p21), en el apogeo del período crítico canónico para la plasticidad de la dominancia ocular en el hurón V1 (p46), cerca del final del período crítico (p60) y en la adolescencia tardía (>p90).
Cada volumen de imagen se escaló utilizando Fiji, y se identificó cada botón presináptico excitatorio dentro de los cuatro volúmenes FIB-SEM. Las sinapsis excitadoras se identificaron por la presencia de membranas presinápticas y postsinápticas paralelas, una prominente densidad postsináptica asimétrica (Tipo I de Gray)27 y ≥3 vesículas presinápticas. Cada botón presináptico excitatorio se evaluó a lo largo de su volumen 3D para detectar la presencia de una o más espínulas con base en criterios conservadores (consultar 4.5) y para determinar si la densidad postsináptica (PSD) tenía una discontinuidad denominada perforación que se asocia con tasas más altas de plasticidad28,29. Además, se siguió a cada espílnula hasta su estructura original para determinar la proporción de espínulas que emanan de distintas neuritas o glía. Con este fin, se delinearon regiones de interés (ROI) utilizando la herramienta ovalada en Fiji para cada botón presináptico excitatorio rodeando toda la sinapsis (ver 2.2.1 - 2.2.3). Cada ROI contenía el número de botones (en orden secuencial) y anotaba la presencia/ausencia de una espínula, el objetivo postsináptico (por ejemplo, espina dendrítica o eje dendrítico) y la presencia/ausencia de una PSD perforada (véase la nota después de 2.2.3). Al examinar cada botón excitador dentro de estos cuatro volúmenes de imágenes, se determinó que los porcentajes de botones presinápticos que contenían espínulas aumentaban a lo largo del desarrollo. Sin embargo, las relaciones tridimensionales de las espínulas con los botones presinápticos aún no se habían determinado.
Examinando la relación entre las espínulas sinápticas y sus botones presinápticos excitatorios encapsulantes
Para examinar la relación entre los dos tipos de espínulas más abundantes (es decir, las espínulas que emanan de las espinas postsinápticas y las que se proyectan desde los axones adyacentes) y sus botones presinápticos (SBB) que contienen espílnulas excitatorias, se analizó el volumen de imagen >p90 para determinar los tamaños de estos tipos de espínulas y si estas espínulas fueron engullidas por SBB de tamaño similar. Utilizando los ROI de la cuantificación de la prevalencia de espílpagos en Fiji descrita anteriormente, Se identificaron SBBs que contenían espinillas de espinas postsinápticas o axones/botones adyacentes. Después de examinar estos SBB y espínulas en 3D dentro de Fiji, se determinó que un espesor de sección de 8 nm era suficiente para resolver la invaginación delgada de cada espícula en su respectiva SBB. Como tal, se hicieron subpilas que contenían todas las demás secciones (es decir, con una resolución z de 8 nm), con un rango de subpilas que incluía la extensión 3D completa de los SBB y sus espínulas (ver 3.3). Después de transferir estas subpilas a Reconstruct, se trazaron y reconstruyeron tridimensionalmente 11 SBB que contenían espinillas de espinas postsinápticas y 14 SBB que contenían espinas de axones adyacentes (ver 4.4-4.7). Estos análisis revelaron que las espinas postsinápticas de la espina eran 2,7 veces más grandes que las espinillas de los axones adyacentes (0,016 ± 0,005 μm3 vs. 0,0059 ± 0,001 μm3, media ± SEM, espina postsináptica frente a espinillas axónicas adyacentes, respectivamente; Figura 3C). Sin embargo, dado el pequeño tamaño de la muestra para este estudio piloto, estos datos no fueron estadísticamente significativos a p < 0,05 (prueba U de Mann Whitney, dos colas, U = 56, p = 0,26). Utilizando un software de análisis de tamaño de efecto y potencia estadística (G*Power)30 de libre acceso30, se estima que si este tamaño de efecto medio (0,596) para la diferencia en los volúmenes de la espínula se mantiene, se necesitarán ~60 reconstrucciones más de la espina postsináptica y de las espinillas axónicas adyacentes para obtener un resultado estadísticamente significativo con α = 0,05 y una potencia estadística de 0,95. Curiosamente, estos análisis también encontraron que, si bien los volúmenes de SBB que contenían espinas de la columna vertebral postsináptica eran similares a los de las SBB que contenían espinillas de axón adyacentes (0,21 ± 0,04 μm3 frente a 0,18 ± 0,02 μm3, espina postsináptica frente a SBB que contenían axones adyacentes, respectivamente), el volumen que ocupaban las espinillas de los axones adyacentes dentro de sus SBB envolventes era casi idéntico al volumen que ocupaban las espinillas de la columna postsináptica dentro de sus SBB (19,3 ± 3,2 % frente a 17,5 ± 2,4 %, espina postsináptica vs. espínulas axónicas adyacentes, respectivamente; Prueba U de Mann Whitney, dos colas, U = 68, p = 0,64; Figura 3D). En resumen, estos datos piloto sugieren que las espinas postsinápticas de la espina pueden ser más grandes que sus contrapartes axónicas adyacentes, y que las espinillas axónicas adyacentes pueden invaginarse preferentemente en una población de botones relativamente pequeños en el hurón V1.
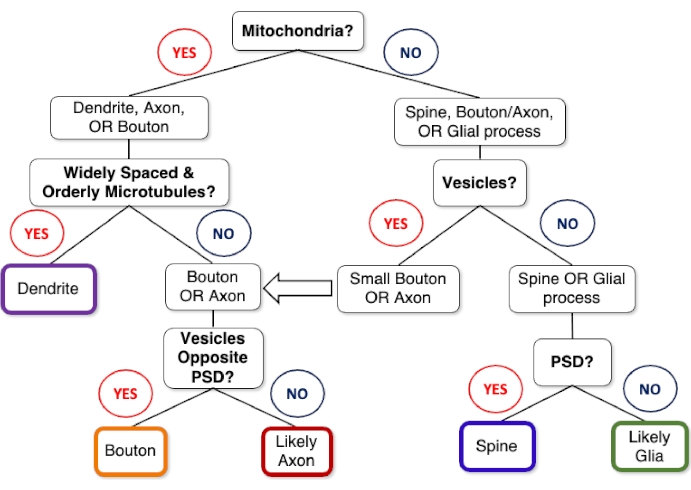
Figura 1: Árbol de decisión para la identificación de neuritas dentro de imágenes FIB-SEM. Las secciones transversales de neuritas (por ejemplo, secciones longitudinales y transversales) dentro de las imágenes FIB-SEM se pueden identificar en función de la presencia/ausencia de algunos orgánulos clave. Por ejemplo, las neuritas que contienen mitocondrias incluyen dendritas, axones y botones presinápticos. Sin embargo, según la mayoría de los protocolos de tinción FIB-SEM, solo las dendritas tendrán una disposición prominente, ampliamente espaciada (~55-70 nm de espaciado)31 y ordenada de los microtúbulos. Por el contrario, los axones y los botones muestran vesículas que contienen neurotransmisores, y los botones exhiben un denso grupo de estas vesículas sinápticas en su(s) zona(s) activa(s) opuesta(s) a la PSD, mientras que los axones contienen microtúbulos densos (~13-30 nm espaciados)31 y neurofilamentos de menor contraste. Las espinas dendríticas casi siempre carecen de mitocondrias, microtúbulos y vesículas y, por lo tanto, se pueden diferenciar más fácilmente de los procesos gliales (por ejemplo, astrocitos) en función de la presencia de una PSD. Sin embargo, las espinas también son en su mayoría más grandes que los procesos gliales, se conectan a su dendrita madre y, a veces, contienen un aparato espinal. Abreviaturas: FIB-SEM = microscopía electrónica de barrido con haz de iones focalizado; PSD = densidad postsináptica. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Cartilla de identificación de la sección transversal de neuritas, que muestra las dendritas y axones neuronales del hipocampo CA1 de rata adulta y el hurón adolescente tardío V1. Como las dendritas y los axones neuronales tienen una gama de tamaños y recorren el cerebro en ángulos tangenciales al plano de sección, las dendritas y los axones de tamaño similar cortados en diferentes ángulos tienen apariencias únicas dentro de las imágenes FIB-SEM. Los dibujos animados centrales muestran una dendrita (arriba) y un axón (abajo) seccionados en un plano longitudinal (amarillo) y un plano transversal (azul). (A, B) Dendritas neuronales seccionadas en un plano longitudinal (es decir, a lo largo de su eje longitudinal). Las flechas apuntan a distinguir las mitocondrias (MC) y los microtúbulos (MT) ordenados y muy espaciados. Nótese que las dendritas seguidas a lo largo de su profundidad también deberían exhibir PSD (puntas de flecha; regiones oscuras y densas en electrones a lo largo de la dendrita y la espina en B). (C, D) Dendritas neuronales seccionadas en un plano transversal (es decir, a lo largo de su eje corto). Las flechas apuntan a MT cortado en un plano transversal que aparece vesicular y MC que parece circular u ovoide. Las dendritas se pueden diferenciar de los axones seccionados transversalmente por sus microtúbulos ordenados y muy espaciados, ~1,25-2,75 × diámetro mayor32, ausencia de vesículas sinápticas y la presencia de una o más PSD. (E, F) Axones neuronales seccionados a lo largo de un plano longitudinal, mostrando MC y vesículas (V). Hay que tener en cuenta que el axón de E contiene microtúbulos ordenados y densamente espaciados, lo que puede llevar a su identificación errónea como dendrita, pero también contiene vesículas prominentes que contienen neurotransmisores. Las vesículas sinápticas agrupadas también se observan en el axón en F en dos zonas activas opuestas a las PSD. Los axones seguidos a través de la profundidad de la mayoría de los volúmenes de imágenes mostrarán botones sinápticos en paso a lo largo de su longitud, como en F. (G, H) Axones neuronales seccionados en un plano transversal, mostrando vesículas prominentes. Los axones seccionados en un plano transversal a menudo parecen ovoides y tienen regiones a lo largo de su profundidad que se encuentran entre las estructuras neuronales de menor diámetro en el neurópilo. Barras de escala en A-H = 0,5 μm. Abreviaturas: FIB-SEM = microscopía electrónica de barrido con haz de iones focalizado; PSD = densidad postsináptica; MC = mitocondrias; MT = microtúbulos; V = vesículas. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Estudio piloto utilizando la línea de análisis descrita para cuantificar los volúmenes de la espina postsináptica y la espínula axonal adyacente dentro de los botones presinápticos en el hurón V1. (A 1-A 3) Dos imágenes secuenciales que muestran la invaginación (A1) y la envoltura (A2) de una espínula de la espina postsináptica (púrpura) en un botón presináptico excitador (gris). Reconstrucción 3D completa (A3) que muestra la espina postsináptica (púrpura) proyectando una gran espínula en forma de ancla en su compañero presináptico (gris, hecho transparente). (B 1-B 3) Dos imágenes secuenciales que muestran un axón adyacente (es decir, no sináptico) (cian) invaginando en (B1) y quedando completamente encapsulado (B2) dentro de un botón presináptico (gris). Nótese que el botón presináptico tiene una sinapsis con una columna vertebral postsináptica a la izquierda de ambas imágenes, indicada por el prominente PSD asimétrico. Reconstrucción completa en 3D (B3) de esta espínula axonal adyacente (cian) dentro de un botón presináptico (gris, hecho transparente). (C) Las espinas postsinápticas de la columna vertebral muestran una tendencia a ser más grandes que las espínulas que se proyectan desde los axones adyacentes (n = 11 y 14, para las espinulas PSS y AdjAx, respectivamente; las barras de error indican el error estándar de la media (SEM); Prueba U de Mann Whitney; p = 0,26). (D) Las espínulas AdjAx ocupan una porción similar de sus botones presinápticos que las espínulas PSs (las barras de error indican SEM; Prueba U de Mann Whitney, p = 0,64.). Barras de escala para A2 y B2 = 0,5 μm; cubos de escala para A3 y B3 = 0,5 μm/lado. Abreviaturas: PSD = densidad postsináptica; PSS = espínulas postsinápticas de la columna vertebral (PS); AdjAx = axones adyacentes. Haga clic aquí para ver una versión más grande de esta figura.
| Software | Compatibilidad con el sistema operativo | Segmentación semiautomatizada | Experiencia de usuario fácil de usar | Funciones de medición 3D | Sitio de descarga | |
| Espina | Windows; Linux | +++ | +++ | ++++ | https://cajalbbp.es/espina/#started | |
| IMOD | Windows; Linux; Mac | + | + | ++++ | https://bio3d.colorado.edu/imod/ | |
| Neuromorph (plugin de Blender) | Windows; Linux; Mac | + | ++ | +++ | https://neuromorph.epfl.ch/software/ | |
| Reconstruir | Windows | + | ++++ | ++ | https://synapseweb.clm.utexas.edu/software-0 | |
| TrakEM2 (Complemento de ImageJ) | Windows; Linux; Mac | ++ | +++ | +++ | https://www.ini.uzh.ch/~acardona/trakem2.html | |
Tabla 1: Software de análisis de volumen de imágenes 3D. Selección de programas de software libre y de código abierto para el registro, visualización, reconstrucción y medición de estructuras subcelulares delgadas dentro de volúmenes de imágenes FIB-SEM. Se presenta una evaluación cualitativa del grado en que cada plataforma de software contiene tres características destacadas para los usuarios (segmentación semiautomática, interfaz de usuario fácil de usar (UX) y características de medición 3D), con "++++" como el grado/cantidad más alto y "+" como el grado/cantidad más bajo. Abreviatura: FIB-SEM = microscopía electrónica de barrido por haz de iones focalizados.
Discusión
Esta canalización de análisis de volumen de imágenes FIB-SEM puede producir reconstrucciones 3D fiables y mediciones cuantitativas de estructuras subcelulares delgadas. Si bien las técnicas semiautomatizadas actuales que utilizan redes neuronales profundas y algoritmos de segmentación pueden aumentar la velocidad y la eficiencia en la reconstrucción de estructuras celulares que poseen un contraste de membrana relativamente alto dentro de grandes volúmenes de imágenes33, muchas estructuras subcelulares (por ejemplo, espínulas, retícula endoplásmica lisa, endosomas) seguirán siendo difíciles de capturar de manera confiable utilizando métodos automatizados debido a su menor contraste de membrana y/o tortuosidad. Aunque los métodos más nuevos han comenzado a capturar mitocondrias de alto contraste y retícula endoplásmica rugosa34,35. Además, las sutiles distinciones entre estructuras subcelulares no marcadas (por ejemplo, endocitosis mediada por clatrina frente a espícula con punta recubierta de clatrina, o endosoma frente a retículo endoplásmico) requerirán una revisión manual recurrente de cualquier técnica de reconstrucción automatizada36. Por lo tanto, este protocolo detalla la importancia de establecer criterios claros para la identificación positiva de objetos subcelulares antes de iniciar un análisis de las estructuras subcelulares dentro de las imágenes FIB-SEM.
Con estos fines, es fundamental familiarizarse con la apariencia de una estructura subcelular particular en un rango de ángulos seccionados aleatoriamente (por ejemplo, Figura 2) y los parámetros de imagen para identificar adecuadamente los objetos de interés y producir rastros precisos y confiables de estas estructuras dentro de volúmenes de imágenes 3D. Además, los criterios y la diversidad estructural aparente de una estructura subcelular de interés deben ser acordados por todos los evaluadores (es decir, analizadores) de los datos a través de pruebas robustas de confiabilidad entre evaluadores utilizando datos reales del terreno previamente anotados. Por ejemplo, una rutina sugerida es entrenar a los evaluadores en volúmenes de imágenes idénticos previamente anotados hasta que la confiabilidad entre evaluadores para el rastreo de objetos subcelulares sea >95% antes de analizar un nuevo conjunto de datos.
También es crucial elegir un protocolo de fijación e incrustación FIB-SEM que maximice el contraste del objeto de interés. Cada protocolo de congelación y/o fijación FIB-SEM tiene distintas ventajas y desafíos en su accesibilidad a la obtención de imágenes y localización de estructuras subcelulares 24,37,38. Para facilitar las reconstrucciones de la estructura subcelular unida a la mícula delgada y otras mínbulas delgadas a partir de volúmenes de imágenes FIB-SEM, es beneficioso utilizar un protocolo de fijación que utilice tanto formaldehído como glutaraldehído, incubaciones largas de metales pesados y/o acetato de uranilo, y un protocolo de inclusión de resina epoxi 4,37. Si bien la congelación a alta presión es la técnica "estándar de oro" para preservar la estructura de las proteínas nativas dentro de muestras de tejido grueso sin inducir deshidratación o artefactos de reticulación de proteínas24,39, la identificación y reconstrucción de estructuras delgadas unidas a membranas dentro de imágenes FIB-SEM requiere un protocolo que ofrezca un contraste más alto que el que ofrecen la mayoría de los protocolos de congelación.
Este protocolo se centra en la identificación, reconstrucción y análisis de espínulas delgadas que se invaginan y se encapsulan completamente en botones presinápticos. La identificación y el análisis de estructuras subcelulares delgadas conectadas a una estructura principal en un extremo, e incrustadas dentro de una estructura separada en su otro extremo, ofrece desafíos únicos. Por ejemplo, es crucial establecer criterios para lo que constituye una envoltura de la proyección subcelular delgada. Si bien es concebible que se pueda establecer que la envoltura está casi completamente rodeada por otra estructura, como una península en un océano, este protocolo requiere que las espínulas se observen como objetos unidos a la membrana completamente incrustados dentro de botones presinápticos envolventes (ver 4.5 y Figura 3). Esta distinción separa las estructuras delgadas que se proyectan parcialmente en otros objetos de las estructuras delgadas que invaginan y están rodeadas por la membrana del objeto envolvente, como una mano entrelazada alrededor de un dedo. Funcionalmente, este último caso implica un mayor contacto de área de superficie, un mayor potencial para que los orgánulos de la estructura de proyección a la envoltura interactúen y una mayor inversión estructural/energética entre la proyección subcelular invaginante delgada y el objeto envolvente. Además, al reconstruir y analizar estos objetos incrustados delgados, es importante determinar si el software de reconstrucción 3D puede restar automáticamente el volumen de las estructuras incrustadas delgadas de los objetos circundantes. Por ejemplo, usando Reconstruct, uno puede simplemente restar el volumen de la espílnela del volumen del botón post-hoc, produciendo un aumento de ~ 100% (después vs. antes de la resta) en la relación entre el volumen de la espílnela y el volumen del botón.
Es importante destacar que este protocolo se centra en el análisis preciso y eficiente de estructuras subcelulares delgadas con imágenes de un tamaño de vóxel pequeño (por ejemplo, <15 nm/vóxel) que permite el muestreo fiel de su ultraestructura 3D. Como tal, estos pasos son susceptibles de un análisis relativamente rápido de cientos de estructuras subcelulares dentro de volúmenes de imágenes FIB-SEM dentro de un marco de tiempo razonable (por ejemplo, semanas o meses). Sin embargo, dado que este protocolo utiliza la segmentación manual para reconstruir tridimensionalmente objetos de interés, sería difícil y probablemente llevaría mucho tiempo identificar y reconstruir miles de objetos subcelulares delgados para un solo experimento. Además, si los objetos subcelulares de interés, o algún subconjunto de ellos, son relativamente escasos dentro del volumen de la imagen, puede ser difícil identificar suficientes de estos objetos para las comparaciones estadísticas. En estos casos, sería ventajoso escribir/utilizar un algoritmo para acelerar la identificación y/o segmentación de las estructuras subcelulares, seguido de una revisión manual del proceso de identificación y segmentación.
El análisis cuantitativo de estructuras subcelulares delgadas dentro de volúmenes de imágenes FIB-SEM es una técnica robusta, confiable y adaptable, que se utiliza para investigar estructuras tan dispares como la disposición compleja de un parásito Plasmodium chabaudi incrustado dentro de un eritrocito40, la organización 3D de proyecciones de telópodos delgados dentro de telocitos cardíacos41 y los procesos citoplasmáticos delgados de osteocitos y osteoblastos42. Aquí, se describe una línea de análisis que puede servir como punto de entrada para la identificación, reconstrucción y análisis de estructuras subcelulares delgadas dentro de volúmenes de imágenes FIB-SEM. Si bien este protocolo se centró en el análisis de las espínulas sinápticas dentro de los botones presinápticos en el tejido cerebral, estos pasos generalizables son susceptibles de la reconstrucción y el análisis de proyecciones subcelulares delgadas en una variedad de tipos de tejidos y células.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Este trabajo fue apoyado por el Fondo Puente de la Universidad de Washington y el Fondo Piloto RRF de la Universidad de Washington Tacoma. Muchas gracias a la Dra. Claudia López y la Dra. Jessica Riesterer del MMC de la Universidad de Salud y Ciencias de Oregón por el apoyo técnico FIB-SEM, al Dr. Graham Knott por el uso del volumen de imagen CA1 FIB-SEM, y a los estudiantes de UW Tacoma en el curso de Reconstrucciones Neuronales (TBIOMD 495) por su paciencia y excelencia al trabajar con este protocolo.
Materiales
| Name | Company | Catalog Number | Comments |
| Fiji (ImageJ) | https://imagej.net/software/fiji/downloads | ||
| Reconstruct | https://synapseweb.clm.utexas.edu/software-0 | ||
| Blender | Blender Foundation | https:/www.blender.org |
Referencias
- Holler, S., Kostinger, G., Martin, K. A. C., Schuhknecht, G. F. P., Stratford, K. J. Structure and function of a neocortical synapse. Nature. 591 (7848), 111-116 (2021).
- Xu, C. S., Pang, S., Hayworth, K. J., Hess, H. F. Transforming FIB-SEM. Volume microscopy: Multiscale imaging with photons, electrons, and ions. Wacker, I., Hummel, E., Burgold, S., Schroder, R. , Springer. US. 221-243 (2020).
- Merchan-Perez, A., Rodriguez, J. R., Alonso-Nanclares, L., Schertel, A., Defelipe, J. Counting synapses using FIB/SEM microscopy: a true revolution for ultrastructural volume reconstruction. Frontiers in Neuroanatomy. 3, 18(2009).
- Wu, Y., et al. Contacts between the endoplasmic reticulum and other membranes in neurons. Proceedings of the National Academy of Sciences of the United States of America. 114 (24), 4859-4867 (2017).
- Weigel, A. V., et al. ER-to-Golgi protein delivery through an interwoven, tubular network extending from ER. Cell. 184 (9), 2412-2429 (2021).
- Rodriguez-Moreno, J., et al. Quantitative 3D ultrastructure of thalamocortical synapses from the "lemniscal" ventral posteromedial nucleus in mouse barrel cortex. Cerebral Cortex. 28 (9), 3159-3175 (2018).
- Khanmohammadi, M., Waagepetersen, R. P., Sporring, J. Analysis of shape and spatial interaction of synaptic vesicles using data from focused ion beam scanning electron microscopy (FIB-SEM). Frontiers in Neuroanatomy. 9, 116(2015).
- Cali, C., et al. The effects of aging on neuropil structure in mouse somatosensory cortex-A 3D electron microscopy analysis of layer 1. PLoS One. 13 (7), 0198131(2018).
- Vogelstein, J. T., et al. A community-developed open-source computational ecosystem for big neuro data. Nature Methods. 15 (11), 846-847 (2018).
- Morales, J., et al. Espina: A tool for the automated segmentation and counting of synapses in large stacks of electron microscopy images. Frontiers in Neuroanatomy. 5, 18(2011).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Jorstad, A., Blanc, J., Knott, G. NeuroMorph: a software toolset for 3D analysis of neurite morphology and connectivity. Frontiers in Neuroanatomy. 12, 59(2018).
- Fiala, J. C. Reconstruct: a free editor for serial section microscopy. Journal of Microscopy. 218, Pt 1 52-61 (2005).
- Cardona, A., et al. TrakEM2 software for neural circuit reconstruction. PLoS One. 7 (6), 38011(2012).
- Petralia, R. S., Wang, Y. X., Mattson, M. P., Yao, P. J. Invaginating structures in mammalian synapses. Frontiers in Synaptic Neuroscience. 10, 4(2018).
- Petralia, R. S., Wang, Y. X., Mattson, M. P., Yao, P. J. Structure, distribution, and function of neuronal/synaptic spinules and related invaginating projections. Neuromolecular Medicine. 17 (3), 211-240 (2015).
- Pappas, G., Purpura, D. Fine structure of dendrites in the superficial neocortical neuropil. Experimental Neurology. 4, 507-530 (1961).
- Case, N. M., Gray, E. G., Young, J. Z. Ultrastructure and synaptic relations in the optic lobe of the brain of Eledone and Octopus. Journal of Ultrastructure Research. 39 (1), 115-123 (1972).
- Bailey, C. H., Thompson, E. B., Castellucci, V. F., Kandel, E. R. Ultrastructure of the synapses of sensory neurons that mediate the gill-withdrawal reflex in Aplysia. Journal of Neurocytology. 8 (4), 415-444 (1979).
- Wagner, H., Djamgoz, M. B. A. Spinules: a case for retinal synaptic plasticity. Trends in Neuroscience. 16 (6), 201-206 (1993).
- Campbell, C., Lindhartsen, S., Knyaz, A., Erisir, A., Nahmani, M. Cortical presynaptic boutons progressively engulf spinules as they mature. eNeuro. 7 (5), (2020).
- Spacek, J., Harris, K. M. Trans-endocytosis via spinules in adult rat hippocampus. Journal of Neuroscience. 24 (17), 4233-4241 (2004).
- Knott, G., Rosset, S., Cantoni, M. Focussed ion beam milling and scanning electron microscopy of brain tissue. Journal of Visualized Experiments: JoVE. (53), e2588(2011).
- Steyer, A. M., Schertel, A., Nardis, C., Möbius, W. FIB-SEM of mouse nervous tissue: Fast and slow sample preparation. Methods in Cellular Biology. 152, 1-21 (2019).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Westfall, J. A. Ultrastructure of synapses in a primitive coelenterate. Journal of ultrastructure research. 32 (3), 237-246 (1970).
- Gray, E. G. Axo-somatic and axo-dendrritic synapses of the cerebral cortex: An electron microscope study. Journal of Anatomy. 93 (4), 420-433 (1959).
- Jones, D. G., Calverley, R. K. Perforated and non-perforated synapses in rat neocortex: three-dimensional reconstructions. Brain Research. 556, 247-258 (1991).
- Geinisman, Y., et al. Structural synaptic correlate of long-term potentiation: Formation of axospinous synapses with multiple, completely partitioned transmission zones. Hippocampus. 3 (4), 435-445 (1993).
- Faul, F., Erdfelder, E., Lang, A. -G., Buchner, A. G*Power 3: A flexible statistical power analysis program for the social, behavioral, and biomedical sciences. Behavior Research Methods. 39, 175-191 (2007).
- Chen, J., Kanai, Y., Cowan, N. J., Hirokawa, N. Projection domains of MAP2 and tau determine spacings between microtublules in dendrites and axons. Nature. 360, 674-677 (1992).
- Benavides-Piccione, R., et al. Differential structure of hippocampal CA1 pyramidal neurons in the human and mouse. Cerebral Cortex. 30 (2), 730-752 (2020).
- Kornfeld, J., Denk, W. Progress and remaining challenges in high-throughput volume electron microscopy. Current Opinion Neurobiology. 50, 261-267 (2018).
- Liu, J., et al. Automatic reconstruction of mitochondria and endoplasmic reticulum in electron microscopy volumes by deep learning. Frontiers in Neuroscience. 14, 599(2020).
- Lucchi, A., Smith, K., Achanta, R., Knott, G., Fua, P. Supervoxel-based segmentation of mitochondria in EM image stacks with learned shape features. IEEE Transactions on Medical Imaging. 31 (2), 474-486 (2012).
- Chklovskii, D. B., Vitaladevuni, S., Scheffer, L. K. Semi-automated reconstruction of neural circuits using electron microscopy. Current Opinion in Neurobiology. 20 (5), 667-675 (2010).
- Knott, G., Marchman, H., Wall, D., Lich, B. Serial section scanning electron microscopy of adult brain tissue using focused ion beam milling. Journal of Neuroscience. 28 (12), 2959-2964 (2008).
- Takahashi-Nakazato, A., Parajuli, L. K., Iwasaki, H., Tanaka, S., Okabe, S. Ultrastructural observation of glutamergic synapses by focused ion beam scanning electron microscopy (FIB-SEM). Glutamate Receptors: Methods and Protocols. Burger, C., Velardo, M. J. , Humana Press. 17-27 (2019).
- Korogod, N., Petersen, C. C., Knott, G. W. Ultrastructural analysis of adult mouse neocortex comparing aldehyde perfusion with cryo fixation. Elife. 4, 05793(2015).
- Soares Medeiros, L. C., De Souza, W., Jiao, C., Barrabin, H., Miranda, K. Visualizing the 3D architecture of multiple erythrocytes infected with Plasmodium at nanoscale by focused ion beam-scanning electron microscopy. PLoS One. 7 (3), 33445(2012).
- Cretoiu, D., Hummel, E., Zimmermann, H., Gherghiceanu, M., Popescu, L. M. Human cardiac telocytes: 3D imaging by FIB-SEM tomography. Journal of Cellular and Molecular Medicine. 18 (11), 2157-2164 (2014).
- Hasegawa, T., et al. Three-dimensional ultrastructure of osteocytes assessed by focused ion beam-scanning electron microscopy (FIB-SEM). Histochemistry and Cell Biology. 149 (4), 423-432 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados