Method Article
Calcio de pericitos cerebrales e imágenes hemodinámicas en ratones transgénicos in vivo
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo presenta pasos para adquirir y analizar imágenes de calcio fluorescente de pericitos que envuelven el cerebro y datos de flujo sanguíneo de vasos sanguíneos cercanos en ratones anestesiados. Estas técnicas son útiles para estudios de fisiología de células murales y se pueden adaptar para investigar transitorios de calcio en cualquier tipo de célula.
Resumen
Los avances recientes en biología de proteínas y genética de ratones han hecho posible medir las fluctuaciones de calcio intracelular de las células cerebrales in vivo y correlacionar esto con la hemodinámica local. Este protocolo utiliza ratones transgénicos que han sido preparados con una ventana craneal crónica y expresan el indicador de calcio codificado genéticamente, RCaMP1.07, bajo el promotor de actina del músculo liso α para etiquetar específicamente las células murales, como las células del músculo liso vascular y los pericitos envolventes. Se describen los pasos sobre cómo preparar un catéter de vena de cola para la inyección intravenosa de tintes fluorescentes para rastrear el flujo sanguíneo, así como cómo medir el calcio de pericitos cerebrales y la hemodinámica de los vasos sanguíneos locales (diámetro, velocidad de los glóbulos rojos, etc.) mediante microscopía de fotones in vivo a través de la ventana craneal en ratones anestesiados con ketamina / xilazina. Finalmente, se proporcionan detalles para el análisis de las fluctuaciones de calcio y las películas de flujo sanguíneo a través de los algoritmos de procesamiento de imágenes desarrollados por Barrett et al. 2018, con énfasis en cómo estos procesos se pueden adaptar a otros datos de imágenes celulares.
Introducción
La vasculatura del sistema nervioso central consiste en arteriolas penetrantes, capilares y vénulas ascendentes. Dentro de esta red, las células murales como las células del músculo liso vascular encierran arteriolas y pericitos extienden procesos celulares a lo largo de las primeras ramas y capilares de la arteriola1. Los pericitos parecen tener varias funciones dentro del cerebro, incluido el mantenimiento de la barrera hematoencefálica1,2,la migración y la motilidad3,las propiedades potenciales de las células madre y la regulación del flujo sanguíneo cerebral4,5,6. Muchas de las funciones funcionales de los pericitos se han relacionado con fluctuaciones en el calcio intracelular que pueden regular la dilatación o contracción de estas células4,5,6.
Varios estudios recientes han establecido criterios para identificar diferentes tipos de pericitos cerebrales7,8. Las células murales dentro de las primeras 4 ramas de las arteriolas penetrantes son pericitos envolventes en función de su expresión de la proteína contráctil α-actina del músculo liso (αSMA) y sus somata ovoides sobresalientes con procesos que envuelven alrededor de los vasos7,8,9. Para visualizar las fluctuaciones de calcio en los pericitos de envainamiento, este protocolo utiliza una nueva línea de ratón transgénico, Acta2-RCaMP1.07, también conocida como Tg(RP23-370F21-RCaMP1.07)B3-3Mik/J10. Estos ratones expresan el indicador de calcio rojo codificado genéticamente, RCaMP1.07, en células que expresan αSMA (células del músculo liso vascular y pericitos envolventes). Las colonias de cría se mantienen cruzando animales no pastores con hemicigotas. RCaMP1.07 es una proteína fluorescente roja con un dominio de unión a calmodulina, que aumenta la fluorescencia al unirse al calcio intracelular10,11. Este protocolo describe los pasos para la obtención combinada de imágenes de calcio de pericitos envainados y mediciones del flujo sanguíneo mediante microscopía de dos fotones, incluidos los procedimientos para la inyección de colorantes fluorescentes en la vena de la cola, la adquisición de imágenes con microscopio en ratones anestesiados y el análisis de datos con plataformas de programación(Figura 1). Estas técnicas son útiles para abordar preguntas sobre la fisiología de las células murales, pero se pueden adaptar para estudiar los transitorios de calcio en cualquier tipo de célula en el cerebro u otro sistema de órganos.
Se utilizó un ratón Hembra Acta2-RCaMP1.07 de 10 meses de edad para el experimento presentado en este artículo. El ratón se sometió a una cirugía para la ventana craneal crónica y la cabeza después de la implantación dos meses antes. Los detalles para el protocolo quirúrgico se discuten en estudios previos12,13 y procedimientos similares se han realizado en otros protocolos publicados anteriormente14,15. La vasculatura se marca con fluoresceína-dextrano verde (70.000 MW, solución adiónica, 2,5% p/v) inyectada por vía intravenosa. Este tinte es rentable y está fácilmente disponible en fuentes comerciales, pero tiene un espectro de emisión más amplio que puede superponerse con la emisión de RCaMP y sangrar durante la adquisición de imágenes de microscopio. Los pasos para la desmezcla espectral se describen en la Sección 4 a continuación para eludir esto, pero también se pueden usar otros tintes verdes con espectros de emisión más estrechos, como los basados en EGFP.
Protocolo
Todos los procedimientos que involucran animales de experimentación que se describen a continuación han sido aprobados por el Comité de Cuidado de Animales de la Universidad de Manitoba, que se rige por el Consejo Canadiense de Cuidado de Animales.
1. Configuración y preparación del procedimiento
NOTA: Se requieren los siguientes elementos para una inyección de catéter de vena de cola: jeringas de insulina, una pieza de tubo de PE10 de 15 cm, agujas de 30 G, gasa, solución salina, fórceps, tinte dextrano de fluoresceína verde, alicates y tijeras. Además, tenga una aguja lista con anestesia con ketamina / xilazina que se inyectará antes de la sesión de imágenes.
CRÍTICO: Todos los materiales y equipos en los Pasos 1 y 2 deben ser esterilizados antes del uso mediante autoclave o enjuague con etanol al 70%. Si los alicates no se pueden esterilizar adecuadamente, se recomienda el uso de un par de portaagujas grandes. El montaje del catéter debe hacerse con alicates y pórceps para evitar punciones accidentales con aguja.
- Corte aproximadamente 15-20 cm de tubos de polietileno, PE10 (I.D. 28 mm; D.O. 61 mm).
- Llene una jeringa de insulina de 27 G con solución salina al 0,9% y ate la aguja de la jeringa en la punta del tubo de polietileno. Empuje la solución salina a través del tubo, asegurándose de que no haya fugas.
- Con alicates, doble una aguja de 30 G (0,3 mm x 25 mm) hacia adelante y hacia atrás hasta que se rompa del cubo. La aguja debe estar limpia sin curvas.
- Sosteniendo la aguja con forzadores, inserte cuidadosamente la aguja en el extremo del tubo de PE10 que está conectado a la jeringa llena de solución salina y retire las burbujas de aire. Este es el catéter para inyección.
- Filtrar 30 μL de 2,5% (p/v) de fluoresceína dextrano a través de un filtro de 13-25 μm antes de la inyección.
- Llene otra jeringa de insulina con la alícuota de 30 μL de dextrano y asegúrese de que no haya burbujas en la jeringa llena.
2. Inyección de la vena de la cola
- Anestesiar al ratón con isoflurano (4% de inducción, 1,5% de mantenimiento) o ketamina/xilazina (60 mg/kg; 10 mg/kg; p.i.) y aplicar gel lubricante para los ojos. La ketamina/xilazina se recomienda para mediciones de flujo sanguíneo más estables durante las imágenes y la dosis se puede aumentar a 90 mg/kg; 10 mg/kg de ketamina/xilazina para sesiones de diagnóstico por imágenes más largas.
- Cuando el ratón esté en el plano quirúrgico de la anestesia, coloque un guante lleno de agua tibia en la cola para dilatar la vena lateral.
- Retire el guante después de 30 s y limpie la cola con etanol.
- Coloque la cola entre el pulgar y el dedo medio. Proporcionar presión con el dedo índice en la cola para dilatar la vena. Con la otra mano, recoge la aguja del catéter con las pórceps orientando el bisel hacia arriba hacia el techo.
- Después de limpiar la cola con etanol al 70%, inserte suavemente la aguja en la vena en un ángulo de 0 ° e inyecte suavemente solución salina a través del catéter para asegurarse de que la aguja se coloque correctamente.
NOTA: Si no hay resistencia en el émbolo y no hay hinchazón de la cola, entonces la aguja está en la vena. Si hay resistencia o hinchazón significativas, se debe retirar la aguja. Los pasos 2.2-2.5 se pueden repetir hasta 3 veces a cada lado de la cola, reemplazando la aguja 30G al final del catéter cada segundo ensayo hasta que la colocación sea correcta. - Una vez que la aguja esté en la vena, cambie la jeringa salina al final del catéter con la jeringa que contiene fluoresceína dextrano (Paso 1.6). Inyecte lentamente el dextrano en el tubo del catéter, asegurándose de que no entren burbujas en el tubo. Si hay una burbuja de aire aparente, corte el tubo que contiene la burbuja para retirarla y vuelva a colocar la jeringa.
- Cuando se haya inyectado todo el dextrano (alícuota de 30 μL), retire la jeringa y reemplácela con la jeringa salina. Inyecte el dextrano restante del tubo en el ratón hasta que no quede ningún tinte en el tubo.
- Retire la aguja de la cola y proporcione presión con una gasa durante 10-30 s hasta que el sangrado se detenga.
CRÍTICA: Si después de 6 intentos la inyección de la vena de la cola no tiene éxito, el animal debe ser fotografiado en otra sesión. Además, el volumen total (solución salina y dextrano) inyectado en el ratón no debe exceder los 100 μL.
3. Microscopía de dos fotones
- Centrarse en la ventana craneal
NOTA: Use anestesia con ketamina/xilazina durante la adquisición de datos porque tiene menos efectos vasculares (vasodilatación) que el isoflurano. Si usa isoflurano en los pasos anteriores, inyecte al ratón ketamina/xilazina i.p. (dosis recomendada descrita anteriormente) antes de la obtención de imágenes.- Fije el ratón con un tornillo a través de su poste de la cabeza a una plataforma con una almohadilla térmica bajo el microscopio.
- Aplique lubricante para los ojos en los ojos del ratón.
- Limpie la ventana craneal con aplicadores dentales húmedos. Asegúrese de que no queden partículas que puedan interferir con el proceso de obtención de imágenes.
- Aplique gel de ultrasonido en la ventana.
- Enfoque a través del objetivo del microscopio de dos fotones hasta que los vasos sanguíneos piales se puedan ver debajo de la ventana.
- Compruebe la respiración del ratón y asegúrese de que la almohadilla térmica proporciona suficiente soporte de temperatura.
- Adquisición de imágenes
NOTA: El microscopio de dos fotones utilizado en este experimento tiene un láser Ti-Sapphire sintonizable para la excitación de fluorescencia con una célula Pockel que controla la cantidad de láser que llega a la muestra. La luz emitida se divide por un dicroico de paso largo de 565 a dos tubos fotomultiplicadores GaAsP (PMT) con filtro de paso de banda 595/50 (rojo) y filtro de paso de banda 525/70 (verde) para la detección.
Los procedimientos descritos en los pasos 3.2 a 3.4 se realizan utilizando un software específico del microscopio de dos fotones de este protocolo (ver Tabla de Materiales). Estos pasos se pueden adaptar a otro software y equipo de microscopio.- Con las luces de la habitación apagadas, ajuste la longitud de onda deseada en el software del microscopio a 990 nm para excitar tanto RCaMP como fluoresceína-dextrano haciendo clic en el cuadro láser 2-P.
- Ajuste la potencia del láser haciendo clic en la sección Power/Gain box/Lasers y ajustando el voltaje de la celda Pockels 1 al 30% o un valor de 300 en una escala de 1000. La potencia del láser que llega a la muestra en esta configuración se determinó previamente que era de ~ 30 mW.
- Configure la sensibilidad del detector PMT haciendo clic en la sección Power/Gain box/PMTs y ajustando el valor a 700-800.
NOTA: Estos valores se pueden ajustar en relación con la intensidad de la muestra fluorescente y deben establecerse en cero antes de encender las luces de la habitación. - Vaya a la sección Resolución de imagen y haga clic en la resolución de 512 x 512 para obtener un tamaño de imagen más grande.
- Haga clic en Láser 2-P/Abrir para abrir el obturador láser 2-P.
- Vaya a la sección Escaneo y haga clic en el botón Escaneo en vivo.
NOTA: Se puede ver el escaneo en vivo con estos parámetros y una resolución más alta, las células murales RCaMP positivas y el plasma sanguíneo etiquetado fluorescentemente. Si la señal es débil, el valor de pockel se puede aumentar hasta que la imagen sea clara.
CRÍTICO: En las capas de tejido superficial, la potencia del láser no debe exceder los 50 mW, que es aproximadamente 600 en la configuración de las células de Pockels en este ejemplo.
- Adquirir una pila de profundidad de las células murales y la red vascular
NOTA: Se recomienda la adquisición de una pila de profundidad para localizar adecuadamente los pericitos en la red vascular. Los pericitos de envainamiento se encuentran en la primera a cuarta ramas de la arteriola penetrante7,8,9. El software de microscopio utilizado en este protocolo se refiere a las pilas de profundidad como "serie Z".- Moviendo el objetivo del microscopio en los planos X, Y y Z, localizar una arteria grande en la superficie del cerebro en función del etiquetado de células musculares lisas de RCaMP.
- Haga clic en el cuadro Z-Series.
- Concéntrese en la parte superior del tejido cerca de los vasos piales, establezca esto como el punto cero y la parte superior de la pila de la serie Z haciendo clic en la sección Actual de la serie Z / Posición de inicio [μm]. Haga clic en el cuadro con cuatro rayas negras y una franja roja en la parte superior.
- Concéntrese hacia abajo en el tejido a la profundidad deseada y configúre esto como la parte inferior de la pila haciendo clic en la sección Actual de la serie Z / Posición de parada [μm]. Haga clic en el cuadro con cuatro rayas negras y una franja roja en la parte inferior.
- Establezca el grosor de cada plano de imagen (tamaño de paso) en 1-2 μm escribiendo el valor deseado en el cuadro debajo del botón " Tamaño depaso" (el botónTamaño de paso está localizado en la sección serie Z/Serie Z actual). Esto definirá el número de imágenes que se adquieren en la pila.
- Configure la potencia del láser para que aumente exponencialmente a medida que el microscopio se mueve más profundamente a través de la pila haciendo clic en el cuadro Compensación láser / PMT y seleccionando Relativo (gradiente exponencial).
- Asigne un nombre al archivo, elija una carpeta para guardarlo y haga clic en Iniciar Z-series.
- Después de la adquisición, abra la serie Z en el software de procesamiento de imágenes.
- Fusione los dos canales como imágenes coloreadas y escanee a través de la pila en busca de pericitos y vasos sanguíneos de interés haciendo clic en el cuadro Imagen | | de color Canales divididos; Imagen | | de color Combinar canales.
- Seleccione regiones de interés (ROI) que contengan pericitos y guarde las posiciones para ayudar a localizar estos puntos nuevamente en futuras sesiones de imágenes.
- Adquisición de películas de imágenes de calcio de la serie T (tiempo)
- Usando la pila de profundidad y los ROI desde arriba como referencia, mueva el objetivo del microscopio en los ejes X, Y y Z durante el modo de escaneo en vivo hasta que se encuentre un pericito de interés.
- Para recopilar una película de eventos de calcio pericito, aumente la velocidad de fotogramas de adquisición (>10 fotogramas por segundo) yendo a la sección Resolución de imagen y haciendo clic en el cuadro 128x128.
- Establezca la duración de la imagen en 60 s haciendo clic en el cuadro de la serie T e ingresando el tiempo en el cuadro de duración.
- Junto al cuadro Guardar ruta, haga clic en el botón con tres puntos para actualizar la ruta de guardado con un nombre de archivo único.
- Zoom óptico en el recipiente para tener en cuenta la resolución más baja y obtener una vista más cercana del pericito ajustando el valor en la sección Zoom óptico [mag].
- Adquiera la serie T haciendo clic en Iniciar la serie T.
- Mediciones hemodinámicas con kymographs (escaneos de línea)
- Concéntrese en el recipiente de interés a una resolución de 512 x 512píxeles en el modo Live Scan.
- Para medir el diámetro de los vasos sanguíneos y la velocidad de los glóbulos rojos, haga clic en el Escaneo de línea para iniciar un escaneo unidimensional con el microscopio.
- Establezca la duración del escaneo (30-60 s) en milisegundos.
- Dibuje una línea que divida el recipiente de interés y se mueva paralelo a lo largo del recipiente. Esto generará un kymograph del diámetro del vaso a la izquierda y rayas de los glóbulos rojos que se mueven a través del vaso a la derecha.
NOTA: Se pueden medir varios vasos sanguíneos con la misma línea, siempre y cuando estén en el mismo plano de imagen. - Asigne un nombre al archivo y haga clic en las líneas de inicio para adquirir los datos.
4. Análisis de imágenes
- Análisis de películas de calcio.
NOTA: Este protocolo describe los pasos para la desmezcla espectral (Figura 2) y dos métodos diferentes para analizar los eventos de calcio de pericitos que envuelven utilizando ROI manuales seleccionados a mano (Figura 3) y la selección automatizada del ROI basada en actividades (Figura 4)16,17. Para detectar y clasificar los picos de señal con la traza de calcio normalizada de cada ROI, los datos se filtran de paso largo y paso de banda, lo que ayuda a suavizar los datos para las estimaciones de amplitud y anchura y también a identificar picos de diferentes formas: picos individuales, picos múltiples y mesetas (Figura 3B). Los parámetros para este análisis se pueden optimizar para detectar diferentes tipos de señales celulares dinámicas. Los pasos a continuación requerirán el uso de software de procesamiento de imágenes y software de programación con paquetes de procesamiento de imágenes que contienen diferentes códigos para analizar películas de calcio como se mencionó anteriormente. Consulte la tabla de materiales para obtener una lista completa de los programas y paquetes utilizados en este protocolo. Los datos de imágenes de diferentes tipos de microscopios se pueden importar con estos paquetes manteniendo los metadatos de las imágenes.
NOTA: Los pasos 4.1.1-4.1.7 describen cómo seleccionar el ROI a mano en el software de procesamiento de imágenes para su posterior uso en el método de análisis manual de calcio (Paso 4.1.16)- Cargue la serie T de imágenes de calcio en el software de procesamiento de imágenes, arrastrando el archivo de .xml a la barra de herramientas del software. Haga clic en el cuadro Aceptar.
- Tome el promedio de la pila (el promedio de la pila está etiquetado como "proyección Z" por el software de procesamiento de imágenes). Esto se puede hacer haciendo clic en imagen | Pilas | Proyección Z en la barra de herramientas.
- Haga una imagen coloreada desde ambos canales como en el paso 3.3.9.
- Abra la ventana del administrador de ROI haciendo clic en el cuadro Analizar | Herramientas | ROI Manager,o simplemente pulsando la letra"T"en el teclado.
- Seleccione la herramienta de polígono haciendo clic en la forma del polígono en la barra de herramientas del software de procesamiento de imágenes y delinee las estructuras de pericitos de envainamiento visibles, como el soma y los procesos.
- Haga clic en el botón Agregar ubicado en la ventana del administrador de ROI para agregar ROI seleccionados en el administrador de ROI.
- Asigne a cada región de interés un nombre único haciendo clic en el botón Cambiar nombre y guárdelos como una carpeta zip que se puede cargar más adelante en el software de programación haciendo clic en Más>> | Guardar.
NOTA: Los pasos 4.1.8-4.1.14 describen cómo importar la serie T de calcio a la plataforma de programación y cómo desmezclar los diferentes fluoróforos detectados por los PMT del microscopio en diferentes canales(Figura 2). - Abra el software de programación y asegúrese de que las carpetas de los paquetes de procesamiento de imágenes estén en la ruta (consulte tabla de materiales).
- Importe la serie T de calcio en el software de programación llamando a la función BioFormats en la ventana de comandos de la plataforma de programación, que abre automáticamente la ventana de selección de archivos.
- Defina lo que hay en cada canal ingresando el número deseado. En estos datos de ejemplo, Channel 1 answer=6 (cellular_signal), Channel 2 answer=1 (blood_plasma).
- Traza los datos como una película dentro del software de programación para facilitar la visualización llamando a la función de trama.
- Para eliminar la fluorescencia verde de la fluoresceína-dextrano que sangra en el canal RCaMP rojo, desmezcla los canales en el paquete de procesamiento de imágenes llamando a la función unmix_chs en la ventana de comandos de la plataforma de programación.
- Seleccione una región que solo contenga fluorescencia de este fluoróforo en el canal 1, como RCaMP en este caso.
- Seleccione una región que solo contenga fluorescencia del fluoróforo 2, como la fluoresceína en el plasma sanguíneo en este ejemplo.
- Seleccione un área de fondo que no tenga fluorescencia de ninguno de los fluoróforos. Esto genera una matriz de contribución espectral que se aplica a cada píxel en cada canal. Mejora significativamente la localización de la señal RCaMP, lo que mejorará la detección de eventos de calcio en estas estructuras.
NOTA: Como se mencionó anteriormente, hay varias formas en que los datos de imágenes de calcio se pueden analizar dentro de los paquetes de procesamiento de imágenes. Los pasos 4.1.16-4.1.23 describen el método para analizar los eventos de calcio de pericitos que envuelven utilizando ROI manuales seleccionados a mano. - Ejecute el análisis de señalización celular en la película de calcio sin mezclar llamando a la función CellScan en la ventana de comandos de la plataforma de programación.
- El código preguntará "¿Qué método de detección de ROI le gustaría usar?". Escriba el número 2 para cargar los ROI seleccionados a mano en la plataforma de programación.
- Cargue las regiones de interés de la carpeta zip que se seleccionaron de los pericitos a mano anteriormente (Paso 4.1.6).
- El código preguntará "¿Cuál es el factor de escala?". Determine el factor de escala para los ROI seleccionados a mano en relación con la serie de imágenes que se está analizando y escriba el número de la escala. En este ejemplo, el factor de escala es 1 porque no es necesario cambiar el tamaño de los ROI, ya que se seleccionaron en imágenes con 128x128 píxeles, la misma resolución que la película de calcio original.
- Genere gráficos de cada ROI y los rastros de calcio normalizados en diferentes colores(Figura 3A)llamando a las funciones de proceso y trazado en la ventana de comandos.
- Si el código no detecta la mayoría de los eventos de calcio en los seguimientos individuales, modifique los parámetros integrados dentro del cuadro de optimización de la configuración llamando a la función opt_config y ajustando los valores, como disminuir el umbral para los datos filtrados de paso corto a tres veces la desviación estándar del período de referencia, que son los primeros 30 fotogramas de la serie T.
- Seleccione el botón Procesar en el cuadro de optimización para aplicar los nuevos parámetros.
NOTA: Para detectar y clasificar las señales, la traza de calcio normalizada es de paso largo y paso de banda filtrada, lo que ayuda a suavizar los datos para las estimaciones de amplitud y anchura, pero también para determinar si las señales son picos individuales, picos múltiples o mesetas(Figura 3B). - Genere los datos como un archivo .csv que contiene información espacial sobre las regiones de interés y los picos que se identificaron llamando a la función output_data en la ventana de comandos. Asigne al archivo un nombre único para su posterior análisis en un programa de estadísticas.
NOTA: Los pasos 4.1.24-4.1.31 describen el método para analizar los eventos de calcio pericito de envainamiento mediante el análisis de ROI basados en la actividad. - Repita los pasos 4.1.8.-4.1.16 para importar la película de calcio, desmezclar los canales y llamar a la función CellScan en la plataforma de programación.
- El código preguntará "¿Qué método de detección de ROI le gustaría usar?". Escriba el número 6 para seleccionar la identificación automatizada de la región de interés en función de la actividad y el cambio en la fluorescencia en 3 dimensiones (x, y y tiempo; "Algoritmo FLIKA 3D").
- Trace los resultados procesados para ver las regiones de interés identificadas como diferentes colores llamando a las funciones de proceso y trazado en la ventana de comandos. Cada ROI se distingue en tiempo y espacio y se representa como una máscara sobreabreada (Figura 4).
- Si el algoritmo no detecta ROI que son claramente visibles a simple vista, modifique los parámetros incorporados dentro del cuadro de optimización llamando a la función opt_config y ajustando los valores, como aumentar el filtro gaussiano que suaviza los datos en el tiempo (en 2 s) y disminuir el umbral para encontrar ROI a 3 veces la desviación estándar de la línea de base.
- Seleccione el botón de proceso en el cuadro de optimización para aplicar los nuevos parámetros. Con el proceso de optimización se deben identificar más ROI(Figura 4B).
- Traza los ROI como una película para identificar claramente las áreas de actividad (delineadas en colores del arco iris) cambiando el modo del cuadro predeterminado a película dentro de la ventana de optimización para una mayor visualización.
- Genere los datos como un archivo csv llamando a la función output_data en la ventana de comandos. Este archivo se puede analizar más a fondo en un programa de estadísticas.
NOTA: Los parámetros de análisis se pueden ajustar para adaptarse a cualquier tipo de señal celular dinámica (calcio, relaciones FRET, etc.). Todos los pasos anteriores se pueden automatizar con un código de programación simple para procesar por lotes muchas películas de calcio con la misma configuración.
- Análisis de flujo sanguíneo de exploración de línea.
- Importe el archivo de datos kymograph de escaneo de línea adquirido en la sección 3.5 en el software de programación.
- El código preguntará "¿Qué se muestra en los canales 1 y 2?". Defina lo que hay en cada canal cuando se le solicite. En este ejemplo, el canal 1 está en blanco (tipo 0)y el canal 2 está blood_plasma (tipo 1).
- Ejecute la función de análisis de diámetro en el escaneo de línea llamando a la función LineScanDiam, que abre un cuadro para seleccionar el área que corresponde al diámetro en el kymograph(Figura 5B,izquierda).
- Dibuje una caja fuera de los límites de fluorescencia del kymograph que corresponda al diámetro del vaso.
- Procese esta clase de datos llamando a la función de proceso para medir el ancho total a la mitad de los máximos para el diámetro del recipiente y genere una gráfica (Figura 5C) con la función de gráfica.
- Genere los datos como un archivo csv llamando a la función output_data en la ventana de comandos. Este archivo se puede analizar más a fondo en un programa de estadísticas.
- Ejecute el análisis de transformación de radón de velocidad llamando a la función LineScanVel, que abre un cuadro para seleccionar el área que corresponde a la velocidad de RBC en el kymograph(Figura 5B,derecha).
- Dibuje una caja dentro del borde de la fluorescencia del kymograph que corresponda a la velocidad del vaso.
- Procese esta clase de datos llamando a la función de proceso para calcular la velocidad, el flujo y la densidad lineal de los glóbulos rojos desde el ángulo de las rayas en la fluorescencia. Genere una gráfica (Figura 5D) con la función de gráfica.
- Genere los datos como un archivo csv llamando a la función output_data en la ventana de comandos. Este archivo se puede analizar más a fondo en un programa de estadísticas.
NOTA: Los kymographs deben tener fluorescencia clara con aristas bien definidas entre los espacios negros para que el análisis de diámetro y velocidad sea preciso(Figura 5A,B). Es muy importante dibujar las líneas ortogonales y paralelas de manera precisa, de lo contrario no será posible un análisis confiable de los kymographs. Al igual que el análisis de calcio con los algoritmos de procesamiento de imágenes, se pueden optimizar los parámetros para los cálculos de diámetro y velocidad.
Resultados
La fluoresceína-dextrano tiene un amplio espectro de emisión que puede sangrar a través del canal rojo, lo que afecta la detección de RCaMP en pericitos de envainamiento(Figura 2A). La desmezcla espectral después de la adquisición de datos en el programa de software reduce el sangrado de fluoresceína(Figura 2B,más abajo), mejorando la detección de señales de calcio en los pasos de análisis posteriores.
El análisis de calcio con los algoritmos de procesamiento de imágenes utilizados en este protocolo permite varios enfoques diferentes para identificar el ROI y las fluctuaciones intracelulares del calcio (es decir, las señales de calcio). La selección manual de estructuras celulares permite la detección de fluctuaciones de calcio dentro de estas regiones(Figura 3A),incluidos diferentes tipos de picos de señal, como picos individuales y picos múltiples, después de que los rastros de calcio normalizados se filtran de paso bajo y paso de banda(Figura 3B). Además, los ROI se identifican agrupando píxeles activos donde la intensidad de la fluorescencia cambia con el tiempo utilizando algoritmos de procesamiento de imágenes desarrollados por Ellefsen et al. 201416 y Barrett et al. 201817 (Figura 4). Esto se puede aplicar a cualquier señal celular dinámica ajustando el tiempo, el umbral y los parámetros espaciales para abarcar el tamaño y la forma esperados de la señal. La disminución del umbral para la identificación de señales encuentra más regiones de interés (Figura 4B).
Se pueden analizar kymographs hemodinámicos brillantes y claros para medir el diámetro y la velocidad de los glóbulos rojos en los vasos sanguíneos cerca de los pericitos de envainamiento(Figura 5A,B). El diámetro se calcula a partir del ancho total a la mitad como máximo de la fluorescencia(Figura 5C). La velocidad de los glóbulos rojos se aproxima a partir de las rayas hechas de glóbulos rojos sin etiquetar, donde el ángulo se introduce en una transformación de radón para calcular la velocidad, el flujo (células/s) y la densidad lineal (células/mm; Figura 5D). Los kymographs de mala calidad donde hay saturación de fluorescencia, mala relación señal/ruido o movimiento del campo de imagen(Figura 6A)crean gráficos poco confiables con puntos de error (cruces rojas) donde no se pueden determinar los datos(Figura 6B,C). La calidad de los datos adquiridos es fundamental para un buen resultado y seguir los pasos descritos en este protocolo asegura buenos resultados.

Figura 1. Resumen del protocolo. El protocolo presenta los pasos para adquirir y analizar imágenes fluorescentes de calcio de pericitos que envuelven el cerebro y datos de flujo sanguíneo de vasos sanguíneos cercanos en ratones anestesiados. El protocolo se divide en 4 pasos. 1) Preparación del procedimiento: configuración del equipo y preparación del catéter; 2) Inyección de la vena de la cola; 3) Adquisición de datos por microscopía de dos fotones; 4) Análisis de datos con algoritmos de procesamiento de imágenes. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2. Desmezcla espectral de fluoróforos. A) Imagen media representativa de los vasos sanguíneos etiquetados con pericitos y fluoresceína-dextrano RCaMP de una adquisición de la serie T. Barra de escala = 10 μm.B) Superior: Cuando se consideran canales individuales, el sangrado desde el Canal 2 es evidente en el Canal 1 (izquierda). Inferior: Después de la desmezcla espectral, el sangrado se reduce y la señal de RCaMP es más prominente en la estructura del pericito. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. ROI seleccionados a mano y trazas de calcio optimizadas. A) Las regiones de interés seleccionadas en el software de procesamiento de imágenes utilizado (formas de arco iris) se pueden utilizar para identificar rastros de señales de calcio. B) Los picos de señal de trazas normalizadas se identifican mediante el paso bajo y el paso de banda filtrando los datos. Definimos el umbral de señal como 3 veces la desviación estándar del período basal (primeros 30 fotogramas) y cualquier pico por encima de este umbral se consideró una señal (traza inferior). Haga clic aquí para ver una versión más grande de esta figura.
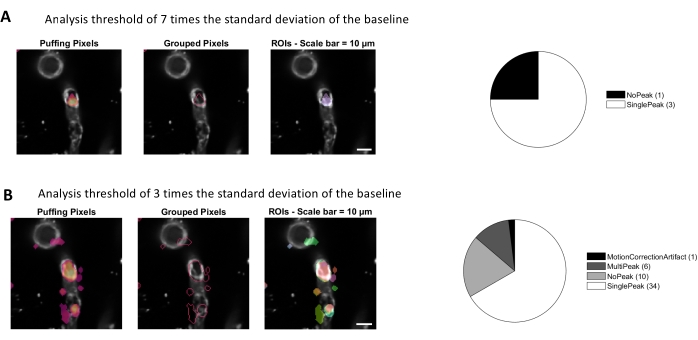
Figura 4. ROI automatizados basados en la actividad para el análisis de calcio. Los mismos datos se analizaron con un umbral de 7 veces la desviación estándar de la línea de base (A) y 3 veces la desviación estándar de la línea de base (B). Al disminuir el umbral para identificar píxeles activos, se encuentran más ROI (B) y picos de señal (gráfico circular) dentro de los pericitos. Haga clic aquí para ver una versión más grande de esta figura.
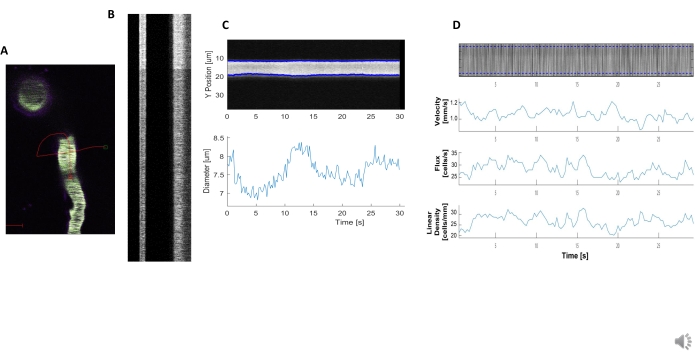
Figura 5. Mediciones hemodinámicas del kymógrafo. A) Ejemplo de escaneo de línea a través del buque. B) Ejemplo de kymographs bien definidos para diámetro (izquierda) y velocidad (derecha). Las rayas negras dentro de la banda derecha de fluorescencia corresponden a glóbulos rojos.C) Análisis de diámetro con claras fluctuaciones de vasomoción. D) Análisis de velocidad con gráficos para el eje Y = flujo de glóbulos rojos (celdas / s), densidad de línea (celdas / mm), velocidad (mm / s) y ángulo de raya (grado), relación señal a ruido (unidades arbitrarias, a.u.), eje X = tiempo (seg). Haga clic aquí para ver una versión más grande de esta figura.

Figura 6. Representación de mediciones hemodinámicas de mala calidad. A) Ejemplos de kymographs de mala calidad con saturación de fluorescencia, mala relación señal/ruido o movimiento del campo de imágenes durante la adquisición. B y C) Gráficos similares a la Figura 7 de datos de diámetro y velocidad que tienen puntos de error (puntos rojos) debido a la mala calidad de los kymographs. (imagen E, eje Y = Diámetro (μm), eje X = tiempo (seg); imagen F, eje Y = flujo de glóbulos rojos (celdas / s), densidad de línea (celdas / mm), velocidad (mm / s) y ángulo de raya (grado), relación señal a ruido (a.u.), eje X = tiempo (seg). Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El presente método proporciona detalles sobre la inyección de vena de cola de ratón con un catéter, adquisición de imágenes de microscopio de dos fotones para pilas de profundidad, películas de señalización de calcio celular, creación de kymographs hemodinámicos y análisis de calcio y hemodinámico con nuestros algoritmos de procesamiento de imágenes17 (Figura 1). Hay varias ventajas en estas técnicas que mejoran el resultado de las imágenes in vivo y reducen el tiempo, los recursos y el estrés animal durante la sesión. En primer lugar, el uso de un catéter para la inyección de la vena de la cola proporciona un mayor control sobre la aguja, la jeringa y la cantidad de sustancia inyectada en la circulación del ratón. Además, evita la inyección de tinte en el tejido de la cola, ahorrando costosos reactivos. En segundo lugar, utilizamos ratones transgénicos que expresan sensores de calcio codificados genéticamente en pericitos de envainamiento y demuestran cómo localizarlos dentro de la red vascular cerebral con una pila z de profundidad, lo que facilita la identificación celular y la reubicación en sesiones de imágenes posteriores a largo plazo. Este es un factor importante en los estudios de pericitos y asegura una clasificación celular adecuada6,7. En tercer lugar, proporcionamos nuestros parámetros para recopilar películas de calcio y datos de escaneo de líneas hemodinámicas que son un buen punto de partida para medir señales celulares dinámicas. Finalmente, presentamos nuestros algoritmos de procesamiento de imágenes17,una caja de herramientas integral de procesamiento de imágenes que contiene múltiples enfoques para el preprocesamiento de imágenes (como la desmezcla espectral), el análisis de imágenes de calcio y el análisis hemodinámico (diámetro, velocidad, etc.). Estos algoritmos pueden generar gráficos para una visualización rápida y fácil de los datos, al tiempo que minimizan el nivel de experiencia del usuario requerido para analizar los resultados. Además, se puede automatizar con unas pocas líneas de código para procesar rápidamente por lotes múltiples conjuntos de datos con los mismos parámetros. Esto puede mejorar potencialmente la visualización de datos y la inversión de tiempo del investigador.
La clave para recopilar buenos datos de imágenes de calcio es ajustar la potencia del láser y la configuración de PMT para una clara adquisición de señales de fluorescencia, pero también recopilar datos a una velocidad de fotogramas suficiente para capturar todo el evento de calcio. Los datos en este protocolo se adquirieron a 10-11 cuadros por segundo, lo que captura las oscilaciones de calcio más lentas en los pericitos de envainamiento. También hay varios pasos durante el análisis que pueden mejorar el resultado del análisis. En primer lugar, la desmezcla espectral es beneficiosa si existe una superposición significativa entre los espectros de emisión de fluoróforos (Figura 2). Fluoresceína-dextrano se utilizó en este protocolo porque es un conjugado de dextrano rentable y disponible comercialmente que se usa comúnmente para mediciones hemodinámicas5. La desmezcla espectral ayuda a limpiar los datos para una mejor detección de señales de calcio, pero también se podrían usar fluoróforos alternativos con espectros de emisión más estrechos. En segundo lugar, la selección manual de estructuras celulares como ROI(Figura 3)es útil para clasificar los eventos de calcio en diferentes regiones subcelulares, como el soma o las ramas de proceso. La selección del ROI basada en la actividad (Figura 4)16 proporciona más información espacial y temporal sobre los eventos individuales de calcio. Esto puede ser útil al determinar la frecuencia de eventos de calcio en un área determinada o la propagación de eventos a otras áreas celulares. El uso de software de programación para analizar datos de imágenes puede ahorrar a los investigadores horas de tiempo cuando los datos se procesan por lotes, pero requiere una inversión de tiempo inicial para ajustar los parámetros para obtener resultados óptimos. Los factores más importantes son el tamaño esperado (en μm2) de la región activa, así como la duración de la señal (se debe definir el tiempo mínimo de señal y el tiempo máximo de señal). Los investigadores deben examinar primero algunas películas de la serie T de ejemplo para determinar mejor qué parámetros se ajustan a sus datos. Finalmente, los datos de mala calidad adquiridos en el microscopio pueden dificultar en gran medida el análisis del calcio y la hemodinámica (Figura 6). Por lo tanto, se debe tener cuidado de optimizar la configuración de adquisición del microscopio al principio. Con estos factores en mente, este protocolo que se puede adaptar para adaptarse a imágenes de calcio o análisis de otras señales celulares dinámicas (por ejemplo, sodio fluorescente, potasio, metabolito o fluctuaciones de voltaje) en otros tejidos o tipos de células.
Hay varias limitaciones para este protocolo. Primero, los datos se recopilan bajo anestesia, lo que afecta la actividad cerebral y podría afectar el flujo sanguíneo. Se pueden hacer imágenes similares en ratones despiertos que están entrenados para aceptar la fijación de la cabeza para obtener resultados más fisiológicos. Además, es importante recordar que recopilamos imágenes en 2 dimensiones de una célula y un vaso sanguíneo de 3 dimensiones in vivo. Por lo tanto, solo podemos capturar una facción de los eventos de calcio dentro de estas células o el flujo sanguíneo en una sola sección de un vaso sanguíneo a la vez.
Otra limitación a tener en cuenta es que las imágenes de calcio de dos fotones son sensibles a los artefactos de movimiento, donde el movimiento dentro y fuera del plano focal puede confundirse con fluctuaciones de calcio. Este protocolo se realizó bajo anestesia, lo que limita el movimiento del animal; sin embargo, los artefactos de movimiento pueden ser introducidos por la frecuencia respiratoria del ratón, la frecuencia cardíaca, la posible hinchazón del tejido y, en el caso de pericitos de envainamiento, contracción de vasos o vasomoción 4,6,18,19. Los artefactos de movimiento pueden ser mitigados por varias estrategias. Los paquetes de procesamiento de imágenes utilizados en este protocolo incluyen un paso de corrección de movimiento opcional, que utiliza un motor de convolución 2D para alinear las imágenes dentro de la serie T basada en la vasculatura visible13,17. Los fotogramas con cambios significativos en el plano focal son identificados por este algoritmo y pueden ser excluidos del análisis. Además, es posible utilizar estrategias estadísticas dentro de los paquetes de procesamiento de imágenes, como una puntuación Z al generar los rastros de fluorescencia para normalizar el movimiento inducido por las fluctuaciones de calcio20. El enfoque más robusto para tener en cuenta los artefactos de movimiento en las imágenes de dos fotones es combinar la expresión de dos indicadores fluorescentes dentro de la misma célula, como un indicador de calcio (por ejemplo, GCaMP) y un reportero fluorescente (por ejemplo, mCherry) que es independiente del calcio. Las fluctuaciones en el reportero fluorescente se pueden atribuir al movimiento y se restan de la señal indicadora de calcio para normalizar los artefactos de movimiento.
El propósito de este protocolo es proporcionar una comprensión clara de cómo recopilar imágenes óptimas de calcio y datos de flujo sanguíneo in vivo y presentar nuevos métodos y herramientas de análisis que los investigadores pueden implementar para mejorar sus resultados. Estas técnicas se pueden aplicar para estudiar el papel de diferentes poblaciones de pericitos en el control del flujo sanguíneo o en diferentes estados de enfermedad cerebral. Estos parámetros de imagen también se pueden utilizar para estudiar el calcio y el flujo sanguíneo en otros tipos de células y sistemas de órganos y principios similares se aplican a otras técnicas de imagen dinámica que son posibles gracias a otros sensores codificados genéticamente, más allá del calcio.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
J. Meza cuenta con el apoyo de becas de Mitacs e Research Manitoba. El financiamiento para este trabajo fue proporcionado por los Institutos Canadienses para la Investigación de la Salud, Research Manitoba, manitoba Medical Service Foundation, fondos iniciales de la Universidad de Manitoba y Brain Canada a través del Fondo de Investigación del Cerebro de Canadá, con el apoyo financiero de Health Canada y la Fundación Azrieli. Las opiniones expresadas en el presente documento no representan necesariamente las opiniones del Ministro de Salud o del Gobierno del Canadá.
Materiales
| Name | Company | Catalog Number | Comments |
| Acta2-RCaMP1.07 | The Jackson Laboratory | 28345 | In the video protocol the animal model used is a female mouse of 10 months, 1 day old. |
| Applicators (Regular) | Bisco | X-80250P | |
| BioFormats package for MATLAB | NA | NA | Denominated in this protocol as "image processing packages". Available in: https://docs.openmicroscopy.org/bio-formats/ |
| CHIPS MATLAB toolbox | NA | NA | Denomitaded in this protocol as "image processing algorithms". Barrett MJP, Ferrari KD, Stobart JL, Holub M, Weber B. CHIPS: an Extensible Toolbox for Cellular and Hemodynamic Two-Photon Image Analysis. Neuroinformatics. 2018;16(1):145-147. doi:10.1007/s12021-017-9344-y. Available in: https://github.com/EIN-lab/CHIPS |
| Clear Ultrasound Gel, Medium viscosity | HealthCare Plus | UGC250 | |
| Dextran, fluorescein, 70,000 MW, anionic | Thermo Fisher Scientific | D1823 | |
| Dextran, Texas Red, 70,000 MW, neutral | Thermo Fisher Scientific | D1830 | |
| Eye Lube Plus | Optixcare | NA | |
| FIJI | Image J | NA | Denominated in this protocol as "image processor software". Available in: https://imagej.net/Fiji/Downloads |
| GCaMP6sfl/fl | The Jackson Laboratory | ||
| Head Post fixing platform | University of Zurich | NA | |
| Ketamine (Narketan 100 mg/mL) | Vetoquinol | 440893 | |
| MATLAB R2020b | NA | Denominated in this protocol as "programming platform ". Available in: https://www.mathworks.com/downloads/ | |
| Needle 0.3mmx25mm | BD PrecisionGlide | 305128 | |
| Objective XLUMPLFLN20XW | Olympus | NA | https://www.olympus-lifescience.com/en/objectives/lumplfln-w/ |
| PDGFRβ-CreERT2 | The Jackson Laboratory | 30201 | |
| Polyethylene Tubing, PE10 I.D. 28mm (0.11”) O.D. 61mm (.024”) | BD Intramedic | 427401 | |
| Prairie View | Bruker Fluorescence Microscopy | NA | https://www.bruker.com/en/products-and-solutions/fluorescence-microscopy/multiphoton-microscopes/ultima-in-vitro.html |
| Ultima In Vitro Multiphoton Microscope | Bruker Fluorescence Microscopy | NA | https://www.bruker.com/en/products-and-solutions/fluorescence-microscopy/multiphoton-microscopes/ultima-in-vitro.html |
| Under Tank Heater | Reptitherm U.T.H | E169064 | |
| Xylazine (Rompun 20 mg/mL) | Bayer HealthCare | 2169592 |
Referencias
- Armulik, A., Genové, G., Betsholtz, C. Pericytes: Developmental, physiological, and pathological perspectives, problems, and promises. Developmental Cell. 21 (2), 193-215 (2011).
- Armulik, A., et al. Pericytes regulate the blood-brain barrier. Nature. 468 (7323), 557-561 (2010).
- Berthiaume, A. -A., et al. Dynamic remodeling of pericytes in vivo maintains capillary coverage in the adult mouse brain. Cell Reports. 22 (1), 8-16 (2018).
- Rungta, R. L., Chaigneau, E., Osmanski, B. -F. F., Charpak, S. Vascular compartmentalization of functional hyperemia from the synapse to the pia. Neuron. 99 (2), 362-375 (2018).
- Shen, Z., Lu, Z., Chhatbar, P. Y., O'Herron, P., Kara, P. An artery-specific fluorescent dye for studying neurovascular coupling. Nature Methods. 9 (3), 273-276 (2012).
- Gonzales, A. L., et al. Contractile pericytes determine the direction of blood flow at capillary junctions. Proceedings of the National Academy of Sciences of the United States of America. 117 (43), 27022-27033 (2020).
- Hartmann, D. a, et al. Pericyte structure and distribution in the cerebral cortex revealed by high-resolution imaging of transgenic mice. Neurophotonics. 2 (4), 041402(2015).
- Grant, R. I., et al. Organizational hierarchy and structural diversity of microvascular pericytes in adult mouse cortex. Journal of Cerebral Blood Flow & Metabolism. 39 (3), 411-425 (2017).
- Hill, R. A., Tong, L., Yuan, P., Murikinati, S., Gupta, S., Grutzendler, J. Regional blood flow in the normal and ischemic brain is controlled by arteriolar smooth muscle cell contractility and not by capillary pericytes. Neuron. 87 (1), 95-110 (2015).
- 28345 - STOCK Tg(RP23-370F21-RCaMP1.07)B3-3Mik/J. Jackson Laboratory. , Available from: https://www.jax.org/strain/028345 (2021).
- Ohkura, M., Sasaki, T., Kobayashi, C., Ikegaya, Y., Nakai, J. An improved genetically encoded red fluorescent Ca2+ indicator for detecting optically evoked action potentials. PLoS ONE. 7 (7), (2012).
- Stobart, J. L., et al. Long-term in vivo calcium imaging of astrocytes reveals distinct cellular compartment responses to sensory stimulation. Cerebral Cortex. 28 (1), 184-198 (2018).
- Stobart, J. L., et al. Cortical circuit activity evokes rapid astrocyte calcium signals on a similar timescale to neurons. Neuron. 98 (4), 726-735 (2018).
- Mostany, R., Portera-Cailliau, C. A craniotomy surgery procedure for chronic brain imaging. JoVE. (12), e680(2008).
- Lin, X., et al. Imaging neural activity in the primary somatosensory cortex using Thy1-GCaMP6s transgenic mice. JoVE. (143), e56297(2019).
- Ellefsen, K. L., Settle, B., Parker, I., Smith, I. F. An algorithm for automated detection, localization and measurement of local calcium signals from camera-based imaging. Cell Calcium. 56 (3), 147-156 (2014).
- Barrett, M. J. P., Ferrari, K. D., Stobart, J. L., Holub, M., Weber, B. CHIPS: an extensible toolbox for cellular and hemodynamic two-photon image analysis. Neuroinformatics. 16, 145-147 (2018).
- Hall, C. N., et al. Capillary pericytes regulate cerebral blood flow in health and disease. Nature. 508 (1), 55-60 (2014).
- Nilsson, H., Aalkjaer, C. Vasomotion: mechanisms and physiological importance. Molecular interventions. 3 (2), 79-89 (2003).
- Rungta, R. L., et al. Vascular arbors in layer II / III somatosensory cortex. Communications Biology. , (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados