Method Article
Análisis de transcriptoma de alto rendimiento para investigar las interacciones huésped-patógeno
En este artículo
Resumen
El protocolo presentado aquí describe una tubería completa para analizar los datos del transcriptoma de secuenciación de ARN desde lecturas sin procesar hasta análisis funcionales, incluidos los pasos de control de calidad y preprocesamiento para enfoques analíticos estadísticos avanzados.
Resumen
Los patógenos pueden causar una amplia variedad de enfermedades infecciosas. Los procesos biológicos inducidos por el huésped en respuesta a la infección determinan la gravedad de la enfermedad. Para estudiar tales procesos, los investigadores pueden utilizar técnicas de secuenciación de alto rendimiento (RNA-seq) que miden los cambios dinámicos del transcriptoma del huésped en diferentes etapas de la infección, los resultados clínicos o la gravedad de la enfermedad. Esta investigación puede conducir a una mejor comprensión de las enfermedades, así como a descubrir posibles objetivos farmacológicos y tratamientos. El protocolo presentado aquí describe una tubería completa para analizar los datos de secuenciación de ARN desde lecturas sin procesar hasta análisis funcionales. La tubería se divide en cinco pasos: (1) control de calidad de los datos; (2) mapeo y anotación de genes; (3) análisis estadístico para identificar genes expresados diferencialmente y genes coexpresados; (4) determinación del grado molecular de la perturbación de las muestras; y (5) análisis funcional. El paso 1 elimina los artefactos técnicos que pueden afectar a la calidad de los análisis posteriores. En el paso 2, los genes se mapean y anotan de acuerdo con los protocolos de biblioteca estándar. El análisis estadístico en el paso 3 identifica genes que se expresan diferencialmente o coexpresan en muestras infectadas, en comparación con las no infectadas. La variabilidad de la muestra y la presencia de posibles valores biológicos atípicos se verifican utilizando el enfoque de grado molecular de perturbación en el paso 4. Finalmente, el análisis funcional en el paso 5 revela las vías asociadas con el fenotipo de la enfermedad. La tubería presentada tiene como objetivo apoyar a los investigadores a través del análisis de datos de ARN-seq de estudios de interacción huésped-patógeno e impulsar futuros experimentos in vitro o in vivo , que son esenciales para comprender el mecanismo molecular de las infecciones.
Introducción
Los arbovirus, como el dengue, la fiebre amarilla, el chikungunya y el zika, se han asociado ampliamente con varios brotes endémicos y se han convertido en uno de los principales patógenos responsables de infectar a los humanos en las últimas décadas1,2. Las personas infectadas con el virus chikungunya (CHIKV) a menudo tienen fiebre, dolor de cabeza, erupción cutánea, poliartralgia y artritis3,4,5. Los virus pueden subvertir la expresión génica de la célula e influir en varias vías de señalización del huésped. Recientemente, los estudios de transcriptoma sanguíneo utilizaron RNA-seq para identificar los genes expresados diferencialmente (DEG) asociados con la infección aguda por CHIKV en comparación con la convalecencia6 o los controles sanos7. Los niños infectados con CHIKV tenían genes regulados al alza que están involucrados en la inmunidad innata, como los relacionados con los sensores celulares para el ARN viral, la señalización JAK/STAT y las vías de señalización del receptor tipo toll6. Los adultos infectados agudamente con CHIKV también mostraron inducción de genes relacionados con la inmunidad innata, como los relacionados con los monocitos y la activación de las células dendríticas, y con las respuestas antivirales7. Las vías de señalización enriquecidas con genes regulados a la baja incluyeron las relacionadas con la inmunidad adaptativa, como la activación y diferenciación y enriquecimiento de células T en células T y B7.
Se pueden utilizar varios métodos para analizar los datos del transcriptoma de los genes huésped y patógeno. A menudo, la preparación de la biblioteca RNA-seq comienza con el enriquecimiento de transcripciones maduras de poli-A. Este paso elimina la mayor parte del ARN ribosómico (ARNr) y, en algunos de los casos, los ARN virales/bacterianos. Sin embargo, cuando la cuestión biológica involucra la detección de la transcripción del patógeno y el ARN se secuencia independientemente de la selección anterior, se podrían detectar muchas otras transcripciones diferentes mediante secuenciación. Por ejemplo, se ha demostrado que los ARNm subgenómicos son un factor importante para verificar la gravedad de las enfermedades8. Además, para ciertos virus como CHIKV y SARS-CoV-2, incluso las bibliotecas enriquecidas con poli-A generan lecturas virales que se pueden utilizar en análisis posteriores9,10. Cuando se centran en el análisis del transcriptoma del huésped, los investigadores pueden investigar la perturbación biológica a través de las muestras, identificar genes expresados diferencialmente y vías enriquecidas, y generar módulos de coexpresión7,11,12. Este protocolo destaca los análisis de transcriptomas de pacientes infectados por CHIKV e individuos sanos utilizando diferentes enfoques bioinformáticos (Figura 1A). Se utilizaron datos de un estudio publicado previamente7 que consta de 20 individuos sanos y 39 infectados agudamente por CHIKV para generar los resultados representativos.
Protocolo
Las muestras utilizadas en este protocolo fueron aprobadas por los comités de ética tanto del Departamento de Microbiología del Instituto de Ciencias Biomédicas de la Universidad de São Paulo como de la Universidad Federal de Sergipe (Protocolos: 54937216.5.0000.5467 y 54835916.2.0000.5546, respectivamente).
1. Instalación de escritorio de Docker
NOTA: Los pasos para preparar el entorno de Docker son diferentes entre los sistemas operativos (SO). Por lo tanto, los usuarios de Mac deben seguir los pasos enumerados como 1.1, los usuarios de Linux deben seguir los pasos enumerados como 1.2 y los usuarios de Windows deben seguir los pasos enumerados como 1.3.
- Instalar en MacOS.
- Acceda al sitio web de Get Docker (Tabla de materiales), haga clic en Docker Desktop para Mac y luego haga clic en el enlace Descargar desde Docker Hub .
- Descargue el archivo de instalación haciendo clic en el botón Obtener Docker .
- Ejecute el archivo Docker.dmg para abrir el instalador y, a continuación, arrastre el icono a la carpeta Aplicaciones . Localice y ejecute el Docker.app en la carpeta Aplicaciones para iniciar el programa.
NOTA: El menú específico del software en la barra de estado superior indica que el software se está ejecutando y que es accesible desde un terminal.
- Instale el programa contenedor en el sistema operativo Linux.
- Acceda al sitio web Get Docker Linux (Tabla de materiales) y siga las instrucciones para la instalación mediante la sección repositorio disponible en el enlace Repositorio de Docker Linux .
- Actualice todos los paquetes de Linux mediante la línea de comandos:
sudo apt-get update - Instale los paquetes necesarios en Docker:
sudo apt-get install apt-transport-https ca-certificates curl gnupg lsb-release - Cree un archivo de llavero de archivo de software:
curl -fsSL https://download.docker.com/linux/ubuntu/gpg | sudo gpg --dearmor -o /usr/share/keyrings/docker-archive-keyring.gpg - Agregue la información de docker deb en el archivo source.list:
echo "deb [arch=amd64 signed-by=/usr/share/keyrings/docker-archive-keyring.gpg] https://download.docker.com/linux/ubuntu $(lsb_release -cs) stable" | sudo tee /etc/apt/sources.list.d/docker.list > /dev/null - Actualice todos los paquetes de nuevo, incluidos los que se agregaron recientemente:
sudo apt-get update - Instale la versión de escritorio:
sudo apt-get install docker-ce docker-ce-cli containerd.io - Seleccione el área geográfica y la zona horaria para finalizar el proceso de instalación.
- Instale el programa contenedor en el sistema operativo Windows.
- Acceda al sitio web de Get Docker (Tabla de materiales) y haga clic en Comenzar. Busque el instalador de Docker Desktop para Windows. Descargue los archivos e instálelos localmente en el equipo.
- Después de la descarga, inicie el archivo de instalación (.exe) y mantenga los parámetros predeterminados. Asegúrese de que las dos opciones Instalar componentes de Windows necesarios para WSL 2 y Agregar acceso directo al escritorio estén marcadas.
NOTA: En algunos casos, cuando este software intenta iniciar el servicio, muestra un error: la instalación de WSL está incompleta. Para averiguar este error, acceda al sitio web WSL2-Kernel (Tabla de materiales). - Descargue e instale el último kernel de Linux WSL2.
- Acceda al terminal de PowerShell como administrador y ejecute el comando:
dism.exe /online /enable-feature /featurename:Microsoft-Windows-Subsystem-Linux /all /norestart - Asegúrese de que el software Docker Desktop se haya instalado correctamente.
- Descargue la imagen del repositorio CSBL en el hub de Docker (Tabla de materiales).
- Abra el escritorio de Docker y verifique que el estado se esté "ejecutando" en la parte inferior izquierda de la barra de herramientas.
- Vaya a la línea de comandos del terminal de Windows PowerShell. Descargue la imagen de contenedor de Linux para este protocolo desde el repositorio CSBL en el concentrador de Docker. Ejecute el siguiente comando para descargar la imagen:
docker pull csblusp/transcriptome
NOTA: Después de descargar la imagen, el archivo se puede ver en el escritorio de Docker. Para crear el contenedor, los usuarios de Windows deben seguir el paso 1.5, mientras que los usuarios de Linux deben seguir el paso 1.6.
- Inicialice el contenedor del servidor en el sistema operativo Windows.
- Vea el archivo de imagen de Docker en el Administrador de aplicaciones de escritorio desde la barra de herramientas y acceda a la página Imágenes.
NOTA: Si la imagen de la canalización se descargó correctamente, habrá una imagen csblusp/transcriptome disponible. - Inicie el contenedor desde la imagen csblusp/transcriptome haciendo clic en el botón Ejecutar . Expanda la Configuración opcional para configurar el contenedor.
- Defina el nombre del contenedor (por ejemplo, servidor).
- Asocie una carpeta en el equipo local con la carpeta dentro de la ventana acoplable. Para ello, determine la ruta de acceso del host. Establezca una carpeta en la máquina local para almacenar los datos procesados que se descargarán al final. Establezca la ruta del contenedor. Defina y vincule la carpeta contenedora csblusp/transcriptome a la ruta de acceso del equipo local (utilice el nombre "/opt/transferdata" para la ruta del contenedor).
- Después de esto, haga clic en Ejecutar para crear el contenedor csblusp/transcriptome.
- Para acceder al terminal Linux desde el contenedor csblusp/transcriptome, haga clic en el botón CLI.
- Escriba el terminal bash para tener una mejor experiencia. Para ello, ejecute el comando:
juerga - Después de ejecutar el comando bash, asegúrese de que el terminal muestre (root@:/#):
root@ac12c583b731:/ #
- Vea el archivo de imagen de Docker en el Administrador de aplicaciones de escritorio desde la barra de herramientas y acceda a la página Imágenes.
- Inicialice el contenedor del servidor para el sistema operativo Linux.
- Ejecute este comando para crear el contenedor de Docker basado en la imagen:
docker run -d -it --rm --name server -v :/opt/transferdata csblusp/transcriptome
NOTA: : defina una ruta de acceso de la máquina de carpetas locales. - Ejecute este comando para acceder al terminal de comandos del contenedor docker:
docker exec -it server bash - Garantice la disponibilidad de un terminal Linux para ejecutar cualquier programa/script utilizando la línea de comandos.
- Después de ejecutar el comando bash, asegúrese de que el terminal muestre (root@:/#):
root@ac12c583b731:/ #
NOTA: La contraseña raíz es "transcriptome" de forma predeterminada. Si lo desea, la contraseña raíz se puede cambiar ejecutando el comando:
passwd - Primero, ejecute el comando source en addpath.sh para asegurarse de que todas las herramientas estén disponibles. Ejecute el comando:
source /opt/addpath.sh
- Ejecute este comando para crear el contenedor de Docker basado en la imagen:
- Compruebe la estructura de la carpeta de secuenciación de ARN.
- Acceda a la carpeta de scripts de canalización de transcriptoma y asegúrese de que todos los datos de la secuenciación de ARN se almacenen dentro de la carpeta: /home/transcriptome-pipeline/data.
- Asegúrese de que todos los resultados obtenidos del análisis se almacenen dentro de la carpeta de la ruta /home/transcriptome-pipeline/results.
- Asegúrese de que los archivos de referencia de genoma y anotación se almacenen dentro de la carpeta de la ruta /home/transcriptome-pipeline/datasets. Estos archivos ayudarán a soportar todos los análisis.
- Asegúrese de que todos los scripts estén almacenados en la carpeta de la ruta /home/transcriptome-pipeline/scripts y separados por cada paso como se describe a continuación.
- Descarga la anotación y el genoma humano.
- Acceda a la carpeta de scripts:
cd /home/transcriptome-pipeline/scripts - Ejecute este comando para descargar el genoma humano de referencia:
downloadGenome.sh bash - Para descargar la anotación, ejecute el comando:
downloadAnnotation.sh bash
- Acceda a la carpeta de scripts:
- Cambiar la anotación o la versión del genoma de referencia.
- Abra downloadAnnotation.sh y downloadGenome.sh para cambiar la URL de cada archivo.
- Copie los archivos downloadAnnotation.sh y downloadGenome.sh en el área de transferencia y edítelos en el sistema operativo local.
cd /home/transcriptome-pipeline/scripts
cp downloadAnnotation.sh downloadGenome.sh /opt/transferdata - Abra la carpeta Ruta de acceso del host , que está seleccionada para vincular entre el host y el contenedor de Docker en el paso 1.5.4.
- Edite los archivos utilizando el software de edición preferido y guárdelos. Finalmente, coloque los archivos modificados en la carpeta de script. Ejecute el comando:
cd /opt/transferdata
cp downloadAnnotation.sh downloadGenome.sh /home/transcriptome-pipeline/scripts
NOTA: Estos archivos se pueden editar directamente utilizando vim o nano Linux editor.
- A continuación, configure la herramienta fastq-dump con la línea de comandos:
vdb-config --interactivo
NOTA: Esto permite descargar archivos de secuenciación de los datos de ejemplo.- Navegue por la página Herramientas con la tecla de tabulación y seleccione la opción de carpeta actual. Navegue hasta la opción Guardar y haga clic en Aceptar. A continuación, salga de la herramienta fastq-dump.
- Inicie la descarga de las lecturas del artículo publicado anteriormente7. Se requiere el número de acceso SRA de cada muestra. Obtenga los números SRA del sitio web de SRA NCBI (Tabla de Materiales).
NOTA: Para analizar los datos de RNA-Seq disponibles en bases de datos públicas, siga el paso 1.12. Para analizar los datos privados de RNA-seq, siga el paso 1.13. - Analizar datos públicos específicos.
- Acceda al sitio web del Centro Nacional de Información Biotecnológica (NCBI) y busque palabras clave para un tema específico.
- Haga clic en el enlace Resultado de BioProject en la sección Genomas .
- Elija y haga clic en un estudio específico. Haga clic en los experimentos SRA. Se abre una nueva página, que muestra todas las muestras disponibles para este estudio.
- Haga clic en el número de acceso "Enviar a:" anterior. En la opción "Elegir destino", seleccione la opción Archivo y formato , seleccione RunInfo. Haga clic en "Crear archivo" para exportar toda la información de la biblioteca.
- Guarde el archivo SraRunInfo.csv en la ruta de acceso host definida en el paso 1.5.4 y ejecute el script de descarga:
cp /opt/transferdata/SraRunInfo.csv /home/transcriptome-pipeline/data
cd /home/transcriptome-pipeline/scripts
downloadAllLibraries.sh bash
- Analice datos de secuenciación privados y no publicados.
- Organice los datos de secuenciación en una carpeta denominada Lecturas.
NOTA: Dentro de la carpeta Lecturas , cree una carpeta para cada ejemplo. Estas carpetas deben tener el mismo nombre para cada ejemplo. Agregue datos de cada muestra dentro de su directorio. En caso de que se trate de un RNA-Seq de extremo pareado, cada directorio de muestra debe contener dos archivos FASTQ, que deben presentar nombres que terminen de acuerdo con los patrones {sample}_1.fastq.gz y {sample}_2.fastq.gz, secuencias directa e inversa, respectivamente. Por ejemplo, un ejemplo denominado "Healthy_control" debe tener un directorio con el mismo nombre y archivos FASTQ denominados Healthy_control_1.fastq.gz y Healthy_control_2.fastq.gz. Sin embargo, si la secuenciación de la biblioteca es una estrategia de un solo extremo, solo se debe guardar un archivo de lectura para el análisis posterior. Por ejemplo, la misma muestra, "Control saludable", debe tener un archivo FASTQ único denominado Healthy_control.fastq.gz. - Cree un archivo fenotípico que contenga todos los nombres de ejemplo: asigne a la primera columna el nombre 'Muestra' y a la segunda columna como 'Clase'. Rellene la columna Muestra con nombres de muestra, que deben ser el mismo nombre para los directorios de muestra y llene la columna Clase con el grupo fenotípico de cada muestra (por ejemplo, control o infectado). Finalmente, guarde un archivo con el nombre "metadata.tsv" y envíelo al directorio /home/transcriptome-pipeline/data/. Consulte el metadata.tsv existente para comprender el formato del archivo fenotípico.
cp /opt/transferdata/metadata.tsv
/home/transcriptome-pipeline/data/metadata.tsv - Acceda al directorio Host Path definido en el paso 1.5.4 y copie los nuevos ejemplos de directorios estructurados. Por último, mueva los ejemplos de /opt/transferdata al directorio de datos de canalización.
cp -rf /opt/transferdata/reads/*
/home/transcriptome-pipeline/data/reads/
- Organice los datos de secuenciación en una carpeta denominada Lecturas.
- Observe que todas las lecturas se almacenan en la carpeta /home/transcriptome-pipeline/data/reads.
2. Control de calidad de los datos
NOTA: Evalúe, gráficamente, la probabilidad de errores en las lecturas de secuenciación. Elimine todas las secuencias técnicas, por ejemplo, adaptadores.
- Acceda a la calidad de secuenciación de las bibliotecas con la herramienta FastQC.
- Para generar los gráficos de calidad, ejecute el programa fastqc. Ejecute el comando:
FastQC.sh bash
NOTA: Los resultados se guardarán en la carpeta /home/transcriptome-pipeline/results/FastQC. Dado que los adaptadores de secuencia se utilizan para la preparación y secuenciación de bibliotecas, en algunos casos los fragmentos de secuencia de adaptadores pueden interferir con el proceso de mapeo.
- Para generar los gráficos de calidad, ejecute el programa fastqc. Ejecute el comando:
- Elimine la secuencia del adaptador y las lecturas de baja calidad. Acceda a la carpeta Scripts y ejecute el comando de la herramienta Trimmomatic:
cd /home/transcriptome-pipeline/scripts
trimmomatic.sh bash
NOTA: Los parámetros utilizados para el filtro de secuenciación son: Quitar plomo de baja calidad o 3 bases (por debajo de la calidad 3) (LEADING:3); Retire el arrastre de baja calidad o 3 bases (por debajo de la calidad 3) (TRAILING: 3); Escanee la lectura con una ventana deslizante de 4 bases de ancho, cortando cuando la calidad promedio por base caiga por debajo de 20 (SLIDINGWINDOW: 4: 20); y Drop se lee por debajo de las 36 bases de largo (MINLEN:36). Estos parámetros podrían modificarse editando el archivo de script Trimmomatic.- Asegúrese de que los resultados se guardan en la siguiente carpeta: /home/transcriptome-pipeline/results/trimreads. Ejecute el comando:
ls /home/transcriptome-pipeline/results/trimreads
- Asegúrese de que los resultados se guardan en la siguiente carpeta: /home/transcriptome-pipeline/results/trimreads. Ejecute el comando:
3. Cartografía y anotación de muestras
NOTA: Después de obtener las lecturas de buena calidad, estas deben ser mapeadas al genoma de referencia. Para este paso, se utilizó el asignador STAR para asignar los ejemplos de ejemplo. La herramienta STAR mapper requiere 32 GB de memoria RAM para cargar y ejecutar las lecturas y el mapeo del genoma. Para los usuarios que no tienen 32 GB de memoria RAM, se pueden usar lecturas ya asignadas. En tales casos, vaya al paso 3.3 o use el mapeador Bowtie2. Esta sección tiene scripts para STAR (resultados que se muestran en todas las figuras) y Bowtie2 (mapeador de baja memoria requerida).
- Primer índice del genoma de referencia para el proceso de mapeo:
- Acceda a la carpeta Scripts mediante la línea de comandos:
cd /home/transcriptome-pipeline/scripts - Para el asignador STAR, ejecute:
indexGenome.sh bash - Para Bowtie mapper, ejecute:
indexGenomeBowtie2.sh bash
- Acceda a la carpeta Scripts mediante la línea de comandos:
- Ejecute el siguiente comando para asignar lecturas filtradas (obtenidas del paso 2) al genoma de referencia (versión GRCh38). Los mapeadores STAR y Bowtie2 se realizan utilizando parámetros predeterminados.
- Para el asignador STAR, ejecute:
mapSTAR.sh bash - Para el mapeador Bowtie2, ejecute:
mapBowtie2.sh bash
NOTA: Los resultados finales son archivos de mapa de alineación binaria (BAM) para cada muestra almacenada en /home/transcriptome-pipeline/results/mapreads.
- Para el asignador STAR, ejecute:
- Anote lecturas asignadas mediante la herramienta FeatureCounts para obtener recuentos sin procesar para cada gen. Ejecute los scripts que anotan las lecturas.
NOTA: La herramienta FeatureCounts es responsable de asignar lecturas de secuenciación asignadas a las entidades genómicas. Los aspectos más importantes de la anotación del genoma que se pueden cambiar después de la cuestión biológica incluyen, detección de isoformas, múltiples lecturas mapeadas y uniones exón-exón, correspondientes a los parámetros, GTF.attrType="gene_name" para el gen o no especificar los parámetros para el nivel de meta-característica, allowMultiOverlap=TRUE y juncCounts=TRUE, respectivamente.- Acceda a la carpeta de scripts mediante la línea de comandos:
cd /home/transcriptome-pipeline/scripts - Para anotar las lecturas asignadas para obtener recuentos sin procesar por gen, ejecute la línea de comandos:
Anotación Rscript. R
NOTA: Los parámetros utilizados para el proceso de anotación fueron: nombre corto del gen de retorno (GTF.attrType="gene_name"); permitir múltiples superposiciones (allowMultiOverlap = TRUE); e indique que la biblioteca está emparejada al final (isPairedEnd=TRUE). Para la estrategia de un solo extremo, utilice el parámetro isPairedEnd=FALSE. Los resultados se guardarán en la carpeta /home/transcriptome-pipeline/countreads.
- Acceda a la carpeta de scripts mediante la línea de comandos:
- Normalizar la expresión génica.
NOTA: Normalizar la expresión génica es esencial para comparar los resultados entre los resultados (por ejemplo, muestras sanas e infectadas). También se requiere la normalización para realizar la coexpresión y el grado molecular de los análisis de perturbación.- Acceda a la carpeta Scripts mediante la línea de comandos:
cd /home/transcriptome-pipeline/scripts - Normalizar la expresión génica. Para ello, ejecute la línea de comandos:
Rscript normalizamuestras. R
NOTA: La expresión de recuentos brutos, en este experimento, se normalizó utilizando los métodos Trimmed Mean of M-values (TMM) y Count Per Million (CPM). Este paso tiene como objetivo eliminar las diferencias en la expresión génica debido a la influencia técnica, haciendo la normalización del tamaño de la biblioteca. Los resultados se guardarán en la carpeta /home/transcriptome-pipeline/countreads.
- Acceda a la carpeta Scripts mediante la línea de comandos:
4. Genes expresados diferencialmente y genes coexpresados
- Identifique genes expresados diferencialmente utilizando el paquete EdgeR de código abierto. Esto implica encontrar genes cuya expresión es mayor o menor en comparación con el control.
- Acceda a la carpeta Scripts mediante la línea de comandos:
cd /home/transcriptome-pipeline/scripts - Para identificar el gen expresado diferencialmente, ejecute el script DEG_edgeR R utilizando la línea de comandos:
Rscript DEG_edgeR.R
NOTA: Los resultados que contienen los genes expresados diferencialmente se guardarán en la carpeta /home/transcriptome-pipeline/results/degs. Los datos se pueden transferir a una computadora personal.
- Acceda a la carpeta Scripts mediante la línea de comandos:
- Descargue los datos del contenedor csblusp/transcriptome.
- Transfiera los datos procesados desde /home/transcriptome-pipeline a la carpeta /opt/transferdata (equipo local).
- Copie todos los archivos en el equipo local ejecutando la línea de comandos:
cp -rf /home/transcriptome-pipeline/results /opt/transferdata/pipeline
cp -rf /home/transcriptome-pipeline/data /opt/transferdata/pipeline
Nota : ahora, vaya al equipo local para asegurarse de que todos los resultados, conjuntos de datos y datos están disponibles para descargar en la ruta de acceso del host.
- Identificar módulos de coexpresión.
- Acceda al sitio web de la Herramienta de identificación de módulos de coexpresión (CEMiTool) (Tabla de
Materiales). Esta herramienta identifica los módulos de coexpresión de los datasets de expresiones proporcionados por los usuarios. En la página principal, haga clic en Ejecutar en la parte superior derecha. Esto abrirá una nueva página para cargar el archivo de expresión. - Haga clic en Elegir archivo debajo de la sección Archivo de expresión y cargue la matriz de expresión génica normalizada 'tmm_expression.tsv' desde la ruta del host.
NOTA: Paso 4.4. no es obligatorio.
- Acceda al sitio web de la Herramienta de identificación de módulos de coexpresión (CEMiTool) (Tabla de
- Explore el significado biológico de los módulos de coexpresión.
- Haga clic en Elegir archivo en la sección Fenotipos de muestra y cargue el archivo con fenotipos de muestra metadata_cemitool.tsv desde el paso Descargar datos 4.2.2. para realizar un análisis de enriquecimiento de conjuntos de genes (GSEA).
- Presione Elegir archivo en la sección Interacciones genéticas para cargar un archivo con interacciones genéticas (cemitool-interactions.tsv). Es posible utilizar el archivo de interacciones genéticas proporcionado como ejemplo por webCEMiTool. Las interacciones pueden ser interacciones proteína-proteína, factores de transcripción y sus genes transcritos, o vías metabólicas. Este paso produce una red de interacción para cada módulo de coexpresión.
- Haga clic en la sección Elegir archivo en la sección Conjuntos de genes para cargar una lista de genes funcionalmente relacionados en un archivo de formato Gene Matrix Transposed (GMT). El archivo Gene Set permite a la herramienta realizar análisis de enriquecimiento para cada módulo de coexpresión, es decir, un análisis de sobrerrepresentación (ORA).
NOTA: Esta lista de genes puede abarcar vías, términos GO o genes objetivo de miRNA. El investigador puede utilizar los módulos de transcripción sanguínea (BTM) como conjuntos de genes para este análisis. El archivo BTM (BTM_for_GSEA.gmt).
- Establecer parámetros para realizar análisis de coexpresión y obtener sus resultados.
- A continuación, expanda la sección Parámetro , haciendo clic en el signo más para mostrar los parámetros predeterminados. Si es necesario, cámbielos. Marque la casilla Aplicar VST .
- Escriba el correo electrónico en la sección Correo electrónico para recibir los resultados como un correo electrónico. Este paso es opcional.
- Pulse el botón Ejecutar CEMiTool .
- Descargue el informe de análisis completo haciendo clic en Descargar informe completo en la parte superior derecha. Descargará un archivo comprimido cemitool_results.zip.
- Extraiga el contenido de la cemitool_results.zip con WinRAR.
NOTA: La carpeta con el contenido extraído abarca varios archivos con todos los resultados del análisis y sus parámetros establecidos.
5. Determinación del grado molecular de perturbación de las muestras
- Versión web de Grado Molecular de Perturbación (MDP).
- Para ejecutar MDP, acceda al sitio web de MDP (Tabla de materiales). MDP calcula la distancia molecular de cada muestra a partir de la referencia. Haga clic en el botón Ejecutar .
- En el vínculo Elegir archivo , cargue el archivo de expresión tmm_expression.tsv. A continuación, cargue el archivo de datos fenotípicos metadata.tsv desde el paso Descargar datos 4.2.2. También es posible enviar un archivo de anotación de vías en formato GMT para calcular la puntuación de perturbación de las vías asociadas con la enfermedad.
- Una vez cargados los datos, defina la columna Clase que contiene la información fenotípica utilizada por el MDP. A continuación, defina la clase de control seleccionando la etiqueta que corresponde a la clase de control.
NOTA: Hay algunos parámetros opcionales que afectarán a la forma en que se calculan las puntuaciones de la muestra. Si es necesario, el usuario puede cambiar el método promedio de las estadísticas, la desviación estándar y el porcentaje máximo de los genes perturbados. - Después de eso, presione el botón Ejecutar MDP y se mostrarán los resultados de MDP. El usuario puede descargar las figuras haciendo clic en el gráfico de descarga en cada gráfico, así como en el puntaje MDP en el botón Descargar archivo de puntaje MDP .
NOTA: En caso de preguntas sobre cómo enviar los archivos o cómo funciona MDP, simplemente vaya al Tutorial y Acerca de las páginas web.
6. Análisis de enriquecimiento funcional
- Cree una lista de DEG regulados a la baja y otra de DEG regulados al alza. Los nombres de los genes deben estar de acuerdo con los símbolos genéticos de Entrez. Cada gen de la lista debe colocarse en una línea.
- Guarde las listas de genes en formato txt o tsv.
- Acceda a la página web de Enrichr (Tabla de Materiales) para realizar el análisis funcional.
- Seleccione la lista de genes haciendo clic en Elegir archivo. Seleccione uno de los DEGs de la lista y pulse el botón Enviar .
- Haga clic en Pathways en la parte superior de la página web para realizar un análisis de enriquecimiento funcional con el enfoque ORA.
- Elija una base de datos de rutas. La base de datos de vías "Reactome 2016" se utiliza ampliamente para obtener el significado biológico de los datos humanos.
- Haga clic de nuevo en el nombre de la base de datos de rutas. Seleccione Gráfico de barras y compruebe si está ordenado por clasificación de valor p. De lo contrario, haga clic en el gráfico de barras hasta que se ordene por valor p. Este gráfico de barras incluye las 10 vías principales según los valores p.
- Pulse el botón Configuración y seleccione el color rojo para el análisis de genes regulados hacia arriba o el color azul para el análisis de genes regulados hacia abajo. Guarde el gráfico de barras en varios formatos haciendo clic en svg, png y jpg.
- Seleccione Tabla y haga clic en Exportar entradas a la tabla en la parte inferior izquierda del gráfico de barras para obtener los resultados del análisis de enriquecimiento funcional en un archivo txt.
NOTA: Este archivo de resultados de enriquecimiento funcional abarca en cada línea el nombre de una vía, el número de genes superpuestos entre la lista DEG presentada y la vía, el valor p, el valor p ajustado, la razón de probabilidades, la puntuación combinada y el símbolo genético de los genes presentes en la lista DEG que participan en la vía. - Repita los mismos pasos con la lista de otros DEG.
NOTA: El análisis con DEG regulados a la baja proporciona vías enriquecidas para genes regulados a la baja y el análisis con genes regulados al alza proporciona vías enriquecidas para genes regulados al alza.
Resultados
El entorno informático para los análisis de transcriptomas se creó y configuró en la plataforma Docker. Este enfoque permite a los usuarios principiantes de Linux utilizar sistemas de terminal Linux sin conocimientos de gestión a priori. La plataforma Docker utiliza los recursos del sistema operativo host para crear un contenedor de servicios que incluye herramientas de usuarios específicos (Figura 1B). Se creó un contenedor basado en la distribución Linux OS Ubuntu 20.04 y se configuró completamente para análisis transcriptómicos, al que se puede acceder a través del terminal de línea de comandos. En este contenedor, hay una estructura de carpetas predefinida para conjuntos de datos y scripts que es necesaria para todos los análisis de canalización (Figura 1C). Para los análisis se utilizó un estudio publicado por nuestro grupo de investigación7 , que comprendió 20 muestras de individuos sanos y 39 muestras de individuos infectados agudamente por CHIKV (Figura 1D).
El proceso de secuenciación total del ARN puede generar errores de lectura, que pueden ser causados por un cúmulo con dos o más transcripciones o el agotamiento de los reactivos. Las plataformas de secuenciación devuelven un conjunto de archivos "FASTQ" que contienen la secuencia (lectura) y la calidad asociada para cada base de nucleótidos (Figura 2A). La escala de calidad phred indica la probabilidad de una lectura incorrecta de cada base (Figura 2B). Las lecturas de baja calidad pueden generar un sesgo o una expresión génica inadecuada, lo que desencadena errores sucesivos en los análisis posteriores. Se desarrollaron herramientas como Trimmomatic para identificar y eliminar lecturas de baja calidad de muestras y para aumentar la probabilidad de lecturas de mapeo (Figura 2C, D).
El módulo de mapeo fue preconfigurado con el alineador STAR y el huésped humano GRCh38 como genoma de referencia. En este paso, las lecturas de alta calidad recuperadas del paso anterior se utilizan como entrada para alinearse con el genoma humano de referencia (Figura 3A). El alineador STAR genera una alineación de lecturas asignadas a un genoma de referencia en el archivo de formato BAM. Sobre la base de esta alineación, la herramienta FeatureCounts realiza la anotación de entidades (genes) de las lecturas alineadas utilizando la anotación de referencia del host humano en formato de archivo GTF (Figura 3B). Finalmente, se genera la matriz de expresión con cada nombre de gen como una fila, y cada muestra como una columna (Figura 3C). También es necesario proporcionar un archivo de metadatos adicional que contenga los nombres de muestra y los respectivos grupos de muestra para un análisis posterior posterior. La matriz de expresión génica representa el número de recuentos asignados a cada gen entre las muestras, que se puede utilizar como entrada EdgeR para identificar DEG. Además, esta matriz de expresión génica se normalizó utilizando TMM y CPM con el fin de eliminar la variabilidad técnica y corregir la medición de ARN-seq considerando la proporción de genes expresados en el tamaño total de la biblioteca entre las muestras. Esta matriz se utilizó además como insumo para los análisis de coexpresión y MDP.
CEMiTool identifica y analiza los módulos de coexpresión12. Los genes que están en el mismo módulo se coexpresan, lo que significa que exhiben patrones similares de expresión en las muestras del conjunto de datos. Esta herramienta también permite la exploración de la importancia biológica de cada módulo identificado. Para ello, proporciona tres análisis opcionales: análisis de enriquecimiento funcional por GSEA, análisis de enriquecimiento funcional por análisis de sobrerrepresentación (ORA) y análisis de red. El análisis de enriquecimiento funcional realizado por GSEA proporciona información sobre la expresión génica de cada módulo en cada fenotipo (Figura 4A). De acuerdo con esto, permite la identificación de los módulos que son reprimidos o inducidos en cada fenotipo. El análisis ORA muestra las 10 principales funciones biológicas significativamente enriquecidas de cada módulo ordenadas por valores p ajustados. Es posible combinar los resultados de GSEA y ORA para identificar procesos biológicos deteriorados y si están siendo reprimidos o inducidos por el fenotipo de interés. Los análisis de red proporcionan un interactoma de cada módulo (Figura 4A). Permite la visualización de cómo interactúan los genes de cada módulo. Además de esto, el análisis de red proporciona información sobre los genes más conectados, los hubs, que se identifican por sus nombres en la red. El tamaño de los nodos representa el grado de conectividad.
Para identificar los DEG, se desarrolló un script interno para ejecutar un análisis diferencial de extremo a extremo en una línea de comandos concisa y unidireccional. El script realiza todos los pasos necesarios para realizar un análisis DEG, comparando diferentes grupos de ejemplo proporcionados por el usuario en un archivo de metadatos. Además, los resultados de DEG se almacenan en listas separadas de genes regulados a la baja y regulados al alza, y luego se compilan en una figura lista para su publicación (Figura 4B) utilizando el paquete EnhancedVolcano R de Bioconductor.
El análisis del grado molecular de perturbación realizado por la herramienta MDP nos permite identificar muestras perturbadas de individuos sanos e infectados11. La puntuación de perturbación se calcula considerando todos los genes expresados para cada muestra infectada con CHIKV y considerando las muestras sanas como el grupo de referencia (Figura 5A). MDP también realiza el análisis utilizando solo el 25% superior de los genes más perturbados de esas muestras (Figura 5B). Las muestras pueden presentar una gran variabilidad dados los antecedentes genéticos, la edad, el sexo u otras enfermedades previas. Estos factores pueden cambiar el perfil del transcriptoma. Sobre la base de esto, MDP sugiere qué muestras son valores atípicos biológicos potenciales para eliminarlas y mejorar los resultados posteriores (Figura 5A, B).
Se puede realizar un análisis de enriquecimiento funcional por ORA utilizando Enrichr para identificar el significado biológico de los DEG. Los resultados proporcionados en base a la lista de genes regulados a la baja indican los procesos biológicos reprimidos en el fenotipo estudiado, mientras que los resultados proporcionados en base a la lista de genes regulados al alza presentan los procesos biológicos que se inducen en el fenotipo de interés. Los procesos biológicos mostrados en el gráfico de barras generado por Enrichr son los 10 mejores conjuntos de genes enriquecidos basados en la clasificación del valor p (Figura 6).

Figura 1: Docker de entorno y estudio de ejemplo. (A) La plataforma Docker utiliza los recursos del host del sistema operativo para crear "contenedores" para el sistema Linux que contienen herramientas para análisis de transcriptomas. (B) El contenedor Docker simula un sistema Linux para ejecutar scripts de canalización. (C) La estructura de carpetas de la canalización del transcriptoma se creó y organizó para almacenar conjuntos de datos y scripts para su análisis. (D) El estudio de nuestro grupo se utilizó como ejemplo de análisis de transcriptomas. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Control de calidad de la secuenciación. (A) El archivo de formato FASTQ se utiliza para representar la calidad de la secuencia y la base de nucleótidos. (B) Ecuación de puntuación de Phred, donde cada 10 aumenta una base de probabilidad de lectura errónea de registro. (C) y (D) El Boxplot representa una distribución de calidad de cada base de nucleótidos antes y después de la ejecución trimmomática, respectivamente. Haga clic aquí para ver una versión más grande de esta figura.
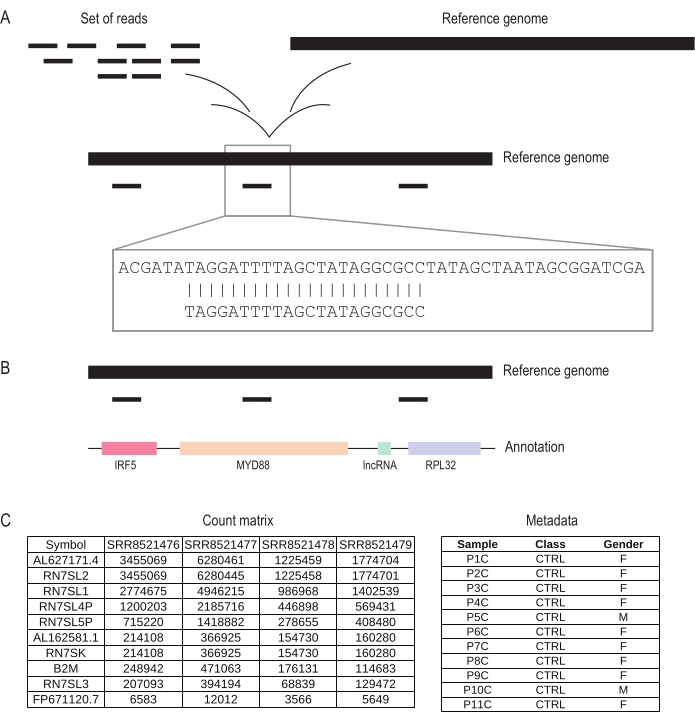
Figura 3: Proceso de mapeo y anotación desde la secuencia hasta la expresión del recuento de genes. (A) El mapeo consiste en alinear la secuencia de la transcripción y la secuencia del genoma para identificar la localización genómica. (B) Las lecturas mapeadas al genoma de referencia se anotan en función de su localización genómica de superposición. (C) Sobre la base de las herramientas de archivo de mapeo como featureCounts, se resume la expresión génica. Haga clic aquí para ver una versión más grande de esta figura.
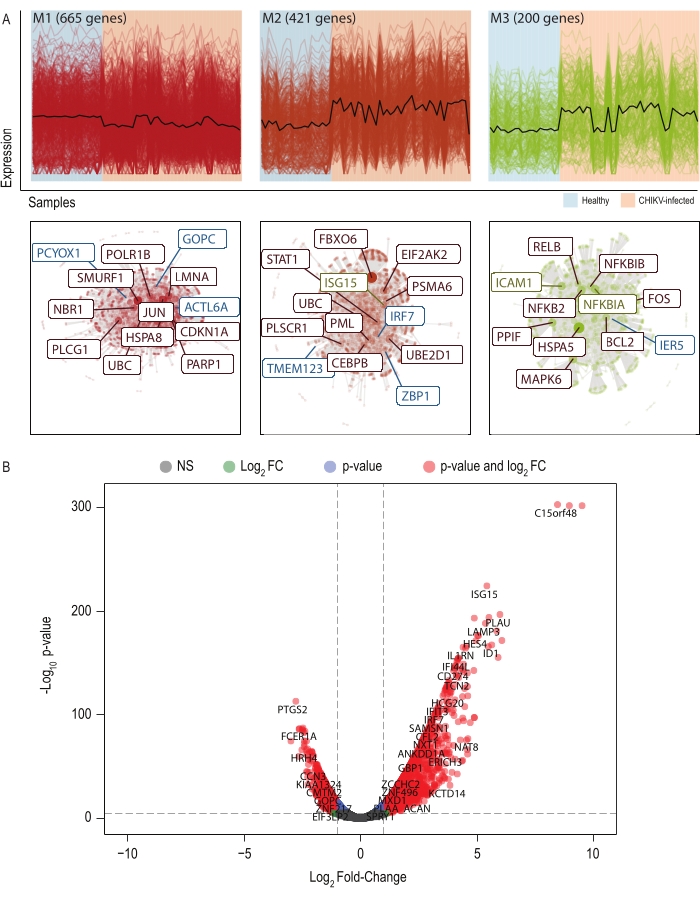
Figura 4: Red de genes coexpresados y análisis estadístico de DEGs. (A) Módulos de coexpresión basados en la expresión génica y la red de interacciones proteína-proteína a partir de genes módulo. (B) Análisis estadístico de individuos sanos e infectados agudamente por CHIKV, y expresión génica diferencial en rojo (criterios p-value y log2FC), púrpura (solo p-value), verde (solo log2FC) y gris (sin significación). Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Grado molecular de perturbación (MDP) de individuos sanos e infectados agudamente por CHIKV. (A) Puntuación de MDP para cada muestra utilizando todos los genes expresados del transcriptoma. (B) Puntuación MDP para cada muestra utilizando solo el 25% superior de los genes más perturbados. Haga clic aquí para ver una versión más grande de esta figura.
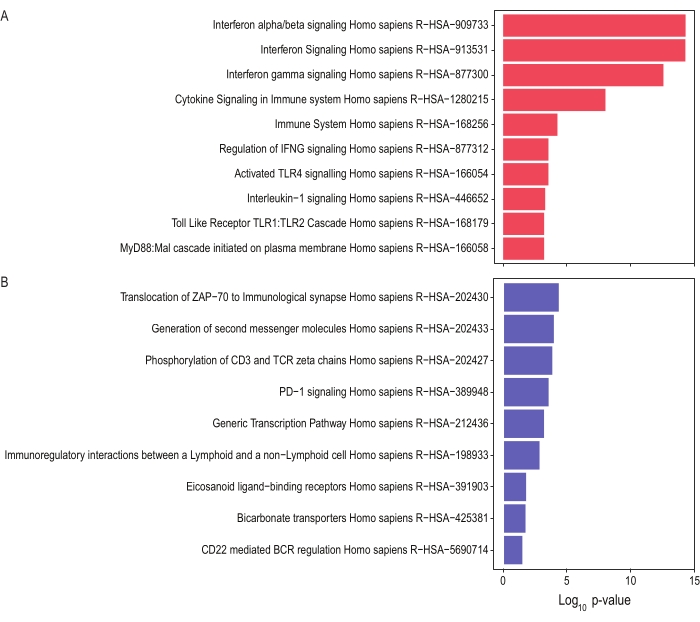
Figura 6: Análisis funcional para DEG. (A) Los genes regulados hacia arriba y (B) regulados hacia abajo se enviaron a la herramienta del sitio web de Enrichr para evaluar las vías biológicas o los conjuntos de genes representativos. Se calcularon los valores de P para cada vía y solo se mostraron diferencias significativas en el gráfico. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
La preparación de las bibliotecas de secuenciación es un paso crucial para responder a las preguntas biológicas de la mejor manera posible. El tipo de transcripciones de interés del estudio guiará qué tipo de biblioteca de secuenciación se elegirá e impulsará los análisis bioinformáticos. Por ejemplo, a partir de la secuenciación de un patógeno y la interacción del huésped, de acuerdo con el tipo de secuenciación, es posible identificar secuencias de ambos o solo de las transcripciones del huésped.
El equipo de secuenciación de próxima generación, por ejemplo, la Plataforma Illumina, mide las puntuaciones de calidad de secuenciación, lo que representa la probabilidad de que una base se llame incorrectamente. Los análisis posteriores son muy sensibles a las secuencias de baja calidad y conducen a una expresión génica poco leída o mal leída. Otro obstáculo para realizar análisis e interpretación correctos son las secuencias adaptadoras. Las secuencias de adaptadores ayudan en la preparación y secuenciación de bibliotecas, y en la mayoría de los casos, los adaptadores también se secuencian. Estudios recientes han identificado que el impacto de la herramienta de mapeo en los resultados finales es mínimo13. Sin embargo, en los estudios de patógenos-huéspedes, el proceso de mapeo puede generar resultados ligeramente mejores al probar diferentes umbrales para minimizar el problema de las secuencias de locus mapeados múltiples.
Los resultados de la expresión génica diferencial deben interpretarse con cierta precaución, especialmente cuando el número de muestras por grupo es muy pequeño y las muestras provienen de diferentes ensayos e interfieren por efectos de lotes en el resultado de los DEG. Estos resultados son sensibles a varios factores: (i) el filtrado de datos aplicado, como la eliminación de genes de baja expresión y el número de muestras a mantener; (ii) diseño del estudio, para comparar solo entre grupos de muestra o cada paciente infectado vs todos los pacientes de control, como se ilustra en el estudio CHIKV7; y iii) método estadístico utilizado para identificar los DEG. Aquí, ilustramos un ejemplo básico con EdgeR para identificar DEG asumiendo un valor p umbral de 0.05. También se sabe en la literatura que, en comparación con otros métodos de referencia, EdgeR puede tener un amplio rango de variabilidad en la identificación de DEGs14. Se podría considerar la disyuntiva entre estos diferentes métodos y tener en cuenta el número de réplicas disponibles y la complejidad del diseño experimental14.
CEMiTool realiza análisis de módulos de coexpresión12. Esta herramienta está disponible a través del paquete R en el repositorio bioconductor y también está disponible en una versión fácil de usar a través de webCEMiTool; esta última es la versión utilizada en este protocolo actual. Se trata de un software alternativo en relación con WGCNA15 que presenta varios beneficios en comparación con este último16, entre ellos el hecho de que es más fácil de usar17. Además, esta herramienta tiene un método automático para filtrar genes, mientras que en WGCNA el usuario debe filtrar los genes antes del uso de WGCNA. Además, esta herramienta tiene establecidos parámetros por defecto, mientras que en WGCNA el usuario debe seleccionar manualmente los análisis de parámetros. La selección manual de parámetros perjudica la reproducibilidad; por lo tanto, la selección automática de parámetros garantiza una mejor reproducibilidad.
En ciertos casos, CEMiTool no es capaz de encontrar un umbral suave apropiado, también llamado valor de β. En este caso, el usuario debe comprobar si los datos de RNA-seq presentan una fuerte dependencia de la varianza media. Si la media exhibe una fuerte relación lineal con la varianza (considerando todos los genes), el usuario debe volver a ejecutar los análisis verificando el parámetro "Apply VST" para eliminar la dependencia media-varianza de los datos transcriptómicos. Siempre es fundamental comprobar si existe una fuerte dependencia de la varianza media en los datos y eliminarlos cuando estén presentes.
CEMiTool se ha utilizado ampliamente para identificar y explorar el significado biológico de los módulos de coexpresión. Un estudio de infección aguda por CHIKV mostró un módulo con mayor actividad en pacientes después de 2 a 4 días de la aparición de los síntomas7. El enriquecimiento funcional de este módulo por ORA exhibió un aumento de monocitos y neutrófilos7. Un estudio de vacunación contra la influenza utilizando transcriptoma sanguíneo desde el inicio hasta el día 7 después de la vacunación presentó módulos de coexpresión funcionalmente enriquecidos para procesos biológicos relacionados con T, B y células asesinas naturales, monocitos, neutrófilos, respuestas de interferón y activación plaquetaria18.
Teniendo en cuenta la variabilidad de los datasets transcriptómicos, identificar y cuantificar la heterogeneidad de los datos puede ser un desafío ya que muchas variables pueden influir en el perfil de expresión génica7,11. MDP proporciona una forma de identificar y cuantificar muestras perturbadas de sujetos sanos e infectados siguiendo estos pasos: (i) calcular un método de centralidad (mediana o media) y desviación estándar de muestras de control; ii) utilizar los valores obtenidos para calcular la puntuación z de todos los genes; iii) establecer un umbral z-score absoluto superior a 2, indicando desviaciones representativas de las muestras de control; y (iv) calcular el promedio de los valores genéticos utilizando las puntuaciones filtradas para cada muestra. A pesar de tener algunas limitaciones para el análisis scRNA-seq, esta herramienta fue funcional para determinar la puntuación de perturbación a partir de datos de microarrays y ARN-seq11. Además, un estudio previo ha utilizado esta herramienta para demostrar el grado molecular de perturbación elevado en el transcriptoma sanguíneo en pacientes con tuberculosis y diabetes mellitus19. En este trabajo, se ha demostrado la perturbación de muestras de control y CHIKV infectadas agudamente utilizando individuos sanos como grupo de referencia.
El análisis de enriquecimiento funcional realizado por Enrichr es el ORA20,21. ORA es un tipo de análisis de enriquecimiento funcional en el que el usuario debe proporcionar la lista de DEG a la herramienta. La lista de DEG generalmente se separa en una lista de DEG regulada a la baja y en una lista de DEG regulada al alza. Existen otras herramientas para realizar ORA, entre ellas, el gProfiler, que está disponible en una versión web fácil de usar22 y el goseq23 que está disponible como un paquete R en Bioconductor. Otro tipo de análisis de enriquecimiento funcional es GSEA. Para realizar GSEA, el usuario debe proporcionar todos los genes en una lista clasificada. Esta lista generalmente se clasifica de acuerdo con la expresión génica en el cambio de pliegue.
Enrichr siempre proporciona los 10 mejores conjuntos de genes enriquecidos en función de sus valores p en el resultado del gráfico de barras. Por lo tanto, el usuario debe estar alerta al interpretar los resultados, si hay menos de 10 conjuntos de genes enriquecidos, el gráfico de barras también mostrará procesos biológicos no enriquecidos. Para evitar este error, el usuario debe establecer un punto de corte para el valor p y observar los valores p de las vías antes de asumir que todos los conjuntos de genes del gráfico de barras están enriquecidos. Además, el usuario debe ser consciente de que el orden de los 10 conjuntos de genes que se muestran en el gráfico de barras es de acuerdo con los valores p, no con los valores p ajustados. En caso de que el usuario quiera mostrar todas las rutas enriquecidas en un gráfico de barras o incluso reordenar de acuerdo con los valores p ajustados, se recomienda que el usuario cree su propio gráfico de barras utilizando la tabla descargada. El usuario puede hacer un nuevo gráfico de barras utilizando Excel o incluso el software R.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
HN es financiado por la FAPESP (números de subvención: #2017/50137-3, 2012/19278-6, 2018/14933-2, 2018/21934-5 y 2013/08216-2) y CNPq (313662/2017-7).
Estamos particularmente agradecidos a las siguientes becas para becarios: ANAG (Proceso FAPESP 2019/13880-5), VEM (Proceso FAPESP 2019/16418-0), IMSC (Proceso FAPESP 2020/05284-0), APV (Proceso FAPESP 2019/27146-1) y, RLTO (Proceso CNPq 134204/2019-0).
Materiales
| Name | Company | Catalog Number | Comments |
| CEMiTool | Computational Systems Biology Laboratory | 1.12.2 | Discovery and the analysis of co-expression gene modules in a fully automatic manner, while providing a user-friendly HTML report with high-quality graphs. |
| EdgeR | Bioconductor (Maintainer: Yunshun Chen [yuchen at wehi.edu.au]) | 3.30.3 | Differential expression analysis of RNA-seq expression profiles with biological replication |
| EnhancedVolcano | Bioconductor (Maintainer: Kevin Blighe [kevin at clinicalbioinformatics.co.uk]) | 1.6.0 | Publication-ready volcano plots with enhanced colouring and labeling |
| FastQC | Babraham Bioinformatics | 0.11.9 | Aims to provide a simple way to do some quality control checks on raw sequence data coming from high throughput sequencing |
| FeatureCounts | Bioinformatics Division, The Walter and Eliza Hall Institute of Medical Research | 2.0.0 | Assign mapped sequencing reads to specified genomic features |
| MDP | Computational Systems Biology Laboratory | 1.8.0 | Molecular Degree of Perturbation calculates scores for transcriptome data samples based on their perturbation from controls |
| R | R Core Group | 4.0.3 | Programming language and free software environment for statistical computing and graphics |
| STAR | Bioinformatics Division, The Walter and Eliza Hall Institute of Medical Research | 2.7.6a | Aligner designed to specifically address many of the challenges of RNA-seq data mapping using a strategy to account for spliced alignments |
| Bowtie2 | Johns Hopkins University | 2.4.2 | Ultrafast and memory-efficient tool for aligning sequencing reads to long reference sequences |
| Trimmomatic | THE USADEL LAB | 0.39 | Trimming adapter sequence tasks for Illumina paired-end and single-ended data |
| Get Docker | Docker | 20.10.2 | Create a bioinformatic environment reproducible and predictable (https://docs.docker.com/get-docker/) |
| WSL2-Kernel | Windows | NA | https://docs.microsoft.com/en-us/windows/wsl/wsl2-kernel |
| Get Docker Linux | Docker | NA | https://docs.docker.com/engine/install/ubuntu/ |
| Docker Linux Repository | Docker | NA | https://docs.docker.com/engine/install/ubuntu/#install-using-the-repository |
| MDP Website | Computational Systems Biology Laboratory | NA | https://mdp.sysbio.tools |
| Enrichr Website | MaayanLab | NA | https://maayanlab.cloud/Enrichr/ |
| webCEMiTool | Computational Systems Biology Laboratory | NA | https://cemitool.sysbio.tools/ |
| gProfiler | Bioinformatics, Algorithmics and Data Mining Group | NA | https://biit.cs.ut.ee/gprofiler/gost |
| goseq | Bioconductor (Maintainer: Matthew Young [my4 at sanger.ac.uk]) | NA | http://bioconductor.org/packages/release/bioc/html/goseq.html |
| SRA NCBI study | NCBI | NA | https://www.ncbi.nlm.nih.gov/bioproject/PRJNA507472/ |
Referencias
- Weaver, S. C., Charlier, C., Vasilakis, N., Lecuit, M. Zika, Chikungunya, and Other Emerging Vector-Borne Viral Diseases. Annual Review of Medicine. 69, 395-408 (2018).
- Burt, F. J., et al. Chikungunya virus: an update on the biology and pathogenesis of this emerging pathogen. The Lancet. Infectious Diseases. 17 (4), 107-117 (2017).
- Hua, C., Combe, B. Chikungunya virus-associated disease. Current Rheumatology Reports. 19 (11), 69 (2017).
- Suhrbier, A., Jaffar-Bandjee, M. -. C., Gasque, P. Arthritogenic alphaviruses-an overview. Nature Reviews Rheumatology. 8 (7), 420-429 (2012).
- Nakaya, H. I., et al. Gene profiling of chikungunya virus arthritis in a mouse model reveals significant overlap with rheumatoid arthritis. Arthritis and Rheumatism. 64 (11), 3553-3563 (2012).
- Michlmayr, D., et al. Comprehensive innate immune profiling of chikungunya virus infection in pediatric cases. Molecular Systems Biology. 14 (8), 7862 (2018).
- Soares-Schanoski, A., et al. Systems analysis of subjects acutely infected with the Chikungunya virus. PLOS Pathogens. 15 (6), 1007880 (2019).
- Alexandersen, S., Chamings, A., Bhatta, T. R. SARS-CoV-2 genomic and subgenomic RNAs in diagnostic samples are not an indicator of active replication. Nature Communications. 11 (1), 6059 (2020).
- Wang, D., et al. The SARS-CoV-2 subgenome landscape and its novel regulatory features. Molecular Cell. 81 (10), 2135-2147 (2021).
- Wilson, J. A. C., et al. RNA-Seq analysis of chikungunya virus infection and identification of granzyme A as a major promoter of arthritic inflammation. PLOS Pathogens. 13 (2), 1006155 (2017).
- Gonçalves, A. N. A., et al. Assessing the impact of sample heterogeneity on transcriptome analysis of human diseases using MDP webtool. Frontiers in Genetics. 10, 971 (2019).
- Russo, P. S. T., et al. CEMiTool: a Bioconductor package for performing comprehensive modular co-expression analyses. BMC Bioinformatics. 19 (1), 56 (2018).
- Costa-Silva, J., Domingues, D., Lopes, F. M. RNA-Seq differential expression analysis: An extended review and a software tool. PloS One. 12 (12), 0190152 (2017).
- Seyednasrollah, F., Laiho, A., Elo, L. L. Comparison of software packages for detecting differential expression in RNA-seq studies. Briefings in Bioinformatics. 16 (1), 59-70 (2015).
- Zhang, B., Horvath, S. A general framework for weighted gene co-expression network analysis. Statistical Applications in Genetics and Molecular Biology. 4, (2005).
- Cheng, C. W., Beech, D. J., Wheatcroft, S. B. Advantages of CEMiTool for gene co-expression analysis of RNA-seq data. Computers in Biology and Medicine. 125, 103975 (2020).
- Cardozo, L. E., et al. webCEMiTool: Co-expression modular analysis made easy. Frontiers in Genetics. 10, 146 (2019).
- de Lima, D. S., et al. Long noncoding RNAs are involved in multiple immunological pathways in response to vaccination. Proceedings of the National Academy of Sciences of the United States of America. 116 (34), 17121-17126 (2019).
- Prada-Medina, C. A., et al. Systems immunology of diabetes-tuberculosis comorbidity reveals signatures of disease complications. Scientific Reports. 7 (1), 1999 (2017).
- Chen, E. Y., et al. Enrichr: interactive and collaborative HTML5 gene list enrichment analysis tool. BMC Bioinformatics. 14, 128 (2013).
- Kuleshov, M. V., et al. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. Nucleic Acids Research. 44, 90-97 (2016).
- Raudvere, U., et al. g:Profiler: a web server for functional enrichment analysis and conversions of gene lists (2019 update). Nucleic Acids Research. 47, 191-198 (2019).
- Young, M. D., Wakefield, M. J., Smyth, G. K., Oshlack, A. Gene ontology analysis for RNA-seq: accounting for selection bias. Genome Biology. 11 (2), 14 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados