Method Article
Evaluación de la reprogramación cardíaca mediante el análisis de imágenes de alto contenido
En este artículo
Resumen
Presentamos un protocolo para cuantificar células similares a cardiomiocitos (iCMs) inducidas directamente reprogramadas in vitro utilizando análisis de imagen de alto contenido. Este método nos permite cuantificar la eficiencia de la reprogramación cardíaca de forma automatizada y visualizar directamente los iCMs.
Resumen
El objetivo de este protocolo es describir un método para cuantificar las células similares a cardiomiocitos (iCMs) inducidas, que son reprogramadas directamente in vitro mediante una técnica de reprogramación. La reprogramación cardíaca proporciona una estrategia para generar nuevos cardiomiocitos. Mediante la introducción de factores de transcripción cardiogénicos centrales en los fibroblastos; los fibroblastos se pueden convertir en iCM sin transición a través del estado de células madre pluripotentes. Sin embargo, la tasa de conversión de fibroblastos en iCMs sigue siendo baja. En consecuencia, ha habido numerosos enfoques adicionales para mejorar la eficiencia de la reprogramación cardíaca. La mayoría de estos estudios evaluaron la eficiencia de la reprogramación cardíaca mediante citometría de flujo, mientras que al mismo tiempo realizaron inmunocitoquímica para visualizar los iCM. Por lo tanto, se requieren al menos dos conjuntos separados de experimentos de reprogramación para demostrar el éxito de la reprogramación de iCM. Por el contrario, el análisis automatizado de imágenes de alto contenido proporcionará tanto la cuantificación como la calificación de la reprogramación de iCM con un número relativamente pequeño de células. Con este método, es posible evaluar directamente la cantidad y la calidad de los iCM con un solo experimento de reprogramación. Este enfoque podrá facilitar futuros estudios de reprogramación cardíaca que requieran experimentos de reprogramación a gran escala, como el cribado de factores genéticos o farmacológicos, para mejorar la eficiencia de la reprogramación. Además, la aplicación del protocolo de análisis de imágenes de alto contenido no se limita a la reprogramación cardíaca. Se puede aplicar a la reprogramación de otros linajes celulares, así como a cualquier experimento de inmunotinción que necesite tanto la cuantificación como la visualización de células inmunoteñidas.
Introducción
La reprogramación cardíaca se ha desarrollado como un enfoque alternativo a los enfoques mediados por células madre para generar nuevos cardiomiocitos. Dado que no pasa por el estado de células madre, tiene un alto potencial para eludir algunas limitaciones heredadas en los enfoques mediados por células madre. Se ha demostrado que la infección viral de al menos tres o cuatro factores de transcripción cardiogénicos en fibroblastos puede convertir los fibroblastos en un destino cardíaco mediante la eliminación de los programas genéticos de fibroblastos y la reconstrucción de las redes transcripcionales cardiogénicas en los fibroblastos 1,2,3,4,5,6,7,8,9,10, 11,12,13,14,15,16,17.
Desde el primer estudio de referencia que demostró la reprogramación cardíaca in vitro1, el protocolo de reprogramación cardíaca ha sido optimizado por numerosos estudios 3,5,6,7,9,11,12,13,14,15,16,18 . Los enfoques técnicos comunes para evaluar los fenotipos cardíacos en fibroblastos después de la reprogramación cardíaca han sido el análisis de citometría de flujo para cuantificar las células que expresan marcadores específicos de cardiomiocitos y la inmunocitoquímica para visualizar esas células a nivel de una sola célula. Aunque ambos experimentos (es decir, citometría de flujo e inmunocitoquímica) deben demostrar la expresión de marcadores de cardiomiocitos utilizando los mismos anticuerpos, deben realizarse por separado. Además, la citometría de flujo necesita un número relativamente mayor de células, lo que aumenta la cantidad de reactivos necesarios para el experimento. Alternativamente, las células positivas para marcadores de cardiomiocitos se pueden cuantificar mediante recuento manual siguiendo la inmunocitoquímica. Sin embargo, es muy laborioso y tiende a ser menos preciso.
El propósito de este protocolo es describir el método que puede cuantificar y visualizar los iCMs mediante un único experimento de inmunotinción utilizando el análisis automatizado de imágenes de alto contenido. Requiere un número relativamente pequeño de celdas de inicio porque este protocolo se realiza en una placa de 24 pocillos. Se pueden utilizar hasta tres marcadores diferentes al mismo tiempo. Las células positivas simples, dobles y triples se pueden cuantificar automáticamente. Además de la cuantificación de células inmunoteñidas, el análisis de imágenes de alto contenido proporciona imágenes objetivas de alta calidad de 2 a 100x. Si es necesario, las mismas células inmunoteñidas que se utilizan en el análisis de imágenes de alto contenido se pueden reutilizar para estudios de imagen adicionales, como la microscopía confocal. La principal ventaja de este protocolo es que no solo proporciona una cuantificación imparcial de los iCM con un número mucho menor de celdas, sino también la visualización de los iCM. Además, este protocolo se puede utilizar para evaluar la reprogramación del linaje no cardíaco (por ejemplo, iPSC, reprogramación de neuronas y hepatocitos).
Protocolo
Todos los procedimientos con animales se realizaron con la aprobación del Comité Institucional de Cuidado y Uso de Animales del Centro Médico de la Universidad de Vanderbilt.
1. Generación de retrovirus y reprogramación cardíaca in vitro
- Cultivo de células Platinum E en DMEM suplementado con 10% de FBS, 1% de penicilina/estreptomicina, 1 μg/mL de puromicina y 10 μg/mL de blasticidina hasta que la confluencia de células Platinum E alcance 70-80%.
- El día 1, siembre ~0.55 x 106 celdas (primer pocillo) y ~0.18 x 106 celdas (segundo pocillo) en dos pocillos separados de placa de 12 pocillos con 1 mL de DMEM suplementado con 10% de FBS y 1% de penicilina/estreptomicina un día antes de la primera transfección.
NOTA: Se pueden utilizar placas de cultivo de diferentes tamaños dependiendo de la cantidad de medios virales necesarios para los experimentos. Consulte la Tabla 1 para ver otras escalas de experimentos. - En el día 2, transfecte el constructo retroviral M-G-T-H cuádruple cistrónico que codifica Mef2c, Gata4, Tbx5 y Hand2 descrito en el estudio anterior9 o vector vacío en células Platinum E del primer pocillo. Añadir 3 μL de reactivo de transfección a 30 μL de medio sérico reducido. Cinco minutos más tarde, agregue 1 μg de construcción retroviral o el vector vacío a la mezcla de reactivo de transfección y medio sérico reducido. Después de 20 minutos de incubación a temperatura ambiente, agregue la mezcla a las células Platinum E.
- El día 3, 16-20 h después de la transfección, retire el medio y reponga el DMEM fresco suplementado con un 10% de FBS y un 1% de penicilina/estreptomicina.
- El día 3, 24 h después de la primera transfección, realice la segunda transfección en el segundo pocillo en el que se sembraron las células de platino E el día 1 como se describe en el paso 1.3).
- En el día 3, sembraron ~5 x 104 fibroblastos embrionarios (MEF) de ratón congelados aislados de ratones knock-in19 reporteros de Titin-GFP en un pocillo de placa de 24 pocillos. Se necesita un total de dos pocillos de placa de 24 pocillos (es decir, control no infectado e infección M-G-T-H).
NOTA: Es posible que sea necesario ajustar el número de MEF plateados, ya que la tasa de recuperación de los MEF congelados puede variar según las condiciones de congelación de las celdas. Alrededor del 10% de confluencia al día después de sembrar las células es adecuado para la reprogramación. La eficiencia de la reprogramación podría mejorarse utilizando MEF frescos sin congelar. - El día 4, 16-20 h después de la segunda transfección, retire el medio y reponga el DMEM fresco suplementado con un 10% de FBS y un 1% de penicilina/estreptomicina.
- El día 4, 48 h después de la primera transfección, recoja los medios virales en el primer pocillo de células Platinum E con una jeringa de 5 ml y fíltrelos a través de un filtro de membrana de polietersulfona (PES) de 0,45 μm. Retire los medios de crecimiento de fibroblastos en los MEF y reemplácelos con los medios virales suplementados con polibreno a 6 μg/mL (primera infección).
- El día 5, 48 h después de la segunda transfección, realizar la segunda infección utilizando los medios virales en el segundo pocillo como se describe en el paso 1.8).
- El día 6, 24 h después de la segunda infección, los medios virales se sustituyen por medios de inducción cardíaca compuestos por DMEM/199 (4:1), 10% de FBS, 5% de suero de caballo, 1% de penicilina/estreptomicina, 1% de aminoácidos no esenciales, 1% de aminoácidos esenciales, 1% de B-27, 1% de insulina-selenio-transferrina, 1% de mezcla de vitaminas y 1% de piruvato de sodio, 1 μM de SB431542 y 0,5 μm de A83-01. Cambie el medio de inducción cardíaca cada tres días hasta que se recolecten las células.
2. Inmunotinción
- A los 14-15 días después de la infección, fije las células en una placa de 24 pocillos con paraformaldehído al 2% durante 15 minutos.
- Permeabilizar las células fijas con tampón de permeabilización (0,05% Triton-X en PBS) lavando las células tres veces con tampón de permeabilización cada 5 min.
- Incubar las células con tampón de bloqueo universal durante 45 min.
- Incubar las células con α-actinina de ratón (dilución 1:400) y anticuerpos GFP de pollo (dilución 1:400) durante 1,5 h a temperatura ambiente.
NOTA: Aproximadamente 150 μL de solución de anticuerpos pueden cubrir toda el área de un pocillo de placa de 24 pocillos. - Lave las células con tampón de permeabilización durante 5 min tres veces.
- Incubar las células con anticuerpos secundarios anti-ratón Alexa-555 y anti-pollo Alexa-488 (dilución 1:400) durante 1 h a temperatura ambiente
- Lave las células con tampón de permeabilización durante 5 min tres veces.
- Añadir 2,5 μL de solución de DAPI en 250 μL de tampón de permeabilización.
3. Imágenes de alto contenido
- Encienda el sistema de imágenes.
- Abra el software asociado.
- Inicie sesión en el sistema.
- En la barra de tareas, seleccione Ejecutar un plato.
- Haga clic en Abrir placa de expulsión de puerta para abrir la puerta de la máquina y colocar la placa. Asegúrese de que la placa esté en la dirección correcta. Haga clic en Cerrar placa de carga de puerta para cerrar la puerta.
- Haga clic en Configuración de la placa de carga. Seleccione un protocolo de plantilla y, a continuación, haga clic en Cargar desde DB.
- Haga clic en Configuración de adquisición.
- En el cuadro de diálogo Configuración de adquisición de placas , haga clic en Configurar.
- En la pestaña Objetivo y cámara , seleccione Objetivo 10x.
- En la pestaña Placa , seleccione una placa de 24 pocillos.
- En la pestaña Sitios para visitar en Opciones de sitio, elija "Número fijo de sitios" para determinar el número de sitios de imágenes eligiendo el número en columnas y filas. Se utilizan un total de 36 sitios de imágenes seleccionando 6 en columnas y 6 en filas. Seleccione el espaciado entre cada sitio de imagen (es decir, 500 μm).
- En la pestaña Adquisición , establezca el número de longitud de onda en 3.
- En la pestaña Longitudes de onda de Iluminación, seleccione DAPI, FITC o Texas Red para cada longitud de onda por separado.
- Haga clic en la pestaña Ejecutar y luego configure el "Nombre de la carpeta" y el "Nombre de la placa" para la placa. Haga clic en los pocillos del diagrama de placas para obtener imágenes y, a continuación, en Calcular para configurar el desplazamiento de enfoque para cada longitud de onda. Configure primero el desplazamiento de foucs para DAPI. Haga clic en Exposición automática para configurar el tiempo de exposición automáticamente para cada longitud de onda. A continuación, haga clic en Adquirir placa para empezar a crear imágenes de los sitios seleccionados.
4. Análisis de imágenes de alto contenido
- Una vez que se completen las imágenes de alto contenido, haga clic en el menú Cribado y seleccione Revisar datos de placa para seleccionar una placa para el análisis.
- Haga clic en Seleccionar placa. En el cuadro de diálogo Seleccionar placa para revisión , abra la carpeta y seleccione la placa guardada en la base de datos y luego haga clic en Seleccionar.
NOTA: Para mostrar las imágenes, seleccione DAPI, FITC y Texas Red en el campo Longitudes de onda . En el campo Sitios , seleccione Todos los sitios. Haga clic en un sitio entre los 36 sitios seleccionados para mostrarlo en cada ventana de imagen de longitud de onda y, a continuación, haga clic en Tabla de búsqueda para seleccionar un color para la longitud de onda. Utilice la tecla Imprimir pantalla del teclado y pegue la imagen en un software de edición de fotos (por ejemplo, Paint y Photoshop) para guardarla. - Haga clic en Ejecutar análisis y seleccione una configuración de plantilla.
- Haga clic en Configurar ajustes y luego en Número de longitudes de onda. Seleccione tres longitudes de onda (es decir, DAPI para la tinción de núcleos, Teaxs Red para la α-actinina y FITC para Titin-GFP).
- Con la herramienta Línea de la barra de herramientas, mida el ancho en el eje corto de una celda. En función de los anchos medidos de las celdas, establezca el "Ancho mínimo aproximado" y el "Ancho máximo aproximado" para incluir la mayoría de las celdas en el sitio de imagen seleccionado.
- Haga clic en Vista previa para comprobar si casi todos los núcleos están seleccionados. Los núcleos seleccionados exhiben color blanco, mientras que los núcleos no seleccionados permanecen azules (teñidos con DAPI). Si es necesario, realice ajustes para "Ancho mínimo aproximado" y "Ancho máximo aproximado".
- Para establecer la intensidad sobre el fondo local, coloque un cursor del ratón dentro y fuera de una celda. El valor de intensidad aparece en la parte inferior de la ventana. Reduzca ligeramente la intensidad de una celda tenue para exhibir uniformemente la intensidad en toda el área de cada celda. Defina este valor de intensidad como el valor de Intensidad por encima del fondo local . Este valor debe establecerse por separado para cada canal.
- Haga clic en el menú Cribado y seleccione Utilidades de datos de placas. En el cuadro de diálogo Utilidades de datos de placa , haga clic en Ejecutar análisis para seleccionar la placa.
- En el campo Configuración , seleccione la configuración guardada para el análisis; seleccione Agregar a la lista de ejecución automática y, a continuación, haga clic en Aceptar para ejecutar el análisis.
- Una vez completado el análisis, haga clic en el menú Cribado y seleccione Utilidades de datos de placas. A continuación, en el cuadro de diálogo Utilidades de datos de placa , haga clic en Exportar mediciones para exportar los resultados del análisis.
- Haga clic en Mediciones de celdas e imágenes y luego en Aceptar.
- En la página Asistente para exportar medidas - Paso 1 , seleccione la placa y haga clic en Siguiente.
- En la página Asistente para exportar medidas - Paso 2 , haga clic en Finalizar.
- En la página Configurar exportación de datos , seleccione los tipos de datos (es decir, nombre del pozo, número de celda total, número de celda subtotal para DAPI+, número de celda subtotal para Texas Red+, número de celda subtotal para FITC+, número de celda subtotal para DAPI+Texas Red+, número de celda subtotal para DAPI+FITC+, número de celda subtotal para DAPI+FITC+Texas Red+, porcentaje de celdas DAPI+ , porcentaje de células DAPI+Texas Red+ , porcentaje de células DAPI+FITC+ y porcentaje de células DAPI+Texas Red+FITC+ ) y luego haga clic en Aceptar.
- En la página Exportar como archivo de texto , seleccione el destino para guardar el archivo y haga clic en Aceptar.
- Abra el archivo en Microsoft Excel. Los resultados serán presentados por cada sitio (Tabla 2). Hay 36 sitios por pozo. Utilizando la herramienta de análisis, Tabla dinámica, resuma los datos de los 36 sitios (es decir, las sumas de los números de celdas y los promedios del porcentaje de celdas indicado en los 36 sitios) (Tabla 3).
Resultados
Después de los experimentos de reprogramación, cuantificamos los iCM utilizando el análisis de imágenes de alto contenido como se describió anteriormente. En la Figura 1 se mostraron imágenes compuestas de 36 sitios de imágenes que se utilizaron para el análisis de imágenes de alto contenido. Las iCMs se definen como células dobles positivas (α-actinina+Titina-eGFP+) en estos experimentos. El análisis de imágenes de alto contenido muestra que ~ 26% de las células exhibieron ambos marcadores cardíacos después de la transducción M-G-T-H, mientras que ~ 1% de las células de control transducidas con vector vacío muestran células dobles positivas. Los iCMs individuales se visualizaron en imágenes de 10x tomadas por el sistema de imágenes de alto contenido (Figura 2).
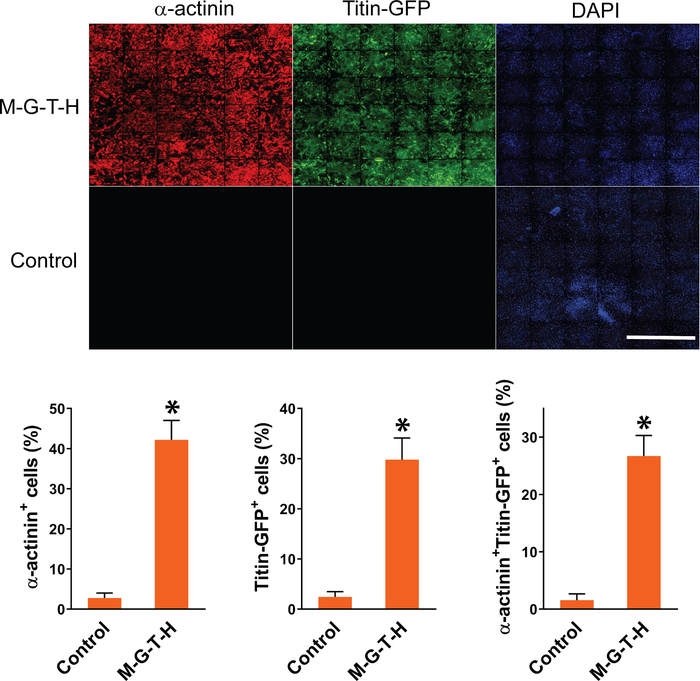
Figura 1: Imágenes compuestas representativas del análisis automatizado de imágenes de alto contenido. Los MEFs aislados de ratones knock-in de Titina-GFP se transducieron con la construcción M-G-T-H o vector vacío. Se utilizaron células transducidas vectoriales vacías como control. Quince días después de la transducción, los MEFs fueron inmunoteñidos para GFP y α-actinina. Las células inmunoteñidas se analizaron mediante un sistema automatizado de imágenes de alto contenido para cuantificar las células DAPI+GFP+, DAPI+α-actinina+ o DAPI+GFP+α-actinina+ . El gráfico muestra un resumen de los análisis de imágenes de alto contenido. Tres experimentos independientes se presentan con una media ±S.D. *p < 0,005. Barra de escala = 2,5 mm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Imágenes representativas de imágenes de objetivos de 10x utilizadas para el análisis de imágenes de alto contenido. Estas imágenes se eligieron de las imágenes compuestas de 36 sitios de imagen, como se muestra en la Figura 1. Cada imagen representa un sitio de imagen individual por cada canal. Barra de escala = 400 μm. Haga clic aquí para ver una versión más grande de esta figura.
| Placa de cultivo celular | Células E de platino (miilión/placa) | Medios (ml) | Fugene 6 (μl) | Opti-MEM (μl) | ADN (μg) |
| 15 cm | 13.75 | 25 | 75 | 750 | 25 |
| 10 cm | 5.5 | 10 | 30 | 300 | 10 |
| 6 cm | 2.2 | 4 | 12 | 120 | 4 |
| 35 mm (6 pocillos) | 1.1 | 2 | 6 | 60 | 2 |
| 12 pozos | 0.55 | 1 | 3 | 30 | 1 |
| 24 pozos | 0.275 | 0.5 | 1.5 | 15 | 0.5 |
Tabla 1: Escala de transfección en función del tamaño de una placa celular.
Tabla 2: Conjunto de datos brutos analizados por un sistema de imágenes de alto contenido. Haga clic aquí para descargar esta tabla.
Tabla 3: Conjunto de datos resumidos. Haga clic aquí para descargar esta tabla.
Discusión
Los estudios de reprogramación anteriores evaluaron la eficiencia de la reprogramación mediante citometría de flujo y demostraron la calidad estructural de los iCM mediante inmunocitoquímica en dos experimentos separados. El análisis de citometría de flujo requiere un número mucho mayor de células iniciales, lo que aumenta la escala de los experimentos. Por el contrario, el análisis de imágenes de alto contenido puede evaluar tanto la calidad como la cantidad de la reprogramación de iCM mediante un solo experimento con un número relativamente pequeño de células. Por lo tanto, este nuevo método puede proporcionar una nueva plataforma técnica eficiente para futuros estudios de reprogramación. En particular, este nuevo método será útil para los experimentos de reprogramación para el cribado de nuevos factores genéticos o farmacológicos. Para probar un gran número de factores, el formato de análisis se puede reducir a un formato de 384 pocillos.
La eficiencia de la transducción está directamente relacionada con la eficiencia de la reprogramación. En estudios previos, hemos demostrado que asegurar la expresión de todos los factores de reprogramación en fibroblastos es importante para lograr una alta eficiencia de reprogramación 8,9. Dado que un número relativamente pequeño de fibroblastos expresaba todos los factores de reprogramación mediante la transducción de vectores virales individuales, el uso de un solo vector policistrónico que albergue todos los factores de reprogramación es beneficioso para mejorar la eficiencia de la reprogramación 7,9. Además, para aumentar la eficiencia de la transducción, realizamos dos transducciones secuenciales utilizando dos transfecciones separadas de vectores de reprogramación en células Platinum E. Encontramos que este método es superior a la transducción secuencial anterior con una sola transfección mediante la reutilización de células de Platino E transfectadas para la segunda transducción.
El éxito de la reprogramación cardíaca se puede definir de diferentes maneras. El método más utilizado fue cuantificar el porcentaje de células que expresan una proteína estructural cardíaca, que está ausente en los fibroblastos iniciales. En este estudio, utilizamos la α-actinina específica del músculo cardíaco, así como la titina específica del músculo estriado como marcadores de la inducción de proteínas estructurales cardíacas durante la reprogramación cardíaca. La razón para el uso de Titin fue que pudimos demostrar más claramente la formación de sarcómeros utilizando fibroblastos aislados de la línea de ratón knock-in Titin-GFP 8,9. En cualquier caso, definimos las iCM como una célula doble positiva (actinina + Titina +) para excluir las células musculares no cardíacas que expresan titina, si las hay. Es importante destacar que cualquier combinación de proteínas estructurales cardíacas se puede utilizar para el análisis de imágenes de alto contenido, siempre que haya anticuerpos específicos disponibles contra esas proteínas. Aunque el método que describimos aquí se basa únicamente en la expresión de proteínas estructurales, es importante reconocer que las células que expresan una proteína estructural cardíaca no son necesariamente cardiomiocitos funcionales. En nuestros estudios previos, hemos demostrado que el ensamblaje de los sarcómeros es necesario para que los iCMs sean funcionales 4,8,9. Es importante destacar que el desarrollo estructural de los iCM se puede examinar directamente a través del análisis de imágenes de alto contenido. Además, se puede realizar posteriormente una evaluación estructural adicional (p. ej., microscopía confocal) utilizando las mismas células inmunoteñidas para el análisis de imágenes de alto contenido. Sin embargo, la verdadera funcionalidad de los MCi debe determinarse mediante el registro de la contracción espontánea y las propiedades electrofisiológicas (es decir, los transitorios de calcio y los potenciales de acción) de los MCI. Además, sería necesario demostrar cambios transcriptómicos completos de fibroblastos a cardiomiocitos para definir la reprogramación exitosa de iCM (p. ej., secuenciación de ARN de una sola célula). Por lo tanto, el objetivo principal del análisis de imágenes de alto contenido para evaluar la reprogramación cardíaca no es cuantificar los iMC funcionales, sino evaluar los avances estructurales generales de la reprogramación del iMC como evaluación inicial.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
El análisis de imágenes de alto contenido se llevó a cabo en las instalaciones centrales de Vanderbilt High-Throughput Screening (HTS) con la asistencia de David Westover y Joshua Bauer. El HTS Core recibe apoyo del Instituto de Biología Química de Vanderbilt y del Centro Oncológico Vanderbilt Ingram (P30 CA68485). Este trabajo fue apoyado por el Premio al Proyecto Innovador de la AHA 18IPA34110341 y el NIH R01 HL146524 (Y-.J. N.), y el premio de beca postdoctoral de la AHA 20POST35210170 (Z.Z).
Materiales
| Name | Company | Catalog Number | Comments |
| A83-01 | Tocris | 2939 | |
| anti-chicken Alexa 488 | Thermofisher | A11039 | |
| anti-GFP antibody | Invitrogen | A10262 | |
| anti-mouse Alexa 555 | Thermofisher | A21422 | |
| anti-α-actinin antibody | Sigma | A7811 | |
| DAPI solution | Vector labs | H1200 | |
| Fugene 6 | Promega | E2691 | |
| Insulin-Transferrin-SeleniumG supplement | Invitrogen | 41400-045 | |
| Medium 199 | Invitrogen | 11150059 | |
| MEM vitamin solution | Invitrogen | 11120-052 | |
| MetaXpress software | Molecular device | ||
| Micro XL automated cell imagining system | Molecular device | ||
| Minimal essential amino acid solution | Sigma | M7145 | |
| Opti-MEM | Gibco | 31905-070 | |
| PES filter (0.45 µm) | Thomas scientific | 1159T84 | |
| Platninum E cells | Cell Biolabs | RV-101 | |
| Polybrene | Sigma | H9268 | |
| SB431542 | Sigma | S4317 | |
| Universal blocking buffer | BiogeneX | HK083-50K |
Referencias
- Ieda, M., et al. Direct reprogramming of fibroblasts into functional cardiomyocytes by defined factors. Cell. 142 (3), 375-386 (2010).
- Song, K., et al. Heart repair by reprogramming non-myocytes with cardiac transcription factors. Nature. 485 (7400), 599-604 (2012).
- Ifkovits, J. L., Addis, R. C., Epstein, J. A., Gearhart, J. D. Inhibition of TGFbeta signaling increases direct conversion of fibroblasts to induced cardiomyocytes. PLoS One. 9 (2), 89678(2014).
- Nam, Y. J., et al. Induction of diverse cardiac cell types by reprogramming fibroblasts with cardiac transcription factors. Development. 141 (22), 4267-4278 (2014).
- Muraoka, N., et al. MiR-133 promotes cardiac reprogramming by directly repressing Snai1 and silencing fibroblast signatures. The EMBO Journal. 33 (14), 1565-1581 (2014).
- Umei, T. C., et al. Single-construct polycistronic doxycycline-inducible vectors improve direct cardiac reprogramming and can be used to identify the critical timing of transgene expression. International Journal of Molecular Sciences. 18 (8), 1805(2017).
- Wang, L., et al. Stoichiometry of Gata4, Mef2c, and Tbx5 influences the efficiency and quality of induced cardiac myocyte reprogramming. Circulation Research. 116 (2), 237-244 (2015).
- Zhang, Z., Zhang, A. D., Kim, L. J., Nam, Y. J. Ensuring expression of four core cardiogenic transcription factors enhances cardiac reprogramming. Science Reports. 9 (1), 6362(2019).
- Zhang, Z., Zhang, W., Nam, Y. J. Stoichiometric optimization of Gata4, Hand2, Mef2c, and Tbx5 expression for contractile cardiomyocyte reprogramming. Science Reports. 9 (1), 14970(2019).
- Zhao, Y., et al. High-efficiency reprogramming of fibroblasts into cardiomyocytes requires suppression of pro-fibrotic signalling. Nature Communications. 6, 8243(2015).
- Zhou, H., Dickson, M. E., Kim, M. S., Bassel-Duby, R., Olson, E. N. Akt1/protein kinase B enhances transcriptional reprogramming of fibroblasts to functional cardiomyocytes. Proceedings of the National Academy of Sciences of the United States of America. 112 (38), 11864-11869 (2015).
- Zhou, H., et al. ZNF281 enhances cardiac reprogramming by modulating cardiac and inflammatory gene expression. Genes & Development. 31 (17), 1770-1783 (2017).
- Zhou, Y., et al. Bmi1 is a key epigenetic barrier to direct cardiac reprogramming. Cell Stem Cell. 18 (3), 382-395 (2016).
- Muraoka, N., et al. Role of cyclooxygenase-2-mediated prostaglandin E2-prostaglandin E receptor 4 signaling in cardiac reprogramming. Nature Communications. 10 (1), 674(2019).
- Yamakawa, H., et al. Fibroblast growth factors and vascular endothelial growth factor promote cardiac reprogramming under defined conditions. Stem Cell Reports. 5 (6), 1128-1142 (2015).
- Abad, M., et al. Notch inhibition enhances cardiac reprogramming by increasing MEF2C transcriptional activity. Stem Cell Reports. 8 (3), 548-560 (2017).
- Protze, S., et al. A new approach to transcription factor screening for reprogramming of fibroblasts to cardiomyocyte-like cells. Journal of Molecular and Cell Cardiology. 53 (3), 323-332 (2012).
- Addis, R. C., et al. Optimization of direct fibroblast reprogramming to cardiomyocytes using calcium activity as a functional measure of success. Journal of Molecular and Cell Cardiology. 60, 97-106 (2013).
- da Silva Lopes, K., Pietas, A., Radke, M. H., Gotthardt, M. Titin visualization in real time reveals an unexpected level of mobility within and between sarcomeres. The Journal of Cell Biology. 193 (4), 785-798 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados