Method Article
Herramientas de ARNi específicas de tejidos para identificar componentes para la señalización de estrés sistémico
En este artículo
Resumen
El mantenimiento de la proteostasis del organismo requiere la coordinación de las respuestas de control de calidad de las proteínas, como la expresión de chaperonas, de un tejido a otro. Aquí, proporcionamos herramientas utilizadas en C. elegans que permiten monitorear la capacidad de proteostasis en tejidos específicos y determinar las respuestas de señalización intercelular.
Resumen
Durante la última década se ha producido un aumento transformador en el conocimiento en torno a la regulación de los procesos de control de calidad de las proteínas, lo que revela la importancia de los procesos de señalización intercelular en la regulación de la proteostasis no autónoma de las células. Estudios recientes están comenzando a descubrir los componentes de señalización y las vías que coordinan el control de la calidad de las proteínas de un tejido a otro. Por lo tanto, es importante identificar los mecanismos y componentes de la red de proteostasis (NP) no autónoma de las células y su relevancia para el envejecimiento, las respuestas al estrés y las enfermedades de mal plegamiento de proteínas. En el laboratorio, utilizamos la eliminación genética por ARNi específico de tejido en combinación con reporteros de estrés y sensores de proteostasis específicos de tejido para estudiar esto. Describimos metodologías para examinar e identificar componentes de la NP celular no autónoma que pueden actuar en los tejidos que perciben una condición de estrés y en las células que responden para activar una respuesta protectora. En primer lugar, describimos cómo generar construcciones de ARNi de horquilla para la eliminación genética constitutiva en tejidos específicos y cómo realizar la eliminación genética específica de tejido alimentando ARNi en diferentes etapas de la vida. Los reporteros de estrés y los ensayos de comportamiento funcionan como lecturas valiosas que permiten la detección rápida de genes y condiciones que modifican los procesos de señalización del estrés sistémico. Finalmente, se utilizan sensores de proteostasis expresados en diferentes tejidos para determinar cambios en la capacidad tisular específica de la NP en diferentes etapas de desarrollo y envejecimiento. Por lo tanto, estas herramientas deberían ayudar a clarificar y permitir el seguimiento de la capacidad de la NP en tejidos específicos, al tiempo que ayudan a identificar los componentes que funcionan en diferentes tejidos para mediar la NP no autónoma de células en un organismo.
Introducción
La proteostasis celular es monitoreada por una intrincada red de componentes de control de calidad de proteínas, como chaperonas moleculares, respuestas al estrés y mecanismos de degradación, incluido el sistema de proteasoma de ubiquitina (UPS) y la autofagia 1,2. La activación de las vías de respuesta al estrés, como la respuesta de choque térmico mediada por HSF-1 (HSR), la respuesta proteica desplegada del retículo endoplásmico (UPRER) y las mitocondrias (UPRmito) es vital para la adaptación celular y la supervivencia durante los desafíos ambientales o la enfermedad de plegamiento incorrecto de proteínas que conducen a la agregación de proteínas tóxicas 1,2,3,4,5, 6.
La proteostasis celular está coordinada por una capa adicional en organismos multicelulares, como C. elegans, que requiere la orquestación de respuestas de estrés celular en diferentes tejidos para activar componentes protectores de control de calidad de proteínas, como las chaperonas moleculares7. En la última década, se ha observado la activación no autónoma de las vías de respuesta al estrés "celulares" para la respuesta al choque térmico (HSR), elRE UPR y elmito UPR, así como la señalización de chaperonas transcelulares (TCS)3,4,7,8,9,10. En cada caso, el sistema nervioso, así como la señalización del intestino, desempeña un papel crucial en el control de la activación de chaperonas a través de los tejidos, para proteger contra las consecuencias tóxicas del estrés agudo y crónico por plegamiento incorrecto de proteínas 3,5,9,11. Esta transmisión de las neuronas al intestino y a otras células de la periferia puede ser lograda por neurotransmisores, como es el caso delRE UPR y el HSR 6,8,11. En una forma de señalización de estrés no autónoma de las células, TCS, que se activa por el aumento de la expresión de HSP-90 en las neuronas, los péptidos inmunes secretados desempeñan un papel en la activación de la expresión de chaperonas hsp-90 de las neuronasal músculo. En otra forma de TCS, la reducción de la expresión de la chaperona molecular principal hsp-90 en el intestino conduce a un aumento de la expresión de hsp-70 inducible por calor a temperatura permisiva en el músculo de la pared corporal 5,10. Sin embargo, en este caso particular, se desconocen las moléculas de señalización específicas activadas en el intestino que percibe el estrés y las células musculares que responden.
Por lo tanto, para identificar cómo se activa la expresión de chaperonas de un tejido a otro, se requiere un enfoque que permita monitorear la capacidad de la red de proteostasis (NP) y la activación de la respuesta al estrés a nivel específico del tejido. Para investigar qué vía de respuesta al estrés se activa en los tejidos individuales, se puede utilizar una selección disponible de reporteros de chaperonas transcripcionales fusionados con marcadores de proteínas fluorescentes (véase también la Tabla 3). Estos incluyen reporteros transcripcionales hsp-90, hsp-70 y hsp-16.2 marcados con fluorescencia que indican la inducción del HSR, hsp-4 que indica la activación delRE UPR y hsp-6, que indica elmito UPR. La combinación de estos reporteros con una condición de estrés específica del tejido permite una lectura potente que identificará los tejidos individuales que responden a un desequilibrio de la NP en un tejido "emisor" distal que percibe el estrés. Para inducir una condición de estrés o desequilibrio de la NP en un tejido específico, se pueden tomar diferentes enfoques. Por ejemplo, uno de estos enfoques es mediante la expresión ectópica de la forma activada de un factor de transcripción de estrés (p. ej., xbp-1s) y otro es mediante la reducción de los niveles de expresión de una chaperona molecular esencial (p. ej., hsp-90) utilizando promotores específicos de tejido 8,10. Para agotar los componentes de PN en un solo tipo de célula, la eliminación específica de tejidos por ARNi es una herramienta útil.
Sin embargo, en C. elegans, el ARNi es sistémico; el ARN bicatenario en el medio ambiente puede entrar y propagarse por todo el animal para silenciar un gen objetivo12,13. Esta diseminación sistémica del dsRNA ingerido está mediada por proteínas SID (ARNi sistémico defectuoso), como las proteínas SID-1 y SID-2 que son transportadoras de dsRNA, así como por el SID-5, que se colocaliza con las proteínas del endosoma tardío y está implicado en la exportación del dsRNAingerido 14,15,16. SID-1 es una proteína transmembrana de múltiples pasos en todas las células, excepto en las neuronas, y es necesaria para la exportación e importación de dsRNA a las células17. La expresión de SID-2 está restringida al intestino, donde funciona como un receptor endocítico para el dsRNA ingerido desde la luz intestinal hacia el citoplasma de las células intestinales16. Las neuronas carecen de una respuesta al ARNi sistémico, y esto se correlaciona con una expresión reducida de la proteína transmembrana SID-1 en las neuronas, que es esencial para que el dsRNA sea importado15,18. Por lo tanto, para que el ARNi específico de tejido sea eficaz en un solo tipo de célula, es necesario evitar la propagación sistémica del dsRNA. Esto se puede lograr utilizando el mutante sid-1 resistente al ARNi (pk3321) que impide la liberación y absorción de dsRNA a través de los tejidos15. La expresión de una construcción de ARNi de horquilla específica de tejido en este mutante o la expresión ectópica de SID-1 en un tejido específico puede complementar la función de SID-1 mutante y permitirá un ARNi19 específico de tejido.
Entonces, ¿cómo es el dsRNA ingerido por el intestino en un mutante de pérdida de función SID-1 y cómo puede llegar a las neuronas o células musculares que expresan ectópicamente una construcción SID-1? En un modelo actual que explica este mecanismo, el dsRNA endocitado se absorbe en el citoplasma intestinal a través de SID-2 y luego se exporta al pseudoceloma por otro mecanismo independiente de SID-1, que involucra SID-5 y transcitosis17. Por lo tanto, debido a que SID-1 es necesario para la importación de dsRNA17, solo las células que expresan SID-1 de tipo salvaje podrán absorber el dsRNA liberado desde el intestino en el pseudoceloma.
Aquí demostramos el uso de un conjunto de herramientas que permiten el ARNi específico de tejido. Utilizamos el ejemplo de la chaperona molecular Hsp90 para describir la construcción de ARNi de horquilla que puede ser útil para derribar constitutivamente la expresión génica en un tejido específico10. El enfoque descrito podría utilizarse para cualquier gen diana de interés. La respuesta de otros tejidos al desequilibrio de la proteostasis causado por el ARNi hsp-90 específico del tejido puede sondearse mediante el seguimiento de la expresión de reporteros de estrés marcados con fluorescencia en otros tejidos. Como segundo método para el ARNi específico de tejido, demostramos cómo el sistema mutante sid-1 se puede adaptar para alimentar a bacterias que expresan ARNi en lugar de expresar una construcción de ARNi de horquilla. Esto puede ser útil cuando se realiza un cribado de ARNi candidato o de todo el genoma para identificar los componentes necesarios para una respuesta específica de tejido. Del mismo modo, los defectos de desarrollo asociados con el agotamiento de un componente vital de la NP requerirán la eliminación mediada por ARNi en tejidos específicos en etapas posteriores del desarrollo. Demostramos cómo se puede utilizar un sistema de complementación de SID-1 en un cribado de ARNi candidato para modificadores TCS específicos de tejido. En el ejemplo, nuestro objetivo es identificar los componentes de señalización que, al ser golpeados en el tejido emisor "que percibe el estrés" (intestino) y el tejido que afecta el estrés (músculo) conducen al cambio en la expresión de un reportero hsp-70 marcado con fluorescencia en las células musculares.
Protocolo
1. ARNi específico de tejido de dos maneras: ARNi de horquilla y complementación de SID-1 específico de tejido
- Generación de construcciones de ARNi de horquilla para la expresión específica de tejido en mutantes sid-1
- Amplifique la secuencia del gen diana (por ejemplo, la secuencia hsp-90 aislada del clon de ARNi hsp-90 de la biblioteca de ARNi de Ahringer20) mediante PCR. Coloque una secuencia no palindrómica en el extremo 3' de la secuencia hsp-90 , es decir, un sitio SfiI (ATCTA)21.
NOTA: Los cebadores utilizados para clonar el hsp-90 con la secuencia SfiI (subrayados) son:
as-hsp90-SfiI 5'-GGCCATCTAGGCCCTGGGTTGATTTCGAGATGCT-3'
as-hsp90 5' TCATGGAGAACTGCGAAGAGC-3'. - Subclonar la secuencia amplificada en el vector del kit de clonación comercial (por ejemplo, TOPO pCR BluntII).
- Aislar la secuencia hsp-90 invertida del clon de ARNi hsp-90 (biblioteca de ARNi de Ahringer)20 mediante digestión de restricción utilizando sitios de restricción XbaI y PstI y colocarla aguas abajo de la secuencia hsp-90-SfiI en el vector (desde el paso 1.1.1), dando como resultado una construcción de horquilla hsp-90 (Figura 1).
- Subclonar la construcción de horquilla en un vector de entrada de Gateway pDONR221 y fusionarla con clones de entrada de Gateway que contienen promotores específicos de tejido para cualquiera de las expresiones en neuronas (rgef-1p); en el intestino (VHA-6P); o el músculo de la pared corporal (unc-54p) y el unc-54 3'UTR (o cualquier otro 3'UTR de elección) en una reacción de puerta de enlace como se describe en el protocolo del proveedor.
- Linealizar las construcciones de ARNi de horquilla resultantes (Figura 1) utilizando un sitio de restricción único fuera de la secuencia codificante y microinyectar como una matriz compleja a una concentración de 1 ng/μL de construcción de ARNi de horquilla, mezclado con 100 ng/μL de ADN genómico N2 Bristol (digerido con ScaI) en una cepa de C. elegans que expresa el reportero hsp-70p::RFP (cepa AM722) y cruzada con el fondo genético de sid-1 (pk3321) mutantes (cepa NL3321). Para obtener un protocolo sobre cómo realizar la microinyección de matrices complejas, sigael 22.
- Como control negativo, utilice construcciones de horquilla de vector vacío que expresen la secuencia que contiene SfiI no palindrómica (GGCCATCTAGGCC) bajo el control de un promotor específico de tejido.
- Utilice el aumento de la expresión de hsp-70p::RFP del reportero como lectura para puntuar los transformantes positivos que expresan el ARNi de horquilla de hsp-90 (Figura 3). Para un enfoque más general para verificar la eliminación específica del tejido de cualquier gen de interés, mida los niveles de ARNm de animales enteros mediante qRT-PCR del gen de interés.
- Integrar la matriz extracromosómica de la cepa resultante que expresa la construcción de horquilla hsp-90 específica del intestino (PVH2; ver Tabla 1) mediante irradiación gamma. Para la integración de las matrices extracromosómicas en el genoma, véase22.
- Amplifique la secuencia del gen diana (por ejemplo, la secuencia hsp-90 aislada del clon de ARNi hsp-90 de la biblioteca de ARNi de Ahringer20) mediante PCR. Coloque una secuencia no palindrómica en el extremo 3' de la secuencia hsp-90 , es decir, un sitio SfiI (ATCTA)21.
- Expresión de SID-1 específica de tejido para permitir ARNi específico de tejido mediante la alimentación de bacterias que expresan dsRNA
- Subclonar el ADN genómico sid-1 del vector TU867 (unc-119p::SID-1)19 en el vector de entrada de puerta de enlace pDONR221. Los cebadores para la clonación de ADN sid-1 se pueden encontrar en19. Fusionar la construcción sid-1 pDONR221 con clones de entrada Gateway que contengan promotores específicos de músculo (myo-3p) o intestino (vha-6p) y el unc-54 3'UTR (o cualquier otro 3'UTR de elección) en la reacción de Gateway como se describe anteriormente en 1.1.4.
- Microinyecte las construcciones resultantes vha-6p::SID-1::unc-54 3'UTR o myo-3p::SID-1::unc-54 3'UTR a una concentración de 30 ng/μL junto con un marcador de coinyección faríngeo fluorescente rojo (p. ej., myo-2p::RFP; 5 ng/μL) en mutantes sid-1(pk3321).
- Integrar las matrices extracromosómicas de sid-1 específicas del intestino o del músculo en el genoma como se describe en elartículo 22. En este caso, esto dio lugar a las cepas PVH5 [myo-3p::SID-1; myo-2p::RFP]; sid-1(pk3321) y PVH65 [vha-6p::SID-1; myo-2p::RFP]; SID-1 (PK3321).
- Para la expresión neuronal específica de sid-1 en el mutante sid-1(pk3321), use la cepa TU3401 uIs3401[unc-119p::SID-1; myo-2p::RFP]; sid-1(pk3321) que fue generado previamente por Calixto et al.19.
- Como se mencionó en 1.1.7, asegure la eliminación específica del tejido del gen de interés midiendo los niveles de ARNm del gen diana deseado mediante qRT-PCR. Alternativamente, confirme la sensibilidad del ARNi específico del tejido mediante el uso de una proteína fluorescente (por ejemplo, GFP o RFP) expresada en el mismo tejido y trate los gusanos con GFP o RFP RNAi. Exponga los nematodos a GFP/RFP RNAi como larvas sincronizadas en etapa L1 y crezca en las bacterias RNAi hasta el día 1 de la edad adulta (ver Figura 2). En nuestro caso, utilizamos cepas que expresan SID-1 en las neuronas, el músculo o el intestino y las cruzamos en cepas que expresan HSP-90::RFP en las neuronas (AM987), en el intestino (AM986) y en el músculo (AM988).
2. Uso de reporteros de estrés y sensores de proteostasis para monitorear la proteostasis celular autónoma y no autónoma de la célula
NOTA: Para monitorizar la capacidad de la NP en tejidos específicos, utilice sensores de proteostasis específicos de tejido (como las cepas que expresan Q44 en el intestino o Q35 en el músculo – ver Tabla 3) y reporteros de estrés (como el reportero hsp-70p::mCherry inducible por calor; Tabla 3).
- Cruzando genéticamente el alelo mutante sid-1 (pk3321) en una cepa sensora de proteostasis y confirmando la presencia de sid-1 (pk3321) alimentando ARNi
- Cruzar genéticamente la cepa del sensor de proteostasis/reportero de estrés con el fondo genético de la cepa mutante sid-1 (pk3321). Para establecer cruzamientos genéticos entre diferentes cepas transgénicas, siga el23 para obtener un protocolo detallado.
NOTA: Los mutantes sid-1(pk3321) son resistentes a la alimentación con ARNi y, por lo tanto, el tratamiento de embriones con ARNi contra un gen esencial (como elt-2 o hsp-90) solo conducirá a detenciones del desarrollo o letalidad larvaria en cepas heterocigóticas o de tipo salvaje para el gen sid-1 . - Deje que 10 hermafroditas grávidos pongan huevos en placas de ARNi contra elt-2 o hsp-90 y controle (vector vacío; EV) Placas de ARNi a 20 °C. Retire las madres después de 1 a 2 h. Utilice N2 Bristol y el mutante sid-1(pk3321) como controles.
- Observe el desarrollo de las larvas en las placas de ARNi durante los próximos 2-3 días. ELT-2 El ARNi dará lugar a la detención de las larvas L1, mientras que el ARNi de hsp-90 dará lugar a la detención de las larvas L3 en N2 Bristol. Los mutantes sid-1 no se verán afectados por el tratamiento con ARNi y se convertirán en adultos grávidos.
NOTA: El homocigoto de C. elegans para sid-1 (pk3321) mostrará una población uniforme que se desarrolla hasta la edad adulta. Los heterocigotos estarán indicados por poblaciones mixtas de algunos animales que muestran detención larvaria, y algunos animales que se convierten en adultos.
- Cruzar genéticamente la cepa del sensor de proteostasis/reportero de estrés con el fondo genético de la cepa mutante sid-1 (pk3321). Para establecer cruzamientos genéticos entre diferentes cepas transgénicas, siga el23 para obtener un protocolo detallado.
- Confirmación de la presencia de sid-1(pk3321) por genotipado
- Recoger 15-20 gusanos de la cepa F2 candidata seleccionada en un tubo de PCR que contenga 15 μL de tampón de lisis de lombrices (Tabla 2).
- Coloque el tubo a -80 °C durante al menos 10 minutos o toda la noche.
- Incubar el tubo en la máquina de PCR utilizando el siguiente programa:
- 65 °C durante 60 min (gusano de lisis); 95 °C durante 15 min (inactiva la proteinasa K); mantener a 4 °C.
- Utilice 2 μL del lisado de lombriz como "plantilla" para realizar la reacción de PCR para el genotipado, utilizando los siguientes cebadores para sid-1: sid-1 forw: 5'-agctctgtacttgtattcg-3' y sid-1 rev: 5'-gcacagttatcagatttg-3'.
- Utilice el siguiente programa para el genotipado por PCR: 1 ciclo a 95 °C durante 3 min; luego 30 ciclos de 95 °C durante 10 , 55 °C durante 30 s y 72 °C durante 30 s; 1 ciclo a 72 °C durante 10 min, mantener a 4 °C.
- Purifique el producto de PCR de ~650 pb utilizando un kit de purificación de PCR (Tabla de materiales) y secuencie el producto de PCR sid-1 para identificar la mutación puntual G-a-A del alelo sid-1 (pk3321). Alternativamente, la mutación puntual G-a-A crea un sitio de restricción de ApoI, que se puede utilizar en el producto de PCR para el genotipado, como se describe en elpunto 24.
- Uso de iQ44::YFP como sensor de proteostasis para el intestino
- Sincronizar C. elegans expresando Q44::YFP en el intestino (cepa OG412) o cruzado con el fondo mutante sid-1(pk3321) mediante blanqueo, siguiendo el protocolo descrito en25. Coloque las larvas L1 sincronizadas en una placa de agar y medio de crecimiento de nematodos (NGM) de 9 cm que contenga la bacteria OP50 y crezca hasta la etapa L4 a 20 °C.
- Recoge animales L4 lavando los gusanos del plato con 5 mL de tampón M9. Transfiera el tampón M9 que contiene lombrices L4 a un tubo de 15 ml con una pipeta de vidrio o una pipeta de plástico siliconado, y centrifugue a 1000 x g durante 1 minuto a temperatura ambiente para granular suavemente las lombrices. Retire el sobrenadante con cuidado, asegurándose de dejar la pelletza de lombriz intacta.
- Paso crítico: Para transferir o eliminar nematodos, use una pipeta de vidrio o una punta de pipeta de plástico que haya sido tratada con un agente siliconante (por ejemplo, SigmaCote) siguiendo las instrucciones del fabricante. Esto evita que los gusanos se peguen a la superficie de plástico de la punta de una pipeta.
- Repita el paso 2.3.2 tres veces más para lavar todas las bacterias OP50 de los gusanos.
- Tome el gránulo de lombriz en 5 mL de tampón M9 y cuente el número de gusanos presentes en 10 μL.
- Colocar los animales L4 en placas de agar NGM de 6 cm que contengan bacterias de control de vectores vacíos (EV) o ARNi hsp-90 a una densidad de 10 gusanos por placa (preparar 5 placas por punto de tiempo y replicar biológicamente) e incubar durante 24 -48 horas a 20 °C.
- Después de 24 horas (=Día 1 adultos) y 48 horas (=Día 2 adultos) se cuenta el número de focos Q44 en los intestinos de los nematodos que exhiben agregados. Puntúa un total de 30-50 nematodos por réplica biológica.
3. Cribado de ARNi candidato a tejido específico para modificadores de la proteostasis celular no autónoma
NOTA: Para el cribado de ARNi específico de tejido, utilizamos la cepa PVH172, que permite el ARNi específico del intestino mediante la alimentación de bacterias RNAi y la cepa PVH171, que permite el ARNi específico del músculo (véase la Tabla 1 para el genotipo).
- Preparación de las placas candidatas de ARNi
- Prepare placas de agar NGM de 6 cm suplementadas con 100 μg/mL de ampicilina, 12,5 μg/mL de tetraciclina y 1 mM de IPTG de acuerdo con los métodos estándar25.
- Utilice la biblioteca de ARNi de Ahringer para obtener los clones de ARNi candidatos para el cribado de ARNi20.
- Inocular 3 mL de medio LB-amp (50 μg/mL de ampicilina en medio LB) en un tubo de 15 mL con el clon de ARNi deseado utilizando una punta de pipeta de plástico. Crecer a 37 °C durante la noche con agitación.
- Al día siguiente, agregue isopropil-b-D-tiogalactopiranosida (IPTG) (de un stock de 1 M) a una concentración final de 1 mM en el cultivo bacteriano durante la noche.
- Agitar los cultivos durante 3 h más a 37 °C.
- Placa de 300 μL de cultivo de ARNi bacteriano en una placa de agar NGM de 6 cm suplementada con 100 μg/mL de ampicilina, 12,5 μg/mL de tetraciclina y 1 mM de IPTG. Deje que las placas se sequen en el banco durante 2 días a temperatura ambiente, cubiertas con papel de aluminio para protegerlas de la luz. Una vez secas, las placas de ARNi pueden almacenarse en una caja a 4 °C durante varias semanas.
- Sincronización de C. elegans y tratamiento con bacterias RNAi
- Para sincronizar las cepas de gusanos, elija 15 adultos grávidos en placas de ARNi y deje poner huevos durante 1 h. A continuación, retira los adultos del plato.
- Paso crítico: En este caso, se evita la sincronización por blanqueo, ya que puede inducir el reportero hsp-70p::RFP , ya que es una condición estresante para C. elegans.
- Elija las larvas sincronizadas en etapa L4 y transfiéralas a una placa de ARNi fresca.
- Permitir que los nematodos crezcan en el ARNi relevante durante dos generaciones para garantizar una absorción eficiente del dsRNA, asegurándose de que la temperatura se mantenga a 20 °C.
- Para la obtención de imágenes y la cuantificación de fluorescencia hsp-70p::RFP , utilice adultos del día 1.
- Preparación de portaobjetos de microscopio
- Prepare los portaobjetos del microscopio colocando ~250 μL de una solución de agarosa al 2% (en tampón M9) sobre un portaobjetos de microscopio de vidrio y coloque un segundo portaobjetos encima para crear un disco plano.
- Coloque 5 μL de solución de 5 mM de levamisol (en tampón M9) en la almohadilla de agarosa establecida y transfiera 5 gusanos adultos del Día 1 a la gota de Levamisol. Deja que los nematodos se paralicen durante 5 min.
- Una vez que C. elegans esté paralizado, alinéelo cuidadosamente con un pico de alambre de platino y elimine el exceso de levamisol con una toallita de laboratorio antes de agregar un cubreobjetos.
- Paso crítico: Asegúrese de tomar imágenes de los gusanos dentro de los 30 minutos posteriores a la preparación de los portaobjetos del microscopio. Los nematodos paralizados en el portaobjetos del microscopio pueden secarse y reventar, lo que puede comprometer las mediciones de fluorescencia.
- Ajustes del microscopio y análisis de imágenes
NOTA: Las imágenes se obtienen utilizando un microscopio confocal equipado con una cámara EM-CCD y un software de automatización y análisis de imágenes de microscopía.- Tome imágenes con aumentos de 10x utilizando un láser de 561 nm para la excitación de fluorescencia RFP. Asegúrese de que todas las imágenes se tomen con los mismos ajustes de potencia láser, tamaño de orificio y ganancia de fluorescencia para permitir las comparaciones.
- Guarde todas las imágenes como archivos TIFF.
- Realice análisis de imágenes con ImageJ. Mida la intensidad de fluorescencia en cada imagen como píxeles por unidad de área, restando la fluorescencia de fondo. Normalice la intensidad de fluorescencia de cada imagen al área de la imagen, así como la longitud de los gusanos.
- Mida la intensidad media con Analizar | Medir en ImageJ. Normalice el valor de intensidad resultante al área de la imagen dividiendo la intensidad por el área.
- Para normalizar la intensidad de la longitud del gusano, mida el gusano dibujando una línea a lo largo de la longitud del gusano en ImageJ y utilizando Analizar | Medir. La razón para normalizar la intensidad de la fluorescencia a la longitud del gusano es que los gusanos pueden variar en tamaño, dependiendo del gen que es derribado por el ARNi, y esto podría afectar la intensidad media.
- Normalizar las intensidades de fluorescencia medidas a controles no tratados (es decir, C transgénico . elegans cultivados en placas de ARNi de control (EV). Agrupe los valores normalizados para comparar las intensidades medias de fluorescencia para cada condición de ARNi. El objetivo es obtener imágenes de 20 gusanos por réplica biológica y recopilar al menos 3 imágenes de réplicas biológicas.
- Calcule los valores P de los valores medios de intensidad de fluorescencia utilizando la prueba t de Student y realice una corrección para pruebas múltiples utilizando el método de Benjamini-Hochberg, utilizando una tasa de falsos descubrimientos de 0,05.
Resultados
ARNi específico de tejido de dos maneras: Expresión de construcciones de horquilla o complementación de SID-1 específica de tejido
La expresión de construcciones de ARNi de horquilla específicas de tejido permite la eliminación constitutiva de un gen a lo largo del desarrollo. Sin embargo, esto a veces puede ser poco práctico cuando el gen estudiado es necesario para la organogénesis de ese tejido en particular, como elt-2 , que se requiere para el desarrollo del intestino26. La expresión de SID-1 específica de tejido en los mutantes sid-1 resistentes al ARNi tiene la ventaja particular de que la eliminación de genes específicos de tejido puede programarse en etapas posteriores del desarrollo. En ambos casos (para la expresión de una construcción de horquilla o la complementación de SID-1 específica de tejido), es necesario validar la eficiencia del ARNi específico de tejido para confirmar que solo el tejido objetivo está afectado por el ARNi. Esto se logra mediante la coexpresión de una proteína marcada con fluorescencia, como HSP-90, fusionada con RFP (HSP-90::RFP) en diferentes tejidos.
Cruzamos genéticamente mutantes de sid-1(pk3321) solos o mutantes de sid-1 que expresan SID-1 en neuronas, intestino o músculo de la pared corporal con C. elegans que expresan HSP-90::RFP en las neuronas (Figura 2A), el intestino (Figura 2B) o el músculo (Figura 2C). Las cepas resultantes se trataron con ARNi de hsp-90 en estadio L4 durante 24 horas y la expresión de HSP-90::RFP en tejidos específicos se examinó mediante microscopía de fluorescencia.
Losanimales neuro HSP-90::RFP que expresan SID-1 en las neuronas (unc-119p::SID-1) exhiben una expresión reducida de HSP-90::RFP en las neuronas de la región de cola ampliada (Figura 2A). Del mismo modo, los animales HSP-90::RFPint que expresan SID-1 en el intestino (vha-6p::SID-1) muestran una expresión reducida de HSP-90::RFP en el intestino como se esperaba (Figura 2B), mientras que los mutantes de sid-1(pk3321) que expresan HSP-90::RFP en el intestino no se ven afectados por la alimentación con ARNi de hsp-90. La expresión intestinal de HSP-90::RFP tampoco se ve afectada en los animales que expresan SID-1 en las neuronas o en el músculo de la pared corporal, lo que indica que el dsRNA no se está propagando desde el músculo o las neuronas al intestino (Figura 2B). Por el contrario, los animalesmusculares HSP-90::RFP que expresan SID-1 en el músculo (myo-3p::SID-1) exhiben una expresión reducida de HSP-90::RFP en el músculo durante el ARNi de hsp-90, mientras que los niveles de HSP-90::RFP no se ven afectados en los gusanos que expresan SID-1 en las neuronas o el intestino (Figura 2C).
Uso de un indicador de estrés para un cribado de ARNi candidato a tejido específico
La reducción de la función o expresión de la chaperona molecular principal HSP-90 induce la respuesta al choque térmico y la expresión de la chaperona HSP-7027. El ARNi de hsp-90 específico de tejido en el intestino de C. elegans da lugar a una fuerte regulación positiva de hsp-70 en el músculo, así como en otros tejidos, como lo indica el reportero de hsp-70 inducible por estrés (hsp-70p::RFP) (Figura 3A)10. hsp-70 es un chaperona inducible por calor, por lo que no se puede observar expresión en animales cultivados a temperatura permisiva (20 °C) (Figura 3A)10,28. La exposición al estrés térmico (35 °C) induce la expresión de hsp-70, como lo indica la expresión de RFP del reportero en la faringe, la espermateca, el intestino y el músculo de la pared corporal (Figura 3A). Esto se puede cuantificar midiendo la intensidad de fluorescencia de RFP utilizando el software Image J (Figura 3B), como se describe en el paso 3.4 de la sección de protocolo.
La eliminación constitutiva de hsp-90 en el intestino por el ARNi de horquilla de hsp-90 activa TCS10 y da lugar a una regulación ascendente de 20 veces del reportero hsp-70p::RFP principalmente en las células musculares de la pared corporal (pero no detectable en otros tejidos) a temperatura permisiva (Figura 3A, 3B).
Para identificar los componentes que desencadenan la inducción mediada por TCS de la expresión de hsp-70 desde el intestino hasta el músculo, realizamos un cribado de ARNi candidato específico de tejido de 58 genes candidatos (véase la Figura 4A para ver un diagrama de flujo experimental). Los genes candidatos se identificaron en un análisis previo de cribado genético directo y transcriptoma como posibles modificadores de TCS en la cepa hp-RNAi hp-90int (Figura 4); y consistía en componentes implicados en los procesos de señalización celular, como quinasas, factores de transcripción y proteínas de membrana. A continuación, queríamos determinar en qué tejido los genes candidatos actuaban como potenciadores o supresores de la expresión de hsp-70 inducida por TCS en el músculo. Esto se logra midiendo la intensidad de fluorescencia reducida (potenciadora) o aumentada (supresor) de hsp-70p::RFP del reportero.
Para realizar el cribado de ARNi específico de tejido, primero cruzamos genéticamente la cepa hsp-90int hp-RNAi con C. elegans que expresa SID-1 en el intestino o en el músculo. La expresión intestinal de SID-1 permite detectar posibles componentes de señalización de TCS que actúan en el intestino para mediar el TCS, que es el tejido que percibe el estrés como niveles reducidos de hsp-90. Del mismo modo, la expresión de SID-1 específica del músculo permite el cribado de los componentes de TCS necesarios en el tejido muscular que responde. Los 58 genes candidatos utilizados para el cribado de ARNi específico de tejido se denominaron txt (tcs-(x)cross-tissue).
Los animales se cultivaron en placas de ARNi durante dos generaciones hasta el día 1 de la edad adulta y la intensidad de la fluorescencia de hsp-70p::RFP en el músculo se midió mediante el software ImageJ. Como se muestra en la Figura 4, la eliminación mediada por ARNi de 58 genes txt candidatos en el intestino (Figura 4B) o el músculo (Figura 4C) dio lugar a una serie de modificadores que suprimen o mejoran la inducción de hsp-70 en el músculo. El ARNi de los candidatos que dan lugar a un aumento significativo de la intensidad de la fluorescencia de hsp-70p::RFP indica que el gen actúa como un supresor celular no autónomo de TCS, mientras que una reducción de la intensidad de la fluorescencia de RFP indica que el gen candidato funciona como un potenciador. Los resultados obtenidos (potenciadores/supresores) pueden confirmarse midiendo su efecto sobre los niveles de ARNm de hsp-70 endógenos mediante qRT-PCR y utilizando sensores de proteostasis.
Uso de sensores de proteostasis para monitorizar la capacidad de NP específica del tejido.
La inducción de la expresión de hsp-70 mediada por TCS protege contra el mal plegamiento y la agregación de proteínas de forma no autónoma 5,10. Los sensores de proteostasis se pueden utilizar para estudiar la capacidad de plegamiento en diferentes tejidos en condiciones de estrés. Entre ellas se encuentran las proteínas endógenas y metaestables como, por ejemplo, una proteína condicional (sensible a la temperatura; ts) mutante de miosina expresado exclusivamente en el músculo de la pared corporal (unc-54(ts))29 o proteínas que contienen tramos expandidos de glutamina (PolyQ)30,31,32 (ver Tabla 3 para una lista de cepas). Las proteínas con una longitud de 35-40 glutaminas son particularmente útiles para este propósito, ya que se agregan de manera dependiente de la edad y el estrés y, por lo tanto, son muy adecuadas para informar sobre el entorno de plegamiento en tejidos específicos. Estos incluyen cepas que expresan Q40::YFP en las neuronas o Q35::YFP en el músculo y Q44::YFP en el intestino 30,31,32. Además de utilizar la agregación de PolyQ como lectura, las cepas que expresan Q40::YFP o Q35::YFP también exhiben un defecto de motilidad dependiente de la edad30, lo que permite la cuantificación de la motilidad mediante la medición de las tasas de agitación de manera automatizada (consulte 33 para obtener un ejemplo detallado).
Aquí, coexpresamos Q44::YFP32 intestinal en cepas que permiten ARNi específico de tejido a través de la complementación de SID-1. La eliminación mediada por ARNi de hsp-90 en la etapa L4 en las neuronas, el intestino o el músculo de la pared corporal, que induce TCS10, resultó en una menor acumulación de agregados intestinales de Q44 en los adultos del día 2 en comparación con los animales de control (Figura 5). Por lo tanto, esto indica que la regulación positiva no autónoma de la expresión de hsp-70 en células mediada por TCS protege contra el mal plegamiento de proteínas asociado a la edad en múltiples tejidos de C. elegans.

Figura 1. ARNi de horquilla para la eliminación de genes constitutivos en tejidos específicos. (A) Las repeticiones invertidas de hsp-90 se generan por ligadura cabeza-cabeza a través de un sitio SfiI (azul) introducido en un extremo de cada repetición. Las repeticiones invertidas están bajo el control de un promotor específico de tejido para la expresión específica del músculo (unc-54p), la neurona (rgef-1p) o el intestino (vha-6p). (B) La expresión específica de tejido de las repeticiones hsp-90 invertidas producirá ARN de bucle de horquilla que induce ARNi específico de tejido en una cepa con un fondo genético mutante sid-1 (pk3321). Haga clic aquí para ver una versión más grande de esta figura.
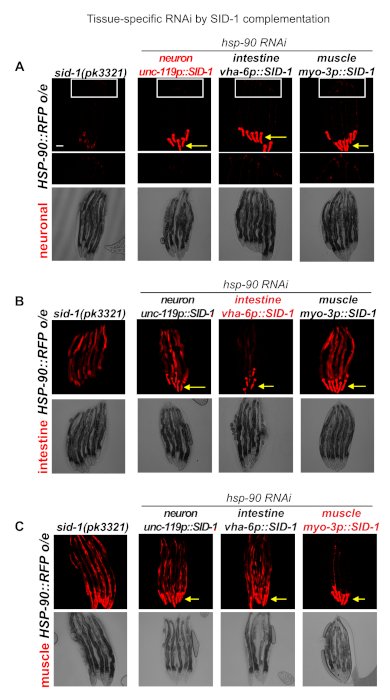
Figura 2. Expresión tisular específica de SID-1 para mejorar la eliminación mediada por ARNi selectivo en tejidos. (A) Sobreexpresión de HSP-90::RFP en las neuronas de mutantes sid-1(pk3321) resistentes al ARNi. Expresión de SID-1 en las neuronas (unc-119p::SID-1) (cepa PVH16); en el intestino (vha-6p::SID-1) (cepa PVH17); y músculo (myo-3p::SID-1) (cepaPVH18) mejora la sensibilidad del ARNi en estos tejidos específicos. Los animales fueron expuestos a bacterias hsp-90 RNAi después de la etapa L4 durante 24 horas, lo que llevó a una reducción visible de la intensidad de fluorescencia de HSP-90::RFP neuronal-específica en los animales que expresan unc-119p::SID-1 solamente. Las neuronas en la región de la cola de los nematodos se magnifican. (B) Sobreexpresión de HSP-90::RFP en el intestino de mutantes sid-1(pk3321) resistentes al ARNi. Expresión de SID-1 en las neuronas (unc-119p::SID-1) (cepa PVH19); en el intestino (vha-6p::SID-1) (cepa PVH20); y músculo (myo-3p::SID-1) (cepa PVH21) mejora la sensibilidad al ARNi en estos tejidos específicos. Los animales fueron expuestos a la bacteria hsp-90 RNAi después de la etapa L4 durante 24 horas, lo que llevó a una reducción visible de la intensidad de fluorescencia HSP-90::RFP específica del intestino en el intestino de los animales que expresan vha-6p::SID-1 solamente. (C) Sobreexpresión de HSP-90::RFP en el músculo de la pared corporal de mutantes sid-1 (pk3321) resistentes al ARNi. Expresión de SID-1 en las neuronas (unc-119p::SID-1) (cepa PVH22); en el intestino (vha-6p::SID-1) (cepa PVH23); y músculo (myo-3p::SID-1) (cepa PVH24) mejora la sensibilidad al ARNi en estos tejidos específicos. Esto se indica por una intensidad de fluorescencia HSP-90::RFP visiblemente reducida en el músculo de los animales que expresan myo-3p::SID-1 , pero no en los animales que expresan unc-119p::SID-1 o vha-6p::SID-1 ni en los animales control (sid-1(pk3321)) que son resistentes al ARNi en todos los tejidos. Las flechas amarillas indican el marcador de coinyección faríngea fluorescente rojo (myo-2p::RFP). Barra de escala = 50 mm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. La expresión de ARNi de horquilla hsp-90 específica del intestino induce el reportero hsp-70p::RFP inducible por calor a temperatura permisiva en el músculo. (A) Imágenes de microscopio fluorescente de animales que expresan el promotor hsp-70 fusionado con la proteína fluorescente roja (RFP) y en el fondo de mutantes sid-1 (pk3321) (control) (cepa AM994). Los animales se cultivaron a 20 °C (sin HS) o se trataron con un choque térmico de 1 hora a 35 °C (HS) y se les permitió recuperarse durante 6 horas después de la HS. Los animales hp-RNAidel intestino hsp-90 (cepa PVH2) expresan una construcción de ARNi de horquilla hsp-90 bajo el control del promotor específico del intestino (vha-6p) en el fondo genético de hsp-70p::RFP; SID-1 (PK3321). Barra de escala = 100 μm (B) Cuantificación de la intensidad de fluorescencia de RFP de animales control cultivados a 20 °C (sin HS), tratados con una HS de 1 h a 35 °C (HS) o que expresan ARNi de horquilla hsp-90 específica del intestino a temperatura permisiva (20 °C). Los gráficos de barras representan el promedio de 3 réplicas biológicas; Las barras de error representan S.E.M. Los valores p se calcularon mediante la prueba t de Student. *P < 0,05. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4. Cribado de ARNi específico de tejido para identificar modificadores de la señalización transcelular de chaperonas. (A) Diagrama de flujo que demuestre el protocolo de cribado de ARNi específico de tejido utilizando reporteros de estrés y sensores de proteostasis específicos de tejido. (B) Cribado de ARNi específico del intestino alimentando con bacterias dsRNA a animales hp-RNAi delintestino hsp-90 que expresan vha-6p::SID-1 (cepa PVH172; ver Tabla 1 para el genotipo). Se muestran genes candidatos que actúan como posibles modificadores de TCS (txt para tcs-(x)cross-tissue) suprimiendo o mejorando la intensidad de fluorescencia de hsp-70p::RFP en el músculo de la pared corporal cuando se derriba en el intestino. (C) Cribado de ARNi específico del músculo mediante el tratamiento de animales con ARNi hp-ARNien el intestino hsp-90 que expresan myo-3p::SID-1 (cepa PVH171; ver Tabla 1) con ARNi candidato al gen candidato txt. (B y C) La intensidad de fluorescencia de los genes candidatos se indica como barras grises y se normaliza para controlar el ARNi (EV), que se indica como una barra negra. Las barras de error son S.E.M. de 5 réplicas biológicas. La significación estadística de la disminución o el aumento de la intensidad de fluorescencia de RFP entre el gen txt RNAi en comparación con el ARNi de control de vector vacío (EV) se calculó mediante la prueba t de Student, y la corrección de múltiples pruebas se realizó mediante el método de Benjamini-Hochberg con una tasa de falsos descubrimientos de 0,05. * P < 0,05. Haga clic aquí para ver una versión más grande de esta figura.
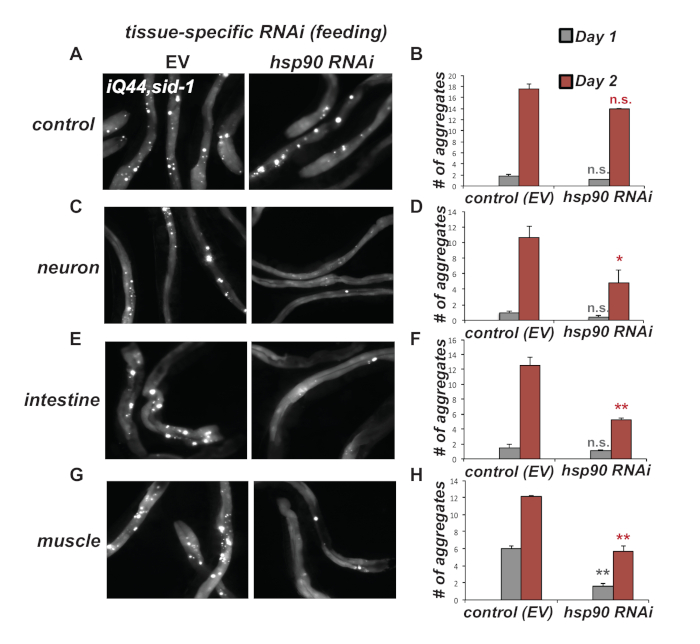
Figura 5. El ARNi hsp-90 específico de tejido reduce la agregación intestinal de Q44::YFP (iQ44). (A y B) La expresión intestinal de Q44::YFP en el fondo del alelo mutante sid-1(pk3321) resistente al ARNi (cepa PVH228) conduce a la acumulación de focos Q44 en el día 2 de la edad adulta. La eliminación mediada por ARNi mediante la alimentación de E. coli que expresa dsRNA hsp-90 desde la etapa L4 en adelante es ineficaz en comparación con el ARNi (EV) de control. (C y D) El ARNi hsp-90 específico de la neurona (cepa PVH229), específico del intestino (E y F) específico del intestino (cepa PVH230) o (G & H) específico del músculo (cepa 231) conduce a una acumulación reducida de focos de iQ44 en el día 2 de la edad adulta. (B, D, F, H) Cuantificación del número de focos de Q44 en gusanos que presentan agregación de Q44 dependiente de la edad en el día 1 y el día 2 de la edad adulta. Las barras de error son S.E.M de 3 réplicas biológicas. La significación estadística entre hsp-90 y el ARNi de vector vacío (EV) en el día 1 o el día 2 de la edad adulta se calculó mediante una prueba t de Student. n.s. no significativo; *P < 0,05; **P < 0,01. Haga clic aquí para ver una versión más grande de esta figura.
| Nombre de la cepa | Genotipo | Fuente |
| N2 | Tipo salvaje | CGC |
| AM986 | rmIs346[vha-6p::HSP-90::RFP] | Laboratorio PVO |
| AM987 | rmIs347[rgef-1p::HSP-90::RFP] | Laboratorio PVO |
| AM988 | rmIs347[unc-54p::HSP-90::RFP] | Laboratorio PVO |
| TU3401 | uIs69 [pCFJ90 (myo-2p::mCherry) + unc-119p::sid-1]; SID-1 (PK3321) | CGC |
| PVH2 | pccIs002[vha-6p::hsp-90 RNAi::unc-54 3'UTR]; rmIs288[hsp-70p::mCherry; myo-2p::CFP]; SID-1 (PK3321) | Laboratorio PVO |
| PVH5 | pccIs005[myo-3p::SID-1::unc-54 3'UTR +myo-2p::RFP]; SID-1 (PK3321) | Laboratorio PVO |
| PVH65 | pccIs004[vha-6p::SID-1::unc-54 3'UTR + myo-2p::RFP]; SID-1 (PK3321) | Laboratorio PVO |
| NL3321 | SID-1 (PK3321) | CGC |
| AM722 | rmIs288[hsp-70p::mCherry; myo-2p::CFP] | Laboratorio de Morimoto |
| AM799 | hsp-90p::GFP | Laboratorio de Morimoto |
| OG412 | vha-6p::Q44::YFP | CGC |
| PVH228 | vha-6p::Q44::YFP; SID-1 (PK3321) | Laboratorio PVO |
| PVH171 | SID-1 (PK3321); rmIs288 (myo-2p::CFP; C12C8.1p::mCereza); pccIs002 (vha-6p::hsp-90 RNAi::unc-54 3'-UTR); pccIs005 (myo-3p::SID-1::unc-54 3'UTR; myo-2p::RFP) | Laboratorio PVO |
| PVH172 | SID-1 (PK3321); rmIs288 (myo-2p::CFP; C12C8.1p::mCereza); pccIs002 (vha-6p::hsp-90 RNAi::unc-54 3'-UTR); pccIs004 (vha-6p::SID-1::unc-54 3'UTR; myo-2p::RFP) | Laboratorio PVO |
| PVH26 | rmIs345[F25B3.3p::HSP-90::RFP]; rmIs317[hsp-90pr::GFP; pCeh361]; SID-1 (PK3321) | Laboratorio PVO |
| PVH16 | rmIs345[F25B3.3p::HSP-90::RFP]; rmIs317[hsp-90pr::GFP; pCeh361]; SID-1 (PK3321); uIs69 [pCFJ90 (myo-2p::mCherry) + unc-119p::sid-1] | Laboratorio PVO |
| PVH17 | rmIs345[F25B3.3p::HSP-90::RFP]; rmIs317[hsp-90pr::GFP; pCeh361]; SID-1 (PK3321); pccIs004[vha-6p::SID-1::unc-54 3'UTR + myo-2p::RFP] | Laboratorio PVO |
| PVH18 | rmIs345[F25B3.3p::HSP-90::RFP]; rmIs317[hsp-90pr::GFP; pCeh361]; SID-1 (PK3321); pccIs005[myo-3p::SID-1::unc-54 3'UTR +myo-2p::RFP; | Laboratorio PVO |
| PVH14 | rmIs346[vha-6p::HSP-90::RFP]; rmIs317[hsp-90pr::GFP; pCeh361]; SID-1 (PK3321) | Laboratorio PVO |
| PVH19 | rmIs346[vha-6p::HSP-90::RFP]; rmIs317[hsp-90pr::GFP; pCeh361]; SID-1 (PK3321); uIs69 [pCFJ90 (myo-2p::mCherry) + unc-119p::sid-1] | Laboratorio PVO |
| PVH20 | rmIs346[vha-6p::HSP-90::RFP]; rmIs317[hsp-90pr::GFP; pCeh361]; SID-1 (PK3321); pccIs004[vha-6p::SID-1::unc-54 3'UTR + myo-2p::RFP | Laboratorio PVO |
| PVH21 | rmIs346[vha-6p::HSP-90::RFP]; rmIs317[hsp-90pr::GFP; pCeh361]; SID-1 (PK3321); pccIs005[myo-3p::SID-1::unc-54 3'UTR +myo-2p::RFP | Laboratorio PVO |
| PVH15 | rmIs347[unc-54p::HSP-90::RFP]; rmIs317[hsp-90pr::GFP; pCeh361]; SID-1 (PK3321) | Laboratorio PVO |
| PVH22 | mIs347[unc-54p::HSP-90::RFP]; rmIs317[hsp-90pr::GFP; pCeh361]; sid-1(pk3321) uIs69 [pCFJ90 (myo-2p::mCherry) + unc-119p::sid-1] | Laboratorio PVO |
| PVH23 | rmIs347[unc-54p::HSP-90::RFP]; rmIs317[hsp-90pr::GFP; pCeh361]; SID-1 (PK3321); pccIs004[vha-6p::SID-1::unc-54 3'UTR + myo-2p::RFP | Laboratorio PVO |
| PVH24 | rmIs347[unc-54p::HSP-90::RFP]; rmIs317[hsp-90pr::GFP; pCeh361]; SID-1 (PK3321); pccIs005[myo-3p::SID-1::unc-54 3'UTR +myo-2p::RFP | Laboratorio PVO |
| AM994 | rmIs288[hsp-70p::mCherry; myo-2p::CFP]; SID-1 (PK3321) | Laboratorio PVO |
| PVH229 | vha-6p::Q44::YFP; SID-1 (PK3321); uIs69 [pCFJ90 (myo-2p::mCherry) + unc-119p::sid-1] | Laboratorio PVO |
| PVH230 | vha-6p::Q44::YFP; SID-1 (PK3321); pccIs004[vha-6p::SID-1::unc-54 3'UTR + myo-2p::RFP | Laboratorio PVO |
| PVH231 | vha-6p::Q44::YFP; SID-1 (PK3321); pccIs005[myo-3p::SID-1::unc-54 3'UTR +myo-2p::RFP | Laboratorio PVO |
Tabla 1. Lista de cepas utilizadas en este trabajo.
| Solución | Reactivo | Concentración final |
| Tampón de lisis de lombriz | 1 M Tris pH 8.9 | 10 mM |
| 2 M KCl | 50 mM | |
| 50 mM MgCl2 | 1 mM | |
| Preadolescente 20 | 0.5% | |
| 2% Gelatina | 0.25% | |
| Proteinasa K | 0,1 mg/mL |
Tabla 2. Tampón de lisis de gusanos.
| Nombre de la cepa | Genotipo | Fuente | Uso |
| Reporteros de estrés | |||
| AM722 | hsp-70p::mCereza; myo-2p::CFP | Laboratorio de Morimoto | HSR |
| AM446 | hsp-70p::GFP; ROL-6 | Laboratorio de Morimoto | HSR |
| AM799 | hsp-90p::GFP | Laboratorio de Morimoto | HSR |
| TJ375 | hsp-16.2p::GFP | CGC | HSR |
| TJ3001 | hsp-16.2p::GFP; Cbr-unc-119p(+) | CGC | HSR |
| CF1553 | sod-3p::GFP; ROL-6 | CGC | Estrés oxidativo |
| CL2166 | gst-4p::GFP::NLS | CGC | Estrés oxidativo |
| LD1 | skn-1p::SKN-1::GFP; ROL-6 | CGC | Estrés oxidativo |
| SJ4005 | hsp-4p::GFP | CGC | UPR ER |
| SJ4100 | hsp-6p::GFP | CGC | UPRmito |
| SJ4058 | hsp-60p::GFP | CGC | UPRmito |
| Reporteros de proteostasis | |||
| Sensor de plegado (transgén) | |||
| AM140 | unc-54p::Q35::YFP | Laboratorio de Morimoto | expresión de polyQ específica del músculo |
| AM141 | unc-54p::Q40::YFP | Laboratorio de Morimoto | expresión de polyQ específica del músculo |
| OG412 | vha-6p::Q44::YFP | CGC | expresión de polyQ específica del intestino |
| FUH55 | unc-54p::FLUC::EGFP; ROL-6 | Laboratorio Hartl | Músculo específico de la luciferasa WT |
| FUH134 | unc-54p::FLUCSM::EGFP; ROL-6 | Laboratorio Hartl | expresión muscular específica de la luciferasa mutante R188Q |
| FUH135 | unc-54p::FLUCDM::EGFP; ROL-6 | Laboratorio Hartl | expresión muscular específica de la luciferasa doble mutante R188Q,R261Q |
| FUH48 | rgef-1::FLUC::EGFP; ROL-6 | Laboratorio Hartl | Expresión neuronal específica de la luciferasa WT |
| FUH136 | rgef-1::FLUCSM::EGFP; ROL-6 | Laboratorio Hartl | expresión neuronal específica de la luciferasa mutante R188Q |
| FUH137 | rgef-1::FLUCDM::EGFP; ROL-6 | Laboratorio Hartl | expresión neuronal específica de la luciferasa doble mutante R188Q,R261Q |
| Sensores de plegado condicional (endógenos) | |||
| CB1301 | UNC-54(E1301) I | CGC | miosina(ts), mutante sensible a la temperatura (músculo) |
| CB1157 | UNC-54(E1157) I | CGC | miosina(ts), mutante sensible a la temperatura (músculo) |
| CB1402 | UNC-15 (E1402) | CGC | Paramiosina(ts), mutante sensible a la temperatura (músculo) |
| HE250 | unc-52 (e669su250) II | CGC | Perlecan(ts), mutante sensible a la temperatura (músculo) |
| SD551 | let-60 (GA89) | CGC | Ras(ts), mutante sensible a la temperatura (múltiples tejidos) |
| CX51 | DYN-1(KY51) | CGC | Dinamina(ts), mutante sensible a la temperatura (múltiples tejidos) |
| CW152 | GAS-1 (FC21) | CGC | Gas-1(ts), sensible a la temperatura, sensible al EtOH |
| Reportero de autofagia | |||
| MAH215 | lgg-1p::mCereza::GFP::lgg-1; ROL-6 | CGC | Tándem etiquetado Autophagy Reporter |
| DA2123 | lgg-1p::LGG-1::GFP; ROL-6 | CGC | Ubicuo reportero de autofagia |
| DLM10 | myo-3p::CERULEAN-VENUS::lgg-1 + unc-119(+) | CGC | Reportero de autofagia muscular específica |
| DLM12 | rab-3p::CERULEAN-VENUS::lgg-1 + unc-119(+) | CGC | Reportero de autofagia neuronal específica |
| DLM4 | vha-6p::CERULEAN-VENUS::lgg-1 + unc-119(+) | CGC | Reportero de autofagia específica del intestino |
| DLM2 | eft-3p::CERULEAN-VENUS::lgg-1 + unc-119(+) | CGC | Reportero ubicuo de autofagia |
| Reportero del Sistema de Proteasoma de Ubiquitina (UPS) | |||
| rgef-1p::UbG76V::D endra2 | Laboratorio Holmberg (Ref. 38) | Reportero de UPS específico de neuronas | |
| AGD1033 | unc-54p::UbG76::D endra2 | CGC | Reportero de UPS específico para músculos |
| dat-1p::UbG76::D endra2 | Laboratorio Holmberg (Ref. 38) | Reportero de UPS específico de neuronas dopaminérgicas |
Tabla 3. Lista de cepas de sensores de proteostasis y reporteros de estrés.
Discusión
Los métodos aquí descritos demuestran el uso de herramientas que permiten la eliminación tisular específica de los componentes de la NP de una manera constitutiva y temporal. Hemos identificado previamente TCS, un mecanismo de respuesta al estrés no autónomo de las células que es inducido por la alteración específica del tejido de los niveles de expresión de Hsp9010. La eliminación tisular específica de hsp-90 por la expresión de ARNi de horquilla conduce a una regulación positiva no autónoma de la expresión de chaperonas protectoras de hsp-70 en los tejidos distales, lo que aumenta la resistencia al estrés del organismo10. Sin embargo, no sabemos qué componentes de señalización en el tejido que percibe o responde al estrés se activan para iniciar esta respuesta protectora. Para identificar los componentes de señalización que median este proceso, los cribados genéticos inversos específicos de tejido son uno de los métodos importantes de elección.
Aunque el derribo específico de tejido por expresión de una construcción de horquilla puede ser efectivo, esto tiene desventajas cuando es necesario estudiar un mayor número de genes. El uso de mutantes sid-1 resistentes al ARNi complementados con la expresión de SID-1 en el intestino, las neuronas o el músculo permite la eliminación de genes específicos de tejido mediante la alimentación de ARNi y, por lo tanto, es una herramienta susceptible para los cribados genéticos específicos de tejido. Si bien aquí describimos un cribado de ARNi a pequeña escala de 58 genes candidatos, el sistema SID-1 específico de tejido puede adaptarse para cribados de ARNi a mayor escala o de todo el genoma. Para ello, se requerirá el crecimiento de C. elegans en un formato de placa de 96 pocillos y la puntuación automatizada de la intensidad de fluorescencia mediante un lector de placas.
Si bien el sistema sid-1 puede ser efectivo para el ARNi específico de tejido, un método alternativo aprovecha rde-1, una proteína de Argonaute que funciona de manera no autónoma para mediar la capacidad sistémica de ARNi13. Los promotores específicos de tejido que impulsan las construcciones de rescate rde-1 también permiten que el ARNi sea efectivo en tejidos específicos, de manera similar al sistema sid-1 34. Sin embargo, las mutaciones rde-1 utilizadas en el sistema rde-1 se basan en una mutación de cambio de sentido RDE-1 E411K que puede no derogar completamente la función de RDE-1 y, por lo tanto, podría conducir a fugas de la actividad de ARNi en otros tejidos34,35 . Sin embargo, este problema parece eliminarse mediante el uso de una mutación indel35 rde-1(mkc36) recién generada. Una ventaja actual particular del sistema rde-1 en comparación con el sistema sid-1 es la reciente adaptación del sistema rde-1 para un ARNi específico y eficaz en la línea germinal35. Esto es importante, ya que otras cepas de ARNi específicas de la línea germinal actualmente existentes también pueden exhibir eficiencia de ARNi en el soma. Por lo tanto, el sistema rde-1 que permite el ARNi de la línea germinal podría ser una herramienta útil para los investigadores que investigan la importancia de la línea germinal en diversos procesos biológicos, como por ejemplo la investigación del envejecimiento.
Este método se basa en la expresión multicopia de matrices SID-1 específicas de tejido integradas. Para lograr niveles más fisiológicos de expresión de SID-1 en los tejidos específicos, se podría adaptar un método de knock-in de copia única mediado por CRISPR-Cas9 en loci genómicos definidos para el uso futuro del sistema SID-1 y para expresar SID-1 bajo el control de promotores específicos de tejido36.
Para investigar la activación de la vía del estrés, se puede elegir entre una gran selección de reporteros de chaperonas transcripcionales fusionados con proteínas fluorescentes verdes o rojas (Tabla 3). El estrés tisular específico (intracelular), a diferencia del estrés ambiental, también puede conducir a un perfil de expresión tisular diferencial de los reporteros chaperonas, como se muestra en los resultados de la Figura 3. Por ejemplo, mientras que el estrés por calor conduce a la inducción del reportero hsp-70p::RFP en múltiples tejidos (músculo, espermateca, faringe, intestino), el ARNi de horquilla hsp-90 en el intestino da como resultado una fuerte regulación positiva de hsp-70 en el músculo (Figura 3A). Esto puede indicar que las células musculares son más sensibles a los cambios en los niveles de hsp-90 no autónomos, sin embargo, no se puede excluir que la hsp-70 también se induzca en otros tejidos, aunque esto no es visiblemente detectable con el reportero transcripcional fusionado a una proteína fluorescente roja.
Por lo tanto, los sensores de proteostasis son una alternativa importante, ya que informan sobre el entorno de plegamiento real o la capacidad de la NP en un tejido específico. El entorno de plegamiento no solo depende de la expresión de chaperonas, sino también de los componentes de la PN que regulan la eliminación de proteínas mal plegadas, como la autofagia o el UPS. Por ejemplo, si uno de los sensores de plegamiento bien establecidos indica una mayor capacidad de plegamiento, pero esto no se superpone con una mayor expresión de chaperonas, entonces esto puede sugerir que se activan otros componentes de la NP que aumentan la proteostasis en un tejido específico. Por ejemplo, la hsp-4 se induce principalmente en el intestino cuando la célula UPR no autónoma se activa en las neuronas, sin embargo, la acumulación de proteínas mal plegadas expresadas en las células musculares también se suprime, posiblemente a través de señales de activación de lisosomas del intestino3. Asimismo, los datos muestran que el ARNi de hsp-90 en el intestino retrasa la agregación de iQ44 en el intestino (Figura 5), aunque no se haya detectado la expresión del reportero de hsp-70 en el mismo tejido (Figura 3). Por lo tanto, además de los sensores de plegamiento que informan sobre el entorno de plegamiento en un tejido dado, los indicadores de flujo autofágico como Cherry::GFP::LGG-1 37 o los reporteros que indican la actividad del UPS como UbG76V::D endra238 expresada en diferentes tejidos son igual de cruciales.
En conjunto, hemos descrito un sistema de ARNi específico de tejido que permite examinar la capacidad de NP en diferentes tejidos en respuesta a un mecanismo de respuesta al estrés activado de forma no autónoma.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos al Dr. Richard I. Morimoto por proporcionar la cepa AM722. Algunas cepas de C. elegans utilizadas en esta investigación fueron proporcionadas por el Centro de Genética de Caenorhabditis, financiado por la Oficina de Programas de Infraestructura de Investigación de los NIH (P40 OD010440). P.V.O.-H. fue financiado por subvenciones de las NC3R (NC/P001203/1) y por un premio Wellcome Trust Seed Award (200698/Z/16/Z). J.M. contó con el apoyo de una asociación de formación doctoral DiMeN del MRC (MR/N013840/1).
Materiales
| Name | Company | Catalog Number | Comments |
| Ampicillin | Merck | A0166-5G | Protocol Section 3.1. |
| DNA Clean & Concentrator-500 | Zymo Research | D4031 | Protocol Section 2.2. |
| IPTG Isopropyl-β-D-thiogalactoside | Merck | 367-93-1 | Protocol Section 3.1. |
| Multisite Gateway Cloning Kit | Thermo Fisher | 12537100 | Protocol Section 1.2. |
| SigmaCote | Merck | SL2-25mL | Protocol Section 2.3. |
| Tetracycline | Merck | T7660-5G | Protocol Section 3.1. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Fisher | K280002 | Protocol Section 1.1. |
Referencias
- Morimoto, R. I. The heat shock response: systems biology of proteotoxic stress in aging and disease. Cold Spring Harbor Symposia on Quantitative Biology. 76, 91-99 (2011).
- Hipp, M. S., Kasturi, P., Hartl, F. U. The proteostasis network and its decline in ageing. Nature Reviews Molecular Cell Biology. 20, 421-435 (2019).
- Imanikia, S., Özbey, N. P., Krueger, C., Casanueva, M. O., Taylor, R. C. Neuronal XBP-1 Activates Intestinal Lysosomes to Improve Proteostasis in C. elegans. Current Biology. 29, 2322-2338 (2019).
- Berendzen, K. M., et al. Neuroendocrine Coordination of Mitochondrial Stress Signaling and Proteostasis. Cell. 166, 1553-1563 (2016).
- O'Brien, D., et al. A PQM-1-Mediated Response Triggers Transcellular Chaperone Signaling and Regulates Organismal Proteostasis. Cell Reports. 23, 3905-3919 (2018).
- Tatum, M. C., et al. Neuronal Serotonin Release Triggers the Heat Shock Response in C. elegans in the Absence of Temperature Increase. Current Biology. 25, 163-174 (2015).
- Miles, J., Scherz-Shouval, R., van Oosten-Hawle, P. Expanding the Organismal Proteostasis Network: Linking Systemic Stress Signaling with the Innate Immune Response. Trends in Biochemical Sciences. 44, 927-942 (2019).
- Taylor, R. C., Dillin, A. XBP-1 Is a Cell-Nonautonomous Regulator of Stress Resistance and Longevity. Cell. 153, 1435-1447 (2013).
- Prahlad, V., Cornelius, T., Morimoto, R. I. Regulation of the Cellular Heat Shock Response in Caenorhabditis elegans by Thermosensory Neurons. Science. 320, 811-814 (2008).
- van Oosten-Hawle, P., Porter, R. S., Morimoto, R. I. Regulation of Organismal Proteostasis by Transcellular Chaperone Signaling. Cell. 153, 1366-1378 (2013).
- Frakes, A. E., et al. Four glial cells regulate ER stress resistance and longevity via neuropeptide signaling in C. elegans. Science. 367, 436-440 (2020).
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854(1998).
- Tabara, H., Grishok, A., Mello, C. C. RNAi in C. elegans: soaking in the genome sequence. Science. 282, 430-431 (1998).
- Hinas, A., Wright, A. J., Hunter, C. P. SID-5 is an endosome-associated protein required for efficient systemic RNAi in C. elegans. Current Biology. 22, 1938-1943 (2012).
- Winston, W. M., Molodowitch, C., Hunter, C. P. Systemic RNAi in C. elegans Requires the Putative Transmembrane Protein SID-1. Science. 295, 2456-2459 (2002).
- McEwan, D. L., Weisman, A. S., Hunter, C. P. Uptake of extracellular double-stranded RNA by SID-2. Molecular Cell. 47, 746-754 (2012).
- Whangbo, J. S., Weisman, A. S., Chae, J., Hunter, C. P. SID-1 Domains Important for dsRNA Import in Caenorhabditis elegans. G3 GenesGenomesGenetics. 7, 3887-3899 (2017).
- Feinberg, E. H., Hunter, C. P. Transport of dsRNA into cells by the transmembrane protein SID-1. Science. 301, 1545-1547 (2003).
- Calixto, A., Chelur, D., Topalidou, I., Chen, X., Chalfie, M. Enhanced neuronal RNAi in C. elegans using SID-1. Nature Methods. 7, 554-559 (2010).
- Kamath, R. S., et al. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421, 231-237 (2003).
- Lee, Y. S., Carthew, R. W. Making a better RNAi vector for Drosophila: use of intron spacers. Methods (San Diego CA). 30, 322-329 (2003).
- Evans, T. Transformation and microinjection. WormBook. , (2006).
- Fay, D. Genetic mapping and manipulation: Chapter 1-Introduction and basics. WormBook. , (2006).
- Lim, M. A., et al. Reduced Activity of AMP-Activated Protein Kinase Protects against Genetic Models of Motor Neuron Disease. Journal of Neuroscience. 32, 1123-1141 (2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , (2006).
- Hawkins, M. G., McGhee, J. D. elt-2, a second GATA factor from the nematode Caenorhabditis elegans. Journal of Biological Chemistry. 270, 14666-14671 (1995).
- Bharadwaj, S., Ali, A., Ovsenek, N. Multiple components of the HSP90 chaperone complex function in regulation of heat shock factor 1 In vivo. Molecular and Cellular Biology. 19, 8033-8041 (1999).
- Guisbert, E., Czyz, D. M., Richter, K., McMullen, P. D., Morimoto, R. I. Identification of a tissue-selective heat shock response regulatory network. PLoS Genetics. 9, 1003466(2013).
- Ben-Zvi, A., Miller, E. A., Morimoto, R. I. Collapse of proteostasis represents an early molecular event in Caenorhabditis elegans aging. Proceedings of the National Academy of Sciences of the United States of America. 106, 14914-14919 (2009).
- Morley, J. F., Brignull, H. R., Weyers, J. J., Morimoto, R. I. The threshold for polyglutamine-expansion protein aggregation and cellular toxicity is dynamic and influenced by aging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 99, 10417-10422 (2002).
- Brignull, H. R., Moore, F. E., Tang, S. J., Morimoto, R. I. Polyglutamine Proteins at the Pathogenic Threshold Display Neuron-Specific Aggregation in a Pan-Neuronal Caenorhabditis elegans Model. Journal of Neuroscience. 26, 7597-7606 (2006).
- Mohri-Shiomi, A., Garsin, D. A. Insulin Signaling and the Heat Shock Response Modulate Protein Homeostasis in the Caenorhabditis elegans Intestine during Infection. Journal of Biological Chemistry. 283, 194-201 (2008).
- Nussbaum-Krammer, C. I., Neto, M. F., Brielmann, R. M., Pedersen, J. S., Morimoto, R. I. Investigating the spreading and toxicity of prion-like proteins using the metazoan model organism C. elegans. Journal of Visualized Experiments. , e52321(2015).
- Qadota, H., et al. Establishment of a tissue-specific RNAi system in C. elegans. Gene. 400, 166-173 (2007).
- Zou, L., et al. Construction of a germline-specific RNAi tool in C. elegans. Scientific Reports. 9, 1-10 (2019).
- Silva-García, C. G., et al. Single-Copy Knock-In Loci for Defined Gene Expression in Caenorhabditis elegans. G3 Bethesda Md. 9, 2195-2198 (2019).
- Chang, J. T., Kumsta, C., Hellman, A. B., Adams, L. M., Hansen, M. Spatiotemporal regulation of autophagy during Caenorhabditis elegans aging. eLife. 6, 18459(2017).
- Hamer, G., Matilainen, O., Holmberg, C. I. A photoconvertible reporter of the ubiquitin-proteasome system in vivo. Nature Methods. 7, 473-478 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados