Method Article
Utilizando tecnología de impresión 3D se fusiona con la RM Histología: Un protocolo para el Cerebro de seccionamiento
En este artículo
Resumen
El objetivo general de este protocolo es para alinear con precisión los volúmenes de imágenes magnéticas resonancia nuclear (RMN) con secciones histológicas a través de la creación de los titulares del cerebro en 3D impresos personalizados y cajas de la máquina de cortar.
Resumen
Magnetic resonance imaging (MRI) allows for the delineation between normal and abnormal tissue on a macroscopic scale, sampling an entire tissue volume three-dimensionally. While MRI is an extremely sensitive tool for detecting tissue abnormalities, association of signal changes with an underlying pathological process is usually not straightforward. In the central nervous system, for example, inflammation, demyelination, axonal damage, gliosis, and neuronal death may all induce similar findings on MRI. As such, interpretation of MRI scans depends on the context, and radiological-histopathological correlation is therefore of the utmost importance. Unfortunately, traditional pathological sectioning of brain tissue is often imprecise and inconsistent, thus complicating the comparison between histology sections and MRI. This article presents novel methodology for accurately sectioning primate brain tissues and thus allowing precise matching between histology and MRI. The detailed protocol described in this article will assist investigators in applying this method, which relies on the creation of 3D printed brain slicers. Slightly modified, it can be easily implemented for brains of other species, including humans.
Introducción
In vivo MRI provides a noninvasive and sensitive measure of tissue integrity at the macroscopic level. Changes in MRI signal intensity seen in vivo are outcome measures in many ongoing clinical trials.1 While the intensity changes seen via MRI can identify areas of abnormality in the context of the whole brain, they are often not sufficiently specific to differentiate pathological processes. This is especially true of dynamic processes involving multiple pathologies. For example, in multiple sclerosis (MS) or its animal model, experimental autoimmune encephalomyelitis (EAE), inflammation, edema, myelin degradation, axonal destruction, gliosis, and neuronal death overlap. 2, 3 To obtain the necessary specificity regarding the underlying pathology, context must be taken into account, together with knowledge of the histology of the MRI-identified abnormal tissues.
However, even in well-controlled animal experiments, matching histology with in vivo MRI is fundamentally challenging for various reasons. First, the difference in dimensional scales between histology sections and MRI is of several orders of magnitude.4 Second, for proper comparison, the orientation of MRI slice plane must match the sectioning plane of the brain tissue when cut. Due to the shape of the brain, it is very difficult to make consistently straight and accurate cuts when the brain is sitting on a flat surface. Third, the large size of the brain relative to a potentially small area of interest (lesion, tumor, etc.) creates a "needle-in-a-haystack" scenario for the pathologist processing the tissue. Fourth, even when the target tissue is found, it is commonly processed in such a way as to render virtually impossible an association with the original MRI data. Finally, traditional pathological sectioning of brain tissue is often imprecise and inconsistent, further complicating the comparison between histology sections and MRI images.
Previous attempts to overcome these challenges relied on the use of deformational algorithms to coregister the data and/or placement of fiducial markers within or around the tissue as a reference.5, 6, 7, 8 The former approach requires complex computational models that are particularly susceptible to complications due to data formatting, imaging artifacts, and changes caused by tissue processing.4 On the other hand, the latter approach introduces the possibility of contaminating or otherwise harming the tissue itself.9
The approach described here improves the transition between modalities through the use of postmortem MRI to bridge the gap between in vivo MRI and histology. Postmortem MRI provides three-dimensional (3D) images of the brain at higher resolution than can be achieved in vivo and furthermore provides the data needed for producing a morphologically accurate model of the brain surface. This digital model can then be used to create a 3D-printed custom holder for the brain. With careful positioning, the brain holder allows for precise, MRI-oriented brain sectioning, reducing the need for complex mathematical algorithms, and enables a focus on specific regions for targeted sampling.
Our laboratory recently introduced new methods for creating custom brain holders and slicers using postmortem MRI and 3D-printing technology for human10 and marmoset brains.4 The two methods allow for a more accurate correlation between MRI and histology in a research setting, and ultimately allow a deeper understanding of the specific pathology underlying MRI abnormalities. Carefully designed experiments, in which the brain is sampled repeatedly over time in vivo, can provide context for interpretation of the pathology, which in turn can add specificity to interpretation of the MRI. Here, we present a modified protocol in a unified framework that can be applied to any brain tissue, whether it derive from nonhuman primates, rodents, or humans. We provide detailed instructions, and a corresponding video, for the marmoset sectioning. Although the overall protocol applies to any type of brain, due to differences in MRI acquisition and tissue size, as well as the challenges encountered when dealing with specific brain types, there are some differences in the approach depending on the type of brain being processed. In this presentation, sections with "human" will denote differences in protocol specific to the human brain.
Protocolo
Toda la manipulación y procedimientos descritos en el presente documento con animales se realizaron de acuerdo con un protocolo aprobado por el Instituto Nacional de Trastornos Neurológicos y Accidentes Cerebrovasculares Cuidado de Animales y el empleo Comisión. Los cerebros fueron recogidos de los titíes comunes (Callithrix jacchus) inducidos a desarrollar EAE. 11 Los cerebros se almacenaron en formalina al 10% de entre 3 semanas y un año después de la eutanasia mediante perfusión transcardial de paraformaldehído al 4%.
1. Postmortem MRI Preparación y Adquisición
- cerebro tití
- Preparar una estación de trabajo con una gasa de algodón, un tubo de centrífuga de 50 ml, pequeñas espátulas, ~ 30 ml de un aceite fluorado, parafina, y el cerebro tití.
- Llenar el tubo con el aceite fluorado y gasa hasta la marca de 20 ml. Comprimir la gasa para eliminar las burbujas de aire a lo largo del camino.
- formol secar suavemente desde la superficie del cerebro con una toalla de papel. Insertar el cerebro con el polo frontal hacia la parte inferior deel tubo. asegurar cuidadosamente el cerebro en el tubo usando más gasa alrededor de los lados para fijar su posición. Consulte la sección suplementaria 11 para un método para crear una cuna resonancia magnética del cerebro para la configuración de imágenes por resonancia magnética adicionales.
- Llenar el resto del tubo con una gasa y aceite fluorado. Retirar con cuidado las burbujas de aire en el camino. Asegure la tapa y sella el tubo con parafina.
- Marque la tapa de acuerdo con la fisura interhemisférica. Envolver el tubo en una toalla de papel y la inserta en la bobina con la marca superior central.
- Adquirir 2D girar T2 eco. Los parámetros se dan en la Tabla 1.
- Abra los 10 a 150 micras adquisiciones T2-weigted anatómicas en Mipav y registrarse para la adquisición 6º.
NOTA: El registro es un algoritmo de registro 3.5D automático optimizado con 9 grados de libertad, con ventanas de interpolación de sincronismo, la función de coste correlación cruzada normalizada, con llamando algoritmo de búsqueda de Brent del Powell. Las rotaciones se tomaron muestras de 106; a -10 ° con rotaciones incrementales gruesas 5 grados y la rotación de finos de incremento de 1 °. Entonces promediar las imágenes registradas: Utilidades, de imágenes Las imágenes de la calculadora-granel, Normal.
- Cerebro humano
- Separar el cerebro anterior del tronco cerebral usando un corte en el nivel del mesencéfalo. 10 Los hemisferios también se pueden separar con un corte por la línea media.
- Coloque el cerebro anterior en un tubo cilíndrico con una cúpula hemisférica en un extremo y una boquilla en el otro extremo.
- Llenar el tubo con un aceite fluorado través de la boquilla. Eliminar las burbujas de aire mediante una succión suave durante ~ 30 min a través de la boquilla.
- Adquirir 3D T1-MPRAGE. Los parámetros se dan en la Tabla 1.
2. La extracción del cerebro Superficie: 7,2 Mipav
- Abra la resonancia magnética en la orientación coronal.
- Seleccionar algoritmos, herramientas de transformación, Transform, Volver a muestrear. Seleccione el tamaño definido por el usuario y volver a muestrear a isotvoxels Ropic: 0,1 x 0,1 x 0,1 mm. Guarde el MRI resampled como Brain_MRI_Resampled. Humano: Volver a muestrear a isotrópica voxels 0,3 x 0,3 x 0,3 mm.
- Seleccionar Algoritmos, Filtros (espacial), reducción de ruido no lineal. Utilice la configuración predeterminada, haga clic en OK.
- Seleccionar Muestra tabla de búsqueda y hacer clic en el botón de doble umbral. Arrastre el control deslizante en el gráfico para cubrir la totalidad del cerebro.
- Seleccionar Algoritmos, Segmentación, Umbral, Umbral usando min / max. Introduzca el valor situado en la esquina inferior izquierda del gráfico de intensidad (justo por debajo de la escala) en el cuadro de "límite inferior". Seleccione "binario" en el tipo de imagen de salida y desactive "umbral invertido."
- Seleccionar Algoritmos, morfológico, Llene los agujeros. Marque "Proceso de 2.5D."
- Debido a que esta es una resonancia magnética del cerebro completo, hay algo de espacio vacío entre la parte posterior del cerebro y la corteza que necesita ser llenado. El cerebro humano: omitir este paso.
- El uso de la línea VOI, dibujar en una conexiónentre la parte posterior del cerebro y la corteza en ambos lados del cerebro en el punto más lateral. Continuar este a través del cerebro.
- Seleccione la conversión VOI, VOI, todo para máscara binaria. Seleccione Utilidades, calculadora de imágenes. Por favor, seleccione en el menú desplegable y seleccionar el operador de la máscara cerebro. Seleccione "Promover el tipo de imagen de destino"
- Seleccionar Algoritmos, morfológico, Llene los agujeros. Marque "Proceso en 2.5D"
- Guardar la máscara binaria como Brain_Model.nii.
- Seleccionar Algoritmos, la superficie del extracto (cubos de marcha). Seleccionar imagen máscara, Guardar como Brain_Model.ply.
3. Selección de la rebanada Ubicaciones: 7.2 Mipav
- Identificar el tejido de interés o posición de partida. Calcular el espesor de la losa previsto. En el cerebro del tití, contando 30 rebanadas de resonancia magnética por sección, y 5 rebanadas de resonancia magnética por hueco de rasqueta, crea secciones de 3 mm con lagunas cuchilla 0,5 mm, lo que resulta en losas ~ 3,5 mm. El cerebro humano: 20 rebanadas de resonancia magnética por sección, y 4 sl RMhielos por hueco de rasqueta, crea secciones 6 mm con lagunas de hoja 1,2 mm, lo que resulta en losas ~ 7,2 mm.
- En el lugar del primer espacio de la hoja, haga clic en "caja VOI" y dibuje un rectángulo sobre el cerebro. Haga clic dentro del cuadro para seleccionarlo. Copia y pega el contorno de cada rebanada de resonancia magnética correspondiente a la brecha de la cuchilla.
- Vaya directamente por el número de rebanadas de resonancia magnética que corresponden al espesor de la sección y copiar y pegar el contorno correspondiente a la siguiente brecha de la cuchilla. Repita este proceso a través del cerebro.
- Seleccionar VOI, VOI conversión, todos con máscara binaria. Guardar como Blade_Gaps.nii.
- Seleccionar imagen Máscara Algoritmos, Extracto de Superficie (cubos de marcha),, Blade_Gaps.ply.
4. Creación de resonancia magnética Mapa de la hoja: 7,2 Mipav
- Abra la Brain_MRI_Resampled y las imágenes Blade_Gaps.nii.
- Con la imagen seleccionada Blade_Gaps.nii, Utilidades imagen Matemáticas. Seleccione Promover tipo de imagen y multiplicarse.Introduce 10.000 como valor.
- Seleccione Utilidades, calculadora de imágenes. Seleccione Agregar y, a continuación, seleccione la imagen Brain_MRI_Resampled de la caja de la imagen desplegable. Promover Seleccionar tipo de imagen de destino.
- Guarde esta imagen como Brain_BladeMap.nii.
- Al hacer clic en la vista TRIPLANAR, los lugares donde se cortó el cerebro pueden verse en tres vistas ortogonales.
5. Importación de Cerebro y superficies hueco de rasqueta: Netfabb Profesional
- Seleccione la Parte, Añadir parte. Elija los archivos Brain_Model.ply y Blade_Gaps.ply
- Seleccione el cerebro y hacer clic en el modo de reparación. En el modo de reparación:
- Haga clic en el botón de selección de concha y, a continuación, haga clic en el cerebro.
- Haga clic en el botón de selección de palanca para seleccionar las otras mallas. Haga clic en Quitar para eliminar las otras mallas.
- Haga clic en Aplicar reparar y eliminar la parte antigua.
- Clic derecho en el cerebro. Seleccionar movimiento. Clickea en elPara botón de origen. Registrar los parámetros que aparecen XYZ. Se necesitan estos parámetros de traslación para mantener la posición de la hoja creada en Mipav. Haga clic en Traducir. A continuación, cierre la ventana. (No haga clic en traducir más de una vez. Esta opción convertirá de nuevo utilizando los mismos parámetros.)
- Seleccione el modelo de Blade_Gaps. Haga clic derecho sobre la parte y seleccione Mover. Introduzca los valores XYZ grabadas previamente en las casillas de parámetros XYZ. Ahora haga clic en Traducir y cerrar la ventana.
- Seleccione el modelo de Brain_Model y haga clic en modo de reparación. En el modo de reparación:
- Haga clic en el botón de selección de concha y, a continuación, haga clic en el cerebro.
- Haga clic derecho y seleccione triángulos lisos. Introduzca 4-5 iteraciones. Compruebe prevenir la contracción de volumen. El cerebro humano: 1-2 iteraciones.
- Clic derecho, seleccione Reducir triángulos. Introduzca 200000 en la cuenta del triángulo de destino y haga clic en Ejecutar.
- Haga clic en la reparación automática, reparación de defecto. Luego haga clic en Aplicar Reparación
- Haga clic derecho, en Cambiar nombre. Cambie el nombre del cerebro alisada como Smoothed_Brain_Model.
- Seleccionar Smoothed_Brain_Model. Haz clic derecho, Exportación, STL.
6. Edición de curvas de nivel cerebral: Meshmixer
- Importe el Smoothed_Brain_Model en Meshmixer.
- Utilice las herramientas para esculpir y selección para realizar ajustes en la malla. Las modificaciones incluyen:
- Utilice herramientas para esculpir, robusto lisas. Suavizar el área que corresponde a la línea VOI. El cerebro humano: omitir este paso.
- Alisar la superficie de la corteza que se boca abajo en la caja.
- Suavizar pequeños terrones que podrían haber sido creados en el proceso de mallado y edición.
- Seleccione Análisis, inspector, Autorepair todos.
- Exportar el Smoothed_Brain_Model como Smoothed_Edited_Brain_Model.
7. Creación de la máquina de cortar Brain Box: Netfabb Profesional
- Seleccione la Parte, la Parte Agregar. Elija el file Smoothed_Edited_Brain_Model.
- Seleccione la Parte, la Parte Agregar. A continuación, seleccione el archivo STL cerebro máquina de cortar Parts_Marmoset y haga clic en Abrir. El cerebro humano: Cerebro Máquina de cortar Parts_Human (archivos de código suplementario).
- Haga clic derecho sobre la parte y seleccione extendidas, depósitos de piezas. Seleccione cada parte por separado y hacer clic derecho para cambiar el nombre de ellos. (Al hacer clic en el ojo junto a un objeto oculta o hace que sea visible.) Cambiar el nombre de la gran caja principal, la pequeña caja Sub, y la caja de corte Box_Cutout, la forma del hexágono Blade_Holder_Main, la pequeña caja plana Micrótomo hoja, y la media tubo de la horquilla objeto. El cerebro humano: no Blade_Holder_Main, Micrótomo Blade, o la base.
- Hide principal, secundario, Cuchilla, Blade_Holder_Main, Micrótomo Blade, y la horquilla y utilizar el cambio de selección para seleccionar los seis y los Box_Cutout. Sólo el Box_Cutout y la Smoothed_Edited_Brain_Model debe ser visible, pero el Smoothed_Edited_Brain_Model no se debe elegir.
- Haga clic y arrastrar las piezas seleccionadas para ajustar la posición de la caja en relación con el cerebro.
- Coloque el cerebro en el centro de la caja. Postion el cerebro lo suficientemente profunda en el cuadro que se agarró con fuerza, pero no demasiado profunda para crear salientes que impiden la colocación adecuada.
- Una vez colocado, el contorno cuadro cerebro puede ser probado para voladizos. Seleccione la Box_Cutout y la Smoothed_Edited_Brain_Model. Seleccione la operación booleana.
- Haga clic en el Smoothed_Edited_Brain_Model para convertirlo rojo.
- Seleccionar booleana Resta, y aplicar los cálculos.
- Marque la casilla del cerebro para voladizos que impedirían el tejido cerebral de ser colocado de manera segura en la caja. Si estas proyecciones están presentes, ajuste el cerebro por lo que es menos profunda dentro de la caja. Si el cerebro se encuentra en la profundidad y voladizos deseadoestán presentes, consulte la sección complementaria 10 para una solución para la eliminación de los voladizos.
- Seleccione el modelo de Blade_Gaps. Haz clic derecho, seleccione Mover. Registrar el valor Z posición, a continuación, cierre la ventana. Esta será la posición de la brecha de la cuchilla más posterior.
- Seleccione la STL lámina que vino de la máquina de cortar el cerebro de piezas. Haz clic derecho, seleccione Mover.
- Seleccionar traducción absoluta. Introduzca el valor Z desde el 7.8. Para los valores de X e Y, introduzca el valor correspondiente de las casillas de parámetro posición actual
- Seleccione la STL Blade. Clic derecho, clic en Duplicar.
- Compruebe Organizar partes. Introduce el número total de láminas en el recuento total. Introduzca el mismo número en el cuadro de recuento Z. Introduzca el espesor de la losa en el hueco Z. Haga clic en Duplicar. Si el espesor de la losa se varió, tendrán que ser posicionado de forma individual las cuchillas. Duplicar cada nueva cuchilla de la anterior (movimiento de posterior a anterior)con la diferencia z igual al espesor de la sección de esa sección.
- Coloque las piezas de Micrótomo Blade en exactamente los mismos intervalos que las cuchillas de la máquina de cortar repitiendo el paso 7.9 y 7.10 para el microtomo Blade. El cerebro humano: omitir este paso.
- Seleccione la hoja microtomo junto con la parte Blade_Holder_Main y seleccione la operación booleana.
- Seleccione todos los módulos de alta densidad de la hoja microtomo para destacarlos rojo. Haga clic en booleana Resta, y aplicar los cálculos.
- Seleccione el modo de reparación. Realizar una reparación extendida. Seleccione aplicar el reparar y eliminar la parte antigua.
- Cambiar el nombre del titular de la parte de la hoja. Exportar la pieza como STL.
- Shift-select Smoothed_Edited_Brain_Model, todos los modelos de la lámina creado en el paso anterior, y el Secundario y Principal. Haga clic en la operación booleana.
- Hacer todas las partes excepto el rojo principal por sí mismanando y haciendo clic en la flecha debajo de la caja verde para mover a rojo.
- Seleccionar booleana Resta y luego seleccione aplicar cálculos.
- Haga clic en el modo de reparación. En el modo de reparación:
- Marque la casilla para voladizos y puntos agudos. Estos pueden ser suavizadas en Netfabb o Meshmixer.
- Haga clic en la reparación automática, reparación extendido. A continuación, aplicar la reparación y quitar la parte antigua
- Haga clic derecho sobre el cuadro de cerebro reparado y cambiarle el nombre Cerebro Máquina de cortar la caja. Exportar como un STL.
8. Impresión de la máquina de cortar Brain Box en el Ultimaker 2
- Tití Cerebral: Cura
- Importe la máquina de cortar Brain Box en Cura.
- Seleccione Rotar y arrastre el círculo para girar el cuadro de lo que es plana en la cama.
- Ajustar la configuración de impresión: la resolución capa de 0,1 mm, el 50% de relleno de densidad, Raft.
- Seleccione Guardar herramienta de ruta a la tarjeta SD. (Imprimir la ~ 12 h.)
- Titular de la importación de la lámina enal Cura y girarlo de modo que las ranuras están en los lados y la cara del hexágono se encuentra en la XZ o plano YZ.
- Duplicar el objeto.
- Ajustar la configuración de impresión: la resolución capa de 0,2 mm, 20% de densidad de relleno, borde.
- Seleccione Guardar herramienta de ruta a la tarjeta SD. (El tiempo de impresión ~ 3 h)
- Cerebro Humano: Cura
- Importe la máquina de cortar Brain Box en Cura y girar como en 8.1.2.
- Ajustar la configuración de impresión: Resolución de 0,2 mm de capa, un 30-35% de la densidad de llenado, balsa.
- Seleccionar trayectoria de la herramienta Guardar en la tarjeta SD. (El tiempo de impresión ~ 70 h para la caja única hemisferio.)
- En la Ultimaker 2
- Aplicar una capa fina de pegamento barra de pegamento para construir la placa.
- Inserte la tarjeta SD. Seleccione Imprimir y seleccionar la parte.
9. Cortar el cerebro
- cerebro tití
- Preparar una estación de trabajo con el cerebro fija, la máquina de cortar de cerebro, dos soportes de cuchilla, cuchillas de microtomo, 1 ml de aceite fluorado, flat pinzas, guantes protectores, y cassettes de inclusión.
- Coloque nuevas cuchillas de microtomo en las ranuras de los soportes de las aspas. Asegúrese de que el borde biselado de cada cuchilla se oriente en la misma dirección. Use guantes de protección al manipular las cuchillas de microtomo.
- Retire el cerebro de formalina y séquelo cuidadosamente.
- Coloque el cerebro en la máquina de cortar. Unas pocas gotas de aceite fluorado se pueden aplicar al cerebro y la máquina de cortar para permitir un fácil posicionamiento. Asegúrese de que el cerebro está firmemente en su lugar.
- Coloque los soportes de cuchilla con las cuchillas en las ranuras correspondientes de cuchilla.
- Empuje hacia abajo en los soportes de cuchilla con firmeza y aplicar presión equilibrio lento para cortar a través del cerebro.
- Retire cada losa, de una en una, a partir de la parte delantera del cerebro. Ayuda a eliminar la hoja microtomo en frente de una losa antes de retirar la propia losa. Prestar mucha atención a la orientación anterior / posterior de cada losa.
- Tomar fotografías de la dirección anterod superficie posterior de cada losa. Las placas posteriores más probable contendrán piezas separadas, así que presta atención a la orientación de las piezas para incrustar. Coloque cada losa en un casete de inclusión y poner a todos en una solución de formalina al 10%.
- Cerebro humano
- Con cuidado probar el ajuste del cerebro en la caja.
- Cortar el cerebro empezando por un extremo mediante un corte en ángulo, lenta pero firmemente rebanar. Cortar el cerebro a través de cada hueco de la hoja.
- Retire cada losa de uno a la vez, prestando especial atención al número y la orientación anterior / posterior de cada losa.
- Tomar fotografías de la superficie anterior y posterior de cada losa. Coloque losas en sellados 10% bolsas formalina. bloques de tejido se cortan a partir de las losas y se colocaron en casetes para incrustar.
10. Eliminación de aleros en Brain Box (sección complementaria)
- Sacando rebanadas: Meshmixer
- Importe el Smoothed_Edited_Brain_Model.
- Seleccione Editar, Hacer rodajas. En rodajas de Marca:
- Seleccione apilada en 3D, Z, introduzca 1-2 mm de espesor. Haga clic en Calcular. Cuando se cargan las rebanadas, haga clic en Aceptar.
- Selecciona de 1 a 2 rebanadas con grandes perímetros cerca de la parte inferior de la corteza. Estas rebanadas deben estar por debajo del nivel de la caja de Sub.
- Exportar cada uno de estos cortes como Brain_Slice_ #.
- La ampliación de las rebanadas hasta eliminar voladizos: Netfabb Profesional
- Importe los Brain_Slice_ # rodajas.
- Duplicar cada Brain_Slice_ # (desactiva organizar las piezas si se activa).
- Haga clic derecho sobre una copia de cada Brain_Slice_ # y seleccione Escala.
- relación de escala fija Deseleccionar. A continuación, ampliar el corte de cerebro en la dirección Y para que se alcance el nivel de la parte inferior del cuadro Sub.
- Cambiar el nombre de estas rodajas Brain_Slice_Big_ #.
- Y comprobar la posición o,f Brain_Slice_ el original # haciendo clic derecho sobre la pieza y la selección de movimiento. Registrar la posición Y para cada una de las rebanadas # Brain_Slice_ originales.
- Realizar el cálculo: Brain_Slice_ # [y posición] - (Brain_Slice_Big_ # [y tamaño] - Brain_Slice _ # [tamaño y])
- Seleccione cada uno de los Brain_Slice_Big_ # individualmente, haga clic derecho y seleccione Mover.
- Introduzca el valor calculado a partir 10.2.6 en el cuadro de parámetros traducción Y. Para las direcciones X y Z traducción parámetros, introduzca los valores ubicados en las casillas de parámetro posición actual. Seleccione Absoluto traducción. Haga clic en Traducir y cerrar la ventana.
NOTA: Las rebanadas # Brain_Slice_Big_ se restarán junto con el cerebro y las cuchillas al hacer la caja.
- Introduzca el valor calculado a partir 10.2.6 en el cuadro de parámetros traducción Y. Para las direcciones X y Z traducción parámetros, introduzca los valores ubicados en las casillas de parámetro posición actual. Seleccione Absoluto traducción. Haga clic en Traducir y cerrar la ventana.
11. Tití RM cerebral Cuna para poder analizarlo
- La creación de la Cuna RM cerebral
- Asegúrese de que la superficie superior del CrADLE está en la misma altura que el Box_Cutout. La profundidad y la posición del cerebro en la cuna debe ser configurado igual que lo es para la máquina de cortar.
- Shift-select para seleccionar el Smoothed_Edited_Brain_Model y Cuna.
- Seleccionar operación booleana. Seleccione el cerebro para resaltarlo rojo, a continuación, seleccione la resta de Boole. A continuación, aplicar los cálculos. (También seleccione las rebanadas # Brain_Slice_Big_ si es aplicable).
- Entre en el modo de reparación para eliminar los puntos agudos en el contorno de soporte como se hizo anteriormente para la máquina de cortar. Seleccione la reparación extendido. Aplicar la reparación, y quitar la parte antigua.
- Haga clic derecho para cambiar el nombre de la parte del cerebro MRI de la horquilla. Seleccione Exportar, STL.
- Impresión de la cuna: Cura
- Importe la RM cerebral cuna en Cura y girarlo para que la parte plana con el recorte del cerebro está boca arriba.
- Ajustar la configuración de impresión: la resolución capa de 0,1 mm, 100% de densidad de llenado, balsa.
- Seleccionar trayectoria de la herramienta Guardar en la tarjeta SD. (El tiempo de impresión ~ 10 h)
- Imprimir en el Ultimaker 2 como se describe en 8.3.
- La adquisición de alta resolución T2 * resonancia magnética se utiliza la base
- formol secar suavemente desde la superficie del cerebro con una toalla de papel.
- Coloque el cerebro en la cuna como se describe para la máquina de cortar.
- Deslizar el cerebro y el soporte en el tubo de centrífuga de 50 ml. Llenarlo hasta el borde con aceite fluorado.
- Apriete suavemente el tubo para permitir que las burbujas de aire que se escapan del cerebro. Inserte el casquillo de inserción en el recuadro de la tapa del tubo para evitar que se formen burbujas de aire allí. Asegure la tapa y sella el tubo con parafina.
- Colocar el tubo en la bobina como se ha descrito anteriormente. parámetros 3D T2 * se dan en la Tabla 1.
- Abrir las 18 anatómicas 100 micras T2 * adquisiciones ponderadas en Mipav y registrarse para la adquisición 10º. parámetros de registro son los mismos que en 1.1.7. Promediar las imágenes registradas: Utilities, de imágenes Las imágenes de la calculadora-granel, Normal.
Resultados
El flujo de trabajo de este método se resume en la figura 1. Una vez que se corta el cerebro, una comparación visual entre las imágenes de RM e imágenes de las superficies superficiales de las losas muestra un buen partido orientación a través de múltiples placas (Figura 2). Después de que las losas se incluyeron en parafina, que se seccionan en un microtomo y se tiñeron. Una comparación más exhaustiva entre el post-mortem de resonancia magnética de alta resolución y de las secciones histológicas teñidas demuestra una coincidencia exacta y consistente a través de todas las estructuras del cerebro tití (Figura 3).
En este modelo animal de la esclerosis múltiple, los animales desarrollan lesiones de sustancia blanca repartidos por toda la sustancia blanca cerebral. Estas lesiones se pueden detectar de manera no invasiva mediante la realización de resonancia magnética. Figura 4 demuestra la capacidad de esta técnica para dilucidar el sustrato patológico de los resultados de la RM. Las pequeñas lesiones detectadas en la RM in vivo puederealizar un seguimiento tanto en la autopsia resonancia magnética y la histología. Como se muestra en las inserciones, desmielinización dentro de las lesiones es uno de los principales componentes que impulsan el cambio de señal MR (hiperintensidad en comparación con el tejido circundante). La histología postmortem y resonancia magnética también puede mostrar lesiones perdido en la RM in vivo (Figura 4).

Figura 1. Flujo de trabajo para la creación de un cuadro de la máquina de cortar tití cerebro. El cerebro se fija con formalina (A1) y un MRI ponderada en T2 se adquiere con voxels isotrópicos de 150 micras en el borde (A2). Las imágenes se procesan y thresholded para crear una máscara binaria (A3). La superficie se vuelve entonces en el software de modelado 3D (A4). Una resta booleano entre una plantilla de la máquina de cortar y el modelo de cerebro crea un modelo digital de la máquina de cortar de cerebro (B1). El cuadro de la máquina de cortar el cerebro se imprime en una impresora 3D (B2). Entonces, el cerebro se coloca firmemente en la caja de la máquina de cortar dede corte (B3). Haga clic aquí para ver una versión más grande de esta figura.
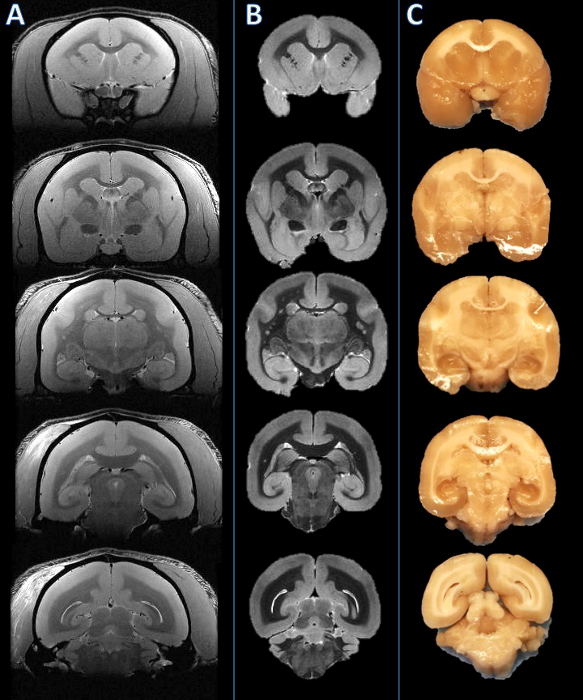
Figura 2. De izquierda a derecha: En vivo resonancia magnética, MRI post mortem, y la fotografía losa de tejido. Se establecieron planos de cortado basado en el MRI postmortem (B) y visualmente en comparación con el correspondiente in vivo MRI rebanada (A). a continuación, se cortó el cerebro, y se encontraron las losas resultantes para ser coherente (C). Haga clic aquí para ver una versión más grande de esta figura.
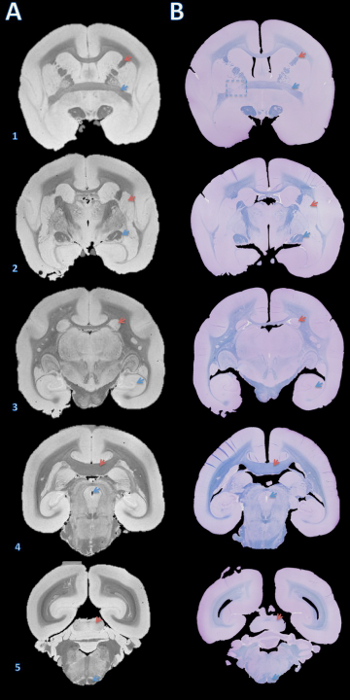
Figura 3. RM de alta resolución y la histología postmortemsección de acople. Las losas fueron incluidos en parafina, cortadas con un microtomo en 4 secciones micras, y se tiñeron con violeta rápido de cresilo y azul (B). Las secciones fueron emparejados visualmente con el 100 micras T2 * ponderadas MRI basado en estructuras del cerebro (A). Los detalles de la adquisición de esta imagen están en la sección complementaria del protocolo y en la Tabla 1. Las estructuras cerebrales: (1) la flecha roja = cápsula interna, flecha azul = comisura anterior; (2) Houthaven putamen, flecha azul = tracto óptico; (3) Houthaven caudado, flecha azul = hipocampo; (4) Houthaven cuerpo calloso, flecha azul = acueducto cerebral; (5) flecha roja = colículo inferior, flecha azul = tracto piramidal. El recuadro de trazos en B1 indica una rebanada cuando, de forma durante el corte de cerebro o de la inclusión en parafina, un error causado una ligera rotación alrededor del eje Y, lo que lleva a falta de coincidencia de la comisura anterior a la izquierda. Haga clic aquí para ver unaversión más grande de esta figura.
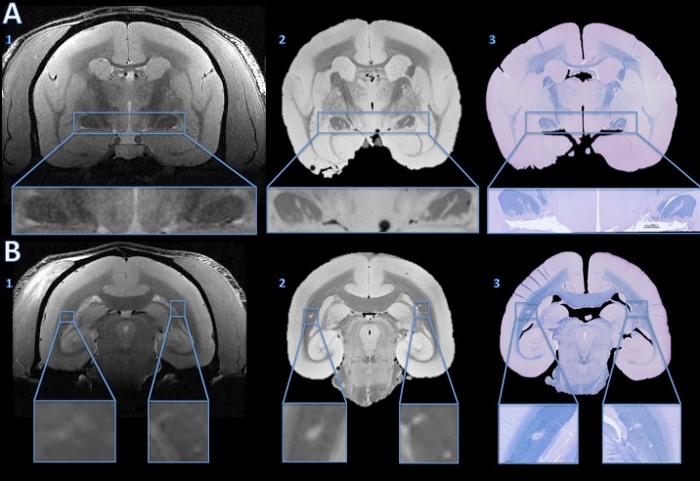
Figura 4. El seguimiento de las lesiones de resonancia magnética in vivo para la sección histología. La resonancia magnética in vivo mostró ninguna evidencia convincente de hiperintensidad de señal anormal que sugiera lesiones, ya sea en el tracto óptico (A1). Sin embargo, la alta resolución de la RM postmortem muestra líneas intensas hiper claras en ambas vías ópticas (A2). El ayuno azul / violeta de cresilo mancha de una sección de la histología 4 micras, muestra que las áreas hiperintensas visto en la ex vivo de resonancia magnética se mielina (A3). En la sustancia blanca cerebral, la resonancia magnética in vivo muestra sutil hiperintensidad bilateral (B1, ampliada en los recuadros). Las áreas hiperintensas son más evidentes en la autopsia resonancia magnética (B2) de alta resolución. La mancha LFB de una sección histología 4 micras muestra que estas áreas se desmielinizadas (B3). Después de la comparación con la línea base in vivo resonancia magnética y una mancha de hematoxilina-eosina y, del lado derecho se determinó que es una anormalidad anatómica, no una lesión desmielinizante. Haga clic aquí para ver una versión más grande de esta figura.

Archivos de código suplementarios. Brain_Slicer_Parts_Marmoset.stl: Haga clic aquí para descargar este archivo. Brain_Slicer_Parts_Human.stl: Haga clic aquí para descargar este archivo. Cap_Insert.stl: Haga clic aquí para descargar este archivo.
Discusión
El protocolo descrito aquí permite una comparación exacta entre la RM y secciones histológicas. El protocolo se presenta en un formato unificado que se puede aplicar a los cerebros de seres humanos o animales pequeños, tales como monos tití o roedores. Las diferencias específicas a grande (humana) y pequeñas (de primates no humanos y roedores) cerebros se ponen de relieve, y en el video y las figuras adjuntas que muestran la aplicación en el tití. Aunque el enfoque es sencillo, el método requiere muchos pasos, así como el uso de varios tipos de software. Por otra parte, hay varias cuestiones que puedan afectar a la exactitud de este método son importantes de mencionar.
La calidad de imagen de la resonancia magnética in vivo es un factor importante. Para minimizar la disparidad en la resolución de la imagen entre la RM y la histología imágenes digitalizadas, el tamaño de resonancia magnética voxel más pequeño posible se debe utilizar. Este concepto se aplica también a la calidad de imagen de la MRI postmortem. Mientras que el aumento de latiempo de adquisición en MRI postmortem permite mucho más alta resolución de la imagen, la preparación puede introducir artefactos de imagen tales como pérdidas de señal de coordinación relacionados con las burbujas de aire. Estos artefactos pueden ocultar áreas del tejido, así como afectar a su contorno. Por otra parte, las dimensiones del tejido en la autopsia MRI son propensos a ser afectados por el proceso de fijación y duración. Mientras que la in vivo para ex vivo partido de resonancia magnética se puede aproximar estrechamente mediante la utilización de puntos de referencia anatómicos en la configuración de la geometría rebanada durante la adquisición, el registro no lineal todavía sería necesario alcanzar un mayor grado de precisión en la adecuación de esas dos imágenes de resonancia magnética.
El diseño del soporte de la máquina de cortar cerebro y es también un paso crucial. En la creación del modelo digital del cerebro, se aplica un algoritmo de suavizado que agranda ligeramente el modelo en relación con el cerebro fijo. Esto permite una fácil inserción del cerebro en su soporte y la máquina de cortar y reduce los bordes afilados en el soporte y# 39; s contorno. Sin embargo, si el modelo es demasiado grande (por ejemplo, en más de un 5%), el cerebro podría moverse durante el postmortem MRI y / o el seccionamiento. Otro punto importante es adaptar el diseño del modelo de cerebro, de modo que el cerebelo está correctamente colocado en el interior del objeto 3D impreso. Esto puede ser particularmente difícil cuando el cerebelo ha sido dañado durante la extracción del cerebro en la autopsia.
Cuando la impresión de la máquina de cortar el cerebro y el soporte, del tipo de impresora 3D también deben ser elegidos cuidadosamente. Algunas impresoras de chorro múltiple requieren post-procesado utilizando un horno para eliminar el material de soporte. Si bien estas impresoras pueden producir objetos que son a prueba de agua y relativamente más duradero que las impresoras de escritorio fusionado modelado por deposición (FDM), el proceso de calentamiento para eliminar soportes pueden deformar ligeramente la caja, la creación de lagunas de cuchilla que no son perfectamente perpendicular al contorno cerebro.
El proceso cerebral de seccionamiento es otro paso crucial. Antes de cortar THe de todo el cerebro en losas, es importante asegurarse de que el cerebro está sentado firmemente dentro de la máquina de cortar de cerebro: no debe haber movimiento cuando se aplica una ligera presión en el cerebro. Esto hará posible que las cuchillas corten a través del cerebro en el lugar preciso establecido por los investigadores. Una presión continua, equilibrada debe aplicarse a ambos soportes de cuchilla durante el corte. Dependiendo del filo de las cuchillas y la rigidez del tejido, un ligero movimiento de corte transversal podría ser ventajoso para el mantenimiento de superficies de corte planas.
El proceso de inclusión en parafina también puede ser una fuente de falta de alineación entre la resonancia magnética y la histología. Si la losa tejido no está sentado plana contra la casete durante el proceso de incrustación, habrá una inclinación entre el plano de corte de la micrótomo y el lugar superficie de la losa. Esto requerirá cortar secciones inutilizables para encontrar una superficie plana en la que está expuesta todo el tejido. Una manera de corregir la inclinaciónes cambiando el ángulo del plano de visualización en el MRI postmortem de alta isotrópico-resolución. Sin embargo, esto es casi imposible de realizar en el vivo MRI en que por lo general se adquiere con la resolución anisotrópico (típicamente gruesas rebanadas coronales).
Finalmente, el tejido puede experimentar una cierta deformación durante el período de la fijación con formalina y la incrustación de parafina (contracción), así como durante la preparación de diapositivas (plegable, grietas, arrugas). Algunas de estas deformaciones se pueden corregir poniendo las secciones 4-5 micras en un baño de agua antes de ser transferido en portaobjetos. Otros deformaciones se pueden resolver parcialmente mediante la realización de coregistration imagen deformable de las imágenes digitalizadas histológicos a las imágenes de resonancia magnética post mortem. Sin embargo, lo que minimiza las deformaciones con la práctica cuidadosa y hábil es el método más eficaz de igualar los volúmenes de resonancia magnética a la histología secciones.
En conclusión, la metodología presentada aquí permite investigators para evaluar con precisión la patología subyacente de los resultados de la RM. Más en general, es un enfoque prometedor para identificar y / o validar nuevos biomarcadores de resonancia magnética para estudios de investigación que se dirigen a procesos patológicos específicos, tales como la inflamación o la remielinización.
Divulgaciones
The authors declare that they have no competing financial interests.
Agradecimientos
The Intramural Research Program of NINDS supported this study. We thank the NIH Functional Magnetic Resonance Imaging Facility. We thank Jennifer Lefeuvre and Cecil Chern-Chyi Yen for assistance with postmortem MRI acquisition. We thank John Ostuni and the Section on Instrumentation Core Facility for assistance with 3D printing. Figure 1 of this work used snapshots from MeshLab, a tool developed with the support of the 3D-CoForm project.
Materiales
| Name | Company | Catalog Number | Comments |
| 7T/30cm USR AVIII Bruker MRI | Bruker Biospin | ||
| 38 mm Bruker Biospin volume coil | Bruker Biospin | ||
| Fomblin | Solvay Solexis | ||
| 50 ml Falcon Centrifuge Tubes, Polypropylene, Sterile | Corning | 21008-951 | |
| Fisherbrand Gauze Sponges | Fisher Scientrific | 13-761-52 | |
| Parafilm M All-Purpose Laboratory Film | Bemis | ||
| Leica RM2235 rotary microtome | Leica | ||
| Leica Disposable Blades, low profile (819) | Leica | ||
| Cresyl Violet Acetate, 0.1% Aqueous | Electron Microscopy Sciences | 26089-01 | |
| Luxol Fast Blue, 0.1% in 95% Alcohol | Electron Microscopy Sciences | 26056-15 | |
| ETOH | |||
| Ultimaker 2 Extended | Ultimaker | ||
| .75 kg Official Ultimaker Branded PLA Filament, 2.85 mm, Silver Metallic | Ultimaker | ||
| Axio Observer.Z1 | Zeiss | ||
| Zen 2 (Blue Edition) | Zeiss | ||
| Netfabb Professional 5.0.1 | Netfabb | http://www.netfabb.com/professional.php | |
| Meshmixer 10.9.332 | Autodesk | http://www.meshmixer.com/download.html | |
| Mipav 7.2 | NIH CIT | http://mipav.cit.nih.edu | |
| Cura | Ultimaker | https://ultimaker.com/en/products/cura-software |
Referencias
- Evans, A. C., Frank, J. A., Antel, J., Miller, D. H. The role of MRI in clinical trials of multiple sclerosis: comparison of image processing techniques. Ann Neurol. 41 (1), 125-132 (1997).
- 't Hart, B. A., van Kooyk, Y., Geurts, J. J. G., Gran, B. The primate autoimmune encephalomyelitis model; a bridge between mouse and man. Ann Clin Transl Neurol. 2 (5), 581-593 (2015).
- Ontaneda, D., Hyland, M., Cohen, J. A. Multiple sclerosis: new insights in pathogenesis and novel therapeutics. Annu Rev Med. 63, 389-404 (2012).
- Guy, J. R., Sati, P., Leibovitch, E., Jacobson, S., Silva, A. C., Reich, D. S. Custom fit 3D-printed brain holders for comparison of histology with MRI in marmosets. J Neurosci Methods. 257, 55-63 (2016).
- Breen, M. S., Lazebnik, R. S., Wilson, D. L. Three-dimensional registration of magnetic resonance image data to histological sections with model-based evaluation. Ann Biomed Eng. 33 (8), 1100-1112 (2005).
- Dauguet, J., et al. Three-dimensional reconstruction of stained histological slices and 3D non-linear registration with in-vivo MRI for whole baboon brain. J Neurosci Methods. 164 (1), 191-204 (2007).
- McGrath, D. M., Vlad, R. M., Foltz, W. D., Brock, K. K. Technical note: fiducial markers for correlation of whole-specimen histopathology with MR imaging at 7 tesla. Med Phys. 37, 2321-2328 (2010).
- Schormann, T., Zilles, K. Three-Dimensional linear and nonlinear transformations: An integration of light microscopical and MRI data. Hum Brain Mapp. 6, 339-347 (1998).
- Jiang, L., et al. Combined MR, fluorescence and histology imaging strategy in a human breast tumor xenograft model. NMR Biomed. 26 (3), 285-298 (2013).
- Absinta, M., et al. Postmortem magnetic resonance imaging to guide the pathologic cut: individualized, 3-dimensionally printed cutting boxes for fixed brains. J Neuropathol Exp Neurol. 73 (8), 780-788 (2014).
- Gaitán, M. I., et al. Perivenular brain lesions in a primate multiple sclerosis model at 7-tesla magnetic resonance imaging. Mult Scler. 20 (1), 64-71 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados