Method Article
Mejora de la reacción en cadena de polimerasa-polimorfismo de longitud de fragmentos de restricción Genotipado de Pez globo Tóxico por cromatografía líquida / espectrometría de masas
En este artículo
Resumen
Se describe un método de longitud de los fragmentos de restricción polimórficos-reacción en cadena de polimerasa mejorado para el genotipado de especies pufferfish por espectrometría de cromatografía / masa líquida. Una columna monolito de sílice de fase inversa se emplea para la separación de amplicones digeridos. Este método puede dilucidar las masas monoisotópicas de oligonucleótidos, que es útil para la identificación de la composición base.
Resumen
Una versión mejorada de un método para la genotipificación de las especies pufferfish tóxicos por cromatografía / ionización por electrospray espectrometría de masa líquida (LC / ESI-MS) reacción en cadena de la polimerasa (PCR) fragmento -restriction polimorfismo de la longitud (RFLP) se describe. La extracción de ADN se lleva a cabo utilizando un kit de extracción de ADN basado en membrana de sílice. Después de la amplificación por PCR usando un tampón de PCR libre de detergente, enzimas de restricción se añaden a la solución sin la purificación de la solución de reacción. Una columna monolito de sílice de fase inversa y una transformada de Fourier espectrómetro de masas de alta resolución con un analizador de trampa de Kingdon modificado se emplean para la separación y detección, respectivamente. La fase móvil, que consiste en 400 mM 1,1,1,3,3,3-hexafluoro-2-propanol, 15 trietilamina mM (pH 7,9) y metanol, se entrega a una velocidad de flujo de 0,4 ml / min. El tiempo de ciclo para el análisis / ESI-MS LC es 8 min incluyendo equilibrado de la columna. software de deconvolución que tiene un modelo de distribución de isótopos of el oligonucleótido se utiliza para calcular la masa monoisotopic correspondiente del espectro de masas. Para el análisis de los oligonucleótidos (rango 26-79 nucleótidos), exactitud de la masa fue de 0,62 ± 0,74 ppm (n = 280) y una excelente precisión y equivalencia se mantiene durante 180 horas sin el uso de un patrón de masa de bloqueo.
Introducción
Espectrometría de masas (MS) es una tecnología aceptada para la identificación rápida y fiable de ácidos nucleicos, láser asistida por matriz de desorción / ionización (MALDI) y la ionización por electrospray (ESI) que se utiliza para la ionización 1,2. La técnica MALDI se combina típicamente con un tiempo de vuelo (TOF) analizador; Sin embargo, la aplicación de MALDI-TOF MS se limita a oligonucleótidos cortos (~ 25 nucleótidos (nt)) debido a la eficiencia de la posterior fragmentación, la formación de aductos y baja ionización 1,2. En contraste, ESI es aplicable a oligonucleótidos más largos (> 100 nt), pero muchos estados de carga de múltiples iones cargados ([M-NH] n-) se producen de forma simultánea desde biomacromoléculas y, por lo tanto, la masa del analito puede exceder de la parte superior límite del rango de m / z del espectrómetro. Esto requiere la interpretación de los espectros de enrollado, es decir, la transformación de una serie estado de carga en la masa correspondientea través de deconvolución.
En el caso de medición de la masa de una molécula pequeña, el pico de la masa monoisotópica, es decir, la masa de la molécula que contiene sólo el isótopo más común de cada elemento es el más abundante y se observa naturalmente 3. A medida que aumenta el peso molecular, los cambios de distribución isotópicas a mayor m / z y el pico monoisotopic se oscurece por el ruido de línea de base 3-5. Una vez que el pico monoisotopic ya no es detectable para masas mayores que 10 kDa 3, la masa molecular media se utiliza para la medición en lugar de la masa monoisotópica 5. En tales casos, la distribución isotópica en la que cada uno de los picos individuales se separa por 1 Da sólo puede ser observada cuando un analizador de masas de alta resolución tales como TOF, una resonancia de ciclotrón de iones de transformada de Fourier modificada Kingdon analizador de trampa 6, o una transformada de Fourier analizador se utiliza para el análisis. Sin embargo, el pico más abundante es slgunas veces no es consistente con la masa molecular media 5. Estos problemas pueden conducir a la dificultad para determinar con exactitud los analitos.
Teniendo en cuenta la variación en la abundancia natural de isótopos estables y las dificultades en la determinación de la masa molecular promedio, medida de la masa monoisotópica es ideal para la caracterización de masas de las biomoléculas 3,4. En la práctica, si un pico monoisotopic se puede observar o no, la masa monoisotópica se puede estimar comparando el patrón de la distribución isotópica observado que la teórica calculada a partir de un modelo de analito 4,7-10. El algoritmo de ajuste 8 se ha incorporado en el software propietario.
En el contexto de la ESI-MS, se requiere que la disociación de un dúplex de ADN, purificación y desalinización para la medición directa para evitar el fracaso de ionización debido a la supresión de iones y la formación de aducto de 2,10-14. Estos procedimientos son troublesome, sin embargo, los sistemas de análisis completamente automatizados se han desarrollado comercialmente que implica la reacción en cadena de polimerasa (PCR), preparación de muestras y ESI-MS para la detección de patógenos 15-20. Otro enfoque es la introducción de cromatografía líquida (LC) para la separación. LC proporciona una separación en línea de los analitos a partir de sustancias coexistentes y no requiere una laboriosa preparación de la muestra antes de la ionización 21,22. Sin embargo, la separación de ácidos nucleicos usando una fase móvil compatible-MS es más difícil que para la mayoría de otros compuestos tales como fármacos, péptidos y proteínas debido a la naturaleza polianiónica de los nucleótidos. Los ejemplos más exitosos de LC / ESI-MS implican el uso de pares de iones en fase inversa LC. Una fase móvil de 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP) -triethylamine (TEA) metanol fue propuesto inicialmente por Apffel et al. Para la separación y detección de oligonucleótidos cortos 23. Aplicación de LC / ESI-MS para el genotipado differentiation de las especies de patógenos, polimorfismos de un solo nucleótido y repeticiones cortas en tándem ha informado utilizando una columna capilar de polímero monolito que puede separar oligonucleótidos más largos 1,21,24-33.
En Japón y Estados Unidos, la intoxicación grave de alimentos se ha producido debido a errores de identificación y preparación inadecuada de pez globo, y esto es a pesar del hecho de que la distribución y preparación de pez globo está estrictamente controlado por la legislación de seguridad alimentaria 34,35. Por otra parte, un homicidio intencional uso de extracto de pez globo se produjo en Japón 36. Por lo tanto, se requiere la diferenciación de las especies pufferfish de la salud pública y puntos de vista de investigación forense. Además, debido a Takifugu rubripes es mucho más caro que otros tipos de pez globo, también se necesita una capacidad de diferenciación en el contexto de las investigaciones de fraude alimentos.
Aquí, un método detallado para determinar la mse describe masa onoisotopic de un producto de PCR por LC / ESI-MS usando una columna de monolito de sílice de fase inversa y un espectrómetro de masas de alta resolución. En concreto, el enfoque ha sido desarrollado para permitir la diferenciación de las especies pufferfish tóxicos basados en el uso del método de polimorfismo de la longitud 37 del fragmento de restricción PCR (RFLP), que es el primer ejemplo para la diferenciación de especies de animales utilizando MS.
Protocolo
1. Extracción de ADN
Nota: Utilice una habitación separada para los exámenes de extracción de ADN y post-PCR tales como electroforesis en gel y LC / ESI-MS. Añadir etanol al tampón de lavado 1 (que contiene hidrocloruro de guanidina) y tampón de lavado 2 (que no contiene hidrocloruro de guanidina) de acuerdo con el protocolo del kit de extracción de ADN. Las muestras de peces se obtuvieron de mayoristas de pescado y mercados al por menor en Japón.
- Coloque 30-50 mg de tejido de los peces en un tubo de 0,5 ml o 1.5 ml. Squash el tejido utilizando un micro-espátula (a excepción de la aleta y la piel).
Nota: En el caso de la aleta, utilizar un tubo de 0.5 ml para el remojo. - Añadir 180 l de tampón de lisis y 40 l de proteinasa K y vórtice brevemente. Se incuba a 56 ° C en un calentador de bloque durante 2 horas o durante la noche. Mezclar por agitación brevemente cada 15 minutos durante la incubación.
Nota: la lisis completa no es necesaria. - Centrifugar durante 5 min a 13.000 x g. Después de la centrifugación, si una pequeña amount de aceite existe en la superficie en el caso de una muestra grasos tales como el hígado, descartarlo. Transferir el sobrenadante a un nuevo tubo de microcentrífuga de 1,5 ml.
- Añadir 200 l del tampón caotrópico que contiene hidrocloruro de guanidina y mezclar mediante agitación. Añadir 200 l de etanol (99,5%) y mezclar mediante agitación de nuevo.
- Transferir la mezcla en la columna de centrifugación en un tubo de recogida de 2 ml. Centrifugar durante 1 min a 13.000 x g. Desechar el flujo continuo.
Precaución: No añadir cloro para los residuos de guanidina, porque puede formar compuestos altamente reactivos con lejía. - Añadir 500 l de tampón de lavado 1 (que contiene hidrocloruro de guanidina) y centrifugar durante 1 min a 13.000 x g. Desechar el flujo a través y el tubo de recogida.
- Colocar la columna de centrifugación en un nuevo tubo de recogida de 2 ml provisto con el kit. Añadir 500 l de tampón de lavado 2 y centrifugar durante 3 minutos a 20.000 x g. Desechar el flujo continuo y el tubo de recogida. Transferir la columna de centrifugación a un nuevo1,5 ml tubo de microcentrífuga.
- Pipetear 200 l de tampón de elución al centro de la membrana de la columna de centrifugación. Después de 1 min, se centrifuga durante 1 min a 6.000 x g.
- Pipetear 50 l de eluato a una cubeta UV desechable y medir el espectro de UV del eluato a 220 a 300 nm y la absorbancia a 260 nm usando un espectrofotómetro.
- Verificar el espectro UV del ADN con el pico a 260 nm. Calcular la concentración de ADN aproximada usando la ecuación simple "concentración de ADN (ng / l) = absorbancia a 260 nm x 50".
Nota: la concentración de ADN típico extraído de las aletas del pez globo es 63 ± 30 ng / l (n = 20). - Si la concentración ADN es mayor que 10 ng / l, diluir las muestras de ADN con EDTA-Tris tampón (TE) (pH 8) a 5 ng / l.
- Conservar la muestra a -20 ° C o proceder a la amplificación por PCR (sección 2).
2. PCR
- Colocada a 25 l de PCR para cada extractuado muestra de ADN, control positivo (0,2 M de oligonucleótidos sintetizados que tiene la secuencia diana en tampón TE pH 8,0) y el control negativo (agua ultrapurificada en lugar de muestra de ADN) en hielo según las Tablas 1 y 2 en un banco limpio para evitar la contaminación.
Nota: Para una fácil operación, haga cantidad suficiente de cóctel que contiene todos los reactivos sin ADN molde y prescindir de él. Utilice la polimerasa que tiene actividad de corrección de pruebas de ADN para evitar la adición de 3 'salientes para el producto de PCR. - Llevar a cabo la amplificación por PCR en un termociclador usando el programa del ciclo se muestra en la Tabla 3.
3. La digestión enzimática
- Añadir 2 l de la solución que contiene 100 mM Tris-HCl (pH 7,5), 100 mM MgCl 2, ditiotreitol 10 mM y NaCl 500 mM a la solución de PCR.
- Añadir 1 l de cada enzima de restricción (Dra I y Msp I) y mezclar suavemente. Incubar durante 30 min a 37 ° C en el THciclador ermal.
- Incubar durante 5 min a 72 ° C para activar la polimerasa de DNA restante, que llena el extremo cohesivo generado por Msp I.
- Opcional: analizar cada 2,5 l de la solución de reacción por electroforesis en gel de poliacrilamida utilizando una relación transversal enlace (% C, porcentaje de bisacrilamida) de 3,33 (% en peso) y una concentración de gel de poliacrilamida (% T) de 15 (% w / v) 38. Teñir el gel utilizando la mancha de ADN fluorescente y observar el ADN de doble cadena en un transiluminador de luz azul utilizando una pantalla de color ámbar.
- Se filtra la solución de reacción con un dispositivo de filtro centrífugo 0,2-0,5 micras para evitar la obstrucción, lo que dañaría la columna analítica.
- Transferir el filtrado a un vial de polipropileno cónico y se almacena a -20 ° C hasta su análisis.
4. LC / ESI-MS Analysis
- Pesar 67,2 g (aproximadamente 42 ml) de HFIP y 1,52 g (aproximadamente 2,0 ml) de TEA en una botella de 1 litro. Añadir 955 ml de agua ultrapurificada (LC / MS grade) y se agita utilizando un agitador magnético hasta que los reactivos se disolvieron completamente. Tomar una alícuota de la solución y comprobar el pH (entre 7,85 y 7,95, lo ideal pH = 7,9) utilizando un medidor de pH.
Nota: No devuelva la alícuota a la botella para evitar la contaminación por iones metálicos. Si el pH no es 7,9, añadir una pequeña cantidad de cualquiera de HFIP o TEA para ajustar el pH y medir el pH de nuevo. - Conectar la botella a la línea A del cromatógrafo líquido con el enfriamiento de la botella usando una nevera portátil de enfriamiento directo (para uso doméstico) para evitar la vaporización. Conectar otra botella llena de metanol a la línea B.
- Conectar la precolumna y la columna analítica (2,0 x 50 mm, de mesoporos size = 30 nm) en el cromatógrafo de líquidos. Comienzan a fluir la fase inicial móvil (95% de A y 5% de B) para equilibrar las columnas.
- Preparar 0,5 mg / ml de sodio trifluoroacetato (pH 3,5) como un calibrador 39.
- Pesar 50 mg (aproximadamente 34 l) de corriente alterna trifluoroacéticoIdentificación en un vaso de precipitados.
- Añadir 30 ml de agua ultrapurificada y se valora a pH 3,5 con hidróxido de sodio 10 mM con la ayuda de un medidor de pH.
- Llenar hasta 50 ml con agua ultrapurificada. Añadir 50 ml de acetonitrilo (LC / MS de grado) y mezclar.
- Tomar una porción de la solución de trabajo y almacenarlo a temperatura ambiente. Almacenar el resto de la solución a 4 ° C.
- Reducir la presión de nitrógeno de la C-trampa de la siguiente manera.
Nota: Recent transformada de Fourier espectrómetros de masas, que son adecuados para el análisis de la proteína intacta, tener un software de control para controlar la presión.- Retire la cubierta superior izquierdo del espectrómetro de masas. La válvula C-trampa está en la esquina de la izquierda del primer plano.
- Compruebe el valor de presión de alto vacío en la ventana de estado del instrumento del software de control.
Nota: El valor es normalmente, 3 x 10 -8 bar. - Tire de la perilla para desbloquear y girar el mando en sentido antihorario muy lentamente para reducir el alto prensa de vacíoUre a 1/3 (típicamente, 1 x 10 -8 bares). La presión indicada debe cambiar gradualmente de una manera retardada. Ignorar el mensaje de advertencia acerca de baja presión. Empuje el botón para cerrar.
- Restaurar la cubierta superior para la seguridad.
- Calibrar el espectrómetro de masas usando trifluoroacetato de sodio 39.
Nota: La calibración de masa diaria puede ser sustituido por el presente protocolo, sin embargo, la calibración recomendada sugerido por el fabricante debería haberse completado antes de esta calibración.- Establecer parámetros de análisis y parámetros calentados fuente de ESI en el cuadro de control de instrumentos del software de ajuste de la siguiente manera: Tipo de escaneo completo de MS; rango de exploración m / z 500-4.000; la fragmentación (en fuente de disociación inducida por colisión (CID) Tensión de 60 eV; polaridad negativa; control automático de ganancia (AGC) Objetivo 1 x 10 6, el tiempo máximo de inyección 50 ms; caudal de gas 5 vaina; aux caudal de gas 0; gas de barrido tasa de flujo 0; rocíe tensión de 3 kV; temperatura del capilar 320 ° C; S-lente de radiofrecuencia (RF) de nivel 100; temperatura del calentador de 30 ° C.
- Entrada de la lista de los iones negativos deben controlarse de m / z 792,85908, 1064.80870, 1336.75832, 1608.70794, 1880.65755, 2152.60717, 2424.55679, 2696.50640, 2968.45602, 3376.38045.
- Mover la sonda ESI calienta más cerca de la interfaz del espectrómetro de masas (posición B). Conectar la sonda y una jeringa con un tubo.
- Infundir el calibrante constantemente a una velocidad de flujo de 10 l / min utilizando la bomba de jeringa incrustado. Comprobar sólo los 6 iones de baja masa (m / z) 792,85908-2.152,60717 en la tabla de calibración personalizado.
- Proceder con la calibración después de la intensidad de las señales se estabilizan. Después de la calibración, comprobar sólo los seis iones de mayor masa (m / z) 1880,65755-3.376,38045 y proceder con la calibración. Evitar la calibración con todos los iones a la vez para evitar el fracaso.
- Establecer parámetros de análisis y parámetros calentados fuente de ESI en el cuadro de control de instrumentos del software de ajuste de la siguiente manera: Tipo de escaneo completo de MS; rango de exploración m / z 500-4.000; la fragmentación (en fuente de disociación inducida por colisión (CID) Tensión de 60 eV; polaridad negativa; control automático de ganancia (AGC) Objetivo 1 x 10 6, el tiempo máximo de inyección 50 ms; caudal de gas 5 vaina; aux caudal de gas 0; gas de barrido tasa de flujo 0; rocíe tensión de 3 kV; temperatura del capilar 320 ° C; S-lente de radiofrecuencia (RF) de nivel 100; temperatura del calentador de 30 ° C.
- Mover la sonda a la apropiaciónPosición TE para una velocidad de flujo de 0,4 ml / min (posición D) y conectar al cromatógrafo líquido con un tubo de metal inerte.
- Establecer los parámetros de fuente ESI calentados en el cuadro de control de instrumentos del software de ajuste de la siguiente manera: vaina de caudal de gas 50; caudal de gas auxiliar 15; barrer caudal de gas 1; rocíe tensión de 2,5 kV; temperatura capilar 350 ° C; S-lente nivel de RF 100; temperatura del calentador de 350 ° C.
- parámetros establecidos de adquisición del espectrómetro de masas en la ventana de configuración instrumental como se indica a continuación: Duración método 8 min; desviar la válvula, 3,5-6 min a espectrómetro de masas, 0-3.5 y 6-8 min a perder; tiempo de ejecución de 3,5 a 6 min; polaridad negativa; en el voltaje de fuente CID 15,0 eV; microscans 3; poder de resolución nominal (en m / z 200) 140 000; AGC objetivo 1 x 10 6; tiempo de iones máximo de 50 ms; número de exploraciones 1; rango de exploración m / z 700-3,500; espectro perfil tipo de datos.
- Establecer parámetros de cromatógrafo de líquidos de la siguiente manera: la columna de temperatura del horno 20 ° C; samplTemperatura en la bandeja de correo de 10 ° C; programa de gradiente 0-0,5 min B al 5%, 0,5-1 min 5-30% de B, 1-3,5 min 30-40% de B, 3,5-5 min 40-98% de B, 5-6 min 98% de B, 6- 6,05 min 98-5% de B, 6,05-8 min 5% de B; velocidad de flujo 0,4 ml / min.
- Purga de burbujas tocando el fondo de los viales. Establecer los viales de muestra en el soporte de muestras e inyectar 1,0 l de una muestra utilizando el inyector automático para iniciar el análisis.
- Abra archivos de datos brutos con el software del navegador y confirman que todas las adquisiciones están terminados con éxito, incluyendo los controles positivos y negativos.
Nota: Típicamente, se observarían dos o tres picos en el cromatograma corriente de iones total en un tiempo de retención de 4 a 5,5 min cuando el ADN pez globo se amplifica y se digirió con éxito.
5. La deconvolución e Interpretación
- Ejecutar el software de deconvolución. Seleccione el tipo de experimento de "extracto de automóviles (con isótopos resueltos)".
- Editar un método que utiliza los siguientes parámetros: masa M de salida; S / N threshold 3, umbral de abundancia relativa 0; carga negativa que sí; Calcular extraída de iones cromatograma actual (XIC) sí; m / z gama 700-3,500; portador de carga H +; número mínimo de carga detectado 3; la proporción de isótopos de nucleótidos; factor de ajuste de 80%; umbral resto 0%; en consideración los solapamientos sí; cargar rango 3-50; intensidad mínima 1; error de intensidad esperada 3.
- Guardar como un nuevo método y seleccionarlo.
- Seleccione el directorio donde se encuentran los archivos de datos en bruto. Seleccione los archivos de datos en bruto que hay que analizar y haga clic en "Agregar a la cola" botón.
- Haga clic en el botón "Ejecutar" en la pestaña "Ejecutar cola 'para iniciar la deconvolución.
- Una vez completada la cola, seleccione una fila y haga clic en "Resultado abierto 'en el título superior para obtener los resultados de deconvolución.
- Interpretar los resultados de las masas monoisotópicas derivados de los picos a un tiempo de retención de 4-5,5 min usando la Tabla 4.
Resultados
Se evaluaron tres columnas disponibles en el mercado para la separación de oligonucleótidos largos a velocidades de flujo de 100 a 400 l / min. Columna de sílice en partículas -bonded A octadecilo amplia de poros cadena de carbono (C 18) (a), un poli disponible comercialmente (estireno-divinilbenceno) (PS-DVB) columna monolito (b) y una columna de sílice monolito -bonded C 18 ( c) se compararon (Figura 1). Tres pares de los dúplex de ADN (26, 37 y 53 pb) se han separado con todas las columnas, y se utilizó la columna de sílice monolito -bonded el C18 en estudios posteriores.
Los resultados representativos para el análisis de una muestra de ADN extraída del músculo de T. rubripes se muestra en la Figura 2. picos isotópicamente resueltas, como se muestra en la Figura 2a, se requieren para el cálculo con éxito la masa monoisotópica. El teórico y measmonoisotopic masa ured de T. rubripes se muestran en la Figura 2b. Debido a que la digestión con las endonucleasas se lleva a cabo justo después de la amplificación por PCR sin purificación, el extremo 3'-pegajoso generada por la endonucleasa de Msp I se llena con la polimerasa de ADN restante. Según el análisis de los amplicones derivados de las plantillas de ADN sintéticos, exactitud de masa varió -2,48 a 2,40 ppm (0,62 ± 0,74 ppm en promedio, n = 280). Por lo tanto, se usó una tolerancia de masa de 3 ppm para la diferenciación de especies (Tabla 4). Utilizando la tabla, todas las muestras obtenidas de pufferfish mercados y tiendas se diferenciaron adecuadamente como especies específicas 37.
De acuerdo con el análisis periódico de la muestra obtenida del molde de ADN sintético de T. chrysops, todas las masas monoisotopic de los analitos se determinaron con éxito dentro de ± 3 ppm para en least 180 horas sin ningún tipo de calibración de masa (Figura 3). Esto significa que sería necesario realizar una calibración de masa sobre una base semanal.
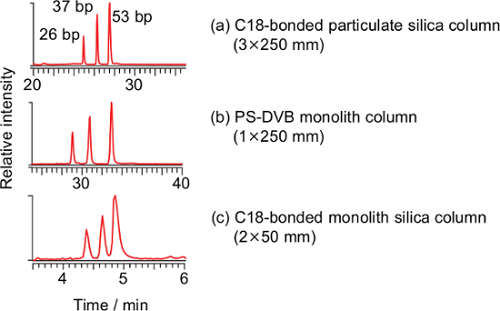
Figura 1: Número total de cromatogramas de corriente de iones del producto de PCR-RFLP derivado de la plantilla de ADN sintetizada de T. pardalis usando una columna C 18 -bonded sílice en partículas (a, 3 × 250 mm, tamaño de partícula 3 micras), un poli (estireno-divinilbenceno) (PS-DVB) columna monolito (b, 1,0 x 250 mm) y un C 18 -bonded columna de sílice monolítico (c, presente método) Condiciones para (a):. velocidad de flujo de 0,2 ml / min; proporción de metanol, 0-2 min 5%, 2-5 min 5% -25%, 5-20 min 25% -40%, 20-35 min 40% -98%, 35-50 min 98%, 50- 51 min 98% -5%, 51 a 65 min 5%. Condiciones para la (b):velocidad de flujo 0,1 ml / min; proporción de metanol, 0-2 min 5%, 2-5 min 5% -25%, 5-35 min 25% -40%, 35-40 min 40% -98%, 40-55 min 98%, 55- 56 min 98% -5%, 56 a 70 min 5%. La temperatura para ambas columnas fue de 20 ° C. Por favor, haga clic aquí para ver una versión más grande de esta figura.
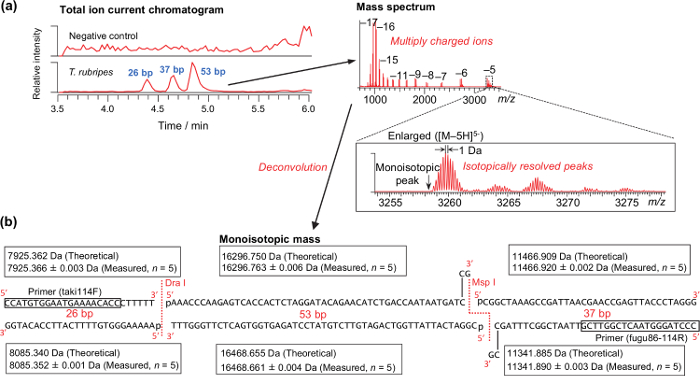
Figura 2: Los resultados representativos para el análisis muscular cruda de T. Rubripes. (a) cromatogramas de iones totales actuales típicos y espectro de masas. (B) masas monoisotopic teóricos y de medición de los productos de PCR-RFLP derivados de la plantilla de ADN sintetizada de T. Rubripes. Haga clic aquí para ver una granr Versión de esta figura.

Figura 3:. Ensayo de estabilidad digerido amplicones derivados de ADN sintético de T. chrysops se analizaron cada 10 hr. La calibración de masa no se llevó a cabo durante la prueba. Por favor, haga clic aquí para ver una versión más grande de esta figura.
| Nombre | Secuencia |
| lago86F | 5'-3'-CCATGTGGAATGAAAACACC |
| taki114F | 5'-3'-AAAAACAAGAGCCACAGCTCTAA |
| fugu86-114R | 5'-3'-CCCTAGGGTAACTCGGTTCG |
Cuadro 1: cebadores de PCR.
| reactivo de PCR | volumen utilizado | La concentración final |
| agua ultrapurificada | 15,75 l | |
| -Detergente libre 5x Buffer | 5.0 l | 1x |
| mezcla de dNTP (10 mM cada uno) | 0,5 l | 0,2 mM |
| mezcla de cebadores (10 mM de cada uno de lago86F, taki114F y fugu86-114R en tampón TE pH 8,0) | 1.0 l | 0,4 mM cada uno |
| ADN polimerasa (2,5 unidades / l) | 0,25 l | 0,1 unidades / l |
| Plantilla de ADN en TE tampón de pH 8,0 (5,0 a 10 ng / l para la muestra extraída, 0,2 M de ADN sintético como control positivo) | 2,5 l | 0,5-1,0 ng / l de ADN extraído y 20 nM para el ADN sintético |
| Total: 25 l |
Tabla 2: Componentes de una escala de 25 l de PCR.
| Ciclo | Condición | Función |
| 1 | 2 min a 95 ° C | La desnaturalización inicial |
| 2 | 30 s a 95 ° C | La desnaturalización |
| 3 | 30 s a 56 ° C | Recocido |
| 4 | 30 s a 72 ° C | Alargamiento |
| 5 | Repita 2-4 (en total 30 ciclos) | |
| 6 | 7 min a 72 ° C | elongación final |
| 7 | Se mantiene a 12 ° C hasta la retirada de la muestra |
Tabla 3: programa de PCR.
| número pico a un tiempo de retención de 4,0 a 5,5 min | monoisotopic masa del tercer pico en el rango (Da) | monoisotopic masa del segundo pico en el rango (Da) | pufferfish especies | ||
| cadena más pequeña | cadena más grande | cadena más pequeña | cadena más grande | ||
| 2 | → | → | 15.890,707-15.890,803 | 16.177,531-16177,629 | L. inermis |
| 15.921,702-15.921,797 | 16146,537-16146,634 | L. gloveri | |||
| 15.930,713-15.930,809 | 16.137,525-16137,622 | L. lunaris | |||
| 15937,697-15.937,792 | 16131,537-16.131,634 | L. wheeleri | |||
| 24.173,034-24.173,179 | 24.566,905-24.567,053 | T. chrysops | |||
| 3 | 16295,706-16295,804 | 16.467,610-16.467,709 | 11341,851-11341,919 | 11466,875-11466,944 | T. pardalis T. snyderi T. ocellatus T. xanthopterus T. stictonotus |
| 11654,908-11654,978 | 11.770,920-11.770,991 | T. niphobles | |||
| 16296,701-16296,799 | 16.468,605-16.468,704 | → | → | T. rubripes | |
| 16.311,701-16.311,799 | 16452,610-16.452,709 | → | → | T. poecilonotus T. exascurus | |
| 16.320,713-16.320,811 | 16.443,599-16443,697 | → | → | T. porphyreus T. obscurus | |
| 16.599,751-16.599,851 | 16.780,667-16.780,767 | → | → | T. vermicularis | |
Tabla 4: Diferenciación de pez globo de masas monoisotopic derivados de LC / ESI-MS.

Tabla 5: Amplified secuencia de ADN (a) la secuencia es idéntica a.la de las otras especies pufferfish como se describe en la Tabla 4. Haga clic aquí para ver una versión más grande de esta tabla.
Discusión
Para extraer el ADN, un kit comercial para la extracción de ADN de la sangre y los tejidos se utiliza de acuerdo con el protocolo del fabricante, con modificación menor (cantidad de proteinasa K y la centrifugación de la solución de lisis). Sin embargo, cualquier kit de extracción se puede utilizar siempre y cuando el ADN celular se puede extraer con la recuperación y la pureza apropiada para PCR. Este método ha sido probado usando muscular, aleta, hígado, ovario, y piel 37. Fin es especialmente adecuado debido a la gran superficie, lo que permite una rápida lisis con proteinasa K. frescos, congelados, secos, hervidos y las muestras pufferfish horneados fueron probados con éxito 37.
El conjunto de cebadores de PCR (Tabla 1) se ha diseñado para el pez globo y amplifica el gen mitocondrial 16S ribosomal RNA de pez globo. El ADN diana para amplificar es 114 a 115 pb (género Takifugu) y 86 pb (género Lagocephalus) (Tabla 5). Para facilitar método development, se utilizaron oligonucleótidos sintetizados que tienen las secuencias diana para la referencia en lugar de la recogida de muestras pufferfish auténticos. El conjunto de cebadores puede ser sustituido por otro conjunto de cebadores en respuesta a la finalidad, sin embargo, la longitud final de ADN después de la digestión enzimática debe ser menos de 100 nt en términos de mantener la calidad del espectro de masas requerida para deconvolución éxito. Además, la presión de nitrógeno en el C-trampa debe reducirse cuando el oligonucleótido diana es más largo que aproximadamente 75 nt y la calidad del espectro de masas es insuficiente para deconvolución éxito. Estas limitaciones se pueden controlar a través de la evolución reciente de software del instrumento, de lo contrario la válvula para el C-trampa en el interior del chasis debe ser sintonizado como se describe en la sección de protocolo. En cuanto a la preparación de la muestra, el uso de reactivos libres de detergentes es crítica para la LC subsiguiente en cuanto a la forma de pico adecuada, suficiente intensidad máxima y tiempo de retención estable 31.
A columna PS-DVB monolito capilar 21,24-33, una columna de sílice en partículas de fase inversa C 18 23,40,41 y una cromatografía de interacción hidrófoba (HILIC) columna 42 se han utilizado para la LC / análisis ESI-MS de oligonucleótidos. Entre ellos, la columna de la monolito capilar es superior a los otros en términos de capacidad de separación, sin embargo, la columna de la monolito capilar utilizado en estudios anteriores se hizo en la casa y funcionar a una velocidad de flujo baja (2 l / min), que requiere instrumentación dedicado a LC micro. Para facilitar la operación fácil, se evaluaron columnas disponibles en el mercado para la separación de oligonucleótidos largos a velocidades de flujo más altas (100 a 400 l / min). Tres pares de dúplex de ADN (26, 37 y 53 pb) se separaron usando las columnas mencionadas anteriormente, sin embargo, los tiempos de ciclo de la columna de sílice en partículas -bonded C 18 y la columna de la PS-DVB monolito eran 65 y 70 min, respectivamente , mientras que la del C 18 monolito columna de sílice -bonded fue de 8 minutos (Figura 1). Tomando en consideración el análisis rápido, la columna monolito de sílice C 18 -bonded fue elegido para nuestros propósitos a pesar de la capacidad de separación limitada; sin embargo, las dos columnas restantes se pueden emplear cuando se requiere una mejor separación. Teóricamente, en el caso de una columna de monolito, no hay volumen intersticial y la fase móvil se vería obligada a fluir a través de los poros de la fase sólida mientras se mantiene una longitud de camino constante, lo que permite una separación eficiente 27. Tales procesos se manifiestan, en particular en el análisis de biomacromoléculas tal como un oligonucleótido, ya que la difusión lenta de las moléculas grandes. Una de las ventajas prácticas de una columna de monolito es que la presión de retorno es más baja que la de una columna de sílice de partículas 27. A pesar de la alta velocidad de flujo (400 l / min) y baja temperatura de la columna (20 ° C), la contrapresión máxima de lasistema fue de 12,5 MPa 37. Esta es la primera demostración de la ventaja de una columna de sílice monolito -bonded C 18 para el análisis rápido de un largo oligonucleótido. Debido a la alta velocidad de flujo, no se requiere un instrumento dedicado a las micro LC y una alineación precisa en la interfaz. En lugar de ello, se requiere una sonda ESI calienta para disociar el dúplex de ADN y ayudar a la ionización de ADN como se describe más adelante.
cromatografía de par iónico se utiliza comúnmente para la separación compatible-MS de oligonucleótidos. Sin embargo, un reactivo de par iónico interfiere generalmente con el proceso de ESI y disminuye la sensibilidad de ESI-MS. Por lo tanto, HFIP se utiliza con frecuencia para la fase móvil para mejorar la sensibilidad del oligonucleótido. Sin embargo, HFIP (punto 59 ° C de ebullición) se vaporiza rápidamente en la interfase antes (punto de ebullición 65 ° C) de metanol y, por lo tanto, esta pérdida de disolvente aumenta el pH y promueve la disociación del reactivo de par iónico (es decir, TEA)del oligonucleótido. Debido a que el presente método emplea una sonda calentada ESI, que nebuliza el eluato con gas nitrógeno caliente a 350 ° C, este efecto puede ser hincapié sobre-. En vez de tampón de HFIP-TEA, Erb y Oberacher recomienda acetato cyclohexyldimethylammonium (CycHDMAA; pH 8,4) para el análisis de genotipos debido a una reducción en la formación de aductos con los iones metálicos traza 33. Los autores dedujeron que sí CycHDMAA suprimida la formación del aducto de metal. A pesar de la literatura, la formación significativa aducto no se ha observado en el presente método. Además, el beneficio notable del sistema de HFIP-TEA-metanol es que el área del pico obtenido con el sistema de HFIP-TEA-metanol fue de 17 veces mayor que la obtenida a partir del sistema de CycHDMAA-acetonitrilo en el análisis de un amplicón 86 pb de T. poecilonotus (datos no mostrados). Una desventaja del sistema de HFIP-TEA-metanol, sin embargo, es el coste aumentado en relación con el sistema de CycHDMAA-acetonitrilo.
Cálculo de la masa monoisotopic requiere la separación de los picos isotópicos de iones cargados se multiplican. Por lo tanto, la resolución es crítica para el presente análisis. Aunque el poder de resolución requerida depende de la longitud de pares de bases del analito, analizadores TOF convencionales, que tienen un poder de resolución de varias decenas de millares, pueden estar limitados para el análisis de oligonucleótidos cortos.
Las masas teóricas monoisotopic mostrados en la Tabla 4 se calcularon a partir de las composiciones de base correspondientes de los analitos. Alternativamente, Muddiman et al. Desarrollado una aplicación de software para calcular la composición de base de la masa exacta 43. Un programa similar se integró en el sistema automatizado ESI-MS 16-18. El uso de estos algoritmos puede mejorar la robustez de la presente método porque una masa monoisotopic medido no siempre se corresponde con una base única composición oala a la tolerancia de masa de 3 ppm resultante de la medición de error inevitable. Por desgracia, no pudimos obtener estos productos de software para el presente estudio.
La determinación especies basado en la masa monoisotopic presente puede ser adecuado no sólo para la diferenciación de pez globo, sino también para la detección de otros polimorfismos de ADN debido a que el presente método se basa en el análisis de la composición de base y no implica un procedimiento diseñado específicamente para el pez globo. En cuanto a la detección de polimorfismo de ADN, el sistema ESI-MS dedicado es totalmente automatizado y fácil de operar, que puede ser adecuado para uso en diagnóstico tales como detección de patógenos 15,17,18,20,30. Por el contrario, el presente método es factible con instrumentos comunes de investigación y aparatos y, por lo tanto, adecuados para usos de investigación. ESI-MS ya se ha aplicado a polimorfismo de ADN humano, como único 13,28,32 polimorfismo de nucleótido, la repetición corta en tándem 26, Y el ADN mitocondrial análsis 16,19. Micro RNA también se analizó mediante capilar LC / ESI-MS 44. Estas aplicaciones publicadas también se pueden realizar mediante el presente método. Además, este método puede ser adecuado para el control de la interacción entre los compuestos de oligonucleótidos y de bajo peso molecular, tales como la interacción de los antibióticos y el ARN ribosomal 45 debido a la separación a baja temperatura. En tales casos, oligonucleótidos y compuestos de bajo peso molecular debe ser detectado simultáneamente, lo que es una ventaja de usar LC / ESI-MS.
Hay una limitación que la instrumentación no es adecuado para la realización de análisis en paralelo como en las técnicas convencionales, tales como la secuenciación de Sanger y PCR en tiempo real. Además, el presente método sólo identifica la composición de bases y cualquier sustitución de bases dentro de la misma molécula no se pueden distinguir. Sin embargo, el análisis de ADN basado en MS descrito aquí todavía puede tener mérito en plazos de exactitud en comparación con las técnicas basadas en colorantes, tales como electroforesis en gel y PCR en tiempo real de unión a ADN.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
This work was supported by a Grant-in-Aid for Scientific Research by the Japanese Society for the Promotion of Science (15K08060).
Materiales
| Name | Company | Catalog Number | Comments |
| DNeasy Blood & Tissue Kit (50) | Qiagen | 69504 | DNA extraction kit |
| Proteinase K | Qiagen | 19131 | |
| Ethanol (99.5+ vol%) | Wako | 054-07225 | For dilution of wash buffers |
| TE buffer (pH 8.0) | Wako | 310-90023 | For dilution of DNA sample |
| PCR primer | Fasmac | NA | Purified with reverse-phase cartridge column by the supplier |
| Template DNA | Eurofins Genomics | NA | Purified by HPLC by the supplier |
| Ultrapurified water | NA | NA | Generated with a Milli-Q Direct water purification system (Merck Millipore), used for sample preparation |
| Detergent Free 5x Phusion HF Buffer | Thermo Fisher | F-520L | Use instead of the provided buffer of DNA polymerase |
| Pfu-X DNA polymerase | Jena Bioscience | PCR-207S | |
| dNTP mix (20 mM each) | Jena Bioscience | NA | Supplied with the DNA polymerase |
| 10× Universal buffer M | Takara Bio | NA | Containing 100 mM Tris-HCl (pH 7.5), 100 mM MgCl2, 10 mM dithiothreitol and 500 mM NaCl |
| Dra I | Thermo Fisher | FD0224 | Restriction enzyme |
| Msp I | Thermo Fisher | FD0544 | Restriction enzyme |
| 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP) | Fluka | 42060-50ML | Eluent additive for LC-MS grade. |
| Triethylamine (TEA) | Fluka | 65897-50ML | Eluent additive for LC-MS grade. |
| Trifluoroacetic acid | Sigma-Aldrich | T6508 | For preparation of calibrant. |
| Sodium hydroxide (1.0 M) | Fluka | 72082 | Dilute to 10 mM with ultrapurified water and use for titration of trifluoroacetic acid. |
| Acetonitrile | Fluka | 34967 | For preparation of calibrant, LC/MS grade. |
| Methanol | Kanto | 25185-76 | For mobile phase, LC/MS grade. |
| Water | Thermo Fisher | W6-1 | For mobile phase, LC/MS grade. |
| Microcentrifuge tube (0.5 ml) | Eppendorf | 0030123301 | PCR clean grade |
| Microcentrifuge tube (1.5 ml) | Eppendorf | 0030123328 | PCR clean grade |
| UVette | Eppendorf | 0030106300 | Disposable UV cuvette. |
| Gel Green | Biotium | 41004 | Fluorescent DNA stain |
| Cosmospin filter G (0.2 μm) | Nakalai Tesque | 06549-44 | Made of hydrophilic polytetrafluoroethylene (PTFE) membrane. Any centrifugal filter unit (pore size, 0.2–0.5 μm) made of hydrophilic PTFE or another low-binding membrane is applicable. |
| 300 μl PP screw vial | American Chromatography Supplies | V0309P-1232 | |
| Preassembled screw cap and septa | Finneran | 5395-09R | |
| Monobis C18 analytical column (2.0×50 mm, mesopore size = 30 nm) | Kyoto Monotech | 2050H30ODS | Outside Japan, available for purchase from GL Sciences via its distributors. (http://www.glsciences.com/distributors/) |
| Monobis C18 guard column | Kyoto Monotech | GCSET-ODS3210 | Holder included. |
| Cadenza CW-C18 (3.0×250 mm) | Imtakt | CW036 | C18-bonded particulate silica column |
| ProSwift RP-4H (1.0×250 mm) | Thermo Fisher | 066640 | Poly(styrene-divinylbenzene) monolith column |
| Themo Mixer C | Eppendorf | 5382 000.023 | For digestion of fish tissues. |
| Spectrophotometer | Shimadzu | UV-3150 | Quantification of DNA concentration. |
| Thermal cycler | Bio-Rad | T100 | |
| Portable refrigerator | Twinbird | D-CUBE | For aqueous mobile phase to avoid evaporation of HFIP. This can be replaced by an ice box. |
| Ultimate 3000 liquid chromatograph | Thermo Fisher | NA | |
| Q Exactive mass spectrometer | Thermo Fisher | NA | Fourier transform mass spectrometer equipped with the modified Kingdon trap analyzer. |
| Protein deconvolution 3.0 | Thermo Fisher | NA | Use version 3.0 or higher having an isotopic patter model of nucleotide. |
Referencias
- Oberacher, H. On the use of different mass spectrometric techniques for characterization of sequence variability in genomic DNA. Anal. Bioanal. Chem. 391, 135-149 (2008).
- Banoub, J. H., Miller-Banoub, J., Jahouh, F., Joly, N., Martin, P. Chapter 1, Overview of recent developments in the mass spectrometry of nucleic acid and constituents. Mass spectrometry of nucleosides and nucleic acids. Banoub, J. H., Limbach, P. A. 1, CRC Press. 1-90 (2010).
- Yergey, J., Heller, D., Hansen, G., Cotter, R. J., Fenselau, C. Isotopic distributions in mass spectra of large molecules. Anal. Chem. 55, 353-356 (1983).
- Zubarev, R. A., Demirev, P. A. Isotope depletion of large biomolecules: Implications for molecular mass measurements. J. Am. Soc. Mass Spectrom. 9, 149-156 (1998).
- Null, A. P., Muddiman, D. C. Determination of a correction to improve mass measurement accuracy of isotopically unresolved polymerase chain reaction amplicons by electrospray ionization Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 17, 1714-1722 (2003).
- Hu, Q., Noll, R. J., Li, H., Makarov, A., Hardman, M., Cooks, R. G. The Orbitrap: a new mass spectrometer. J Mass Spectrom. 40, 430-443 (2005).
- Senko, M. W., Beu, S. C., McLaffertycor, F. W. Determination of monoisotopic masses and ion populations for large biomolecules from resolved isotopic distributions. J. Am. Soc. Mass Spectrom. 6, 229-233 (1995).
- Horn, D. M., Zubarev, R. A., McLafferty, F. W. Automated reduction and interpretation of high resolution electrospray mass spectra of large molecules. J. Am. Soc. Mass Spectrom. 11, 320-332 (2000).
- Frahm, J. L., Mason, C. J., Muddiman, D. C. Utility of accurate monoisotopic mass measurements to confidently identify lambda exonuclease generated single-stranded amplicons containing 7-deaza analogs by electrospray ionization FT-ICR mass spectrometry. Int. J. Mass Spectrom. 234, 79-87 (2004).
- Frahm, J. L., Muddiman, D. C. Nucleic Acid analysis by fourier transform ion cyclotron resonance mass spectrometry at the beginning of the twenty-first century. Curr. Pharm. Des. 11, 2593-2613 (2005).
- Null, A. P., George, L. T., Muddiman, D. C. Evaluation of sample preparation techniques for mass measurements of PCR products using ESI-FT-ICR mass spectrometry. J. Am. Soc. Mass Spectrom. 13, 338-344 (2002).
- Null, A. P., Benson, L. M., Muddiman, D. C. Enzymatic strategies for the characterization of nucleic acids by electrospray ionization mass spectrometry. Rapid Commun. Mass Spectrom. 17, 2699-2706 (2003).
- Manduzio, H., Ezan, E., Fenaille, F. Evaluation of the LTQ-Orbitrap mass spectrometer for the analysis of polymerase chain reaction products. Rapid Commun. Mass Spectrom. 24, 3501-3509 (2010).
- Manduzio, H., Martelet, A., Ezan, E., Fenaille, F. Comparison of approaches for purifying and desalting polymerase chain reaction products prior to electrospray ionization mass spectrometry. Anal. Biochem. 398, 272-274 (2010).
- Hofstadler, S. A., et al. TIGER: the universal biosensor. Int. J. Mass Spectrom. 242, 23-41 (2005).
- Eduardoff, M., et al. Mass spectrometric base composition profiling: Implications for forensic mtDNA databasing. Forensic Sci. Int. Genet. 7, 587-592 (2013).
- Tang, Y. W., et al. Clinical accuracy of a PLEX-ID flu device for simultaneous detection and identification of influenza viruses A and B. J. Clin. Microbiol. 51, 40-45 (2013).
- Legoff, J., et al. Broad-range PCR-electrospray ionization mass spectrometry for detection and typing of adenovirus and other opportunistic viruses in stem cell transplant patients. J. Clin. Microbiol. 51, 4186-4192 (2013).
- Kiesler, K. M., Coble, M. D., Hall, T. A., Vallone, P. M. Comparison of base composition analysis and Sanger sequencing of mitochondrial DNA for four U.S. population groups. Forensic Sci. Int. Genet. 8, 226-232 (2014).
- Bacconi, A., et al. Improved sensitivity for molecular detection of bacterial and Candida infections in blood. J. Clin. Microbiol. 52, 3164-3174 (2014).
- Huber, C. G., Oberacher, H. Analysis of nucleic acids by on-line liquid chromatography-mass spectrometry. Mass Spectrom. Rev. 20, 310-343 (2001).
- Pourshahian, S., McCarthy, S. M. Chapter 4, Analysis of oligonucleotides by liquid chromatography and mass spectrometry. Handbook of Analysis of Oligonucleotides and Related Products. Bonilla, J. V., Srivatsa, G. S. , CRC Press, Chapter. 137-166 (2011).
- Apffel, A., Chakel, J. A., Fischer, S., Lichtenwalter, K., Hancock, W. S. Analysis of Oligonucleotides by HPLC-Electrospray Ionization Mass Spectrometry. Anal. Chem. 69, 1320-1325 (1997).
- Premstaller, A., Oberacher, H., Huber, C. G. High-performance liquid chromatography-electrospray ionization mass spectrometry of single- and double-stranded nucleic acids using monolithic capillary columns. Anal. Chem. 72, 4386-4393 (2000).
- Oberacher, H., Oefner, P. J., Parson, W., Huber, C. G. On-Line Liquid Chromatography Mass Spectrometry: A Useful Tool for the Detection of DNA Sequence Variation. Angew. Chem. Int. Ed. Engl. 40, 3828-3830 (2001).
- Oberacher, H., Parson, W., Muhlmann, R., Huber, C. G. Analysis of polymerase chain reaction products by on-line liquid chromatography-mass spectrometry for genotyping of polymorphic short tandem repeat loci. Anal. Chem. 73, 5109-5115 (2001).
- Oberacher, H., Huber, C. G. Capillary monoliths for the analysis of nucleic acids by high-performance liquid chromatography-electrospray ionization mass spectrometry. Trends Anal. Chem. 21, 166-174 (2002).
- Oberacher, H., et al. Re-sequencing of multiple single nucleotide polymorphisms by liquid chromatography-electrospray ionization mass spectrometry. Nucleic Acids Res. 30, e67(2002).
- Oberacher, H., Parson, W., Holzl, G., Oefner, P. J., Huber, C. G. Optimized suppression of adducts in polymerase chain reaction products for semi-quantitative SNP genotyping by liquid chromatography-mass spectrometry. J. Am. Soc. Mass Spectrom. 15, 1897-1906 (2004).
- Mayr, B. M., et al. Identification of bacteria by polymerase chain reaction followed by liquid chromatography-mass spectrometry. Anal. Chem. 77, 4563-4570 (2005).
- Oberacher, H., Niederstatter, H., Casetta, B., Parson, W. Some guidelines for the analysis of genomic DNA by PCR-LC-ESI-MS. J. Am. Soc. Mass Spectrom. 17, 124-129 (2006).
- Beer, B., et al. CYP2D6 genotyping by liquid chromatography-electrospray ionization mass spectrometry. Anal. Bioanal. Chem. 400, 2361-2370 (2011).
- Erb, R., Oberacher, H. Comparison of mobile-phase systems commonly applied in liquid chromatography-mass spectrometry of nucleic acids. Electrophoresis. 35, 1226-1235 (2014).
- Cohen, N. J., et al. Public health response to puffer fish (Tetrodotoxin) poisoning from mislabeled product. J. Food Prot. 72, 810-817 (2009).
- Cole, J. B., et al. Tetrodotoxin poisoning outbreak from imported dried puffer fish--Minneapolis, Minnesota 2014. MMWR Morb. Mortal. Wkly. Rep. 63, 1222-1225 (2015).
- Ohno, Y., et al. The influence of tetrodotoxin on the toxic effects of aconitine in vivo. Tohoku J. Exp. Med. 167, 155-158 (1992).
- Miyaguchi, H., Yamamuro, T., Ohta, H., Nakahara, H., Suzuki, S. Genotyping of Toxic Pufferfish Based on Specific PCR-RFLP Products As Determined by Liquid Chromatography/Quadrupole-Orbitrap Hybrid Mass Spectrometry. J. Agric. Food Chem. 63, 9363-9371 (2015).
- Sambrook, J. F., Russell, D. W. Neutral polyacrylamide gel electrophoresis. in: Molecular Cloning: A Laboratory Manual. 1, 3rd, Cold Spring Harbor Laboratory Press. 40-46 (2001).
- Moini, M., Jones, B. L., Rogers, R. M., Jiang, L. Sodium trifluoroacetate as a tune/calibration compound for positive- and negative-ion electrospray ionization mass spectrometry in the mass range of 100-4000 Da. J. Am. Soc. Mass Spectrom. 9, 977-980 (1998).
- Fountain, K. J., Gilar, M., Gebler, J. C. Analysis of native and chemically modified oligonucleotides by tandem ion-pair reversed-phase high-performance liquid chromatography/electrospray ionization mass spectrometry. Rapid Commun. Mass Spectrom. 17, 646-653 (2003).
- Chen, B., Bartlett, M. G. Evaluation of mobile phase composition for enhancing sensitivity of targeted quantification of oligonucleotides using ultra-high performance liquid chromatography and mass spectrometry: application to phosphorothioate deoxyribonucleic acid. J. Chromatogr. A. 1288, 73-81 (2013).
- Gong, L., McCullagh, J. S. Analysis of oligonucleotides by hydrophilic interaction liquid chromatography coupled to negative ion electrospray ionization mass spectrometry. J. Chromatogr. A. 1218, 5480-5486 (2011).
- Muddiman, D. C., Anderson, G. A., Hofstadler, S. A., Smith, R. D. Length and base composition of PCR-amplified nucleic acids using mass measurements from electrospray ionization mass spectrometry. Anal. Chem. 69, 1543-1549 (1997).
- Kullolli, M., Knorf, E., Arampatzidou, M., Tewari, M., Pitteri, S. J. Intact MicroRNA analysis using high resolution mass spectrometry. J. Am. Soc. Mass Spectrom. 25, 80-87 (2013).
- Cummins, L. L., Chen, S., Blyn, L. B., Sannes-Lowery, K. A., Drader, J. J., Griffey, R. H., Hofstadler, S. A. Multitarget affinity/specificity screening of natural products: finding and characterizing high-affinity ligands from complex mixtures by using high-performance mass spectrometry. J. Nat. Prod. 66, 1186-1190 (2003).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados