Method Article
Purificación de Complejos nativo para el estudio estructural se utiliza un método Tag Tandem Affinity
En este artículo
Resumen
The Tandem Affinity Purification (TAP) method has been used extensively to isolate native complexes from cellular extract, primarily eukaryotic, for proteomics. Here, we present a TAP method protocol optimized for purification of native complexes for structural studies.
Resumen
enfoques de purificación por afinidad han tenido éxito en el aislamiento de los complejos nativos para la caracterización proteómica. La heterogeneidad estructural y un grado de heterogeneidad de composición de un complejo por lo general no impiden el progreso en la realización de dichos estudios. En contraste, un complejo destinado a la caracterización estructural debe ser purificado en un estado que es a la vez de composición y estructuralmente homogéneo, así como a una concentración mayor que la requerida para la proteómica. Recientemente, se han producido avances significativos en la aplicación de la microscopía de electrones para la determinación de la estructura de grandes complejos macromoleculares. Esto ha aumentado el interés en los enfoques para purificar complejos nativas de calidad y cantidad suficiente para la determinación estructural mediante microscopía electrónica. El método de purificación de afinidad en tándem (TAP) ha sido optimizado para extraer y purificar un joven de 18 subunidad, ~ 0,8 ensamblaje ribonucleoproteína MDA a partir de levadura en ciernes (Saccharomyces cerevisiae)
Introducción
Muchos de los principales procesos celulares se llevan a cabo por gran proteína y proteína-ARN complejos 1. Un cuello de botella significativo para la realización de estudios biofísicos y estructurales de tales complejos de ellos es la obtención de una calidad adecuada (es decir, homogeneidad) y a una concentración apropiada. El aislamiento de un complejo de una fuente nativa tiene muchas ventajas, incluyendo la retención de las modificaciones post-transcripcionales y / o traduccionales relevantes de subunidades y asegurar complejo montaje adecuado. Sin embargo, grandes complejos celulares están a menudo presentes en una célula en un bajo número de copias y la purificación deben ser altamente eficiente y producir en condiciones fisiológicas cerca para asegurar que se mantiene la integridad del complejo. La purificación de un complejo de una fuente eucariota es particularmente difícil y puede ser económicamente prohibitivo. Por lo tanto, es muy deseada estrategias o métodos que sean eficientes y producen un complejo homogéneo.
Una estrategia que ha sidoéxito en la purificación de los complejos nativos de células eucariotas para su caracterización inicial es el tándem de purificación de afinidad (TAP) Método de 2,3. El método TAP fue ideado inicialmente para purificar una proteína nativa de la levadura en ciernes (S. cerevisiae) en el complejo con la interacción de los factores (s) 2. El método TAP utiliza dos etiquetas, cada etiqueta fusionado en tándem para la misma secuencia del gen codificante de la proteína. Las etiquetas fueron seleccionados a fin de equilibrar la necesidad de apretado y selectiva la unión a una resina de afinidad con un deseo de mantener cerca de las condiciones de solución fisiológica. Este equilibrio sirve para preservar la interacción estable (s) de la proteína de la etiqueta con el factor de interacción (s) para la caracterización posterior a la purificación. La etiqueta TAP genómicamente incorporada se coloca en el extremo (C-terminal) de un gen que codifica la proteína y consistía en una secuencia que codifica para un calmodulina de unión al péptido (CBP) seguido de proteína A - una adición de un poco más de 20 kDa a la etiquetado proteína. CBP es corta, 26 aminoácidos, y reconocidos por el ~ 17 kDa proteína calmodulina (CaM) en presencia de calcio con una K D del orden de 10 -9 M 4. La etiqueta de la proteína A es más grande, que consiste en dos repeticiones de 58 residuos con un enlazador corto entre las repeticiones. Cada repetición de 58 aminoácidos es reconocido por la inmunoglobulina G (IgG) con una K D ~ 10 -8 M 5. Entre estas dos etiquetas se incorporó un sitio de reconocimiento para la proteasa TEV, una endopeptidasa de la Virus del grabado del tabaco 6,7. Como se ilustra en la Figura 1, en la primera etapa de afinidad del método de la TAP proteína marcada se une a una resina de IgG a través de la proteína A. La proteína marcada se eluye por escisión en la columna tras la adición de la proteasa TEV, específicamente en el sitio de escisión entre las dos etiquetas. Este es un paso necesario como la interacción de IgG y proteína A es muy fuerte y sólo puede ser perturbado adecuadamente bajo condiciones de solución de desnaturalización. A falta de un PrOtein Una etiqueta, la proteína se une a la resina de CaM en presencia de calcio y se eluyó de esta resina con adición de EGTA el quelante de iones metálicos (ácido etilen glicol) (Figura 1).
Poco después de la introducción del método de TAP, que se utilizó en un estudio a gran escala para generar un "mapa" de las interacciones complejas en S. 8 cerevisiae. Es importante destacar que, como consecuencia de este esfuerzo una biblioteca entera del marco de levadura-TAP Tagged de lectura abierto (ORF), así como TAP individuo etiquetado ORFs 9 están disponibles de una fuente comercial. Por lo tanto, se puede obtener cualquier cepa de levadura con una proteína etiquetada para cualquier complejo de levadura. El método TAP también estimuló modificaciones o variaciones de la etiqueta TAP, incluyendo: su uso para la purificación de complejos de otros eucariotas, así como células bacterianas 10,11; el diseño de una "etiqueta dividir", en el que la proteína A y CBP se colocan en diferentes proteínas de 12; y la tags cambiado, así como para dar cabida a la necesidad del investigador, tales como la sensibilidad del complejo de Ca 2+ o EGTA 13.
Los recientes avances, tanto en instrumentación y metodología han dado lugar a avances significativos en la aplicación de la microscopía electrónica (EM) para la determinación de la estructura, que han conducido a alta, cerca de imágenes de resolución atómica de complejos macromoleculares 14. La resolución obtenible de un complejo por EM, sin embargo, sigue dependiendo de la calidad del complejo en estudio. En este estudio se ha utilizado la estrategia de etiquetas de TAP para purificar a partir de S. cerevisiae U1 snRNP, un joven de 18 subunidad (~ 0,8 MDA) bajo número de copias ribonucleoprotein complejo que forma parte de la spliceosome 15,16. Una serie de medidas se han adoptado para purificar este complejo tal que es homogéneo y de una concentración adecuada. Los problemas potenciales encontrados en diversas etapas de la purificación se describen y estrategias adoptadas para superar challEnges resaltados. Mediante la evaluación y optimización de las operaciones en la purificación cuidado, el U1 snRNP purificada es de una calidad y en cantidad adecuada para la tinción negativa y crio (crio-EM) los estudios de microscopía de electrones. Un protocolo TAP método optimizado para la purificación de complejos nativas para estudios estructurales se describe en este documento.
Protocolo
Nota: El siguiente protocolo se diseñó para la purificación de un complejo de 4 l de cultivo de células, aproximadamente 40 g de peso húmedo de células. Una vez preparada, todos los tampones deben almacenarse a 4 ° C y utilizarse dentro de un mes de su preparación. Los inhibidores de la proteasa y de agentes reductores sólo se incorporan al tampones justo antes de su uso.
1. Preparación del extracto de células enteras para Tandem Purificación por Afinidad
- El crecimiento de S. Las células cerevisiae
- Pasar la derivación deseada etiquetada S. cerevisiae cepa de almacenamiento (-80 ° C) en una solución de dextrosa de levadura peptona (YPD) plato. Incubar durante 3 - 5 días (30 ° C), hasta que las células de levadura aparecen en blanco y esponjoso.
- Inocular una racha de aproximadamente 2 mm 2 de las células frescas de la placa en 10 ml de medio YPD en un tubo de centrífuga de polipropileno cónico de 50 ml. Agitar el tubo a 180 revoluciones por min (rpm) durante la noche (30m ° C).
- Inocular 2 ml de la máscultura noche por litro de medio YPD en un Erlenmeyer de 2 L o frasco cónico. Se agita a 180 rpm (30 ° C). Monitor de crecimiento celular a una densidad óptica de 600 nm (OD 600) hasta la fase logarítmica tardía, OD 600 de 1,8 a 2, típicamente 20 a 24 hr.
- La recolección S. Las células cerevisiae
- Cosecha células por centrifugación a 5.400 xg durante 15 min (4 ° C).
- decantar rápidamente el sobrenadante y mantener las células a 4 ° C o en hielo.
- Suspender cada litro de sedimento celular con 2 ml refrigerados tampón de lisis (10 mM Tris-HCl, pH 8,0; NaCl 300 mM; KCl 10 mM; 0,2 mM de EDTA, pH 8,0; imidazol 5 mM, pH 8,0; 10% de glicerol; 0,1% v NP-40 / v). Suspender las células agitando el recipiente y / o repetición de pipeteado utilizando una pipeta serológica de 10 ml.
Nota: La importancia de NP-40 en lo que respecta a la calidad del complejo y el análisis posterior a la purificación se discuten a continuación. - Transferir la suspensión de células en un vacío de 50 ml cónico de polipropileno tubo de centrífuga kEPT en el hielo. Lavar cada botella de centrífuga con un adicional de 2 ml de tampón de lisis enfriado y añadir a la suspensión de células en el tubo de 50 ml.
- Determinar el peso húmedo de sedimento celular pesando el tubo y restando el peso del tubo vacío, así como el añadido tampón de lisis.
Nota: Un litro de cultivo celular que tiene un OD 600 de 1,8 es de aproximadamente 10 g. - Crear un baño de nitrógeno líquido en un tubo de centrífuga cónico de polipropileno de 50 ml. Dibuje células suspendidas en una jeringa de 5 ml. Conectar una aguja 16 G a la jeringa. Congelación de la suspensión de células haciéndolo pasar a través de la jeringa / aguja para generar células "gotas". Velocidad de congelación debe ser de aproximadamente 1 min / ml.
- Almacenar las gotitas de células congeladas (-80 ° C) o proceder a la lisis.
- Las células de lisis y clarificación de lisado
- Lyse las células de levadura utilizando un molinillo de café. lisar células de ocho a nueve veces con 25 segundos de molienda ráfagas, mientras se agita la cmolinillo de offee. Antes de su uso, pre-enfriar el molinillo de café con nitrógeno líquido. Cada dos rondas de molienda, añaden una capa superficial de nitrógeno líquido para el molino y deje que se evapore.
- Remover con una espátula de nitrógeno líquido refrigerado, según sea necesario para mantener las células se agrupen. Tenga precaución para evitar la descongelación de las células, lo que puede dar lugar a la formación de grumos de células. Las células deben aparecer como un polvo blanco fino después de la lisis. Un sedimento celular máximo de 4 L de cultivo de levadura (~ 40 g) se puede moler a la vez.
Nota: Observaciones de método de lisis se discuten a continuación. - Evaluar el grado de lisis celular utilizando un hemocitómetro o método de recuento celular alternativo. Para proporcionar un análisis adecuado, tome las siguientes muestras: (1) antes de la lisis; (2) después de cuatro rondas de lisis; y (3) después de la lisis. Para comprobar estas muestras, tomar una pequeña cantidad de células con una espátula de nitrógeno líquido refrigerado y colocar en un tubo de 1,5 ml.
- Una vez que se descongelan las células, de pipeta 5 l de célulasy mezclar con 495 l de agua. Si el 40 - 60% se observó lisis de las células, continúe con el siguiente paso en el protocolo.
Nota: Observaciones de efecto de largos períodos de lisis se discuten a continuación. - , almacenar células lisadas Una vez que se observa lisis adecuada (-80 ° C) o proceder al siguiente paso.
- Preparar dos 50 ml tubos de centrífuga de polipropileno cónico de 15 ml cada uno con tampón de lisis, incluyendo 1 mM de fluoruro de fenilmetanosulfonilo (PMSF), ditiotreitol 1 mM (DTT), y un cóctel disponible en el mercado de los inhibidores de la proteasa.
Nota: Si se observa la proteólisis significativa, considere el uso de una cepa deficiente en proteasa. - Use una espátula de nitrógeno líquido refrigerado para sacar el polvo de células lisadas congelada en los preparados tubos de 50 ml. Añadir el polvo de células / sólido de forma incremental al tampón de lisis, que contiene tanto un reductor y el inhibidor (s) proteasa.
- Rotar los tubos de 50 ml suavemente a TA con el fin de descongelar / disuelva las células, sino también para evitar la formación de burbujas. UNEs el polvo celular / sólido se disuelve, añadir celular adicional sólido / polvo.
- Llene cada uno de los dos tubos de 50 ml hasta que cada una tiene aproximadamente 20 g de células o todas las células añadidas. Continuar descongelación / disolución hasta que no haya grumos de células congeladas se observan en los tubos de 50 ml. Esto debe tomar aproximadamente 50 minutos.
- Centrifugar el lisado de células en suspensión durante 20 minutos a 25.000 xg (4 ° C). células lisadas bien pueden exhibir una pequeña cantidad de precipitado negro.
- La transferencia de los 50 ml de sobrenadante a tubos de policarbonato ultra-centrífuga (26,3 ml tubos utilizados) y añadir 10 l de PMSF 200 mM a cada tubo completo.
- Centrifugar durante 1 hora a 100.000 xg (4 ° C). Cuatro capas deben ser visibles, de abajo hacia arriba: (1) un sedimento duro clara, que contiene complejos ribosómicos; (2) una pastilla suave rica en lípidos; (3) una gran capa de color amarillo teñido transparente que contiene la mayor parte de las proteínas y los complejos solubles de la célula; y (4) una 'escamas' capa superior que consiste en lípidos.
- Realizar todos los subsequent pasos en una habitación fría (4 ° C). Eliminar la mayor cantidad de la capa de lípidos top 'escamas' como sea posible usando una pipeta de 1 ml y desechar. Utilice una pipeta serológica de 10 ml a recuperar la mayor parte de la capa amarilla, clara.
- Recuperar los últimos ml de esta capa usando una pipeta de 1 ml para no molestar a las dos capas inferiores. De 40 g sedimento celular húmedo, aproximadamente 48 ml de la capa del medio es típicamente recuperados y 8 ml de capa lipídica eliminan / desechado.
Nota: La purificación en columna de flujo se ve obstaculizado en gran medida por la presencia de lípidos, que también pueden reducir la calidad del complejo, se discute a continuación.
2. Columna etapa de purificación 1: Cromatografía de IgG
- Resina de equilibrado y Encuadernación
- Preparar un 300 l IgG Sepharose suspensión de 40 g de sedimento celular. Cortar el extremo de una punta de pipeta 1 ml de facilitar pipeteando la suspensión. Wash / equilibrar la resina IgG tres veces, cada vez con 5 ml IgGD150 Buffer (10 mM Tris-HCl, pH 8,0; NaCl 150 mM; KCl 150 mM; 1 mM MgCl 2; imidazol 5 mM, pH 8; 0,1% v / v de NP-40; DTT 1 mM; una tableta de inhibidor de la proteasa por 50 volumen ml).
- Centrifugado para sedimentar a 160 xg (4 ° C) entre los lavados y eliminar el sobrenadante.
- Girar la resina de IgG con el sobrenadante de fase media y dos tabletas de mini inhibidores de la proteasa por 40 g de células, utilizando un rotador apropiado, por 2 hr (4 ° C). Esta es la solución de proceso por lotes IgG.
- Preparar dos 10 ml columnas poli-de preparación para el uso al cortar el extremo de la columna para producir un corte plano. Para acelerar el embalaje y el lavado de la columna por flujo de gravedad, cargar un máximo de 200 l de resina de IgG acondicionamiento o de ~ 25 ml de la solución de lote IgG por 10 ml de la columna.
- Se vierte la solución de lotes de IgG en la columna y dejar sedimentar por gravedad. Si la sedimentación tarda más de 30 minutos, no era más probable la contaminación de lípidos cuando se recupera la fase media de la centrífuga tUBE.
- Se lava la columna rellena con cuatro sucesivos volúmenes de 10 ml de Tampón IgGD150.
- Preparar un 300 l IgG Sepharose suspensión de 40 g de sedimento celular. Cortar el extremo de una punta de pipeta 1 ml de facilitar pipeteando la suspensión. Wash / equilibrar la resina IgG tres veces, cada vez con 5 ml IgGD150 Buffer (10 mM Tris-HCl, pH 8,0; NaCl 150 mM; KCl 150 mM; 1 mM MgCl 2; imidazol 5 mM, pH 8; 0,1% v / v de NP-40; DTT 1 mM; una tableta de inhibidor de la proteasa por 50 volumen ml).
- La escisión en la columna TEV
- Sellar la parte inferior de la columna y se añade 1 ml de tampón de IgGD150 y 100 l de proteasa TEV (0,6 mg / ml de stock, la variante mutante S219V de TEV de producción propia). Sellar la parte superior de la columna y se mezcla durante 20 min (18 ° C) usando un mezclador térmico, agitando a 750 rpm. Resuspender resina y mezclar de nuevo durante 20 min, repita una vez más para un total de 1 h de incubación.
Nota: Es muy importante para mantener el tiempo de incubación a un mínimo. - Volver la columna a 4 ° C y se eluye la proteína / complejo por la gravedad. Eluir el volumen muerto con un 200 l adicionales de tampón de IgGD150.
- Sellar la parte inferior de la columna y se añade 1 ml de tampón de IgGD150 y 100 l de proteasa TEV (0,6 mg / ml de stock, la variante mutante S219V de TEV de producción propia). Sellar la parte superior de la columna y se mezcla durante 20 min (18 ° C) usando un mezclador térmico, agitando a 750 rpm. Resuspender resina y mezclar de nuevo durante 20 min, repita una vez más para un total de 1 h de incubación.
3. La purificación en columna Paso 2: cromatografía de afinidad calmodulina
- Resina de equilibrado y Encuadernación
- Preparar 200 l de resina de afinidad de calmodulina por 40 g de célulasbolita. La resina se lava tres veces, cada vez con 5 ml de calmodulina tampón de unión (10 mM Tris-HCl, pH 8,0; NaCl 150 mM; KCl 10 mM; 1 mM MgCl 2; imidazol 5 mM, pH 8; 2 mM CaCl 2; 0.1 % v / v de NP-40; 10 mM β-mercaptoetanol), centrifugando a 160 xg (4 ° C) para sedimentar para cada lavado.
- Para cada 1 ml de eluido IgG, añadir 3 volúmenes de tampón de unión de calmodulina. Para mantener constante el nivel de calcio, añadir CaCl2 y β-mercaptoetanol para dar cuenta de la falta de estos componentes en la elución de IgG. Las concentraciones finales deben ser 2 mM CaCl 2 y 10 mM β-mercaptoetanol.
- Añadir la muestra a la resina de calmodulina y se unen para 1 hr (4 ° C) utilizando un rotador tubo.
- Embalaje y que eluye de la resina de calmodulina
- Preparar una columna de poli-prep 2 ml por 100 l de suspensión calmodulina cortando el extremo de la columna para crear una abertura plana. No cargue más de 100 l calmodulina suspensión por columna para reducir al mínimo la cantidad de resina de la muestra entra en contacto con durante la elución.
- Paquete de la columna por gravedad y se lava la resina en la columna tres veces con 5 ml de calmodulina tampón de unión.
- Eluir la proteína con la adición de 200 l calmodulina tampón de elución (10 mM Tris-HCl, pH 8,0; NaCl 150 mM; KCl 10 mM; 1 mM MgCl 2; imidazol 5 mM, pH 8,0; EGTA 4 mM, pH 8,0; 0,08% v / v NP-40; 10 mM β-mercaptoetanol). Repita seis veces la adición de 200 l de tampón de elución calmodulina.
- Para eluciones 1 a 3, aplicar el volumen a la columna y recoger el eluido inmediatamente. Para la elución 4, sellar la parte inferior de la columna y se incuba con tampón de elución para 2,5 min y luego quitar el sello y permitir que para eluir por gravedad. Para la elución 5, incubar durante 5 min y luego quitar el sello y eluir. Para la elución 6, incubar durante 10 min y luego quitar el sello y eluir.
Nota: La integridad del complejo puede variar entre eluciones / fracciones (ver sección de resultados representativos) <./ Li> - Dializar eluciones 2-6 individualmente durante la noche utilizando un método de diálisis micro apropiado. Se dializa contra las fracciones diálisis Buffer (10 mM Tris-HCl, pH 8,0; NaCl 150 mM; KCl 10 mM; 1 mM MgCl 2; imidazol 5 mM, pH 8; 10 mM β-mercaptoetanol).
Nota: NP-40 no pasa sensiblemente, en todo caso, a través de la membrana de diálisis.
4. Los análisis post-purificación para evaluar la calidad del Complejo
- Nativo del gel y tinción con plata
- Preparar un 4 - gel nativo prefabricado 16% para el análisis del complejo según las instrucciones del fabricante. Utilice un patrón de proteína de peso molecular adecuado y la carga de 15 l de cada uno de los calmodulina de elución / fracciones dializadas en el gel.
- La electroforesis el gel a 150 V durante aproximadamente 3 hr (4 ° C).
- La tinción con plata del gel para evaluar la calidad de cada uno de los seis eluciones. Como alternativa, el uso comercial igualmente sensiblesmancha fluorescente (s). En esta etapa, es más probable por debajo del nivel de detección de tinción con azul de Coomassie de la concentración del complejo.
- Métodos adicionales análisis en gel
- Para detectar la proteína mediante inmunotransferencia de tipo Western, resolver el complejo mediante SDS o PAGE nativa y, a continuación sondear el gel para el epítopo de unión al péptido La calmodulina (CBP) utilizando un anticuerpo tag TAP según las instrucciones del fabricante.
- El uso de una mancha fluorescente específico (s), confirmar la presencia y la integridad de la proteína y / o ácido nucleico en un complejo TAP purificado. Tenga cuidado de que no se detecta ningún solapamiento espectral entre estas manchas.
- La visualización de la mancha negativa Microscopía Electrónica
- Utilizar rejillas continuas de carbono-resplandor descarga a -15 mA durante 30 segundos, a los 30 minutos de aplicación compleja.
- Aplicar 3 l de complejo TAP purificado (típicamente a una concentración de 1 nM) a la red. Se incuba durante un minuto. f blotesde el lado de la parrilla para eliminar el exceso de tampón.
- Lavar dos veces con 20 gotas l de agua desionizada. blot lateral entre lavados.
- Aplicar cuadrícula a un 50 l gota de tinción negativa y se incuba durante un minuto. Se recomienda la tinción con acetato de uranilo (2%) o formato de uranilo (0,75%).
Precaución: sales de uranilo y soluciones son débilmente radiactivos y contienen metales pesados. Estos deben ser manejados con cuidado y todo el material que entra en contacto con el mismo deberán eliminarse siguiendo las directrices de residuos recomendados estas soluciones institucionales. - Aplicar rejilla a un nuevo máximo de 50 l gota de tinción negativa, sin goteo. Se incuba durante un minuto, y luego por transferencia lateral, y finalmente secarse al aire y visualizar.
- Cryo Microscopía Electrónica (crio-ME) Visualización
- Realizar la crio-EM complejo con TAP purificado, aunque la optimización puede ser necesario, de acuerdo con el protocolo del fabricante.
Nota: La presencia de detergente en altas concentraciones may obstaculizan los avances y se discute a continuación.
- Realizar la crio-EM complejo con TAP purificado, aunque la optimización puede ser necesario, de acuerdo con el protocolo del fabricante.
5. Complejo de almacenamiento
- Preparación de Complejo de almacenamiento
- Se concentra el complejo, si es necesario, usando un filtro centrífugo que tiene un peso molecular de corte apropiado para el complejo. Girar a 14.000 xg durante 1 min incrementos hasta que se alcanza la concentración deseada.
Nota: la concentración de NP-40 se incrementará en la concentración durante la concentración, ya que no pasa a través de la membrana de filtro ni de diálisis. - Flash congelar el complejo en 30 ml de alícuotas en nitrógeno líquido o hielo seco se enfrió baño de etanol y luego se almacena a -80 ° C.
- Se concentra el complejo, si es necesario, usando un filtro centrífugo que tiene un peso molecular de corte apropiado para el complejo. Girar a 14.000 xg durante 1 min incrementos hasta que se alcanza la concentración deseada.
- La eliminación opcional post-purificación de detergente
Nota: La presencia de un detergente tal como NP-40 y a una concentración por encima de su concentración micelar crítica pueda obstaculizar o inhibir el progreso para algunas aplicaciones. Las consecuencias de su retirada de la protocol así como la sustitución con otros aditivos o co-disolventes se discuten a continuación. Si no se desea NP-40, una opción es utilizar una aplicación comercial para la eliminación de, por ejemplo, Bio-perlas.
- Pre-Equilibrar disponibles en el mercado de detergentes de absorción de los granos en la diálisis de amortiguamiento. Mezclar cinco perlas por 30 l de complejo (por lo general en una concentración de 10 nM).
- Incubar cuentas equilibradas desde el paso 5.2.1 con el complejo purificado TAP durante 15 minutos en hielo. Utilice complejo dentro de unas pocas horas después de la eliminación de detergente.
Resultados
Un método TAP modificado se utiliza para purificar a partir de S. cerevisiae, el U1 snRNP, un complejo de ribonucleoproteína 18 de la subunidad. Una purificación TAP inicial del complejo después de la 2,3 protocolo publicado produjo un complejo que apareció heterogénea, migrando como tres bandas en un plata manchadas gel de poliacrilamida nativo (Figura 2A). Múltiples rondas de optimización del método de TAP, produjeron un complejo que migró como principalmente una sola banda en un gel nativo indicativo de un conjunto más homogéneo (Figura 2A). El uso de colorantes fluorescentes para teñir para proteínas y ácidos nucleicos, se estableció que el complejo purificado tenía ambos biopolímeros presente (Figura 2B).
El complejo purificado también se analizó mediante SDS PAGE y transferencia Western utilizando un anticuerpo tag TAP (Figura 3A). En consonancia con las Native resultado gel, el complejo purificado de acuerdo con el protocolo publicado exhibió la proteólisis de la proteína marcada TAP (Snu71). La cantidad de la proteolisis, sin embargo, se redujo significativamente con modificaciones del método TAP. Casi la totalidad de las diecisiete proteínas en el complejo U1 snRNP se resolvieron mediante SDS PAGE y identificado positivamente por espectrometría de masas MALDI (Figura 3B). Así como nativo y SDS PAGE, la calidad del complejo se evaluó en diferentes etapas de la purificación por microscopía electrónica con tinción negativa (Figura 4). Las partículas monodispersas observados en la Figura 4A y B, pero ausente de 4C son ejemplos de buena muestra para el estudio estructural. Este método de análisis proporciona una visión crítica en cuanto al impacto de los cambios en el método de TAP.
El método TAP pide la inclusión del detergente no iónico NP-40 y a una concentration encima de la de su concentración micelar crítica (CMC). La robustez de los métodos de evaluación de la calidad del complejo descrito en el protocolo son bien demostró mediante un experimento en el que el ion híbrido CHAPS surfactante, glicerol o sin co-disolvente se sustituyeron por NP-40 en todos los tampones (Figura 5). Por PAGE nativa y tinción negativa EM, el U1 snRNP aparece más homogénea y estable en NP-40 con la disminución de la estabilidad de CHAPS a glicerol a no co-disolvente añadido. Podría ser que la snRNP levadura U1 tiene una cara hidrofóbica y la presencia de NP-40 evita la agregación mediante el recubrimiento de esta superficie. Desafortunadamente, NP-40 no puede ser eliminado por diálisis y la concentración del complejo entonces concentrar la NP-40. Mientras mayor es NP-40 no pareció perturbar el complejo, que afectó negativamente a la congelación de la partícula en hielo vítreo para la crio-EM y grandes agregados de tipo micela se observaron. Detergentes perlas absorbentes se utilizaron con éxito para remove la mayoría de la NP-40 de la muestra U1 snRNP sin que parezca que un impacto en el complejo estructuralmente.
Después de la purificación del complejo con las modificaciones detalladas en el protocolo, un complejo adecuado tanto para tinción negativa y crio-EM se obtuvo, como se evidencia por las imágenes de la césped de partículas y los promedios de clase representativas distintivos (Figura 6).
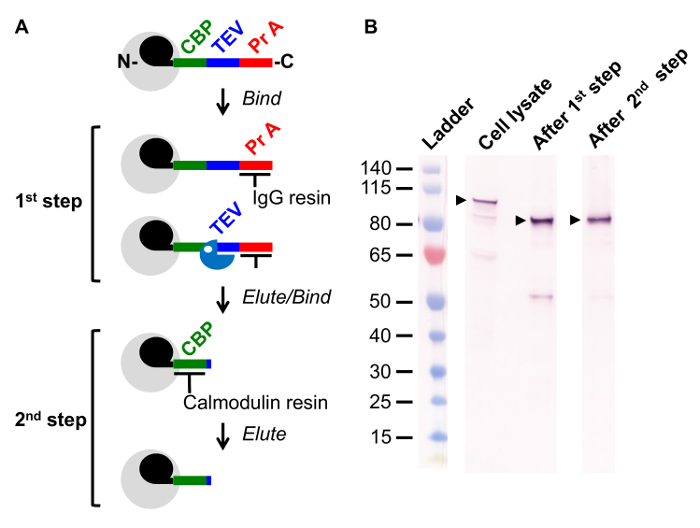
Figura 1. Visión general de Tandem Purificación por Afinidad (TAP) Método. (A) Representación esquemática del método de TAP dos pasos. Complejo de interés (gris) incluye una TAP etiquetado subunidad (negro; U1 snRNP subunidad Snu71) genómicamente fusionado con una secuencia compuesta por un calmodulina de unión del péptido (CBP, verde), un sitio de reconocimiento para la proteasa específica del sitio TEV (azul), IgG y dos regiones de la proteína A de unión (Pr Un , Rojo). En la primera etapa, se incubó el lisado celular con una resina de IgG y el complejo es retenido a través de su interacción con la proteína de una secuencia. El complejo se libera de la resina por escisión mediada TEV. En el segundo paso, se incuba el complejo con una resina de calmodulina, una reacción dependiente de Ca2 +. El complejo se libera a través de la adición de la EGTA quelante de Ca2 +. (B) transferencia de Western después de un TAP proteína marcada a través de las etapas de purificación se ilustra en (A). Se tomaron muestras de lisado celular, después de la primera etapa, y después de la segunda etapa del método de TAP. La proteína reactiva observada en lisado de células migra más rápido después de la 1ª etapa, después de la escisión de la proteasa TEV y por lo tanto la pérdida de la proteína A secuencia. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Ure 2 "src =" / files / ftp_upload / 54389 / 54389fig2.jpg "/>
Figura 2. Optimización del método TAP. (A) se tiñeron con plata gel nativo de complejos de red purificada. PÁGINA materno: patrón de proteína (carril 1); complejo purificado según el método original Toque 3 (carril 2), tres bandas observadas (puntas de flecha); tres eluciones consecutivos a partir de la misma purificación TAP siguientes alteraciones para amortiguar, observaron dos bandas (puntas de flecha) (carriles 3 - 5); más cambios en el método de TAP, produciendo principalmente una sola banda compleja que migraba a ~ 800 kDa (carril 6). (B) Análisis de la composición de un complejo purificado mediante tinción con un único carril de gel con proteínas y ARN manchas reactivas. PÁGINA materno: proteína estándar (carril 1) teñidas con una tinción fluorescente de proteínas; complejo fotografiado con la proteína tinción fluorescente (carril 2), dos bandas observadas; mismo carril en el carril 2, ahora fotografiada con una mancha fluorescente de ácido nucleico (carril 3), dos bandas observadas; y la señal fluorescente durantesentar del complejo de colores en las calles 2 y 3 (carril 4). Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Optimización del método tap. (A) Western blot de mejora de la calidad compleja, siguiendo TAP etiquetado subunidad Snu71 sondeó con anticuerpos α-TAP. Varias bandas proteolizada son visibles inicialmente (carril 1) pero las mejoras reducen la cantidad de bandas inferiores considerablemente (carriles 2 y 3). (B) un gel de SDS representante manchado con una mancha de gel de proteína fluorescente. Gel resuelve 12 de las 17 subunidades de proteínas del complejo. La identidad de la proteína en las bandas fue establecido por espectrometría de masas. Por favor, haga clic aquí para ver una larger versión de esta figura.
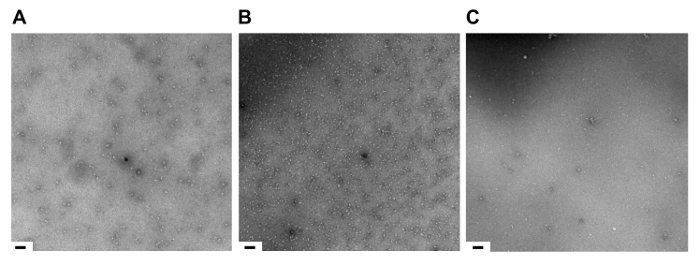
Figura 4. Muestra la calidad en las etapas en el método TAP Evaluado por negativa de la mancha de Microscopía Electrónica de evaluación por microscopía electrónica con tinción negativa del complejo después de:. (A) del st el paso 1 del TAP; (B) la 2ª etapa de TAP; (C ) complejo desestabilizado. Barra de escala, 125 nm. Haga clic aquí para ver una versión más grande de esta figura.
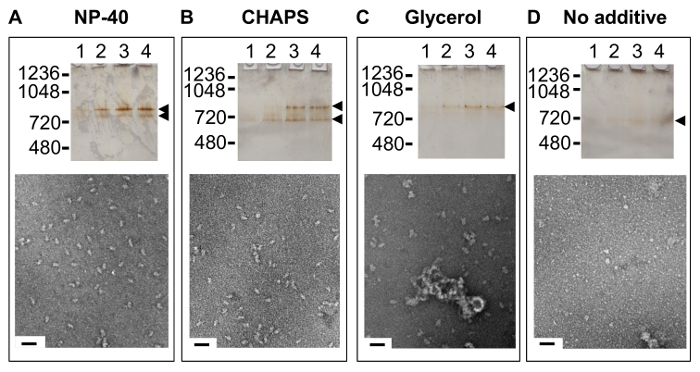
Figura 5. Impacto en la calidad de la muestra de aditivos presentes en el método Tap Tampones Los carriles 1 -. 4 son los primeros cuatro eluciones de la etapa final o 2º de la purificación TAP de complex purificado en presencia de: (A) el detergente no iónico nonil phenoxypolyethoxylethanol (NP-40), analizada por PAGE nativa (arriba) y EM tinción negativa (inferior); (B) el detergente zwitteriónico 3 - [(3- colamidopropil) dimetilamonio] -1-propanosulfonato (CHAPS), analizada por PAGE nativa (arriba) y tinción negativa EM (parte inferior), (C) de glicerol, analizada por PAGE nativa (arriba) y EM tinción negativa (inferior); y (D) sin aditivo, analizada por PAGE nativa (arriba) y tinción negativa EM (parte inferior). Barra de escala, 50 nm. Haga clic aquí para ver una versión más grande de esta figura.
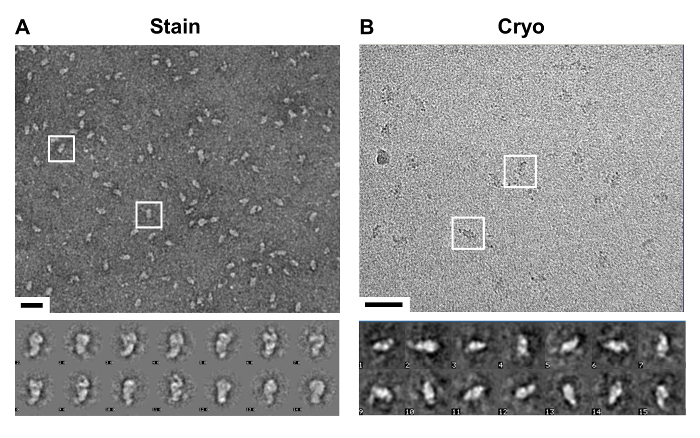
Figura 6. Visualización del método de red purificada complejo. (A) Arriba, una imagen representativa de un electrón microsco tinción negativapy (EM) micrografía. Observado es un césped de partículas de forma similar, dos están en caja, purificado por el método TAP optimizado. En pocas palabras, una selección de los promedios de clase tomadas de las partículas que ponen de relieve las características distintivas de las partículas. Barra de escala, 50 nm. (B) superior, una imagen representativa de una micrografía de crio-EM. Observadas son las regiones de mayor contraste representante de partículas, ver dos de estas regiones en caja. , promedios de clase inferior de partículas seleccionadas congelados en el hielo vítreo. Barra de escala, 50 nm. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El método TAP utiliza dos etiquetas que equilibren la necesidad de más rigurosa y selectiva la unión a una resina de afinidad con el deseo de mantener cerca de condiciones de solución fisiológica. Este equilibrio sirve para preservar la interacción estable (s) de la proteína de la etiqueta con el factor de interacción (s) para la caracterización posterior a la purificación. Además, TAP individuo etiquetada ORFs están disponibles de una fuente comercial, de modo que se puede obtener cualquier cepa de levadura con una proteína etiquetada para cualquier complejo de levadura. La preservación de la integridad de un complejo y la disponibilidad de un recurso para poner a prueba el uso de diferentes subunidades de proteínas etiquetadas en un complejo para la purificación son dos ventajas para la utilización del método de TAP para purificar un complejo nativo. Sin embargo, el protocolo del método TAP original no fue ideado para asegurar que el complejo purificado es de composición y estructuralmente homogéneo. Por consiguiente, el método de TAP se ha modificado, tal como se detalla anteriormente, para lograr este objetivo.
Variospasos del método TAP se evaluaron para establecer el enfoque y las condiciones óptimas para purificar una composición homogénea y complejo estructural. Un paso crítico temprano es la lisis celular. Este paso requiere un equilibrio entre la de un deseo para un alto rendimiento y por lo tanto la lisis máxima, mientras que asegurar que el complejo obtenido es de alta calidad, es decir, homogénea. Diferentes enfoques para la lisis de las células de levadura fueron explorados, incluyendo el uso de un cordón-batidora, procesador de alta fluido pura y molino de bolas. El uso del molino de café menos costoso era menos eficiente que los métodos alternativos (40 - 60% de lisis), pero el complejo aislado y purificado apareció monodisperso mientras que varios de estos enfoques apareció a cualquiera de integridad de las partículas perturb o causar un cierto grado de agregación. Al final, un número significativo de células no fueron lisadas a fin de asegurar que se obtuvo complejo de alta calidad. Mientras que 40 - 60% de lisis es ideal para la levadura snRNP U1, la optimización se sugiere en la aplicación de este protocolo a una nuevo complejo biológico.
Era esencial para identificar los pasos en el método que podría acelerar la purificación para asegurar que la integridad del complejo no se vio comprometida por las proteasas celulares, la disociación de la subunidad, como resultado de la dilución, y se extendió de incubación a 18 ° C durante TEV escisión de la proteasa. Un solo paso para lograr la purificación eficiente momento era asegurar que los lípidos no se transfieren de la clarificación de lisado temprana, ya que los lípidos impiden el flujo durante la purificación de la gravedad. Otros pasos incluyen la identificación de un equilibrio óptimo de la resina de cantidad y de columna de tamaño / dimensiones para lograr la purificación eficiente y una velocidad de flujo máxima. Un paso especialmente crítico fue para reducir el tiempo de la escisión por la proteasa TEV columna. La proteasa TEV utilizado se purificó en casa para asegurarse de que era proteasa / nucleasa libre y en una alta concentración 17. Cantidades significativas de la proteasa TEV se utilizaron para obtener la escisión completa en menos de una hora.
se observó ONTENIDO "> En purificaciones TAP inicial proteólisis. proteólisis durante la purificación puede ser limitado por trabajo de manera eficiente, la adición de una batería de inhibidores, y en particular en los altos lavados de sal de purificación. Una amplia gama de inhibidores de la proteasa suministrados sobre la primera mitad de la purificación mejora en gran medida la estabilidad de la TAP proteína marcada y complejo. Además, la proteolisis se redujo al mínimo aumentando la concentración monovalente de 300 a 500 mM durante la lisis celular y pasos de IgG de purificación. Incluso con las modificaciones anteriores en el método de TAP, sucesiva eluido fracciones fuera de la resina de afinidad CBP varía en su grado de homogeneidad de composición. por lo tanto, la agrupación selectiva de las fracciones de la segunda etapa de cromatografía, la CBP, se justifica.Un aspecto de método de TAP que recibió especial atención fue la importancia de que el detergente no iónico NP-40 a la purificación compleja y a una concentración por encima de su CMC. La presencia de NP-40 a una concentración de 0,08% o superior tenían un efecto beneficioso global sobre la integridad del complejo y el rendimiento final. El efecto beneficioso aparente (s) de NP-40 puede ser debido en parte a reducir las interacciones no específicas entre la resina y el complejo. Mientras NP-40 parece tener un efecto beneficioso durante la purificación, podría ser eliminado después de la purificación sin causar agregación compleja.
Finalmente, crítica para optimizar el método de TAP para el estudio estructural eran varios ensayos complementarios usados para evaluar la integridad y la homogeneidad del complejo. Estos dispersión de la luz incluido, SDS PAGE nativa y probaron utilizando el anticuerpo TAP por Western blot y / o teñido con colorantes fluorescentes, así como microscopía electrónica con tinción negativa. Los cambios realizados en el método TAP se detalla en el presente documento, produjeron un complejo homogéneo en cantidades suficientes para microscopía electrónica. Los cambios adicionales pueden necesitar ser realizado con el método TAP para otros complejos, en particular una disuadirminación de la importancia de NP-40.
En resumen, el método de purificación TAP se puede usar para purificar con éxito complejos macromoleculares de una fuente eucariota de calidad adecuada para el estudio estructural. Hemos establecido un enfoque adecuado para la purificación de la S. cerevisiae U1 snRNP y el detalle de las medidas adoptadas, así como la justificación de muchos de estos cambios. Para otros complejos, se sugiere que el investigador explorar de manera similar a los pasos en el protocolo, lo que puede producir cantidades y calidad de complejo adecuados. Los pasos que deben ser examinados incluyen la lisis celular, la sal y el tipo de aditivo y concentraciones incluyendo detergente, y la sensibilidad del complejo de calcio y metal quelante EGTA. Además, es fundamental que los estudios a largo plazo que el complejo puede ser congelado y descongelado sin perturbar la estructura. Es importante destacar que uno debe tener varias formas alternativas de ensayo de la estructura del complejo durante y después de la purificación. un nativond SDS PAGE, tinción negativa EM, y la dispersión de la luz nos han servido bien en nuestra investigación de la mejor manera de purificar un complejo homogéneo utilizando el método de TAP.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
The authors are grateful for the support and advice of Nikolaus Grigorieff. We thank Anna Loveland, Axel Brilot, Chen Xu, and Mike Rigney for helpful discussions and EM guidance. This work was funded by the National Science Foundation, Award No. 1157892. The Brandeis EM facility is supported by National Institutes of Health grant P01 GM62580.
Materiales
| Name | Company | Catalog Number | Comments |
| S. cerevisiae TAP tagged strain | Open Biosystems | YSC1177 | This is the primary yeast strain used to develop the TAP protocol. Its background is S288C: ATCC 201388: MATa, his3Δ1, leu2Δ0, met15Δ0, ura3Δ0, SNU71::TAP::HIS3MX6 |
| Coffee grinder | Mr. Coffee | IDS77 | Used for cell lysis |
| Hemocytometer, Bright Line | Hausser Scientific | 3120 | Used to assess cell lysis |
| JA 9.100 centrifuge rotor | Beckman Coulter, Inc. | Used to harvest the yeast cells | |
| JA 20 fixed-angle centrifuge rotor | Beckman Coulter, Inc. | Used to clear the cell extract of non-soluble cellular material | |
| Ti 60 fixed-angle centrifuge rotor | Beckman Coulter, Inc. | Used to further clear the soluble cell extract | |
| Thermomixer | Eppendorf | R5355 | Temperature controlled shaker |
| Novex gel system | Thermo Fisher Scientific | ||
| IgG resin | GE Healthcare | 17-0969-01 | Sepharose 6 fast flow |
| Calmodulin resin | Agilent Technologies, Inc. | 214303 | Affinity resin |
| Protease inhibitor cocktail, mini tablets | Sigma Aldrich | 589297 | Mini cOmplete ultra EDTA-free tablets |
| Protease inhibitor cocktail, large tablets | Sigma Aldrich | 5892953 | cOmplete ultra EDTA-free tablets |
| Phenylmethanesulfonyl fluoride (PMSF) | Dissolved in isopropanol | ||
| 2 mL Bio-spin column | Bio-Rad Laboratories, Inc. | 7326008 | Used to pack and wash the Calmodulin resin |
| 10 mL poly-prep column | Bio-Rad Laboratories, Inc. | 7311550 | Used to pack and wash the IgG resin |
| Precast native PAGE Bis-Tris gels | Life Technologies | BN1002 | Novex NativePAGE Bis-Tris 4-16% precast polyacrylamide gels |
| NativeMark protein standard | Thermo Fisher Scientific | LC0725 | Unstained protein standard used for native PAGE. Load 7.5 μL for a silver stained gel and 5 μL for a SYPRO Ruby stained gel |
| Precast SDS PAGE Bis-Tris gels | Life Technologies | NP0321 | Novex Nu-PAGE Bis-Tris 4-12% precast polyacrylamide gels |
| PageRuler protein standard | Thermo Fisher Scientific | 26614 | Unstained protein standard used for Western blotting |
| SDS running buffer | Life Technologies | NP0001 | 1x NuPAGE MOPS SDS Buffer |
| TAP antibody | Thermo Fisher Scientific | CAB1001 | Primary antibody against CBP tag |
| Secondary antibody | Thermo Fisher Scientific | 31341 | Goat anti-rabbit alkaline phosphatase conjugated |
| BCIP/NBT | Thermo Fisher Scientific | 34042 | 5-bromo-4-chloro-3-indolyl-phosphate/nitro blue tetrazolium |
| Dialaysis units | Thermo Fisher Scientific | 88401 | Slide-A-Lyzer mini dialysis units |
| Centrifugal filter units, 100kDa MWCO | EMD Millipore | UFC5100008 | Amicon Ultra-0.5 Centrifugal Filter Unit with Ultracel-100 membrane |
| Detergent absorbing beads | Bio-Rad Laboratories, Inc. | 1523920 | Bio-bead SM-2 absorbants |
| SYBR Green II | Thermo Fisher Scientific | S-7564 | Flourescent dye for nucleic acid staining, when detecting with SYPRO Ruby present, use excitation wavelength of 488 nm and emission wavelength of 532 nm |
| SYPRO Ruby | Molecular Probes | S-12000 | Flourescent dye for protein staining, when detecting with SYBR Green II present, use excitation wavelength of 457 nm and emission wavelength of 670 nm |
| Copper grids | Electron Microscopy Sciences | G400-CP |
Referencias
- Gavin, A. C., et al. Functional organization of the yeast proteome by systematic analysis of protein complexes. Nature. 415 (6868), 141-147 (2002).
- Rigaut, G., Shevchenko, A., Rutz, B., Wilm, M., Mann, M., Seraphin, B. A generic protein purification method for protein complex characterization and proteome exploration. Nature Biotechnology. 17 (10), 1030-1032 (1999).
- Puig, O., et al. The tandem affinity purification (TAP) method: A general procedure of protein complex purification. Methods. 24 (3), 218-229 (2001).
- Vaillancourt, P., Zheng, C. F., Hoang, D. Q., Breister, L. Affinity purification of recombinant proteins fused to calmodulin or to calmodulin-binding peptides. Methods Enzymol. 326, 340-362 (2000).
- Braisted, A. C., Wells, J. A. Minimizing a binding domain from protein A. Proc Natl Acad Sci U S A. 93 (12), 5688-5692 (1996).
- Kapust, R. B., et al. Tobacco etch virus protease: mechanism of autolysis and rational design of stable mutants with wild-type catalytic proficiency. Protein Eng. 14 (12), 993-1000 (2001).
- Nallamsetty, S., et al. Efficient site-specific processing of fusion proteins by tobacco vein mottling virus protease in vivo and in vitro. Protein Expr Purif. 38 (1), 108-115 (2004).
- Ghaemmaghami, S., et al. Global analysis of protein expression in yeast. Nature. 425 (6959), 737-741 (2003).
- Howson, R., et al. Construction, verification and experimental use of two epitope-tagged collections of budding yeast strains. Comp Funct Genomics. 6 (1-2), 2-16 (2005).
- Cox, D. M., Du, M., Guo, X., Siu, K. W., McDermott, J. C. Tandem affinity purification of protein complexes from mammalian cells. Biotechniques. 33 (2), 267-268 (2002).
- Gully, D., Moinier, D., Loiseau, L., Bouveret, E. New partners of acyl carrier protein detected in Escherichia coli by tandem affinity purification. FEBS Lett. 548 (1-3), 90-96 (2003).
- Tharun, S. Purification and analysis of the decapping activator Lsm1p-7p-Pat1p complex from yeast. Methods Enzymol. 448, 41-55 (2008).
- Xu, X., Song, Y., Li, Y., Chang, J., Zhang, H., An, L. The tandem affinity purification method: an efficient system for protein complex purification and protein interaction identification. Protein Expr Purif. 72 (2), 149-156 (2010).
- Cheng, Y., Grigorieff, N., Penczek, P. A., Walz, T. A Primer to Single-Particle Cryo-Electron Microscopy. Cell. 161 (3), 438-449 (2015).
- Gottschalk, A., et al. A comprehensive biochemical and genetic analysis of the yeast U1 snRNP reveals five novel proteins. RNA. 4 (4), 374-393 (1998).
- Neubauer, G., Gottschalk, A., Fabrizio, P., Seraphin, B., Luhrmann, R., Mann, M. Identification of the proteins of the yeast U1 small nuclear ribonucleoprotein complex by mass spectrometry. Proc Natl Acad Sci U S A. 94 (2), 385-390 (1997).
- Lucast, L. J., Batey, R. T., Doudna, J. A. Large-scale purification of a stable form of recombinant tobacco etch virus protease. Biotechniques. 30 (3), 544-546 (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados