Method Article
ADN de isótopos estables de palpado (ADN-SIP)
En este artículo
Resumen
ADN de isótopos estables de sondeo es un método de cultivo independiente para identificar y caracterizar las comunidades activas de microorganismos que son capaces de utilizar sustratos específicos. Asimilación de sustrato enriquecido en los isótopos pesados lleva a la incorporación de los átomos marcados en la biomasa microbiana. Ultracentrifugación en gradiente de densidad recupera la etiqueta de ADN para posterior análisis molecular.
Resumen
ADN de isótopos estables de sondeo (ADN-SIP) es una poderosa técnica para la identificación de microorganismos activos que asimilar ciertos sustratos de carbono y nutrientes en la biomasa celular. Como tal, esta técnica de cultivo-independiente ha sido una importante metodología para la asignación de la función metabólica de las diversas comunidades que habitan en una amplia gama de ambientes terrestres y acuáticos. Después de la incubación de una muestra ambiental con isótopos estables compuestos marcados, el ácido nucleico extraído se somete a ultracentrifugación en gradiente de densidad y el fraccionamiento del gradiente posteriores para separar los ácidos nucleicos de diferentes densidades. La purificación del ADN a partir de cloruro de cesio recupera no marcado y marcado de ADN para posterior caracterización molecular (por ejemplo, huellas dactilares, microarrays, bibliotecas clon, la metagenómica). Este protocolo de vídeo JoVe proporciona visuales paso a paso las explicaciones del protocolo de gradiente de densidad gradiente de fraccionamiento de ultracentrifugación, y la recuperación de ADN marcado. El protocolo también incluye la muestra de datos SIP y pone de relieve importantes consejos y precauciones que se deben considerar para asegurar el éxito de ADN-SIP análisis.
Protocolo
1. Preparación de los reactivos
ADN-SIP requiere el uso de reactivos que se deben preparar con antelación del procedimiento actual. Las instrucciones para la preparación de cada reactivo se muestran en esta sección y se modifican a partir de una anterior protocolo SIP 1.
- Cloruro de cesio (CsCl) solución para la preparación de los gradientes de SIP - Preparar una solución 7.163 M CsCl gradualmente disolviendo 603,0 g de CsCl en agua destilada y desionizada (DDC 2 O) a un volumen final de 500 ml. Tenga cuidado de no exceder de 500 ml! El calentamiento de la solución mientras se agita ligeramente ayudará a disolver todos los CsCl. Alícuota de la solución final en alícuotas de sellado. En nuestro laboratorio, una práctica común de almacenamiento es para preparar 100 ml alícuotas de 125 mL viales de suero, que luego son sellados con precinto de tapones de caucho butilo. Las alícuotas de sellado se puede almacenar indefinidamente a temperatura ambiente (20 ° C). Los sellos de ayudar a evitar la evaporación y la formación de CsCl "corteza". Determinar la densidad de la solución de un peso por triplicado, de 100 l alícuotas, o mediante el uso de un refractómetro digital (por ejemplo, Reichert AR200) que ha sido cuidadosamente calibrado para las soluciones de CsCl. Una vez calibrado correctamente, el AR200 Reichert es consistente y proporciona mediciones precisas desde hace varios años. A temperatura ambiente (20 ° C), la densidad final de esta solución suele oscilar desde 1.88 hasta 1.89 g -1 ml. La densidad varía ligeramente cada vez que se prepara un nuevo stock.
- Solución de cloruro de cesio para la preparación de gradientes con bromuro de etidio (EtBr) - Combinar 250 g de CsCl con 250 ml de agua estéril ddH 2 O. Alícuota de esta solución en distintos viales de suero que se han engarzado sellado con los sellos de goma de butilo como se describe en 1.1.
- Buffer gradiente - Combine 50 ml de 1 M Tris-HCl, 3,75 g de KCl y 1 ml de EDTA 0,5 M a 400 ml de agua. Disolver el KCl, a continuación, añadir ddH2O a 500 ml. Filtro de esterilización y autoclave. La solución final es de 0,1 M Tris, 0,1 M KCl y 1 mM EDTA.
- Polietilenglicol (PEG) - Prepare la solución de PEG por disolución de 150 g de polietilenglicol 6000 y 46,8 g de NaCl en agua estéril ddH 2 O hasta un volumen total de 500 ml (PEG 30%, 1,6 M NaCl). Autoclave.
Nota: Esta solución se separa en dos fases con autoclave. Incluye una barra de agitación en la botella de autoclave para que la solución puede estar bien mezclado cuando esto ocurre. - TE Buffer - Preparar una solución de 10 mM Tris-HCl (pH 8,0) y 1 mM EDTA (pH 8,0) en estériles ddH 2 O del agua, el uso de soluciones de archivo de un autoclave EDTA M Tris-HCl (pH 8,0) y 0,5 M ( pH 8,0). Filtro de esterilización y autoclave.
- 70% de etanol - Combine 350 ml de etanol de alta pureza con 150 ml de agua estéril ddH2O.
2. Incubación de muestras y extracción de ADN
Para SIP ADN incubaciones, las muestras se incuban normalmente con pesados de isótopos de carbono (13 C) del sustrato. Períodos de incubación y las condiciones (por ejemplo, la suplementación de nutrientes, humedad, luz) puede variar dependiendo del tipo de muestra que se incuba y la naturaleza del sustrato. ADN-SIP experimentos se han realizado exitosamente usando una variedad de compuestos de carbono solo 2,3, multi-compuestos de carbono 4,5,6, y el uso de nitrógeno u oxígeno etiquetados 7,8 9. Sin embargo, una desventaja de usar N-15 o el 18 compuestos O-etiqueta es la separación física disminuida de ácido nucleico marcado, sobre todo debido a la presencia de menos de nitrógeno y átomos de oxígeno en el ADN y en relación con los átomos de carbono de ARN.
Un crítico de control para el ADN-SIP experimentos de incubación idénticos establecido con nativos (por ejemplo, 12 C) del sustrato. Esta incubación se ofrece una comparación posterior para asegurarse de que cualquier etiquetado aparente de ácido nucleico que no era un artefacto de la ultracentrifugación o G + C las diferencias de densidad de contenido en el ADN que contribuyen a la separación 10. También es importante para mantener el material de muestra congelada para la comparación de "luz" y el ADN "pesado", y vale la pena incluir un control sin sustrato para evaluar los cambios de fondo en toda la población de la incubación SIP.
- Incubar las muestras ambientales en el microcosmos que contiene sustrato marcado (Figura 1). En nuestra experiencia, hemos encontrado que una incorporación mínima de entre 500 a 500 mol de carbono 13 C por gramo de muestra adecuado para muestras que contienen biomasa como una muestras de suelo. Para las muestras que contienen menos de la biomasa acuática que los suelos, 100 a 100 mol de carbono 13 incorporado C por litro puede producir un nivel detectable de gran isotópica firma 1. La cantidad de modificación de carbono, la proporción de carbono incorporado en la biomasa y la necesidad de adición de nutrientes suplementarios para la asimilación de todo dependerá de las características de las muestras que se analizaron y el objetivo organisms de interés. Un único conjunto de directrices de incubación de la muestra no será de aplicación para todas las muestras. Es importante destacar que la concentración de sustrato utilizado para la incubación SIP idealmente debería ser lo más cercano posible a la concentración que normalmente se encuentran in situ, el sesgo experimental puede ser una consecuencia de las condiciones de enriquecimiento de la cultura 10.
- Después de la incubación de la muestra con el sustrato de isótopos estables etiquetados, extraer el ADN de un microcosmos con un riguroso protocolo de extracción (por PCR o la clonación de insertar pequeñas) o una lisis enzimática de confianza para la clonación de alto peso molecular (por ejemplo, de gran inserción metagenómica). ARN co-extracción generalmente no afecta el análisis, por lo que los protocolos de producción de ARN, así como el ADN puede ser utilizado. La ultracentrifugación del ADN extraído no se corte los fragmentos más cortos que ~ 50 pares kb 1.
- Cuantificar el ADN extraído antes de la instalación de los tubos de ultracentrifugación gradiente CsCl. Cuantificar el ADN con un espectrofotómetro (por ejemplo, Nanodrop 2000) si el protocolo de extracción de ADN sólo produce (por ejemplo, basada en columnas kits). Por otra parte, cuantificar mediante electroforesis en gel de agarosa.
3. La preparación de soluciones de gradiente de ultracentrifugación
Este procedimiento consiste en la adición de ADN para ultracentrífuga tubos. Hay más de un tipo de manguera y el rotor por lo que el protocolo exacto será variable y dependerá de las instrucciones del fabricante. Dicho esto, se recomienda el uso de un rotor vertical y para asegurar la máxima separación posible de ADN ligeros y pesados. Utilizamos un Beckman-Coulter Vti 65,2 rotor con 16 pozos para la celebración de los tubos de 5,1 ml Polyallomer Quickseal y el protocolo se describen los pasos y las consideraciones para estas condiciones.
- Utilizando las concentraciones de ADN determinada en el paso 2.3, calcular el volumen necesario de ADN extraído lo que se requiere para proporcionar 0,5 mg - 5 mg de ADN en los tubos de ultracentrífuga.
- Combinar el ADN extraído (0,5 - 5 mg) con el tampón de degradado (véase el paso 1.3) y 4.8 ml de 7.163 M CsCl a un volumen total de aproximadamente 6 ml en una estéril desechable de 15 ml tubo. Tenga en cuenta que la densidad de la solución de CsCl puede variar incluso en la misma molaridad (véase el paso 1.1). La siguiente ecuación se puede utilizar para determinar el volumen de Gradient Buffer / ADN mezcla de lo que se requiere para generar una relación de mezcla adecuada:
Gradiente de volumen de solución tampón y el ADN (ml) = (stock CsCl densidad de la solución - la densidad final deseado) x volumen de la solución madre CsCl añadido x 1,52
Especificar el volumen de la solución madre CsCl en 4.80 ml. La densidad deseada final debe ser 1,725 g ml -1. La densidad de la solución madre se determinó en el paso 1.1.
Tenga en cuenta también que los volúmenes relativos de CsCl y gradiente de Buffer / ADN se traducirá en un volumen combinado de más de 5,1 ml. Preparación de volúmenes superiores a la capacidad del volumen máximo de los tubos de ultracentrífuga (más de 5,1 ml) se asegurará de que no hay una solución suficiente para llenar completamente el tubo. - Mezclar invirtiendo 10 veces. El ADN es estable a temperatura ambiente en CsCl.
4. La creación de un gradiente de control EtBr (opcional)
Debido a que EtBr es un colorante intercalante que los complejos con el ADN por lo que es visible bajo luz ultravioleta, los gradientes de control que contiene EtBr son útiles porque proporcionan la confirmación visual inmediata de la formación de pendiente antes de fraccionamiento de los tubos de muestra (por ejemplo, Figura 1). La inclusión de un tubo de control conteniendo EtBr y una mezcla de ambos C-12 y el 13 de ADN-ADN C (o 14, N-ADN y 15 N-DNA) permite la visualización inmediata de la formación de la banda dentro de los tubos sobre la terminación de ultracentrifugación. Esto es importante porque un tubo roto en la ultracentrifugación o condiciones de funcionar correctamente programada puede resultar en la formación del gradiente no. Unido al ADN, EtBr disminuye la densidad del ADN y, en consecuencia, un protocolo diferente es seguir en la preparación gradientes. Tenga en cuenta que otras manchas de ácido nucleico puede ser utilizado en lugar de EtBr 11, pero el protocolo requiere la optimización con fluoróforos otros.
- El gradiente de control requiere de dos volúmenes de ADN genómico: una totalmente marcado con isótopos estables y uno sin etiqueta. Por lo general utilizar Sinorhizobium meliloti cultivadas en medios que contienen 13 o C-12 C-glucosa como única fuente de carbono, o Methylococcus baño cepa capsulatus cultivadas en la presencia de 13 o C-12 C-metano como nuestros controles.
- Combinar una cantidad de entre 5 y 10 mg tanto de la 12 C-ADN y 13 C-ADN con el tampón de degradado para un volumen final de 1,00 ml de una desechable de 15 ml con tapón de rosca del tubo.
- Añadir 1,00 g de CsCl sólido para el mismo tubo. Mezclar por inversión.
- Añadir 110 lde una solución de 10 mg ml -1 EtBr y 4,3 ml de una solución de 1 g ml -1 acciones CsCl a la misma tapa de rosca del tubo utilizado en el paso 4.2. La densidad final de la solución se aproximará a la de la solución madre de CsCl original.
- Adicionales "en blanco" solución de control que contiene EtBr también será necesario para contrarrestar la solución creada en el paso 4.4. Combine 1,00 ml de tampón de gradiente, 1,00 g de CsCl, 110 l de una solución de 10 mg ml -1 EtBr y 4,3 ml de una solución de 1 g ml -1 CsCl acciones por separado en un 15 ml con tapón de rosca del tubo y mezclar por inversión.
5. Ultracentrifugación
- El uso de un bulbo y pipeta Pasteur, llenar cuidadosamente los tubos de ultracentrifugación con gradiente de soluciones preparadas en el paso 3.2 (o 4.4, cuando la preparación de los pasos de un gradiente de control EtBr). Añadir con cuidado las soluciones a los tubos con una pipeta Pasteur. Marque los tubos en el hombro de tubo con un marcador permanente fino. PRECAUCIÓN: Asegúrese de que los tubos se llenan exactamente a la base del cuello del tubo. Tubos de llenado insuficiente es probable que estalló durante la ultracentrifugación.
- Cuando todos los tubos necesarios están llenos de soluciones de la muestra, registra la masa exacta de cada tubo. Tubos de par y el equilibrio que dentro de 0-10 mg. Para el equilibrio, encontrar parejas casi igualó y añadir o eliminar pequeñas cantidades de solución hasta que se equilibran, manteniendo el nivel de la solución lo más cercano a la base del cuello del tubo como sea posible. Tenga en cuenta que para el pesaje de los tubos, se utiliza una invertida de 15 ml con tapón de rosca del tubo que ha sido cortada por la mitad como un soporte de tubo para el equilibrio.
- Sellado de los tubos con un "tubo de Topper" de acuerdo con las instrucciones del fabricante.
- Compruebe que los tubos están sellados adecuadamente, invirtiendo y aplicando una presión moderada. Pesar los tubos de nuevo para comprobar que siguen siendo equilibrado después de sellar dentro de 0-10 mg.
- Compruebe cada rotor y con cuidado para garantizar que los pozos estén limpios y libres de escombros y polvo que pueden perforar los tubos durante la ultracentrifugación.
- Insertar los tubos en el rotor con los pares de equilibrio frente a la otra. Anote la ubicación del rotor de cada muestra porque el proceso de ultracentrifugación puede hacer que las etiquetas de marcadores para ser dañado o borrado. Selle cuidadosamente los pozos de rotor según lo indicado por el fabricante.
- Cargar el rotor en la ultracentrífuga. Cierre la puerta de ultracentrifugación y aplicar el vacío. Si se utiliza un Vti 65,2 rotor, ajuste la velocidad de rotación de 44.100 rpm (177.000 xg ~ av), la temperatura a 20 ° C el tiempo, y ultracentrifugación de 36 a 40 horas. Seleccione vacío, la máxima aceleración, y apagar el freno (asegura gradiente no interrumpida por la desaceleración). Tenga en cuenta que desactivar el freno se sumarán a las 1-2 horas de tiempo de ejecución. También tenga en cuenta que los tiempos de ejecución más corto, no puede lograr la resolución de banda suficiente. Tandas largas ultracentrifugación se recomiendan, ya que conducen a una mayor resolución de las distintas bandas de ácidos nucleicos.
- Inmediatamente después de la finalización del procedimiento de ultracentrifugación, extraer el rotor con cuidado. Evitando cualquier inclinación o golpes del rotor, retire suavemente los tubos del rotor para evitar molestias a los gradientes dentro de los tubos. En raras ocasiones, un tubo se rompió durante la carrera. Si es así, existe la posibilidad de que los gradientes en las trompas de otros no está bien formada. Si un gradiente de control se incluyó, marque este tubo cuidadosamente a la luz ultravioleta para confirmar la formación de pendiente. Si el gradiente no se ha formado adecuadamente en el tubo de control, lo mejor es repetir todo el paso 5. Tenga en cuenta que el tubo de control EtBr y su control en blanco se puede almacenar en la oscuridad y volver a utilizar durante un máximo de seis meses. Tenga cuidado de limpiar el rotor cuidadosamente de acuerdo a las instrucciones del fabricante una vez que el tubo de explosión se ha eliminado. No utilice cepillos metálicos o abrasivos para limpiar los pozos del rotor para evitar arañazos a los pozos de rotor! Rotor específicos cepillos y solución de limpieza se pueden comprar de Beckman.
6. Gradiente de Fraccionamiento
Hay dos métodos que se utilizan actualmente para recuperar el ADN de los tubos de ultracentrífuga: el fraccionamiento y la extracción de la aguja. Este protocolo sólo se describirá el proceso de extracción de ADN mediante la técnica de fraccionamiento. Esto es así porque para la mayoría de los experimentos SIP, la etiqueta de ADN no pueden ser visualizados con bromuro de etidio y en su lugar deben ser detectadas mediante la comparación de la luz equivalentes y fracciones pesadas de los tubos de muestras múltiples. Una bomba de jeringa es muy recomendable para recuperar la igualdad de las fracciones de gradiente de densidad de los tubos de ultracentrífuga. Utilizamos un modelo BSP bomba de infusión (Braintree Scientific Inc.). Una bomba peristáltica de bajo flujo o una bomba de HPLC también puede ser utilizado.
- Llenar un recipiente estéril de 60 ml jeringa con agua estéril ddH2O contiene suficiente colorante azul de bromofenol para proporcionar un color azul oscuro. Colocar la jeringa en el brazo de carga de la bomba de jeringa.Conecte el tubo de bomba con un calibre 23 una "aguja y pasar a la bomba hasta que algún ddH2O ha llegado hasta el final de la Nota de la aguja. Que cualquier burbuja de aire en el suministro de ddH2O afectará negativamente al proceso de fraccionamiento.
- Fijar uno de los tubos de ultracentrifugación en un soporte de sujeción. Asegúrese de que la pinza es lo suficientemente apretado para evitar que el tubo se desplace, pero no de tal manera que la presión en el tubo que causan la liberación de la solución de CsCl cuando el tubo se perfora. Perforar el fondo del tubo a lo largo de la costura del tubo con un nuevo calibre 23 una "aguja. Para obtener los mejores resultados, perforar el tubo de una manera controlada, rápida y segura. Esto es muy difícil de hacer bien, la práctica varias veces antes de Este es el primer intento de tubos de muestras.
- Para cada muestra, preparar 12 tubos de microcentrífuga estéril de 1,5 ml con etiquetas que indiquen el número de muestra y de la fracción (1-12; pesado a ligero). Utilizando la aguja unida a la tubería de la bomba (paso 6.1), perforar la parte superior del tubo en el tubo superior del hombro, a lo largo de la costura. Recoger la solución degradado con los tubos de microcentrífuga. Tal como se realizó para la parte inferior del tubo, perfore el tubo de una manera rápida y controlada. La práctica de antemano y ser muy cuidadoso en usar un movimiento controlado para evitar tirar la aguja obligadas a pasar por el tubo y en un dedo! Utilizar un tipo de bomba previamente calibrado que producirá 12 x 425 fracciones l en 12 minutos (425 minutos l -1).
- Use un refractómetro digital (por ejemplo, Reichert AR200, recomendado) o una balanza analítica para comprobar la densidad de las fracciones de un gradiente para confirmar la formación de pendiente adecuada. Usted tendrá que utilizar aproximadamente 50 l de muestra para esta prueba. A menudo incluyen el ADN cultivo puro en un tubo (como se describe para la preparación de los gradientes de control EtBr) para servir como un control para el fraccionamiento y la usamos para determinar la densidad. Esperamos que la densidad en un rango de 1,690 a 1,760 ~ -1 g ml, con una densidad media de ~ 1,725 g -1 ml.
7. Precipitaciones ADN
- Precipitar el ADN de todas las fracciones, en primer lugar la adición de 20 mg de poliacrilamida lineal como un portador de la precipitación. Mezclar por inversión. Agregar 2 volúmenes de solución de PEG (ver paso 1) y mezclar por inversión. Tenga en cuenta que un portador de las precipitaciones (por ejemplo, el glucógeno o poliacrilamida lineal) es fundamental para la recuperación cuantitativa del ADN de las facciones del gradiente, pero se debe tener precaución si el glucógeno se utiliza como soporte para la precipitación de este protocolo. Los preparativos de glucógeno se ha demostrado que estar contaminados con bacterias de ácido nucleico y la contaminación pueden confundir la interpretación de las fracciones del gradiente SIP 12.
- Deje los tubos a temperatura ambiente durante 2 horas para permitir que el ADN precipitado. Si lo desea, los tubos se puede dejar toda la noche a temperatura ambiente.
- Se centrifuga a 13.000 g durante 30 minutos con la parte de atrás de los tubos hacia el exterior por un tubo de orientación constante en el rotor. Con cuidado aspirar y descartar el sobrenadante. Una pastilla debe ser visible, pero puede ser muy difícil de ver en esta etapa. Trabajo en una fuente de luz brillante (por ejemplo, la lámpara de escritorio) para ayudar en la visualización de la pastilla.
- Lavar el precipitado con 500 l de etanol al 70%. Se centrifuga a 13.000 g durante 10 minutos. Con cuidado aspirar y descartar el sobrenadante. El pellet por lo general será más visible en este paso, pero se disocian de la pared del tubo más fácilmente.
- Dejar que el precipitado se seque a temperatura ambiente durante 15 minutos.
- Suspender cada pellet en 50 l de buffer TE (véase el paso 1.5). Carrera de 5 l de cada fracción en un gel de agarosa de acuerdo a los protocolos estándar de laboratorio.
8. Caracterización de la fracción
El método utilizado para caracterizar las fracciones del gradiente para evaluar el éxito de la incubación SIP puede variar dependiendo del laboratorio y la disponibilidad de equipos. El uso de un método de toma de huellas dactilares para la selección del gen 16S ARNr es un enfoque común y métodos tales como el polimorfismo de longitud de fragmentos de restricción terminales (T-RFLP) o la electroforesis en gel de gradiente desnaturalizante (DGGE) son apropiados (Figura 1). Siguiendo el protocolo descrito anteriormente, esperamos que el ADN de la luz que se asocia con las fracciones 9-11 (~ 1.705-1.720 g ml -1) y las huellas dactilares de ADN pesado para ser asociada a las fracciones 5-8 (~ 1.720-1.735 g ml -1 ). Huellas digitales únicas asociadas con las fracciones 5-8 de isótopos estables muestras incubadas, pero no con los controles nativos de sustrato incubado proporciona una fuerte evidencia la vinculación de organismos específicos con el metabolismo de sustrato marcado particular. Si el ADN sigue siendo insuficiente etiquetados para algunas aplicaciones (hibridación, la metagenómica), la amplificación de desplazamiento múltiple se puede utilizar para producir mayores cantidades de 13 a 15, pero esto puede introducir quimeras en el ADN amplificado 14,16.
9. Resultados
SIP típica de ADN resultados demuestran una separación de ADN no marcado y marcado en el gradiente formado por ultracentrifugación. Lo ideal sería que la resolución completa de material de alto peso molecular genética (por ejemplo, 13 C, 15 N) a partir de materiales no etiquetados se logrará. La resolución puede ser testigo visual mediante la observación de formación de la banda en los tubos de control EtBr. Las concentraciones de ADN genómico recuperado contenidas en las fracciones de gradiente individuo también puede ser utilizado para confirmar la formación de pendiente adecuada.
Para este protocolo, se incluyen los resultados representativos de ultracentrifugación gradiente a cabo utilizando ácido nucleico a partir de dos cultivos puros (Figura 2). El gradiente fraccionado se incluye aquí se preparó utilizando ADN genómico extraído de S. meliloti (ATCC 1021), y marcado con 13 C M. str capsulatus. Bath. Después de la ultracentrifugación, el fraccionamiento y la recuperación de ADN, no marcado y marcado de ADN genómico se separan en fracciones de gradiente respectivos con diferentes densidades (Figura 2A). Pesado isótopo de ADN marcado se puede observar en las fracciones 4-5, mientras que el ADN no marcado se encuentra en altas concentraciones en las fracciones 90-10. El ADN de cada fracción se caracterizó por electroforesis en gel de gradiente desnaturalizante 17 y los productos de la PCR-amplificación generada discretos patrones de bandas correspondientes a los dos organismos incluidos en el gradiente (Figura 2B). La densidad de las fracciones oscilaron entre ~ 1,580 a 1,759 g ml -1, y se muestran en orden decreciente de la densidad de izquierda a derecha.
A pesar de la separación de puro 13 C-12 y C-ADN puede ser pronunciada (Figura 2), incubaciones muestras ambientales puede ser más difícil de interpretar. Por ejemplo, se incubaron los suelos de la tundra Resolute Bay (Nunavut, Canadá), ya sea con 12 o C-13 C-glucosa etiquetada por un período de 14 días a 15 ° C. Los geles de agarosa de ADN purificado fracción gradiente demostrado que el ADN genómico fue "manchada" a través de las fracciones 70-10 para los 12 C y 13 C-incubación (Figura 3A y 3C, respectivamente). En este caso, 13 C-enriquecimiento de la biomasa microbiana de los taxones en particular sólo puede ser determinado con un enfoque como el DGGE de genes del 16S rRNA. El 12 C-glucosa ADN suelo incubadas genera patrones similares en todas las fracciones del gradiente (Figura 3b), pero la 13 C-glucosa muestra incubada genera las huellas dactilares de DGGE que están asociadas únicamente con las fracciones 5-8 (Figura 3D). De particular interés son las bandas de conservación indicada por las flechas. Esta dominante filotipo es consistente a través de todas las fracciones de gradiente, pero se desplaza a fracciones más pesadas de ADN obtenido a partir de 13 C-glucosa suelo incubadas. ADN posterior secuenciación de esta banda y / o clonar el análisis de la biblioteca que confirmaría la identidad de este gen particular 16S ARNr y guía de metagenómica posterior o enfoques basados en el cultivo.

Figura 1. Esquema de un experimento de ADN-SIP participación de incubación de la muestra, extracción de ADN, CsCl ultracentrifugación en gradiente de densidad y la caracterización del ADN con técnicas moleculares.
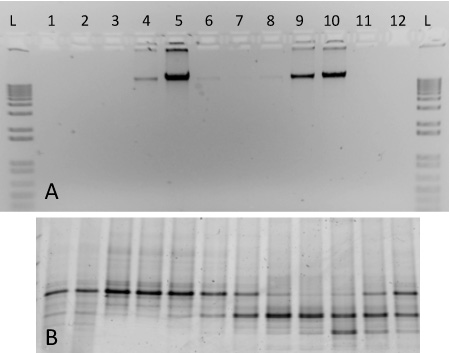
Figura 2. Resultados esperados para un fraccionamiento gradiente SIP como el ADN a partir de dos cultivos puros. (A) Las alícuotas de ADN 1 a 12 fracciones del gradiente se ejecutan en un 1% en gel de agarosa de un degradado que contiene marcado con 13 C M. capsulatus baño cepa (fracciones 4-6) y 12 C-etiquetados S. meliloti (fracciones 8-10). Una escala de 1 kb se incluye para la comparación (B) PCR-amplificación de ADN de las mismas fracciones se ejecutan en un gel de DGGE del 10%. Los patrones de huellas dactilares revelan claras diferencias entre las fracciones 5 y 9, por ejemplo.

Figura 3. Resultados esperados para fraccionamientos gradiente SIP de incubación de la muestra del suelo. Alícuotas de las fracciones del gradiente de la edad de 12 C-glucosa en suelo enmendado (A) y 13 C-glucosa en suelo enmendado (C) se llevaron a cabo el 1% en geles de agarosa y una escala de 1 kb se incluye para la comparación. Las huellas dactilares correspondientes DGGE para cada una de estas muestras se muestran en (B) y (D). Toma de huellas dactilares de las fracciones revela el enriquecimiento de los taxones de bacterias en particular en la 13 C-glucosa de la muestra modificada en las fracciones 5-8 (D).
Discusión
El diseño apropiado de isótopos estables experimentos de sondeo es de importancia crítica para la obtención de ADN marcado por encima de la comunidad de fondo sin etiquetar. Consideraciones relacionadas con la muestra de los tiempos de incubación, las concentraciones de sustrato, las condiciones de incubación (por ejemplo nutrientes, contenido de humedad del suelo), cruzar la alimentación y la replicación se han discutido en otros lugares 10,18 y se recomienda al lector consultar estas publicaciones en el diseño de una incubación de SIP. Relacionados con el protocolo actual, vale la pena comentar sobre otras consideraciones relacionadas con la interpretación de los datos de los gradientes de SIP. Debido a la naturaleza del proceso de ultracentrifugación, es importante incluir, además, los controles, tales como cultivos puros y nativos-sustrato muestras incubadas para asegurarse de que las bandas que aparecen o desaparecen en fracciones particulares no son artefactos del protocolo en sí. Por ejemplo, el ADN dentro de un gradiente de ultracentrífuga puede no ser visible en un gel de agarosa (Figura 2A), pero aún así pueden contaminar a todo lo largo del gradiente (Figura 2B). Aunque M. capsulatus patrones son más distintas en las fracciones densas (5-7) del gel se muestra en la Figura 2B, el patrón de DGGE se observó la misma aún en el más ligero de la fracción (12). Con los controles considerados cuidadosamente, la interpretación de los datos SIP fracción gradiente es posible.
Debido a la naturaleza de algunos experimentos bien diseñados SIP (por ejemplo, cerca de las concentraciones de sustrato in situ, tiempos cortos de incubación), la incorporación de isótopos puede ser muy baja 10. Además, la mayoría de los microorganismos en ambientes terrestres o acuáticos tienen largos tiempos generacionales frente a un crecimiento en el laboratorio, y requieren largos tiempos de incubación para alcanzar niveles detectables de enriquecimiento isotópico. Otras poblaciones pueden ser capaces de metabolizar una gran variedad de sustratos, y no se pueden crecer completamente el sustrato marcado. También hay comunidades (por ejemplo, el agua subterránea) que pueden estar asociados con los niveles de biomasa de baja y generan rendimientos bajos de ácido nucleico extraído. En todos estos casos, la recuperación cuantitativa de la etiqueta de los ácidos nucleicos puede ser un desafío.
Para sortear estas limitaciones, una variedad de moléculas portadoras naturales y sintéticos existentes que ayudan en la precipitación y la recuperación de ADN de los gradientes de CsCl. Moléculas portadoras pueden ser de origen biológico como el glucógeno o de ADN de un organismo de archaea 19, o sintéticos en la naturaleza, tales como poliacrilamida lineal. La ventaja de utilizar moléculas portadoras de este tipo cuando se realiza el ADN SIP es que pueden permitir la visualización de las bandas en los gradientes de CsCl que normalmente no serían visibles y garantizar la recuperación cuantitativa de las concentraciones de ADN de baja. Éxito de la recuperación de las cantidades de nanogramos de baja de ADN de los gradientes de CsCl requiere realmente el uso de una molécula portadora 1,12. Estudios recientes han indicado que las moléculas de transporte obtenidos a partir de fuentes biológicas a menudo pueden estar contaminados con ADN del organismo fuente 12 y los resultados son muy difíciles de distinguir de los patrones asociados con el marcado con 13 C de ADN (datos no mostrados). Por lo tanto se recomienda que las moléculas sintéticas como el portador de poliacrilamida lineal se utiliza para el ADN-SIP. Además, el uso de múltiples desplazamientos de amplificación (MDA) puede generar altos rendimientos fidelidad de la etiqueta de ADN para posteriores análisis molecular 13,14, a pesar de las quimeras pueden ser generados por la amplificación y se detecta en los análisis moleculares abajo 14.
Una de las aplicaciones más poderosas de la ADN-SIP que aún no se ha explotado plenamente el potencial de recuperación de ADN de miembros de la comunidad activa para el análisis de la biblioteca metagenómica. Esperamos que los grandes avances en el descubrimiento de la enzima será el resultado de la integración de los isótopos estables de sondeo en los actuales estudios de metagenómica de diversos ambientes terrestres y acuáticos. El protocolo de visualizar aquí se produce la etiqueta de ADN de suficiente calidad para estas aplicaciones basada en el descubrimiento.
Divulgaciones
Agradecimientos
Este trabajo fue apoyado por el Proyecto Estratégico de Subvenciones y descubrimiento de JDN de las Ciencias Naturales e Ingeniería de Investigación de Canadá (NSERC).
Materiales
| Name | Company | Catalog Number | Comments | |
| Bromophenol Blue | Reagent | Fisher Scientific | BP115-25 | |
| Cesium chloride | Reagent | Fisher Scientific | BP210-500 | |
| Ethanol, reagent grade | Reagent | Sigma-Aldrich | 652261 | |
| Ethidium bromide | Reagent | Sigma-Aldrich | E1510 | |
| Hydrochloric acid | Reagent | Fisher Scientific | 351285212 | |
| Linear polyacrylamide | Reagent | Applichem | A6587 | |
| Polyethylene Glycol 6000 | Reagent | VWR international | CAPX1286L-4 | |
| Potassium Chloride | Reagent | Fisher Scientific | AC42409-0010 | |
| Sodium Chloride | Reagent | Fisher Scientific | S2711 | |
| Sodium Hydroxide pellets | Reagent | Fisher Scientific | S3181 | |
| Tris base | Reagent | Fisher Scientific | BP1521 | |
| Dark Reader | Equipment | Clare Chemical | DR46B | |
| Microcentrifuge | Equipment | Eppendorf | 5424 000.410 | |
| Nanodrop 2000 | Equipment | Fisher Scientific | 361013650 | |
| Infusion pump | Equipment | Braintree Scientific, Inc. | N/A | Model Number: BSP See www.braintreesci.com for ordering details. |
| Tube sealer | Equipment | Beckman Coulter Inc. | 358312 | |
| Ultracentrifuge | Equipment | Beckman Coulter Inc. | ||
| Ultracentrifuge rotor | Equipment | Beckman Coulter Inc. | 362754 | |
| Ultraviolet light source | Equipment | UVP Inc. | 95-0017-09 | Any UV source will suffice |
| Ultraviolet light face shield | Equipment | Fisher Scientific | 114051C | |
| Butyl rubber stoppers, gray | Material | Sigma-Aldrich | 27232 | |
| Centrifuge tubes | Material | Beckman Coulter Inc. | 342412 | |
| Hypodermic needle, 23 gauge, 2” length | Material | BD Biosciences | 305145 | |
| Microfuge tubes, 1.5 mL | Material | DiaMed | AD151-N500 | |
| Open center seals, 20 mm diameter | Material | Sigma-Aldrich | 27230-U | |
| Pasteur pipettes, glass | Material | Fisher Scientific | 13-678-6C | |
| Pipet tips | Material | DiaMed | BPS340-1000 | Catalogue number is for 200 μl tips. 10 or 20 μl tips may be purchased from the same source |
| Pump tubing 1.5 mm bore x 1.5 mm wall | Material | Appleton Woods | ||
| Screw-cap tubes, 15 mL | Material | DiaMed | AD15MLP-S | |
| Serum vials, 125 mL volume | Material | Sigma-Aldrich | Z114014 | |
| Syringe, 60 mL | Material | BD Biosciences | 309653 |
Referencias
- Neufeld, J. D. DNA stable-isotope probing. Nat. Protocols. 2, 860-866 (2007).
- Neufeld, J. D., Boden, R., Moussard, H., Schäfer, H., Murrell, J. C. Substrate-specific clades of active marine methylotrophs associated with a phytoplankton bloom in a temperate coastal environment. Appl. Environ. Microbiol. 74, 7321-7328 (2009).
- Nercessian, O., Noyes, E., Kalyuzhnaya, M. G., Lidstrom, M. E., Chistoserdova, L. Bacterial populations active in metabolism of C1 compounds in the sediment of Lake Washington, a freshwater lake. Appl. Environ. Microbiol. 71, 6885-6899 (2005).
- Padmanabhan, P. Respiration of 13C-labelled substrates added to soil in the field and subsequent 16S rRNA gene analysis of 13C-labelled soil DNA. Appl. Environ. Microbiol. 69, 1614-1622 (2003).
- Bernard, L. Dynamics and identification of soil microbial populations actively assimilating carbon from 13C-labelled wheat residue as estimated by DNA- and RNA-SIP techniques. Environ. Microbiol. 9, 752-764 (2007).
- Haichar, elZ. ahar, F, Identification of cellulolytic bacteria in soil by stable isotope probing. Environ. Microbiol. 9, 625-634 (2007).
- Addison, S., McDonald, I., Lloyd-Jones, G. Stable isotope probing: Technical considerations when resolving 15N-labelled RNA in gradients. J. Microbiol. Meth. 80, 70-75 (2009).
- Buckley, D. H., Huangyutitham, V., Hsu, S. -F., Nelson, T. A. Stable isotope probing with 15N achieved by disentangling the effects of genome G + C content and isotope enrichment on DNA density. Appl. Environ. Microbiol. 73, 3189-3195 (2007).
- Schwartz, E. Characterization of growing microorganisms in soil by stable isotope probing with H218O. Appl. Environ. Microbiol. 73, 2541-2546 (2007).
- Neufeld, J. D., Dumont, M. G., Vohra, J., Murrell, J. C. Methodological considerations for the use of stable isotope probing in microbial ecology. Microb. Ecol. 53, 435-442 (2007).
- Martineau, C., Whyte, L., Greer, C. Development of a SYBR safe technique for the sensitive detection of DNA in cesium chloride density gradients for stable isotope probing assays. J. Microbiol. Meth. 73, 199-202 (2008).
- Bartram, A. K., Poon, C., Neufeld, J. D. Nucleic acid contamination of glycogen used in nucleic acid precipitation and assessment of linear polyacrylamide as an alternative co-precipitant. Biotechniques. 47, 1019-1022 (2009).
- Chen, Y. Revealing the uncultivated majority: combining DNA stable-isotope probing, multiple displacement amplification and metagenomic analyses of uncultivated Methylocystis in acidic peatlands. Environ. Microbiol. 10, 2609-2622 (2008).
- Neufeld, J. D., Chen, Y., Dumont, M. G., Murrell, J. C. Marine methylotrophs revealed by stable-isotope probing, multiple displacement amplification and metagenomics. Environ. Microbiol. 10, 1526-1535 (2008).
- Kalyuzhnaya, M. High-resolution metagenomics targets specific functional types in complex microbial communities. Nat. Biotechnol. 26, 1029-1034 (2008).
- Binga, E. K., Lasken, R. S., Neufeld, J. D. Something from (almost) nothing: the impact of multiple displacement amplification on microbial ecology. ISME J. 2, 233-241 (2008).
- Green, S. J., Leigh, M. B., Neufeld, J. D. Microbiology of Hydrocarbon and Lipid Microbiology. Timmis, K. N. , Springer-Verlag. Berlin Heidelberg. 4137-4158 (2010).
- Neufeld, J. D., Wagner, M., Murrell, J. C. Who eats what, where and when? Isotope-labelling experiments are coming of age. ISME J. 1, 103-110 (2007).
- Gallagher, E., McGuinness, L., Phelps, C., Young, L. Y., Kerkhof, L. J. DNA shortens the incubation time needed to detect benzoate-utilizing denitrifying bacteria by stable-isotope probing. Appl. Environ. Microbiol. 71, 5192-5196 Forthcoming.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados