Method Article
In vitro Chemische Kartierung von G-Quadruplex-DNA-Strukturen durch Bis-3-Chlorpiperidine
In diesem Artikel
Zusammenfassung
Bis-3-Chloropiperidine (B-CePs) sind nützliche chemische Sonden, um G-Quadruplex-Strukturen in DNA-Matrizen in vitro zu identifizieren und zu charakterisieren. Dieses Protokoll beschreibt das Verfahren zur Durchführung von Sondierungsreaktionen mit B-CePs und zur Auflösung von Reaktionsprodukten durch hochauflösende Polyacrylamid-Gelelektrophorese.
Zusammenfassung
G-Quadruplexe (G4s) sind biologisch relevante, nicht-kanonische DNA-Strukturen, die eine wichtige Rolle bei der Genexpression und bei Krankheiten spielen und wichtige therapeutische Ziele darstellen. Für die in vitro Charakterisierung von DNA innerhalb potenzieller G-Quadruplex-bildender Sequenzen (PQS) werden zugängliche Methoden benötigt. B-CePs sind eine Klasse von Alkylierungsmitteln, die sich als nützliche chemische Sonden für die Untersuchung der Struktur höherer Ordnung von Nukleinsäuren erwiesen haben. In dieser Arbeit wird ein neuer chemischer Mapping-Assay beschrieben, der die spezifische Reaktivität von B-CePs mit dem N7 von Guaninen ausnutzt, gefolgt von einer direkten Strangspaltung an den alkylierten Gs.
Um G4-Falten von ungefalteten DNA-Formen zu unterscheiden, verwenden wir B-CeP 1, um das Thrombin-bindende Aptamer (TBA) zu untersuchen, eine 15-mer-DNA, die in der Lage ist, die G4-Anordnung anzunehmen. Die Reaktion von B-CeP-antwortenden Guaninen mit B-CeP1 führt zu Produkten, die durch hochauflösende Polyacrylamid-Gelelektrophorese (PAGE) auf Einzelnukleotidebene aufgelöst werden können, indem einzelne Alkylierungsaddukte und DNA-Strangspaltung an den alkylierten Guaninen lokalisiert werden. Die Kartierung mit B-CePs ist ein einfaches und leistungsfähiges Werkzeug für die In-vitro-Charakterisierung von G-Quadruplex-bildenden DNA-Sequenzen, das die genaue Lokalisierung von Guaninen ermöglicht, die an der Bildung von G-Tetrades beteiligt sind.
Einleitung
Neben der typischen Watson-Crick-Doppelhelix können Nukleinsäuren aufgrund ihrer Guanin-reichen Sequenzen verschiedene Sekundärstrukturen annehmen, wie z.B. die alternative G-Quadruplex (G4)-Form. Die G4-Struktur basiert auf der Bildung von planaren Tetrameren, sogenannten G-Tetraden, in denen vier Guanine durch Hoogsteen-Wasserstoffbrückenbindungen wechselwirken. G-Tetraden werden durch monovalente Kationen, die im Zentrum des Guaninkerns koordiniert sind, gestapelt und weiter stabilisiert (Abbildung 1)1.
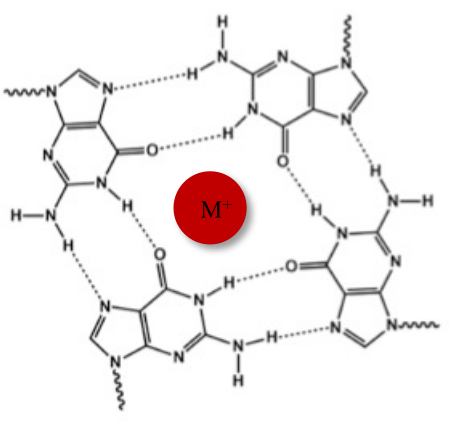
Abbildung 1: Schematische Darstellung einer G-Quadruplex-Struktur. (A) Schematische Darstellung einer G-Tetrade. Das planare Array wird durch Hoogsteen-Basenpaarung und durch ein zentrales Kation (M+) stabilisiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Sequenzen mit vier oder mehr Durchläufen von mindestens zwei aufeinanderfolgenden Guanin-Nukleotiden sind potentielle G-Quadruplex-bildende Sequenzen (PQS), die sich in G-Quadruplex-Strukturen falten können. PQS befinden sich in vielen verschiedenen zellulären Kontexten, wie z.B. an Telomeren, Genpromotoren, ribosomaler DNA und Rekombinationsstellen, und sind an der Regulation vieler biologischer Prozesse beteiligt2. Daher ist die Identifizierung und experimentelle Validierung von G4s im menschlichen Genom, die derzeit hauptsächlich mit computergestützten Werkzeugen durchgeführt wird, ein biologisch relevantes Thema3. Um computergestützte Vorhersagen zu unterstützen oder unvorhergesehene G4-Strukturen zu erkennen, wird hier eine auf chemischer Kartierung basierende Methode zur Identifizierung der G4-Bildung in einem DNA-Template gezeigt, die die präzise Identifizierung von Guaninen ermöglicht, die die G-Tetradenstruktur bilden.
Der beschriebene chemische Mapping-Assay nutzt die unterschiedliche Reaktivität von Bis-3-Chlorpyperidinen (B-CePs) mit Guaninen nach der Bildung von G4-Strukturen. Aufgrund ihrer hohen Reaktivität mit Nukleophilen 4,5,6,7,8,9 sind B-CePs Nukleinsäure-Alkylanzien mit der Fähigkeit, sehr effizient mit der N7-Position von Guanin-Nukleotiden10 zu reagieren. Auf die Alkylierung folgt die Depurinierung und die Strangspaltung in einzel- und doppelsträngigen DNA-Konstrukten. Im Gegensatz dazu sind Guanine, die an der Bildung der G-Tetraden in G4-Anordnungen beteiligt sind, unempfindlich gegen die B-CeP-Alkylierung, da die N7-Positionvon Guanin an den Hoogsteen-Wasserstoffbrückenbindungen beteiligt ist. Diese spezifische Reaktivität von B-CePs ermöglicht nicht nur den Nachweis von G4-Strukturen, sondern auch die Identifizierung der Guanine, die die Tetrade bilden, da sie aus ihrem relativen Schutz vor Alkylierung im Vergleich zu Guaninen in einzel- und doppelsträngiger DNA abgeleitet werden können.
Das chemische Kartierungsprotokoll wird hier unter Verwendung von B-CeP 1 (Abbildung 2A) als Sonde für die Charakterisierung von Thrombin-bindendem Aptamer (TBA) vorgestellt, einer 15-mer-DNA, die in der Lage ist, die G4-Anordnung in Gegenwart von Kaliumkationen anzunehmen11,12. Die G4-Anordnung von TBA (G4-TBA) wird direkt mit zwei Kontrollen verglichen, nämlich TBA in der einzelsträngigen Form (ssTBA) und TBA, die zu ihrer komplementären Sequenz getempert wurde, um das doppelsträngige Konstrukt (dsTBA) zu bilden (Tabelle 1). Die Produkte der Sondierungsreaktionen werden durch hochauflösende Polyacrylamid-Gelelektrophorese (PAGE) auf Einzelnukleotidebene aufgelöst, indem einzelne Alkylierungsaddukte und DNA-Strangspaltungen an den alkylierten Guaninen lokalisiert werden. Die Visualisierung auf dem Gel wird durch Konjugation des TBA-Oligonukleotids mit einem Fluorophor an seinem 3'-Ende ermöglicht (Tabelle 1). Dieses Protokoll zeigt, wie TBA in seinen verschiedenen Konformationen (G4 und Kontrollen) gefaltet wird und wie Sondierungsreaktionen mit B-CePs gefolgt von PAGE durchgeführt werden.
Protokoll
1. Nukleinsäure- und chemische Sondenvorbereitung
- Nukleinsäuren
HINWEIS: Das Oligonukleotid mit der Bezeichnung "TBA" ist die 15-mer-DNA-Sequenz 5'-GGT-TGG-TGT-GGT-TGG-3', die am 3'-Ende durch das Fluorophor 5-Carboxyfluorescein (FAM) markiert wird, um die Visualisierung auf dem Gel zu ermöglichen. Das unmarkierte Oligonukleotid "cTBA" ist seine DNA-komplementäre Sequenz 5'-CCA-ACC-ACA-CCA-ACC-3'. TBA und cTBA werden verwendet, um die drei verschiedenen Strukturen zu erhalten, wie in Tabelle 1 dargestellt.- Autoklavenspitzen und 0,5-ml-Röhrchen, um sterile Einwegartikel zu erhalten und Kontaminationen zu vermeiden.
- Bereiten Sie Stammlösungen vor, in denen jedes Oligonukleotid in Reinstwasser auf eine Endkonzentration von 100 μM gelöst wird. Bestimmen Sie die genaue Oligonukleotidkonzentration mit einem UV-Vis-Spektralphotometer (UV-Vis) unter Verwendung des vom Hersteller angegebenen Extinktionskoeffizienten bei 260 nm.
ANMERKUNG: Extinktionskoeffizienten: 164.300 M-1 cm-1 und 138.600 M-1 cm-1 wurden für TBA bzw. cTBA verwendet. - TBA- und cTBA-Stammlösungen bei -20 °C lagern (monatelang unter diesen Bedingungen).
- Verbindung B-CeP 1
HINWEIS: Die Verbindung B-CeP 1 wird wie zuvor berichtetsynthetisiert 6.- Bereiten Sie die B-CeP 1 Stammlösung bei ~10 mM vor. Wiegen Sie ~1 mg der lyophilisierten Verbindung mit einer Analysenwaage in einem Abzug und lösen Sie sie in 100% Dimethylsulfoxid (DMSO).
- Berechnen Sie die genaue Wirkstoffkonzentration auf der Grundlage der tatsächlich verwendeten Menge an Verbindung und DMSO (d = 1,1 g/cm3).
HINWEIS: Behandeln Sie die Verbindung immer mit Handschuhen (sowohl bei Gefriertrocknung als auch beim Auflösen in DMSO)13,14.
Tabelle 1: In diesem Protokoll verwendete Oligonukleotidstrukturen. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
2. Faltung von Nukleinsäurekonstrukten
- Vorbereitung von Puffern
- Es wird eine BPE-Pufferlösung (Biphosphat-Ethylendiamintetraessigsäure [EDTA], 5x: 2 mM NaH2PO 4, 6 mM Na2HPO4, 1 mMNa2EDTA, pH7,4) und eine Lösung von 500 mM KCl in Reinstwasser hergestellt. Filtern Sie die Lösungen durch Filter mit einer Porengröße von 0,22 μm.
HINWEIS: Die besten Ergebnisse erzielen Sie, wenn Sie frisch zubereitete Lösungen verwenden. BPE-Puffer kann bei 4 °C bis zu 15 Tage gelagert werden.
- Es wird eine BPE-Pufferlösung (Biphosphat-Ethylendiamintetraessigsäure [EDTA], 5x: 2 mM NaH2PO 4, 6 mM Na2HPO4, 1 mMNa2EDTA, pH7,4) und eine Lösung von 500 mM KCl in Reinstwasser hergestellt. Filtern Sie die Lösungen durch Filter mit einer Porengröße von 0,22 μm.
- Falten von G4-TBA-, ssTBA- und dsTBA-Proben im Heat-Refolding-Verfahren
HINWEIS: Kaliumkationen sind notwendig, um die G4-Struktur (G4-TBA) zu falten. Kaliumkationen dürfen nicht in die Faltungslösung der Kontrollen ssTBA und dsTBA gegeben werden.- Bereiten Sie 40 μl einer 4 μM-Lösung von G4-TBA in 1x BPE und 100 mM KCl vor. Denaturieren Sie die Oligonukleotid-G4-TBA-Lösung, indem Sie das Röhrchen 5 Minuten lang auf 95 °C erhitzen und langsam auf Raumtemperatur (RT) abkühlen, damit sich die TBA in G-Quadruplexe falten kann.
- Bereiten Sie 40 μl einer 4 μM Lösung von ssTBA in 1x BPE vor. Führen Sie die in Schritt 2.2.1 beschriebene Hitzerückfaltung durch, um TBA in seiner einzelsträngigen Form zu falten.
- Bereiten Sie 40 μl einer 4 μM-Lösung von dsTBA vor, indem Sie äquimolare Mengen von TBA und cTBA in 1x BPE mischen. Führen Sie das oben beschriebene Verfahren der Wärmerückfaltung (Schritt 2.2.1) durch, damit TBA zu seiner komplementären Sequenz cTBA glüht und die doppelsträngige Form von TBA bildet.
HINWEIS: Das endgültige Volumen jeder Faltlösung basiert auf der Anzahl der Proben für die Sondierungsreaktionen, wobei zu berücksichtigen ist, dass für jede Probe 5 μl 4 μM-Lösung benötigt werden. Bereiten Sie von jeder Lösung ein wenig überschüssiges Volumen vor, um Pipettierfehler zu vermeiden.
3. Sondierungsreaktionen
HINWEIS: Sondierungsreaktionen müssen unmittelbar nach dem Hitzerückfaltungsverfahren durchgeführt werden.
- Wenn die Faltlösungen von G4-TBA, ssTBA und dsTBA auf RT abgekühlt sind, wird eine Kurzschleuderzentrifugation (7.000 × g für 5-8 s bei RT) durchgeführt und die Sondierungsreaktionen gestartet.
- Bereiten Sie 21 leere 0,5-ml-Autoklavierröhrchen vor. Organisieren Sie sie in drei Sätzen mit jeweils sieben Röhrchen im Rack für Laborproben, wie in Tabelle 2 angegeben.
HINWEIS: Jeder Spaltensatz entspricht den drei verschiedenen TBA-Faltungsbedingungen G4-TBA, ssTBA und dsTBA. Jede Reihe entspricht drei verschiedenen Inkubationszeiten. Jede Zelle innerhalb der Säule entspricht der endgültigen B-CeP 1-Sondenkonzentration (Tabelle 2). Achten Sie darauf, die Röhrchen deutlich zu beschriften. - Geben Sie 3 μl Reinstwasser in jedes Röhrchen.
- Geben Sie 5 μl gefaltetes G4-TBA in jedes Röhrchen des ersten Satzes (Schritt 3.2). Geben Sie 5 μl gefaltetes ssTBA in jedes Röhrchen des zweiten Sets. Geben Sie 5 μl gefaltetes dsTBA in jedes Röhrchen des dritten Sets.
- Verdünnen Sie die B-CeP 1 Stammlösung auf 250 μM und 25 μM in Reinstwasser.
HINWEIS: Verdünnungen der chemischen B-CeP 1-Sonde müssen frisch zubereitet und sofort mit dem DNA-Substrat umgesetzt werden, um konkurrierende Reaktionen mit Wasser zu vermeiden. - Die Proben werden mit 2 μl der entsprechenden B-CeP 1-Verdünnung (25 μM und 250 μM) versetzt. Die Verbindung wird in den drei Kontrollproben (C) durch Reinstwasser ersetzt, um die unterschiedlich gefalteten TBAs in Abwesenheit der Verbindung zu analysieren. Alle Proben werden bei 37 °C inkubiert.
- Nach 1 h, 4 h und 15 h Inkubation wird die Reaktion gestoppt, indem die Röhrchen bis zum nächsten Schritt bei -20 °C platziert werden.
HINWEIS: Die Proben können unter diesen Bedingungen einige Tage gelagert werden. - Trocknen Sie die Proben in einer Vakuumzentrifuge.
HINWEIS: Die getrockneten Proben können wochenlang bei -20 °C gelagert werden, bevor mit der PAGE-Analyse fortgefahren wird.
Tabelle 2: Proben für die Sondierungsreaktionen (Strukturen, Sondenkonzentrationen und Inkubationszeit). Jeder Spaltensatz entspricht den drei verschiedenen TBA-Faltungsbedingungen (G4-TBA, ssTBA und dsTBA). Jede Reihe entspricht drei verschiedenen Inkubationszeiten (1, 4, 15 h). Jede Zelle innerhalb der Säule entspricht der endgültigen B-CeP 1-Sondenkonzentration (5 oder 50 μM). Die Kontrolle (C) für jeden Satz entspricht einer Probe der unterschiedlich gefalteten TBAs, die für die längere Zeit (15 h) in Abwesenheit von Verbindung inkubiert wurden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
4. Hochauflösende SEITE
- Herstellung der denaturierenden Polyacrylamidlösung
HINWEIS: Bereiten Sie im Voraus 500 ml 20%ige denaturierende Polyacrylamid-Gellösung vor. Etwa 80 ml dieser Lösung werden für jedes Experiment verwendet. Verwenden Sie eine Braunglasflasche oder decken Sie eine Glasflasche mit Aluminiumfolie ab, um die Lösung bei RT aufzubewahren.
ACHTUNG: Polyacrylamid ist neurotoxisch. Tragen Sie bei allen Schritten der Gelvorbereitung und des Gießens Handschuhe und einen Laborkittel. Entsorgen Sie polymerisiertes Acrylamid in einem geeigneten Karton für kontaminierte Materialien.- 210 g Harnstoff werden in einem 1-Liter-Becherglas eingewogen. 250 ml 40%ige Acrylamid/Bisacrylamid-Lösung (19/1) und 50 ml 10x FSME (890 mM Tris-HCl, 890 mM Borat, 20 mM EDTA, pH 8) zugeben.
- Stellen Sie das Becherglas auf eine Rührplatte und mischen Sie die Lösung mit einem Rührstab. Decken Sie das Becherglas während des Mischens mit Aluminiumfolie ab, um Spritzer und Verunreinigungen zu vermeiden.
- Mischen Sie die Lösung, bis sich der Harnstoff vollständig aufgelöst hat und die Lösung klar ist.
HINWEIS: Dieser Schritt kann viele Stunden dauern. Um die Auflösung von Harnstoff zu fördern, fügen Sie ein kleines Aliquot Wasser hinzu, ohne das gewünschte Endvolumen zu überschreiten. - Entfernen Sie den Rührstab. Gießen Sie die Lösung in einen Zylinder und fügen Sie Wasser zu einem genauen Endvolumen von 500 ml hinzu.
- Aufbau der Gel-Apparatur
- Reinigen Sie zwei Platten (eine gekerbte und eine ungekerbte) mit 70%igem Ethanol, lassen Sie sie trocknen und behandeln Sie die Platten dann mit einer Dimethyldichlorsilanlösung.
HINWEIS: Die Silanisierung kann übersprungen werden, obwohl sie die Freisetzung des Gels von einer der Platten unterstützt, wenn das Gel-Sandwich auseinandergenommen wird.
VORSICHT: Fassen Sie die Silanisierungslösung mit Handschuhen an und führen Sie die Plattenbehandlung mit dieser Lösung in einem Abzug durch. - Platzieren Sie die 0,4-mm-Abstandshalter entlang der langen Kanten der längeren Platte, legen Sie die kurze Platte aufeinander und richten Sie die beiden Platten unten aus.
- Lege mehrere Lagen Papierklebeband entlang aller Kanten außer der Oberseite.
- Um Leckagen während des Gießens zu vermeiden, fügen Sie eine zusätzliche Schicht Klebeband auf die Unterseite des Gels auf.
- Befestigen Sie die Seiten des Glassandwiches mit sauberen Klammern gemäß den Anweisungen des Lieferanten (verschiedene Lieferanten verwenden leicht unterschiedliche Apparaturen, Sandwichklemmen und Dichtungen).
- Reinigen Sie zwei Platten (eine gekerbte und eine ungekerbte) mit 70%igem Ethanol, lassen Sie sie trocknen und behandeln Sie die Platten dann mit einer Dimethyldichlorsilanlösung.
- Gießen des Gels
HINWEIS: Gießen Sie das Gel bei RT (25 °C), da die Polyacrylamidpolymerisation temperaturempfindlich ist.- Unmittelbar vor Gebrauch werden 80 ml der zuvor hergestellten denaturierenden Polyacrylamidlösung (Schritt 4.1), 450 μl einer 10%igen m/V-Ammoniumpersulfatlösung (APS) und 45 μl Tetramethylethylendiamin (TEMED) in ein Becherglas gegeben.
- Mischen Sie die Lösung und gießen Sie sie mit einer 50-ml-Spritze schnell zwischen die Glasplatten. Führen Sie den Kamm mit der gewünschten Anzahl von Vertiefungen zwischen die Glasplatten ein, um Blasen zu vermeiden. Füge bei Bedarf eine Gellösung hinzu, um das Sandwich vollständig zu füllen. Setzen Sie vier Klammern auf den Kamm, um sie nach unten zu drücken und eine gleichmäßige Verteilung der Vertiefungen zu ermöglichen.
- Lassen Sie das Gel mindestens 45 Minuten lang polymerisieren.
- Das Gel laufen lassen
- Entfernen Sie nach der Polymerisation alle Klammern und Papierbandschichten. Entfernen Sie den Kamm langsam und spülen Sie die Vertiefungen gründlich mit destilliertem Wasser aus.
- Befolgen Sie die Anweisungen des jeweiligen Lieferanten, um das Gel-Sandwich korrekt in das vertikale Gelelektrophoresegerät zu legen.
- FSME-Laufpuffer (1x: 89 mM Tris-HCl, 89 mM Borat, 2 mM EDTA, pH 8) in deionisiertem Wasser vorbereiten und sowohl den oberen als auch den unteren Behälter mit dem Puffer füllen.
- Erwärmen Sie die Platten, indem Sie einen Vorlauf der Gelelektrophorese für mindestens 30 Minuten bei 50 W durchführen.
- Denaturierungsgel-Ladepuffer (DGLB: 1 M Tris-HCl, 80 % Formamid, 50 % Glycerin, 0,05 % Bromphenolblau) in Reinstwasser herstellen.
HINWEIS: GLB hilft dabei, die Bewegung der Oligonukleotidproben in das Gelsystem zu verfolgen und ermöglicht es, die Proben in die Vertiefungen des Gels zu laden. Das Vorhandensein des Denaturierungsmittels Formamid ermöglicht es DNA-Spezies, sich nach Größe zu trennen, selbst in einem nicht denaturierenden PAGE. - Die getrockneten Proben (Proben aus Schritt 3.8) werden in 5 μl DGLB resuspendiert.
- Reinigen Sie vor dem Laden der Proben die Vertiefungen mit einer kleinen Spritze und dem TBE-Puffer in der oberen Pufferkammer, um den Harnstoff aus den Vertiefungen zu entfernen.
HINWEIS: Wiederholen Sie diesen Schritt mehrmals, um die Vertiefungen genau zu reinigen und so schwer zu interpretierende Bänder zu vermeiden. - Laden Sie die Proben in die sauberen Vertiefungen und notieren Sie sich die Reihenfolge der Beladung.
- Lassen Sie die Gelelektrophorese 2 Stunden lang bei 50 W laufen, oder zumindest so lange, bis der Bromphenolblaufarbstoff 2/3 des Gels heruntergelaufen ist.
- Bildgebung des Gels
- Schalten Sie nach der Elektrophorese die Stromversorgung aus, entfernen Sie das Glassandwich und reinigen Sie die Gläser.
- Detektieren Sie die Fluoreszenz der FAM-markierten Oligonukleotidbanden durch Scannen mit einem Gel-Imager.
Ergebnisse
Abbildung 2 zeigt ein repräsentatives Ergebnis eines durchgeführten chemischen Mapping-Assays, wie im Protokoll beschrieben, mit B-CeP 1 auf dem TBA-Oligonukleotid, das in drei verschiedenen Strukturen gefaltet ist. Die G-Quadruplex-Anordnung von TBA (G4-TBA) wurde durch Faltung des Oligonukleotids in BPE und in Gegenwart des K+-Kations erhalten, während die einzelsträngige Form derselben TBA-Sequenz (ssTBA) in Abwesenheit von Kalium gefaltet wurde. Das doppelsträngige Konstrukt (dsTBA) wurde durch Annealing von TBA zu seiner komplementären Sequenz im BPE-Puffer hergestellt. Entweder eine Endkonzentration von 5 oder 50 μM von B-CeP 1 wurde mit 2 μM Proben von G4-TBA (linke Spuren von Abbildung 2B), ssTBA (mittlere Spuren von Abbildung 2B) und dsTBA (rechte Spuren von Abbildung 2B) für 1 h, 4 h und 15 h umgesetzt. Zusätzliche Kontrollen sind die DNA-Konstrukte, die nicht mit der Sonde reagiert haben. Die Zirkulardichroismus-Analyse (CD) der drei verschiedenen TBA-Faltungen bestätigte ihre korrekte Struktur (Ergänzende Abbildung S1). Das CD-Spektrum von dsTBA war in der Tat durch ein positives langwelliges Band bei ~260-280 nm und ein negatives Band bei ~245 nm15 charakterisiert, was mit einer DNA-Helix in ihrer B-Form übereinstimmt. Es wurde festgestellt, dass die ssTBA in Abwesenheit von K+-Ionen schlecht strukturiert ist, während sich G4-TBA in Gegenwart von K+-Ionen in ein antiparalleles G4 faltet, das durch eine negative Bande bei 260 nm und eine positive Bande bei 290 nm gekennzeichnet ist15.
Die hochauflösenden PAGE-Ergebnisse, die in Abbildung 2B dargestellt sind, bieten einen umfassenden Überblick über die zeit- und konzentrationsabhängigen Reaktionen von B-CeP 1 gegenüber G4-TBA, ssTBA und dsTBA und zeigen deutlich ein deutlich anderes Alkylierungsmuster für das G4-TBA im Vergleich zu dem, was für die ssTBA- und dsTBA-Konstrukte beobachtet wurde. Banden, die mit geringerer Mobilität migrieren als die unbehandelten Proben (C), stimmen mit der Alkylierung des DNA-Substrats überein. Im Falle des G4-TBA-Substrats ist nur ein Alkylierungsaddukt eindeutig zu beobachten, da nur ein Guanin (G8) für die Alkylierung zur Verfügung steht (Nummerierung der Guanine in Abbildung 2C). Im Gegensatz dazu werden mehrere Addukte unterschieden, wenn das Substrat entweder ssTBA oder dsTBA ist, wobei alle Guanine, die die N7-Position besitzen, zur Sondierung frei sind. Schneller wandernde Banden werden stattdessen auf die Produkte der Strangspaltung zurückgeführt. Im Falle des G4-TBA-Substrats sind die Guanine (G1, G2, G5, G6, G10, G11, G14, G15), die an G-Quadruplex-Tetraden beteiligt sind, unempfindlich gegen Alkylierung, während G8 durch Spaltung als folgenden Schritt der Alkylierung beeinflusst wird. In den ssTBA- und dsTBA-Konstrukten sind alle Guanine anfällig für Spaltung, wie die zahlreichen detektierten Banden zeigen, die bei längeren Inkubationszeiten zunehmend intensiver werden.
Dieses Protokoll ermöglicht die Unterscheidung zwischen den Guaninen, die an der Bildung der G-Quadruplex-Tetraden beteiligt sind, und den Guaninen, die nicht an einer solchen Anordnung beteiligt sind, und klärt so die G4-TBA-Struktur auf (Abbildung 2C).
Ein möglicher Nachteil des Experiments ist das Vorhandensein unerwünschter Nukleophile in der Reaktionsumgebung. Zum Beispiel konkurrieren Nukleophile, die sich von Salzen im Reaktionspuffer ableiten, mit der DNA um die Reaktivität mit B-CePs, was zu einer ineffizienten Reaktion der Sonde mit dem Substrat führt. Abbildung 3 zeigt die Ergebnisse der in Tris-Puffer durchgeführten Sondierungsreaktionen, die die nukleophile Tris-Base enthalten. B-CeP 1 (5 und 50 μM) wird mit 2 μM Proben von G4-TBA (linke Spuren in Abbildung 3), ssTBA (mittlere Spuren in Abbildung 3) und dsTBA (rechte Spuren von Abbildung 3) für 1 h, 4 h, 15 h und 24 h umgesetzt. Unter diesen Bedingungen ist die Reaktivität der Sonde gegenüber allen drei DNA-Substraten deutlich verringert. Bei den G4-TBA-Proben können wir nur die Bildung von Addukten beobachten, ohne eine Strangspaltung zu erkennen. In den Fällen von ssTBA und dsTBA ist die DNA-Fragmentierung erst nach 24 h vergleichbar mit derjenigen, die nach 15 h Inkubation in Abbildung 2B beobachtet wurde. Die unerwünschte konkurrierende Reaktivität von B-CePs mit Tris führt zu einem Mangel an Nukleinsäure-Strukturinformationen.
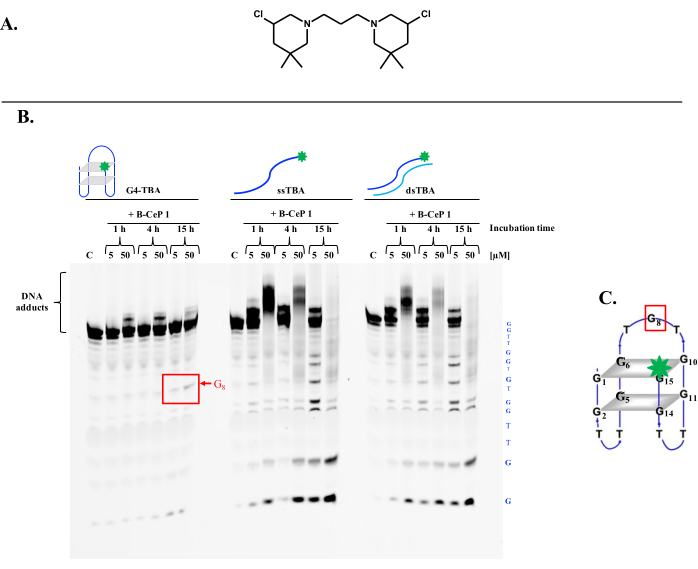
Abbildung 2: Chemische Kartierung einer TBA-G-Quadruplex-Struktur durch B-CeP 1 in BPE-Puffer. (A) Chemische Struktur von B-CeP 1. (B) Zeit- und Konzentrationsabhängigkeit der Sondierungsreaktionen zwischen B-CeP 1 und TBA im BPE-Puffer. Die Aliquots von TBA wurden unter drei verschiedenen Bedingungen gefaltet, um die G-Quadruplex-Anordnung (G4-TBA), das einzelsträngige Oligonukleotid (ssTBA) und TBA zu seiner komplementären Sequenz zu geglühen, um das doppelsträngige Konstrukt (dsTBA) zu bilden. Das TBA-Oligonukleotid wurde am 3'-Ende mit einem Fluorophor (FAM) markiert, um eine Visualisierung auf dem Gelsystem zu ermöglichen. Aliquots (2 μM) von G4-TBA, ssTBA und dsTBA wurden mit B-CeP 1 in einer Endkonzentration von entweder 5 oder 50 μM in BPE-Puffer behandelt und bei 37 °C für die angegebene Zeit inkubiert. Das Ergebnis der Sondierungsreaktionen wurde mittels hochauflösender denaturierender PAGE (20% Polyacrylamid [PAA], 7 M Harnstoff, 1x FSME) analysiert. "C" gibt die Kontrollstichprobe jedes TBA-Konstrukts an. (C) Karikatur der G-Quadruplex-Anordnung von G4-TBA und Nummerierung der Guanine in der TBA-Sequenz. Das einzige Guanin, das nicht an der G4-Struktur beteiligt war, also mit B-CeP 1 reagierte, ist rot markiert. Der grüne Stern steht für den Fluorophor FAM. Abkürzungen: TBA = Thrombin-bindendes Aptamer; B-CeP 1 = Bis-3-chlorpiperidin 1; BPE = Biphosphat-EDTA; G = Guanin; G4 = Guanin-Quadruplex; ss = einzelsträngig; ds = doppelsträngig; FAM = 5-Carboxyfluorescein; PAA = Polyacrylamid; PAGE = Polyacrylamid-Gelelektrophorese; FSME = Tris-Borat-EDTA. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Ineffiziente chemische Kartierung in Gegenwart von Tris-Puffer. Zeit- und Konzentrationsabhängigkeit der Sondierungsreaktionen zwischen B-CeP 1 und TBA im Tris-Puffer. Die Aliquots von TBA wurden unter drei verschiedenen Bedingungen gefaltet, um die G-Quadruplex-Anordnung (G4-TBA), das einzelsträngige Oligonukleotid (ssTBA) und TBA zu seiner komplementären Sequenz zu geglühen, um das doppelsträngige Konstrukt (dsTBA) zu bilden. Das TBA-Oligonukleotid wurde am 3'-Ende mit einem Fluorophor (FAM) markiert, um eine Visualisierung auf dem Gelsystem zu ermöglichen. Aliquots (2 μM) von G4-TBA, ssTBA und dsTBA wurden mit B-CeP 1 in einer Endkonzentration von 5 oder 50 μM in 10 mM Tris-HCl-Puffer behandelt und für die angegebene Zeit bei 37 °C inkubiert. Das Ergebnis der Sondierungsreaktionen wurde mittels hochauflösender denaturierender PAGE (20% PAA, 7 M Harnstoff, 1x FSME) analysiert. "C" gibt die Kontrollstichprobe jedes TBA-Konstrukts an. Die Nukleophilie von Tris, die als Puffer während der Sondierungsreaktionen verwendet wird, führt zu niedrigeren Signalen und einem Verlust struktureller Informationen. Abkürzungen: TBA = Thrombin-bindendes Aptamer; B-CeP 1 = Bis-3-chlorpiperidin 1; BPE = Biphosphat-EDTA; G = Guanin; G4 = Guanin-Quadruplex; ss = einzelsträngig; ds = doppelsträngig; FAM = 5-Carboxyfluorescein; PAA = Polyacrylamid; PAGE = Polyacrylamid-Gelelektrophorese; FSME = Tris-Borat-EDTA. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung S1: CD-Spektren von G4-TBA, ssTBA und dsTBA. Nach der korrekten Faltung der drei unterschiedlichen Strukturen wurden CD-Spektren bei 25 °C aufgenommen. Das CD-Spektrum von dsTBA, das durch positive langwellige Banden bei ~260-280 nm und eine negative Bande bei ~245 nm16 gekennzeichnet ist, stimmt mit einer B-Form der DNA-Helix überein. Im Vergleich zum ssTBA, das sich in Abwesenheit von K+-Ionen als schlecht strukturiert erwies, faltet sich G4-TBA in Gegenwart von K+-Ionen in ein antiparalleles G4, das durch eine negative Bande bei 260 nm und eine positive Bande bei 290 nm gekennzeichnet ist15. Abkürzungen: TBA = Thrombin-bindendes Aptamer; B-CeP 1 = Bis-3-chlorpiperidin 1; BPE = Biphosphat-EDTA; G = Guanin; G4 = Guanin-Quadruplex; ss = einzelsträngig; DS = doppelsträngig. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
G-Quadruplexe sind Nukleinsäure-Sekundärstrukturen, die sich typischerweise innerhalb von Guanin-reichen DNA-Sequenzen falten und aufgrund ihrer Assoziation mit genetischer Kontrolle und Krankheiten wichtige Forschungsziele sind. Die chemische Kartierung durch B-CePs ist ein nützliches Protokoll für die Charakterisierung von DNA-G4s, das verwendet werden kann, um die Guaninbasen zu identifizieren, die an der Bildung von G-Tetrades unter physiologischen Salzbedingungen beteiligt sind.
Die in diesem Protokoll verwendete chemische Sonde ist B-CeP 1 (Abbildung 2A), die durch spezifische Reaktion mit dem N7 der Guanin-Nukleobasen zwischen den Guaninen, die an der Bildung der G-Quadruplex-Tetraden beteiligt sind, und den Guaninen, die nicht an einer solchen Anordnung beteiligt sind, unterscheiden kann. Tatsächlich beeinträchtigen Hoogsteen-Wasserstoffwechselwirkungen zwischen den Guaninen in G-Tetrade (Abbildung 1) die Reaktion mit der Sonde. Die Produkte von Sondierungsreaktionen werden durch hochauflösendes PAGE (Abbildung 2B) aufgelöst, ein im Labor zugängliches Verfahren, das häufig zum Nachweis von Alkylierungsaddukten und Spaltstellen in DNA-Sequenzen eingesetzt wird. Darüber hinaus ermöglicht die Verwendung einer DNA-Sequenz, die mit einem Fluorophor markiert ist, den Anwendern, das Experiment unter sicheren Bedingungen durchzuführen, wobei radioaktiv markierte Nukleinsäuren und der herkömmliche Färbeschritt mit Interkalatoren vermieden werden.
Die B-CeP 1-Sonde ist sowohl in fester Form als auch in nichtwässriger Stammlösung sehr stabil und es können nur wenige Probleme bei der Durchführung der Sondierungsreaktion auftreten. Um Nachteile im Protokoll zu vermeiden, ist es sehr wichtig, das Vorhandensein unerwünschter Nukleophile in den Sondierungslösungen zu vermeiden, um konkurrierende Reaktionen zu vermeiden, die den Titer der aktiven Sonde verringern könnten, die zur Bildung des Addukts mit der DNA zur Verfügung steht. Da die am häufigsten verwendeten Puffer Trisbase enthalten, erinnern wir den Leser daran, dass sein nukleophiles primäres Amin mit dem elektrophilen Zentrum der Sonde reagieren kann, das mit der Reaktivität der Guanine des DNA-Substrats10 konkurriert, wie in Abbildung 3 für B-CeP 1 gezeigt. Dies ist der Grund für die Durchführung der Sondierungsreaktionen in Phosphatpuffer, wie im Protokoll beschrieben. Um Reaktionen mit Wasser7 zu vermeiden, empfehlen wir außerdem, alle Verdünnungen von B-CeP 1 frisch herzustellen und sofort mit dem Nukleinsäuresubstrat umzusetzen.
Die hier beschriebene chemische Kartierung durch B-CePs ist eine geeignete Alternative zum Dimethylsulfat (DMS)-Footprinting-Protokoll, das den Mainstream-Assay für die anfängliche Charakterisierung von G4s16 darstellt. Die beiden Protokolle liefern im Wesentlichen die gleichen Informationen, nämlich die Bildung der G4-Struktur und die Identifizierung der an G4 beteiligten Guanine, unabhängig von der G4-Konformation. Das chemische Mapping mit B-CePs besitzt jedoch einen entscheidenden Vorteil in Bezug auf das DMS-Footprinting - die Einfachheit des Protokolls. Nach den Sondierungsreaktionen benötigt das hier beschriebene Protokoll keine zusätzlichen Schritte, während das DMS-Footprinting nach der Reaktion mit der Sonde eine anschließende Spaltung durch heißes Piperidin erfordert, was eine zusätzliche Probenreinigung und einen zusätzlichen Pufferaustausch impliziert16. Die Reduzierung der Anzahl der Schritte im Protokoll mit B-CePs impliziert die Verwendung kleinerer Mengen des ursprünglichen DNA-Substrats. Schließlich wird die chemische Kartierung durch B-CePs, die hier mit der TBA-Sequenz als Proof-of-Concept beschrieben wird, zu neuen Experimenten führen, die auf jede andere potenzielle G-Quadruplex-bildende Sequenz anwendbar sind.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Arbeit wurde vom Institut für Pharmazeutische und Pharmakologische Wissenschaften der Universität Padua (PRIDJ-BIRD2019) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Acrylamide/bis-acrylamide solution 40% | Applichem | A3658 | R45-46-20/21-25-36/38-43-48/23/ 24/25-62 |
| Ammonium per-sulfate (APS) | Sigma Aldrich | A7460 | |
| Analytical balance | Mettler Toledo | ||
| Autoclave | pbi international | ||
| Boric acid | Sigma Aldrich | B0252 | |
| Bromophenol blue Brilliant blue R | Sigma Aldrich | B0149 | |
| di-Sodium hydrogen phosphate dodecahydrate | Fluka | 71649 | |
| DMSO | Sigma Aldrich | 276855 | |
| DNA oligonucleotides | Integrated DNA Technologies | synthesis of custom sequences | |
| EDTA disodium | Sigma Aldrich | E5134 | |
| Formamide | Fluka | 40248 | H351-360D-373 |
| Gel imager | GE Healtcare | STORM B40 | |
| Glycerol | Sigma Aldrich | G5516 | |
| Micro tubes 0.5 mL | Sarstedt | 72.704 | |
| Potassium Chloride | Sigma Aldrich | P9541 | |
| Sequencing apparatus | Biometra | Model S2 | |
| Silanization solution I | Fluka | 85126 | H225, 314, 318, 336, 304, 400, 410 |
| Sodium phosphate monobasic | Carlo Erba | 480086 | |
| Speedvac concentrator | Thermo Scientific | Savant DNA 120 | |
| TEMED | Fluka | 87689 | R11-21/22-23-34 |
| Tris-HCl | MERCK | 1.08387.2500 | |
| Urea | Sigma Aldrich | 51456 | |
| UV-Vis spectrophotometer | Thermo Scientific | Nanodrop 1000 |
Referenzen
- Davis, J. T. G-quartets 40 years later: from 5'-GMP to molecular biology and supramolecular chemistry. Angewandte Chemie. 43 (6), 668-698 (2004).
- Varshney, D., Spiegel, J., Zyner, K., Tannahill, D., Balasubramanian, S. The regulation and functions of DNA and RNA G-quadruplexes. Nature Reviews Molecular Cell Biology. 21 (8), 459-474 (2020).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nature Biotechnology. 33 (8), 877-881 (2015).
- Zuravka, I., Sosic, A., Gatto, B., Gottlich, R. Synthesis and evaluation of a bis-3-chloropiperidine derivative incorporating an anthraquinone pharmacophore. Bioorganic & Medicinal Chemistry Letters. 25 (20), 4606-4609 (2015).
- Zuravka, I., Roesmann, R., Sosic, A., Gottlich, R., Gatto, B. Bis-3-chloropiperidines containing bridging lysine linkers: Influence of side chain structure on DNA alkylating activity. Bioorganic & Medicinal Chemistry. 23 (6), 1241-1250 (2015).
- Zuravka, I., et al. Synthesis and DNA cleavage activity of bis-3-chloropiperidines as alkylating agents. ChemMedChem. 9 (9), 2178-2185 (2014).
- Sosic, A., Gottlich, R., Fabris, D., Gatto, B. B-CePs as cross-linking probes for the investigation of RNA higher-order structure. Nucleic Acids Research. 49 (12), 6660-6672 (2021).
- Sosic, A., et al. Bis-3-chloropiperidines targeting TAR RNA as a novel strategy to impair the HIV-1 nucleocapsid protein. Molecules. 26 (7), 1874 (2021).
- Sosic, A., et al. In vitro evaluation of bis-3-chloropiperidines as RNA modulators targeting TAR and TAR-protein interaction. International Journal of Molecular Sciences. 23 (2), 582 (2022).
- Sosic, A., et al. Direct and topoisomerase II mediated DNA damage by bis-3-chloropiperidines: The importance of being an earnest G. ChemMedChem. 12 (17), 1471-1479 (2017).
- Bock, L. C., Griffin, L. C., Latham, J. A., Vermaas, E. H., Toole, J. J. Selection of single-stranded DNA molecules that bind and inhibit human thrombin. Nature. 355 (6360), 564-566 (1992).
- Paborsky, L. R., McCurdy, S. N., Griffin, L. C., Toole, J. J., Leung, L. L. The single-stranded DNA aptamer-binding site of human thrombin. The Journal of Biological Chemistry. 268 (28), 20808-20811 (1993).
- Carraro, C., et al. Behind the mirror: chirality tunes the reactivity and cytotoxicity of chloropiperidines as potential anticancer agents. ACS Medicinal Chemistry Letters. 10 (4), 552-557 (2019).
- Carraro, C., et al. Appended aromatic moieties in flexible bis-3-chloropiperidines confer tropism against pancreatic cancer cells. ChemMedChem. 16 (5), 860-868 (2021).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Onel, B., Wu, G., Sun, D., Lin, C., Yang, D. Electrophoretic mobility shift assay and dimethyl sulfate footprinting for characterization of G-quadruplexes and G-quadruplex-protein complexes. Methods in Molecular Biology. 2035, 201-222 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten