Method Article
Optimierung der Leistungsparameter des TAGGG Telomerlängen-Assays
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier beschreiben wir detailliert das Protokoll zur Quantifizierung der Telomerlänge mittels nicht-radioaktiver Chemilumineszenzdetektion, wobei der Schwerpunkt auf der Optimierung verschiedener Leistungsparameter des TAGGG-Telomerlängen-Assay-Kits liegt, wie z.B. Puffermengen und Sondenkonzentrationen.
Zusammenfassung
Telomere sind sich wiederholende Sequenzen, die an den Chromosomenenden vorhanden sind. Ihre Verkürzung ist ein charakteristisches Merkmal menschlicher Körperzellen. Die Verkürzung tritt aufgrund eines Problems mit der Endreplikation und des Fehlens des Telomerase-Enzyms auf, das für die Aufrechterhaltung der Telomerlänge verantwortlich ist. Interessanterweise verkürzen sich Telomere auch als Reaktion auf verschiedene interne physiologische Prozesse wie oxidativen Stress und Entzündungen, die durch extrazelluläre Stoffe wie Schadstoffe, Infektionserreger, Nährstoffe oder Strahlung beeinflusst werden können. Somit dient die Telomerlänge als hervorragender Biomarker für das Altern und verschiedene physiologische Gesundheitsparameter. Das TAGGG Telomerlängen-Assay-Kit wird zur Quantifizierung der durchschnittlichen Telomerlängen mit dem Telomerrestriktionsfragment (TRF)-Assay verwendet und ist hochgradig reproduzierbar. Es handelt sich jedoch um eine teure Methode, die daher nicht routinemäßig für große Stichprobenzahlen eingesetzt wird. In dieser Arbeit beschreiben wir ein detailliertes Protokoll für eine optimierte und kostengünstige Messung der Telomerlänge mittels Southern Blots oder TRF-Analyse und nicht-radioaktiver Chemilumineszenz-basierter Detektion.
Einleitung
Telomere sind die sich wiederholenden DNA-Sequenzen, die am Ende der Chromosomen vorhanden sind. Sie haben Tandemwiederholungen von TTAGGG und bewahren die Genomintegrität, indem sie das Chromosom sowohl vor dem Ausfransen als auch vor dem Endreplikationsproblem schützen, was bedeutet, dass ein Teil des 3'-Überhangs nicht von der DNA-Polymerase 1,2 repliziert werden kann. Kurze Telomere führen zu Chromosomenanomalien in den Zellen, wodurch die Zellen in einem Stadium, das als replikative Seneszenz bezeichnetwird, dauerhaft blockiert werden 3. Kurze Telomere verursachen auch eine Vielzahl anderer Probleme, wie z. B. die Dysfunktion der Mitochondrien 4,5 und die Dysfunktion der Zellen.
DNA-Telomerwiederholungen gehen verloren, wenn sich die Zelle teilt, mit einem durchschnittlichen Verlust von 25 bis 200 bp pro Jahr 6, was nach einer bestimmten Anzahl von Teilungen zu einer zellulären Seneszenz führt6. Das Altern ist mit einer höheren Häufigkeit von Komorbiditäten verbunden, die durch eine Verkürzung der Telomerlänge gekennzeichnet ist7. Die Analyse von Telomerrestriktionsfragmenten (TRF), wie sie von Mender beschrieben wird, ist eine sehr teure Methode8. Aus diesem Grund wird es in den meisten Studien bei der Quantifizierung der Telomerlänge nicht implementiert.
Gegenwärtig verwenden die meisten epidemiologischen Studien quantitative Messungen der Telomerlänge auf Basis der quantitativen Polymerase-Kettenreaktion (qPCR). Die qPCR-basierte Methode ist jedoch eine relative Messmethode, da sie das Verhältnis zwischen Telomeren und Einzelkopien-Genamplifikationsprodukten und nicht die absolute Telomerlänge misst. Die Telomerlängenmessung mit dem TRF-Protokoll ist die Goldstandardmethode, da sie die Telomerlängenverteilung in der Probe messen kann und die Messungen in absoluten Werten in Kilobasen (kb) ausgedrückt werden können. Seine Verwendung ist jedoch begrenzt, da es umständlich, arbeitsintensiv und kostspielig ist. In dieser Arbeit stellen wir ein optimiertes Protokoll zur Telomerlängenmessung mit Chemilumineszenz-basierten TRFs vor.
Die TRF-Analyse umfasst sieben Hauptschritte: 1) Kultivierung von Zellen für die genomische DNA-Extraktion, 2) genomische DNA-Extraktion mit der Phenol:Chloroform:Isoamylalkohol (P:C:I)-Methode, 3) Restriktionsverdauung genomischer DNA, 4) Agarose-Gelelektrophorese, 5) Southern-Blotting des Restriktionsverdau-DNA-Fragments, 6) Hybridisierung und Detektion über Chemilumineszenz-Die immobilisierte Telomersonde wird durch ein hochempfindliches chemilumineszentes Substrat für alkalische Phosphatase, Dinatrium-2-Chlor-5-(4-methoxyspiro[1,2-dioxetan-3,2′-(5-chlortricyclo[3.3.1.13.7]decan])-4-yl]-1-phenylphosphat (CDP-Star)-und 7)-Analyse visualisiert, um Informationen über die mittlere Telomerlänge und -reichweite aus diesen Telomerausstrichen zu erhalten.
Protokoll
HINWEIS: In der Materialtabelle finden Sie Details zu allen Reagenzien, die im Protokoll unten verwendet werden. Tabelle 1 enthält im Labor hergestellte Reagenzien zusammen mit optimierten Volumina und Tabelle 2 zeigt die Arbeitskonzentrationen kommerziell erhältlicher Reagenzien.
1. Zellkultur
- Zellen, deren Telomerlänge gemessen werden soll (hier wurden A2780-Zellen verwendet, bei denen es sich um eine ovarielle Adenokarzinom-Zelllinie handelt), werden in Dulbeccos modifiziertem Eagle-Medium (DMEM) Komplettmedium mit 10 % fötalem Kälberserum (FBS), Streptomycin, Penicillin und Amphotericin B in einer 6-cm-Petrischale gehalten. Inkubieren Sie bei 37 °C in einer befeuchteten und kontrollierten Umgebung mit 5 % Kohlendioxid, bis die Zellen zu 80 % bis 100 % konfluierend sind.
- Entfernen Sie das Medium und waschen Sie es mit 5 ml 1x phosphatgepufferter Kochsalzlösung (PBS).
- Behandeln Sie die Zellen mit 1 ml Trypsin-Ethylendiamintetraessigsäure (EDTA), um die Zellen abzulösen, indem Sie sie bei 37 °C für 3-5 Minuten inkubieren.
- Fügen Sie 2 ml DMEM-Komplettmedium hinzu, um das Trypsin zu inaktivieren und die Zellen in einem Zentrifugationsröhrchen zu sammeln.
- Pelletieren Sie die Zellen bei 2.348 × g für 5 Minuten.
- Das Pellet mit 1x PBS waschen und bei 2.348 × g 5 min zentrifugieren.
- Lagern Sie das Pellet bis zur weiteren Verwendung bei -80 °C.
HINWEIS: Die Zellzahl kann je nach Zelllinie variieren. Wir erhielten ca. 2,5 × 106 Zellen im Fall der A2780-Zelllinie, die in weiteren Schritten verwendet wird.
2. Genomische DNA-Isolierung
- Geben Sie 500 μl Lysepuffer (10 mM Tris-Cl, pH 8,0; 25 mM EDTA, pH 8,0; 100 mM NaCl; 0,5 % w/v Natriumdodecylsulfat) in das Zellpellet und mischen Sie es vorsichtig mit einer abgeschnittenen Spitze (mit einem Öffnungsdurchmesser von mindestens 2 mm). Fügen Sie 20 μg/mL frisch zubereitete RNase A hinzu und mischen Sie vorsichtig durch Inversion.
- Bei 37 °C für 30 min inkubieren und das Röhrchen während der Inkubation gelegentlich umdrehen.
- Proteinase K bis zu einer Endkonzentration von 100 μg/ml zugeben und 10-mal vorsichtig durch Inversion mischen. Bei 55 °C 2 h inkubieren. Drehen Sie das Röhrchen während der Inkubation in regelmäßigen Abständen (alle 10 Minuten) um.
- Fügen Sie 500 μl Phenol:Chloroform:Isoamylalkohol-Reagenz (25:24:1) hinzu und mischen Sie vorsichtig durch Inversion 20 Mal. Bei 9.391 × g für 15 min bei Raumtemperatur (ca. 25 °C) zentrifugieren.
Anmerkungen: Abbildung 1A zeigt die drei Schichten, die nach dem Zentrifugieren erhalten werden. - Entfernen Sie die viskose obere wässrige Schicht aus den drei sichtbaren Schichten, legen Sie sie mit einer abgeschnittenen Spitze in ein frisches Röhrchen und fügen Sie die gleiche Menge Chloroform hinzu. 20 Mal durch sanfte Umkehrung mischen.
- Zentrifugieren Sie die Röhrchen bei 9.391 × g für 15 min bei Raumtemperatur.
- Sammeln Sie die wässrige Schicht und fügen Sie eine entsprechende Menge von 5 M NaCl hinzu, so dass die Endkonzentration von NaCl 0,2 M beträgt.
- Fügen Sie zwei Volumina 100%iges Ethanol hinzu. 20-25 Mal durch sanfte Umkehrung mischen. Bei 15.871 × g 5 min bei Raumtemperatur zentrifugieren.
- Entfernen Sie den Überstand und fügen Sie 500 μl 70%iges Ethanol hinzu, um das Pellet zu waschen. Bei 15.871 × g für 5 min bei Raumtemperatur zentrifugieren.
- Entfernen Sie den Überstand, lassen Sie das Pellet einige Minuten an der Luft trocknen und fügen Sie 50 μl steriles nukleasefreies Wasser hinzu. Lassen Sie die DNA 1-2 Tage bei Raumtemperatur rehydrieren. Mischen Sie durch Pipettieren mit der abgeschnittenen Spitze.
- Messen Sie die DNA-Konzentration mit ultravioletter (UV)-Spektrophotometrie. Verdünnen Sie die Proben bei Bedarf mit sterilem nukleasefreiem Wasser, um eine Mindestkonzentration von 300-500 ng/μl zu erhalten.
- Überprüfen Sie die Unversehrtheit der DNA, indem Sie sie mit einem 1%igen Agarose-Gel ausführen (Abbildung 1B).
- Lagern Sie die verdünnte DNA bis zur weiteren Verwendung bei -20 °C.
3. Verdauung genomischer DNA
- Bereiten Sie eine Enzymmischung aus Rsa1 (20 U) und Hinf1 (20 U) vor und fügen Sie pro Reaktion 2 μl Restriktionsaufschlusspuffer hinzu.
- Nehmen Sie ein angemessenes Volumen genomischer DNA, so dass die Gesamtmenge der DNA 1,5 μg beträgt. Das Volumen wird mit sterilem nukleasefreiem Wasser aufgefüllt, so dass das Gesamtvolumen nach der Enzymzugabe 20 μL beträgt.
- Durch Klopfen gut mischen, gefolgt von einer Impulsdrehung.
- Inkubieren Sie die Mischung bei 37 °C für 2 h.
4. Agarose-Gelelektrophorese
- Bereiten Sie ein 10 cm × 15 cm großes 0,8%iges Agarose-Gel in 1x Trisacetat-EDTA-Puffer (TAE) unter Verwendung von hochwertigem Agarose vor.
- Geben Sie 5 μl Beladungsfarbstoff zu jeder verdauten genomischen DNA-Probe, so dass das endgültige Volumen 25 μl beträgt, und laden Sie die Proben auf das Gel.
- Bereiten Sie eine molekulare Markermischung vor, indem Sie 1 μl molekularen Marker (Leiter), 3 μl steriles nukleasefreies Wasser und 1 μl 5-fachen Beladungsfarbstoff verwenden.
- Laden Sie 5 μl molekulare Markermischung auf beide Seiten der genomischen DNA-verdauten Proben, wenn eine höhere Anzahl von Proben (~10) vorhanden ist, oder nur auf einer Seite, wenn weniger als oder gleich fünf Proben vorhanden sind.
- Lassen Sie das Gel 6 Stunden lang bei 5 V/cm laufen. Sobald der Lauf abgeschlossen ist, machen Sie eine Kerbe an einer Ecke des Gels, um die Ladereihenfolge zu markieren.
- Tauchen Sie das Gel für 10 min bei Raumtemperatur unter leichtem Rühren in 0,25 M HCl.
- Spülen Sie das Gel zweimal mit destilliertem Wasser ab.
- Die DNA wird denaturiert, indem das Gel zweimal für jeweils 15 min bei Raumtemperatur unter leichtem Rühren in eine Lösung von 0,5 M NaOH und 1,5 M NaCl getaucht wird.
- Spülen Sie das Gel zweimal mit destilliertem Wasser ab.
- Neutralisieren Sie die DNA, indem Sie das Gel zweimal für 15 min bei Raumtemperatur unter leichtem Rühren in eine Lösung aus 0,5 M Tris-HCl und 3 M NaCl (pH 7,5) tauchen.
5. Südliches Blotting
- Schneiden Sie eine Nylonmembran von 10 cm × 12 cm Größe zu und machen Sie eine Kerbe in der gleichen Position wie das Gel.
- Aktivieren Sie die Membran durch Eintauchen in destilliertes Wasser, gefolgt von 20-fachem Natriumsalzcitratpuffer (SSC) (siehe Tabelle 1).
- Richten Sie die Übertragung ein.
- Nehmen Sie ein sauberes Glastablett und stellen Sie ein weiteres Tablett in umgekehrter Position hinein. Erstellen Sie einen Docht mit normalem Filterpapier so, dass die Enden den Boden der äußeren Schale berühren.
- Legen Sie das Gel auf das Filterpapier. Legen Sie die aktivierte Nylonmembran vorsichtig über das Gel, wobei sich die Kerben überlappen, und entfernen Sie alle Luftblasen, indem Sie sie über einen Glasstab rollen.
- Legen Sie einen 2 cm großen Haufen von 9,5 cm × 11,5 cm Zellulosefilterpapier, gefolgt von einem 6 cm großen Haufen normalem Filterpapier mit den gleichen Abmessungen. Platzieren Sie ein Gewicht auf diesem Setup, so dass darunter eine gleichmäßige Gewichtsverteilung besteht. Füllen Sie das äußere Fach mit 20x SSC-Puffer (siehe Abbildung 2).
- Überlassen Sie das Setup für den Transfer über Nacht.
- Nach dem Südtransfer fixieren Sie die übertragene DNA auf der Membran durch UV-Vernetzung auf einem UV-Transilluminator oder UV-Vernetzer. Verwenden Sie eine 302 nm 8 W Lampe für 5 min oder eine 254 nm 8 W Lampe für 2 min, was ungefähr 120 mJ entspricht. Achten Sie darauf, dass die Membranseite, auf der die DNA übertragen wird, der Lampe zugewandt ist.
- Waschen Sie die Membran zweimal mit 25 ml 2x SSC-Puffer.
- Unterbrechen Sie das Protokoll bei Bedarf, indem Sie den Blot vollständig trocknen und nach dem lockeren Einwickeln in Folie bis zur Weiterverarbeitung bei 2-8 °C lagern.
6. Hybridisierung und Chemilumineszenz-Detektion
- Den Vorhybridisierungspuffer auf 42 °C vorwärmen.
Anmerkungen: Die folgenden Schritte umfassen die Volumina der Lösungen/Puffer, die pro 100cm2 Blot zu verwenden sind. - Inkubieren Sie die Membran in 10 ml Prähybridisierungspuffer für 1 h bei 42 °C unter sanftem Rühren zur Prähybridisierung.
- Fügen Sie 0,5 μl Telomersonde pro 5 ml vorgewärmten Vorhybridisierungspuffer hinzu, um die Hybridisierungslösung herzustellen.
- Inkubieren Sie den Blot in 10 ml Hybridisierungslösung bei 42 °C für 3 h unter sanftem Rühren.
- Waschen Sie den Blot mit dem strengen Puffer 1 (Tabelle 1) zweimal für 10 Minuten (je 25 ml) bei Raumtemperatur unter leichtem Rühren.
- Stringenter Puffer 2 (Tabelle 1) für 30 min bei 50 °C vorwärmen.
- Waschen Sie den Blot zweimal mit strengem Puffer 2 für 15 min (je 25 ml) bei 50 °C unter leichtem Rühren.
- Mit 15 ml Waschpuffer 5 min lang unter leichtem Rühren bei RT spülen.
- Inkubieren Sie die Membran in 10 mL frisch zubereiteter 1x-Blockierlösung für 30 min bei Raumtemperatur unter leichtem Rühren.
- Die Membran wird in 10 ml Anti-Digioxigenin, konjugiert mit alkalischer Phosphatase-Arbeitslösung (siehe Tabelle 2), für 30 min bei RT unter leichtem Rühren inkubiert.
- Waschen Sie den Blot zweimal mit Waschpuffer bei Raumtemperatur für 15 Minuten unter leichtem Rühren.
- Inkubieren Sie in 10 ml Detektionspuffer für 5 min bei RT unter sanftem Rühren.
- Entfernen Sie den überschüssigen Nachweispuffer und halten Sie die Membran mit der DNA nach oben auf einem Hybridisierungsbeutel oder einer Acetatfolie. Geben Sie ~1-1,5 ml Substratlösung tropfenweise auf die Membran, legen Sie sofort eine weitere Folie darauf und inkubieren Sie sie 5 Minuten lang bei RT.
- Drücken Sie die überschüssige Substratlösung für die Bildgebung aus.
- Bilden Sie den Blot ca. 20 Minuten lang in einem Gel-Dokumentations-Bildgebungssystem ab, wobei mehrere Bilder zu unterschiedlichen Zeitpunkten aufgenommen werden. Wählen Sie ungesättigte Bilder für die weitere Analyse aus.
HINWEIS: Bei schwachem Signal kann die Bildgebung über einen längeren Zeitraum durchgeführt werden.
7. Würdigung
- Nachdem Sie die Bilddateien erhalten haben, exportieren Sie sie zur Veröffentlichung in .tif Format mit der höchstmöglichen Auflösung (in diesem Fall 600 dpi).
- Installieren Sie die Telotool-Software . Dies erfordert eine bestimmte Version (R2012a) von MATLAB (frei verfügbar), um ausgeführt zu werden.
- Öffnen Sie die Software und klicken Sie oben rechts auf die Schaltfläche Bilddatei laden .
- Nachdem das Bild geladen wurde, klicken Sie auf Bild umkehren, sodass der Bildhintergrund schwarz und die Schlieren/Streifen weiß sind.
- Schneiden Sie das Bild so zu, dass die oberen Ecken von den Vertiefungen ausgehen. Nachdem Sie die Auswahl des Zuschnitts getroffen haben, klicken Sie mit der rechten Maustaste hinein und wählen Sie Bild zuschneiden.
- Nachdem das Bild zugeschnitten wurde, klicken Sie auf Fahrspuren berechnen und lassen Sie die Fahrspurerkennung automatisch erfolgen.
- Wenn die Fahrspuren nicht richtig erkannt wurden, z. B. wenn bestimmte Fahrspuren überhaupt nicht erkannt wurden oder wenn zusätzliche oder teilweise Fahrspuren erkannt wurden, führen Sie die Fahrspuranpassung wie unten beschrieben durch.
- Klicken Sie auf Bahnen anpassen und fügen Sie im sich öffnenden Popup-Fenster nach Bedarf Bahnen hinzu und/oder passen Sie sie an und klicken Sie auf Übernehmen und Schließen.
- Stellen Sie nach dem Anpassen der Bahnen sicher, dass auf jeder der Bahnen ein roter Punkt zu sehen ist, und passen Sie die Leitern mit der Schaltfläche Ladder Fit an, wobei Sie sicherstellen, dass sich die Anzahl der Spitzen in beiden Ladder-Bahnen an der gleichen Position befindet. Entfernen Sie alle überflüssigen Peaks mit der Schaltfläche Extremum löschen .
- Nachdem die Kontaktpläne angepasst wurden, wählen Sie Fahrspurprofile, klicken Sie auf Kontaktplananpassung und wählen Sie Polynompassung.
- Klicken Sie auf Trendlinie, wie von der Software empfohlen, und wählen Sie dann in der nächsten Option Korrigiert aus.
- Klicken Sie auf Get Results , um die Ergebnisse als Tabelle anzuzeigen, in der die Zeile Mean TRF die Telomerlängenmessung der jeweiligen Fahrspurproben darstellt.
- Klicken Sie auf Alle speichern, um die Ergebnisse in Form einer Tabelle zu speichern .
Ergebnisse
Die extrahierte genomische DNA (gDNA), die auf einem 1%igen Agarose-Gel ausgeführt wurde, zeigte eine gute Integrität, wie in Abbildung 1B dargestellt, was darauf hindeutet, dass die Probe für die weitere Weiterverarbeitung von TRFs verwendet werden könnte. Der TRF-Assay wurde dann durchgeführt, indem die in jedem Schritt erforderlichen Lösungsvolumina modifiziert wurden (siehe Tabelle 1 und Tabelle 2). Das TRF-Signal war deutlich sichtbar (Abbildung 3). So konnten durch die Modifikation der Lösungsvolumina und -konzentrationen mehr Proben verarbeitet werden, ohne dass sich dies negativ auf die Ergebnisse auswirkte, und die Telomerlänge konnte mit frei verfügbarer Software wie Telotool10 erfolgreich bestimmt werden.

Abbildung 1: Isolierung und Qualitätsprüfung genomischer DNA. (A) Drei verschiedene Trennphasen, die durch Zentrifugation nach Zugabe von Phenol:Chloroform:Isoamylalkohol erhalten werden. Die obere wässrige Schicht enthält die gDNA, die Interphase die Proteine und die untere, organische Phase die abgebaute RNA, Zelltrümmer und Lipide. (B) Ein Bild eines 1%igen Agarosegels, das intakte, unverdaute gDNA zeigt. Bahn 1 zeigt eine 1 kb große Leiter und Bahn 2 zeigt unverdaute gDNA der Krebszelllinie A2780. Abkürzung: gDNA = genomische DNA. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
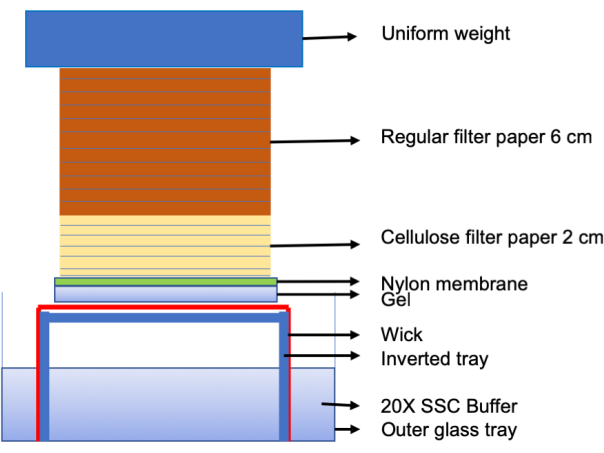
Abbildung 2: Illustration des Southern-Blotting-Transfer-Setups. Repräsentatives Diagramm, das die Systemanordnung für die Übertragung von Telomer-Wiederholungsabstrichen vom Gel auf die Nylonmembran durch Kapillarwirkung zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
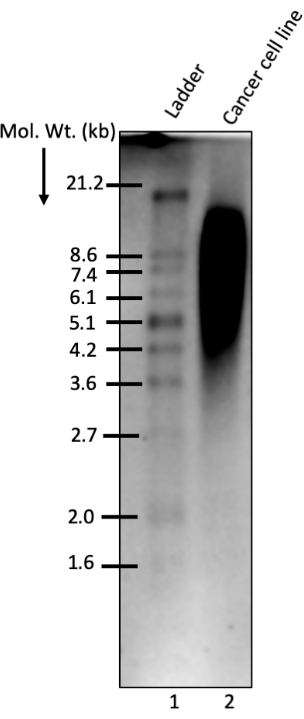
Abbildung 3: Chemilumineszenz-Detektion von TRFs nach Southern-Blot und Hybridisierung. Blot, der eine Reihe von Telomerwiederholungen als Abstrich in Spur 2 zeigt. Spur 1 zeigt einen molekularen Marker, wobei die Molekulargewichte (kb) der Banden auf der linken Seite angegeben sind. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Tabelle 1: Liste der in diesem Protokoll verwendeten Reagenzien. Die Tabelle enthält die im Labor hergestellten Reagenzien zusammen mit ihren Lagerungsdetails, der empfohlenen Verwendung der Reagenzien des TAGGG-Telomerlängen-Assay-Kits und ihren modifizierten Verwendungsvolumina gemäß der Optimierung in diesem Protokoll. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Liste der kommerziell erhältlichen Reagenzien mit modifizierten Gebrauchsanweisungen. Die Tabelle enthält kommerziell erhältliche Reagenzien zusammen mit verschiedenen Verdünnungen, ihren Lagerungsdetails und ihrer empfohlenen Verwendung durch das TAGGG-Telomerlängen-Assay-Kit und modifizierten Verwendungsvolumina gemäß der Optimierung in diesem Protokoll. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Wir beschreiben ein detailliertes Verfahren für eine nicht-radioaktive, Chemilumineszenz-basierte Methode zur Telomerlängenmessung mittels Southern Blotting. Das Protokoll wurde getestet, um die vernünftige Verwendung mehrerer Reagenzien ohne Kompromisse bei der Qualität der Ergebnisse zu ermöglichen. Der Vorhybridisierungs- und Hybridisierungspuffer kann bis zu fünfmal wiederverwendet werden. Die Enzymkonzentration kann zwischen 10 und 20 U pro 1,5 bis 2 μg genomischer DNA variieren, ohne dass die Ergebnisse beeinträchtigt werden. Mehrere andere Kit-Komponenten, wie z. B. der DIG-markierte Molekulargewichtsmarker und die Hybridisierungssonde, können in niedrigeren Konzentrationen als empfohlen verwendet werden. Die optimierten Volumina sind in Tabelle 2 angegeben. Wir haben sie mit der Hälfte der empfohlenen Mengen/Konzentrationen getestet und eine optimale Leistung festgestellt. Das Befolgen dieser Schritte reduziert die Kosten pro Probe enorm und ermöglicht es den Forschern, die Telomerlänge mit dieser Methode in größerem Maßstab zu messen.
Es gibt mehrere veröffentlichte TRF-Protokolle. Wir haben das kommerziell erhältliche Kit-Protokoll optimiert, um es kostengünstig zu machen. Dieses Protokoll unterscheidet sich von einigen der veröffentlichten Protokolle, da es keine Radioaktivität verwendet11,12. Es ist auch relativ einfach, da es die Kit-Komponenten verwendet, anstatt hauseigene Reagenzien, insbesondere die Telomersonde13, herzustellen.
Wenn das endgültige Bild lückenhaft ist, ist die Membran während der Inkubation teilweise getrocknet. Um dies zu beheben, sollte man entweder das Lösungsvolumen erhöhen oder mit einer höheren Geschwindigkeit rühren. Wenn auf dem Blot keine Schlieren sichtbar sind, liegt möglicherweise ein Problem bei der Übertragung nach Süden vor. Darüber hinaus könnte es ein Problem mit der DNA-Quantität oder -Qualität gegeben haben.
Es gibt einige Einschränkungen für diese Methode. Erstens dauert die genomische DNA-Extraktion und -Quantifizierung mehr als 4 Tage, basierend auf der Quelle der DNA14. DNA mit höherem Molekulargewicht benötigt eine längere Zeit, um zu rehydrieren und zu homogenisieren, was die Quantifizierung und das genaue Pipettieren erschwert. Zweitens ist das TRF-Protokoll langwierig (mindestens 2 Tage) und erfordert qualifizierte Labormitarbeiter, um es umzusetzen. Es ist auch eine teure Methode aufgrund der Reagenzien und der benötigten Mengen. Das TRF-Protokoll misst auch die durchschnittliche Telomerlänge in jeder Probe, im Gegensatz zur kürzesten Telomerlänge, die TESLA misst, oder der Telomerlänge pro Zelle, die durchflusszytometrische Methoden liefern15. Eine weitere Einschränkung der TRF-Analyse mit den hier verwendeten Enzymen ist eine Überschätzung der Telomerlänge, da diese Enzyme keine Restriktionsstelle in den subtelomeren Regionen aufweisen15.
Trotz der Einschränkungen gibt es jedoch Gründe, warum diese Methode immer noch als Goldstandard der Telomerlängenmessung gilt. Telomerlängen werden genau in absoluten Werten in Kilobasenpaaren (kbp) wiedergegeben.
Mit dieser Methode kann die durchschnittliche Telomerlänge in einer Vielzahl von Zelltypen gemessen werden, von Zelllinien16,17 bis hin zu Buffy-Coat-Pellets18,19. Dies macht es zu einer robusten Methode, die in verschiedenen Arten von Forschungsstudien eingesetzt werden kann. Es kann auch in groß angelegten epidemiologischen Studien sowie in Studien eingesetzt werden, in denen zellbasierte Therapeutika entwickelt werden.
Offenlegungen
Die Autoren haben keine Interessenkonflikte.
Danksagungen
Wir möchten uns bei Frau Prachi Shah dafür bedanken, dass sie uns anfangs bei der Optimierung des Protokolls geholfen hat. Wir bedanken uns bei Dr. Manoj Garg für die Bereitstellung der A2780 Eierstockkrebszelllinie. Das EK wird durch ein Forschungsstipendium des Departements für Biotechnologie (Nr. BT/RLF/Re-entry/06/2015), des Departements für Wissenschaft und Technologie (ECR/2018/002117) und des NMIMS Seed Grant (IO 401405) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Cell Line | |||
| A2780 (Ovarian adenocarcinoma cell line) | Received as a gift | ||
| Equipment | |||
| ChemiDoc XRS+ (for imaging and UV cross linking) | Biorad | Universal hood II (721BR14277) | |
| Nanodrop (Epoch 2) | Biotek | EPOCH2 | |
| Software | |||
| TeloTool | Version 1.3 | ||
| Materials | |||
| Acetic Acid | Molychem | 64-19-7 | |
| Agarose | MP | 180720 | |
| Amphotericin B | Gibco, ThermoFisher Scientific, USA | 15240062 | |
| DMEM | HyClone, Cytiva, USA | SH30243.01 | |
| Ethylenediamine tetraacetic acid | Molychem | 6381-92-6 | |
| HI FBS | Gibco, ThermoFisher Scientific, USA | 10270106 | |
| HCl | Molychem | 76-47-01-0 | |
| NaCl | Molychem | 7647-14-5 | |
| NaOH | Molychem | 1310-73-2 | |
| Nylon membrane | Sigma | 11209299001 | |
| Penicillin | Gibco, ThermoFisher Scientific, USA | 15240062 | |
| Sodium dodecyl sulfate | Affymetrix | 151-21-3 | |
| Streptomycin | Gibco, ThermoFisher Scientific, USA | 15240062 | |
| Tris | BIORAD | 77-86-1 | |
| Tris HCl | Sigma Aldrich | 1185-53-1 | |
| Whatman paper | GE healthcare lifesciences | 1001-917 | |
| Reagents | |||
| 1 kb ladder | NEB | N3232S | |
| 20x SSC | Invitrogen | 15557-036 | |
| Anti DIG AP | Telo TAGGG Telomere Length Assay kit | 12209136001 | |
| Blocking solution 10x | Telo TAGGG Telomere Length Assay kit | 12209136001 | |
| Cutsmart Buffer | NEB | B6004 | |
| Detection buffer 10x | Telo TAGGG Telomere Length Assay kit | 12209136001 | |
| Dig easy hyb | Telo TAGGG Telomere Length Assay kit | 12209136001 | |
| Digestion Buffer | Telo TAGGG Telomere Length Assay kit | 12209136001 | |
| Hinf 1 | Telo TAGGG Telomere Length Assay kit | 12209136001 | |
| Hinf 1 (alternative to kit) | NEB | R0155T | |
| Loading Dye | BIOLABS | N3231S | |
| Maleic acid buffer 10x | Telo TAGGG Telomere Length Assay kit | 12209136001 | |
| Molecular marker | Telo TAGGG Telomere Length Assay kit | 12209136001 | |
| Probe | Telo TAGGG Telomere Length Assay kit | 12209136001 | |
| Rsa 1 | Telo TAGGG Telomere Length Assay kit | 12209136001 | |
| Rsa 1 (alternative to kit) | NEB | R0167L | |
| Substrate | Telo TAGGG Telomere Length Assay kit | 12209136001 | |
| Wash buffer | Telo TAGGG Telomere Length Assay kit | 12209136001 |
Referenzen
- Greider, C. W. Telomere length regulation. Annual Review of Biochemistry. 65, 337-365 (1996).
- Valdes, A. M., et al. Obesity, cigarette smoking, and telomere length in women. Lancet. 366 (9486), 662-664 (2005).
- Allsopp, R. C., et al. Telomere length predicts replicative capacity of human fibroblasts. Proceedings of the National Academy of Sciences. 89 (21), 10114-10118 (1992).
- Epel, E. S., et al. Accelerated telomere shortening in response to life stress. Proceedings of the National Academy of Sciences. 101 (49), 17312-17315 (2004).
- Canela, A., Vera, E., Klatt, P., Blasco, M. A. High-throughput telomere length quantification by FISH and its application to human population studies. Proceedings of the National Academy of Sciences. 104 (13), 5300-5305 (2007).
- Révész, D., Milaneschi, Y., Verhoeven, J. E., Penninx, B. W. Telomere length as a marker of cellular aging is associated with prevalence and progression of metabolic syndrome. The Journal of Clinical Endocrinology and Metabolism. 99 (12), 4607-4615 (2014).
- Rizvi, S., Raza, S. T., Mahdi, F. Telomere length variations in aging and age-related diseases. Current Aging Science. 7 (3), 161-167 (2014).
- Mender, I., Shay, J. W. Telomere restriction fragment (TRF) analysis. Bio-Protocol. 5 (22), e1658 (2015).
- Zhu, Y., Liu, X., Ding, X., Wang, F., Geng, X. Telomere and its role in the aging pathways: telomere shortening, cell senescence and mitochondria dysfunction. Biogerontology. 20 (1), 1-16 (2019).
- Göhring, J., Fulcher, N., Jacak, J., Riha, K. TeloTool: a new tool for telomere length measurement from terminal restriction fragment analysis with improved probe intensity correction. Nucleic Acids Research. 42 (3), 21 (2014).
- Jenkins, F. J., Kerr, C. M., Fouquerel, E., Bovbjerg, D. H., Opresko, P. L. Modified terminal restriction fragment analysis for quantifying telomere length using in-gel hybridization. Journal of Visualized Experiments. (125), e56001 (2017).
- Fojtová, M., Fajkus, P., Sováková, P. P., Fajkus, J. Terminal restriction fragments (TRF) method to analyze telomere lengths. Bio-protocol. 5 (23), e1671 (2015).
- Kimura, M., et al. Measurement of telomere length by the Southern blot analysis of terminal restriction fragment lengths. Nature Protocols. 5 (9), 1596-1607 (2010).
- Trigodet, F., et al. High molecular weight DNA extraction strategies for long-read sequencing of complex metagenomes. Molecular Ecology Resources. 22 (5), 1786-1802 (2022).
- Lai, T. P., Wright, W. E., Shay, J. W. Comparison of telomere length measurement methods. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 373 (1741), 20160451 (2018).
- Mochida, A., et al. Telomere size and telomerase activity in Epstein-Barr virus (EBV)-positive and EBV-negative Burkitt's lymphoma cell lines. Archives of Virology. 150 (10), 2139-2150 (2005).
- Gupta, N., et al. Replicative senescence, telomere shortening and cell proliferation rate in Gaddi goat's skin fibroblast cell line. Cell Biology International. 31 (10), 1257-1264 (2007).
- Michaeli, J., et al. Leukocyte telomere length correlates with extended female fertility. Cells. 11 (3), 513 (2022).
- Lesmana, A., et al. Continuous reference intervals for leukocyte telomere length in children: the method matters. Clinical Chemistry and Laboratory Medicine. 59 (7), 1279-1288 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten