Method Article
Protokoll zur Analyse und Konsolidierung von TrackMate-Ausgängen zur Messung der zweidimensionalen Zellmotilität mittels Nuclear Tracking
In diesem Artikel
Zusammenfassung
Dieses Protokoll zur Hochdurchsatzmessung der Zellmotilität in HaCaT-Keratinozyten beschreibt Verfahren zur Erfassung und Verarbeitung von Bildern von Zellkernen und zur Durchführung von Partikelverfolgung mit dem ImageJ-Plugin TrackMate.
Zusammenfassung
Die kollektive zelluläre Migration spielt eine Schlüsselrolle in vielen grundlegenden biologischen Prozessen, einschließlich der Entwicklung, der Wundheilung und der Metastasierung von Krebs. Um die Regulation der Zellmotilität zu verstehen, müssen wir in der Lage sein, sie unter verschiedenen Bedingungen einfach und konsistent zu messen. In dieser Arbeit beschreiben wir eine Methode zur Messung und Quantifizierung der Einzelzell- und Bulk-Motilität von HaCaT-Keratinozyten mit Hilfe einer Kernfärbung. Diese Methode umfasst ein MATLAB-Skript zur Analyse von TrackMate-Ausgabedateien, um Verschiebungen, Motilitätsraten und Trajektorienwinkel in einzelnen Zellen und in großen Mengen für eine Bildgebungsstelle zu berechnen. Dieses Skript zur Motilitätsanalyse ermöglicht eine schnelle, unkomplizierte und skalierbare Analyse der Zellmotilitätsraten aus TrackMate-Daten und könnte auf breiter Basis verwendet werden, um die Regulation der Motilität in Epithelzellen zu identifizieren und zu untersuchen. Wir stellen auch ein MATLAB-Skript zur Verfügung, um Mikroskopievideos, die auf einem Mikroskop gesammelt wurden, neu zu organisieren und in TIF-Stacks zu konvertieren, die mit dem ImageJ TrackMate-Plugin in großen Mengen analysiert werden können. Mit dieser Methodik zur Untersuchung der Rolle von Adhärens-Verbindungen und der Aktin-Zytoskelettdynamik bei der Regulierung der Zellmotilität in HaCaT-Keratinozyten zeigen wir, dass die Arp2/3-Aktivität für die erhöhte Motilität erforderlich ist, die nach einer α-Catenin-Depletion in HaCaT-Keratinozyten beobachtet wurde.
Einleitung
Eine präzise, reaktionsschnelle Regulation der zellulären Motilität in Epithelzellen ist entscheidend für die Wundheilung und die Wiederauffüllung der Epithelschicht. Das Versagen der Aufrechterhaltung der Motilität kann zu Problemen mit der Embryonalentwicklung und der Wundheilung führen1 und eine überaktive Motilitätssignalisierung ist ein wichtiger Faktor für die Metastasierung von Krebs2.
Das Verständnis der zellulären Kontrolle der Motilität in HaCaT-Keratinozyten bietet wichtige Einblicke in diese Prozesse. Die hier beschriebenen Verfahren liefern konsistente Messungen und Berechnungen der durchschnittlichen Größe der zellulären Motilität auf Einzelzell- oder Populationsebene. Wir haben diese Methode verwendet, um die Motilität in HaCaT-Keratinozyten nach genetischer Störung oder Behandlung mit niedermolekularen Inhibitoren zu messen, um die Zellsignalisierung zu verstehen, die die Motilitätsraten steuert. Das bereitgestellte MATLAB-Skript misst sowohl die durchschnittliche Geschwindigkeit als auch die durchschnittliche Richtung der Motilität für jede Zelle.
Die kollektive Zellmotilität ist entscheidend für den epithelial-mesenchymalen Übergang sowohl in der Entwicklung als auch bei der Krebsmetastasierung3. Zellen erreichen Beweglichkeit durch Koordination von Adhäsion, Polarisation, Protrusion und Retraktion4. Diese Prozesse beruhen in hohem Maße auf einer koordinierten Regulation des dynamischen Aktin-Zytoskeletts. Die Aktivierung von Rac1-GTPase stromabwärts von PI3K oder verschiedenen Rezeptor-Tyrosinkinasen polarisiert die Zelle, was zu einer Aktinpolymerisationführt 5. Die Dynamik des Aktin-Zytoskeletts wird durch diese und andere Rho-vermittelte GTPasen reguliert, die durch nachgeschaltete verschiedene Wachstumsfaktoren aktiviert werden. Diese Rho-vermittelten GTPasen aktivieren dann den Arp2/3-Komplex, der die Aktinverzweigungstimuliert 6. Diese Dynamik des Aktin-Zytoskeletts steht in engem Zusammenhang mit einem weiteren wichtigen Regulator der Motilität: den interzellulären Verbindungen. Wir interessieren uns besonders dafür, wie die Adhärentionsverbindungen, die starke Adhäsionen zwischen den Zellen bilden, mit dem Aktin-Zytoskelett koordinieren, um die Gewebeintegrität zu erhalten7.
Wir haben bereits gezeigt, dass HaCaT-Keratinozyten, die einen shRNA-vermittelten α-Catenin-Knockdown exprimieren, höhere Motilitätsraten aufweisen als solche, die eine nicht-zielgerichtete shRNA-Kontrolle exprimieren8. Wir möchten die von uns entwickelten Motilitätsanalysewerkzeuge nutzen, um den Mechanismus dieser erhöhten Motilität bei Erschöpfung von α-Catenin besser zu verstehen. α-Catenin ist eine erforderliche zytosolische Komponente der Adhärensverbindungen9. Es ist durch seine Wechselwirkung mit ß-Catenin, das es als Monomer10,11 bindet, an Adhärensverbindungen beteiligt. α-Catenin kann jedoch auch von Adhärentverbindungen dissoziieren, um ein Homodimer zu bilden, das Aktinfilamente bindet und bündelt, wodurch die Arp2/3-Aktivität gehemmtwird 10,11. Obwohl α-Catenin nicht direkt mit Aktin interagiert, während es an ß-Catenin gebunden ist, kann es dennoch die Koordination zwischen Adhärensverbindungen und dem Zytoskelett durch Bindung an Vinculin, das Aktinfilamente stabilisiert, erleichtern oder verstärken12.
Obwohl das TrackMate-Plugin für ImageJ eine etablierte Methode zur Partikelverfolgung ist, die zur Verfolgung von Zellkernen verwendet werden kann, stellten wir fest, dass die verfügbaren Tools zum Parsen, Analysieren und Konsolidieren von TrackMate-Ausgaben zur Berechnung der Zellmotilität für mehrere Bildgebungsstellen nicht integriert waren und für Personen ohne Kenntnisse in mehreren Programmiersprachen schwierig zu bedienen waren. Tinevez und Herbert bieten eine Einführung in die Verwendung von TrackMate, aber der Abschnitt zur Analyse der mittleren quadratischen Verschiebung erfordert beträchtliche MATLAB-Kenntnisse13. Bestehende Methoden zur Extraktion von Motilitätsraten aus Zeitraffer-Mikroskopievideos bieten komplexere dreidimensionale Analysen, erfordern aber mehr Programmierkenntnisse 14,15. Pathfinder, eine Software zur Zellverfolgung und Motilitätsanalyse, die zuvor in unserem Labor entwickelt wurde, misst die Geschwindigkeit und Richtung der Zellmigration, ist aber nur auf Windows-Rechnern lauffähig und erfordert eine einschränkende Java-Laufzeitumgebung16. Da TrackMate ein so zuverlässiges und gut dokumentiertes Tool ist, haben wir eine unkomplizierte Methode zum Generieren, Analysieren und Organisieren großer zweidimensionaler TrackMate-Datensätze mit MATLAB entwickelt. Unser Skript entfernt auch die wiederholten ganzzahligen Datenpunkte, die manchmal in TrackMate-Ausgaben auftreten, nachdem eine verfolgte Zelle die Bildgebungsstelle verlassen hat, und ermöglicht so die Einbeziehung von Zellen mit hoher Motilität und/oder Richtungsabhängigkeit in die Analyse, ohne dass diese falschen Datenpunkte einbezogen werden. Wir bieten auch ein Skript zum Reorganisieren von Bildern, die auf einem ImageXpress Micro XL gesammelt wurden, in TIFF-Stacks, die mit TrackMate in großen Mengen analysiert werden können.
Bei diesem Protokoll werden die Zellen in eine 96-Well-Bildgebungsfolie ausgesät. Nachdem wir den Zellen genügend Zeit gelassen haben, sich am Boden der Platte festzusetzen, behandeln wir sie mit Hoechst-Kernfärbung und allen kleinen Molekülen, deren Auswirkungen auf die Beweglichkeit von Interesse sind. Wir sammeln Bilder der Zellkerne über einen Zeitraum von fünf oder mehr Stunden, danach werden die Bildsequenzen mit Hilfe von MATLAB zu hintergrundsubtrahierten TIFF-Stacks verarbeitet. Diese TIFF-Stapel werden mit dem ImageJ TrackMate-Plug-in analysiert, das jede einzelne Zelle über die Zeitpunkte17 hinweg registriert und verfolgt. Nachdem wir Zellspuren für alle Bildgebungsstellen generiert haben, verwenden wir ein benutzerdefiniertes MATLAB-Skript, um falsche Datenpunkte zu entfernen, die nach dem Verlassen des Bildgebungsfeldes auftreten, und um die durchschnittlichen Motilitätsraten für jede Bildgebungsstelle zu berechnen. Das Skript analysiert nur Zellen, die für eine vom Benutzer festgelegte Mindestanzahl von Zeitpunkten verfolgt werden, und ermöglicht es dem Benutzer, Zellen nach Gesamtentfernung und/oder zurückgelegter Richtung zu filtern. Das Ergebnis sind die durchschnittliche Verschiebung, die durchschnittliche Bewegungsrichtung, die Gesamtverschiebung und die Gesamtbewegungsrichtung für jede Zelle, die zur Berechnung der Massendurchschnitte dieser Werte für alle Zellen in einer Bildgebungsstelle verwendet werden. Dieses Protokoll kann in großen Bildgebungsexperimenten in großen Mengen durchgeführt werden, was sich für eine Motilitätsanalyse mit relativ hohem Durchsatz eignet (Abbildung 1).
Protokoll
1. Vorbereitung der Zellkultur und der Speicherfolie
- Kultur von HaCaT-Zellen in Zellkulturmedium (Dulbecco's Modified Eagle's Medium, ergänzt mit 2 mM L-Glutamin, 100 U/mL Penicillin/Streptomycin und 10 % (v/v) fötalem Rinderserum) bei 37 °C und 5 % Kohlendioxid in einem befeuchteten Inkubator.
- Aussaat der gewünschten Anzahl von Zellen pro Well (wir verwenden 5000 - 20.000 Zellen/Well) in einer 96-Well-Speicherfolie, z. B. einer schwarzen Polystyrol-Mikroplatte mit 96 Wells. Geben Sie in drei Vertiefungen nur Medium (keine Zellen).
- Legen Sie die Platte 20 - 30 Minuten lang auf eine völlig ebene Fläche, damit sich die Zellen mit gleichmäßiger räumlicher Verteilung am Boden der Platte befestigen können.
- Die Platte für 4-6 Stunden in einen befeuchteten Zellkultur-Inkubator (37 °C, 5 % Kohlendioxid) umfüllen, damit die Zellen vollständig am Boden der Platte haften können.
- Nehmen Sie das Medium von der Platte und ersetzen Sie es durch Zellkulturmedium mit 2 μg/ml Hoechst-Farbstoff (eine Verdünnung von 1:5000 von 1 mg/ml Stammlösung).
- Rückführplatte in den befeuchteten Zellkultur-Inkubator (37 °C, 5 % Kohlendioxid) für 2 h.
- Waschen Sie die Platte dreimal vorsichtig mit 100 μl phosphatgepufferter Kochsalzlösung pro Vertiefung, bevor Sie 90 μl Bildgebungsmedium (phenolrotfreies Dulbecco's Modified Eagle's Medium, ergänzt mit 2 mM L-Glutamin, 100 U/mL Penicillin/Streptomycin und 10 % (v/v) fötalem Rinderserum) in jede Vertiefung geben.
- Wenn Sie die Wirkung von niedermolekularen Inhibitoren oder anderen Effektoren messen, behandeln Sie die Zellen unmittelbar vor der Bildgebung.
2. Bildgebung
- Nehmen Sie die Platte (einschließlich reiner Medienvergrößerungen) bei 10-facher Vergrößerung in einem Mikroskop mit einer befeuchteten Klimakammer bei 37 °C und 5 % Kohlendioxid ab.
HINWEIS: Die Analyseskripte in diesem Protokoll wurden für Daten geschrieben, die mit einem ImageXpress Micro XL Weitfeldmikroskop von Molecular Devices erfasst wurden. - Sammeln Sie Bilder entsprechend der zeitlichen Auflösung, die das Experiment benötigt. Um beste Ergebnisse zu erzielen, zeichnen Sie mindestens 4 Zeitpunkte pro Stunde für mindestens 5 Stunden auf. Wenn die Wirkung von niedermolekularen Inhibitoren oder anderen Stoffen auf Zellen gemessen wird, ist eine Aufnahme für mindestens 5 Stunden nach der erwarteten Zeit durchzuführen, um eine Wirkung zu erzielen. Für unsere Zwecke bilden wir 100 8-Minuten-Zeitpunkte ab, was einer Gesamtbildzeit von 13 h 20 min entspricht.
3. Bildverarbeitung
- Verwenden Sie das bereitgestellte benutzerdefinierte MATLAB-Skript "ImageReorganizer.m", um Miniaturbilder zu entfernen und die Bilddateien nach Well und nicht nach Zeitpunkt neu zu organisieren. ImageXpress gibt Dateien in der folgenden Ordnerorganisation aus: "[Name des Experiments] > [JJJJ-MM-TT] > [Laufnummer] > Timepoint_[Zeitpunkt] > [Bilddateien]." Nachdem die Bilder neu organisiert wurden, führt dieses Skript eine Hintergrundsubtraktion für Bilder von jedem Zeitpunkt durch, indem es die gemittelten und Gaußschen verschwommenen Bilder der reinen Medien-Wells von diesem Zeitpunkt verwendet. Dieses Skript organisiert dann die vom Hintergrund subtrahierten Bilder in einem TIFF-Stapel für jede Imaging-Site. Jeder TIFF-Stack hat den Namen "[Well]_[Site].tif".
- Wenn sich das Experiment über mehr als einen Tag erstreckt hat, kombinieren Sie den Inhalt der Ordner [Laufnummer] für alle Tage, sodass sich alle Unterordner "Timepoint_[Zeitpunkt]" im selben Ordner "[Laufnummer]" befinden.
- Beachten Sie, dass dieses Skript die MATLAB-Funktion int2strz18 benötigt. Geben Sie den lokalen Pfad zu dieser Funktion nach "addpath" in Zeile 2 ein.
- Geben Sie die Zeilen für reine Medienbereiche in Zeile 9 als "emptyrows" und in Zeile 10 als "emptycolumns" ein.
- Geben Sie die Zeilen für Versuchsbohrungen als "Zeilen" in Zeile 15 und die Spalten als "Spalten" in Zeile 16 ein. Geben Sie die Anzahl der Bildgebungsstellen pro Vertiefung sowie "Standorte" in Zeile 17 ein.
- Geben Sie den Pfadnamen für den Ordner "[Run number]" in die Pfadleiste in MATLAB ein und führen Sie das Skript aus.
4. Verfolgung von Zellen
HINWEIS: TrackMate kann auf allen TIFF-Stacks in einem bestimmten Verzeichnis mit einer leicht modifizierten Version des Run_TrackMate_Headless.groovy-Skripts von Tinevez et al.17 ausgeführt werden oder kann manuell für jeden einzelnen TIFF-Stack ausgeführt werden. Wir führen FIJI manuell auf einigen TIFF-Beispielstacks aus, um sicherzustellen, dass wir die richtigen Parameter verwenden, bevor wir das Groovy-Skript auf allen TIFF-Stacks mit diesen Parametern ausführen.
- Um das TrackMate-Plugin in FIJI für alle TIFF-Stacks in einem Ordner auszuführen, laden Sie das Skript Run_TrackMate_Headless.groovy von Tinevez et al.17 herunter und verwenden Sie es gemäß dem bereitgestellten Protokoll. Wir verwenden eine leicht modifizierte Version dieses Skripts, die es uns ermöglicht, sowohl die Eingabedateien als auch die Ausgabedateinamen einfach zu stapeln und alle Parameter über eine grafische Benutzeroberfläche für die Eingabe einzugeben. Im Folgenden finden Sie detaillierte Anweisungen zur Verwendung dieses modifizierten Skripts, das hier als TrackMate_Headless_Mod.groovy bereitgestellt wird.
- Öffnen Sie das Skript in FIJI, indem Sie Datei>Öffnen auswählen und dann zum Speicherort des Skripts TrackMate_Headless_Mod.groovy navigieren.
- Wählen Sie Ausführen aus.
- Geben Sie das Verzeichnis, in dem sich die TIFF-Stacks befinden (die Standardausgabe von "StackGenerator.m" sollte "[Experiment Name] > [YYYY-MM-DD] >> Stacks") als "Eingabeverzeichnis" ein.
- Erstellen Sie ein Ausgabeverzeichnis und geben Sie den Pfadnamen als "Ausgabeverzeichnis" ein. Die Standardeinstellung für "Dateierweiterung sollte ".tif" sein; wenn nicht, geben Sie ".tif" als "Dateierweiterung" ein.
- Um TrackMate nur für eine Teilmenge der Dateien im Verzeichnis auszuführen, verwenden Sie das Eingabefeld nach "Dateiname enthält", um diese einzuschließen. Andernfalls lassen Sie dieses Feld leer.
- Wählen Sie "Verzeichnisstruktur beim Speichern beibehalten".
- Geben Sie "Spot-Radius", "Qualitätsschwellenwert", "Maximaler Frame-Abstand", "Maximaler Abstand beim Verknüpfen" und "Maximaler Abstand zum Schließen von Lücken" in Pixel entsprechend den gewünschten Einstellungen ein. In der Regel verwenden wir 3,5, 18, 2, 15 bzw. 40 für HaCaT-Kerne bei 10-facher Vergrößerung.
- Wählen Sie OK aus. Dieses Skript sollte eine XML-Datei für jeden TIFF-Stack im angegebenen Ausgabeverzeichnis ausgeben.
- Alternativ können Sie das TrackMate-Plugin in FIJI manuell für jeden TIFF-Stack gemäß dem in Tinevez et al. beschriebenen Protokoll ausführen.13
- Öffnen Sie einen TIFF-Stack in FIJI und öffnen Sie das TrackMate-Plugin, indem Sie in der Menüleiste Plugins >Tracking > TrackMate auswählen.
- Wenn in einem Popup-Fenster die Eingabeaufforderung "Es scheint, dass dieses Bild 1 Zeitpunkt hat, aber [Anzahl der Zeitpunkte] Scheiben hat. Möchten Sie Z und T tauschen?" Wählen Sie Ja aus.
- In TrackMate v4.0.1 verwenden Sie die Standardeinstellungen mit Ausnahme von "Geschätzter Blob-Durchmesser" (wir verwenden 7.000 Pixel für HaCaT-Kerne bei 10-facher Vergrößerung) und Schwellenwert (wir verwenden 18.000, aber dies variiert je nach verwendetem Mikroskop und Intensität des Hoechst-Farbstoffs).
- Verwenden Sie die Vorschaufunktion , um eine optimierte Partikelerkennung zu gewährleisten und die Einstellungen für Blob-Durchmesser und Schwellenwerte nach Bedarf anzupassen.
- Bei der Option "Maximale Lückenschließung" funktionieren die Standardeinstellungen häufig für die basale HaCaT-Motilität, aber die maximale Lückenschließungsentfernung muss unter Bedingungen, die die Beweglichkeit verbessern, möglicherweise auf bis zu 40,0 Pixel erhöht werden.
- Bestätigen Sie visuell, dass die Nachverfolgung mit minimalen Lücken in den Zellspuren optimiert ist.
- Wählen Sie unter Aktion auswählen [sic] die Option Tracks in XML-Datei exportieren und ausführen.
- Wiederholen Sie diesen Vorgang für alle TIFF-Stapel.
5. Quantifizierung der Motilitätsraten
HINWEIS: Verwenden Sie das bereitgestellte benutzerdefinierte MATLAB-Skript "TrackMateAnalysis.m", um fehlgeschlagene Kurven zu entfernen und die Motilität für alle TrackMate-Ausgabedateien zu quantifizieren. Das Skript entfernt Spuren, die außerhalb des Frames verschoben werden (deren Positionsspuren standardmäßig Ganzzahlen sind), sowie Spuren, die für weniger als eine bestimmte Anzahl von Zeitpunkten verfolgt werden. Das Skript entfernt Spuren, die außerhalb des Frames verschoben werden (deren Positionsspuren standardmäßig Ganzzahlen sind), sowie Spuren, die für weniger als eine designierbare Anzahl von Zeitpunkten verfolgt werden. Die Ausgabe ist die durchschnittliche Verschiebung pro Zeitpunkt für einzelne Zellen und alle Zellen an einer Bildgebungsstelle. (Beispielausgabe: 'Output.mat')
- Beachten Sie, dass dieses Skript die bereitgestellte MATLAB-Funktion "import_trackmate_xml.m." benötigt. Geben Sie den lokalen Pfad zu dieser Funktion nach "addpath" in Zeile 4 ein.
- Geben Sie in Zeile 7 die Mindestanzahl von Zeitpunkten an, über die ein Partikel verfolgt werden soll, um in die konsolidierte Ausgabe einbezogen zu werden.
- Geben Sie in den Zeilen 12 und 13 den Bereich der zurückgelegten Entfernungen (in Pixel) an, der in die gefilterte Analyse einbezogen werden soll. Um alle möglichen zurückgelegten Entfernungen einzubeziehen, geben Sie einen Bereich von 0 bis zur Breite Ihres Bildgebungsfeldes ein.
- Geben Sie in den Zeilen 14 und 15 den Bereich der Leitkurven (in Grad) an, die in die gefilterte Analyse einbezogen werden sollen. Um alle möglichen Trajektorien einzuschließen, geben Sie einen Bereich von 0 bis 360 ein.
- Führen Sie das Skript in dem Ordner aus, der alle TrackMate-Ausgabe-XML-Dateien enthält.
HINWEIS: Das Skript generiert und speichert eine MATLAB-Zellenstruktur, die alle in dieser Analyse generierten Daten enthält. Tabellen{1,:} listet die Namen der Trackmate-Ausgabe-XML-Dateien für jede Imaging-Site auf, die von diesem Skript analysiert wird. Tabellen{2,:} ist eine Unterstruktur, die die Positionen (Pixel), Verschiebungen (Pixel) und Bewegungswinkel (Grad, wobei 0° eine Rechtsrichtung innerhalb der Bildgebungsstelle angibt) für alle einzelnen Zellen zu jedem Zeitpunkt enthält. Er enthält auch die zurückgelegte Gesamtstrecke (Pixel), die durchschnittliche Motilität (Pixel/Zeitpunkt), den durchschnittlichen Bewegungswinkel (Grad) für jede Zelle über alle Zeitpunkte hinweg sowie den Gesamtbewegungswinkel zwischen dem ersten und dem letzten Zeitpunkt für jede Zelle. Die 3. bzw. 4. Zeile der Tabellen enthält die Werte für die durchschnittliche und die Standardabweichung (Pixel/Zeitpunkt) für alle Zellen zu allen Zeitpunkten an jeder Bildgebungsstelle. Die 5. bzw. 6. Zeile enthält die Durchschnitts- und Standardabweichungswerte für die Gesamtstrecke (Pixel), die von allen Zellen zu allen Zeitpunkten an jedem Bildgebungsort zurückgelegt wurde. Die 7. und 8. Zeile enthalten die Durchschnitts- und Standardabweichungswerte für den Gesamtverfahrwinkel (Grad) zwischen dem ersten und dem letzten Zeitpunkt für alle Zellen in einer Bildgebungsstelle. Während die Zeilen 3 bis 8 der Tabellen diese Werte für alle Zellen enthalten, die die Mindestkriterien für die Zeitpunktverfolgung erfüllen, enthalten die Zeilen 9 bis 14 dieselben Werte, jedoch nur für Zellen, die die vom Benutzer angegebenen Filterkriterien erfüllen.
Ergebnisse
Um sicherzustellen, dass unser Analyseskript zuverlässig und konsistent ist, haben wir die Motilität von HaCaT-Keratinozyten in drei unabhängigen Experimenten gemessen. Wir fanden heraus, dass die Standardabweichung der Zellmotilitäten zwar zwischen den Experimenten variabel war (möglicherweise aufgrund der Empfindlichkeit der HaCaT-Zellen gegenüber Konfluenz und mechanischen Reizen), die durchschnittliche Motilität jedoch über mehrere Experimente hinweg konsistent repliziert wurde, ohne dass es zu statistisch signifikanten Unterschieden zwischen den Replikaten kam, wie der t-Test eines zweiseitigen Studenten ergab (Abbildung 2).
Aufgrund der Rolle des Arp2/3-Komplexes bei der Vermittlung der Aktindynamik, der bekannten inhibitorischen Wirkung von α-Catenin auf Arp2/3 und unserer früheren Beobachtung, dass eine Depletion von α-Catenin zu einer erhöhten Motilitätführt 3, untersuchten wir, ob die Hemmung des Arp2/3-Komplexes mit dem niedermolekularen Inhibitor CK-666 die erhöhte Motilität bei α-Catenin-Depletion aufheben würde. Zwei HaCaT-Keratinozyten-Zelllinien, die stabil entweder einen shRNA-Knockdown exprimieren, der auf α-Catenin abzielt, oder eine nicht zielgerichtete shRNA-Kontrolle, die wie zuvor beschrieben erzeugt und validiert wurden8, wurden auf einer 96-Well-Bildgebungsplatte ausgesät.
Wir haben unsere Methode verwendet, um die Motilität von HaCaT-Keratinozyten, die einen α-Catenin-Knockdown exprimieren, mit der Motilität von Kontrollzellen bei einer einzigen Konfluenz zu vergleichen (Abbildung 3). In Übereinstimmung mit einem Modell, in dem α-Catenin die Aktindynamik und die Zellmotilität hemmen kann, indem es mit dem Arp2/3-Komplex um die Aktinbindung konkurriert, beobachteten wir eine signifikante Abnahme der Motilität von α-Catenin-Knockdown-Zellen, aber nicht in Kontrollzellen nach der Behandlung mit dem Arp2/3-Inhibitor CK-666, wenn die Zellen eine moderate bis hohe Konfluenz aufwiesen. Dies deutet darauf hin, dass eine Arp2/3-vermittelte Aktindynamik für die erhöhte Motilität erforderlich ist, die wir in HaCaT-Keratinozyten nach Depletion von α-Catenin sehen, aber nicht für die basale Motilität in Kontrollzellen.
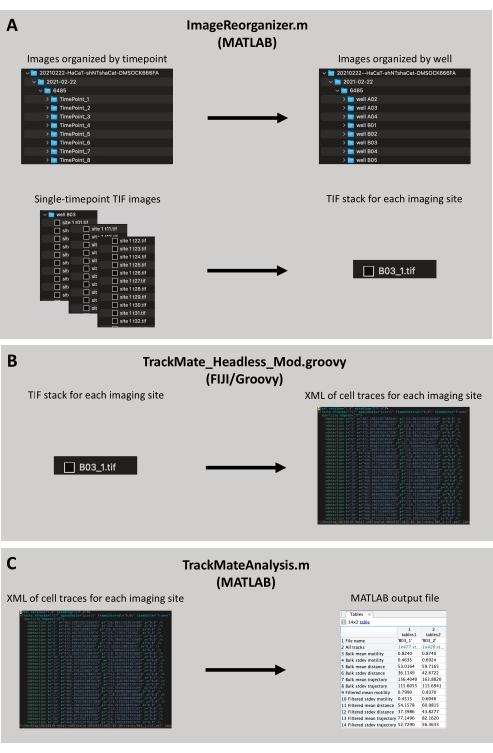
Abbildung 1. Flussdiagramm für das Protokoll der Motilitätsanalyse. (A) Reorganisieren Sie die Rohdateistruktur und konvertieren Sie TIF-Bilder mit einem einzigen Zeitpunkt in einen TIF-Stapel aller Zeitpunkte für jeden Imaging-Standort. (B) Führen Sie TrackMate aus, um eine XML-Datei mit Zellspuren für jede Bildgebungsstelle zu generieren. (C) Filtern Sie Spuren nach der minimalen Anzahl der aufgezeichneten Zeitpunkte und führen Sie eine vergleichende Motilitätsanalyse für alle Bildgebungsstellen durch. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
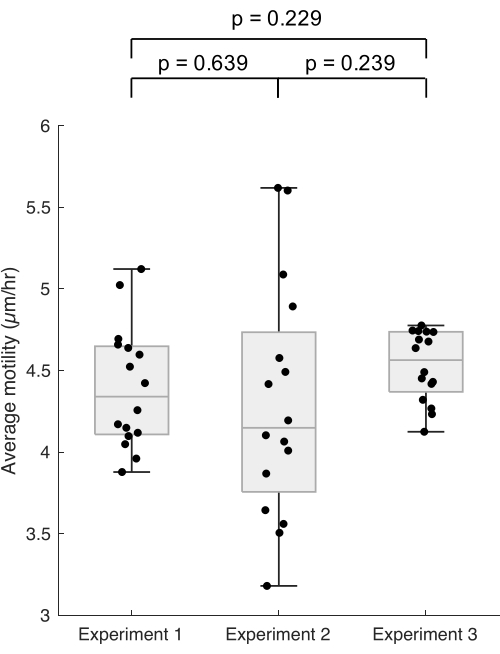
Abbildung 2. Diese Methode ermöglicht eine konsistente und zuverlässige Messung der Motilität in HaCaT-Keratinozyten. HaCaT-Keratinozyten wurden in eine 96-Well-Platte ausgesät und mit einem Zeitpunkt alle 8 Minuten für 100 Zeitpunkte (insgesamt 13 h 20 min) abgebildet. Die Bilder wurden gemäß dem obigen Protokoll analysiert. Dieses Diagramm zeigt die durchschnittliche Motilität an sechzehn replizierten Bildgebungsstellen aus drei unabhängigen Experimenten. Beachten Sie, dass die y-Achse bei 3 μm/h beginnt. In jedem Experiment wurden Messungen in doppelten oder dreifachen Vertiefungen mit zwei Bildgebungsstellen pro Vertiefung durchgeführt, und jeder Datenpunkt repräsentiert die durchschnittliche Motilität aller Zellen in einer Bildgebungsstelle. Die Fehlerbalken stellen die Standardabweichung für alle vier Bildgebungsstellen dar. Die p-Werte wurden durch den t-Test der zweiseitigen Studierenden bestimmt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3. Die Arp2/3-Aktivität ist für eine erhöhte Motilität in HaCaT-Zellen erforderlich, die einen α-Catenin-shRNA-Knockdown exprimieren. Die Zellen wurden mit DMSO (Fahrzeugkontrolle) oder 54 μM CK-666 zum Zeitpunkt 0 behandelt und dann alle 8 Minuten für 100 Zeitpunkte (insgesamt 13 h 20 min) mit einem Zeitpunkt abgebildet. Die Bilder wurden gemäß dem obigen Protokoll analysiert. Dieses Diagramm zeigt die durchschnittliche Motilität an sechzehn replizierten Bildgebungsstellen aus drei unabhängigen Experimenten für jede Bedingung, wobei die Zelldichte unter allen Bedingungen abgestimmt ist. In jedem Experiment wurden Messungen in doppelten oder dreifachen Vertiefungen mit zwei Bildgebungsstellen pro Vertiefung durchgeführt, und jeder Datenpunkt repräsentiert die durchschnittliche Motilität aller Zellen in einer Bildgebungsstelle. Die Fehlerbalken stellen die Standardabweichung für alle vier Bildgebungsstellen dar. Die p-Werte wurden durch den t-Test der zweiseitigen Studierenden bestimmt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
MATLAB-Skripte: Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die oben beschriebene Methodik und die Analysewerkzeuge bieten ein einfaches und skalierbares Mittel zur Messung und Quantifizierung der Motilität von HaCaT-Keratinozyten, das MATLAB verwendet und nur minimale Programmierkenntnisse erfordert. Dieses Protokoll sieht vor, dass die Zellkerne über einen Zeitraum von mindestens 5 Stunden gefärbt und abgebildet werden. Während die Datenerfassung auf jedem geeigneten Mikroskop durchgeführt werden kann, bietet dieses Protokoll ein Skript für die Verarbeitung von Bildern, die mit einem ImageXpress Micro XL-Mikroskop aufgenommen wurden, in einen TIFF-Stack für jede Bildgebungsstelle, der einen Zeitraffer der Zellkernpositionen über alle Zeitpunkte zeigt. Nachdem diese TIFF-Stacks über das TrackMate-Plug-in für ImageJ verarbeitet wurden, ermöglicht dieses Protokoll die Konsolidierung von Ausgabespuren in einer einzigen MATLAB-Zellstruktur, die durchschnittliche Motilitäten, Standardabweichungen und Spuren für jede Bildgebungsstelle und für einzelne Zellen enthält.
Während andere Tools zur Analyse der Zellmotilität verfügbar sind, verwendet diese Methode MATLAB, um die Motilitätsraten für Zellen zu bestimmen, die mit dem ImageJ TrackMate-Plugin auf einfache und skalierbare Weise verfolgt werden. Diese Bildanalyse-Pipeline ist rechnerisch effizient genug, um auf einem PC mit einem Windows- oder iOS-Betriebssystem ausgeführt zu werden. Während die bereitgestellten Skripts für die Verwendung auf einem iOS-Computer geschrieben wurden, funktionieren sie auf einem Windows-Computer, sobald die Pfadnamen aktualisiert wurden, um umgekehrte Schrägstriche anstelle von Schrägstrichen zu enthalten. Das Importieren von XML-Dateien für das Analyse- und Konsolidierungsskript nach TrackMate kann zeitintensiv sein, aber das Ergebnis ist eine einzige MATLAB-Zellstruktur, die kleiner zu speichern und leichter zugänglich ist als XML-Dateien für alle Imaging-Standorte.
Die in diesem Manuskript beschriebene Methode schafft Möglichkeiten für umfassende Studien über Regulatoren der zellulären Motilität und könnte ein tieferes Verständnis der Signalübertragung ermöglichen, die die Motilität in HaCaT-Keratinozyten steuert. Aufgrund der Rolle der Keratinozytenmotilität bei der Wundheilung und Tumorgenese hat ein besseres Verständnis ihrer Regulation das Potenzial für klinische Translationen, die die Ergebnisse für die menschliche Gesundheit verbessern.
Offenlegungen
X.L. und die University of Colorado Boulder haben ein finanzielles Interesse an der Entwicklung von HDAC-Inhibitoren für Therapeutika und Eigenkapital an OnKure. X.L. ist Mitbegründer und Mitglied des Vorstands von OnKure, das proprietäre HDAC-Inhibitoren von der University of Colorado Boulder lizenziert hat. OnKure ist weder am Versuchsdesign noch an der Finanzierung dieser Studie beteiligt.
Danksagungen
Wir danken Daniel Messenger, Lewis Baker, Douglas Chapnick, Adrian Ramirez, Quanbin Xu und anderen Mitgliedern der Labore Liu und Bortz für ihre Einblicke und Ratschläge. Wir danken Jian Tay für das Teilen seiner MATLAB-Expertise und für das Schreiben der Funktion für den XML-Import. Wir danken Joseph Dragavon vom BioFrontiers Advanced Light Microscopy Core für seine Unterstützung bei der Mikroskopie und Bildgebung. Wir danken Theresa Nahreini und Nicole Kethley von der Cell Culture Core Facility für ihre Unterstützung bei der Zellkultur. Diese Arbeit wurde durch Zuschüsse des National Cancer Institute und des National Institute of Arthritis and Musculoskeletal and Skin Diseases der National Institutes of Health (R01AR068254) an X. L. und das National Institute of General Medical Sciences (NIGMS) unterstützt, R01GM126559 an D.B. und X. L. G.E.W. und E.N.B. wurden durch ein Predoctoral Training Grant des NIGMS (T32GM08759) unterstützt. Der ImageXpress Micro XL wurde vom National Center for Research Resources (S10 RR026680) unterstützt. FACSAria wurde von den National Institutes of Health (S10OD021601) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 96-well flat clear bottom black polystyrine TC-treated microplates, individually wrapped, with lid, sterile | Corning | 3603 | |
| Dulbecco's modified eagle medium (high D-glucose) | Life Technologies Corporation/Thermo Fisher Scientific | 12800-082 | |
| Fetal bovine serum | Sigma-Aldrich Inc | F0926 | |
| Fluorobrite Dulbecco's modified eagle medium (high D-glucose, 3.7 g/L sodium bicarbonate, no L-glutamine, no phenol red) | Gibco/Thermo Fisher Scientific | A18967-01 | |
| GlutaminePlus | R&D Systems Inc. | R90210 | |
| Hoechst 33342, trihydrochloride, trihydrate | Invitrogen/Thermo Fisher Scientific | H21492 | |
| Penicillin streptomycin | Life Technologies Corporation/Thermo Fisher Scientific | 15140-122 | |
| Phosphate buffered saline | Gibco/Thermo Fisher Scientific | 14190-144 |
Referenzen
- Li, L., He, Y., Zhao, M., Jiang, J. Collective cell migration: Implications for wound healing and cancer invasion. Burns & Trauma. 1 (1), 21-26 (2015).
- Stuelten, C. H., Parent, C. A., Montell, D. J. Cell motility in cancer invasion and metastasis: insights from simple model organisms. Nature Reviews Cancer. 18 (5), 296-312 (2018).
- Campbell, K., et al. Collective cell migration and metastases induced by an epithelial-to-mesenchymal transition in Drosophilaintestinal tumors. Nature Communications. 10 (1), 1-10 (2019).
- Devreotes, P., Horwitz, A. R. Signaling networks that regulate cell migration. Cold Spring Harbor Perspectives in Biology. 7 (8), (2015).
- Parri, M., Chiarugi, P. Rac and Rho GTPases in cancer cell motility control. Cell Communication and Signaling. 8 (1), 23(2010).
- Sumida, G. M., Yamada, S. Rho GTPases and the downstream effectors actin- related protein 2/3 (Arp2/3) complex and myosin II induce membrane fusion at self-contacts. The Journal of Biological Chemistry. 290 (6), 3238-3247 (2015).
- Alberts, B., et al. Cell junctions. Molecular Biology of the Cell., 4th edition. , (2002).
- Bunker, E. N., Wheeler, G. E., Chapnick, D. A., Liu, X. Suppression of α-catenin and adherens junctions enhances epithelial cell proliferation and motility via TACE- mediated TGF-α autocrine/paracrine signaling. Molecular Biology of the Cell. 32 (4), 348-361 (2021).
- Niessen, C. M. Tight junctions/adherens junctions: basic structure and function. Journal of Investigative Dermatology. 127 (11), 2525-2532 (2007).
- Drees, F., Pokutta, S., Yamada, S., Nelson, W. J., Weis, W. I. α-Catenin is a molecular switch that binds E-cadherin-β-catenin and regulates actin-filament assembly. Cell. 123 (5), 903-915 (2005).
- Yamada, S., Pokutta, S., Drees, F., Weis, W. I., Nelson, W. J. Deconstructing the cadherin-catenin-actin complex. Cell. 123 (5), 889-901 (2005).
- Rangarajan, E., Izard, T. The cytoskeletal protein α-catenin unfurls upon binding to vinculin. The Journal of Biological Chemistry. 287 (22), 18492-18499 (2012).
- Tinevez, J. -Y., Herbert, S. The NEMO dots assembly: single-particle tracking and analysis. Bioimage Data Analysis Workflows. , 67-97 (2020).
- Visweshwaran, S. P., Maritzen, T. A simple 3D cellular chemotaxis assay and analysis workflow suitable for a wide range of migrating cells. MethodsX. 6, 2807-2821 (2019).
- Wu, P. -H., Giri, A., Wirtz, D. Statistical analysis of cell migration in 3D using the anisotropic persistent random walk model. Nature Protocols. 10, 517-527 (2015).
- Chapnick, D. A., Jacobsen, J., Liu, X. The development of a novel high throughput computational tool for studying individual and collective cellular migration. PloSOne. 8 (12), 82444(2013).
- Tinevez, J. -Y., et al. TrackMate: an open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Vargas Aguilera, C. A. int2strz.m. MATLAB Commons. , (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten
Wir verwenden Cookies, um Ihre Erfahrung auf unserer Website zu verbessern.
Indem Sie unsere Website weiterhin nutzen oder auf „Weiter“ klicken, stimmen Sie zu, unsere Cookies zu akzeptieren.