Method Article
Optische Pinzette untersucht RNA-Protein-Interaktionen in der Translationsregulation
In diesem Artikel
Zusammenfassung
Dieses Protokoll stellt einen vollständigen experimentellen Workflow für die Untersuchung von RNA-Protein-Interaktionen mit optischen Pinzetten dar. Mehrere mögliche Versuchsanordnungen werden skizziert, einschließlich der Kombination von optischen Pinzetten mit konfokaler Mikroskopie.
Zusammenfassung
RNA nimmt verschiedene strukturelle Falten an, die für ihre Funktionen essentiell sind und dadurch verschiedene Prozesse in der Zelle beeinflussen können. Darüber hinaus kann die Struktur und Funktion einer RNA durch verschiedene transaktive Faktoren wie Proteine, Metaboliten oder andere RNAs moduliert werden. Frameshifting-RNA-Moleküle zum Beispiel sind regulatorische RNAs in kodierenden Regionen, die Ribosomen direkt in einen alternativen offenen Leserahmen übersetzen und dadurch als Genschalter fungieren. Sie können auch verschiedene Falten annehmen, nachdem sie an Proteine oder andere Transfaktoren gebunden sind. Um die Rolle von RNA-bindenden Proteinen bei der Translation zu analysieren und wie sie die RNA-Struktur und -Stabilität modulieren, ist es wichtig, das Zusammenspiel und die mechanischen Eigenschaften dieser RNA-Protein-Komplexe gleichzeitig zu untersuchen. Diese Arbeit zeigt, wie man mit einer einzelnen Molekül-Fluoreszenz-gekoppelten optischen Pinzette die Konformations- und thermodynamische Landschaft von RNA-Protein-Komplexen mit hoher Auflösung erforscht. Als Beispiel wird die Interaktion des SARS-CoV-2 programmierten ribosomalen Frameshifting-Elements mit der trans-wirkenden Faktor-Kurzisoform des antiviralen Zinkfingerproteins herausgearbeitet. Darüber hinaus wurden fluoreszenzmarkierte Ribosomen mit der konfokalen Einheit überwacht, was letztendlich die Untersuchung der Translationsdehnung ermöglichen würde. Der fluoreszenzgekoppelte OT-Assay kann weit verbreitet eingesetzt werden, um verschiedene RNA-Proteinkomplexe oder trans-wirkende Faktoren zu erforschen, die die Translation regulieren, und könnte Studien zur RNA-basierten Genregulation erleichtern.
Einleitung
Die Übertragung genetischer Information von DNA auf Proteine durch mRNAs ist ein komplexer biochemischer Prozess, der auf allen Ebenen durch makromolekulare Interaktionen innerhalb von Zellen präzise reguliert wird. Für die translationale Regulation spielen RNA-Protein-Interaktionen eine entscheidende Rolle, um schnell auf verschiedene Reize und Signale zu reagieren1,2. Einige RNA-Protein-Interaktionen beeinflussen die mRNA-Stabilität und verändern dadurch die Zeit, in der eine RNA translational aktiv ist. Andere RNA-Protein-Interaktionen sind mit Rekodierungsmechanismen wie Stop-Codon-Readthrough, Bypassing oder Programmed Ribosomal Frameshifting (PRF)3,4,5,6,7 assoziiert. Kürzlich wurde gezeigt, dass eine Reihe von RNA-bindenden Proteinen (RBPs) mit stimulierenden mRNA-Elementen und der Translationsmaschinerie interagieren, um zu bestimmen, wann und wie viel Rekodierung in der Zelle stattfinden wird7,8,9,10,11. Um die Rolle von RNA-bindenden Proteinen bei der Translation und deren Modulation der RNA-Struktur und -Stabilität zu analysieren, ist es daher von entscheidender Bedeutung, die Interaktionsprinzipien und mechanischen Eigenschaften dieser RNA-Proteinkomplexe im Detail zu untersuchen.
Jahrzehntelange Arbeit hat den Grundstein für die Untersuchung des mehrstufigen und mehrkomponentigen Prozesses der Translation gelegt, der auf einer komplizierten Kommunikation zwischen den RNA- und Proteinkomponenten der Translationsmaschinerie beruht, um Geschwindigkeit und Genauigkeit zu erreichen12,13,14. Ein entscheidender nächster Schritt zum Verständnis komplexer regulatorischer Ereignisse ist die Bestimmung der Kräfte, Zeitskalen und strukturellen Determinanten während der Translation mit hoher Genauigkeit12,15,16,17. Die Untersuchung der RNA-Konformationsdynamik und insbesondere wie trans-wirkende Hilfsfaktoren während der Translation auf die RNA-Struktur wirken, wurde durch das Aufkommen von Einzelmolekülwerkzeugen, einschließlich optischer Pinzetten oder Nullmodus-Wellenleiter, weiter beleuchtet16,17,18,19,20,21,22,23,24 ,25,26.
Optische Pinzetten (OT) stellen eine hochpräzise Einzelmolekültechnik dar, die angewendet wurde, um viele Arten von RNA-abhängigen dynamischen Prozessen einschließlich Transkription und Translation zu untersuchen26,27,28,29,30,31,32. Die Verwendung einer optischen Pinzette hat es ermöglicht, molekulare Wechselwirkungen, Nukleinsäurestrukturen und thermodynamische Eigenschaften, Kinetik und Energetik dieser Prozesse im Detail zu untersuchen16,17,22,33,34,35,36,37,38,39 . Der optische Pinzetten-Assay basiert auf dem Einklemmen mikroskopischer Objekte mit einem fokussierten Laserstrahl. In einem typischen OT-Experiment wird das interessierende Molekül zwischen zwei transparenten (normalerweise Polystyrol-) Kügelchen gebunden (Abbildung 1A)27. Diese Perlen werden dann von optischen Fallen aufgefangen, die sich wie Federn verhalten. So kann die auf das Molekül ausgeübte Kraft basierend auf der Verschiebung der Perle aus dem Zentrum des fokussierten Laserstrahls (Fallenzentrum) berechnet werden. Kürzlich wurden optische Pinzetten mit konfokaler Mikroskopie kombiniert (Abbildung 1B), was Fluoreszenz- oder Förster-Resonanzenergietransfermessungen (FRET) ermöglicht40,41,42. Dies eröffnet ein völlig neues Feld möglicher Experimente, die eine gleichzeitige Messung und damit eine präzise Korrelation von Kraftspektroskopie und Fluoreszenzdaten ermöglichen.
Hier demonstrieren wir Experimente mit der optischen Pinzette in Kombination mit konfokaler Mikroskopie, um Protein-RNA-Interaktionen zu untersuchen, die die translationale Frameshifting regulieren. Zwischen objektiv und Kondensator ermöglicht eine Durchflusszelle mit fünf Kanälen eine kontinuierliche Probenapplikation mit laminarer Strömung. Durch die mikrofluidischen Kanäle können verschiedene Komponenten direkt injiziert werden, was die Hands-on-Zeit verkürzt und während des gesamten Experiments einen sehr geringen Probenverbrauch ermöglicht.
Zunächst wird eine grundlegende Richtlinie zur Unterstützung des Designs von OT-Experimenten vorgeschlagen und Vorteile sowie Fallstricke verschiedener Setups diskutiert. Als nächstes werden die Aufbereitung von Proben und experimentelle Workflows beschrieben und ein Protokoll für die Datenanalyse bereitgestellt. Um ein Beispiel darzustellen, skizzieren wir die Ergebnisse von RNA-Dehnungsexperimenten zur Untersuchung des SARS-CoV-2 frameshifting RNA-Elements (Abbildung 2A) mit dem trans-agierenden Faktor, der kurzen Isoform des antiviralen Zinkfingerproteins (ZAP), das die Translation der viralen RNA aus einem alternativen Leserahmen verändert43. Darüber hinaus wird gezeigt, dass fluoreszenzmarkierte Ribosomen in diesem OT-konfokalen Assay verwendet werden können, was nützlich wäre, um die Prozessivität und Geschwindigkeit der Translationsmaschinerie zu überwachen. Die hier vorgestellte Methode kann verwendet werden, um die Wirkung verschiedener Puffer, Liganden oder anderer zellulärer Komponenten schnell zu testen, um verschiedene Aspekte der Translation zu untersuchen. Schließlich werden häufige experimentelle Fallstricke und deren Behebung diskutiert. Im Folgenden werden einige entscheidende Punkte im experimentellen Design skizziert.
Konstruktionsdesign
Prinzipiell gibt es zwei gängige Ansätze, um ein OT-kompatibles RNA-Konstrukt zu erstellen. Der erste Ansatz verwendet ein langes RNA-Molekül, das mit komplementären DNA-Handles hybridisiert wird, wodurch ein Konstrukt aus zwei RNA/DNA-Hybridregionen entsteht, die eine einzelsträngige RNA-Sequenz in der Mitte flankieren (Abbildung 2B). Dieser Ansatz wird in den meisten OT-RNA-Experimenten verwendet33,44,45.
Der zweite Ansatz nutzt dsDNA-Griffe mit kurzen (ca. 20 nt) Überhängen15,17. Diese Überhänge werden dann mit dem RNA-Molekül hybridisiert. Obwohl komplizierter im Design, überwindet die Verwendung von dsDNA-Handles einige Einschränkungen des DNA / RNA-Hybridsystems. Prinzipiell können auch sehr lange Griffe (>10kb) realisiert werden, was für konfokale Messungen komfortabler ist. Darüber hinaus kann das RNA-Molekül an DNA-Handles ligiert werden, um die Tether-Stabilität zu erhöhen.
End-Labeling-Strategie
Das Konstrukt muss über eine starke molekulare Wechselwirkung an Perlen gebunden werden. Während es Ansätze für die kovalente Bindung von Griffen an Perlen46 gibt, werden starke, aber nicht-kovalente Wechselwirkungen wie Streptavidin-Biotin und Digoxigenin-Antikörper häufig in OT-Experimenten verwendet15,33,35,45. Im beschriebenen Protokoll wird das Konstrukt mit Biotin oder Digoxigenin markiert, und die Kügelchen sind mit Streptavidin bzw. Antikörpern gegen Digoxigenin beschichtet (Abbildung 1A). Dieser Ansatz wäre für die Anwendung von Kräften bis zu etwa 60 pN (per Tether)47 geeignet. Darüber hinaus ermöglicht die Verwendung unterschiedlicher 5'- und 3'-Markierungsstrategien die Bestimmung der Ausrichtung des zwischen den Kügelchen gebildeten Seils17.
Proteinmarkierung für Fluoreszenzmessungen
Für die konfokale Bildgebung gibt es mehrere häufig verwendete Ansätze zur Fluoreszenzmarkierung. Zum Beispiel können Fluorophore kovalent an Aminosäurereste gebunden sein, die nativ in Proteinen vorkommen oder durch ortsgerichtete Mutagenese durch eine reaktive organische Gruppe eingeführt werden. Thiol- oder Amin-reaktive Farbstoffe können zur Markierung von Cystein- bzw. Lysinresten verwendet werden. Es gibt mehrere reversible Schutzmethoden, um die Spezifität der Markierung zu erhöhen48,49, jedoch würden native Proteine typischerweise an mehreren Rückständen markiert. Obwohl die geringe Größe des Fluorophors einen Vorteil bieten kann, kann eine unspezifische Markierung die Proteinaktivität beeinträchtigen und somit die Signalintensität variieren49. Abhängig von der Markierungseffizienz kann die Signalintensität zwischen verschiedenen Experimenten variieren. Daher sollte vor dem Experiment eine Aktivitätsprüfung durchgeführt werden.
Falls das Protein von Interesse ein N- oder C-terminales Tag enthält, wie z. B. ein His-Tag oder ein Streptokokken-Tag, stellt die spezifische Kennzeichnung dieser Tags einen weiteren beliebten Ansatz dar. Darüber hinaus verringert die markierungsorientierte Markierung die Wahrscheinlichkeit, dass das Fluorophor die Proteinaktivität stört, und kann die Löslichkeit verbessern49. Die Tag-spezifische Markierung führt jedoch in der Regel zu monofluorophor markierten Proteinen, die schwer nachzuweisen sein können. Eine andere Möglichkeit der spezifischen Markierung kann durch den Einsatz von Antikörpern erreicht werden.
Aufbau der Mikrofluidik
Die Kombination von OT mit einem Mikrofluidik-System ermöglicht einen schnellen Übergang zwischen verschiedenen experimentellen Bedingungen. Darüber hinaus nutzen Stromsysteme die Aufrechterhaltung der laminaren Strömung in der Durchflusszelle, was das Mischen von Flüssigkeiten aus anderen Kanälen in senkrechter Richtung relativ zur Strömungsrichtung verhindert. Daher ist die laminare Strömung besonders vorteilhaft für das experimentelle Design. Derzeit werden häufig Flusszellen mit bis zu 5 Kanälen eingesetzt (Abbildung 3).
Protokoll
1. Probenvorbereitung
- Klonen Sie die interessierende Sequenz in den Vektor, der die Lambda-DNA-Fragmente enthält, die als Griffsequenzen dienen (Abbildung 2)43,50.
- Generieren Sie zunächst eine DNA-Vorlage für die anschließende In-vitro-Transkription mittels PCR (Abbildung 2B; Reaktion 1). Bei diesem PCR-Schritt wird der T7-Promotor am 5'-Ende des Sinnes-DNA-Moleküls 32,33,43,50 hinzugefügt. Stellen Sie die PCR-Reaktion gemäß Tabelle 1 ein. Führen Sie die PCR in 50 μL Aliquots mit entsprechenden Zyklen im Thermocycler durch.
- Die Griffe werden durch zwei getrennte PCR-Reaktionen vorbereitet (Tabelle 1, Abbildung 2B; Reaktion 2 und 3). Generieren Sie zuerst den 5'-Griff per PCR. Erzeugen Sie dann den 3'-Griff und markieren Sie ihn gleichzeitig mit Digoxigenin, indem Sie einen 5'-Digoxigenin-markierten Primer verwenden32,33,43,50.
- Nach der PCR reinigen Sie die DNA mit Quarzspinsäulen.
- Durchführung der In-vitro-Transkriptionsreaktion unter Verwendung der T7-RNA-Polymerase (Tabelle 2)32,33,43,50. Inkubieren Sie die Reaktion bei 37 °C für 2-4 h in Abhängigkeit von der Länge der RNA. Als nächstes DNase I in die Reaktion geben und bei 37 °C für 30 min inkubieren, um die DNA-Vorlage zu verdauen. Reinigen Sie die RNA mit Silica-Spin-Säulen.
- Während der Markierungsreaktion des 5'-Griffs (Tabelle 3) Wird Biotin-16-dUTP am 3'-Ende des Griffs durch T4-DNA-Polymerase38,50 zugegeben. Führen Sie die Reaktion bei Raumtemperatur für 1-2 h durch. Anschließend reinigen Sie die DNA mit Silica-Spin-Säulen.
HINWEIS: Da der 5'-Griff an seinem 3'-Ende gekennzeichnet werden muss (Abbildung 2B), kann die Beschriftung während der PCR nicht durchgeführt werden. - Mischen Sie die oben genannten Komponenten - 5' Griff (3' markiert mit Biotin), 3' Griff (5' markiert mit Digoxigenin) und RNA - in einem molaren Verhältnis von 1:1:1 im Glühpuffer (80% Formamid, 400 mM NaCl, 40 mM HEPES, pH 7,5, 0,5 mM EDTA, pH 8), um den gewünschten RNA/DNA-Hybrid zu erhalten (Tabelle 4). Das Glühgemisch 10 min auf 85 °C erhitzen und dann langsam auf 4 °C abkühlen lassen.
- Mischen Sie die geglühte Probe mit 1/10 Volumen von 3 M Natriumacetat (pH 5), 3 Volumen eiskaltem Ethanol und inkubieren Sie bei -80 °C für mindestens 1 h oder bei -20 °C über Nacht.
- Zentrifugieren Sie die Proben bei 15.000 × g für 30 min bei 4 °C. Entsorgen Sie den Überstand und trocknen Sie das Pellet (normalerweise nicht sichtbar) unter Vakuum.
- Zum Schluss resuspend, das Pellet in 50 μL RNase-freiem Wasser resuspend und Aliquots herstellen. Die Aliquots bis zum Gebrauch bei -80 °C lagern. Zur Kurzzeitlagerung können die Proben auch bei -20 °C gelagert werden.
2. Geräteeinrichtung
HINWEIS: Das folgende Protokoll ist für das kommerzielle optische Pinzetteninstrument C-Trap der Firma LUMICKS optimiert. Daher können Anpassungen an den vorgestellten Schritten bei der Verwendung anderer optischer Pinzetteninstrumente erforderlich sein. Wenn es nicht verwendet wird, wird das Mikrofluidiksystem der Maschine in Bleichmittel (Natriumhypochloritlösung) aufbewahrt und muss vor Gebrauch gewaschen werden.
- Entsorgen Sie das Bleichmittel und füllen Sie die Spritzen mit 1 ml RNase-freiem Wasser.
- Fügen Sie 50 μL 0,5 M Natriumthiosulfat zu mindestens 1 mL des RNase-freien Wassers hinzu und waschen Sie das System gründlich (1 bar, mindestens 0,5 ml), um das verbleibende Bleichmittel im System zu entfernen.
- Verwerfen Sie die Natriumthiosulfatlösung aus den Spritzen. Ersetzen Sie die Spritzen durch frische und waschen Sie das System mit mindestens 0,5 ml RNase-freiem Wasser.
HINWEIS: Achten Sie darauf, dass das Mikrofluidiksystem niemals trocken läuft, um Luftblasen im System zu vermeiden. - Geben Sie 2 Tropfen Tauchöl (Brechungsindex von 1,33) oder ca. 70 μL Wasser auf das Objektiv.
- Platzieren Sie die Durchflusszelle in ihrer Position im Halterahmen.
- Geben Sie 2 Tropfen Tauchöl (Brechungsindex von 1,51) auf die Durchflusszelle.
- Schalten Sie das Lasergerät in der Pinzette ein. Sobald es ausgeführt wird, schalten Sie den Überfanglaser in der Softwareoberfläche zu 100% ein.
- Stellen Sie mit Diagnosekameras (Z-Finder) die Z-Achse durch Drehen der Mikroschraube in die Mitte der Kammer zwischen der zweiten und der dritten Reflexion (Grenzflächen) ein, wo die Brechungsringe am größten sind.
HINWEIS: Jedes Mal, wenn das Objektiv näher an die Messkammer herangeführt wird und die Fokusebene des Objektivs die Grenzfläche zwischen zwei Phasen kreuzt, kann im Z-Finder-Modus eine Reflexion erkannt werden. Es sind 4 Schnittstellen möglich: (i) Wasser / Tauchöl und unteres Glas (ii) unteres Glas und Puffer in der Kammer (iii) Puffer in der Kammer und oberes Glas (iv) oberes Glas und Tauchöl für Kondensator. - Stellen Sie die Kondensatorposition ein (Stellen Sie den Fanglaser auf ca. 50%), so dass der Kondensator das Tauchöl oben auf der Messkammer berührt.
- Stellen Sie den Fokus ein, indem Sie sich mit dem Kondensator langsam nach unten/oben bewegen, so dass im Mondmodus ca. 10 Lichtbänder angezeigt werden (Diagnosekameras).
3. Probenmessung
- Inkubieren Sie Anti-Digoxigenin-beschichtete Kügelchen (AD) mit den Probenkonstrukten (3 μL 0,1% (w/v) AD-Perlensuspension + 4 μL Probe) und mit 1 μL RNase-Inhibitoren und 8 μL des Assay-Puffers (300 mM KCl, 5 mM MgCl2, 20 mM HEPES, pH 7,6, 0,05% Tween 20, 5 mM DTT) bei RT für 10-20 min. Nach der Inkubation verdünnen Sie die Probe in 500 μL Assay-Puffer.
HINWEIS: Es wird empfohlen, Sauerstofffänger, insbesondere während Fluoreszenzmessungen, in den Puffer zu geben, um oxidative Schäden zu vermeiden. Hier wurde ein Sauerstofffängersystem verwendet, das Glukose (8,3 mg/ml), Glucoseoxidase (40 U/ml) und Katalase (185 U/ml) enthielt. - Mischen Sie 0,8 μL 1% (w/v) Streptavidin-beschichtete (SA) Perlen mit 1 ml Assay-Puffer.
- Entsorgen Sie Wasser aus den Spritzen und füllen Sie die Spritzen mit entsprechenden Suspensionen/Lösungen. Mindestens 2 Minuten bei ca. 1 bar waschen und dann mit dem Fangen von Perlen beginnen.
HINWEIS: Je nach Versuchsaufbau können unterschiedliche Kanalanordnungen verwendet werden (Abbildung 3). Typischerweise ist ein Strömungskanal mit Anti-Digoxigenin-Kügelchen gefüllt, die das RNA-Molekül tragen. Ein zweiter Kanal ist mit den mit Streptavidin beschichteten Perlen gefüllt. Pufferkanal wird verwendet, um die Tethers zu bilden. Ein vierter Kanal kann verwendet werden, um das RNA-bindende Protein zu laden (Abbildung 3C), oder alternativ kann RBP direkt im Pufferkanal hinzugefügt werden (Abbildung 3B). - Um die Perlen zu erfassen, bewegen Sie die optischen Fallen voneinander getrennt. Bewegen Sie sich zuerst zum AD-Kanal und fangen Sie eine AD-Perle in Falle 1. Als nächstes bewegen Sie die Stufe zum SA-Kanal und fangen eine einzelne SA-Perle durch Falle 2.
HINWEIS: Versuchen Sie, an der Schnittstelle der Puffer- und Perlenkanäle zu bleiben, um zu vermeiden, dass die bereits gefangene Perle verloren geht, oder um zu verhindern, dass mehrere Perlen durch dieselbe Falle gefangen werden. - Sobald die Perlen der richtigen Größe erfasst sind, bewegen Sie sich zum Pufferkanal und stoppen Sie den laminaren Fluss. Führen Sie als Nächstes eine Kraftkalibrierung durch, um die Fallensteifigkeit zu überprüfen. Die jeweiligen Steifigkeitswerte sollten sich in der x/y-Achse nicht um mehr als 10-15% unterscheiden.
HINWEIS: Passen Sie die Laserleistung oder den Lasersplit zwischen den Fallen entsprechend der Perlengröße an. Die Kraftkalibrierung muss nicht für jedes Perlenpaar durchgeführt werden, solange die Perlenschablonen übereinstimmen (Ähnlichkeitswert > 0,9). Es sollte jedoch regelmäßig oder zumindest jedes Mal durchgeführt werden, wenn die Assay-Bedingungen geändert werden. - Fangen Sie an, nach einem Seil zu fischen, indem Sie die Perlen nahe beieinander bewegen, einige Sekunden warten und sie dann wieder auseinander bewegen, wiederholen, bis ein Seil gebildet ist. Eine Tetherbildung führt zu einer Erhöhung der gemessenen Kraft, wenn die beiden Perlen voneinander weggezogen werden.
HINWEIS: Um die Bildung mehrerer Haltegurte zu vermeiden, sollten die Perlen nicht zu nahe bewegt werden. Nach dem Fangen eines Seils zwischen den beiden Perlen kann die Tetherqualität überprüft werden, indem das überdehnte Plateau gefunden wird. Das Plateau sollte zwischen 50 und 60 pN für ein einzelnes Seil liegen. - Nachdem Sie ein Halteband erhalten haben, starten Sie die Messung. Je nach untersuchtem Phänomen sollten unterschiedliche Messaufbauten gewählt werden (Abbildung 1B-D).
HINWEIS: Normalerweise wird zu Beginn des Experiments ein Force-Ramp-Experiment durchgeführt, um die Tether-Qualität zu überprüfen und das Verhalten zu untersuchen. Danach kann man auch die Experimente mit konstanter Kraft oder konstanter Position starten, um die Zustandsübergänge weiter zu untersuchen. Sobald eine ausreichende Anzahl von Messungen an einer RNA-Probe durchgeführt wurde, um ihr Verhalten zu bestimmen, können markierte Faktoren zum System hinzugefügt werden, um konfokale Messungen durchzuführen. - Um Fluoreszenzmessungen durchzuführen, schalten Sie die konfokalen Laser und die Photonenzähleinheit im optischen Pinzetteninstrument ein.
- Schalten Sie den Anregungslaser der gewünschten Wellenlänge in der Softwareschnittstelle ein und stellen Sie die Leistung des Lasers je nach Fluorophor auf 5% oder höher ein.
HINWEIS: Senken Sie während der Nichtmessung die Leistungseinstellung des Anregungslasers auf 0%, um eine übermäßige Photoschädigung der Probe zu vermeiden. - Beginnen Sie mit der Bildgebung des Beispiels mithilfe der Bildfunktionen der Software.
HINWEIS: Um gut fokussierte Bilder zu erhalten, müssen die Brennebene des konfokalen Mikroskops und der optischen Fallen ausgerichtet werden. Zu diesem Zweck kann die Autofluoreszenz der Polystyrolperlen im blauen Laserkanal eingesetzt werden. Die Brennebene optischer Fallen wird in der z-Achse nach oben oder unten bewegt, bis das Bild der Perlen seinen höchsten Durchmesser erreicht. An dieser Stelle kann das Fluoreszenzsignal des zwischen den Kügelchen gebundenen Moleküls gemessen werden. - Um die Kymographenfunktion zu verwenden, geben Sie die x-y-Position der Kymographenachse an, damit sie die Erkennung des Seils zwischen den Kügelchen ermöglicht.
- Während der gesamten Messung kann die Pufferzusammensetzung leicht geändert werden, indem entweder die Kügelchen in verschiedene Kanäle bewegt oder der im Mikrofluidiksystem zugeführte Puffer geändert wird.
4. Datenanalyse
- Rohdaten-Vorverarbeitung
- Mithilfe eines einfachen Skripts können Sie die Rohdaten so weit herunterrechnen, dass (i) eine schnellere nachfolgende Datenverarbeitung möglich ist, aber (ii) dennoch alle kritischen Informationen enthalten sind (Abbildung 4A). Normalerweise sind 100-5000 Hz für diesen Zweck geeignet.
HINWEIS: Die Datenerfassungsfrequenz in optischen Pinzettenexperimenten ist oft höher als für die Analyse erforderlich - in den vorgestellten Experimenten ist die Datenerfassungsfrequenz standardmäßig auf 78 125 Hz eingestellt. Da der Speicherplatz begrenzt ist, ist es praktisch und zeitsparend, die Abtastrate der Daten zu reduzieren. Hier wurden die Rohdaten um den Faktor 30 heruntergerechnet. - Verwenden Sie als Nächstes einen Signalfilter, um das hochfrequente Messrauschen des Signals zu reduzieren (Abbildung 4A). Passen Sie den Filtergrad und die Parameter der Grenzfrequenz entsprechend an, um die Datenausgabe verschiedener Experimente zu optimieren (Abbildung 5).
HINWEIS: Unter den Signalfiltern ist Butterworth filter51 einer der am weitesten verbreiteten. Ein benutzerdefiniertes Python-Skript, das die Vorverarbeitung von Rohdaten ermöglicht, wird in den ergänzenden Daten bereitgestellt. Downsampling- und Signalfilterungsparameter (Grenzfrequenz, Filtergrad) müssen für verschiedene Experimente optimiert werden.
- Mithilfe eines einfachen Skripts können Sie die Rohdaten so weit herunterrechnen, dass (i) eine schnellere nachfolgende Datenverarbeitung möglich ist, aber (ii) dennoch alle kritischen Informationen enthalten sind (Abbildung 4A). Normalerweise sind 100-5000 Hz für diesen Zweck geeignet.
- Führen Sie für die Force-Ramp-Datenanalyse die folgenden Schritte aus.
- Markieren Sie die Schritte entweder manuell, indem Sie die entsprechenden Punkte im Kraftleitkurvendiagramm suchen oder benutzerdefinierte Skripts verwenden. Entfaltungsschritte zeichnen sich durch einen plötzlichen Kraftabfall in Kombination mit einer Erhöhung des Abstands in der Kraft-Weg-Kurve (FD) aus.
- Sobald sich entfaltende Ereignisse markiert sind, passen Sie verschiedene Bereiche der FD-Kurve mit geeigneten Modellen an (Abbildung 4D).
HINWEIS: Für den Bereich vor dem ersten Entfaltungsschritt kann das Seil als "doppelsträngig" betrachtet werden und wird üblicherweise mit einem erweiterbaren Wurm-ähnlichen Kettenmodell (WLC) 47,52,53 angepasst. Die Teile nach dem ersten sich entfaltenden Ereignis gelten als eine Kombination aus doppelsträngigen Nukleotiden (Griffen) und einzelsträngigen Nukleotiden (entfaltetes RNA-Molekül). Daher ist die Datenanpassung komplexer - in der Regel wird eine Kombination aus 2 WLC-Modellen oder WLC- und freigelenkten Kettenmodellen (FJC) verwendet36,39,52. Das erweiterbare WLC-Modell hat zwei Hauptpassungsparameter, die Konturlänge (LC) und die Persistenzlänge (LP). Die Konturlänge entspricht der Länge des vollständig gedehnten Moleküls und die Persistenzlänge definiert die Biegeeigenschaften des interessierenden Moleküls. Das Modell kann mit der folgenden Gleichung (1) beschrieben werden. WLC kann verwendet werden, um das Verhalten sowohl von gefalteten als auch von entfalteten Regionen zu modellieren, obwohl für jeden dieser Bereiche ein separates Modell mit unterschiedlichen Parametern verwendet werden muss.
(1)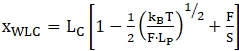
wobei x die Erweiterung, LC die Konturlänge, F die Kraft, LP die Persistenzlänge, kB die Boltzmann-Konstante, T die thermodynamische Temperatur und S der Dehnungsmodul ist.
Das zweite Modell namens Freigelenkte Kette (FJC) wird häufig verwendet, um das Verhalten von entfalteten einzelsträngigen Regionen zu beschreiben. Es verwendet ähnliche Parameter der Polymere, behandelt aber jede Einheit der "Kette" als starren Stab, der hier den Nukleotiden der entfalteten einzelsträngigen Region entspricht. Die folgende Gleichung (2) beschreibt dieses Modell:
(2)
HINWEIS: Unser Labor hat kürzlich einen Algorithmus entwickelt, der die Stapelverarbeitung der rohen Kraftrampendaten mit dem Namen "Practical Optical Tweezers Analysis TOol (POTATO)54" ermöglicht. Der Algorithmus führt die Daten herunter und filtert sie, identifiziert dann mögliche Entfaltungsschritte und führt schließlich die Datenanpassung durch. Der POTATO ist in einer benutzerfreundlichen grafischen Benutzeroberfläche (GUI) (https://github.com/REMI-HIRI/POTATO) eingebaut.
- Verarbeiten Sie Konstantkraftdaten wie folgt:
HINWEIS: Die folgenden Anweisungen können analog auf Konstantlagedaten angewendet werden.- Zeichnen Sie für die Konstantkraftdaten die Entfernung im Zeitverlauf auf (Abbildung 5). Ein Histogramm, das die Häufigkeit (Zählungen) verschiedener Konformationen über die relative Positionsänderung zeigt, ist eine nützliche Möglichkeit, verschiedene dominante und kleine Zustände zu charakterisieren (Abbildung 7).
- Passen Sie das Histogramm mit (mehreren) Gaußschen Funktionen an, um den Gesamtprozentsatz einzelner Konformer bei einer bestimmten Kraft zu schätzen (Abbildung 7C). Die Gaußschen Anpassungen, die mittlere Position und die Standardabweichung umreißen die kraftbezogene Beziehung zwischen verschiedenen Populationen.
HINWEIS: Ein benutzerdefiniertes Python-Skript, das die Vorverarbeitung und grundlegende bimodale Gaußsche Anpassung von Konstantkraftdaten ermöglicht, wird in den ergänzenden Daten bereitgestellt. Parameter (Grenzfrequenz, Filtergrad, erwartete Mittelwerte, Standardabweichungswerte und Amplituden) müssen für verschiedene Experimente optimiert werden. - Als nächstes verwenden Sie das Hidden Markov-Modell, um die Zustände weiter zu analysieren, die zusätzliche faltende Zwischenprodukte (Konformer) aufdecken können55. Für weitere Informationen über das Konstantkraft- und Hidden-Markov-Modell kann man sich auf 55,56,57,58 beziehen.
Ergebnisse
In diesem Abschnitt liegt der Schwerpunkt hauptsächlich auf Messungen von RNA-Protein/Liganden-Wechselwirkungen durch die fluoreszenzoptischen Pinzetten. Für eine Beschreibung allgemeiner Experimente mit optischen RNA-Pinzetten und entsprechenden repräsentativen Ergebnissen siehe 32. Für eine detailliertere Erörterung der RNA/DNA-Protein-Interaktionen siehe auch 1,2,26,59,60.
Grundsätzlich stabilisiert, destabilisiert oder kann die Bindung eines RBP oder eines anderen transaktiven Faktors von Interesse an der RNA die Konformation des Moleküls stabilisieren, destabilisieren oder verändern. Im Folgenden ist eine Darstellung der mechanischen Observablen für jeden Effekt dargestellt. Der tatsächliche Effekt, der für einen bestimmten RNA-Protein-Komplex beobachtet wird, ist jedoch nicht auf diese unten genannten Szenarien beschränkt.
Stabilisierung
Die RNA-Struktur kann spezifisch durch das Protein oder andere Liganden erkannt und gebunden werden45,61,62,63,64. Die Bildung der Bindungen geht mit einer Freisetzung von Energie einher. Daher muss eine extra energetische Barriere überwunden werden, um die gegebene RNA-Struktur zu entfalten. Infolgedessen könnte eine Zunahme der mittleren Entfaltungskraft beobachtet werden50,65. Die Stabilisierung der RNA-Struktur durch Bindung eines externen Mittels (Protein, kleine Moleküle, andere trans-wirkende Faktoren) kann auch zu einer Änderung der Faltungskinetik der Struktur führen45. Dazu können weitere Messungen im Konstantkraftmodus durchgeführt werden, wo weniger häufige Übergänge zwischen den Faltungszwischenprodukten sowie eine Kraftverschiebung im Gleichgewicht beobachtet werden können.
Destabilisierung
Einige Proteine erkennen eher bestimmte Sequenzmotive als spezifische RNA-Strukturen. Die Bindungsstellen können von einem sehr spezifischen Motiv bis hin zu einem allgemeineren Muster wie GC- oder AU-reichen Dehnungen60,66 variieren. Bindet das Protein jedoch bevorzugt an die entfaltete einzelsträngige RNA-Konformation, kann das Gleichgewicht zwischen dem gefalteten und dem entfalteten Zustand in Richtung des entfalteten Zustands verschoben werden36,43,67. In Abbildung 6 und Abbildung 7 sind Beispiele für ein solches Verhalten dargestellt.
Strukturänderung
In einigen Fällen könnten RBPs (oder andere Liganden) beide oben genannten Mechanismen so kombinieren, dass das RBP die zuvor dominante Konformation destabilisiert und das Gleichgewicht in Richtung einer alternativen RNA-Struktur verschiebt44,68,69. Der Wechsel in einen alternativen Zustand kann zu einer Veränderung der beobachteten Konformationspopulationshäufigkeiten sowie zum Auftreten oder Verschwinden einzelner Faltungszustände führen. Diese Änderungen können zuerst in Kraftrampenexperimenten beobachtet und durch die Experimente mit konstanter Kraft (oder konstanter Position) weiter untersucht werden.
Einfluss des trans-wirkenden Faktors auf die RNA-Faltung/-Entfaltung
Hier wurde eine RNA-Sequenz untersucht, die dem -1 programmierten ribosomalen Frameshifting-Element von SARS-CoV-2 entspricht. Es wird vorhergesagt, dass dieses RNA-Element einen Pseudoknoten vom H-Typ bildet70,71. In den Beispiel-Kraft-Distanz-Trajektorien entfaltet sich die RNA und faltet sich in zwei aufeinanderfolgenden Schritten neu (Abbildung 6A). Diese beiden Schritte entsprechen wahrscheinlich den beiden Stammschleifen, die die Voraussetzung für die Pseudoknotenbildung sind. In diesem Fall wurde der Pseudoknoten weder beobachtet, weil sich die RNA nicht vollständig falte oder eine alternative Struktur bildete, die mit dem Pseudoknoten konkurrierte. Bei Zugabe des trans-agierenden Faktors ZAP wurde ein plötzliches Verschwinden der Refolding-Ereignisse und eine enorme Hysterese beobachtet (Abbildung 6B)43. Dies deutet darauf hin, dass das Protein an den einzelsträngigen Zustand der RNA bindet und die Bildung von Sekundärstrukturen behindert. Darüber hinaus bestätigen Konstantkraftexperimente die Ergebnisse von Kraftrampenexperimenten. Während die RNA bei etwa 10 pN vollständig gefaltet ist (Abbildung 7A), verschiebt die Anwesenheit des Proteins die Rückfaltung in Richtung niedrigerer Kräfte, und bei 10 pN nimmt die RNA immer noch größtenteils den entfalteten Zustand ein (Abbildung 7B).
OT-Messungen gekoppelt mit konfokaler Mikroskopie
Als nächstes werden beispielhafte Ergebnisse für die unspezifische sowie spezifische Bindung verschiedener Fluorophore und markierter Ribosomen gezeigt (Abbildung 8). Im ersten Beispiel wurde der Farbstoff SYTOX Green verwendet, um den gebundenen DNA/RNA-Hybriden zu markieren. Mit zunehmender Kraft ist die Farbstoffbindung häufiger, was zu einem höheren Fluoreszenzsignal führt. Sobald die Kraft zu hoch ist, bricht das Seil und das Fluoreszenzsignal geht verloren (Abbildung 8A). Für die Experimente mit bakteriellen Ribosomen (Abbildung 8B) wurde eine unspezifische Markierung der Lysinreste unter Verwendung von N-Hydroxysuccinimid (NHS) verwendet, das zu einem roten Fluoreszenzfarbstoff konjugiert ist. Obwohl das Risiko besteht, die Aktivität des markierten Proteins / Komplexes zu verringern, besteht der große Vorteil darin, dass ein stärkeres Signal erreicht wird, da jedes Ribosom (im Durchschnitt) durch mehrere Fluorophore markiert ist. Das RNA-Konstrukt enthielt eine von bakteriellen Ribosomen-Ribosomen erkannte Ribosomen-Bindungsstelle (RBS), die in der 5'-Nähe der untersuchten RNA-Sequenz platziert wurde. Bei der Bindung der Ribosomen wird das Fluoreszenzsignal am Seil beobachtet. Fluoreszenzdaten können mit Hilfe von Bildanalysewerkzeugen weiter analysiert werden72, und die Ergebnisse können mit den Kraftdaten kombiniert werden, um die Untersuchung von Faltungsübergängen zu ermöglichen.
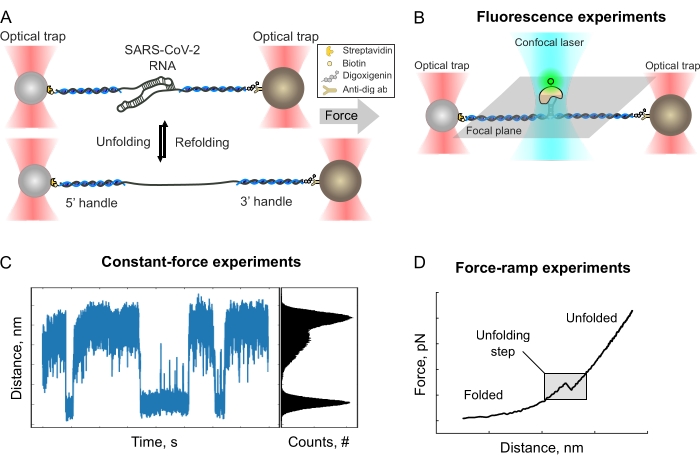
Abbildung 1: Schematische Darstellung des OT-Experiments und mögliche Messansätze. (A) Schematische Darstellung der optischen Pinzettenexperimente mit der SARS-CoV-2 Frameshifting RNA in der Mitte. RNA wird zu ssDNA-Griffen hybridisiert und auf Kügelchen immobilisiert. Diese werden verwendet, um mit einem fokussierten Laserstrahl Zugkraft auf die RNA auszuüben. Die Kraft wird allmählich erhöht, bis die RNA entfaltet wird (unten). (B) Schematische Darstellung der konfokalen Mikroskopie in Kombination mit einer optischen Pinzette zur Überwachung der Bindung des markierten Faktors an RNA. (C) Beispiel konstanter Kraftdaten können erhalten werden, indem die Kraft auf einen konstanten Wert über die Zeit festgelegt wird, der es ermöglicht, die Verweilzeit der Konformer genau zu messen. (D) Beispiel für eine Kraft-Weg-Kurve (FD), die aus einer Kraft-Rampen-Messung gewonnen wurde. Der Entfaltungsschritt wird als plötzlicher Bruch im FD-Profil beobachtet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
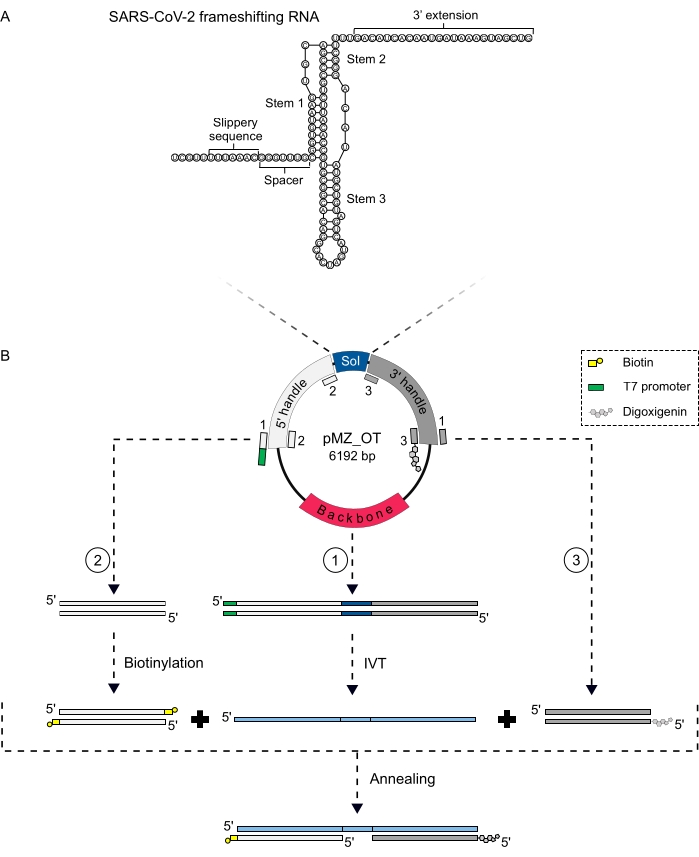
Abbildung 2: Ein allgemeines Schema der OT-Probensynthese. (A) Beispielsequenz und vorhergesagte Sekundärstruktur der untersuchten SARS-CoV-2 Frameshifting RNA, die in der Studie verwendet wurde. (B) Ein Vektor, der die Sequence of Interest (SoI) enthält, flankiert von zwei Handle-Regionen, dient als Vorlage für die Erzeugung des DNA/RNA-Konstrukts in 3 PCR-Reaktionen. Primer werden im Schema entsprechend ihrer Bindungsstellen in der entsprechenden PCR dargestellt und nummeriert. PCR 1 liefert die In-vitro-Transkriptionsvorlage , die anschließend für die In-vitro-Transkriptionsreaktion (IVT) verwendet wird, um das lange RNA-Molekül (hellblau) zu erzeugen. PCR 2 ergibt den 5' Griff, der später 3' mit Biotin markiert wird. PCR 3 unter Verwendung des an Digoxigenin konjugierten Vorwärtsprimers erzeugt den 3'-Digoxigenin-markierten Griff. Schließlich werden die beiden Griffe und die RNA geglüht, um ein DNA/RNA-Hybridkonstrukt zu erhalten, das für optische Pinzettenmessungen geeignet ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Illustration verschiedener Mikrofluidik-Kanal-Setups. (A) Ein Schema der Flusszelle mit 5-Mikrofluidikkanälen. (B) und (C) sind die Zoom-Ins des rot gestrichelten Bereichs von (A). (B) Ein einfaches 3-Kanal-Setup mit AD-Perlen und SA-Perlen in Kanal 1 bzw. 3. Faktor befindet sich in Kanal 2. Dieser Aufbau eignet sich für stabile Proteine mit hoher Affinität, daher wird eine niedrige Konzentration bevorzugt, um einen niedrigen fluoreszierenden Hintergrund zu gewährleisten. Die seitlichen Wulstkanäle ermöglichen eine feste Seilorientierung und bei Bedarf eine schnelle Rekrutierung neuer Kügelchen. (C) 4-Kanal-Setup mit Faktor in Kanal 4. Eine solche Anordnung ist besonders vorteilhaft bei minimalem Probenverbrauch. Die Messung kann direkt in Kanal 4 durchgeführt werden. Alternativ kann der Komplex, um ein Hintergrundfluoreszenzsignal zu vermeiden, in Kanal 4 gebildet werden und dann kann die Messung in Kanal 3 durchgeführt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
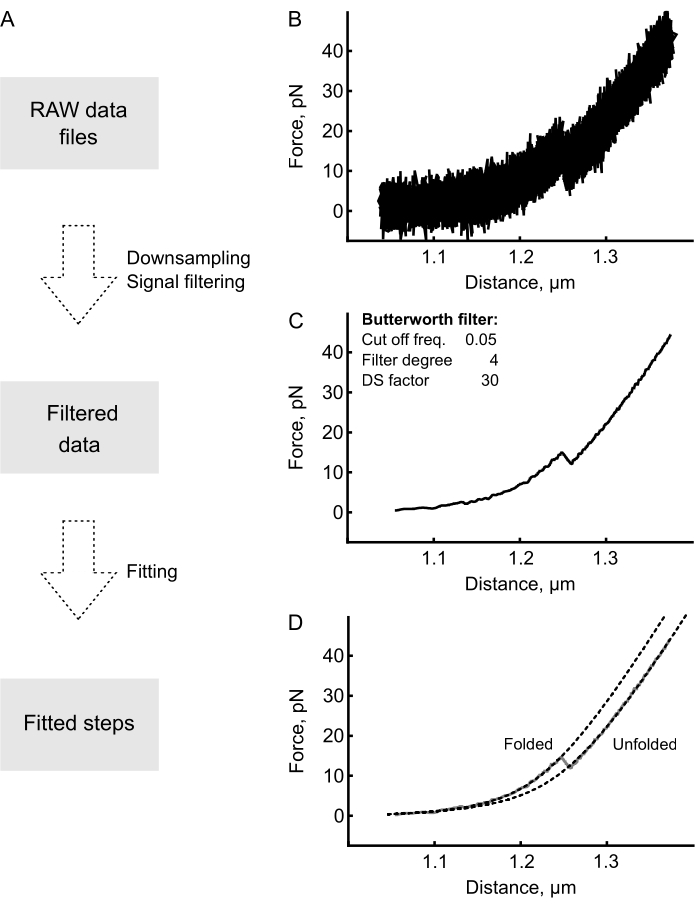
Abbildung 4: Datenanalyse-Workflow für Force-Ramp-Experimente. (A) Flussdiagramm des Datenanalyse-Workflows. Die Rohdatendateien werden zunächst heruntergerechnet und gefiltert, dann Schritte markiert und die einzelnen Zustände an das entsprechende Modell angepasst. (B) Die Rohdaten enthalten eine beträchtliche Menge an Rauschen, die die Identifizierung von sich entfaltenden / neu faltbaren Ereignissen behindern. Außerdem ist in den meisten Experimenten die Häufigkeit der Datenerfassung höher als nötig. (C) Daher werden Downsampling und Signalfiltration eingesetzt, um das Datenprofil zu glätten. (D) Die verarbeiteten Kurven werden schließlich mit dem WLC-Modell angepasst, wenn sich das Molekül noch im gefalteten Zustand befindet (vor dem sich entfaltenden Ereignis), einer Kombination des WLC mit FJC-Modellen oder einem zweiten WLC-Modell, wenn sich das Molekül in einem entfalteten Zustand befindet (nach dem sich entfaltenden Ereignis). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Der Einfluss der Grenzfrequenz auf die Datenausgabe. Während die Rohdatenausgabe mit Signalrauschen belastet sein kann (oben), ist es wichtig, die richtigen Signalfiltrationsparameter für die Datenanalyse zu wählen. Obwohl eine ordnungsgemäße Filtration bei der Identifizierung von Faltungszwischenprodukten (Grenzfrequenz 0,1, Mitte) helfen würde, kann eine Überfiltration (Grenzfrequenz <0,001, unten) zu einem Auflösungsverlust führen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
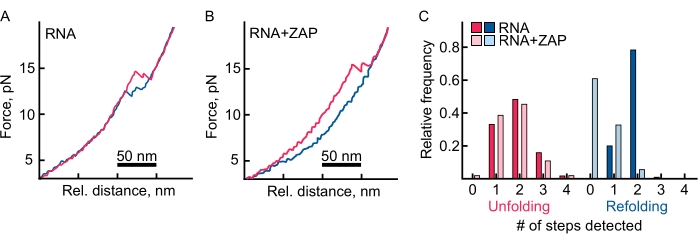
Abbildung 6: Beispiel FD-Trajektorien in Abwesenheit und Anwesenheit von ZAP. (A) Sich entfaltende (rosa) und wiederfaltende (blaue) Spuren der SARS-CoV-2-RNA in Abwesenheit von ZAP. Die Probe zeigt eine leichte Umfaltung mit nur geringer Hysterese. (B) Sich entfaltende (rosa) und umfaltende (blaue) Spuren der RNA in Gegenwart von Transfaktor-ZAP (400 nM). Die Probe zeigt eine enorme Hysterese, was darauf hindeutet, dass das Protein an die einzelsträngige RNA bindet und deren Wiederfaltung verhindert. (C) Ein Balkendiagramm, das die Anzahl der sich entfaltenden (rosa) und neu gefalteten (blau) Schritte in Abwesenheit oder Anwesenheit von ZAP anzeigt. Während die Verteilung der Entfaltungsschritte durch das Vorhandensein von ZAP nahezu unbeeinflusst bleibt, sinkt die Anzahl der Umfaltungsschritte bei ZAP deutlich. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 7: Beispiel konstanter Kraftdaten in Abwesenheit und Anwesenheit von ZAP. (A) Konstantkraftdaten, die bei Kräften zwischen 10 (oben) und 13 (unten) pN erhalten wurden und die Verschiebung des SARS-CoV-2-Frameshifting-RNA-Elements vom vollständig gefalteten Zustand in den vollständig entfalteten Zustand zeigen. Jeder Graph enthält die Position vs. Zeit (links) und ein Histogrammdiagramm (rechts). (B) Konstantkraftdaten, die in Gegenwart von ZAP (400 nM) gewonnen wurden. Bei der Proteinbindung ist die Rückfaltung beeinträchtigt. Bei 10 pN liegt im Gegensatz zu RNA allein in Gegenwart von ZAP RNA meist im entfalteten Zustand vor. Daher ist eine Verschiebung der Gleichgewichtskraft in Richtung niedrigerer Kräfte angezeigt. (C) Das Histogramm der Positionsdaten kann analysiert werden, indem die Daten an Gaußsche Funktionen angepasst werden, um die relative Häufigkeit jedes Zustands zu erhalten (abgeleitet aus der Fläche unter der Kurve für jeden Zustand). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 8: OT kombiniert mit konfokaler Mikroskopie. (A) Ein Beispiel-Kymograph des SYTOX Green beschrifteten Tether (links). Beachten Sie die Zunahme der Signalintensität bei steigenden Kräften. Der schwarze Pfeil markiert das Tetherbruchereignis, das zum Verlust des Signals führt. Darstellung des Seils mit daran gebundenem Farbstoff (Bindung) und nach dem Bruch ohne Farbstoff (Kein Signal) (rechts). (B) Beispiel Kymograph der spezifischen Bindung des Ribosoms an die mRNA (links). Das Bindungsereignis kann als Fluoreszenzsignal auf der angebundenen RNA zwischen den beiden Kügelchen beobachtet werden. Darstellung von Tether ohne (Kein Signal) und mit fluoreszenzmarkierten Ribosomen gebunden (Bindung) (rechts). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Bestandskonzentration | Endkonzentration | Volumen | |
| Reaktionsvolumen | - | - | 500 μL |
| 10× Puffer | 10× | 1× | 50 μL |
| dNTP-Mix | 10 mM | 0,2 mM | 10 μL |
| High-Fidelity-DNA-Polymerase | 1,25 U/μL | 0,025 U/μL | 10 μL |
| Grundierung 1 | 10 μM | 0,4 μM | 20 μL |
| Grundierung 2 | 10 μM | 0,4 μM | 20 μL |
| Schablone | 100 ng/μL | 1 ng/μL | 5 μL |
| Wasser | - | - | 385 μL |
Tabelle 1: Pipettierschema für die PCR zur Erzeugung der optischen Pinzettenkonstrukte.
| Bestandskonzentration | Endkonzentration | Volumen | |
| Reaktionsvolumen | - | - | 300 μL |
| 5× Puffer | 5× | 1× | 60 μL |
| rNTP-Mix | 25 mM | 5 mM | 60 μL |
| RNase-Inhibitor | 40 U/μL | 0,7 U/μL | 5 μL |
| Pyrophosphatase | 100 U/ml | 1,7 mU/μL | 4 μL |
| DVB-T | 100 mM | 3,3 mM | 10 μL |
| T7-RNA-Polymerase | 50 U/μL | 3,3 U/μL | 20 μL |
| Schablone | 120 ng/μL | 2 ng/μL | 5 μL |
| Wasser | - | - | 136 μL |
Tabelle 2: Pipettierschema für die In-vitro-Transkription .
| Bestandskonzentration | Endkonzentration | Volumen | |
| Reaktionsvolumen | - | - | 100 μL |
| 10× Puffer (NEB 2.1) | 10× | 1× | 10 μL |
| BSA | 1 μg/ml | 100 ng/μL | 1 μL |
| Biotin-16-dUTP | 1 mM | 50 μM | 5 μL |
| T4 DNA-Polymerase | 30 HE/μL | 1,5 U/μL | 5 μL |
| DNA 5' Griff (20-60 μg) | 300 ng/μL | 237 ng/μL | 79 μL |
Tabelle 3: Pipettierschema für die 3'-End-Biotin-Markierung.
| Bestandskonzentration | Endkonzentration | Endbetrag | Volumen | |
| Reaktionsvolumen | - | - | - | 300 μL |
| Glühpuffer | 1.25× | 1× | - | 240 μL |
| RNase-Inhibitoren | 40 U/μL | 0,5 U/μL | - | 5 μL |
| 5' biotinylierter DNA-Griff | 300 ng/μL | 10 ng/μL | 3 μg | 10 μL |
| 3' DNA-Griff | 300 ng/μL | 10 ng/μL | 3 μg | 10 μL |
| RNS | 150 ng/μL | 10 ng/μL | 3 μg | 20 μL |
| Wasser | - | - | - | 5 μL |
Tabelle 4: Pipettierschema für das Glühen des optischen Pinzettenkonstrukts.
Diskussion
Hier demonstrieren wir die Verwendung von fluoreszenzgekoppelten optischen Pinzetten, um Wechselwirkungen und dynamisches Verhalten von RNA-Molekülen mit verschiedenen Liganden zu untersuchen. Im Folgenden werden kritische Schritte und Einschränkungen der vorliegenden Technik diskutiert.
Kritische Schritte im Protokoll
Wie bei vielen anderen Methoden ist die Qualität der Probe entscheidend, um zuverlässige Daten zu erhalten. Um Proben von höchstmöglicher Qualität zu erhalten, lohnt es sich daher, Zeit zu investieren, um das Verfahren zur Probenvorbereitung zu optimieren. Die Optimierungsschritte umfassen das richtige Primerdesign, Glühtemperaturen, RNA- und Proteinreinigungsschritte.
Während des gesamten Experiments ist die Verwendung von gefilterten Spitzen und Lösungen von entscheidender Bedeutung, um RNase-freie Bedingungen aufrechtzuerhalten. Darüber hinaus wird das Mikrofluidiksystem bei Nichtgebrauch in Bleichmittel gehalten. Vor Beginn der Messungen ist es wichtig, das System ordnungsgemäß mit Natriumthiosulfat und RNase-freiem Wasser zu waschen, um das Bleichmittel aus dem System zu entfernen.
Falls während des gesamten Experiments die gleich großen Perlen verwendet werden, ist es nicht erforderlich, jedes Mal eine Kraftkalibrierung durchzuführen. Dennoch sollten regelmäßig Kraftkalibrierungsprüfungen für die Reproduzierbarkeit von Experimenten durchgeführt werden.
Änderungen und Fehlerbehebung der Methode
Fluorophorstabilität und Photobleichen
Eine Komplikation bei Fluoreszenzmessungen ist das Photobleichen. Da der Zeitrahmen für die Überwachung der Translation je nach System von Sekunden auf Minuten verlängert werden kann, sollte auch das Photobleichen während der Messungen berücksichtigt und so weit wie möglich minimiert werden73. Eine Möglichkeit besteht darin, stabilere Fluorophore zu verwenden, die weniger anfällig für Photobleiche sind, wie die kürzlich eingeführten Quantenpunkte49,74,75. Weitere Stabilität wird auch durch das Entfernen von Sauerstoffmolekülen mit einem "Sauerstofffänger" -System erreicht, wie z. B. Glukoseoxidase in Verbindung mit Katalase. Glucoseoxidase entfernt Sauerstoff aus der Umgebung, indem sie ihn in Wasserstoffperoxid umwandelt, das dann durch Katalase zersetzt wird. Alternative Sauerstofffängersysteme können ebenfalls eingesetzt werden76,77.
Mikrofluidik
Die Aufrechterhaltung einer kontinuierlichen laminaren Strömung ist für ordnungsgemäße Messungen unerlässlich. Am wichtigsten ist, dass das System niemals trocken laufen sollte. Leider sind RBPs oder andere transaktive Faktoren von Interesse oft nur in kleinen Mengen für die Experimente verfügbar, daher kann die Aufrechterhaltung eines kontinuierlichen Flusses eine Herausforderung und kostenintensiv sein. Werden während der Probenapplikation Luftblasen in das System eingebracht, reicht in der Regel manueller Druck oder Ethanolwäsche für deren Entfernung aus.
Einschränkungen der Methode
Die Kombination von OT mit konfokaler Mikroskopie bringt auch einige Einschränkungen mit sich. Erstens muss die Brennebene der konfokalen Einheit richtig mit Fallenzentren ausgerichtet sein, um eine ordnungsgemäße Aufzeichnung des Fluoreszenzsignals zu ermöglichen. Darüber hinaus werden für konfokale Messungen in der Regel Griffe von mindestens 2 kb an jedem Standort benötigt17. Obwohl prinzipiell die Verwendung längerer Griffe möglich ist, sollte man für die Genauigkeit der Datenanalyse den Energiebeitrag der Griffe und die Änderung der Persistenzlänge berücksichtigen78. Ein weiterer entscheidender Punkt ist, dass die Sauerstofffänger, die zur Erhöhung der Halbwertszeit der Fluorophore eingesetzt werden, ebenfalls zu relativ schnellen Änderungen des pH-Wertes der Lösungen führen76. Diese Veränderungen können teilweise durch Erhöhung der Konzentration der Pufferverbindung kompensiert werden; Während der Messungen sollten die Proben jedoch regelmäßig (alle 30-60 min) aufgefüllt werden, um konsistente Bedingungen während des Experiments zu gewährleisten.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Wir danken Anuja Kibe und Jun. Prof. Redmond Smyth für die kritische Begutachtung des Manuskripts. Wir danken Tatyana Koch für die fachkundige technische Unterstützung. Wir danken Kristyna Pekarkova für die Hilfe bei der Aufnahme experimenteller Videos. Die Arbeit in unserem Labor wird von der Helmholtz-Gemeinschaft unterstützt und vom Europäischen Forschungsrat (ERC) Grant Nr. 948636 (bis NC) gefördert.
Materialien
| Name | Company | Catalog Number | Comments |
| Bacterial Strains | |||
| E. coli HB101 | lab collection | N/A | cloning of the vectors |
| Chemicals and enzymes | |||
| Sodium chloride | Sigma-Aldrich | 31424 | Buffers |
| Biotin-16-dUTP | Roche | 11093070910 | Biotinylation |
| BSA | Sigma-Aldrich | A4737 | Buffers |
| Catalase | Lumicks | N/A | Oxygen scavanger system |
| Dithiothreitol (DTT) | Melford Labs | D11000 | Buffers |
| DNAse I from bovine pancreas | Sigma-Aldrich | D4527 | in vitro transcription |
| dNTPs | Th.Geyer | 11786181 | PCR |
| EDTA | Sigma-Aldrich | E9884 | Buffers |
| Formamide | Sigma-Aldrich | 11814320001 | Buffers |
| Glucose | Sigma-Aldrich | G8270-1KG | Oxygen scavanger system |
| Glucose-oxidase | Lumicks | N/A | Oxygen scavanger system |
| HEPES | Carl Roth | HN78.3 | Buffers |
| Magnesium chloride | Carl Roth | 2189.1 | Buffers |
| Phusion DNA polymerase | NEB | M0530L | Gibson assembly, cloning |
| Potassium chloride | Merck | 529552-1KG | Buffers |
| PrimeSTAR GXL DNA Polymerase | Takara Bio Clontech | R050A | PCR |
| Pyrophosphotase, thermostabile, inorganic | NEB | M0296L | in vitro transcription |
| RNase Inhibitor | Molox | 1000379515 | Buffers |
| rNTPS | life technologies | R0481 | in vitro transcription |
| Sodium thiosulophate | Sigma-Aldrich | S6672-500G | Bleach deactivation |
| Sytox Green | Lumicks | N/A | confocal measurements |
| T4 DNA Polymerase | NEB | M0203S | Biotinylation |
| T5 exonuclease | NEB | M0363S | Gibson assembly, cloning |
| T7 RNA polymerase | Produced in-house | N/A | in vitro transcription |
| Taq DNA polymerase | NEB | M0267S | PCR |
| Taq ligase | Biozym | L6060L | Gibson assembly, cloning |
| TWEEN 20 BioXtra | Sigma-Aldrich | P7949 | Buffers |
| Kits | |||
| Monolith Protein Labeling Kit RED-NHS 2nd Generation (Amine Reactive) | Nanotemper | MO-L011 | Used for ribosome labeling |
| Purefrex 2.0 | GeneFrontier | PF201-0.25-EX | Ribosomes used for the labeling |
| Oligonucleotides | |||
| 5' handle T7 forward | Microsynth | custom order | 5’ - CTTAATACGACTCACTATAGGTC CTTTCTGTGGACGCC - 3’, used to generate OT in vitro transcription template in PCR 1 |
| 3’ handle reverse | Microsynth | custom order | 5' - GTCAAAGTGCGCCCCGTTATCC - 3', used to generate OT in vitro transcription template in PCR 1 |
| 5' handle forward | Microsynth | custom order | 5' - TCCTTTCTGTGGACGCCGC - 3' , used to generate 5' handle in PCR 2 |
| 5’ handle reverse | Microsynth | custom order | 5’ - CATAAATACCTCTTTACTAATATA TATACCTTCGTAAGCTAGCGT - 3’, used to generate 5' handle in PCR 2 |
| 3’ handle forward | Microsynth | custom order | 5' - ATCCTGCAACCTGCTCTTCGCC AG - 3', used to generate 3' handle in PCR 3 |
| 3’ handle reverse 5’labeled with digoxigenin | Microsynth | custom order | 5' -[Dig]-GTCAAAGTGCGCCCCGTTATCC - 3', used to generate 3' handle in PCR 3 |
| DNA vectors | |||
| pMZ_OT | produced in-house | N/A | further description in "Structural studies of Cardiovirus 2A protein reveal the molecular basis for RNA recognition and translational control" Chris H. Hill, Sawsan Napthine, Lukas Pekarek, Anuja Kibe, Andrew E. Firth, Stephen C. Graham, Neva Caliskan, Ian Brierley bioRxiv 2020.08.11.245035; doi: https://doi-org.remotexs.ntu.edu.sg/10.1101/2020.08.11.245035 |
| Software and Algorithms | |||
| Atom | https://atom.io/packages/ide-python | N/A | |
| Bluelake | Lumicks | N/A | |
| Graphpad | https://www.graphpad.com/ | N/A | |
| InkScape 0.92.3 | https://inkscape.org/ | N/A | |
| Matlab | https://www.mathworks.com/products/matlab.html | N/A | |
| POTATO | https://github.com/lpekarek/POTATO.git | N/A | |
| RNAstructure | https://rna.urmc.rochester.edu/RNAstructure.html | N/A | |
| Spyder | https://www.spyder-ide.org/ | N/A | |
| Other | |||
| Streptavidin Coated Polystyrene Particles, 1.5-1.9 µm, 5 ml, 1.0% w/v | Spherotech | SVP-15-5 | |
| Anti-digoxigenin Coated Polystyrene Particles, 2.0-2.4 µm, 2 ml, 0.1% w/v | Spherotech | DIGP-20-2 | |
| Syringes | VWR | TERUMO SS+03L1 | |
| Devices | |||
| C-trap | Lumicks | N/A | optical tweezers coupled with confocal microscopy |
Referenzen
- Balcerak, A., Trebinska-Stryjewska, A., Konopinski, R., Wakula, M., Grzybowska, E. A. RNA-protein interactions: disorder, moonlighting and junk contribute to eukaryotic complexity. Open Biology. 9 (6), 190096 (2019).
- Armaos, A., Zacco, E., Sanchez de Groot, N., Tartaglia, G. G. RNA-protein interactions: Central players in coordination of regulatory networks. BioEssays. 43 (2), 2000118 (2021).
- Firth, A. E., Brierley, I. Non-canonical translation in RNA viruses. Journal of General Virology. 93, 1385-1409 (2012).
- Caliskan, N., Peske, F., Rodnina, M. V. Changed in translation: mRNA recoding by −1 programmed ribosomal frameshifting. Trends in Biochemical Sciences. 40 (5), 265-274 (2015).
- Jaafar, Z. A., Kieft, J. S. Viral RNA structure-based strategies to manipulate translation. Nature Reviews Microbiology. 17 (2), 110-123 (2019).
- Eswarappa, S. M., et al. Programmed translational readthrough generates antiangiogenic VEGF-Ax. Cell. 157 (7), 1605-1618 (2014).
- Rodnina, M. V., et al. Translational recoding: canonical translation mechanisms reinterpreted. Nucleic Acids Research. 48 (3), 1056-1067 (2020).
- Li, Y., et al. Transactivation of programmed ribosomal frameshifting by a viral protein. Proceedings of the National Academy of Sciences. 111 (21), 2172 (2014).
- Napthine, S., et al. Protein-directed ribosomal frameshifting temporally regulates gene expression. Nature Communications. 8 (1), 15582 (2017).
- Patel, A., et al. Molecular characterization of the RNA-protein complex directing -2/-1 programmed ribosomal frameshifting during arterivirus replicase expression. Journal of Biological Chemistry. 295 (52), 17904-17921 (2020).
- Napthine, S., Bell, S., Hill, C. H., Brierley, I., Firth, A. E. Characterization of the stimulators of protein-directed ribosomal frameshifting in Theiler's murine encephalomyelitis virus. Nucleic Acids Research. 47 (15), 8207-8223 (2019).
- Marshall, R. A., Aitken, C. E., Dorywalska, M., Puglisi, J. D. Translation at the Single-Molecule Level. Annual Review of Biochemistry. 77 (1), 177-203 (2008).
- Rodnina, M. V. The ribosome in action: Tuning of translational efficiency and protein folding. Protein science : A publication of the Protein Society. 25 (8), 1390-1406 (2016).
- Rodnina, M. V., Fischer, N., Maracci, C., Stark, H. Ribosome dynamics during decoding. Philosophical Transactions of Royal Society of London B Biological Sciences. 372 (1716), (2017).
- Yan, S., Wen, J. D., Bustamante, C., Tinoco, I. Ribosome excursions during mRNA translocation mediate broad branching of frameshift pathways. Cell. 160 (5), 870-881 (2015).
- Liu, T., et al. Direct measurement of the mechanical work during translocation by the ribosome. eLife. 3, 03406 (2014).
- Desai, V. P., et al. Co-temporal force and fluorescence measurements reveal a ribosomal gear shift mechanism of translation regulation by structured mRNAs. Molecular Cell. 75 (5), 1007-1019 (2019).
- Choi, J., O'Loughlin, S., Atkins, J. F., Puglisi, J. D. The energy landscape of -1 ribosomal frameshifting. Science Advances. 6 (1), (2020).
- Prabhakar, A., Puglisi, E. V., Puglisi, J. D. Single-molecule fluorescence applied to translation. Cold Spring Harbor Perspectives in Biology. 11 (1), 032714 (2019).
- Bao, C., et al. mRNA stem-loops can pause the ribosome by hindering A-site tRNA binding. Elife. 9, 55799 (2020).
- Chen, J., Tsai, A., O'Leary, S. E., Petrov, A., Puglisi, J. D. Unraveling the dynamics of ribosome translocation. Current Opinion in Structural Biology. 22 (6), 804-814 (2012).
- Qu, X., et al. The ribosome uses two active mechanisms to unwind messenger RNA during translation. Nature. 475 (7354), 118-121 (2011).
- Zheng, Q., et al. Ultra-stable organic fluorophores for single-molecule research. Chemical Society Reviews. 43 (4), 1044-1056 (2014).
- Blanchard, S. C. Single-molecule observations of ribosome function. Current Opinion in Structural Biology. 19 (1), 103-109 (2009).
- Juette, M. F., et al. The bright future of single-molecule fluorescence imaging. Current Opinion in Chemical Biology. 20, 103-111 (2014).
- McCauley, M. J., Williams, M. C. Mechanisms of DNA binding determined in optical tweezers experiments. Biopolymers. 85 (2), 154-168 (2007).
- Ashkin, A., Dziedzic, J. M., Bjorkholm, J. E., Chu, S. Observation of a single-beam gradient force optical trap for dielectric particles. Optics Letters. 11 (5), 288-290 (1986).
- Bustamante, C., Smith, S. B., Liphardt, J., Smith, D. Single-molecule studies of DNA mechanics. Current Opinion in Structural Biology. 10 (3), 279-285 (2000).
- Choudhary, D., Mossa, A., Jadhav, M., Cecconi, C. Bio-molecular applications of recent developments in optical tweezers. Biomolecules. 9 (1), 23 (2019).
- Moffitt, J. R., Chemla, Y. R., Smith, S. B., Bustamante, C. Recent advances in optical tweezers. Annual Reviews of Biochemistry. 77, 205-228 (2008).
- Li, P. T. X., Vieregg, J., Tinoco, I. How RNA Unfolds and Refolds. Annual Review of Biochemistry. 77 (1), 77-100 (2008).
- Stephenson, W., Wan, G., Tenenbaum, S. A., Li, P. T. Nanomanipulation of single RNA molecules by optical tweezers. Journal of Visualized Experiments. (90), e51542 (2014).
- Halma, M. T. J., Ritchie, D. B., Cappellano, T. R., Neupane, K., Woodside, M. T. Complex dynamics under tension in a high-efficiency frameshift stimulatory structure. Proceedings of the National Academy of Sciences. 116 (39), 19500 (2019).
- Hansen, T. M., Reihani, S. N. S., Oddershede, L. B., Sørensen, M. A. Correlation between mechanical strength of messenger RNA pseudoknots and ribosomal frameshifting. Proceedings of the National Academy of Sciences of the United States of America. 104 (14), 5830-5835 (2007).
- Zhong, Z., et al. Mechanical unfolding kinetics of the SRV-1 gag-pro mRNA pseudoknot: possible implications for -1 ribosomal frameshifting stimulation. Science Reports. 6, 39549 (2016).
- McCauley, M. J., Rouzina, I., Li, J., Núñez, M. E., Williams, M. C. Significant differences in RNA structure destabilization by HIV-1 GagDp6 and NCp7 proteins. Viruses. 12 (5), 484 (2020).
- de Messieres, M., et al. Single-molecule measurements of the CCR5 mRNA unfolding pathways. Biophysics Journal. 106 (1), 244-252 (2014).
- Yang, L., et al. Single-molecule mechanical folding and unfolding of RNA hairpins: Effects of single A-U to A·C pair substitutions and single proton binding and implications for mRNA structure-induced -1 ribosomal frameshifting. Journal of American Chemical Society. 140 (26), 8172-8184 (2018).
- McCauley, M. J., et al. Targeted binding of nucleocapsid protein transforms the folding landscape of HIV-1 TAR RNA. Proceedings of the National Academy of Sciences of the United States of America. 112 (44), 13555-13560 (2015).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. High-resolution "Fleezers": Dual-trap optical tweezers combined with single-molecule fluorescence detection. Methods in Molecular Biology. 1486, 183-256 (2017).
- Yerramilli, V. S., Kim, K. H. Labeling RNAs in live cells using malachite green aptamer scaffolds as fluorescent probes. ACS Synthetic Biology. 7 (3), 758-766 (2018).
- Gross, P., Farge, G., Peterman, E. J., Wuite, G. J. Combining optical tweezers, single-molecule fluorescence microscopy, and microfluidics for studies of DNA-protein interactions. Methods in Enzymology. 475, 427-453 (2010).
- Zimmer, M. M., et al. The short isoform of the host antiviral protein ZAP acts as an inhibitor of SARS-CoV-2 programmed ribosomal frameshifting. Nature Communications. 12 (1), 7193 (2021).
- Neupane, K., Yu, H., Foster, D. A. N., Wang, F., Woodside, M. T. Single-molecule force spectroscopy of the add adenine riboswitch relates folding to regulatory mechanism. Nucleic acids research. 39 (17), 7677-7687 (2011).
- Ritchie, D. B., Soong, J., Sikkema, W. K., Woodside, M. T. Anti-frameshifting ligand reduces the conformational plasticity of the SARS virus pseudoknot. Journal of the American Chemical Society. 136 (6), 2196-2199 (2014).
- Janissen, R., et al. Invincible DNA tethers: covalent DNA anchoring for enhanced temporal and force stability in magnetic tweezers experiments. Nucleic Acids Research. 42 (18), 137 (2014).
- Smith, S. B., Cui, Y., Bustamante, C. Overstretching B-DNA: The elastic response of individual double-stranded and single-stranded DNA molecules. Science. 271 (5250), 795 (1996).
- Puljung, M. C., Zagotta, W. N. Labeling of specific cysteines in proteins using reversible metal protection. Biophysical Journal. 100 (10), 2513-2521 (2011).
- Toseland, C. P. Fluorescent labeling and modification of proteins. Journal of Chemical Biology. 6 (3), 85-95 (2013).
- Hill, C. H., et al. Structural and molecular basis for Cardiovirus 2A protein as a viral gene expression switch. Nature Communications. 12 (1), 7166 (2021).
- Butterworth, S. On the theory of filter amplifiers. Experimental Wireless and the Wireless Engineer. 7, 536-541 (1930).
- Wang, M. D., Yin, H., Landick, R., Gelles, J., Block, S. M. Stretching DNA with optical tweezers. Biophysics Journal. 72 (3), 1335-1346 (1997).
- Mukhortava, A., et al. Structural heterogeneity of attC integron recombination sites revealed by optical tweezers. Nucleic Acids Research. 47 (4), 1861-1870 (2019).
- Buck, S., Pekarek, L., Caliskan, N. POTATO: An automated pipeline for batch analysis of optical tweezers data. bioRxiv. , (2021).
- Zhang, Y., Jiao, J., Rebane, A. A. Hidden Markov modeling with detailed balance and its application to single protein folding. Biophysical Journal. 111 (10), 2110-2124 (2016).
- Sgouralis, I., Pressé, S. An introduction to infinite HMMs for single-molecule data analysis. Biophysics Journal. 112 (10), 2021-2029 (2017).
- Müllner, F. E., Syed, S., Selvin, P. R., Sigworth, F. J. Improved hidden Markov models for molecular motors, part 1: basic theory. Biophysical Journal. 99 (11), 3684-3695 (2010).
- Elms, P. J., Chodera, J. D., Bustamante, C. J., Marqusee, S. Limitations of constant-force-feedback experiments. Biophysical Journal. 103 (7), 1490-1499 (2012).
- Re, A., Joshi, T., Kulberkyte, E., Morris, Q., Workman, C. T. RNA-protein interactions: an overview. Methods Molecular Biology. 1097, 491-521 (2014).
- Jankowsky, E., Harris, M. E. Specificity and nonspecificity in RNA-protein interactions. Nature reviews. Molecular Cell Biology. 16 (9), 533-544 (2015).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Sunbul, M., Jäschke, A. SRB-2: a promiscuous rainbow aptamer for live-cell RNA imaging. Nucleic Acids Research. 46 (18), 110 (2018).
- Sanchez de Groot, N., et al. RNA structure drives interaction with proteins. Nature Communications. 10 (1), 3246 (2019).
- Zeffman, A., Hassard, S., Varani, G., Lever, A. The major HIV-1 packaging signal is an extended bulged stem loop whose structure is altered on interaction with the Gag polyprotein. Journal of Molecular Biology. 297 (4), 877-893 (2000).
- Mangeol, P., et al. Probing ribosomal protein-RNA interactions with an external force. Proceedings of the National Academy of Sciences. 108 (45), 18272 (2011).
- Luo, X., et al. Molecular mechanism of RNA recognition by Zinc-Finger antiviral protein. Cell Reports. 30 (1), 46-52 (2020).
- Qu, X., Lancaster, L., Noller, H. F., Bustamante, C., Tinoco, I. Ribosomal protein S1 unwinds double-stranded RNA in multiple steps. Proceedings of the National Academy of Science U. S. A. 109 (36), 14458-14463 (2012).
- Chandra, V., Hannan, Z., Xu, H., Mandal, M. Single-molecule analysis reveals multi-state folding of a guanine riboswitch. Nature Chemical Biology. 13 (2), 194-201 (2017).
- Savinov, A., Perez, C. F., Block, S. M. Single-molecule studies of riboswitch folding. Biochimica et Biophysica Acta. 1839 (10), 1030-1045 (2014).
- Kelly, J. A., et al. Structural and functional conservation of the programmed ribosomal frameshift signal of SARS coronavirus 2 (SARS-CoV-2). Journal of Biological Chemistry. 295 (31), 10741-10748 (2020).
- Neupane, K., et al. Structural dynamics of single SARS-CoV-2 pseudoknot molecules reveal topologically distinct conformers. Nature Communications. 12 (1), 4749 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Zheng, Q., Jockusch, S., Zhou, Z., Blanchard, S. C. The contribution of reactive oxygen species to the photobleaching of organic fluorophores. Photochemistry and Photobiology. 90 (2), 448-454 (2014).
- Deerinck, T. J. The application of fluorescent quantum dots to confocal, multiphoton, and electron microscopic imaging. Toxicologic Pathology. 36 (1), 112-116 (2008).
- Rill, N., Mukhortava, A., Lorenz, S., Tessmer, I. Alkyltransferase-like protein clusters scan DNA rapidly over long distances and recruit NER to alkyl-DNA lesions. Proceedings of the National Academy of Science U. S. A. 117 (17), 9318-9328 (2020).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
- Wen, J. -. D., et al. Force unfolding kinetics of RNA using optical tweezers. I. Effects of experimental variables on measured results. Biophysical journal. 92 (9), 2996-3009 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten