Method Article
Nachweis zellfreier DNA in Blutplasmaproben von Krebspatienten
In diesem Artikel
Zusammenfassung
In diesem Beitrag stellen wir ein detailliertes Protokoll für die nichtinvasive flüssige Biopsietechnik vor, einschließlich Blutentnahme, Plasma- und Buffy-Coat-Trennung, cfDNA- und Keimbahn-DNA-Extraktion, Quantifizierung von cfDNA- oder Keimbahn-DNA und cfDNA-Fragmentanreicherungsanalyse.
Zusammenfassung
Die Identifizierung von Mutationen in Tumoren von Krebspatienten ist ein sehr wichtiger Schritt im Krankheitsmanagement. Diese Mutationen dienen als Biomarker für die Tumordiagnose sowie für die Behandlungsauswahl und deren Reaktion bei Krebspatienten. Die aktuelle Goldstandardmethode zum Nachweis von Tumormutationen beinhaltet einen genetischen Test der Tumor-DNA mittels Tumorbiopsien. Diese invasive Methode ist jedoch nur schwer wiederholt als Folgetest des Tumormutationsrepertoires durchzuführen. Flüssigbiopsie ist eine neue und aufkommende Technik, um Tumormutationen als einfach zu bedienenden und nicht-invasiven Biopsieansatz zu erkennen.
Krebszellen vermehren sich schnell. Parallel dazu erkranken zahlreiche Krebszellen einer Apoptose. Trümmer dieser Zellen werden in das Kreislaufsystem eines Patienten freigesetzt, zusammen mit fein fragmentierten DNA-Stücken, sogenannten zellfreien DNA-Fragmenten (cfDNA), die Tumor-DNA-Mutationen tragen. Daher werden zur Identifizierung von cfDNA-basierten Biomarkern mit flüssiger Biopsietechnik Blutproben von den Krebspatienten entnommen, gefolgt von der Trennung von Plasma und buffy Coat. Als nächstes wird Plasma zur Isolierung von cfDNA und das jeweilige Buffy-Mantel zur Isolierung der genomischen DNA eines Patienten verarbeitet. Beide Nukleinsäureproben werden dann auf ihre Menge und Qualität überprüft; und auf Mutationen mit NGS-Techniken der nächsten Generation analysiert.
In diesem Manuskript stellen wir ein detailliertes Protokoll für die flüssige Biopsie vor, einschließlich Blutentnahme, Plasma- und Buffy-Coat-Trennung, cfDNA- und Keimbahn-DNA-Extraktion, Quantifizierung von cfDNA- oder Keimbahn-DNA und cfDNA-Fragmentanreicherungsanalyse.
Einleitung
Technologische Fortschritte haben zur Sequenzierung von Hunderten von Krebsgenomen und Transkriptomen1geführt. Dies hat dazu beigetragen, Landschaften molekularer Veränderungen über verschiedene Krebsarten hinweg zu verstehen2. Weitere Studien zu diesen Landschaften haben dazu beigetragen, die sequenziellen somatischen Veränderungen und Gen-Gen-Fusionen3 zu charakterisieren, die an Krebs oder Tumorprogression beteiligt sind, indem sie die Apoptosewege seriell stören4. Daher können somatische Mutationen und Gen-Gen-Fusionen Informationen über Tumore liefern, indem sie als Biomarker bei einzelnen Patienten für einen bestimmten Tumortyp5dienen, bestehende Primärtumoren-Prognose6identifizieren, sekundäre Tumoren auf der Grundlage molekularer Veränderungen kategorisieren7, und medikamentöse Tumorzieleidentifizieren 8. Solche Informationen können bei der Auswahl einer personalisierten Behandlung für Krebspatienten und bei der Bestimmung positiver und negativer Behandlungsreaktionenerleichtern 9. Die Gewinnung von Tumormaterial zur Identifizierung der genomischen Profilierung von Tumorgewebe ist jedoch ein invasives Verfahren10. Darüber hinaus umfasst eine Tumorbiopsie nur einen kleinen Teil eines heterogenen Tumors; und darf daher nicht repräsentativ für das molekulare Profil des gesamten Tumors11sein. Serielle Überwachung und Tumor-Genotypisierung erfordern eine wiederholte Sammlung von Tumorgeweben, was in der Regel aufgrund der Invasivität des Tumorbiopsieverfahrens und der Sicherheitsprobleme, die sich aus solchen Verfahren ergeben, nicht machbar ist12.
Die flüssige Biopsie-Technik hingegen hat in den letzten zehn Jahren enorme Aufmerksamkeit in der Präzisions-Onkologie erlangt13,14. Es ist vor allem aufgrund der Nicht-Invasivität dieser Technik, und die Möglichkeit, dass es an mehreren Zeitpunkten wiederholt werden, wodurch eine einfach zu bedienende und sichere Überwachungstechnik für die Krankheitskurse15,16ermöglicht wird. Die Flüssigbiopsie basiert auf einem Phänomen, dass sich Tumorzellen schnell vermehren und gleichzeitig viele von ihnen apoptosis und nekrose leiden. Dies führt zur Freisetzung von apoptotischen Zellablagerungen in das Blut der Patienten, zusammen mit den DNA-Fragmenten, die bei Apoptose17in präzisen Größen geschnitten werden. Die Apoptose von nicht-kanzerösen Zellen führt auch zur Freisetzung ihrer zellulären Ablagerungen in das Blut, jedoch ist die Apoptoserate in diesen Zellen relativ viel niedriger als die tumorzellen18. Der Grund der flüssigen Biopsietechnik besteht darin, tumorassoziierte Moleküle wie DNA, RNA, Proteine und Tumorzellen14,19 zu erfassen, die kontinuierlich im Blut zirkulieren. Für die Analyse dieser Moleküle können verschiedene Techniken20 verwendet werden, darunter Next-Generation Sequencing (NGS), digitale Tröpfchenpolymerase-Kettenreaktion (ddPCR), Echtzeit-PCR und enzymgebundener Immunsorbent-Assay (ELISA). Die Flüssigbiopsietechnik ermöglicht die Identifizierung von Biomarkern, die Merkmale von Tumorzellen sind. Diese Biomarkermoleküle werden nicht nur aus bestimmten Teilen eines Tumors freigesetzt, sondern aus allen Teilen des Tumors21. Daher stellen marker, die in der flüssigen Biopsie identifiziert werden, die molekulare Profilierung eines gesamten heterogenen Tumors dar, zusätzlich zu anderen Tumoren im Körper, also Vorteile gegenüber der Gewebebiopsie-basierten Technik22.
Die cfDNA hat eine kurze Halbwertszeit im zirkulierenden Blut von wenigen Minuten bis 1-2 Stunden23. Die kurze Halbwertszeit von cfDNA ermöglicht jedoch Echtzeitanalysen durch die Bewertung der Behandlungsreaktionen und dynamischen Tumorbewertungen. Die tumorabgeleiteten cfDNA-Spiegel zeigen eine Prognose des Tumorstadiums/der Tumorgröße, die durch mehrere Studien belegt wurde, die einen Zusammenhang zwischen cfDNA-Spiegeln und den Überlebensergebnissen24zeigten. Darüber hinaus haben Studien gezeigt, dass die cfDNA eine bessere Vorhersagekapazität als bestehende Tumormarkerhat 25. Die Prognose von cfDNA ist nach der Krebsbehandlung noch ausgeprägter, höhere cfDNA-Konzentrationen nach der Behandlung korrelieren gut mit einer reduzierten Überlebensrate und Resistenz gegen die Behandlung. Während niedrigere cfDNA-Werte nach der Therapie im Allgemeinen mit einem positiven Behandlungsverhalten korrespondiert. Darüber hinaus erleichtert die cfDNA die Früherkennung des Behandlungsreaktionens als die herkömmlichen Nachweismethoden.
Die cfDNA erhöht die Möglichkeit der Früherkennung von krebsassoziierten Mutationen: während der Frühstadium-Krankheit15, das Auftreten der Symptome26 und vor der Krebsdiagnose bis zu 2 Jahre27. Da cfDNA aus mehreren Tumorregionen oder Brennpunkten freigesetzt wird, bietet seine Analyse einen umfassenden Überblick über das Tumorgenom, das28darstellt. Daher ermöglicht die cfDNA, somatische Mutationen zu erkennen, die in den Gewebeproben übersehen worden sein könnten29. Da intratumorliche Heterogenität und subklonale Mutationen durch tiefe Sequenzierung genomischer Regionen, die sich über Tausende von Basen erstrecken, nachgewiesen werden können, ermöglicht die Analyse der cfDNA die Aufdeckung spezifischer molekularer Subtypen mit deutlichen genomischen Signaturen13. Um ein ähnliches Maß an Informationen durch Gewebeproben zu erhalten, wären viele feste Biopsien erforderlich gewesen.
Darüber hinaus erwiesen sich die cfDNA-Spiegel bei Patienten mit einer lokalisierten Erkrankung wie Dickdarm-, Eierstock- und Lungenkrebs nach einer chirurgischen Behandlung und/oder Chemotherapie als ein starker prognostischer Marker für das Wiederauftreten von Krebs und die Behandlungsergebnisse20. Darüber hinaus konnten bei Patienten mit Dickdarm-, Brust- und Lungenkrebs Analysen der cfDNA aus dem Blut die tumorspezifischen Veränderungen erfolgreich erkennen, was zur genauen Vorhersage eines Rezidivs mehrere Monate im Voraus13führte. Darüber hinaus sind die Behandlungsresistenzmarker, wie KRAS-Mutationen bei Patienten mit CRC, die eine Anti-EGFR-Therapieerhalten, 30; VAFs für Gene wie PIK3CA, MED1 oder EGFR bei Patientinnen mit Brustkrebs nach der Behandlung mit verschiedenen Therapien31; und EGFR T790M Resistenzmutation bei Lungenkrebspatienten, die mit EGFR-gezielten TKIs32 behandelt wurden, können auch durch cfDNA-Analyse identifiziert werden.
Zusammenfassend lässt sich sagen, dass die cfDNA-Analyse verwendet werden kann, um präzise Biomarker im Bereich der Onkologie13,33zu identifizieren. In diesem Protokoll wurden Blutproben von 3 Gliom-Patienten und 3 gesunde Kontrollen verarbeitet, um genomische DNA aus WBCs und cfDNA aus dem Plasma zu erhalten. Bei Gliomkrebs dienen Mutationen bei IDH, TERT, ATRX, EGFR und TP53 als diagnostische sowie prognostische Marker, die bei der Frühdiagnose von Gliomtumoren helfen können, verschiedene Arten von Gliomtumoren zu klassifizieren, die genaue Behandlung für den einzelnen Patienten zu leiten und das Behandlungsverhalten zu verstehen34,35. Der Mutationsstatus dieser Gene kann mit blutabgeleiteter cfDNA identifiziert werden. In diesem Manuskript stellen wir ein detailliertes Protokoll der plasmaabgeleiteten cfDNA vor, das zur Untersuchung von Mutationsveränderungen bei Gliomkrebs verwendet wurde12. Ein solches cfDNA-basiertes Flüssigbiopsieprotokoll, das in diesem Artikel erläutert wird, kann zur Untersuchung von Mutationsveränderungen bei vielen anderen Krebsarten verwendet werden. Darüber hinaus hat eine aktuelle Studie gezeigt, dass cfDNA-basierte Flüssigbiopsie 50 verschiedene Krebsarten erkennen kann36.
Die Entnahme, Lagerung und Verbringung von Blutproben sind entscheidende Schritte in diesem Protokoll, da die unkontrollierte Temperatur während dieser Schritte eine Lyse von WBCs verursacht, die zur Freisetzung genomischer DNA aus dem WBC in das Plasma führt und eine Kontamination der cfDNA-Probe verursacht, die den Rest des Verfahrens betrifft37. Hämolyse aufgrund unkontrollierter Temperatur kann nachgeschaltete Probenvorbereitungsprozesse von cfDNA beeinträchtigen, wie z. B. die PCR-Schritte38. Das Serum enthält einen hohen Anteil an Keimbahn cfDNA anstelle von Plasma, obwohl es ein großes Hintergrundrauschen für tumorassoziierte cfDNA39darstellt. Daher ist Plasma zur Isolierung tumorassoziierter cfDNA eine geeignete Probe39. Blut, das in einem mit Blutentnahmeröhrchen haltigen Antikoagulans entnommen wird, sollte sofort oder innerhalb von bis zu zwei Stunden zentrifugiert werden, um das Plasma zu trennen und eine cfDNA-Kontamination zu vermeiden. In diesem Protokoll werden spezielle kommerzielle cfDNA-Konservierungsblutentnahmeröhrchen verwendet (siehe Tabelle der Materialien), die eine Alternative zu gerinnungshemmenden Blutentnahmeröhrchen darstellen. Diese speziellen Blutentnahmeröhrchen bewahren cfDNA und cfRNA und verhindern eine Lyse von WBCs für bis zu 30 Tage bei Umgebungstemperatur und bis zu 8 Tage bei 37 °C. Dies erleichtert die Aufrechterhaltung der entsprechenden Temperatur während einer Blutprobensendung und bis plasma- und WBC-Trennung40.
Derzeit gibt es drei Arten von cfDNA-Extraktionsmethoden: Phasenisolierung, Siliziummembran-basierte Spin-Säule und magnetische Perlen-basierte Isolierung41. Das Siliziummembran-basierte Spinsäulenverfahren lieferte eine hohe Menge an cfDNA mit hoher Integrität im Vergleich zu anderen cfDNA-Extraktionsmethoden42.
Die quantitative Auswertung von DNA ist eine grundlegende Anforderung in der flüssigen Biopsie, es besteht die Notwendigkeit, ein einfaches, erschwingliches und standardisiertes Verfahren für ihre einfache Umsetzung und breite Nutzung zu entwickeln. Drei häufig verwendete Methoden zur cfDNA-Quantifizierung sind spektrophotometrische, fluorimetrische und qPCR. Die fluorimetrische Methode ist besser als die anderen Methoden in Bezug auf die Genauigkeit, Kosten und Leichtigkeit der Leitung43.
Die Integrität und Reinheit der cfDNA kann entweder durch Agaroseelektrophorese oder Kapillarelektrophorese geschätzt werden. Die Agarose-Elektrophorese zeigt weder eine Empfindlichkeit bei geringer CfDNA-Konzentration noch hat sie eine hohe Auflösung, um eine genaue Fragmentgröße von cfDNA zu zeigen. Andererseits hat die Kapillarelektrophorese einen Vorteil gegenüber der Agaroseelektrophorese, indem sie die damit verbundenen Herausforderungen überwindet und daher von den Forschern für die cfDNA-Fragmentgrößenanalyse weit verbreitet ist. In diesem Protokoll wurde die Fragmentgrößenverteilung isolierter cfDNA mit einem automatisierten Kapillarelektrophorese-Instrument geschätzt (siehe Tabelle der Materialien).
Protokoll
Vor der Blutentnahme ist die informierte Zustimmung der an der Forschung beteiligten Personen erforderlich und muss eingeholt werden. Die in diesem Manuskript beschriebene Forschung wurde in Übereinstimmung mit dem Rabin Medical Center, dem israelischen Ethikkomitee (Ethikkodex: 0039-17-RMC) und der Medizinischen Fakultät der Christian-Albrechts-Universität zu Kiel, Deutschland, durchgeführt (Ethikkodex: D 405/14).
1. Blutprobenentnahme und -speicherung in cfDNA- oder cfRNA-Konservierungsröhrchen
- Beschriften Sie die Konservierungsrohre richtig
- Sammeln Sie 8 ml Blut in die cf-DNA-Konservierungsröhre (siehe Tabelle der Materialien),mit einem Blutentnahmeset und einem Halter, wie nach dem unten beschriebenen Standard-institutionellen Protokoll für Venenpunktion.
HINWEIS: Die Verwendung eines Blutentnahmesets kann einen möglichen Rückfluss des Blutes aus der Röhre verhindern.- Richten Sie den Patienten mit dem Arm in einer abwärts gerichteten Position aus.
- Halten Sie die Röhre aufrecht, mit der Kappe nach oben, während Sie sicherstellen, dass der Rohrinhalt die Kappe oder Nadelspitze nicht berührt.
- Wenn Blut in die Röhre zu fließen beginnt, lassen Sie das Tourniquet langsam los.
- Unmittelbar nachdem das Rohr mit Blut gefüllt ist (maximale Kapazität: 8,4 ml Vollblut), das Rohr vorsichtig umkehren (das Handgelenk des Arms, der das Rohr um 180° nach unten und hinten hält) 5 Mal umdrehen, um die Probe zu stabilisieren.
HINWEIS: Inversion stellt sicher, dass das Konservierungsmittel gleichmäßig mit der Probe gemischt wird. Schütteln Sie den Inhalt jedoch nicht noch einmal, auch nicht vor der Plasmazubereitung. Unzureichende Vermischung von Konservierungsstoffen mit der Blutprobe führt zu einer Destabilisierung des Inhalts und der Bildung von Mikrogerinnseln oder Hämolyse. In diesem Stadium kann das Protokoll sofort für die Plasmatrennung fortgesetzt werden oder blutgefüllte Röhren können bis zu 30 Tage bei Umgebungstemperatur (15-25 °C) und bis zu 8 Tage bei 37 °C warten.
2. Plasma- und Buffy-Mantel-Trennung und -Lagerung
- Zentrifugieren Sie das blutgefüllte Konservierungsrohr bei 425 x g für 20 min bei Raumtemperatur, um Plasma zu trennen.
HINWEIS: Die Schritte 2.2 und 2.3 sollten in einem Biosicherheitsschrank durchgeführt werden. - Die obere Plasmaschicht vorsichtig in 1 ml Aliquots in ein frisches Rohr auslagern, ohne die unteren Schichten zu stören.
- Übertragen Sie vorsichtig die nächste Schicht buffy Mantel auf ein frisches Rohr (die Schicht erscheint als Ring über den RBC-Pellets), während RBCs in der unteren Schicht zu vermeiden.
- Fahren Sie mit Schritt 3 mit Plasma und Schritt 4 mit dem buffy Mantel fort. Bei Bedarf den getrennten Inhalt bei -80 °C lagern.
3. Reinigung der zirkulierenden cfDNA aus 1 ml Plasma
HINWEIS: Dieser Schritt wird mit einem kommerziellen Kit durchgeführt (siehe Tabelle der Materialien). Alle Puffer werden mit dem Kit geliefert.
- Herstellung von Puffern und Reagenzien
VORSICHT: Fügen Sie dem Probenvorbereitungsabfall keine sauren Lösungen oder Bleichmittel direkt hinzu. Guanidinsalze, die in Lysispuffer, Bindungspuffer und Waschpuffer-1 in Kombination mit Bleichmittel oder Säuren enthalten sind, können hochreaktive Verbindungen produzieren.- Bindungspuffer: Mischen Sie 300 ml Bindungspufferkonzentrat mit 200 ml 100% Isopropanol, um 500 ml funktionierenden Bindungspuffer zu bilden. Bei Raumtemperatur aufbewahren.
HINWEIS: Der Bindungspuffer ermöglicht die optimale Bindung der zirkulierenden Nukleinsäuren an die Kieselsäuremembran. 500 ml des Bindungspuffers reichen für die Verarbeitung von 276, 138, 92, 69 oder 55 Proben von 1, 2, 3, 4 oder 5 ml Plasma aus und sind für 1 Jahr bei Raumtemperatur stabil. - Waschpuffer-1: Mischen Sie 19 ml Wash Buffer-1 Konzentrat mit 25 ml 96–100% Ethanol, um 44 ml arbeitsfähigen Wash Buffer-1 herzustellen. Bei Raumtemperatur aufbewahren.
HINWEIS: Wash Buffer-1 eliminiert die an die Kieselsäuremembran gebundenen Verunreinigungen. 44 ml Arbeitswaschpuffer-1 ist ausreichend für die Verarbeitung von 73 Proben von 1/2/3/4/5 ml Plasma und ist stabil für 1 Jahr bei Raumtemperatur. - Wash Buffer-2: Mischen Sie gut 13 ml Wash Buffer-2 Konzentrat mit 30 ml 96–100% Ethanol, um 43 ml arbeitsfähigen Wash Buffer-2 zu machen. Bei Raumtemperatur aufbewahren.
HINWEIS: Wash Buffer-2 eliminiert die an die Kieselsäuremembran gebundenen Verunreinigungen. 43 ml Arbeitswaschpuffer-2 reichen für die Verarbeitung von 56 Proben von 1/2/3/4/5 ml Plasma aus und sind für 1 Jahr bei Raumtemperatur stabil. - In ein Rohr, das 310 g lyophilisierte Träger-RNA enthält, fügen Sie 1.550 l Elutionspuffer hinzu, um eine Träger-RNA-Lösung von 0,2 g/l vorzubereiten. Nach gründlicher Auflösung der Träger-RNA die Lösung auf geeignete Aliquots aufteilen und bei -30 °C bis -15 °C lagern. Diese Aliquots nicht mehr als 3 Mal einfrieren. Zum Lysispuffer fügen Sie, wie in Tabelle S1dargestellt, die rekonstituierte Träger-RNA, die im Elution-Puffer gelöst ist, hinzu.
HINWEIS: Da sich träger-RNA nicht direkt im Lysispuffer auflöst, muss sie zuerst in einem Elutionspuffer und dann in Lysis Buffer aufgelöst werden. Erstens wird die Bindung von Kieselsäure-Nukleinsäuren verstärkt, wenn nur sehr wenige Zielmoleküle in der Probe vorhanden sind. Zweitens wird das Risiko eines RNA-Abbaus durch das Vorhandensein großer Mengen von Träger-RNA reduziert.
- Bindungspuffer: Mischen Sie 300 ml Bindungspufferkonzentrat mit 200 ml 100% Isopropanol, um 500 ml funktionierenden Bindungspuffer zu bilden. Bei Raumtemperatur aufbewahren.
- Bevor Sie mit der Isolierung beginnen, bringen Sie die Säulen und Proben auf Raumtemperatur und passen Sie die Probenvolumina bei Bedarf auf 1 ml mit steriler Phosphat-gepufferter Saline (PBS) an. 2 Wasserbäder oder Heizblöcke vorheizen, die 50 ml Zentrifugenrohre und 2 ml Sammelrohre auf 60 °C bzw. 56 °C enthalten.
- Zu einem 50 ml Zentrifugenrohr 100 l Proteinase K, 1 ml Plasma und 0,8 ml Lysispuffer mit 1,0 g Träger-RNA (in Schritt 3.1.4) hinzufügen. Schließen Sie das Zentrifugenrohr mit einer Kappe und mischen Sie den Inhalt durch Pulswirbelung für 30 s, während Sie einen sichtbaren Wirbel in der Röhre gewährleisten. Eine gründliche Vermischung der Inhalte ist wichtig für eine effiziente Lyse.
HINWEIS: Fahren Sie unmittelbar nach dem Wirbeln unverzüglich mit Schritt 3.4 fort. - Inkubieren Sie die Lösung bei 60 °C für 30 min.
- Entfernen Sie die Kappe, fügen Sie 1,8 ml des Bindungspuffers in das Rohr ein, und mischen Sie sie nach dem Platzieren der Kappe gründlich mit Pulswirbelung für 15–30 s.
- Inkubieren Sie das resultierende Gemisch für 5 min auf Eis und legen Sie die Kieselsäure-Membransäule in das Vakuumgerät ein, das mit der Vakuumpumpe verbunden ist. Setzen Sie dann einen 20 ml Rohrverlängerer fest in die offene Säule ein, um ein Leck in der Probe zu verhindern.
- Gießen Sie das inkubierte Gemisch vorsichtig in den Rohrverlängerer der Säule und schalten Sie die Vakuumpumpe ein. Schließlich läuft das Lysatgemisch vollständig durch die Säulen, schaltet die Vakuumpumpe aus, gibt den Druck auf 0 mbar ab und entfernt und entsorgt den Rohrverlängerer.
HINWEIS: Um Kreuzkontaminationen zu vermeiden, sollte der Rohrverlängerer sorgfältig entsorgt werden, um zu verhindern, dass er sich über benachbarte Säulen ausbreitet. - Entfernen Sie die Säule aus dem Vakuumgerät, legen Sie sie in das Sammelrohr ein und zentrieren Sie bei 11.000 x g für 30 s bei Raumtemperatur, um Restlysat zu entfernen. Entsorgen Sie den Durchfluss.
- Fügen Sie 600 l Waschpuffer-1 in die Säule, Zentrifuge bei 11.000 x g für 1 min bei Raumtemperatur, entsorgen Sie den Durchfluss.
- Fügen Sie der Säule 750 l Waschpuffer-2, Zentrifuge bei 11.000 x g für 1 min bei Raumtemperatur, und entsorgen Sie den Durchfluss.
- Fügen Sie 750 l Ethanol hinzu (96–100%) zur Säule zentrifugieren Sie bei 11.000 x g für 1 min bei Raumtemperatur und entsorgen Sie den Durchfluss.
- Zentrifugieren Sie die Säule bei 20.000 x g für 3 min, indem Sie sie in ein sauberes 2 ml Sammelrohr legen.
- Trocknen Sie die Membransäulenbaugruppe vollständig, indem Sie sie in ein neues 2 ml Sammelrohr legen, wobei der Deckel geöffnet ist und bei 56 °C für 10 min inkubiert wird.
- Legen Sie die Säule in ein sauberes 1,5 ml Elutionsrohr. In der Mitte der Säulenmembran 20–150 l Elutionspuffer auftragen und bei Raumtemperatur 3 min mit geschlossenem Deckel inkubieren.
HINWEIS: Stellen Sie sicher, dass der Elution-Puffer der Raumtemperatur gleicht. Bei Verwendung des Elutionspuffers unter 50 l stellen Sie sicher, dass er sorgfältig auf die Mitte der Membran abgegeben wird. Dies hilft bei der vollständigen Elution der gebundenen DNA. Das Elutionsvolumen ist jedoch nicht fixiert und kann gemäß den nachgelagerten Anwendungen geändert werden. Das zurückgewonnene Eluat kann bis zu 5 l und sicherlich kleiner sein als das auf die Säule aufgebrachte Elutionsvolumen. - Zentrifugieren Sie das zurückgewonnene Eluat in einer Mikrozentrifuge bei 20.000 x g für 1 min, um die Nukleinsäuren zu elute, und lagern Sie bei -20 °C.
4. Reinigung der genomischen DNA aus buffy Mantel
HINWEIS: Das in diesem Protokoll verwendete kommerzielle Kit wird in der Tabelle der Materialienerwähnt. Puffer und Reagenzien, die im folgenden Protokoll erwähnt werden, z. B. Lysispuffer A, Lysispuffer B, Waschpuffer X, Waschpuffer Y, Proteinasebuffer, Elutionspuffer und Proteinase K sind Teil dieses kommerziellen Kits.
- Herstellung der Puffer und Reagenzien
VORSICHT: Fügen Sie dem Probenvorbereitungsabfall keine sauren Lösungen oder Bleichmittel direkt hinzu. Guanidinsalze, die in Lysispuffer B und Waschpuffer X in Kombination mit Bleichmittel oder Säuren enthalten sind, können hochreaktive Verbindungen produzieren.- Waschpuffer Y: 12 ml Wash Buffer Y Konzentrat mit 48 ml Ethanol (96–100%) gut mischen um 60 ml Arbeitspuffer Y. bei Raumtemperatur aufbewahren zu können.
HINWEIS: 60 ml Arbeitswaschpuffer Y ist ausreichend für die Verarbeitung von 100 buffy Fellproben und ist stabil für 1 Jahr. - Proteinase K: Bereiten Sie die Proteinase-K-Lösung vor, indem Sie 30 mg lyophilisierte Proteinase K in 1,35 ml Proteinase-Puffer auflösen.
HINWEIS: Die Gesamtarbeitslösung von Proteinase K ist ausreichend für die Verarbeitung von 52 buffy Coat Proben. Die Arbeitslösung Proteinase K kann mindestens 6 Monate bei -20 °C gelagert werden.
- Waschpuffer Y: 12 ml Wash Buffer Y Konzentrat mit 48 ml Ethanol (96–100%) gut mischen um 60 ml Arbeitspuffer Y. bei Raumtemperatur aufbewahren zu können.
- Schritte vor Einleitung des Verfahrens
- Den büffelnden Mantel auf Raumtemperatur ausdemieren.
- Stellen Sie den Wärmeblock oder das Wasserbad auf 56 °C ein.
- Suspendbuffy Mantel in Lysis Puffer A, um ein Endvolumen von 200 l zu erhalten. Dann fügen Sie 25 l Proteinase-K-Lösung und 200 l Lysispuffer B hinzu. Mischen Sie sie durch Wirbeln und inkubieren Sie bei 70 °C für 10–15 min. Stellen Sie sicher, dass die Proben vollständig mit der Lyselösung abgedeckt sind.
HINWEIS: Für die Verarbeitung von Probenreihen können Proteinase K und Lysispuffer A 10 bis 15 Minuten vor dem Eingriff vorgemischt werden, aber nicht mehr davor, da Proteinase K selbstverdaut in Lysispuffer A ohne Substrat. - Fügen Sie 210 l 96–100% Ethanol in das obige Gemisch und Wirbel kräftig.
ANMERKUNG: Die Zugabe von Ethanol kann einen Stringy-Ausscheider bilden; Dies hat jedoch keinen Einfluss auf die DNA-Isolierung. Achten Sie darauf, den Niederschlag auch auf die Spalte zu laden, wie in den folgenden Schritten gezeigt. - Laden Sie die gesamte Probe auf die Kieselsäuresäule, die in einem Sammelrohr platziert ist. Zentrifuge für 1 min bei 11.000 x g. Platzieren Sie die Spalte in einem neuen Sammelrohr, und entsorgen Sie das vorherige Rohr zusammen mit dem Durchfluss.
HINWEIS: Wiederholen Sie den Zentrifugationsschritt, wenn die Probe nicht vollständig durch die Matrix gezogen wird. - Fügen Sie 500 l Waschpuffer X, Zentrifuge für 1 min bei 11.000 x g hinzu, und entsorgen Sie den Durchfluss.
- Legen Sie die Spalte in das Sammelrohr, fügen Sie 600 l Waschpuffer Y auf die Säule, Zentrifuge für 1 min bei 11.000 x g, und entsorgen Sie den Durchfluss.
- Legen Sie die Säule erneut in das Sammelrohr und zentrieren Sie die Säule für 1 min bei 20.000 x g, um die Kieselsäuremembran zu trocknen.
- Inkubieren Sie die Säule bei Raumtemperatur für 1 min, in ein 1,5 ml Mikrozentrifugenrohr gelegt, und fügen Sie dann 100 l Elutionspuffer hinzu. Dann die DNA durch Zentrifugieren für 1 min bei 11.000 x g elute und bei -20 °C lagern.
5. Quantifizierung von cfDNA und genomischer DNA mittels Fluorometer
- Führen Sie vor dem Starten des Protokolls die folgenden Schritte aus.
- Verdünnen Sie 2 l eluierte genomische DNA (ab Schritt 4.9) in 1:10 Proportionen mit ultrareinem Nuklease-freiem Wasser. Aufgrund der zu erwartenden geringen Konzentrationen cfDNA-Proben aus Schritt 3.15 nicht verdünnen.
- Equilibrate the assay Standard #1 und assay Standard #2 auf Raumtemperatur.
- Bereiten Sie insgesamt 6 dünnwandige Klarrohre von 0,5 ml Größe vor.
ANMERKUNG: Das vorgelegte Protokoll dient der Quantifizierung von 2 cfDNA- und 2 genomischen DNA-Proben, daher 4 Tuben für 4 Proben, und dieser Test erfordert 2 Standards. - Beschriften Sie die Deckel des Rohres.
HINWEIS: Die Beschriftung an der Seite des Rohres könnte die Messung beeinträchtigen. Darüber hinaus werden die Assay-Standardröhren sorgfältig beschriftet, da die Kalibrierung des Fluorometers erfordert, dass die Standards in der richtigen Reihenfolge sind. - Verdünnen Sie das Assay-Reagenz in 1:200 mit dem Assay-Puffer, um die Arbeitslösung vorzubereiten. Für 4 Proben und 2 Standards verwenden Sie 6 l Assay-Reagenz plus 1.194 l Assay-Puffer, um 1.200 l (200 l in jedem Rohr) arbeitslösung herzustellen.
HINWEIS: Verwenden Sie keinen Glasbehälter, sondern ein sauberes Kunststoffrohr. Jedes Rohr muss ca. 200 l des Endvolumens enthalten (ein Assay-Standardrohr muss 190 l der Arbeitslösung enthalten, und das Probenrohr muss 180–199 l der Arbeitslösung enthalten). Es muss eine ausreichende Arbeitslösung für alle Assay-Standards und Proben vorbereitet werden. - Fügen Sie in den Arbeitsassay-Standardröhren 190 L Arbeitslösung und 10 L Assay-Standard hinzu und mischen Sie die Lösung durch Wirbeln für 2–3 s. Vermeiden Sie die Bildung von Blasen innerhalb der Lösung.
- In den Probenröhrchen fügen Sie 198 L Arbeitslösung und 2 L cfDNA oder genomische DNA hinzu. Mischen Sie die Lösung, indem Sie 2-3 s wirbeln, und halten Sie sie bei Raumtemperatur für 2 min inkubiert.
- Drücken Sie im 'Home'Bildschirm des Fluorometer-Instruments 'DNA' und wählen Sie 'dsDNA High Sensitivity Assay', um den 'Standards' Bildschirm anzuzeigen, und drücken Sie dann 'Ja' auf dem Fluorometer 'Standards' Bildschirm, um die Standards zu lesen.
- Legen Sie in die Probenkammer den Assay Standard #1 Rohr ein, schließen Sie den Deckel, und drücken Sie 'Lesen'. Entfernen Sie das Rohr, sobald der Messwert abgeschlossen ist (ca. 3 s), und wiederholen Sie den gleichen Schritt für Standard #2.
- Nach Abschluss des Kalibrierungsprozesses wird ein Probenbildschirm angezeigt, dann ein Probenröhrchen ein- und wiederholungsschritt5.8. Der "Probenbildschirm" zeigt dann einen Wert an, der der Konzentration der Probe nach Verdünnung im Probenröhrchen entspricht.
- Für jeden Beispielwiederholungsschritt '5.9', bis alle Proben gelesen werden.
- Verwenden Sie die folgende Gleichung, um die tatsächliche Konzentration der Probe zu berechnen.

HINWEIS: Die Assaywerte sind in ng/mL und entsprechen der Konzentration nach Verdünnung im Assayrohr. Gleichung, die in Schritt 5.11 erwähnt wird, QF-Wert ist der Wert, der vom Fluorometer-Instrument angegeben wird, und x ist die Anzahl der Mikroliter der Probe, die dem Assay-Röhrchen zugesetzt wurde. Die Einheiten für QF-Werte, die durch die Gleichung generiert werden, ähneln dem vom Fluorometer bereitgestellten Wert. Wenn sich der Wert des Fluorometers beispielsweise in ng/mL befindet, sind die Einheiten für die durch die Gleichung berechnete Konzentration ng/mL.
6. DNA-Fragmentgrößenverteilung von cfDNA durch Fragmentanalysator
- Der Schritt vor der Initiierung des Verfahrens: Equilibrate das DNA-Farbstoffkonzentrat und DNA-Gel-Matrix auf Raumtemperatur für 30 min.
- Zubereitung der Gel-Dye-Mischung
VORSICHT: Behandeln Sie Lösungen mit Vorsicht, da DMSO dafür bekannt ist, den Eintritt organischer Moleküle in Gewebe zu erleichtern.- Das DMSO gründlich auftauen, indem man die DNA-Farbstoffkonzentrat-Durchstechflasche für 10 s auftauen lässt. 15 L dieses Konzentrats in eine DNA-Gelmatrix-Durchstechflasche herausbringen und bei 4 °C im Dunkeln lagern.
- Auch hier wirbelt die gekappte Durchstechflasche für 10 s, bis das Mischen von Gel und Farbstoff visualisiert wird.
- Gießen Sie die Mischung auf einem Spin-Filter an die obere Buchse.
- Mikrozentrifugieren Sie den Spinfilter bei 2.240 x g ± 20% für 10 min bei Raumtemperatur.
- Beschriften Sie den vorbereiteten Gelfarbstoff in der Röhre und entsorgen Sie den Filter gemäß guten Laborpraktiken. Beschriften Sie die Röhre und notieren Sie das Herstellungsdatum.
HINWEIS: Entsorgen Sie das Filtrat gemäß guten Laborpraktiken. Der Gel-Dye-Mix kann für 5 HIGH Sensitivity (HS) DNA-Chips verwendet werden. Wenn sie mehr als 1 h unbenutzt sind, bei 4 °C lagern. Die Lagerung im Dunkeln ist bis zu 6 Wochen möglich.
- Um den Gel-Farbstoff-Mix zu laden, stellen Sie die Position der Grundplatte der Span-Grundierstation sicher und stellen Sie den Clip an der niedrigsten Position ein.
- Den Gel-Farbstoff-Mix auf Raumtemperatur für 30 min ausstatten und gleichzeitig die Lichteinstrahlung überwachen.
- Nehmen Sie einen neuen HS-DNA-Chip aus einem versiegelten Beutel und legen Sie ihn auf die Span-Grundierstation, entfernen Sie dann 9,0 l des Gel-Farbstoff-Mixes und geben Sie ihn an der Unterseite des Chips gut, markiert als "G".
HINWEIS: Zeichnen Sie die Gel-Farbstoff-Mischung, um Partikel zu vermeiden, die sich am Boden der Durchstechflasche ansammeln können. Während Sie den Gel-Farbstoff-Mix gut in den HS-DNA-Chip geben, legen Sie die Spitze der Pipette vollständig ein, um die Bildung großer Luftblasen zu verhindern. Darüber hinaus führt das Berühren der Pipette an den Rändern des Brunnens zu schlechten Ergebnissen. - Positionieren Sie den Kolben auf 1 ml und schließen Sie die Span-Grundierstation. Stellen Sie sicher, dass die Verriegelung klickt und stellen Sie den Timer auf 60 s ein, drücken Sie dann den Kolben nach unten, bis er vom Clip gehalten wird, und lassen Sie den Kolben genau nach 60 s mit dem Clip-Release-Mechanismus los.
- Wenn sich der Kolben mindestens auf die 0,3 ml-Marke zurückzieht, warten Sie 5 s und ziehen Sie dann langsam zurück in die 1 ml-Position, öffnen Sie dann die Span-Grundierstation und entfernen Sie erneut 9,0 l des Gel-Farbstoff-Mixes und geben Sie an der Unterseite des HS-DNA-Chips gut, markiert als "G", ab.
- Um den DNA-Marker zu laden, geben Sie 5 l des DNA-Markers in den Brunnen, der mit dem Leitersymbol markiert ist. Wiederholen Sie den Vorgang für alle 11 Probenbohrungen.
- Um die Leiter und die Proben zu laden, geben Sie 1 l der DNA-Leiter in den Brunnen, mit dem Leitersymbol markiert und fügen Sie dann 1 l Probe (verwendete Brunnen) oder 1 l Marker (ungenutzte Brunnen) in allen 11 Probenbrunnen hinzu.
- Wirbeln Sie den HS-DNA-Chip für 60 s bei 2.400 Rpm, indem Sie den Chip horizontal in den Adapter legen. Stellen Sie sicher, dass die Wölbung, die den HS-DNA-Chip fixiert, während des Wirbelns nicht beschädigt wird.
- Um den HS-DNA-Chip in das Fragmentanalysator-Instrument einzufügen, öffnen Sie den Deckel und stellen Sie sicher, dass die Elektrodenpatrone richtig eingesetzt ist und der Chip-Selektor auf "dsHigh Sensitivity DNA" im Fragmentanalysator-Instrument positioniert ist.
- Montieren Sie den HS-DNA-Chip vorsichtig in den Behälter, der nur in eine Richtung passt, und verlieren Sie dann den Deckel, indem Sie sicherstellen, dass die Elektrodenpatrone genau in die Brunnen des HS-DNA-Chips passt.
- Das Display auf dem Bildschirm der Fragmentanalysezeigt zeigt den eingelegten HS-DNA-Chip und den geschlossenen Deckel durch das Chip-Symbol oben links auf dem Bildschirm an.
- Um den HS-DNA-Chiplauf zu initiieren, wählen Sie den dsDNA High Sensitivity Assay aus dem Menü 'Assay' auf dem Instrumentenbildschirm aus, füllen Sie dann die Tabelle der Beispielnamen ordnungsgemäß aus, indem Sie Informationen wie Beispielnamen und Kommentare einspeisen und den Chip lauf starten, indem Sie auf die Schaltfläche"Start" oben rechts auf dem Bildschirm klicken.
- Elektrodenreinigung nach einem HS-DNA-Chiplauf: Entfernen Sie sofort den verwendeten HS-DNA-Chip, sobald der Test abgeschlossen ist, und entsorgen Sie ihn nach guten Laborpraktiken. Führen Sie das folgende Verfahren durch, um sicherzustellen, dass die Elektroden sauber sind, ohne Rückstände aus dem vorherigen Test.
- Füllen Sie langsam 350 l entionisiertes Analysewasser in einen der Elektrodenreinigerbrunnen und legen Sie den Elektrodenreiniger in das Fragmentanalysator-Instrument, indem Sie den Deckel öffnen und dann den Deckel schließen und etwa 10 s warten.
- Entfernen Sie den Elektrodenreiniger, indem Sie den Deckel öffnen und warten Sie weitere 10 s, bis das Wasser auf den Elektroden verdunstet, bevor Sie den Deckel schließen.
Ergebnisse
Plasma-Trennung
8,5-9 ml Blut, das in cfDNA- oder cfRNA-Konservierungsröhrchen gesammelt wird, ergibt ein Volumen von ca. 4 ml Plasma. Das Volumen des Plasmas, das von blutgebundenen Blutinues in EDTA-Röhren getrennt ist, kann je nach Temperatur variieren. Die Exposition von EDTA-Röhren, die Blut bei einer Temperatur von mehr als 37 °C enthalten, führt zu einer verringerten Plasmavolumenausbeute44.
Fluorometer-Assay-Ergebnisse
Die cfDNA-Konzentration in 1 ml Plasma jedes Gliompatienten #1 und #3 und gesunde Kontrollen #H1, #H2 und #H3 137 ng, 12,6 ng, 6,52 ng, 2,26 ng bzw. 2,48 ng betrug. Bei Gliompatienten war #2 die cfDNA-Konzentration jedoch zu niedrig, um in einem Fluorometer nachgewiesen zu werden. Die cfDNA-Konzentration im 1 ml-Plasma eines Krebspatienten reicht von 0,5 bis >1000 ng18mit einem Mittelwert von 20 ng. Bei gesunden Menschen reicht die cfDNA-Konzentration von einer nicht nachweisbaren Konzentration bis zu 16 ng mit einem Mittelwert von 8 ng45. Die genomische DNA, die aus 200 L Buffy Coat (PBMCs) isoliert ist, ergibt etwa 25-50 g DNA.
DNA-Fragmentanalyse-Assay-Ergebnisse
Einer früheren Studie zufolge ist cfDNA in 166-bp-Langenfragmenten angereichert, die 85 % der gesamten cfDNA im Plasma eines Patienten ausmachen. Im Gegensatz dazu machen 332-bp lange Fragmente 10 % und 498 bp lange Fragmente 5 % der cfDNA46aus. Zwei Hauptfaktoren, die die Ergebnisse des cfDNA-Fragmentanalysators beeinflussen, sind die genomische DNA-Kontamination durch PBMCs und die geringe Konzentration von cfDNA. Abbildung 1 zeigt den erwarteten cfDNA-Bioanalysegraphen des Gliompatienten #1, in dem cfDNA-Fragmente mit 166 bp angereichert werden und es keine genomische DNA-Kontamination in der Probe21gibt. Abbildung 2 zeigt das Fragmentanreicherungsdiagramm nach der Vorbereitung der NGS-Bibliothek der cfDNA von Gliompatienten #1 und Fragmentanreicherung wird aufgrund der Befestigung von 125 bp Indizes und Adaptern an sie von 166 bp auf 291 bp verschoben. Abbildung 3 zeigt eine sehr geringe Anreicherung der cfDNA-Fragmente von Gliom-Patienten #2. Trotz der geringen cfDNA-Konzentration bei glioma-Patienten#2 führen die Zugabe von NGS-Adaptern zu den cfDNA- und PCR-Verstärkungsschritten zur sichtbaren cfDNA-Bibliotheksspitze im Fragmentanreicherungsdiagramm Abbildung 4. Daher wurde die Bibliothek erfolgreich aus dieser cfDNA-Probe mit niedriger Konzentration erstellt, wie in Abbildung 4dargestellt. In Abbildung 5werden die cfDNA-Fragmente von Gliom-Patienten #3 in der Nähe von 166 bp nachgewiesen, aber auch eine genomische DNA-Kontamination ist in der Nähe des Referenzleiterspitzen von 10380 bp zu beobachten.

Abbildung 1: Ein idealer cfDNA-Fragmentanalysator-Assay-Graph der Gliomprobe #1 cfDNA. Ein wichtiger Peak wurde mit 166 bp und ein kleinerer Peak bei 332 bp beobachtet. In dieser Probe, die in der Nähe des 10.380 bp-Oberen Markers auftritt, wurde keine genomische Kontamination beobachtet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
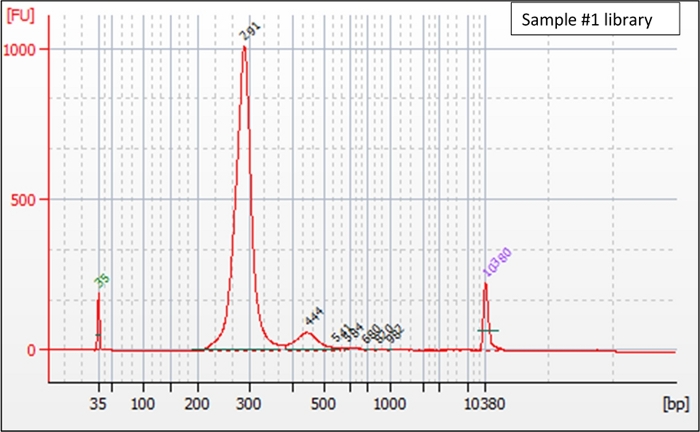
Abbildung 2: Ein ideales cfDNA-Fragmentanalyse-Assay-Diagramm der Gliomprobe #1 cfDNA-Bibliothek. Nach der Ligaierung von 125 bp Sequenzierungsbibliotheksadaptern der nächsten Generation zeigten 166 bp und 332 bp cfDNA-Fragmente einen großen Spitzenwert bei 291 bp und einen kleineren Peak bei 457 bp. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
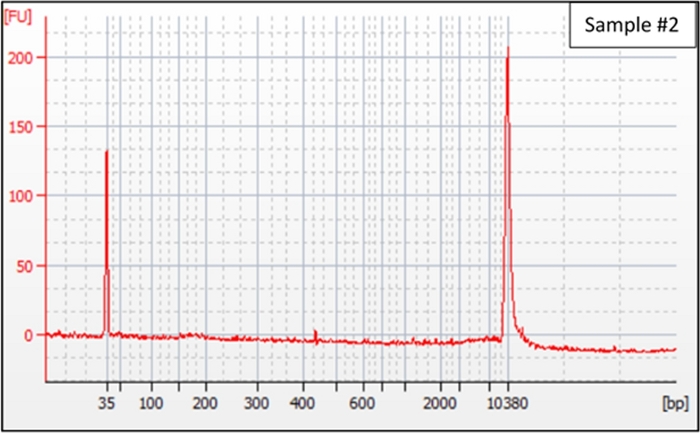
Abbildung 3: cfDNA-Fragmentanalysator-Assay-Diagramm der Gliomprobe #2 cfDNA, die einen sehr kleinen Spitzenwert von 166 bp zeigt. Aufgrund der geringen CfDNA-Konzentration war ein Spitzenwert von 166 bp kaum sichtbar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
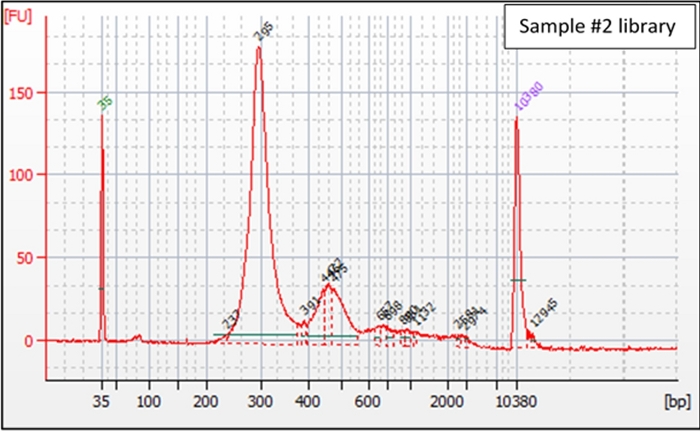
Abbildung 4: cfDNA-Fragmentanalyse-Assay-Diagramm der cfDNA-Bibliothek der Gliom-Probe #2. Nach der Ligaierung von 125 bp Sequenzierungsbibliotheksadaptern der nächsten Generation und der Durchführung von PCR-Zyklen während der Bibliotheksvorbereitung waren die kaum sichtbare cfDNA-Spitze bei 166 bp oder völlig unsichtbare Spitzen bei 332 bp und 498 bp deutlich sichtbar. Ein wichtiger Peak war in der Nähe von 291 bp vorhanden und ein kleinerer Peak bei 457 bp, 623 bp, 789 bp und nahe 2.500 bp wurden beobachtet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
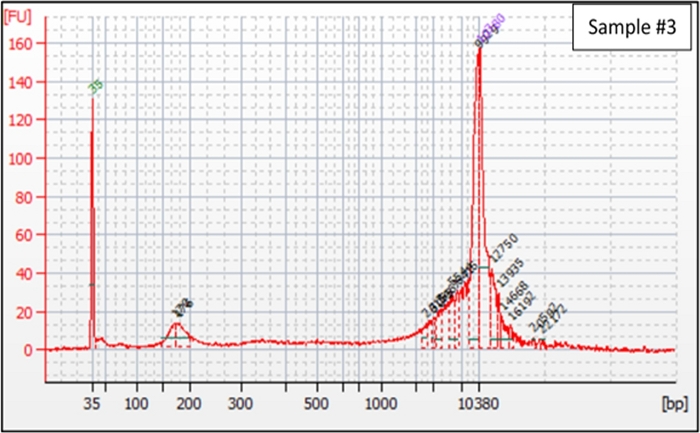
Abbildung 5: cfDNA-Fragmentanalysator-Assay, der eine genomische DNA-Kontamination in Gliomproben #3 cfDNA zeigt. Ein kleiner Peak mit 166 bp deutete auf das Vorhandensein von cfDNA und eine Anreicherung in der Nähe des oberen Markers (10.380 bp) als genomische DNA-Kontamination hin. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle S1: Volumen der Lysepuffer- und Träger-RNA (gelöst in Puffer AVE), die für die Verarbeitung von 1 ml Plasmaproben erforderlich sind. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Die Entnahme des Blutes eines Patienten in einer Röhre, der Versand und die Lagerung sind entscheidende erste Schritte in der flüssigen Biopsie. Unsachgemäße Handhabung kann die Qualität des Plasmas beeinträchtigen und daher die Ergebnisse der flüssigen Biopsiestören 47. Wenn eine Blutprobe in einem EDTA-Blutröhrchen entnommen wird, muss das Plasma innerhalb von zwei Stunden nach der Blutentnahme getrennt werden, um eine Lyse von WBCs und die Freisetzung seiner genomischen DNA in das Plasma zu vermeiden48. WBCs können auch Apoptose in einem EDTA-Rohr durchlaufen, wenn sie länger aufbewahrt werden, und die resultierenden cfDNA-Fragmente können original cfDNA im Plasmaverunreinigen 48. Die Exposition einer Blutprobe bei einer hohen Temperatur (>37 °C) oder übermäßiges Schütteln der Röhre vor der Plasmatrennung verursacht Hämolyse. Das Hämoglobin, das folglich als Kontaminant im Plasma verbleibt, wirkt sich auf die nachgeschalteten Prozesse von cfDNA aus, wie z.B. während der Bibliotheksvorbereitung49. Hohe Temperaturen (>37 °C) beeinflussen auch die Plasmaausbeute in einem EDTA-Blutröhrchen. Daher ist die schnelle Trennung des Plasmas nach seiner Entnahme in einem EDTA-Rohr50 ein wichtiger Schritt in der Qualitätskontrolle des Verfahrens. Wenn Blut in kommerziellen cfDNA oder cfRNA Konservierungstube gesammelt wird, sollte die Konservierungschemikalie sofort nach der Blutentnahme40mit dem Blut vermischt werden. Wenn die Konservierungschemikalie nicht richtig gemischt wird, bilden sich Mikrogerimsen im Plasma. Diese verstopfen die Kieselsäuremembranen in einer Kolonne während des cfDNA-Extraktionsprozesses und senken die Ausbeute von cfDNA. Mäßiges Schütteln von Blut in der Röhre verursacht keine Hämolyse; obwohl übermäßiges Schütteln auch in diesen Röhren zu Hämolyse führen kann40. Daher sollte beim Transport oder der Handhabung dieser Rohre übermäßiges Schütteln vermieden werden. Blutröhrchen werden empfohlen, in ihrer aufrechten Position versendet zu werden.
Die genaue Quantifizierung und Bewertung der cfDNA-Konzentration pro Einheit Plasma ist ein wichtiger Schritt in der flüssigen Biopsie, da die Plasma-cfDNA-Konzentration direkt mit physiologischen und pathologischen Prozessen assoziiert ist51. Die cfDNA-Konzentration ist bei Krebspatienten höher als bei gesunden Probanden52. Erhöhte cfDNA-Konzentration im menschlichen Blut ist nicht spezifisch für Krebs; Schwangere Frauen und Patienten, die Transplantationen erhalten, neigen ebenfalls zu erhöhten cfDNA-Konzentrationen im Blut21. Assoziationen wurden auch für Entzündungen, Gewebetraumata, Diabetes, Myokardinfarkt und Sepsis mit einer erhöhten cfDNA-Konzentration nachgewiesen53. Die Konzentration von Plasma-cfDNA bei Krebspatienten im Frühstadium ist tendenziell niedriger als bei Patienten mit fortgeschrittenen oder metastasierenden Tumoren. Dies unterstützt eine direkte Zuordnung von cfDNA mit einer Tumorbelastung54. Die cfDNA-Konzentration im Blut schwankt signifikant zwischen 0,5 und >1000 ng/ml bei Krebspatienten und zwischen 0 und 100 ng/ml bei gesunden Probanden18. Für die Bibliotheksvorbereitung sind mindestens 0,5 ng cfDNA erforderlich. Um die Mindestbedarfsmenge von cfDNA für die Sequenzierung zu erreichen, kann man mit einem Minimum von 2 ml einer Blutprobe oder einer 1 ml Plasmaprobe beginnen. Darüber hinaus verringern technische Fehler bei der Plasma-cfDNA-Extraktion auch die cfDNA-Ausbeute und die daraus resultierende Konzentration und weichen damit von der tatsächlichen Plasma-cfDNA-Konzentration ab. cfDNA-Abbau kann durch Antikoagulanzien in EDTA-Röhrchen oder durch den Prozess des mehrfachen Auftauens und Einfrierens von Plasmaproben auftreten. Eine schlechte Aktivität der Proteinase K während des Lyseprozesses führt auch zu einer geringen Ausbeute an cfDNA. Die Proteinase-K-Aktivität nimmt aufgrund der längeren Exposition gegenüber hohen Temperaturen ab. Die Verwendung von ethanolmitstatten der niedrigen Konzentration anstelle von 96-100% während des cfDNA-Extraktionswaschschritts führt ebenfalls zu einer geringen Ausbeute an cfDNA. Daher ist die Vermeidung technischer Fehler in der flüssigen Biopsietechnik wichtig, um genaue und zuverlässige Ergebnisse zu erzielen.
Die qualitative Analyse von cfDNA ist ein zentraler Schritt, in dem die tatsächliche Verteilung der in der extrahierten cfDNA-Probe vorhandenen DNA-Fragmente geschätzt wird. Quantitative Ergebnisse von cfDNA sind nur zuverlässig, nachdem sie ihre Reinheit durch qualitative Analyse bestätigt haben. Daher zeigt ein idealer Graph von cfDNA, der im DNA-Fragmentanalysetest erzeugt wird, die Anreicherung des Hauptgipfels von cfDNA-Fragmenten nahe 166 bp und eines kleineren Peaks in der Nähe von 332 bp Länge. Dies deutet darauf hin, dass die extrahierte cfDNA-Probe nicht mit genomischer DNA aus WBCs kontaminiert ist. Wenn ein Fragment von 7.000 bp zur Referenzleiter (10.380bp) und darüber hinaus angereichert ist, deutet dies auf eine Kontamination einer cfDNA-Probe mit der genomischen DNA von WBC hin. Wenn eine genomische DNA-Kontamination in einer cfDNA-Probe nach dna-Fragmentgrößenschätzungstest beobachtet wird, kann die Probe auf 2% Agarose-Gel mit einer 100 bp Leiter ausgeführt werden, und Gel kann zwischen dem Bereich von 150-200 bp ausgeschnitten werden, gefolgt von einem Gelextraktionstest. Während des Agarose-Gel-Größenauswahlprozesses geht eine gewisse Menge an cfDNA verloren. Der Schritt zur Vorbereitung der NGS-Bibliothek erfordert jedoch mindestens 0,5 ng und maximal 1.000 ng Eingabe-DNA-Material, um Bibliotheken für die Sequenzierung vorzubereiten. Wenn also die Gesamtmenge von cfDNA gleich oder mehr als 0,5 ng ist, reicht die Menge für den Bibliotheksvorbereitungsschritt aus. Darüber hinaus durchlaufen solche Samples den Bibliotheksvorbereitungsprozess erfolgreich, und die resultierende Bibliothek zeigt einen großen Peak nahe 291 bp und einen kleineren Peak bei 457 bp, aufgrund der Zugabe von 125 bp Indizes und Adaptern.
Die NGS-Datenanalyse ist der letzte Schritt in der NGS-basierten Flüssigbiopsietechnik, und in diesem Schritt werden Listen von Mutationen und Gen-Gen-Fusionen3,55,56,57 erstellt. Mutationen und Gen-Gen-Fusionen, die identifiziert werden, können nach früheren Studien über Krebsmutationslandschaften58,59, zuvor charakterisierte Genmutationen60 oder Gen-Gen-Fusionen61,62,63bewertet werden. Diese können als prognostische64 und diagnostische7 Biomarker bei Krebspatienten dienen. Beispielsweise ist die R132H-Mutation im Isocitrate-Dehydrogenase 1 (IDH1) Gen ein entscheidender Wegweiser für sekundären Glioblastom-Krebs und unterscheidet sie auch vom primären Glioblastom, bei dem diese Mutation normalerweise nicht vorhanden ist65. Darüber hinaus ist chronische myelogene Leukämie (CML) durch eine BCR/ABL1-Genfusion gekennzeichnet, die aus einer ausgewogenen Translokation zwischen Chromosom 9 und 2261resultiert. Das BCR/ABL1-Gen und seine mRNA und das Fusionsprotein sind einzigartig für CML-Vorläufer und stellen daher ein gutes Ziel für die Therapie66dar.
Die fortgeschrittene Diagnose, die korrekte Inszenierung des Tumors und die Behandlungsüberwachung ist eine aktuelle Strategie für ein effizientes Umgang mit Krebs. Bis vor kurzem war die Gewebebiopsie der Goldstandard für die histologische Bewertung von Tumorgeweben. Doch nach umfangreichen Forschungen auf diesem Gebiet und dem Aufkommen der Technologie hat sie bewiesen, dass eine einzige Biopsie nicht in der Lage ist, die gesamte Mutationslandschaft eines Tumors zu liefern, und dass serielle Biopsien aufgrund ihrer Invasivität, insbesondere bei Glioblastom und Lymphom, wo Biopsien hochinvasiv sind, in der Regel nicht praktikabel durchzuführen sind. Darüber hinaus sind ca. 16% der Nadelbiopsien mit Verfahrenskomplikationen verbunden67. Darüber hinaus erfordert ein hochwertiges genomisches Profiling eine ausreichende Menge an Gewebe, eine Nachfrage, die in der Regel durch die Nadelbiopsie68nicht erfüllt wird.
Die flüssigen Biopsien, aus einer routinemäßigen Blutprobe durch die Analyse von Tumor-abgeleitete DNA wird gefunden, um die routinemäßigen Nadelgewebe Biopsie Verfahren ersetzen oder ergänzen12. Die Analyse von tumorabgeleiteter DNA/cfDNA durch Plasma durch flüssige Biopsie ist ein minimalinvasives und wirksames Verfahren zur molekularen Profilierung von Tumoren. Es kann im Wesentlichen die Überwachung der Behandlung durch serielle Probenahme im Laufe der Zeit ohne die potenziellen Risiken und Komplikationen in der Regel mit traditionellen Tumorgewebe Biopsie aufgrund seiner invasiven Natur69verbunden ermöglichen. Darüber hinaus ergaben Studien, die die Nadelbiopsie mit der cfDNA-Analyse durch flüssige Biopsie einer einzelnen Tumorläsion verglichen, dass cfDNA-Profiling eine vollständige molekulare Heterogenität bieten kann, dass ein Tumor durch mehrere unterschiedliche klonale Populationen behütet werden kann70.
Obwohl die Plasma-cfDNA-basierte Diagnose viele Vorteile gegenüber der Tumorgewebebiopsie hat, gibt es immer noch einige entscheidende Einschränkungen, die ihre Anwendbarkeit in der allgemeinen klinischen Praxis behindern. Einschränkungen der cfDNA-basierten Flüssigbiopsiediagnose umfassen die höheren Kosten des Gesamtverfahrens, den Bedarf an hochwertiger DNA und einen speziellen Bioinformatiker für die Notwendigkeit einer umfangreichen Datenanalyse71. Aufgrund von Problemen im Zusammenhang mit der Datenverwaltung im Zusammenhang mit der cfDNA-Analyse wirft NGS auch Probleme bei der sofortigen Anwendung in der Klinik auf. Die Herausforderungen des Datenmanagements, die NGS darstellt, sind in erster Linie auf die Schwierigkeit zurückzuführen, zwischen dem intrinsischen Hintergrundrauschen der tiefen Sequenzierung und den Aberrationen, die mit dem Tumor verbunden sind, zu unterscheiden72. Schließlich sind sie aufgrund der Nichtverfügbarkeit der klinischen Gültigkeits- und Nutzendaten für die meisten flüssigen Biopsie-Assays derzeit nur auf die klinischen Studien und Grundlagenforschungenbeschränkt 13.
Die zukünftigen Entwicklungen in der flüssigen Biopsietechnik werden durch gemeinsame Anstrengungen durch Grundlagenforschung auf dem Gebiet der Biologie, Physiologie, Molekularbiologie, Assay-Techniken, statistische Analyse für das Datenmanagement und Maschine-Lern-Technologieerwartet 73,74. Darüber hinaus sind zahlreiche klinische Studien auf der Grundlage der cfDNA-Biomarker-Evaluierung im Gange, und ihre Ergebnisse werden dazu beitragen, die Gültigkeit der Technik zu ermitteln. Diese gemeinsamen Anstrengungen werden hoffentlich die Flüssigbiopsie-Plattform durch cfDNA-Analyse als leistungsstarkes prognostisches, diagnostisches und Behandlungsüberwachungsinstrument im onkologischen klinischen Umfeld innerhalb der nächsten fünf Jahreetablieren 70,75.
Abschließend möchte ich sagen, dass die cfDNA-basierte Flüssigbiopsie einen sicheren und nicht-invasiven Ansatz zur Identifizierung genomischer Biomarker bietet, die für das Management von Krebserkrankungen nützlich sind. Um jedoch zuverlässige und genaue Ergebnisse zu erzielen, sind standardisierte Protokolle für die Blutentnahme, Plasma-/WBC-Trennung, cfDNA-Extraktion und cfDNA-quantitative und qualitative Analyse besonders erforderlich74,75.
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen haben.
Danksagungen
Die Autoren danken den Mitgliedern des Laboratory of Cancer Genomics and Biocomputing of Complex Diseases für ihre scharfen Beobachtungsbeiträge und ihre Teilnahme an mehreren Diskussionen in verschiedenen Phasen dieses Projekts. Die Unterstützung umfasst die Israel Cancer Association (ICA-Zuschuss für M.F-M 2017-2019) und kamin Grant der Israel Innovation Authority (für M.F-M.).
Materialien
| Name | Company | Catalog Number | Comments |
| 2100 Bioanalyzer Instrument | Agilent Technologies, Inc. | G2939BA | The 2100 Bioanalyzer system is an established automated electrophoresis tool for the sample quality control of biomolecules. |
| Adjustable Clip for Priming Station | Agilent Technologies, Inc. | 5042-1398 | Used in combination with syringe to apply defined pressure for chip priming. |
| Agilent High Sensitivity DNA Kit | Agilent Technologies, Inc. | 5067-4626 | The High Sensitivity DNA assays are often used for sample quality control for next-generation sequencing libraries |
| cf-DNA/cf-RNA Preservative Tubes | Norgen Biotek Corp. | 63950 | Norgen's cf-DNA/cf-RNA Preservative Tubes are closed, evacuated plastic tubes for the collection and the preservation of cf-DNA, circulating tumor DNA, cf-RNA and circulating tumor cells in human whole blood samples during storage and shipping |
| Chip Priming Station | Agilent Technologies, Inc. | 5065-4401 | Used to load gel matrix into a chip with a syringe provided with each assay kit— used for RNA, DNA, and protein assays. Includes priming station, stop watch, and 1 syringe clip |
| Electrode Cleaner Kit | Agilent Technologies, Inc. | 5065-9951 | Prevents cross-contamination. Removes bacterial or protein contaminants from electrodes. |
| Filters for Gel Matrix | Agilent Technologies, Inc. | 185-5990 | Used for proper mixing of DNA dye concentrate and DNA gel matrix |
| IKA Basic Chip Vortex | IKA-Werke GmbH & Co. KG | MS-3-S36 | Used for proper mixing of DNA ladder and DNA sample on Bioanalyzer assay chips |
| NucleoSpin Tissue kit | MACHEREY-NAGEL | 740952.5 | With the NucleoSpin Tissue kit, genomic DNA can be prepared from tissue, cells (e.g., bacteria), and many other sources. |
| QIAamp circulating nucleic acid kit | Qiagen | 55114 | The QIAamp Circulating Nucleic Acid Kit enables efficient purification of these circulating nucleic acids from human plasma or serum and other cell-free body fluids. |
| QIAvac 24 Plus vacuum manifold | Qiagen | 19413 | The QIAvac 24 Plus vacuum manifold is designed for vacuum processing of QIAGEN columns in parallel. |
| QIAvac Connecting System | Qiagen | 19419 | In combination with the QIAvac Connecting System, the QIAvac 24 Plus vacuum manifold can be used as a flow-through system. The sample flow-through, containing possibly infectious material, is collected in a separate waste bottle. |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | The Qubit 2.0 Fluorometer is an easy-to-use, analytical instrument designed to work with the Qubit assays for DNA, RNA, and protein quantitation. |
| Qubit assay tubes | Thermo Fisher Scientific | Q32856 | Qubit assay tubes are 500 µL thin-walled polypropylene tubes for use with the Qubit Fluorometer. |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific | Q32851 | The Qubit dsDNA HS (High Sensitivity) Assay Kit is designed specifically for use with the Qubit Fluorometer. The assay is highly selective for double-stranded DNA (dsDNA) over RNA and is designed to be accurate for initial sample concentrations from 10 pg/µL to 100 ng/µL. |
| Vacuum Pump | Qiagen | 84010 | used for vacuum processing of QIAGEN columns |
| Miscellaneous | |||
| 50 ml centrifuge tubes | |||
| Crushed ice | |||
| Ethanol (96–100%) | |||
| Heating block or similar at 56 °C (capable of holding 2 ml collection tubes) | |||
| Isopropanol (100%) | |||
| Microcentrifuge | |||
| Phosphate-buffered saline (PBS) | |||
| Pipettes (adjustable) | |||
| Sterile pipette tips (pipette tips with aerosol barriers are recommended to help prevent cross-contamination) | |||
| Water bath or heating block capable of holding 50 mL centrifuge tubes at 60 °C |
Referenzen
- Campbell, P. J., et al. Pan-cancer analysis of whole genomes. Nature. 578 (7793), 82-93 (2020).
- Liotta, L., Petricoin, E. Molecular profiling of human cancer. Nature Reviews Genetics. 1 (1), 48-56 (2000).
- Balamurali, D., et al. ChiTaRS 5.0: the comprehensive database of chimeric transcripts matched with druggable fusions and 3D chromatin maps. Nucleic Acids Research. 48 (1), 825-834 (2019).
- Trédan, O., et al. Molecular screening program to select molecular-based recommended therapies for metastatic cancer patients: Analysis from the ProfiLER trial. Annals of Oncology. 30 (5), 757-765 (2019).
- Oliveira, K. C. S., et al. Current perspectives on circulating tumor DNA, precision medicine, and personalized clinical management of cancer. Molecular Cancer Research. 18 (4), 517-528 (2020).
- Siegal, T. Clinical impact of molecular biomarkers in gliomas. Journal of Clinical Neuroscience. 22 (3), 437-444 (2015).
- Komori, T. The 2016 WHO Classification of Tumours of the Central Nervous System: The Major Points of Revision. Neurologia medico-chirurgica. 57 (7), 301-311 (2017).
- Duffy, M. J., O'Donovan, N., Crown, J. Use of molecular markers for predicting therapy response in cancer patients. Cancer Treatment Reviews. 37 (2), 151-159 (2011).
- Saenz-Antoñanzas, A., et al. Liquid Biopsy in Glioblastoma: Opportunities, Applications and Challenges. Cancers. 11 (7), 950 (2019).
- Marrugo-Ramírez, J., Mir, M., Samitier, J. Blood-Based Cancer Biomarkers in Liquid Biopsy: A Promising Non-Invasive Alternative to Tissue Biopsy. International Journal of Molecular Sciences. 19 (10), 2877 (2018).
- Pantel, K., Alix-Panabières, C. Liquid biopsy in 2016: Circulating tumour cells and cell-free DNA in gastrointestinal cancer. Nature Reviews Gastroenterology & Hepatology. 14 (2), 73-74 (2017).
- Bronkhorst, A. J., et al. The emerging role of cell-free DNA as a molecular marker for cancer management. Biomolecular Detection and Quantification. 17, 100087 (2019).
- Cescon, D. W., Bratman, S. V., Chan, S. M., Siu, L. L. Circulating tumor DNA and liquid biopsy in oncology. Nature Cancer. 1 (3), 276-290 (2020).
- Palmirotta, R., et al. Liquid biopsy of cancer: a multimodal diagnostic tool in clinical oncology. Therapeutic Advances in Medical Oncology. 10, 175883591879463 (2018).
- Cohen, J. D. J., et al. Detection and localization of surgically resectable cancers with a multi-analyte blood test. Science. 359 (6378), 926-930 (2018).
- Wan, J. C. M., et al. Liquid biopsies come of age: towards implementation of circulating tumour DNA. Nature Reviews Cancer. 17 (4), 223-238 (2017).
- Heitzer, E., Haque, I. S., Roberts, C. E. S., Speicher, M. R. Current and future perspectives of liquid biopsies in genomics-driven oncology. Nature Reviews Genetics. 20 (2), 71-88 (2018).
- Thierry, A. R., et al. Origins, structures, and functions of circulating DNA in oncology. Cancer and Metastasis Reviews. 35 (3), 347-376 (2016).
- Buscail, E., et al. High Clinical Value of Liquid Biopsy to Detect Circulating Tumor Cells and Tumor Exosomes in Pancreatic Ductal Adenocarcinoma Patients Eligible for Up-Front Surgery. Cancers. 11 (11), 1656 (2019).
- Heitzer, E., Ulz, P., Geigl, J. B. Circulating Tumor DNA as a Liquid Biopsy for Cancer. Clinical Chemistry. 61 (1), 112-123 (2015).
- Kustanovich, A., Schwartz, R., Peretz, T., Grinshpun, A. Life and death of circulating cell-free DNA. Cancer Biology and Therapy. 20 (8), 1057-1067 (2019).
- Crowley, E., Di Nicolantonio, F., Loupakis, F., Bardelli, A. Liquid biopsy: monitoring cancer-genetics in the blood. Nature Reviews Clinical Oncology. 10 (8), 472-484 (2013).
- Celec, P., et al. Cell-free DNA: the role in pathophysiology and as a biomarker in kidney diseases. Expert Reviews in Molecular Medicine. 20, (2018).
- Gautschi, O., et al. Origin and prognostic value of circulating KRAS mutations in lung cancer patients. Cancer Letters. 254 (2), 265-273 (2007).
- Bidard, F., et al. Detection rate and prognostic value of circulating tumor cells and circulating tumor DNA in metastatic uveal melanoma. International Journal of Cancer. 134 (5), 1207-1213 (2013).
- Chan, K. C. A., et al. Analysis of Plasma Epstein–Barr Virus DNA to Screen for Nasopharyngeal Cancer. New England Journal of Medicine. 377 (6), 513-522 (2017).
- Mao, L., et al. Detection of Oncogene Mutations in Sputum Precedes Diagnosis of Lung Cancer. Cancer Research. 54 (7), 1634-1637 (1994).
- De Mattos-Arruda, L., et al. Cerebrospinal fluid-derived circulating tumour DNA better represents the genomic alterations of brain tumours than plasma. Nature communications. 6 (1), 8839 (2015).
- Khier, S., Lohan, L. Kinetics of circulating cell-free DNA for biomedical applications: critical appraisal of the literature. Future science OA. 4 (4), 295 (2018).
- Misale, S., et al. Resistance to Anti-EGFR therapy in colorectal cancer: From heterogeneity to convergent evolution. Cancer Discovery. 4 (11), 1269-1280 (2014).
- Beddowes, E., Sammut, S. J., Gao, M., Caldas, C. Predicting treatment resistance and relapse through circulating DNA. Breast. 34, 31-35 (2017).
- Sacher, A. G., et al. Prospective Validation of Rapid Plasma Genotyping for the Detection of EGFR and KRAS Mutations in Advanced Lung Cancer. JAMA oncology. 2 (8), 1014-1022 (2016).
- Vaidyanathan, R., et al. Cancer diagnosis: from tumor to liquid biopsy and beyond. Lab on a Chip. 19 (1), 11-34 (2019).
- Kelly, P. Gliomas: Survival, origin and early detection. Surgical Neurology International. 1 (1), 96 (2010).
- Faria, G., Silva, E., Da Fonseca, C., Quirico-Santos, T. Circulating Cell-Free DNA as a Prognostic and Molecular Marker for Patients with Brain Tumors under Perillyl Alcohol-Based Therapy. International Journal of Molecular Sciences. 19 (6), 1610 (2018).
- Liu, M. C., et al. Sensitive and specific multi-cancer detection and localization using methylation signatures in cell-free DNA. Annals of Oncology. 31 (6), 745-759 (2020).
- Enko, D., Halwachs-Baumann, G., Kriegshäuser, G. Plasma free DNA: Evaluation of temperature-associated storage effects observed for roche cell-free DNA collection tubes. Biochemia Medica. 29 (1), 153-156 (2019).
- Streleckiene, G., et al. Effects of Quantification Methods Isolation Kits, Plasma Biobanking, and Hemolysis on Cell-Free DNA Analysis in Plasma. Biopreservation and Biobanking. 17 (6), 553-561 (2019).
- Thress, K. S., et al. Acquired EGFR C797S mutation mediates resistance to AZD9291 in non-small cell lung cancer harboring EGFR T790M. Nature Medicine. 21 (6), 560-562 (2015).
- Ward Gahlawat, A., et al. Evaluation of Storage Tubes for Combined Analysis of Circulating Nucleic Acids in Liquid Biopsies. International Journal of Molecular Sciences. 20 (3), 704 (2019).
- Lu, J. L., Liang, Z. Y. Circulating free DNA in the era of precision oncology: Pre- and post-analytical concerns. Chronic Diseases and Translational Medicine. 2 (4), 223-230 (2016).
- Iyapparaj, P., et al. Optimization of bacteriocin production by Lactobacillus sp. MSU3IR against shrimp bacterial pathogens. Aquatic Biosystems. 9 (1), 12 (2013).
- Ponti, G., et al. The value of fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of cell-free DNA (cfDNA) in malignant melanoma and prostate cancer patients. Clinica Chimica Acta. 479, 14-19 (2018).
- Medina Diaz, I., et al. Performance of Streck cfDNA blood collection tubes for liquid biopsy testing. PLoS One. 11 (11), 0166354 (2016).
- Perkins, G., et al. Multi-Purpose Utility of Circulating Plasma DNA Testing in Patients with Advanced Cancers. PLoS One. 7 (11), 47020 (2012).
- Mouliere, F., et al. Multi-marker analysis of circulating cell-free DNA toward personalized medicine for colorectal cancer. Molecular Oncology. 8 (5), 927-941 (2014).
- Trigg, R. M., Martinson, L. J., Parpart-Li, S., Shaw, J. A. Factors that influence quality and yield of circulating-free DNA: A systematic review of the methodology literature. Heliyon. 4 (7), 00699 (2018).
- Risberg, B., et al. Effects of Collection and Processing Procedures on Plasma Circulating Cell-Free DNA from Cancer Patients. Journal of Molecular Diagnostics. 20 (6), 883-892 (2018).
- Markus, H., et al. Evaluation of pre-analytical factors affecting plasma DNA analysis. Scientific Reports. 8 (1), 7375 (2018).
- Chen, Z., et al. Comprehensive Evaluation of the Factors Affecting Plasma Circulating Cell-Free DNA Levels and Their Application in Diagnosing Nonsmall Cell Lung Cancer. Genetic Testing and Molecular Biomarkers. 23 (4), 270-276 (2019).
- Schwarzenbach, H., Hoon, D. S. B., Pantel, K. Cell-free nucleic acids as biomarkers in cancer patients. Nature Reviews Cancer. 11 (6), 426-437 (2011).
- Malyuchenko, N. V., et al. PARP1 Inhibitors: antitumor drug design. Acta Naturae. 7 (3), 27-37 (2015).
- Fleischhacker, M., Schmidt, B. Circulating nucleic acids (CNAs) and cancer-A survey. Biochimica et Biophysica Acta (BBA) - Reviews on Cancer. 1775 (1), 181-232 (2007).
- Jung, K., Fleischhacker, M., Rabien, A. Cell-free DNA in the blood as a solid tumor biomarker—A critical appraisal of the literature. Clinica Chimica Acta. 411 (21-22), 1611-1624 (2010).
- Frenkel-Morgenstern, M., et al. ChiTaRS: a database of human, mouse and fruit fly chimeric transcripts and RNA-sequencing data. Nucleic Acids Research. 41, 142-151 (2013).
- Frenkel-Morgenstern, M., et al. ChiTaRS 2.1--an improved database of the chimeric transcripts and RNA-seq data with novel sense-antisense chimeric RNA transcripts. Nucleic Acids Research. 43, 68-75 (2015).
- Gorohovski, A., et al. ChiTaRS-3.1-the enhanced chimeric transcripts and RNA-seq database matched with protein-protein interactions. Nucleic Acids Research. 45 (1), 790-795 (2017).
- Tate, J. G., et al. COSMIC: the Catalogue Of Somatic Mutations In Cancer. Nucleic Acids Research. 47 (1), 941-947 (2019).
- Brennan, C. W., et al. The somatic genomic landscape of glioblastoma. Cell. 155 (2), 462-477 (2013).
- Szopa, W., Burley, T. A., Kramer-Marek, G., Kaspera, W. Diagnostic and Therapeutic Biomarkers in Glioblastoma: Current Status and Future Perspectives. BioMed Research International. 2017, 1-13 (2017).
- Salesse, S., Verfaillie, C. M. BCR/ABL: from molecular mechanisms of leukemia induction to treatment of chronic myelogenous leukemia. Oncogene. 21 (56), 8547-8559 (2002).
- Frenkel-Morgenstern, M., Valencia, A. Novel domain combinations in proteins encoded by chimeric transcripts. Bioinformatics. 28 (12), 67-74 (2012).
- Frenkel-Morgenstern, M., et al. Chimeras taking shape: Potential functions of proteins encoded by chimeric RNA transcripts. Genome Research. 22 (7), 1231-1242 (2012).
- Simon, M., et al. TERT promoter mutations: a novel independent prognostic factor in primary glioblastomas. Neuro-Oncology. 17 (1), 45-52 (2015).
- Waitkus, M. S., Diplas, B. H., Yan, H. Isocitrate dehydrogenase mutations in gliomas. Neuro-Oncology. 18 (1), 16-26 (2016).
- Kindler, T., Meyer, R. G., Fischer, T. BCR-ABL as a target for novel therapeutic interventions. Expert Opinion on Therapeutic Targets. 6 (1), 85-101 (2002).
- Overman, M. J., et al. Use of research biopsies in clinical trials: are risks and benefits adequately discussed. Journal of Clinical Oncology Official Journal of the American Society of Clinical Oncology. 31 (1), 17-22 (2013).
- Vanderlaan, P. A., et al. Success and failure rates of tumor genotyping techniques in routine pathological samples with non-small-cell lung cancer. Lung Cancer. 84 (1), 39-44 (2014).
- Stewart, C. M., Tsui, D. W. Y. Circulating cell-free DNA for non-invasive cancer management. Cancer Genetics. 228-229, 169-179 (2018).
- Normanno, N., et al. The liquid biopsy in the management of colorectal cancer patients: Current applications and future scenarios. Cancer Treatment Reviews. 70, 1-8 (2018).
- Hufnagl, C., et al. Evaluation of circulating cell-free DNA as a molecular monitoring tool in patients with metastatic cancer. Oncology Letters. 19 (2), 1551-1558 (2020).
- Petit, J., et al. Cell-Free DNA as a Diagnostic Blood-Based Biomarker for Colorectal Cancer: A Systematic Review. The Journal of Surgical Research. 236, 184-197 (2019).
- Poulet, G., Massias, J., Taly, V. Liquid Biopsy: General Concepts. Acta Cytologica. 63 (6), 449-455 (2019).
- Alix-Panabières, C. The future of liquid biopsy. Nature. 579 (7800), 9 (2020).
- Eisenstein, M. Could liquid biopsies help deliver better treatment. Nature. 579 (7800), 6-8 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten